Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Evaluierung der Substratubiquitylierung mittels E3-Ubiquitin-Ligase in Säugetierzelllysaten
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir bieten ein detailliertes Protokoll für einen Ubiquitylierungsassay eines spezifischen Substrats und einer E3-Ubiquitin-Ligase in Säugetierzellen. HEK293T-Zelllinien wurden zur Proteinüberexpression verwendet, das polyubiquitylierte Substrat wurde durch Immunpräzipitation von Zelllysaten gereinigt und in SDS-PAGE aufgelöst. Immunoblotting wurde verwendet, um diese posttranslationale Modifikation zu visualisieren.
Zusammenfassung
Ubiquitylierung ist eine posttranslationale Modifikation, die in eukaryotischen Zellen auftritt und für die Regulation mehrerer biologischer Signalwege, einschließlich Zellüberleben, -proliferation und -differenzierung, entscheidend ist. Es ist ein reversibler Prozess, der aus einer kovalenten Bindung von Ubiquitin an das Substrat durch eine Kaskadenreaktion von mindestens drei verschiedenen Enzymen besteht, die aus E1 (Ubiquitin-Aktivierungsenzym), E2 (Ubiquitin-konjugierendes Enzym) und E3 (Ubiquitin-Ligase-Enzym) bestehen. Der E3-Komplex spielt eine wichtige Rolle bei der Substraterkennung und Ubiquitylierung. Hier wird ein Protokoll zur Bewertung der Substratubiquitylierung in Säugetierzellen unter Verwendung einer vorübergehenden Co-Transfektion eines Plasmids, das für das ausgewählte Substrat, eine E3-Ubiquitin-Ligase und ein markiertes Ubiquitin kodiert, beschrieben. Vor der Lyse werden die transfizierten Zellen mit dem Proteasom-Inhibitor MG132 (Carbobenzoxy-leu-leu-leucinal) behandelt, um einen proteasomalen Substratabbau zu vermeiden. Darüber hinaus wird der Zellextrakt einer kleinräumigen Immunpräzipitation (IP) unterzogen, um das polyubiquitylierte Substrat für den anschließenden Nachweis durch Western Blotting (WB) unter Verwendung spezifischer Antikörper für den Ubiquitin-Tag zu reinigen. Daher wird ein konsistentes und unkompliziertes Protokoll für den Ubiquitylierungsassay in Säugetierzellen beschrieben, um Wissenschaftler bei der Behandlung der Ubiquitylierung spezifischer Substrate und E3-Ubiquitin-Ligasen zu unterstützen.
Einleitung
Posttranslationale Modifikationen (PTMs) sind ein wichtiger Mechanismus für die Proteinregulation, die für die Zellhomöostase essentiell ist. Die Proteinubiquitylierung ist eine dynamische und komplizierte Modifikation, die eine Auswahl verschiedener Signale erzeugt, was zu mehreren zellulären Ergebnissen in eukaryotischen Organismen führt. Die Ubiquitylierung ist ein reversibler Prozess, der in der Anlagerung eines Ubiquitinproteins mit 76 Aminosäuren an das Substrat besteht und in einer enzymatischen Kaskade auftritt, die aus drei verschiedenen Reaktionen besteht1. Der erste Schritt ist durch eine Ubiquitin-Aktivierung gekennzeichnet, die von einer ATP-Hydrolyse abhängt, um ein hochenergetisches Thioester-verknüpftes Ubiquitin zwischen dem Ubiquitin-C-Terminus und dem im aktiven Zentrum des E1-Enzyms vorhandenen Cysteinrest zu bilden. Anschließend wird das Ubiquitin auf das E2-Enzym übertragen und bildet mit dem Ubiquitin einen Thioester-ähnlichen Komplex. Danach wird das Ubiquitin kovalent durch das E2 oder häufiger durch das E3-Enzym, das das Substrat 2,3 erkennt und mit ihm interagiert, kovalent an das Substratgebunden. Gelegentlich sind E4-Enzyme (Ubiquitin-Kettendehnungsfaktoren) notwendig, um den Multiubiquitin-Kettenaufbauzu fördern 3.
Ubiquitin hat sieben Lysinreste (K6, K11, K27, K29, K33, K48 und K63), was die Bildung von Polyubiquitinketten ermöglicht, die unterschiedliche Verbindungen erzeugen, um verschiedene dreidimensionale Strukturen zu erzeugen, die von mehreren Effektorproteinen erkannt werden 4,5. Daher ist die Art der Polyubiquitinkette, die in das Substrat eingeführt wird, für das Schicksal seiner Zelleunerlässlich 6,7,8. Darüber hinaus könnte das Substrat auch durch seine N-terminalen Reste, die N-Degrons genannt werden, ubiquitiniert werden. Spezifische E3-Ubiquitin-Ligasen sind für die N-Degron-Erkennung verantwortlich und ermöglichen die Polyubiquitylierung des nahegelegenen Lysinrests9.
Heute sind mehr als 40 verschiedene SCF-spezifische Substrate charakterisiert. Unter diesen können wichtige Regulatoren mehrerer biologischer Signalwege, einschließlich Zelldifferenzierung und -entwicklung sowie Zellüberleben und -tod, gefunden werden10,11,12,13. Daher ist die Identifizierung spezifischer Substrate jeder E3-Ubiquitin-Ligase unerlässlich, um eine umfassende Karte verschiedener biologischer Ereignisse zu entwerfen. Auch wenn die Identifizierung echter Substrate biochemisch eine Herausforderung darstellt, ist der Einsatz biochemisch basierter Methoden sehr gut geeignet, um die Kettenspezifität und die Unterscheidung zwischen Mono- und Polyubiquitylierungzu bewerten 14. Diese Studie beschreibt ein vollständiges Protokoll für den Ubiquitylierungsassay unter Verwendung der Säugetierzelllinie HEK293T, die das Substrat UXT-V2 (Ubiquitously exprimierten Prefoldin-like Chaperon-Isoform 2) mit dem E3-Ubiquitin-Ligase-Komplex SCF (Fbxo7) überexprimiert. UXT-V2 ist ein essentieller Co-Faktor für die NF-κB-Signalgebung, und sobald dieses Protein in Zellen niedergeschlagen wird, hemmt es die TNF-α-induzierte NF-κB-Aktivierung11. So wird zum Nachweis von polyubiquityliertem UXT-V2 der Proteasom-Inhibitor MG132 verwendet, da er die Fähigkeit hat, die proteolytische Aktivität der 26S-Untereinheit des Proteasom-Komplexes15 zu blockieren. Darüber hinaus wird der Zellextrakt einem kleinen IP zur Reinigung des Substrats unterzogen, wobei ein spezifischer Antikörper verwendet wird, der zu Agaroseharz immobilisiert ist, um anschließend von WB unter Verwendung ausgewählter Antikörper nachgewiesen zu werden. Dieses Protokoll ist sehr nützlich, um die Substratubiquitylierung in der zellulären Umgebung zu validieren, und es kann auch für verschiedene Arten von Säugetierzellen und anderen E3-Ubiquitin-Ligase-Komplexen angepasst werden. Es ist jedoch notwendig, das getestete Substrat auch durch einen In-vitro-Ubiquitylierungsassay zu validieren, da sich beide Protokolle hinsichtlich der Identifizierung echter Substrate ergänzen.
Protokoll
HINWEIS: Eine Übersicht über das Ubiquitylierungsassay-Protokoll in Säugetierzellen ist in Abbildung 1 dargestellt.
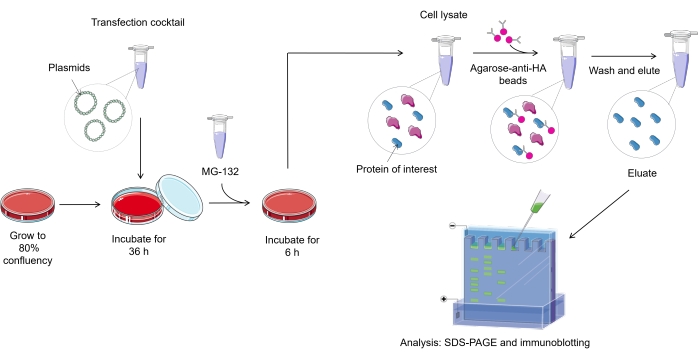
Abbildung 1. Überblick über das Ubiquitylierungsassay-Verfahren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
1. Zellkultur
- Züchten Sie die HEK293T-Zelllinie in einer 100 mm TC-behandelten Kulturschale auf 80%-90% Konfluenz in Wachstumsmedien (Dulbeccos Modified Eagle's Medium (DMEM) hohe Glukose, ergänzt mit 10% fetalem Rinderserum (FBS) und Penicillin (100 Einheiten), Streptomycin (100 μg) und L-Glutamin (0,292 mg/ml)). Inkubieren Sie die Kultur bei 37 °C in einem befeuchteten Zellkultur-Inkubator bei 5% CO2.
- Um die Zellen zu passieren, saugen Sie die Medien mit einer serologischen Pipette aus der Kulturschale ab. Waschen Sie die Zellen einmal mit 1 ml sterilisierter 1x phosphatgepufferter Kochsalzlösung (PBS 1x).
- Lösen Sie die Zellen durch Zugabe von 1 ml Trypsin / EDTA-Lösung (Ethylendiamintetraessigsäure). Die Schale bei 37 °C 5 min inkubieren. Resuspendieren Sie die Zellen mit einer serologischen Pipette in 2 ml Wachstumsmedien.
- Übertragen Sie die Zellsuspension in ein frisches und sauberes 15-ml-Röhrchen und eine Zentrifuge bei 500 x g für 5 min bei Raumtemperatur (RT). Entfernen Sie den Überstand, indem Sie ihn vorsichtig ausgießen. Resuspendieren Sie das Zellpellet vorsichtig in 3 ml Wachstumsmedien durch Auf- und Abpipettieren, um eine homogene Zellsuspension zu erhalten.
- Übertragen Sie 1 ml der Zellsuspension auf eine 100 mm TC-behandelte Kulturschale, die 9 ml Wachstumsmedien enthält.
HINWEIS: Wenn sich die Zellen in gutem Zustand befinden, kann jede Kulturschale von HEK293T, bei der der Zusammenfluss 80% -90% beträgt, 2 Tage nach der Passage drei Kulturschalen mit 80% Konfluenz erzeugen.
2. Zelltransfektion
HINWEIS: Es wird nicht empfohlen, die Zellkultur zu transfizieren, wenn der erreichte Zusammenfluss weniger als 80% beträgt.
- Bestätigen Sie vor der Transfektion, ob die Zellen frei von Kontamination sind und sich an einem angemessenen Zusammenfluss für vorübergehende Transfektionen befinden.
- Für jede Transfektionsprobe werden die DNA-Polyethylenimin (PEI 1 μg/μL bei pH 7,2) Komplexe wie folgt hergestellt:
- Verdünnen Sie 3 μg jedes Plasmids in 100 μL opti-MEM I reduziertem Serummedium ohne Supplementierung und mischen Sie es vorsichtig, indem Sie die Lösung auf und ab pipettieren.
HINWEIS: Hier wurden die Zellen mit 4 μg von jedem Plasmid transfiziert: Leere Vektor (pcDNA3) oder FLAG-Fbxo7-Konstrukte und UXT-V2-HA, mit oder ohne 6xHis-myc-Ubiquitin. Der gesamte DNA-Gehalt betrug 12 μg. - Tauen Sie den PEI bei RT auf und fügen Sie ihn nach einem Anteil von 3 μL PEI pro 1 μg DNA in die Lösung ein. Homogenisieren Sie die Lösung, indem Sie sie nach oben und unten pipettieren. Dann inkubieren Sie es für 15 Minuten bei RT, um die Bildung von DNA-PEI-Komplexen zu ermöglichen.
HINWEIS: Das optimale Verhältnis des PEI-Volumens pro DNA-Menge unterscheidet sich je nach ausgewählter Zelllinie.
- Verdünnen Sie 3 μg jedes Plasmids in 100 μL opti-MEM I reduziertem Serummedium ohne Supplementierung und mischen Sie es vorsichtig, indem Sie die Lösung auf und ab pipettieren.
- Fügen Sie das Gesamtvolumen der DNA-PEI-Komplexe zu jeder Schale hinzu, die die Zellkultur enthält, und mischen Sie sie vorsichtig, indem Sie die Platte hin und her schaukeln. Inkubieren Sie die Zellen bei 37 °C in einem befeuchteten Zellkultur-Inkubator bei 5% CO2.
- Ersetzen Sie das Wachstumsmedium nach 5 h, da eine längere PEI-Exposition für HEK293T-Zellen toxisch sein kann. Inkubieren Sie die Zellen bei 37 °C in einem befeuchteten Zellkultur-Inkubator bei 5% CO2 für 36 h.
3. Zelllyse und Immunpräzipitation
- Nach der Inkubationszeit und 6 h vor der Zelllyse die transfizierten Zellen mit 10 μM des Proteasom-Inhibitors MG-132 behandeln. Inkubieren Sie die Zellen erneut bei 37 °C in einem befeuchteten Zellkultur-Inkubator bei 5%CO2.
- Saugen Sie die Medien mit einer serologischen Pipette aus jeder Kulturschale ab und waschen Sie sie einmal mit 1 ml 1x PBS. Lösen Sie die Zellen, indem Sie 1 ml Trypsin hinzufügen und die Schale bei 37 ° C für 5 Minuten inkubieren. Resuspendieren Sie die Zellen in 1 ml Wachstumsmedien.
- Übertragen Sie die Zellsuspension in ein frisches und sauberes 15-ml-Röhrchen und zentrifugieren Sie es bei 500 x g für 5 min bei RT.
- Entfernen Sie den Überstand, indem Sie ihn vorsichtig ausgießen. Das Zellpellet wird vorsichtig in 200 μL eiskaltem NP-40-Lysepuffer (50 mM Tris-HCl pH 7,2, 225 mM KCl und 1% NP-40) resuspendiert, mit dem Protease- und Phosphatase-Inhibitoren-Cocktail (10 mM NaF und 1 mM Na3VO4) ergänzt und die Lösung in ein sauberes 1,5 mL-Mikroröhrchen übertragen.
- Inkubieren Sie das Zelllysat für 30 min auf Eis. Nach der Inkubation zentrifugieren Sie die Zelllysate bei 16.900 x g für 20 min bei 4 °C.
- In der Zwischenzeit die Agarose-Anti-HA-Perlen mit dem eiskalten NP-40-Lysepuffer ausgleichen. Verwenden Sie 15 μL Agarose-Anti-HA-Perlen für jede Probe. Waschen Sie die Perlen mit 200 μL NP-40-Lysepuffer, indem Sie sie in einem Mikrozentrifugenröhrchen bei 3.000 x g für 1 min bei 4 °C pulsieren. Mit einer Pipette den Überstand sehr vorsichtig absaugen und entsorgen; Wiederholen Sie diesen Vorgang dreimal. Danach halten Sie die Perlen bis zum Gebrauch auf Eis ausgeglichen.
- Nach dem Zentrifugieren der Zelllysate wird der Überstand zurückgewonnen. Quantifizieren Sie den Proteingehalt im Gesamtlysat mit der Bradford-Methode16.
- Stellen Sie sicher, dass jede Probe, die einer Immunpräzipitation unterzogen wird, eine gleiche Menge an Protein enthält. Inkubieren Sie das notwendige Zelllysatvolumen mit den ausgleichenden Agarose-Anti-HA-Perlen für 4 h und drehen Sie sich vorsichtig in einem rotierenden Inkubator bei 4 ° C, wodurch sich der UXT-V2-HA an die Agarose-Anti-HA-Perlen binden kann.
- Sammeln Sie die Agarose-Anti-HA-Perlen, indem Sie sie in einem Mikrozentrifugenröhrchen bei 3.000 x g für 1 min bei 4 °C pulsieren. Den Überstand vorsichtig absaugen und verwerfen. Waschen Sie die Perlen dreimal mit eiskaltem NP-40-Zelllysepuffer und zweimal mit eiskaltem FLAG/HA-Puffer (10 mM Hepes pH 7,9, 15 mM MgCl2, 225 mM KCl und 0,1% NP-40).
- Entfernen Sie nach der letzten Wäsche vorsichtig den gesamten Überstand mit einer Pipette und eluieren Sie das polyubiquitylierte Protein mit HA-Peptid (300 μg / ml), das auf FLAG / HA-Puffer verdünnt ist. Die Agarose-Anti-HA-Perlen mit HA-Peptid für 1 h bei 4 °C in einer schaukelnden Shaker-Plattform inkubieren.
- Drehen Sie die Perlen bei 3.000 x g für 2 min bei 4 °C nach unten und pipettieren Sie vorsichtig den Überstand, der die polyubiquitinierten Proteine enthält. Bei Bedarf das Eluat in einem frischen und sauberen Mikroröhrchen bei -20 °C lagern.
- Die Eluate und die Zelllysate werden in 10% SDS-PAGE (Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese)17 und Immunoblotting aufgelöst.
- In dieser Studie wurde ein Nasstransfer WB durchgeführt. Für diese Art des Transfers legen Sie das Gel in ein Transfersandwich aus Filterpapier-Gel-Membran-Filterpapier, polstern Sie es mit Pads ab und drücken Sie es durch ein Stützgitter zusammen. Platzieren Sie dieses System vertikal in einem mit Transferpuffer gefüllten Tank und zwischen Edelstahl/Platin-Drahtelektroden. Der Transfer erfolgt für 90 min bei 150 V im Nasstransferpuffer (Glycin 192 mM, Tris-Base 25 mM, 0,025% SDS, 20% Methanol).
HINWEIS: Da die Zellextrakte quantifiziert wurden (Schritt 3.7), führen Sie eine SDS-PAGE mit einer gleichen Menge an Protein für jede Probe durch. Um das Eluat aufzulösen, führen Sie außerdem gleiche Volumina jeder Stichprobe aus. - Sondieren Sie die Immunoblot-Membran mit ausgewählten Antikörpern 11. Stellen Sie sicher, dass ein Abstrichsignal im Eluat aus dem polyubiquitylierten Substrat detektiert wird, das im IP-Prozess gezogen wird, indem Sie einen Antimyc-Antikörper in den Proben verwenden, die die Wildtyp-E3-Ligase, das Substrat und Myc-ub enthalten. Stellen Sie im Zelllysat (Input) sicher, dass das Signal des gewählten Substrats, des Fbxo7-Proteins, der ubiquitylierten Proteine und eines Housekeeping-Proteins (z. B. GAPDH und β-Actin) nachgewiesen wird, um die gleiche Menge an Proteinen in jeder Gasse zu gewährleisten.
HINWEIS: Die Verdünnung für jeden verwendeten Antikörper wurde gemäß den Anweisungen des Herstellers vorbereitet.
Ergebnisse
UXT (ubiquitär exprimiertes Transkript) ist ein präfoldinartiges Protein, das ubiquitär exprimierte Proteinfaltungskomplexe in Maus- und menschlichen Geweben wie Herz, Gehirn, Skelettmuskulatur, Plazenta, Bauchspeicheldrüse, Niere und Leberbildet 18. Zwei Spleißisoformen von UXT, die als UXT-V1 und UXT-V2 bezeichnet werden, wurden beschrieben, die unterschiedliche Funktionen und subzelluläre Standorte erfüllen. UXT-V1 ist überwiegend im Zytoplasma und in den Mitochondrien lokalisiert und a...
Diskussion
Die Ubiquitylierung ist eine essentielle posttranslationale Modifikation, die die Spiegel mehrerer Proteine reguliert und eine entscheidende Rolle in vielen Signalwegen und biologischen Prozessen spielt, um eine gesunde intrazelluläre Umgebung zu gewährleisten. Das Ubiquitin-Proteasom-System (UPS) ist einer der Schwerpunkte der neueren pharmazeutischen Forschung und bietet die Möglichkeit, Tumorsuppressoren zu stabilisieren oder den Abbau onkogener Produkte zu induzieren22. Zum Beispiel förder...
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt vorliegt.
Danksagungen
F.R.T wird von der FAPESP-Fördernummer 2020/15771-6 und der CNPq Universal 405836/2018-0 unterstützt. P.M.S.P und V.S werden von CAPES unterstützt. C.R.S.T.B.C wurde durch die FAPESP-Stipendiennummer 2019/23466-1 unterstützt. Wir danken Sandra R. C. Maruyama (FAPESP 2016/20258-0) für die materielle Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
Referenzen
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten