需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
E3泛素连接酶在哺乳动物细胞裂解物中底物泛素化的评价
摘要
我们为哺乳动物细胞中特定底物和E3泛素连接酶的泛素化测定提供了详细的方案。HEK293T细胞系用于蛋白质过表达,通过免疫沉淀从细胞裂解物中纯化多泛型底物,并在SDS-PAGE中分离。免疫印迹用于可视化这种翻译后修饰。
摘要
泛型化是一种发生在真核细胞中的翻译后修饰,对几种生物学途径的调节至关重要,包括细胞存活、增殖和分化。这是一个可逆的过程,由泛素通过至少三种不同酶的级联反应与底物的共价连接组成,这些酶由E1(泛素活化酶),E2(泛素结合酶)和E3(泛素连接酶)组成。E3复合物在底物识别和泛素化中起着重要作用。这里描述了一种方案,用于评估哺乳动物细胞中的底物泛素化,使用编码所选底物的质粒,E3泛素连接酶和标记的泛素的瞬时共转染。在裂解之前,用蛋白酶体抑制剂MG132(羧苄氧基-亮氨酸-亮氨酸)处理转染的细胞,以避免底物蛋白酶体降解。此外,将细胞提取物提交到小规模免疫沉淀(IP)中以纯化多泛素底物,以便随后使用泛素标签的特异性抗体通过免疫印迹(WB)进行检测。因此,描述了哺乳动物细胞中泛素化测定的一致且简单的方案,以帮助科学家解决特定底物和E3泛素连接酶的泛素化。
引言
翻译后修饰(PTMs)是关于蛋白质调控的重要机制,对细胞稳态至关重要。蛋白质泛素化是一种动态且复杂的修饰,可产生各种不同的信号,从而在真核生物中产生多种细胞结果。泛素化是一个可逆的过程,包括将含有76个氨基酸的泛素蛋白附着到底物上,发生在由三个不同反应组成的酶级联反应1中。第一步的特征在于泛素活化,这取决于ATP水解在泛素C端和存在于E1酶活性位点中的半胱氨酸残基之间形成高能硫酯连接的泛素。随后,泛素被转移到E2酶中,与泛素形成硫酯样复合物。之后,泛素通过E2共价连接到底物上,或者更常见的是E3酶,其识别并与底物2,3相互作用。偶尔,E4酶(泛素链伸长因子)对于促进多泛素链组装是必需的3。
泛素有七个赖氨酸残基(K6,K11,K27,K29,K33,K48和K63),允许形成多泛素链,产生不同的连锁反应,产生不同的三维结构,这些结构将被几种效应蛋白4,5识别。因此,在底物中引入的多泛素链对于决定其细胞命运6,7,8至关重要。此外,底物也可以通过其称为N-德隆的N端残基被泛素化。特定的E3泛素连接酶负责N-degron识别,允许附近赖氨酸残基的多泛素化9。
如今,有40多种不同的SCF专用基板被表征。其中,几种生物途径的关键调节因子,包括细胞分化和发育以及细胞存活和死亡,可以找到10,11,12,13。因此,鉴定每个E3泛素连接酶的特定底物对于设计各种生物事件的综合图谱至关重要。尽管鉴定真正的底物在生化上具有挑战性,但使用基于生物化学的方法非常适合评估链特异性以及单泛型和多泛型化之间的区别14。本研究描述了使用哺乳动物细胞系HEK293T过表达底物UXT-V2(普遍表达的预折叠样伴侣同种型2)与E3泛素 - 连接酶复合物SCF(Fbxo7)的泛素化测定的完整方案。UXT-V2是NF-κB信号传导的重要辅助因子,一旦这种蛋白质在细胞中被敲低,它就会抑制TNF-α诱导的NF-κB活化11。因此,为了检测多泛型UXT-V2,使用蛋白酶体抑制剂MG132,因为它具有阻断蛋白酶体复合物15的26S亚基的蛋白水解活性的能力。此外,将细胞提取物提交给小规模IP以纯化底物,利用固定在琼脂糖树脂中的特异性抗体,随后由WB使用选定的抗体进行检测。该协议对于验证细胞环境中的底物泛素化非常有用,并且还可以适应不同类型的哺乳动物细胞和其他E3泛素 - 连接酶复合物。然而,有必要验证通过 体外 泛型化测定测试的底物,因为两种方案在鉴定真实底物方面相互补充。
研究方案
注意: 图1概述了哺乳动物细胞中的泛型化测定方案。
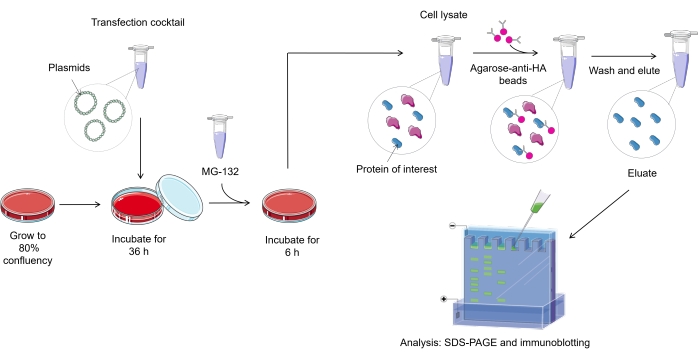
图 1. 泛素化测定程序概述。 请点击此处查看此图的大图。
1. 细胞培养
- 在100毫米TC处理的培养皿中将HEK293T细胞系生长至生长培养基中80%-90%汇合(Dulbecco的改性鹰培养基(DMEM)高葡萄糖,补充10%胎牛血清(FBS)和青霉素(100单位),链霉素(100μg)和L-谷氨酰胺(0.292mg / mL))。将培养物在37°C下在加湿的细胞培养箱中以5%CO2孵育。
- 要传代细胞,请使用血清学移液管从培养皿中吸出培养基。用1mL灭菌的1x磷酸盐缓冲盐水(PBS 1x)洗涤细胞一次。
- 通过加入1mL胰蛋白酶/ EDTA(乙二胺四乙酸)溶液分离细胞。将培养皿在37°C孵育5分钟。使用血清学移液管将细胞重悬于2mL生长培养基中。
- 将细胞悬浮液转移到新鲜干净的15mL管中,并在室温(RT)下以500× g 离心5分钟。小心地倒出上清液,将其取出。通过上下移液将细胞沉淀轻轻重悬于3mL生长培养基中,以获得均匀的细胞悬浮液。
- 将1 mL细胞悬浮液转移到含有9 mL生长培养基的100 mm TC处理的培养皿中。
注意:如果细胞处于良好状态,则HEK293T的每个培养皿,其中汇合度为80%-90%,在传代2天后可以生成三个具有80%汇合度的培养皿。
2. 细胞转染
注意:如果达到的汇合度小于80%,则不建议转染细胞培养物。
- 在转染之前,请验证细胞是否不受污染,以及是否有足够的汇合度进行瞬时转染。
- 对于每个转染样品,制备DNA-聚乙烯亚胺(PEI 1 μg/μL,pH值为7.2)复合物,如下所示:
- 在100μLopti-MEM中稀释3μg每个质粒,I减少血清培养基而不补充,并通过上下移液溶液轻轻混合。
注意:在这里,用4μg每个质粒转染细胞:空载体(pcDNA3)或FLAG-Fbxo7构建体和UXT-V2-HA,有或没有6x-菌丝-泛素。总DNA含量为12微克。 - 在室温下解冻PEI,然后按照每1μgDNA3μLPEI的比例将其添加到溶液中。通过上下移液使溶液均质化。然后在室温下孵育15分钟以允许形成DNA-PEI复合物。
注意:每个DNA数量的PEI体积的最佳比例根据所选的细胞系而有所不同。
- 在100μLopti-MEM中稀释3μg每个质粒,I减少血清培养基而不补充,并通过上下移液溶液轻轻混合。
- 将总体积的DNA-PEI复合物加入到含有细胞培养物的每个培养皿中,并通过来回摇动板来轻轻混合。将细胞在37°C下以5%CO2的加湿细胞培养箱中孵育。
- 5小时后更换生长培养基,因为长时间暴露于PEI可能对HEK293T细胞有毒。将细胞在37°C下在加湿的细胞培养箱中以5%CO2 孵育36小时。
3. 细胞裂解和免疫沉淀
- 在孵育期后和细胞裂解前6小时,用10μM蛋白酶体抑制剂MG-132处理转染的细胞。再次,将细胞在37°C下以5%CO2的加湿细胞培养箱孵育。
- 使用血清学移液管从每个培养皿中吸出培养基,并用1 mL 1x PBS洗涤一次。通过加入1mL胰蛋白酶并在37°C下孵育皿5分钟来分离细胞。将细胞重悬于1 mL生长培养基中。
- 将细胞悬浮液转移到新鲜干净的15mL管中,并在室温下以500× g 离心5分钟。
- 小心地倒出上清液,将其取出。轻轻地将细胞沉淀重悬于200μL冰冷的NP-40裂解缓冲液(50mM Tris-HCl pH 7.2,225mM KCl和1%NP-40)中,补充有蛋白酶和磷酸酶抑制剂混合物(10mM NaF和1mM Na3VO4),并将溶液转移到干净的1.5mL微管中。
- 将细胞裂解物在冰上孵育30分钟。孵育后,将细胞裂解物在16,900× g 下在4°C下离心20分钟。
- 同时,用冰冷的NP-40裂解缓冲液平衡琼脂糖抗HA珠。每个样品使用15μL琼脂糖抗HA珠。用200μLNP-40裂解缓冲液洗涤磁珠,方法是将其在微量离心管中以3,000× g 在4°C下脉冲1分钟。 用移液管,非常小心地吸出并丢弃上清液;重复此过程三次。之后,将珠子保持在冰上平衡,直到使用。
- 离心细胞裂解物后,回收上清液。使用布拉德福德方法16量化总裂解物中的蛋白质含量。
- 确保每个接受免疫沉淀的样品都呈现等量的蛋白质。将必要体积的细胞裂解物与平衡的琼脂糖抗HA珠子孵育4小时,在4°C的旋转培养箱中轻轻旋转,这允许UXT-V2-HA与琼脂糖抗HA珠子结合。
- 通过在微量离心管中以3,000× g 在4°C下脉冲1分钟来收集琼脂糖抗HA珠子。 小心地吸出并丢弃上清液。用冰冷的NP-40细胞裂解缓冲液洗涤珠三次,用冰冷的FLAG / HA缓冲液(10mMHepes pH 7.9,15mMMgCl2,225mM KCl和0.1%NP-40)洗涤两次。
- 最终洗涤后,使用移液管小心地除去所有上清液,并用在FLAG / HA缓冲液上稀释的HA肽(300μg/ mL)洗脱多泛型蛋白。将琼脂糖抗HA珠与HA肽在4°C下在摇动平台中孵育1小时。
- 在4°C下以3,000× g 离心珠2分钟,并小心地移液含有多泛素化蛋白的上清液。如有必要,将洗脱液储存在-20°C的新鲜干净的微管中。
- 在10%SDS-PAGE(十二烷基硫酸钠 - 聚丙烯酰胺凝胶电泳)17 和免疫印迹中溶解洗脱物和细胞裂解物。
- 在这项研究中,进行了湿转移WB。对于这种转印,将凝胶放入由滤纸 - 凝胶 - 膜 - 滤纸组成的转印夹层中,用垫垫垫,并通过支撑网格将其压在一起。将此系统垂直放置在充满转移缓冲液的罐中以及不锈钢/铂丝电极之间。在150 V下在湿转移缓冲液(甘氨酸192mM,三碱基25mM,0.025%SDS,20%甲醇)中进行转移90分钟。
注意:由于细胞提取物已定量(步骤3.7),因此对于每个样品运行具有等量蛋白质的SDS-PAGE。此外,要解决洗脱液,请运行等体积的每个样品。 - 使用选定的抗体 11探测免疫印迹膜。通过在含有野生型E3连接酶,底物和myc-ub的样品中使用抗真菌抗体,确保在IP过程中拉出的多泛型底物的洗脱液中检测到涂片信号。在细胞裂解物(输入)中,确保检测到来自所选底物,Fbxo7蛋白,泛素蛋白和管家蛋白(例如GAPDH和β-肌动蛋白)的信号,以保证每个泳道中相同数量的蛋白质。
注意:所用每种抗体的稀释液是根据制造商的说明制备的。
结果
UXT(普遍表达的转录本)是一种前折叠蛋白样蛋白,在小鼠和人体组织(如心脏、大脑、骨骼肌、胎盘、胰腺、肾脏和肝脏)中形成普遍表达的蛋白质折叠复合物18.已经描述了两种名为UXT-V1和UXT-V2的UXT剪接同种型,它们执行不同的功能和亚细胞位置。UXT-V1主要局限于细胞质和线粒体内,并且与TNF-α诱导的细胞凋亡和抗病毒信号体形成19,20<...
讨论
泛型化是一种基本的翻译后修饰,可调节几种蛋白质的水平,并在许多信号通路和生物过程中起着至关重要的作用,从而确保健康的细胞内环境。泛素-蛋白酶体系统(UPS)是最近药物研究的主要焦点之一,它提供了稳定肿瘤抑制因子或诱导致癌产物降解的可能性22。例如,在多发性骨髓瘤 (MM) 中负责单克隆免疫球蛋白分泌的浆细胞肿瘤的异常增殖促进了病理生理学途径,这是...
披露声明
作者声明不存在利益冲突。
致谢
F.R.T 由 FAPESP 授权号 2020/15771-6 和 CNPq 通用 405836/2018-0 提供支持。公共战略计划和 V.S 由资本保护计划提供支持。C.R.S.T.B.C.获得了FAPESP奖学金编号2019 / 23466-1的支持。我们感谢桑德拉·丸山(FAPESP 2016/20258-0)的物质支持。
材料
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
参考文献
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。