Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Évaluation de l’ubiquitylation du substrat par l’ubiquitine-ligase E3 dans les lysats de cellules de mammifères
Dans cet article
Résumé
Nous fournissons un protocole détaillé pour un test d’ubiquitylation d’un substrat spécifique et d’une ubiquitine-ligase E3 dans des cellules de mammifères. Les lignées cellulaires HEK293T ont été utilisées pour la surexpression des protéines, le substrat polyubiquitylé a été purifié des lysats cellulaires par immunoprécipitation et résolu dans SDS-PAGE. L’immunoblotting a été utilisé pour visualiser cette modification post-traductionnelle.
Résumé
L’ubiquitylation est une modification post-traductionnelle qui se produit dans les cellules eucaryotes et qui est essentielle à la régulation de plusieurs voies biologiques, y compris la survie, la prolifération et la différenciation cellulaires. Il s’agit d’un processus réversible qui consiste en une fixation covalente de l’ubiquitine au substrat par une réaction en cascade d’au moins trois enzymes différentes, composées de E1 (enzyme d’activation de l’ubiquitine), E2 (enzyme conjuguant l’ubiquitine) et E3 (enzyme ubiquitine-ligase). Le complexe E3 joue un rôle important dans la reconnaissance et l’ubiquitylation du substrat. Ici, un protocole est décrit pour évaluer l’ubiquitylation du substrat dans les cellules de mammifères en utilisant la co-transfection transitoire d’un plasmide codant pour le substrat sélectionné, une ligase d’ubiquitine E3 et une ubiquitine marquée. Avant la lyse, les cellules transfectées sont traitées avec l’inhibiteur du protéasome MG132 (carbobenzoxy-leu-leu-leucinal) pour éviter la dégradation protéasomique du substrat. En outre, l’extrait cellulaire est soumis à une immunoprécipitation (IP) à petite échelle pour purifier le substrat polyubiquitylé en vue d’une détection ultérieure par transfert western (WB) à l’aide d’anticorps spécifiques pour l’étiquette ubiquitine. Par conséquent, un protocole cohérent et simple pour le dosage de l’ubiquitylation dans les cellules de mammifères est décrit pour aider les scientifiques à traiter l’ubiquitylation de substrats spécifiques et les ligas d’ubiquitine E3.
Introduction
Les modifications post-traductionnelles (PTM) sont un mécanisme important concernant la régulation des protéines, qui est essentielle pour l’homéostasie cellulaire. L’ubiquitylation des protéines est une modification dynamique et complexe qui crée un assortiment de différents signaux entraînant plusieurs résultats cellulaires chez les organismes eucaryotes. L’ubiquitylation est un processus réversible consistant en la fixation d’une protéine d’ubiquitine contenant 76 acides aminés au substrat, se produisant dans une cascade enzymatique composée de trois réactions distinctes1. La première étape est caractérisée par l’activation de l’ubiquitine, qui dépend d’une hydrolyse de l’ATP pour former une ubiquitine liée au thioester de haute énergie entre l’ubiquitine C-terminus et le résidu de cystéine présent dans le site actif de l’enzyme E1. Par la suite, l’ubiquitine est transférée à l’enzyme E2 formant un complexe semblable à un thioester avec l’ubiquitine. Par la suite, l’ubiquitine est fixée de manière covalente au substrat par l’E2, ou plus souvent, par l’enzyme E3, qui reconnaît et interagit avec le substrat 2,3. Parfois, des enzymes E4 (facteurs d’allongement de la chaîne ubiquitine) sont nécessaires pour favoriser l’assemblage de la chaîne multiubiquitine3.
L’ubiquitine a sept résidus de lysine (K6, K11, K27, K29, K33, K48 et K63), permettant la formation de chaînes de polyubiquitine qui génèrent des liaisons distinctes pour produire différentes structures tridimensionnelles qui vont être reconnues par plusieurs protéines effectrices 4,5. Par conséquent, le type de chaîne de polyubiquitine introduit dans le substrat est essentiel pour décider de son devenir cellulaire 6,7,8. De plus, le substrat pourrait également être ubiquitiné par ses résidus N-terminaux appelés N-dégrons. Les ubiquitine-ligases E3 spécifiques sont responsables de la reconnaissance du N-degron, permettant la polyubiquitylation du résidu de lysine9 à proximité.
De nos jours, il existe plus de 40 substrats différents spécifiques au SCF caractérisés. Parmi ceux-ci, les principaux régulateurs de plusieurs voies biologiques, y compris la différenciation et le développementcellulaires ainsi que la survie et la mort cellulaires, peuvent être trouvés 10,11,12,13. Ainsi, l’identification de substrats spécifiques de chaque ubiquitine-ligase E3 est essentielle pour concevoir une carte complète des différents événements biologiques. Même si l’identification de véritables substrats est biochimiquement difficile, l’utilisation de méthodes basées sur la biochimie est très appropriée pour évaluer la spécificité de la chaîne et la distinction entre mono- et polyubiquitylation14. Cette étude décrit un protocole complet pour le dosage de l’ubiquitylation utilisant la lignée cellulaire de mammifères HEK293T surexprimant le substrat UXT-V2 (Ubiquitously expressed prefoldin-like chaperone isoform 2) avec le complexe E3 ubiquitine-ligase SCF (Fbxo7). L’UXT-V2 est un cofacteur essentiel pour la signalisation NF-κB, et une fois que cette protéine est renversée dans les cellules, elle inhibe l’activation NF-κB induite par le TNF-α11. Ainsi, pour détecter l’UXT-V2 polyubiquitylé, l’inhibiteur du protéasome MG132 est utilisé puisqu’il a la capacité de bloquer l’activité protéolytique de la sous-unité 26S du complexe protéasome15. En outre, l’extrait cellulaire est soumis à une IP à petite échelle pour purifier le substrat, en utilisant un anticorps spécifique immobilisé en résine d’agarose pour une détection ultérieure par WB à l’aide d’anticorps sélectionnés. Ce protocole est très utile pour valider l’ubiquitylation du substrat dans l’environnement cellulaire, et il peut également être adapté à différents types de cellules de mammifères et à d’autres complexes E3 ubiquitine-ligase. Cependant, il est nécessaire de valider le substrat testé par un test d’ubiquitylation in vitro, car les deux protocoles se complètent en ce qui concerne l’identification de véritables substrats.
Protocole
REMARQUE : La figure 1 présente un aperçu du protocole de dosage de l’ubiquitylation dans les cellules de mammifères.
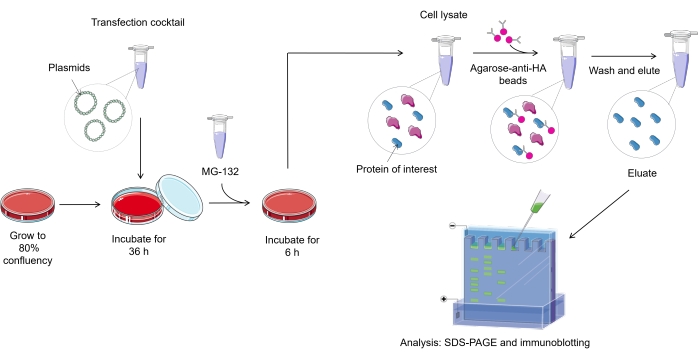
Graphique 1. Vue d’ensemble de la procédure de dosage de l’ubiquitylation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Culture cellulaire
- Cultiver la lignée cellulaire HEK293T dans une boîte de culture traitée par TC de 100 mm jusqu’à une confluence de 80 % à 90 % dans les milieux de croissance (DMEM (Modified Eagle’s Medium) de Dulbecco enrichi en 10 % de sérum fœtal bovin (FBS) et de pénicilline (100 unités), de streptomycine (100 μg) et de L-glutamine (0,292 mg/mL)). Incuber la culture à 37 °C dans un incubateur de culture cellulaire humidifié à 5 % de CO2.
- Pour passer les cellules, aspirer le milieu de la boîte de culture à l’aide d’une pipette sérologique. Lavez les cellules une fois avec 1 mL de solution saline stérilisée 1x tamponnée au phosphate (PBS 1x).
- Détachez les cellules en ajoutant 1 mL de solution de trypsine/EDTA (acide éthylènediaminetétraacétique). Incuber le plat à 37 °C pendant 5 min. Remettre en suspension les cellules à l’aide d’une pipette sérologique dans 2 mL de milieu de croissance.
- Transférer la suspension cellulaire dans un tube frais et propre de 15 mL et centrifuger à 500 x g pendant 5 min à température ambiante (RT). Retirez le surnageant en le versant soigneusement. Remettez doucement en suspension la pastille cellulaire dans 3 mL de milieu de croissance en pipetant de haut en bas pour obtenir une suspension cellulaire homogène.
- Transférer 1 mL de la suspension cellulaire dans une boîte de culture traitée par TC de 100 mm contenant 9 mL de milieu de croissance.
REMARQUE: Si les cellules sont dans de bonnes conditions, chaque plat de culture de HEK293T, dans lequel la confluence est de 80% à 90%, peut générer trois plats de culture avec une confluence de 80% 2 jours après le passage.
2. Transfection cellulaire
REMARQUE: Il n’est pas recommandé de transfecter la culture cellulaire si la confluence atteinte est inférieure à 80%.
- Avant la transfection, certifier si les cellules sont exemptes de contamination et à une confluence adéquate pour les transfections transitoires.
- Pour chaque échantillon de transfection, préparer les complexes ADN-polyéthylène (PEI 1 μg/μL à pH 7,2) comme suit :
- Diluer 3 μg de chaque plasmide dans 100 μL d’opti-MEM I milieu sérique réduit sans supplémentation et mélanger doucement en pipetant la solution de haut en bas.
NOTE: Ici, les cellules ont été transfectées avec 4 μg de chaque plasmide: vecteur vide (pcDNA3) ou FLAG-Fbxo7 construits et UXT-V2-HA, avec ou sans 6xHis-myc-ubiquitine. La teneur totale en ADN était de 12 μg. - Décongeler l’Î.-P.-É. à TA et l’ajouter à la solution en suivant une proportion de 3 μL d’Î.-P.-É. par 1 μg d’ADN. Homogénéiser la solution en pipetant de haut en bas. Ensuite, incubez-le pendant 15 min à TA pour permettre la formation de complexes ADN-PEI.
REMARQUE : La proportion optimale du volume de l’Île-du-Prince-Édouard par quantité d’ADN diffère selon la lignée cellulaire sélectionnée.
- Diluer 3 μg de chaque plasmide dans 100 μL d’opti-MEM I milieu sérique réduit sans supplémentation et mélanger doucement en pipetant la solution de haut en bas.
- Ajoutez le volume total de complexes ADN-PEI à chaque plat contenant la culture cellulaire et mélangez-le doucement en berçant la plaque d’avant en arrière. Incuber les cellules à 37 °C dans un incubateur de culture cellulaire humidifié à 5 % de CO2.
- Remplacez le milieu de croissance après 5 h, car une exposition prolongée à l’Île-du-Prince-Édouard peut être toxique pour les cellules HEK293T. Incuber les cellules à 37 °C dans un incubateur de culture cellulaire humidifié à 5% de CO2 pendant 36 h.
3. Lyse cellulaire et immunoprécipitation
- Après la période d’incubation et 6 h avant la lyse cellulaire, traiter les cellules transfectées avec 10 μM de l’inhibiteur du protéasome MG-132. Encore une fois, incuber les cellules à 37 °C dans un incubateur de culture cellulaire humidifié à 5% de CO2.
- Aspirer le milieu de chaque plat de culture à l’aide d’une pipette sérologique et le laver une fois avec 1 mL de 1x PBS. Détachez les cellules en ajoutant 1 mL de trypsine et en incubant le plat à 37 °C pendant 5 min. Remettre en suspension les cellules dans 1 mL de milieu de croissance.
- Transférer la suspension cellulaire dans un tube frais et propre de 15 mL et centrifuger à 500 x g pendant 5 min à TA.
- Retirez le surnageant en le versant soigneusement. Remettre doucement la pastille cellulaire dans 200 μL de tampon de lyse NP-40 glacé (50 mM Tris-HCl pH 7,2, 225 mM KCl et 1 % NP-40), complété par le cocktail d’inhibiteurs de protéase et de phosphatase (10 mM NaF et 1 mM Na3VO4), et transférer la solution dans un microtube propre de 1,5 mL.
- Incuber le lysat cellulaire pendant 30 min sur de la glace. Après incubation, centrifuger les lysats cellulaires à 16 900 x g pendant 20 min à 4 °C.
- Pendant ce temps, équilibrez les perles d’agarose-anti-HA avec un tampon de lyse NP-40 glacé. Utilisez 15 μL de billes d’agarose-anti-HA pour chaque échantillon. Lavez les billes avec 200 μL de tampon de lyse NP-40 en les pulsant dans un tube de microcentrifugation à 3 000 x g pendant 1 min à 4 °C. Avec une pipette, aspirer et jeter le surnageant très soigneusement; répétez ce processus trois fois. Ensuite, gardez les perles équilibrées sur de la glace jusqu’à utilisation.
- Après avoir centrifugé les lysats cellulaires, récupérez le surnageant. Quantifier la teneur en protéines du lysat total à l’aide de la méthode de Bradford16.
- S’assurer que chaque échantillon soumis à l’immunoprécipitation présente une quantité égale de protéines. Incuber le volume nécessaire de lysat cellulaire avec les billes d’agarose-anti-HA équilibrées pendant 4 h, en tournant doucement dans un incubateur rotatif à 4 °C, ce qui permet à l’UXT-V2-HA de se lier aux billes d’agarose-anti-HA.
- Recueillir les billes d’agarose-anti-HA en les pulsant dans un tube de microcentrifugation à 3 000 x g pendant 1 min à 4 °C. Aspirer soigneusement et jeter le surnageant. Lavez les billes trois fois avec un tampon de lyse cellulaire NP-40 glacé et deux fois avec un tampon FLAG/HA glacé (10 mM Hepes pH 7,9, 15 mM MgCl2, 225 mM KCl et 0,1 % NP-40).
- Après le lavage final, retirer soigneusement tout le surnageant à l’aide d’une pipette et éluer la protéine polyubiquitylée avec le peptide HA (300 μg/mL) dilué sur un tampon FLAG/HA. Incuber les billes d’agarose-anti-HA avec du peptide HA pendant 1 h à 4 °C dans une plate-forme agitatrice à bascule.
- Faire tourner les billes à 3 000 x g pendant 2 min à 4 °C et pipeter soigneusement le surnageant contenant les protéines polyubiquitinées. Si nécessaire, conservez l’éluat dans un microtube frais et propre à -20 °C.
- Résoudre les éluats et les lysats cellulaires dans 10% de SDS-PAGE (électrophorèse sur gel de dodécylsulfate de sodium-polyacrylamide)17 et immunoblotting.
- Dans cette étude, un transfert humide WB a été effectué. Pour ce type de transfert, placez le gel dans un sandwich de transfert composé de papier filtre-gel-membrane-papier filtre, amortissez-le avec des tampons et pressez-le ensemble par une grille de support. Placez ce système verticalement dans un réservoir rempli de tampon de transfert et entre des électrodes en acier inoxydable / fil de platine. Le transfert a lieu pendant 90 min à 150 V dans un tampon de transfert humide (glycine 192 mM, tris-base 25 mM, 0,025% SDS, 20% Méthanol).
REMARQUE: Puisque les extraits cellulaires ont été quantifiés (étape 3.7), exécutez une SDS-PAGE avec une quantité égale de protéines pour chaque échantillon. En outre, pour résoudre l’éluat, exécutez des volumes égaux de chaque échantillon. - Sondez la membrane de l’immunoblot à l’aide d’anticorps sélectionnés 11. Assurez-vous qu’un signal de frottis est détecté dans l’éluat du substrat polyubiquitylé tiré dans le processus IP en utilisant un anticorps anti-myc dans les échantillons contenant la ligase E3 de type sauvage, le substrat et le myc-ub. Dans le lysat cellulaire (entrée), assurez-vous que le signal du substrat choisi, de la protéine Fbxo7, des protéines ubiquitylées et d’une protéine d’entretien ménager (par exemple, GAPDH et β-actine) est détecté pour garantir la même quantité de protéines dans chaque voie.
REMARQUE: La dilution pour chaque anticorps utilisé a été préparée conformément aux instructions du fabricant.
Résultats
L’UXT (transcription exprimée de manière ubiquitaire) est une protéine de type préfoldine qui forme des complexes de repliement des protéines exprimés de manière omniprésente dans les tissus murins et humains tels que le cœur, le cerveau, les muscles squelettiques, le placenta, le pancréas, les reins et le foie18. Deux isoformes d’épissage d’UXT, nommées UXT-V1 et UXT-V2, ont été décrites comme remplissant des fonctions et des emplacements subcellulaires distincts. L’UXT-V1 ...
Discussion
L’ubiquitylation est une modification post-traductionnelle essentielle qui régule les niveaux de plusieurs protéines et joue un rôle crucial dans de nombreuses voies de signalisation et processus biologiques, assurant un environnement intracellulaire sain. Le système ubiquitine-protéasome (UPS) est l’un des principaux axes de la recherche pharmaceutique récente, offrant la possibilité de stabiliser les suppresseurs de tumeurs ou d’induire la dégradation des produits oncogènes22. Par...
Déclarations de divulgation
Les auteurs déclarent qu’il n’y a pas de conflit d’intérêts.
Remerciements
F.R.T est soutenu par le numéro de subvention FAPESP 2020/15771-6 et CNPq Universal 405836/2018-0. P.M.S.P et V.S sont soutenus par CAPES. C.R.S.T.B.C a été soutenu par la bourse FAPESP numéro 2019/23466-1. Nous remercions Sandra R. C. Maruyama (FAPESP 2016/20258-0) pour son soutien matériel.
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
Références
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon