JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
포유동물 세포 용해물에서 E3 유비퀴틴-리가아제에 의한 기질 유비퀴틸화의 평가
요약
우리는 포유동물 세포에서 특정 기질 및 E3 유비퀴틴 리가아제의 유비퀴틸화 검정을 위한 상세한 프로토콜을 제공한다. HEK293T 세포주는 단백질 과발현을 위해 사용되었고, 폴리유비퀴틸화된 기질은 면역침전에 의해 세포 용해물로부터 정제되고, SDS-PAGE에서 분해되었다. 면역블롯팅은 이러한 번역 후 변형을 시각화하기 위해 사용되었다.
초록
유비퀴틸화는 진핵 세포에서 발생하는 번역 후 변형으로, 세포 생존, 증식 및 분화를 포함한 여러 생물학적 경로의 조절에 중요합니다. E1 (유비퀴틴 활성화 효소), E2 (유비퀴틴 접합 효소) 및 E3 (유비퀴틴 리가아제 효소)로 구성된 적어도 3 가지 효소의 캐스케이드 반응을 통해 기질에 유비퀴틴을 공유적으로 부착하는 가역적 인 과정입니다. E3 복합체는 기질 인식 및 유비퀴틸화에 중요한 역할을 한다. 여기서, 선택된 기질을 코딩하는 플라스미드, E3 유비퀴틴 리가아제, 및 태그된 유비퀴틴의 일시적 공동형질감염을 사용하여 포유동물 세포에서 기질 유비퀴틸화를 평가하기 위한 프로토콜이 기재되어 있다. 용해 전에, 형질감염된 세포는 기질 프로테아좀 분해를 피하기 위해 프로테아좀 억제제 MG132 (카르보벤즈옥시-류-류-류알)로 처리된다. 또한, 세포 추출물은 유비퀴틴 태그에 특이적인 항체를 사용하는 웨스턴 블롯팅(WB)에 의한 후속 검출을 위해 폴리유비퀴틸화된 기질을 정제하기 위해 소규모 면역침전(IP)에 제출된다. 따라서, 포유동물 세포에서 유비퀴틸화 분석을 위한 일관되고 복잡하지 않은 프로토콜이 과학자들이 특정 기질 및 E3 유비퀴틴 리가아제의 유비퀴틸화를 다루는 것을 돕기 위해 기술된다.
서문
번역 후 변형 (PTMs)은 세포 항상성에 필수적인 단백질 조절과 관련된 중요한 메커니즘입니다. 단백질 유비퀴틸화는 진핵 생물에서 여러 가지 세포 결과를 초래하는 다양한 신호의 구색을 생성하는 역동적이고 복잡한 변형입니다. 유비퀴틸화는 76개의 아미노산을 함유하는 유비퀴틴 단백질을 기질에 부착시키는 것으로 이루어진 가역적 과정으로, 세 가지 별개의 반응으로 구성된 효소적 캐스케이드에서 발생한다1. 첫 번째 단계는 유비퀴틴 활성화를 특징으로 하는데, 이는 E1 효소의 활성 부위에 존재하는 유비퀴틴 C 말단과 시스테인 잔기 사이에 고에너지 티오에스테르 결합 유비퀴틴을 형성하기 위해 ATP 가수분해에 의존한다. 이어서, 유비퀴틴은 E2 효소로 전달되어 유비퀴틴과 티오에스테르-유사 복합체를 형성한다. 그 후, 유비퀴틴은 E2에 의해 기질에 공유적으로 부착되거나, 또는 더 자주, 기질 2,3을 인식하고 상호작용하는 E3 효소에 의해 공유된다. 때때로, E4 효소 (유비퀴틴 사슬 신장 인자)는 다중 유비퀴틴 사슬 조립을 촉진하기 위해필요하다 3.
유비퀴틴은 7개의 리신 잔기(K6, K11, K27, K29, K33, K48 및 K63)를 가지고 있어, 몇몇 이펙터 단백질 4,5에 의해 인식될 상이한 삼차원 구조를 생성하기 위해 별개의 결합을 생성하는 폴리유비퀴틴 사슬의 형성을 허용한다. 따라서, 기질에 도입된 폴리유비퀴틴 사슬의 종류는 그의 세포 운명 6,7,8을 결정하는 데 필수적이다. 더욱이, 기질은 또한 N-데그론(N-degrons)이라고 불리는 그의 N 말단 잔기를 통해 유비퀴틴화될 수 있다. 특정 E3 유비퀴틴-리가아제는 N-데그론 인식을 담당하며, 근처의 리신 잔기9의 폴리유비퀴틸화를 허용한다.
요즘에는 40 개 이상의 SCF 특정 기판이 특징입니다. 그 중에서도 세포 분화 및 발달뿐만 아니라 세포 생존 및 사망을 포함한 여러 생물학적 경로의 주요 조절인자는10,11,12,13을 찾을 수 있습니다. 따라서, 각 E3 유비퀴틴-리가아제의 특정 기질의 확인은 다양한 생물학적 사건에 대한 포괄적인 지도를 설계하는데 필수적이다. 진정한 기질의 확인이 생화학적으로 도전적이더라도, 생화학 기반 방법의 사용은 사슬 특이성과 모노- 및 폴리유비퀴틸화 사이의 구별을 평가하는데 매우 적합하다(14). 본 연구는 E3 유비퀴틴-리가아제 복합체 SCF(Fbxo7)와 함께 기질 UXT-V2(유비쿼터스로 발현된 프리폴딘-유사 샤페론 이소폼 2)를 과발현하는 포유동물 세포주 HEK293T를 이용한 유비퀴틸화 분석을 위한 완전한 프로토콜을 기술한다. UXT-V2는 NF-κB 신호전달에 필수적인 보조인자이며, 일단 이 단백질이 세포에서 녹다운되면, TNF-α-유도된 NF-κB 활성화(11)를 억제한다. 따라서, 폴리유비퀴틸화된 UXT-V2를 검출하기 위해, 프로테아좀 억제제 MG132는 프로테아좀 복합체15의 26S 서브유닛의 단백질 분해 활성을 차단하는 능력을 갖기 때문에 사용된다. 더욱이, 세포 추출물은 기질을 정제하기 위해 소규모 IP에 제출되고, 선택된 항체를 사용하여 WB에 의한 후속 검출을 위해 아가로스 수지에 고정화된 특이적 항체를 이용한다. 이 프로토콜은 세포 환경에서 기질 유비퀴틸화를 검증하는데 매우 유용하며, 또한 상이한 유형의 포유동물 세포 및 다른 E3 유비퀴틴-리가아제 복합체에 적응될 수 있다. 그러나, 시험관내 유비퀴틸화 분석을 통해 시험된 기질을 검증할 필요가 있는데, 이는 두 프로토콜 모두 진정한 기질의 확인과 관련하여 서로를 보완하기 때문이다.
프로토콜
참고: 포유동물 세포에서 유비퀴틸화 분석 프로토콜의 개요는 도 1에 표시되어 있다.
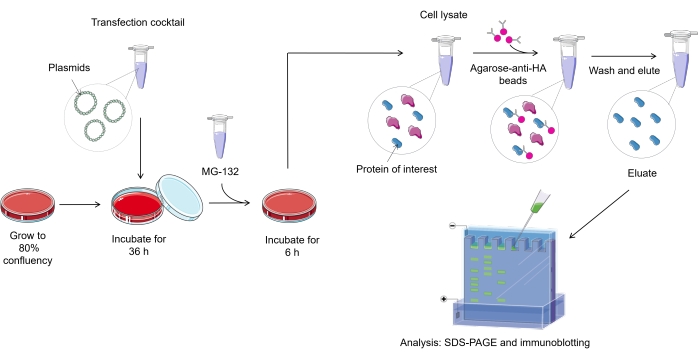
그림 1. 유비퀴틸화 분석 절차의 개요. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
1. 세포 배양
- HEK293T 세포주를 100mm TC 처리 배양 접시에서 성장 배지에서 80%-90% 합류로 성장시킨다(둘베코 변형 이글 배지(DMEM) 고포도당 10% 태아 소 혈청(FBS) 및 페니실린(100 단위), 스트렙토마이신(100 μg) 및 L-글루타민(0.292 mg/mL)으로 보충됨). 배양물을 5%CO2의 가습된 세포 배양 인큐베이터에서 37°C에서 인큐베이션한다.
- 세포를 통과시키기 위해, 혈청학적 피펫을 사용하여 배양 접시로부터 배지를 흡인한다. 세포를 멸균된 1x 포스페이트 완충 식염수 1 mL(PBS 1x)로 1회 세척한다.
- 트립신/EDTA(에틸렌디아민테트라아세트산) 용액 1mL를 첨가하여 세포를 분리합니다. 접시를 37°C에서 5분 동안 인큐베이션한다. 세포를 2 mL의 성장 배지에 혈청학적 피펫을 사용하여 재현탁시킨다.
- 세포 현탁액을 신선하고 깨끗한 15 mL 튜브에 옮기고 실온에서 5분 동안 500 x g 에서 원심분리기(RT)한다. 상층액을 조심스럽게 부어 제거하십시오. 세포 펠릿을 3 mL의 성장 배지에 부드럽게 재현탁시켜 위아래로 피펫팅하여 균질한 세포 현탁액을 얻었다.
- 세포 현탁액 1 mL를 9 mL의 성장 배지를 함유하는 100 mm TC 처리 배양 접시로 옮긴다.
참고 : 세포가 양호한 조건에있는 경우, 합류율이 80 % -90 % 인 HEK293T의 각 배양 접시는 계대 2 일 후에 80 % 합류로 세 가지 배양 접시를 생성 할 수 있습니다.
2. 세포 형질감염
참고: 합류율에 도달한 것이 80% 미만인 경우 세포 배양물을 형질감염시키는 것은 권장되지 않습니다.
- 형질감염 전에, 세포가 오염으로부터 자유롭고 일시적인 형질감염에 대한 적절한 합류에 있는지 인증하십시오.
- 각 트랜스펙션 샘플에 대해 DNA-Polyethylenimine (pH 7.2에서 PEI 1 μg/μL) 복합체를 다음과 같이 준비하십시오.
- 각 플라스미드 3 μg을 100 μL의 opti-MEM I 환원 혈청 배지에 보충 없이 희석하고, 용액을 위아래로 피펫팅하여 부드럽게 혼합한다.
참고: 여기에서, 세포를 4 μg의 각 플라스미드로 형질감염시켰다: 빈 벡터 (pcDNA3) 또는 FLAG-Fbxo7 구축물 및 UXT-V2-HA, 6xHis-myc-유비퀴틴을 포함하거나 포함하지 않는. 총 DNA 함량은 12 μg이었다. - RT에서 PEI를 해동하고 DNA 1 μg 당 PEI의 3 μL의 비율에 따라 용액에 첨가한다. 위아래로 피펫팅하여 용액을 균질화하십시오. 그런 다음 DNA-PEI 복합체의 형성을 허용하기 위해 RT에서 15 분 동안 인큐베이션하십시오.
참고: DNA 양당 PEI 부피의 최적 비율은 선택된 세포주에 따라 다릅니다.
- 각 플라스미드 3 μg을 100 μL의 opti-MEM I 환원 혈청 배지에 보충 없이 희석하고, 용액을 위아래로 피펫팅하여 부드럽게 혼합한다.
- DNA-PEI 복합체의 총 부피를 세포 배양물이 들어있는 각 접시에 넣고 플레이트를 앞뒤로 흔들어 부드럽게 혼합합니다. 세포를 5%CO2의 가습된 세포 배양 인큐베이터에서 37°C에서 인큐베이션한다.
- 5시간 후에 성장 배지를 교체하면, PEI 노출이 길어지면 HEK293T 세포에 독성이 있을 수 있기 때문이다. 세포를 37°C에서 36시간 동안 5%CO2 의 가습된 세포 배양 인큐베이터에서 인큐베이션한다.
3. 세포 용해 및 면역 침전
- 인큐베이션 기간 및 세포 용해 전에 6시간 후, 형질감염된 세포를 10 μM의 프로테아좀 억제제 MG-132로 처리한다. 다시 한번, 세포를 5%CO2의 가습된 세포 배양 인큐베이터에서 37°C에서 인큐베이션한다.
- 혈청학적 피펫을 사용하여 각 배양 접시로부터 배지를 흡인하고, 이를 1 mL의 1x PBS로 1회 세척한다. 트립신 1 mL를 첨가하여 세포를 분리하고, 디쉬를 37°C에서 5분 동안 인큐베이션한다. 세포를 1 mL의 성장 배지에 재현탁시킨다.
- 세포 현탁액을 신선하고 깨끗한 15 mL 튜브로 옮기고 RT에서 5분 동안 500 x g 에서 원심분리한다.
- 상층액을 조심스럽게 부어 제거하십시오. 세포 펠렛을 200 μL의 빙냉 NP-40 용해 완충액 (50 mM Tris-HCl pH 7.2, 225 mM KCl, 및 1% NP-40)에 부드럽게 재현탁시키고, 프로테아제 및 포스파타제 억제제 칵테일 (10 mM NaF 및 1 mMNa3VO4)로 보충하고, 용액을 깨끗한 1.5 mL 마이크로튜브 내로 옮겼다.
- 세포 용해물을 얼음 상에서 30분 동안 인큐베이션한다. 인큐베이션 후, 세포 용해물을 4°C에서 20분 동안 16,900 x g 에서 원심분리한다.
- 한편, 아가로스-항-HA 비드를 빙냉 NP-40 용해 완충액으로 평형화시킨다. 각 샘플에 대해 15 μL의 아가로스 항-HA 비드를 사용한다. 비드를 200 μL의 NP-40 용해 완충액으로 세척하고, 이를 4°C에서 1분 동안 3,000 x g 의 마이크로원심분리 튜브에서 펄싱한다. 피펫으로 상층액을 매우 조심스럽게 흡인하고 버리십시오. 이 과정을 세 번 반복하십시오. 그 후, 사용할 때까지 구슬을 얼음 위에 평형으로 유지하십시오.
- 세포 용해물을 원심분리한 후, 상층액을 회수한다. 브래드포드 방법16을 사용하여 총 용해물 중의 단백질 함량을 정량화한다.
- 면역침전을 실시한 각 샘플이 동일한 양의 단백질을 제시하는지 확인하십시오. 필요한 부피의 세포 용해물을 평형화된 아가로스 항-HA 비드와 함께 4시간 동안 인큐베이션하고, 4°C의 회전 인큐베이터에서 부드럽게 회전시켜 UXT-V2-HA가 아가로스-항-HA 비드에 결합할 수 있게 한다.
- 아가로스 항-HA 비드를 4°C에서 1분 동안 3,000 x g 의 마이크로원심분리 튜브에서 펄싱하여 수집한다. 조심스럽게 상층액을 흡인하고 버리십시오. 빙냉 NP-40 세포 용해 완충액으로 비드를 세 번, 얼음처럼 차가운 FLAG/HA 완충액(10mM Hepes pH 7.9, 15mM MgCl2, 225mM KCl 및 0.1% NP-40)으로 두 번 세척합니다.
- 최종 세척 후, 피펫을 사용하여 모든 상층액을 조심스럽게 제거하고 FLAG/HA 완충액에 희석된 HA 펩티드(300 μg/mL)로 폴리유비퀴틸화된 단백질을 용출시킨다. 아가로스 항-HA 비드를 HA 펩티드와 함께 4°C에서 1시간 동안 락킹 진탕기 플랫폼에서 인큐베이션한다.
- 비드를 4°C에서 2분 동안 3,000 x g 으로 스핀다운하고, 폴리유비퀴틴화 단백질을 함유하는 상등액을 조심스럽게 피펫팅한다. 필요한 경우, 용출액을 -20°C의 신선하고 깨끗한 마이크로튜브에 보관한다.
- 용출액 및 세포 용해물을 10% SDS-PAGE (소듐 도데실 설페이트-폴리아크릴아미드 겔 전기영동)17로 해결하고 면역블롯팅한다.
- 본 연구에서, 습식 전달 WB가 수행되었다. 이러한 종류의 전송을 위해 젤을 여과지 - 젤 - 멤브레인 - 필터 페이퍼로 구성된 전사 샌드위치에 넣고 패드로 쿠션하고 지지대로 함께 누르십시오. 이 시스템을 이송 버퍼로 채워진 탱크와 스테인리스강/백금 와이어 전극 사이에 수직으로 배치합니다. 전달은 습식 전달 완충액 (글리신 192 mM, 트리스-염기 25 mM, 0.025% SDS, 20% 메탄올)에서 150 V에서 90분 동안 발생한다.
참고: 세포 추출물이 정량화되었기 때문에(단계 3.7), 각 샘플에 대해 동일한 양의 단백질로 SDS-PAGE를 실행한다. 또한 용출액을 해결하려면 각 샘플의 동일한 볼륨을 실행하십시오. - 선택된 항체 11을 사용하여 면역블롯 막을 프로브한다. 야생형 E3 리가아제, 기질, 및 myc-ub를 함유하는 샘플에서 항myc 항체를 사용하여 IP 공정에서 뽑아낸 폴리유비퀴틸화된 기질로부터의 용출액에서 도말 신호가 검출되는지 확인한다. 세포 용해물(input)에서, 선택된 기질, Fbxo7 단백질, 유비퀴틸화 단백질, 및 하우스키핑 단백질(예를 들어, GAPDH 및 β-액틴)으로부터의 신호가 검출되어 각 레인에서 동일한 양의 단백질을 보장하는지 확인한다.
주: 사용된 각 항체에 대한 희석은 제조자의 지시에 따라 제조되었다.
결과
UXT (유비쿼터스하게 발현된 전사체)는 마우스와 심장, 뇌, 골격근, 태반, 췌장, 신장 및 간과 같은 인간 조직에서 유비쿼터스하게 발현된 단백질-폴딩 복합체를 형성하는 프리폴딘 유사 단백질이다18. UXT-V1 및 UXT-V2로 명명된 UXT의 두 스플라이싱 이소형이 별개의 기능과 세포하 위치를 수행하는 것으로 설명되었다. UXT-V1은 주로 세포질 및 미토콘드리아 내부에 국소화되며, TNF-α-유?...
토론
유비퀴틸화는 여러 단백질의 수준을 조절하고 많은 신호 전달 경로와 생물학적 과정에서 중요한 역할을하여 건강한 세포 내 환경을 보장하는 번역 후 필수적인 변형입니다. 유비퀴틴-프로테아좀 시스템(UPS)은 최근 제약 연구의 주요 초점 중 하나로서, 종양 억제제를 안정화시키거나 종양원성 생성물(22)의 분해를 유도할 수 있는 가능성을 제공한다. 예를 들어, 다발성 골수종 (MM)...
공개
저자는 이해 상충이 없다고 선언합니다.
감사의 말
F.R.T는 FAPESP 보조금 번호 2020/15771-6 및 CNPq 유니버설 405836/2018-0에서 지원됩니다. P.M.S.P 및 V.S는 CAPES에서 지원됩니다. C.R.S.T.B.C는 FAPESP 장학금 번호 2019/23466-1의 지원을 받았습니다. 재료 지원에 대한 Sandra R. C. Maruyama (FAPESP 2016/20258-0)에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
참고문헌
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유