É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Avaliação da Ubiquização substrato por E3 Ubiquitin-ligase em Lysates de Células Mamárias
Neste Artigo
Resumo
Fornecemos um protocolo detalhado para um ensaio de ubiquização de um substrato específico e uma ubiquitina-ligase E3 em células mamíferas. As linhas celulares HEK293T foram utilizadas para superexpressão de proteínas, o substrato poliubiquizado foi purificado a partir de lisestês celulares por imunoprecipitação, e resolvido em SDS-PAGE. A imunoblotação foi usada para visualizar esta modificação pós-translacional.
Resumo
A ubiquização é uma modificação pós-translacional que ocorre em células eucarióticas que é fundamental para a regulação de várias vias biológicas, incluindo sobrevivência celular, proliferação e diferenciação. É um processo reversível que consiste em um apego covalente da ubiquitina ao substrato através de uma reação em cascata de pelo menos três enzimas diferentes, compostas por E1 (enzima de ativação de ubiquitina), E2 (enzima conjugante de Ubiquitina) e E3 (enzima Ubiquitin-ligase). O complexo E3 desempenha um papel importante no reconhecimento e na ubiquização do substrato. Aqui, um protocolo é descrito para avaliar a ubiquização substrato em células mamíferas usando co-transfecção transitória de um plasmídeo codificando o substrato selecionado, uma liga ligase de ubiquitina E3 e uma ubiquitina marcada. Antes da lise, as células transfeinadas são tratadas com o inibidor proteasome MG132 (carbobenzoxy-leu-leucinal) para evitar a degradação proteasómal substrato. Além disso, o extrato celular é submetido à imunoprecipitação em pequena escala (IP) para purificar o substrato poliubiquítado para posterior detecção por mancha ocidental (WB) usando anticorpos específicos para a tag ubiquitina. Assim, um protocolo consistente e descomplicado para ensaio de onipresença em células de mamíferos é descrito para auxiliar os cientistas no enfrentamento da onipresençação de substratos específicos e ligaduras de ubiquína E3.
Introdução
As modificações pós-translacionais (PTMs) são um mecanismo importante no que diz respeito à regulação da proteína, essencial para a homeostase celular. A ubiquidade proteica é uma modificação dinâmica e intrincada que cria uma variedade de diferentes sinais resultando em vários desfechos celulares em organismos eucarióticos. A ubiquização é um processo reversível que consiste na fixação de uma proteína de ubiquitina contendo 76 aminoácidos ao substrato, ocorrendo em uma cascata enzimática composta por três reações distintas1. O primeiro passo é caracterizado pela ativação da ubiquitina, que depende de uma hidrólise ATP para formar uma oniquitina ligada a tioéster de alta energia entre a ubiquitina C-terminus e o resíduo de cisteína presente no local ativo da enzima E1. Posteriormente, a ubiquitina é transferida para a enzima E2 formando um complexo de tiaster com a ubiquitina. Posteriormente, a ubiquitina é covalentemente ligada ao substrato pelo E2, ou mais frequentemente, pela enzima E3, que reconhece e interage com o substrato 2,3. Ocasionalmente, enzimas E4 (fatores de alongamento da cadeia ubiquitina) são necessárias para promover o conjunto da cadeia multiubiquitina3.
A ubiquitina possui sete resíduos de linfino (K6, K11, K27, K29, K33, K48 e K63), permitindo a formação de cadeias de poliugentina que geram ligações distintas para produzir diferentes estruturas tridimensionais que serão reconhecidas por várias proteínasefeitoras 4,5. Assim, o tipo de cadeia de poliuforma introduzida no substrato é essencial para decidir seu destino celular 6,7,8. Além disso, o substrato também poderia ser ubiquitinado através de seus resíduos n-terminais chamados N-degrons. As ubiquína-ligases específicas E3 são responsáveis pelo reconhecimento de N-degron, permitindo a poliuubiquização do resíduo de liseínanas proximidades 9.
Atualmente, há mais de 40 substratos específicos do SCF caracterizados. Entre elas, podem ser encontrados 10,11,12,13, 12,13. Assim, a identificação de substratos específicos de cada ubiquitina-ligase E3 é essencial para projetar um mapa abrangente de vários eventos biológicos. Embora a identificação de substratos verdadeiros seja bioquimicamente desafiadora, o uso de métodos baseados em bioquímica é muito adequado para avaliar a especificidade da cadeia e a distinção entre mono e poliumipremiado14. Este estudo descreve um protocolo completo para ensaio de onipresença usando a linha celular de mamíferos HEK293T sobreexpressando o substrato UXT-V2 (onipresentemente expresso como acompanhante de ubimia 2) com o complexo e3 ubiquitin-ligase SCF(Fbxo7). O UXT-V2 é um cofator essencial para a sinalização NF-κB, e uma vez que essa proteína é derrubada nas células, inibe a ativação NF-κB induzida por TN α F11. Assim, para detectar o UXT-V2 poliutilítulo, o inibidor proteasome MG132 é utilizado, pois tem a capacidade de bloquear a atividade proteolítica da subunidade 26S do complexoproteasome 15. Além disso, o extrato celular é submetido a um IP de pequena escala para purificar o substrato, utilizando um anticorpo específico imobilizado para resina agarose para posterior detecção por WB usando anticorpos selecionados. Este protocolo é muito útil para validar a ubiquização do substrato no ambiente celular, e também pode ser adaptado para diferentes tipos de células mamíferas e outros complexos de ubiquitina-ligase E3. No entanto, é necessário validar o substrato testado através de um ensaio de onipresença in vitro também, uma vez que ambos os protocolos se complementam em relação à identificação de substratos verdadeiros.
Protocolo
NOTA: Uma visão geral do protocolo de ensaio de onipresença em células de mamíferos está representada na Figura 1.
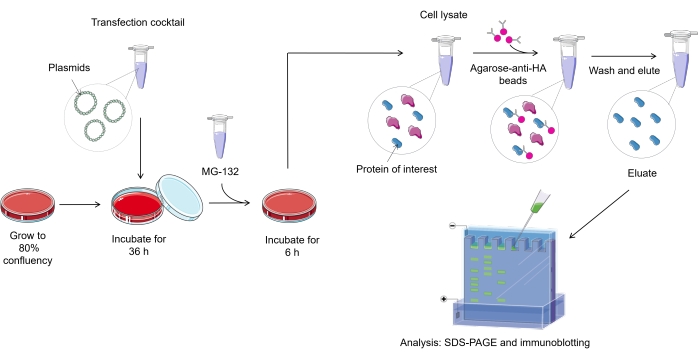
Figura 1. Visão geral do procedimento de ensaio de onipresença. Clique aqui para ver uma versão maior desta figura.
1. Cultura celular
- Cresça a linha de células HEK293T em um prato de cultura tratado com TC de 100 mm para 80%-90% de confluência na mídia de crescimento (Dulbecco's Modified Eagle's Medium (DMEM) alta glicose suplementada com 80%-90% de glicose 10% soro bovino fetal (FBS) e penicilina (100 unidades), estreptomicina (100 μg) e L-glutamina (0,292 mg/mL)). Incubar a cultura a 37 °C em uma incubadora de cultura celular umidificada a 5% de CO2.
- Para passar as células, aspire a mídia do prato de cultura usando uma pipeta sorológica. Lave as células uma vez com 1 mL de soro fisiológico esterilizado 1x tamponado com fosfato (PBS 1x).
- Retire as células adicionando 1 mL de trippsina/EDTA (ácido etilenodiaminetetraacético). Incubar o prato a 37 °C por 5 min. Resuspend as células usando uma pipeta sorológica em 2 mL de mídia de crescimento.
- Transfira a suspensão celular para um tubo fresco e limpo de 15 mL e centrífuga a 500 x g por 5 min a temperatura ambiente (RT). Remova o supernatante derramando-o com cuidado. Resuspenque suavemente a pelota celular em 3 mL de mídia de crescimento, pipetando para cima e para baixo para obter uma suspensão celular homogênea.
- Transfira 1 mL da suspensão celular para um prato de cultura tratado com TC de 100 mm contendo 9 mL de mídia de crescimento.
NOTA: Se as células estiverem em boas condições, cada prato de cultura de HEK293T, em que a confluência é de 80%-90%, pode gerar três pratos de cultura com 80% de confluência 2 dias após a passagem.
2. Transfecção celular
NOTA: Não é recomendável transfetar a cultura celular se a confluência alcançada for inferior a 80%.
- Antes da transfecção, certifique-se se as células estão livres de contaminação e em uma confluência adequada para transfecções transitórias.
- Para cada amostra de transfecção, prepare os complexos DNA-Polietilenimina (PEI 1 μg/μL no pH 7.2) da seguinte forma:
- Diluir 3 μg de cada plasmídeo em 100 μL de opti-MEM reduzi o meio de soro sem suplementação e misture-o suavemente, pipetando a solução para cima e para baixo.
NOTA: Aqui, as células foram transfeinadas com 4 μg de cada plasmídeo: Vetor vazio (pcDNA3) ou construções FLAG-Fbxo7 e UXT-V2-HA, com ou sem 6xHis-myc-ubiquitin. O teor total de DNA foi de 12 μg. - Descongele o PEI na RT e adicione-o à solução seguindo uma proporção de 3 μL de PEI por 1 μg de DNA. Homogeneize a solução se escoando para cima e para baixo. Em seguida, incuba-lo por 15 min na RT para permitir a formação de complexos de DNA-PEI.
NOTA: A proporção ideal do volume de PEI por quantidade de DNA difere de acordo com a linha celular selecionada.
- Diluir 3 μg de cada plasmídeo em 100 μL de opti-MEM reduzi o meio de soro sem suplementação e misture-o suavemente, pipetando a solução para cima e para baixo.
- Adicione o volume total de complexos de DNA-PEI a cada prato contendo a cultura celular e misture-o suavemente balançando a placa para frente e para trás. Incubar as células a 37 °C em uma incubadora de cultura celular umidificada a 5% de CO2.
- Substitua o meio de crescimento após 5h, uma vez que a exposição prolongada ao PEI pode ser tóxica para as células HEK293T. Incubar as células a 37 °C em uma incubadora de cultura celular umidificada a 5% de CO2 por 36 h.
3. Lise celular e imunoprecipitação
- Após o período de incubação e 6h antes da lise celular, trate as células transfeinadas com 10 μM do inibidor proteasome MG-132. Mais uma vez, incubar as células a 37 °C em uma incubadora de cultura celular umidificada a 5% de CO2.
- Aspire a mídia de cada prato de cultura usando uma pipeta sorológica e lave-a uma vez com 1 mL de 1x PBS. Retire as células adicionando 1 mL de trippsina e incubando o prato a 37 °C por 5 min. Resuspense as células em 1 mL de mídia de crescimento.
- Transfira a suspensão da célula para um tubo fresco e limpo de 15 mL e centrífuga a 500 x g por 5 min no RT.
- Remova o supernatante derramando-o com cuidado. Resuspenque suavemente a pelota de célula em 200 μL de tampão de lise NP-40 gelado (50 mM Tris-HCl pH 7.2, 225 mM KCl, e 1% NP-40), complementado com o coquetel de inibidores de protease e fosfatase (10 mM NaF e 1 mM Na3VO4), e transfira a solução para um microtubo limpo de 1,5 mL.
- Incubar a célula lysate por 30 minutos no gelo. Após a incubação, centrifugar as células lísceus a 16.900 x g para 20 min a 4 °C.
- Enquanto isso, equilibre as contas agarose-anti-HA com o tampão de lise NP-40 gelado. Use 15 μL de contas agarose-anti-HA para cada amostra. Lave as contas com 200 μL de tampão de lise NP-40 pulsando-as em um tubo de microcentrífuga a 3.000 x g por 1 min a 4 °C. Com uma pipeta, aspire e descarte o supernatante com muito cuidado; repetir este processo três vezes. Depois, mantenha as contas equilibradas no gelo até o uso.
- Depois de centrifugar as células, recupere o supernatante. Quantifique o teor de proteína no total do lysate utilizando o método Bradford16.
- Certifique-se de que cada amostra submetida à imunoprecipitação apresenta uma quantidade igual de proteína. Incubar o volume necessário de lise celular com as contas agarose-anti-HA equilibradas por 4h, girando suavemente em uma incubadora rotativa a 4 °C, o que permite que o UXT-V2-HA se ligue às contas agarose-anti-HA.
- Colete as contas agarose-anti-HA pulsando-as em um tubo de microcentrifuuge a 3.000 x g por 1 min a 4 °C. Aspire cuidadosamente e descarte o supernatante. Lave as contas três vezes com tampão de lise celular NP-40 gelado e duas vezes com tampão FLAG/HA gelado (10 mM Hepes pH 7.9, 15 mM MgCl2, 225 mM KCl e 0,1% NP-40).
- Após a lavagem final, remova todo o supernatante cuidadosamente usando uma pipeta e elute a proteína poliubiquitylated com peptídeo HA (300 μg/mL) diluído no tampão FLAG/HA. Incubar as contas agarose-anti-HA com peptídeo HA por 1 h a 4 °C em uma plataforma de shaker de balanço.
- Gire as contas a 3.000 x g por 2 min a 4 °C e pipeta cuidadosamente o supernatante contendo as proteínas polibiquitinadas. Se necessário, armazene o eluato em um microtube fresco e limpo a -20 °C.
- Resolva os eluatos e os lises celulares em 10% SDS-PAGE (sulfato de sódio-poliacrilamida eletroforese de gel de poliacrilamida)17 e imunoblotação.
- Neste estudo, foi realizada uma transferência molhada de WB. Para este tipo de transferência, coloque o gel em um sanduíche de transferência composto de papel filtro de papel-gel-papel-filtro-filtro, amorteça-o com almofadas e pressione-o junto por uma grade de suporte. Coloque este sistema verticalmente em um tanque cheio de tampão de transferência e entre eletrodos de aço inoxidável/fio de platina. A transferência ocorre por 90 min a 150 V em tampão de transferência molhada (glicina 192 mM, tris-base 25 mM, 0,025% SDS, 20% metanol).
NOTA: Uma vez que os extratos celulares foram quantificados (etapa 3.7), execute uma SDS-PAGE com uma quantidade igual de proteína para cada amostra. Além disso, para resolver o elunato, execute volumes iguais de cada amostra. - Teste a membrana do imunobloto usando anticorpos selecionados 11. Certifique-se de que um sinal de difamação seja detectado no eluato do substrato poliinubitado puxado no processo IP usando anticorpo anti-mímico nas amostras contendo a liga ligase E3 tipo selvagem, o substrato e o myc-ub. No liseto celular (entrada), certifique-se de que o sinal do substrato escolhido, a proteína Fbxo7, proteínas ubiquizadas e uma proteína de limpeza (por exemplo, GAPDH e β-actin) são detectados para garantir a mesma quantidade de proteínas em cada pista.
NOTA: A diluição de cada anticorpo utilizado foi preparada de acordo com as instruções do fabricante.
Resultados
UXT (transcrição onipresentemente expressa) é uma proteína pré-dobrada que forma complexos onipresentemente expressos de dobra de proteínas em tecidos humanos e camundongos, como coração, cérebro, músculo esquelético, placenta, pâncreas, rim e fígado18. Dois isóformes de emenda do UXT, que são chamados UXT-V1 e UXT-V2, foram descritos executando funções distintas e locais subcelulares. O UXT-V1 é predominantemente localizado no citoplasma e dentro das mitocôndrias, e está impli...
Discussão
A ubiquização é uma modificação pós-translacional essencial que regula os níveis de várias proteínas e desempenha um papel crucial em muitas vias de sinalização e processos biológicos, garantindo um ambiente intracelular saudável. O sistema ubiquitina-proteasome (UPS) é um dos principais focos de pesquisas farmacêuticas recentes, proporcionando a possibilidade de estabilizar supressores tumorais ou induzir a degradação de produtos oncogênicos22. Por exemplo, a proliferação aber...
Divulgações
Os autores declaram que não há conflito de interesses.
Agradecimentos
O F.R.T é apoiado pelo número de bolsas da FAPESP nº 2020/15771-6 e cnpq universal 405836/2018-0. P.M.S.P e V.S são apoiados pela CAPES. C.R.S.T.B.C foi apoiado pela bolsa de estudos da FAPESP número 2019/23466-1. Agradecemos a Sandra R. C. Maruyama (FAPESP 2016/20258-0) pelo apoio material.
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
Referências
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados