Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Оценка убиквитилирования субстрата убиквитин-лигазой E3 в лизатах клеток млекопитающих
В этой статье
Резюме
Мы предоставляем подробный протокол для анализа убиквитилирования конкретного субстрата и убиквитин-лигазы E3 в клетках млекопитающих. Клеточные линии HEK293T использовали для гиперэкспрессии белка, полиубиквитилированный субстрат очищали от клеточных лизатов иммунопреципитацией и разрешали в SDS-PAGE. Иммуноблоттинг использовался для визуализации этой посттрансляционной модификации.
Аннотация
Убиквитилирование - это посттрансляционная модификация, которая происходит в эукариотических клетках, что имеет решающее значение для регуляции нескольких биологических путей, включая выживание клеток, пролиферацию и дифференцировку. Это обратимый процесс, который состоит из ковалентного присоединения убиквитина к субстрату посредством каскадной реакции по меньшей мере трех различных ферментов, состоящих из E1 (фермент активации убиквитина), E2 (убиквитин-конъюгирующий фермент) и E3 (фермент убиквитин-лигаза). Комплекс E3 играет важную роль в распознавании субстрата и убиквитилировании. Здесь описан протокол для оценки убиквитилирования субстрата в клетках млекопитающих с использованием транзиторной котрансфекции плазмиды, кодирующей выбранный субстрат, убиквитин-лигазы E3 и меченого убиквитина. Перед лизисом трансфектированные клетки обрабатывают ингибитором протеасомы MG132 (карбобензокси-лей-лей-лей-лейцинальный), чтобы избежать субстратной протеасомальной деградации. Кроме того, клеточный экстракт подвергают мелкомасштабному иммунопреципитации (IP) для очистки полиубиквитилированного субстрата для последующего обнаружения методом западного блоттинга (WB) с использованием специфических антител для метки убиквитина. Следовательно, описан последовательный и несложный протокол анализа убиквитилирования в клетках млекопитающих, чтобы помочь ученым в решении проблемы убиквитилирования конкретных субстратов и убиквитиновых лигазов E3.
Введение
Посттрансляционные модификации (PTM) являются важным механизмом регуляции белка, который необходим для клеточного гомеостаза. Убиквитилирование белка представляет собой динамическую и сложную модификацию, которая создает набор различных сигналов, приводящих к нескольким клеточным результатам у эукариотических организмов. Убиквитилирование представляет собой обратимый процесс, заключающийся в прикреплении к субстрату белка убиквитина, содержащего 76 аминокислот, происходящий в ферментативном каскаде, состоящем из трех различных реакций1. Первая стадия характеризуется активацией убиквитина, которая зависит от гидролиза АТФ с образованием высокоэнергетического тиоэфир-связанного убиквитина между Убиквитином С-концом и остатком цистеина, присутствующим в активном центре фермента Е1. Впоследствии убиквитин переносится на фермент Е2, образуя тиоэфироподобный комплекс с убиквитином. После этого убиквитин ковалентно присоединяется к субстрату Е2 или, чаще, ферментом Е3, который распознает субстрат 2,3 и взаимодействует с ним. Иногда ферменты E4 (факторы удлинения убиквитиновой цепи) необходимы для стимулирования сборки мультиубиквитиновой цепи3.
Убиквитин имеет семь остатков лизина (K6, K11, K27, K29, K33, K48 и K63), что позволяет формировать полиубиквитиновые цепи, которые генерируют различные связи для получения различных трехмерных структур, которые будут распознаваться несколькими эффекторными белками 4,5. Следовательно, вид полиубиквитиновой цепи, введенный в субстрат, необходим для решения его клеточной судьбы 6,7,8. Кроме того, субстрат также может быть убиквитинирован через его N-концевые остатки, называемые N-дегронами. Специфические убиквитин-лигазы E3 отвечают за распознавание N-дегронов, что позволяет полиубиквитилировать близлежащий остаток лизина9.
В настоящее время существует более 40 различных субстратов, специфичных для SCF. Среди них ключевые регуляторы нескольких биологических путей, включая дифференцировку и развитие клеток, а также выживание и гибель клеток, можно найти 10,11,12,13. Таким образом, идентификация специфических субстратов каждой Е3 убиквитин-лигазы имеет важное значение для разработки всеобъемлющей карты различных биологических событий. Несмотря на то, что идентификация истинных субстратов является биохимически сложной задачей, использование методов, основанных на биохимии, очень подходит для оценки специфичности цепи и различия между моно- и полиубиквитилированием14. Это исследование описывает полный протокол анализа убиквитилирования с использованием клеточной линии млекопитающих HEK293T, сверхэкспрессирующей субстрат UXT-V2 (повсеместно экспрессируемый префолдиноподобный изоформа шаперона 2) с помощью E3 убиквитин-лигазного комплекса SCF (Fbxo7). UXT-V2 является важным кофактором для передачи сигналов NF-κB, и как только этот белок сбивается в клетках, он ингибирует активацию NF-κB, индуцированную TNF-α11. Таким образом, для обнаружения полиубиквитилированного UXT-V2 используется ингибитор протеасомы MG132, поскольку он обладает способностью блокировать протеолитическую активность субъединицы 26S протеасомного комплекса15. Кроме того, клеточный экстракт подают на мелкомасштабный IP для очистки субстрата, используя специфическое антитело, иммобилизованное в агарозную смолу для последующего обнаружения WB с использованием выбранных антител. Этот протокол очень полезен для проверки убиквитилирования субстрата в клеточной среде, а также может быть адаптирован для различных типов клеток млекопитающих и других комплексов убиквитин-лигазы E3. Тем не менее, необходимо также проверить субстрат, протестированный с помощью анализа убиквитилирования in vitro, поскольку оба протокола дополняют друг друга в отношении идентификации истинных субстратов.
протокол
ПРИМЕЧАНИЕ: Обзор протокола анализа убиквитилирования в клетках млекопитающих представлен на рисунке 1.
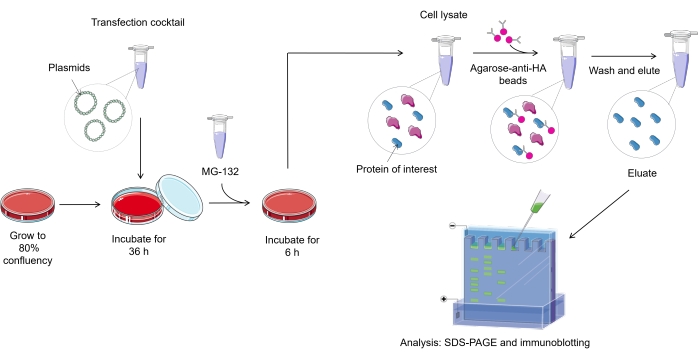
Рисунок 1. Обзор процедуры анализа убиквитилирования. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
1. Клеточная культура
- Вырастите клеточную линию HEK293T в 100-миллиметровой чашке, обработанной TC, до 80%-90% слияния в питательных средах (Dulbecco's Modified Eagle's Medium (DMEM) с высоким содержанием глюкозы, дополненной 10% фетальной бычьей сывороткой (FBS) и пенициллином (100 единиц), стрептомицином (100 мкг) и L-глутамином (0,292 мг / мл)). Инкубируют культуру при 37 °C в увлажненном инкубаторе клеточной культуры при 5% CO2.
- Для прохождения клеток аспирируйте среду из чашки для культивирования с помощью серологической пипетки. Промыть клетки один раз 1 мл стерилизованного 1x фосфатно-буферного физиологического раствора (PBS 1x).
- Отделите клетки, добавив 1 мл раствора трипсина/ЭДТА (этилендиаминтетрауксусной кислоты). Инкубировать блюдо при 37 °C в течение 5 мин. Повторное суспендирование клеток с помощью серологической пипетки в 2 мл питательной среды.
- Переложить клеточную суспензию в свежую и чистую 15 мл пробирку и центрифугу при 500 х г в течение 5 мин при комнатной температуре (RT). Удалите супернатант, тщательно вылив его. Осторожно повторно суспендируют клеточную гранулу в 3 мл питательной среды путем пипетки вверх и вниз для получения однородной клеточной суспензии.
- Переложите 1 мл клеточной суспензии в 100-миллиметровую культуральную чашку, обработанную TC, содержащую 9 мл питательной среды.
ПРИМЕЧАНИЕ: Если клетки находятся в хорошем состоянии, каждая культуральная чашка HEK293T, где слияние составляет 80%-90%, может генерировать три культуральные чашки с 80% слиянием через 2 дня после прохождения.
2. Трансфекция клеток
ПРИМЕЧАНИЕ: Не рекомендуется трансфектировать культуру клеток, если достигнутое слияние составляет менее 80%.
- Перед трансфекцией сертифицируйте, свободны ли клетки от загрязнения и при достаточном слиянии для переходных трансфекций.
- Для каждого образца трансфекции готовят комплексы ДНК-полиэтиленимин (PEI 1 мкг/мкл при рН 7,2) следующим образом:
- Развести 3 мкг каждой плазмиды в 100 мкл восстановленной сывороточной среды opti-MEM I без добавок и осторожно перемешать ее, пипетируя раствор вверх и вниз.
ПРИМЕЧАНИЕ: Здесь клетки были трансфектированы 4 мкг каждой плазмиды: пустый вектор (pcDNA3) или конструкции FLAG-Fbxo7 и UXT-V2-HA, с или без 6xHis-myc-ubiquitin. Общее содержание ДНК составило 12 мкг. - Разморозьте PEI при RT и добавьте его в раствор после пропорции 3 мкл PEI на 1 мкг ДНК. Гомогенизируйте раствор путем пипетирования вверх и вниз. Затем инкубируют его в течение 15 мин на RT, чтобы обеспечить образование комплексов ДНК-PEI.
ПРИМЕЧАНИЕ: Оптимальная пропорция объема PEI на количество ДНК различается в зависимости от выбранной клеточной линии.
- Развести 3 мкг каждой плазмиды в 100 мкл восстановленной сывороточной среды opti-MEM I без добавок и осторожно перемешать ее, пипетируя раствор вверх и вниз.
- Добавьте общий объем комплексов ДНК-PEI в каждое блюдо, содержащее культуру клеток, и аккуратно перемешайте его, раскачивая пластину вперед и назад. Инкубируют клетки при 37 °C в увлажненном инкубаторе клеточной культуры при 5% CO2.
- Замените питательную среду через 5 ч, так как длительное воздействие PEI может быть токсичным для клеток HEK293T. Инкубируют клетки при 37 °C в увлажненном инкубаторе клеточной культуры при 5% CO2 в течение 36 ч.
3. Лизис и иммунопреципитация клеток
- После инкубационного периода и за 6 ч до лизиса клеток обрабатывают трансфектированные клетки 10 мкМ ингибитора протеасомы MG-132. Еще раз, инкубируйте клетки при 37 ° C в увлажненном инкубаторе клеточной культуры при 5% CO2.
- Аспирируйте среду из каждой чашки культуры с помощью серологической пипетки и вымойте ее один раз 1 мл 1x PBS. Отсоедините клетки, добавив 1 мл трипсина и инкубируя блюдо при 37 °C в течение 5 мин. Повторное суспендирование клеток в 1 мл питательной среды.
- Переложите клеточную суспензию в свежую и чистую пробирку объемом 15 мл и центрифугируйте ее при 500 х г в течение 5 мин при РТ.
- Удалите супернатант, тщательно вылив его. Осторожно повторно суспендируют клеточную гранулу в 200 мкл ледяного буфера лизиса NP-40 (50 мМ Tris-HCl pH 7,2, 225 мМ KCl и 1% NP-40), дополняют коктейлем ингибиторов протеазы и фосфатазы (10 мМ NaF и 1 мМ Na3VO4), и переносят раствор в чистую микротрубку объемом 1,5 мл.
- Инкубируйте лизат клеток в течение 30 мин на льду. После инкубации центрифугируют клеточные лизаты при 16 900 х г в течение 20 мин при 4 °C.
- Между тем, уравновешивайте бусины агарозы-анти-ГК ледяным буфером лизиса NP-40. Используйте 15 мкл шариков агарозы-анти-ГК для каждого образца. Вымойте шарики 200 мкл буфера лизиса NP-40, пульсируя его в микроцентрифужной трубке при 3000 х г в течение 1 мин при 4 °C. Пипеткой аспирировать и выбросить супернатант очень осторожно; повторите этот процесс три раза. После этого держите бусины уравновешенными на льду до использования.
- После центрифугирования клеточных лизатов восстанавливают супернатант. Количественно оценить содержание белка в общем лизате с помощью метода16 Брэдфорда.
- Убедитесь, что каждый образец, подвергшийся иммунопреципитации, содержит равное количество белка. Инкубируют необходимый объем клеточного лизата с уравновешенными шариками агароза-анти-ГА в течение 4 ч, осторожно вращаясь во вращающемся инкубаторе при 4 °C, что позволяет UXT-V2-HA связываться с шариками агарозы-анти-HA.
- Соберите шарики агарозы-анти-ГК, пульсируя их в микроцентрифужной трубке при 3000 х г в течение 1 мин при 4 °C. Осторожно аспирировать и выбросить супернатант. Трижды промыть шарики ледяным буфером лизиса NP-40 и дважды ледяным буфером FLAG/HA (10 мМ Hepes pH 7,9, 15 мМ MgCl2, 225 мМ KCl и 0,1% NP-40).
- После заключительной промывки аккуратно удалите весь супернатант пипеткой и элюируйте полиубиквитилированный белок пептидом ГК (300 мкг/мл), разбавленным на буфере FLAG/HA. Инкубировать шарики агарозы-анти-ГК с пептидом ГК в течение 1 ч при 4 °C в качающейся шейкерной платформе.
- Раскрутите шарики при 3000 х г в течение 2 мин при 4 °C и осторожно выложите в пипетку супернатант, содержащий полиубиквитинированные белки. При необходимости храните элюат в свежей и чистой микропробирке при -20 °C.
- Растворяют элюаты и лизаты клеток в 10% SDS-PAGE (додецилсульфат-полиакриламидный гель электрофорез)17 и иммуноблоттинг.
- В этом исследовании был выполнен мокрый перенос WB. Для такого рода переноса поместите гель в бутерброд, состоящий из фильтровальной бумаги-геля-мембраны-фильтровальной бумаги, смягчите его прокладками и прижмите его вместе поддерживающей сеткой. Поместите эту систему вертикально в резервуар, заполненный передаточным буфером, и между электродами из нержавеющей стали / платиновой проволоки. Перенос происходит в течение 90 мин при 150 В во влажном буфере переноса (глицин 192 мМ, трисосное основание 25 мМ, 0,025% SDS, 20% метанол).
ПРИМЕЧАНИЕ: Поскольку клеточные экстракты были количественно определены (шаг 3.7), запустите SDS-PAGE с равным количеством белка для каждого образца. Кроме того, чтобы разрешить элюат, запустите равные объемы каждого образца. - Исследуйте мембрану иммуноблота, используя выбранные антитела 11. Убедитесь, что сигнал мазка обнаружен в элюате из полиубиквитилированного субстрата, вытягиваемого в процессе IP, с использованием антимик-антител в образцах, содержащих лигазу Е3 дикого типа, субстрат и мик-уб. В клеточном лизате (входе) убедитесь, что сигнал от выбранного субстрата, белка Fbxo7, убиквитилированных белков и белка для ведения хозяйства (например, GAPDH и β-актина) обнаружен, чтобы гарантировать одинаковое количество белков в каждой полосе.
ПРИМЕЧАНИЕ: Разведение для каждого использованного антитела готовили в соответствии с инструкциями завода-изготовителя.
Результаты
UXT (вездесущий экспрессированный транскрипт) представляет собой префоллиноподобный белок, который образует повсеместно экспрессируемые белковые складчатые комплексы в тканях мыши и человека, таких как сердце, мозг, скелетные мышцы, плацента, поджелудочная железа, почки и печень
Обсуждение
Убиквитилирование является важной посттрансляционной модификацией, которая регулирует уровни нескольких белков и играет решающую роль во многих сигнальных путях и биологических процессах, обеспечивая здоровую внутриклеточную среду. Убиквитин-протеасомная система (UPS) является одни...
Раскрытие информации
Авторы заявляют, что конфликта интересов нет.
Благодарности
F.R.T поддерживается грантом FAPESP No 2020/15771-6 и CNPq Universal 405836/2018-0. P.M.S.P и V.S поддерживаются CAPES. C.R.S.T.B.C был поддержан стипендией FAPESP No 2019/23466-1. Благодарим Сандру Р. К. Маруяму (FAPESP 2016/20258-0) за материальную поддержку.
Материалы
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
Ссылки
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены