Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Evaluación de la ubiquitilación del sustrato por E3 Ubiquitina-ligasa en lisados de células de mamíferos
En este artículo
Resumen
Proporcionamos un protocolo detallado para un ensayo de ubiquitilación de un sustrato específico y una ubiquitina ligasa E3 en células de mamíferos. Se utilizaron líneas celulares HEK293T para la sobreexpresión de proteínas, el sustrato poliubiquitilado se purificó a partir de lisados celulares por inmunoprecipitación y se resolvió en SDS-PAGE. Se utilizó immunoblotting para visualizar esta modificación post-traduccional.
Resumen
La ubiquitilación es una modificación post-traduccional que ocurre en las células eucariotas que es crítica para la regulación de varias vías biológicas, incluida la supervivencia, proliferación y diferenciación celular. Es un proceso reversible que consiste en una unión covalente de ubiquitina al sustrato a través de una reacción en cascada de al menos tres enzimas diferentes, compuestas de E1 (enzima de activación de ubiquitina), E2 (enzima conjugante de ubiquitina) y E3 (enzima ubiquitina-ligasa). El complejo E3 juega un papel importante en el reconocimiento y ubiquitilación del sustrato. Aquí, se describe un protocolo para evaluar la ubiquitilación del sustrato en células de mamíferos utilizando la coinfección transitoria de un plásmido que codifica el sustrato seleccionado, una ubiquitina ligasa E3 y una ubiquitina etiquetada. Antes de la lisis, las células transfectadas se tratan con el inhibidor del proteasoma MG132 (carbobenzoxy-leu-leu-leucinal) para evitar la degradación proteasómica del sustrato. Además, el extracto celular se somete a inmunoprecipitación a pequeña escala (IP) para purificar el sustrato poliubiquitilado para su posterior detección por western blotting (WB) utilizando anticuerpos específicos para la etiqueta de ubiquitina. Por lo tanto, se describe un protocolo consistente y sin complicaciones para el ensayo de ubiquitilación en células de mamíferos para ayudar a los científicos a abordar la ubiquitilación de sustratos específicos y las ligasas de ubiquitina E3.
Introducción
Las modificaciones post-traduccionales (PTM) son un mecanismo importante con respecto a la regulación de proteínas, que es esencial para la homeostasis celular. La ubiquitilación de proteínas es una modificación dinámica e intrincada que crea una variedad de señales diferentes que resultan en varios resultados celulares en organismos eucariotas. La ubiquitilación es un proceso reversible que consiste en la unión de una proteína de ubiquitina que contiene 76 aminoácidos al sustrato, que se produce en una cascada enzimática compuesta por tres reacciones distintas1. El primer paso se caracteriza por la activación de la ubiquitina, que depende de una hidrólisis de ATP para formar una ubiquitina ligada al tioéster de alta energía entre la ubiquitina C-terminal y el residuo de cisteína presente en el sitio activo de la enzima E1. Posteriormente, la ubiquitina se transfiere a la enzima E2 formando un complejo similar al tioéster con la ubiquitina. Después, la ubiquitina se une covalentemente al sustrato por la E2, o más a menudo, por la enzima E3, que reconoce e interactúa con el sustrato 2,3. Ocasionalmente, las enzimas E4 (factores de elongación de la cadena de ubiquitina) son necesarias para promover el ensamblaje de la cadena de multiubiquitina3.
La ubiquitina tiene siete residuos de lisina (K6, K11, K27, K29, K33, K48 y K63), lo que permite la formación de cadenas de poliubiquitina que generan enlaces distintos para producir diferentes estructuras tridimensionales que van a ser reconocidas por varias proteínas efectoras 4,5. Por lo tanto, el tipo de cadena de poliubiquitina introducida en el sustrato es esencial para decidir su destino celular 6,7,8. Además, el sustrato también podría ser ubiquitinado a través de sus residuos N-terminales llamados N-degrons. Las ubiquitinas-ligasas específicas E3 son responsables del reconocimiento de N-degron, lo que permite la poliubiquitilación del residuo de lisina cercano9.
Hoy en día, hay más de 40 sustratos diferentes específicos de SCF caracterizados. Entre ellos, se pueden encontrar reguladores clave de varias vías biológicas, incluida la diferenciación y el desarrollo celular, así como la supervivencia y la muerte celular, 10,11,12,13. Por lo tanto, la identificación de sustratos específicos de cada ubiquitina-ligasa E3 es esencial para diseñar un mapa completo de diversos eventos biológicos. A pesar de que la identificación de sustratos verdaderos es bioquímicamente desafiante, el uso de métodos basados en la bioquímica es muy adecuado para evaluar la especificidad de la cadena y la distinción entre mono y poliubiquitilación14. Este estudio describe un protocolo completo para el ensayo de ubiquitilación utilizando la línea celular de mamíferos HEK293T que sobreexpresa el sustrato UXT-V2 (isoforma 2 de chaperona similar a la prefoldina expresada ubicuamente) con el complejo E3 ubiquitina-ligasa SCF (Fbxo7). UXT-V2 es un cofactor esencial para la señalización de NF-κB, y una vez que esta proteína es derribada en las células, inhibe la activación de NF-κB inducida por TNF-α11. Así, para detectar UXT-V2 poliubiquitilado, se utiliza el inhibidor del proteasoma MG132 ya que tiene la capacidad de bloquear la actividad proteolítica de la subunidad 26S del complejo proteasoma15. Además, el extracto celular se somete a una IP a pequeña escala para purificar el sustrato, utilizando un anticuerpo específico inmovilizado a resina de agarosa para su posterior detección por WB utilizando anticuerpos seleccionados. Este protocolo es muy útil para validar la ubiquitilación del sustrato en el entorno celular, y también se puede adaptar para diferentes tipos de células de mamíferos y otros complejos de ubiquitina-ligasa E3. Sin embargo, es necesario validar el sustrato probado a través de un ensayo de ubiquitilación in vitro también, ya que ambos protocolos se complementan entre sí en cuanto a la identificación de sustratos verdaderos.
Protocolo
NOTA: En la Figura 1 se representa una descripción general del protocolo de ensayo de ubiquitilación en células de mamíferos.
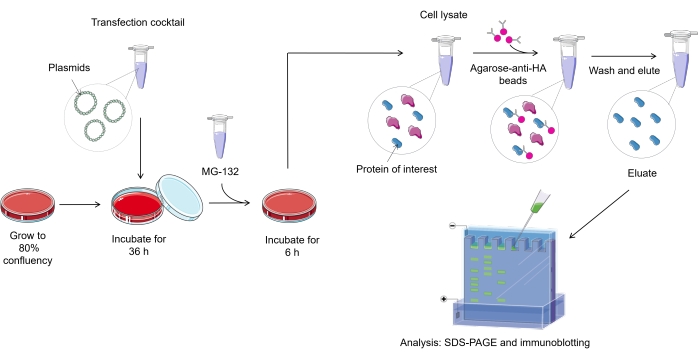
Figura 1. Descripción general del procedimiento de ensayo de ubiquitilación. Haga clic aquí para ver una versión más grande de esta figura.
1. Cultivo celular
- Cultivar la línea celular HEK293T en una placa de cultivo tratada con TC de 100 mm a una confluencia del 80%-90% en los medios de crecimiento (Dulbecco's Modified Eagle's Medium (DMEM) alta glucosa suplementada con 10% de suero fetal bovino (FBS) y penicilina (100 unidades), estreptomicina (100 μg) y L-glutamina (0,292 mg/ml)). Incubar el cultivo a 37 °C en una incubadora de cultivo celular humidificado al 5% de CO2.
- Para pasar las células, aspire los medios de la placa de cultivo utilizando una pipeta serológica. Lave las células una vez con 1 ml de solución salina esterilizada 1x tamponada con fosfato (PBS 1x).
- Separe las células agregando 1 ml de solución de tripsina/EDTA (ácido etilendiaminotetraacético). Incubar el plato a 37 °C durante 5 min. Resuspend las células utilizando una pipeta serológica en 2 ml de medios de crecimiento.
- Transfiera la suspensión celular a un tubo fresco y limpio de 15 ml y centrífuga a 500 x g durante 5 min a temperatura ambiente (RT). Retire el sobrenadante vertiéndolo con cuidado. Resusped suavemente el pellet celular en 3 ml de medios de crecimiento mediante pipeteo hacia arriba y hacia abajo para obtener una suspensión celular homogénea.
- Transfiera 1 ml de la suspensión celular a una placa de cultivo tratada con TC de 100 mm que contenga 9 ml de medios de crecimiento.
NOTA: Si las células están en buenas condiciones, cada plato de cultivo de HEK293T, en el que la confluencia es del 80%-90%, puede generar tres platos de cultivo con 80% de confluencia 2 días después del paso.
2. Transfección celular
NOTA: No se recomienda transfectar el cultivo celular si la confluencia alcanzada es inferior al 80%.
- Antes de la transfección, certifique si las células están libres de contaminación y en una confluencia adecuada para las transfecciones transitorias.
- Para cada muestra de transfección, prepare los complejos ADN-Polietilenimina (PEI 1 μg/μL a pH 7.2) de la siguiente manera:
- Diluir 3 μg de cada plásmido en 100 μL de medio sérico reducido opti-MEM I sin suplementación y mezclarlo suavemente pipeteando la solución hacia arriba y hacia abajo.
NOTA: Aquí, las células fueron transfectadas con 4 μg de cada plásmido: Vector vacío (pcDNA3) o FLAG-Fbxo7 constructos y UXT-V2-HA, con o sin 6xHis-myc-ubiquitin. El contenido total de ADN fue de 12 μg. - Descongele el PEI en RT y agréguelo a la solución siguiendo una proporción de 3 μL de PEI por 1 μg de ADN. Homogeneizar la solución mediante pipeteo hacia arriba y hacia abajo. Luego incubarlo durante 15 min en RT para permitir la formación de complejos ADN-PEI.
NOTA: La proporción óptima de volumen de PEI por cantidad de ADN difiere según la línea celular seleccionada.
- Diluir 3 μg de cada plásmido en 100 μL de medio sérico reducido opti-MEM I sin suplementación y mezclarlo suavemente pipeteando la solución hacia arriba y hacia abajo.
- Agregue el volumen total de complejos de ADN-PEI a cada plato que contenga el cultivo celular y mézclelo suavemente balanceando la placa hacia adelante y hacia atrás. Incubar las células a 37 °C en una incubadora de cultivo celular humidificado al 5% de CO2.
- Reemplace el medio de crecimiento después de 5 h, ya que la exposición prolongada a PEI puede ser tóxica para las células HEK293T. Incubar las células a 37 °C en una incubadora de cultivo celular humidificado al 5% de CO2 durante 36 h.
3. Lisis celular e inmunoprecipitación
- Después del período de incubación y 6 h antes de la lisis celular, tratar las células transfectadas con 10 μM del inhibidor del proteasoma MG-132. Una vez más, incubar las células a 37 °C en una incubadora de cultivo celular humidificado al 5% de CO2.
- Aspirar el medio de cada plato de cultivo utilizando una pipeta serológica y lavarlo una vez con 1 mL de 1x PBS. Separe las células añadiendo 1 ml de tripsina e incubando el plato a 37 °C durante 5 min. Resuspend las células en 1 mL de medios de crecimiento.
- Transfiera la suspensión celular a un tubo fresco y limpio de 15 ml y centrífuga a 500 x g durante 5 minutos a RT.
- Retire el sobrenadante vertiéndolo con cuidado. Resuspender suavemente el pellet celular en 200 μL de tampón de lisis NP-40 helado (50 mM Tris-HCl pH 7.2, 225 mM KCl y 1% NP-40), complementado con el cóctel de inhibidores de proteasa y fosfatasa (10 mM NaF y 1 mM Na3VO4), y transferir la solución a un microtubo limpio de 1.5 mL.
- Incubar el lisado celular durante 30 min sobre hielo. Después de la incubación, centrifugar los lisados celulares a 16.900 x g durante 20 min a 4 °C.
- Mientras tanto, equilibre las perlas de agarosa-anti-HA con un tampón de lisis NP-40 helado. Utilice 15 μL de perlas de agarosa-anti-HA para cada muestra. Lavar las perlas con 200 μL de tampón de lisis NP-40 pulsiéndolo en un tubo de microcentrífuga a 3.000 x g durante 1 min a 4 °C. Con una pipeta, aspira y desecha el sobrenadante con mucho cuidado; repita este proceso tres veces. Después, mantenga las cuentas equilibradas en hielo hasta su uso.
- Después de centrifugar los lisados celulares, recupere el sobrenadante. Cuantificar el contenido de proteínas en el lisado total utilizando el método de Bradford16.
- Asegurar que cada muestra sometida a inmunoprecipitación presente una cantidad igual de proteína. Incubar el volumen necesario de lisado celular con las perlas equilibradas de agarosa-anti-HA durante 4 h, girando suavemente en una incubadora giratoria a 4 °C, lo que permite que el UXT-V2-HA se una a las perlas de agarosa-anti-HA.
- Recoge las perlas de agarosa-anti-HA pulsándolas en un tubo de microcentrífuga a 3.000 x g durante 1 min a 4 °C. Aspira con cuidado y desecha el sobrenadante. Lave las perlas tres veces con tampón de lisis de células NP-40 helado y dos veces con tampón FLAG/HA helado (10 mM Hepes pH 7.9, 15 mM MgCl2, 225 mM KCl y 0.1% NP-40).
- Después del lavado final, retire todo el sobrenadante con cuidado con una pipeta y eluya la proteína poliubiquitilada con péptido HA (300 μg / ml) diluido en tampón FLAG / HA. Incubar las perlas de agarosa-anti-HA con péptido HA durante 1 h a 4 °C en una plataforma agitadora de balanceo.
- Girar las perlas a 3.000 x g durante 2 min a 4 °C y pipetear cuidadosamente el sobrenadante que contiene las proteínas poliubiquitinadas. Si es necesario, guarde el eluido en un microtubo fresco y limpio a -20 °C.
- Resolver los eluidos y los lisados celulares en SDS-PAGE al 10% (electroforesis en gel de dodecil sulfato de sodio-poliacrilamida)17 e inmunoblotting.
- En este estudio, se realizó una transferencia húmeda wb. Para este tipo de transferencia, coloque el gel en un sándwich de transferencia compuesto de papel de filtro-gel-membrana-papel de filtro, acolcharlo con almohadillas y presionarlo junto con una rejilla de soporte. Coloque este sistema verticalmente en un tanque lleno de tampón de transferencia y entre electrodos de alambre de acero inoxidable / platino. La transferencia ocurre durante 90 min a 150 V en tampón de transferencia húmeda (glicina 192 mM, tris-base 25 mM, 0.025% SDS, 20% metanol).
NOTA: Dado que los extractos celulares fueron cuantificados (paso 3.7), ejecute un SDS-PAGE con una cantidad igual de proteína para cada muestra. Además, para resolver el eluido, ejecute volúmenes iguales de cada muestra. - Sondear la membrana de immunoblot utilizando anticuerpos seleccionados 11. Asegúrese de que se detecta una señal de frotis en el eluido del sustrato poliubiquitilado extraído en el proceso IP mediante el uso de anticuerpos anti-myc en las muestras que contienen la ligasa E3 de tipo salvaje, el sustrato y myc-ub. En el lisado celular (entrada), asegúrese de que se detecte la señal del sustrato elegido, la proteína Fbxo7, las proteínas ubiquitiladas y una proteína de limpieza (por ejemplo, GAPDH y β-actina) para garantizar la misma cantidad de proteínas en cada carril.
NOTA: La dilución de cada anticuerpo utilizado se preparó de acuerdo con las instrucciones del fabricante.
Resultados
UXT (transcripción expresada ubicuamente) es una proteína similar a la prefoldina que forma complejos de plegamiento de proteínas expresados ubicuamente en tejidos de ratones y humanos como el corazón, el cerebro, el músculo esquelético, la placenta, el páncreas, el riñón y el hígado18. Se han descrito dos isoformas de empalme de UXT, que se denominan UXT-V1 y UXT-V2, que realizan funciones distintas y ubicaciones subcelulares. UXT-V1 se localiza predominantemente en el citoplasma y en e...
Discusión
La ubiquitilación es una modificación post-traduccional esencial que regula los niveles de varias proteínas y desempeña un papel crucial en muchas vías de señalización y procesos biológicos, asegurando un entorno intracelular saludable. El sistema ubiquitina-proteasoma (SAI) es uno de los principales focos de la investigación farmacéutica reciente, proporcionando la posibilidad de estabilizar supresores tumorales o inducir la degradación de productos oncogénicos22. Por ejemplo, la prol...
Divulgaciones
Los autores declaran que no hay conflicto de intereses.
Agradecimientos
F.R.T cuenta con el apoyo de la subvención FAPESP número 2020/15771-6 y CNPq Universal 405836/2018-0. P.M.S.P y V.S son compatibles con CAPES. C.R.S.T.B.C fue apoyado por la beca FAPESP número 2019/23466-1. Agradecemos a Sandra R. C. Maruyama (FAPESP 2016/20258-0) por el apoyo material.
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
Referencias
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados