A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
عزل وتحديد البكتيريا المقاومة للمضادات الحيوية المنقولة بالماء والتوصيف الجزيئي لجيناتها المقاومة للمضادات الحيوية
In This Article
Summary
نقدم هنا بروتوكولا مفصلا لعزل وتحديد البكتيريا المقاومة للمضادات الحيوية من الماء والتوصيف الجزيئي لجيناتها المقاومة للمضادات الحيوية (ARGs). يوفر استخدام التقنيات القائمة على الثقافة وغير القائمة على الثقافة (التحليل الميتاجينومي) معلومات كاملة حول التنوع البكتيري الكلي والمجموعة الإجمالية ل ARGs المختلفة الموجودة في المياه العذبة من مومباي ، الهند.
Abstract
يعد تطور وانتشار مقاومة المضادات الحيوية (AR) من خلال الكائنات الحية الدقيقة المرتبطة بأجسام المياه العذبة مصدر قلق صحي عالمي كبير. في هذه الدراسة ، تم جمع عينات المياه العذبة وتحليلها فيما يتعلق بالتنوع البكتيري الكلي وجينات AR (ARGs) باستخدام كل من التقنيات التقليدية القائمة على الثقافة ونهج metagenomic عالي الإنتاجية المستقل عن الزراعة. يقدم هذا البحث بروتوكولا منهجيا لتعداد البكتيريا الكلية والمقاومة للمضادات الحيوية من عينات المياه العذبة وتحديد مقاومة النمط الظاهري والوراثي في العزلات القابلة للزراعة. علاوة على ذلك ، أبلغنا عن استخدام التحليل الميتاجينومي الكامل للحمض النووي الميتاجينومي الكلي المستخرج من عينة المياه العذبة لتحديد التنوع البكتيري الكلي ، بما في ذلك البكتيريا غير القابلة للزراعة ، وتحديد المجموعة الإجمالية لمختلف ARGs (المقاومة) في الجسم المائي. باتباع هذه البروتوكولات التفصيلية ، لاحظنا حمولة بكتيريا عالية المقاومة للمضادات الحيوية في نطاق 9.6 × 10 5-1.2 × 109 CFU / mL. كانت معظم العزلات مقاومة للمضادات الحيوية المتعددة التي تم اختبارها ، بما في ذلك سيفوتاكسيم ، والأمبيسلين ، والليفوفلوكساسين ، والكلورامفينيكول ، والسيفترياكسون ، والجنتاميسين ، والنيومايسين ، والتريميثوبريم ، وسيبروفلوكساسين ، مع مؤشرات مقاومة متعددة للمضادات الحيوية (MAR) تبلغ ≥0.2 ، مما يشير إلى مستويات عالية من المقاومة في العزلات. حدد تسلسل 16S rRNA مسببات الأمراض البشرية المحتملة ، مثل Klebsiella pneumoniae ، والبكتيريا الانتهازية ، مثل Comamonas spp. ، Micrococcus spp. ، Arthrobacter spp. ، و Aeromonas spp. أظهر التوصيف الجزيئي للعزلات وجود العديد من ARGs ، مثل blaTEM و blaCTX-M (β-lactams) و aadA و aac (6 ') -Ib (أمينوغليكوزيدات) و dfr1 (trimethoprims) ، وهو ما تم تأكيده أيضا من خلال تحليل الحمض النووي الميتاجينومي بأكمله. كما تم الكشف عن ارتفاع معدل انتشار ARGs الأخرى المشفرة لمضخات تدفق المضادات الحيوية - mtrA و macB و mdtA و acrD و β-lactamases-SMB-1 و VIM-20 و ccrA و ampC و blaZ وجين الكلورامفينيكول أسيتيل ترانسفيراز catB10 ، وجين مقاومة الريفامبيسين rphB- في الحمض النووي الميتاجينومي. بمساعدة البروتوكولات التي تمت مناقشتها في هذه الدراسة ، أكدنا وجود بكتيريا MAR المنقولة بالماء مع سمات النمط الظاهري والوراثي AR المتنوعة. وبالتالي ، يمكن استخدام تحليل الحمض النووي الميتاجينومي الكامل كتقنية تكميلية للتقنيات التقليدية القائمة على الثقافة لتحديد الحالة العامة للواقع المعزز لجسم مائي.
Introduction
تم تحديد مقاومة مضادات الميكروبات (AMR) باعتبارها واحدة من أكثر المشاكل العالمية إلحاحا. يعد التطور السريع لمقاومة مضادات الميكروبات وانتشارها في جميع أنحاء العالم أحد أكبر التهديدات لصحة الإنسان والاقتصاد العالمي من حيث التكاليف الصحية المرتبطة بها1. أدى الإفراط في استخدام المضادات الحيوية وإساءة استخدامها إلى زيادة في الواقع المعزز. وقد تم تسليط الضوء على ذلك من خلال جائحة COVID-19 ، حيث تعرض علاج العدوى الثانوية المصاحبة ، في كثير من الحالات ، للخطر بشكل كبير بسبب مقاومة مضادات الميكروبات في المرضى المصابين2. إلى جانب الاستخدام المباشر / إساءة استخدام المضادات الحيوية من قبل البشر ، فإن الإفراط في استخدام المضادات الحيوية وإساءة استخدامها في الزراعة وتربية الحيوانات وتصريفها بشكل غير مناسب في البيئة ، بما في ذلك المسطحات المائية ، هي مصدر قلق كبير3. إن ظهور سمات مقاومة جديدة ومقاومة للأدوية المتعددة في البكتيريا يسلط الضوء بشكل عاجل على الحاجة إلى فهم أفضل للعوامل التي تؤدي إلى تطوير AR ونشره. يمكن للبكتيريا المتعددة المقاومة للمضادات الحيوية ، والتي غالبا ما تحمل جينات AR متعددة (ARGs) على العناصر الجينية المتنقلة مثل البلازميدات ، نقل جينات المقاومة هذه إلى الكائنات الحية الدقيقة غير المقاومة ، بما في ذلك مسببات الأمراض البشرية المحتملة ، مما يؤدي إلى ظهور الجراثيم الخارقة التي لا يمكن علاجها حتى بالمضادات الحيوية كملاذ أخير4. يمكن لهذه البكتيريا المتعددة المقاومة للمضادات الحيوية ، إذا كانت موجودة في النظم الإيكولوجية للمياه ، أن تدخل الأمعاء البشرية مباشرة عن طريق استهلاك الأطعمة الملوثة القائمة على الماء مثل الأسماك وسرطان البحر والرخويات. أظهرت الدراسات السابقة أن انتشار بكتيريا AR في أنظمة المياه التي تحدث بشكل طبيعي يمكن أن يصل أيضا إلى إمدادات المياه الأخرى ، بما في ذلك مياه الشرب ، وبالتالي يمكن أن يدخل السلسلة الغذائية البشرية5،6،7.
الهدف من هذه الدراسة هو توفير بروتوكول شامل باستخدام مزيج من التقنيات القائمة على الثقافة وغير القائمة على الثقافة (التحليل الميتاجينومي الكامل) للحصول على معلومات كاملة حول التنوع البكتيري الكلي والمجموعة الإجمالية لمختلف ARGs الموجودة في جسم مائي في مومباي ، الهند. تقليديا ، تم استخدام التقنيات القائمة على الثقافة لدراسة التنوع البكتيري في المسطحات المائية. نظرا لأن الكائنات الحية الدقيقة القابلة للزراعة لا تشكل سوى نسبة صغيرة من إجمالي الكائنات الحية الدقيقة في أي مكان ، للحصول على فهم أفضل للحالة العامة للتنوع البكتيري والسمات المقاومة المختلفة السائدة في أي عينة ، يجب استخدام تقنيات مختلفة قائمة على الثقافة ومستقلة عن الثقافة جنبا إلى جنب. إحدى هذه التقنيات القوية والموثوقة المستقلة عن الثقافة هي تحليل الحمض النووي الميتاجينومي الكامل. تم استخدام هذه الطريقة عالية الإنتاجية بنجاح في دراسات مختلفة حول التنوع البكتيري أو التعليقات التوضيحية الوظيفية لمختلف ARGs 8,9. تستخدم هذه التقنية الميتاجينوم (المادة الوراثية الكلية في العينة) كمادة أولية لتحليلات مختلفة ، وبالتالي فهي مستقلة عن الثقافة. يمكن استخدام البروتوكولات في هذه الدراسة لتحليل الحمض النووي الميتاجينومي الكامل للحصول على معلومات حول التنوع البكتيري الكلي ومختلف ARGs (المقاومة) في عينات المياه.
Protocol
1. جمع العينات ومعالجتها
- جمع العينات
- جمع الحجم المناسب من عينة المياه في حاوية (حاويات) عينة معقمة ، مع التأكد من عدم ملء أكثر من 3/4 من الحاوية.
- نقل العينات إلى المختبر تحت ظروف معقمة في أسرع وقت ممكن بعد جمعها ومعالجتها على الفور.
- معالجة العينات
- قم بتصفية عينة الماء بشكل معقم من خلال قطعة قماش موسلين معقمة لإزالة أي جسيمات.
- إجراء التخفيفات التسلسلية المناسبة للمياه المفلترة لمزيد من التحليل.
2. تقدير الحمل البكتيري الكلي وعدد البكتيريا المقاومة للمضادات الحيوية
- تحديد الحمل البكتيري الكلي
- 18.12 جم من R2A Agar ، مسحوق معدل في 1000 مل من الماء المقطر المزدوج ، وقم بإذابة الخليط عن طريق التسخين. الأوتوكلاف الخليط المذاب عند 121 درجة مئوية ، 15 رطل / بوصة مربعة لمدة 20 دقيقة. تحضير R2A أجار ، لوحات معدلة عن طريق صب الكمية المناسبة من الخليط المعقم في ألواح بتري معقمة (على سبيل المثال ، أضف ما يقرب من 20 مل من الوسط المعقم المعقم إلى صفيحة بتري معقمة 90 مم).
- انشر بالتساوي 100 ميكرولتر من التخفيفات المناسبة لعينة الماء المفلترة على R2A Agar ، اللوحة المعدلة بمجرد أن يصلب الوسط. قم بإجراء التجربة من نسختين.
- احتضان جميع الألواح المذكورة أعلاه عند 35-37 درجة مئوية لمدة 48 ساعة (تختلف درجة الحرارة ووقت الحضانة حسب الوسائط المستخدمة للعزل).
- عبر عن الحمل البكتيري الكلي بدلالة وحدات تكوين المستعمرة لكل ملليلتر (CFU / mL) باستخدام المعادلة (1):
 (1)
(1)
- تحديد عدد البكتيريا AR
- اتبع الخطوات 2.1.1-2.1.4. ومع ذلك ، بدلا من R2A Agar ، تستخدم الألواح المعدلة R2A ، الألواح المعدلة المكملة بشكل فردي بخمسة مضادات حيوية مختلفة ، وهي سيفوتاكسيم (3 ميكروغرام / مل) ، سيبروفلوكساسين (0.5 ميكروغرام / مل) ، إريثروميسين (20 ميكروغرام / مل) ، كاناميسين (15 ميكروغرام / مل) ، وفانكومايسين (3 ميكروغرام / مل).
- أضف المضادات الحيوية بشكل منفصل إلى أنابيب تحتوي على 20 مل من أجار R2A المنصهر المعقم ، المعدل (مع درجة حرارة أجار R2A المنصهر ، المعدل عند ≤40 درجة مئوية) لتحقيق تركيز المضادات الحيوية النهائي كما هو مذكور في الخطوة 2.2.1.
- قم بالدوران للخلط المتساوي واسكبه على ألواح بتري المعقمة قبل أن يتجمد الآجار. قم بإجراء التجربة من نسختين.
- احتضان جميع الألواح المذكورة أعلاه عند 35-37 درجة مئوية لمدة 48 ساعة (في حالة استخدام وسائط مختلفة ، قد تختلف درجة الحرارة ووقت الحضانة).
- لمراقبة الجودة والتحقق من فعالية المضادات الحيوية ، انشر 100 ميكرولتر من المعلقات البكتيرية لسلالات الإشريكية القولونية ATCC 25922 والمكورات العنقودية الذهبية ATCC 29213 على ألواح R2A Agar المعدلة المحتوية على المضادات الحيوية (تأكد من أن كثافة الثقافة الطازجة المستخدمة للتلقيح هي OD = 0.5 عند 600 نانومتر).
- تحديد عدد البكتيريا المقاومة للمضادات الحيوية من حيث CFU / mL كما هو موضح في الخطوة 2.1.4.
- مخزون الجلسرين من العزلات
- حدد مستعمرات AR المتميزة شكليا.
- تعليق مستعمرة معزولة واحدة في 2 مل من مرق لوريا-بيرتاني المعقم الذي يحتوي على المضاد الحيوي المعني (على سبيل المثال ، إذا تم اختيار مستعمرة من صفيحة تحتوي على سيفوتاكسيم ، فقم بتلقيح المستعمرة من الخطوة 2.3.1 في مرق لوريا بيرتاني المعقم الذي يحتوي على سيفوتاكسيم عند تركيزه).
- احتضان الأنابيب الملقحة عند 37 درجة مئوية عند 80 دورة في الدقيقة حتى يصل OD600 إلى 0.5.
- تحضير مخزون الجلسرين من العزلات عن طريق خلط 750 ميكرولتر من معلق الثقافة من الخطوة 2.3.3 إلى 250 ميكرولتر من الجلسرين المعقم بنسبة 100٪ تحت ظروف معقمة.
- قم بتخزين مخزون الجلسرين عند -80 درجة مئوية حتى مزيد من التحليل.
ملاحظة: لإحياء الثقافات من مخزون الجلسرين ، قم بإذابة مخزون الجلسرين عند 4 درجات مئوية. قم بتلقيح حلقة من هذا المخزون في 2 مل من مرق لوريا بيرتاني المعقم الذي يحتوي على المضاد الحيوي المعني واتركه ينمو.
3. تحديد البكتيريا القابلة للزراعة عن طريق تسلسل جين 16S rRNA
- تحضير قالب الحمض النووي من العزلات لتفاعل البوليميراز المتسلسل
ملاحظة: تم إعطاء البروتوكول الموصوف لإعداد قالب الحمض النووي لتفاعل البوليميراز المتسلسل لعزل الحمض النووي الخام من البكتيريا بواسطة Carlson et al.10.- باستخدام مسواك معقم ، خذ مستعمرة واحدة معزولة ونقية من العزلة تنمو على صفيحة بتري. المستعمرة البكتيرية في 100 ميكرولتر من الماء المعقم المقطر المزدوج في أنبوب طرد مركزي دقيق معقم ويغلي لمدة 10 دقائق.
- جهاز طرد مركزي للتعليق عند 10000 × جم لمدة 2 دقيقة لحبيبات الحطام ، ونقل الطافي إلى أنبوب طرد مركزي دقيق معقم جديد لاستخدامه كقالب الحمض النووي الخام.
- تضخيم PCR المستهدف لمنطقة V3 لجين 16S rRNA والتسلسل
- تحضير 40 ميكرولتر من خليط التفاعل في أنبوب PCR لتضخيم PCR ، كما هو مذكور في الجدول 1.
ملاحظة: يجب أن يتم تحضير الحمض النووي على كتلة ثلجية مع تقليل فرصة التلوث (ارتداء القفازات أثناء التعامل مع الكواشف ، وتنظيف سطح العمل جيدا باستخدام 70٪ من الإيثانول). - ضع الأنبوب في الكتلة الحرارية ، وقم بتشغيل البرنامج المناسب في جهاز التدوير الحراري PCR. انظر الجدول 2 لمعرفة ظروف دورة PCR الموحدة والمعلومات التمهيدية لتضخيم مناطق V3 لجينات 16S rRNA.
- لحل الأمبليكون والتصور ، قم بإجراء رحلان كهربائي هلام الأغاروز (AGE). امزج 10 ميكرولتر من منتج تفاعل البوليميراز المتسلسل المضخم و 2 ميكرولتر من محلول تحميل الهلام 6x (الجدول 3) ، وقم بتحميل هذا الخليط في الآبار على 1.5٪ هلام الأغاروز (قم بإذابة 1.5 جم من مسحوق الأغاروز في 100 مل من 1x TAE buffer [الجدول 3]) يحتوي على 5 ميكرولتر من 10 مجم / مل من بروميد الإيثيديوم (EtBr) إلى تركيز نهائي قدره 0.5 ميكروغرام / مل EtBr في 100 مل من هلام الأغاروز.
تنبيه: EtBr مادة مسرطنة قوية. يجب ارتداء القفازات في جميع الأوقات أثناء التعامل مع EtBr والمواد الهلامية التي تحتوي على EtBr. - أضف سلم الحمض النووي لتقدير حجم الأمبليكونات.
- نفذ رحلان كهربائي للهلام في مخزن خزان TAE عند 80-100 فولت.
- بمجرد تشغيل صبغة التتبع 3/4 من الجل ، أوقف الرحلان الكهربائي ، وتصور نطاقات amplicon تحت مصباح الأشعة فوق البنفسجية.
- استخدم منتج تفاعل البوليميراز المتسلسل (amplicon) لتسلسل جين 16S rRNA لتحديد العزلة.
- تحديد كمية الأمبليكون عن طريق إخضاعه للتحليل الطيفي باستخدام المعادلة (2).
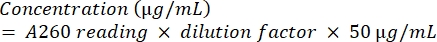 (2)
(2) - للتحقق من نقاء الحمض النووي ، احسب نسبة A260 / A280.
ملاحظة: من الناحية المثالية ، يجب أن يكون هذا الرقم أعلى من 1.5 ، ويفضل أن يكون بين 1.8 و 2.0. - لتحديد العزلات ، قارن التسلسلات التي تم الحصول عليها مع قواعد بيانات التسلسل باستخدام أداة بحث محاذاة مناسبة.
- تحضير 40 ميكرولتر من خليط التفاعل في أنبوب PCR لتضخيم PCR ، كما هو مذكور في الجدول 1.
4. الكشف عن مقاومة المضادات الحيوية في العزلات باستخدام اختبار الحساسية للمضادات الحيوية
ملاحظة: يصف هذا البروتوكول طريقة اختبار الحساسية للمضادات الحيوية (AST) عن طريق نشر القرص. تم استخدام أقراص المضادات الحيوية التالية: سيفوتاكسيم (5 ميكروغرام) ، أمبيسلين (10 ميكروغرام) ، ليفوفلوكساسين (5 ميكروغرام) ، كلورامفينيكول (30 ميكروغرام) ، تيجيسيكلين (15 ميكروغرام) ، سيفترياكسون (30 ميكروغرام) ، إيميبينيم (10 ميكروغرام) ، جنتاميسين (10 ميكروغرام) ، نيومايسين (10 ميكروغرام) ، تريميثوبريم (5 ميكروغرام) ، وسيبروفلوكساسين (5 ميكروغرام).
- تحضير اللقاح ل AST
- قم بتلقيح مستعمرة AR واحدة معزولة ومنقاة باستخدام حلقة معقمة في 2 مل من وسط معقم غير انتقائي ، مثل مرق Luria-Bertani (بدون أي مضاد حيوي) ، واحتضانها عند 37 درجة مئوية عند 80 دورة في الدقيقة طوال الليل.
- أعد التعليق بأخذ 100-150 ميكرولتر من الثقافة المزروعة بين عشية وضحاها (تقريبا ، OD 600 = 1.8-2.0) في 2 مل من وسط مرق Luria-Bertani الطازج غير الانتقائي واحتضانه لمدة 2-4 ساعات (حتى يصل OD600 إلى 0.4-0.5).
- قم بتخفيف هذا المعلق المزروع حديثا باستخدام محلول ملحي معقم بنسبة 0.85٪ بحيث تكون كثافة المزرعة مساوية لمعيار 0.5 McFarland (تقريبا ، OD600 = 0.1) ، وهو ما يتوافق تقريبا مع 1-2 × 108 خلايا / مل.
- امزج المعلق البكتيري برفق لتوزيع الخلايا بشكل متساو.
- استخدم التعليق أعلاه في غضون 15 دقيقة من التخفيف.
- تلقيح لوحات أجار
- قم بإعداد ألواح Mueller-Hinton Agar (MHA) لأداء AST عن طريق خلط 38 جم من MHA في 1000 مل من الماء المقطر المزدوج ، وإذابة الخليط عن طريق التسخين. الأوتوكلاف الخليط المذاب عند 121 درجة مئوية ، 15 رطل / بوصة مربعة لمدة 15 دقيقة.
- تأكد من أن عمق MHA في الألواح هو 4 مم (25 مل من الوسط لكل لوحة).
- في الوقت نفسه ، قم بإزالة أقراص المضادات الحيوية من الثلاجة وتسخينها إلى درجة حرارة الغرفة.
ملاحظة: يجب إذابة أقراص المضادات الحيوية تدريجيا عن طريق إذابة الأقراص في البداية عند 4 درجات مئوية وبعد ذلك في درجة حرارة الغرفة لتقليل أي خطر محتمل للتكثيف على الأقراص ، مما قد يؤثر لاحقا على منطقة التثبيط (ZOI). - في ظل ظروف معقمة ، اغمس قطعة قطن معقمة في اللقاح المحضر في الخطوة 4.1 ، وقم بإزالة التعليق الزائد لتجنب الإفراط في تلقيح الألواح.
- انشر الثقافة بالتساوي على الأطباق ، بدءا من أعلى لوحة MHA والانتقال ذهابا وإيابا من الحافة إلى الحافة. قم بتدوير اللوحة بمقدار 60 درجة أثناء المسح.
- تطبيق أقراص المضادات الحيوية
- بمساعدة ملقط معقم باللهب ، قم بنقل أقراص المضادات الحيوية بشكل معقم إلى ألواح MHA الملقحة ، واضغط على الأقراص برفق لضمان ملامسة المستوى الكامل للأجار.
ملاحظة: يجب أن يتم هذا الإجراء في غضون 15 دقيقة من تلقيح المزرعة على اللوحات. - وضع العدد المناسب من أقراص المضادات الحيوية على صفيحة الآجار مع مراعاة الكائن الحي والمضاد الحيوي المستخدم وحجم الصفيحة لتجنب تداخل مناطق التثبيط.
ملاحظة: يمكن استيعاب أربعة إلى خمسة أقراص على لوحة دائرية مقاس 90 مم.
- بمساعدة ملقط معقم باللهب ، قم بنقل أقراص المضادات الحيوية بشكل معقم إلى ألواح MHA الملقحة ، واضغط على الأقراص برفق لضمان ملامسة المستوى الكامل للأجار.
- حضانة اللوحات
- في غضون 15 دقيقة من تطبيق أقراص المضادات الحيوية ، اقلب الألواح واحتضنها عند 37 درجة مئوية طوال الليل.
- تفسير النتائج
- قم بقياس قطر ZOI بالمليمترات (مم) ، وفسر وفقا لقيم نقطة التوقف التي قدمتها EUCAST11. انظر المثالين الواردين أدناه.
- نقطة توقف قطر المنطقة (مم) لقرص مضاد حيوي سيبروفلوكساسين (5 ميكروغرام) للبكتيريا المعوية هي S ≥ 25 و R < 22 ، مما يعني أنها تعتبر حساسة (S) إذا ≥ ZOI 25 مم ، بينما تكون مقاومة (R) إذا كان ZOI < 22 مم. إذا كان قطر ZOI يتراوح بين 22 و 25 ، فإن العزلة تعتبر متوسطة (I).
- نقطة توقف قطر المنطقة (مم) لقرص مضاد حيوي الكلورامفينيكول (30 ميكروغرام) للمكورات العنقودية هي S ≥ 18 و R < 18 ، مما يعني أنها تعتبر حساسة إذا كان ZOI ≥ 18 مم ، بينما تكون مقاومة إذا كان ZOI < 18 مم.
- تحديد مؤشر المقاومة المتعددة للمضادات الحيوية (MAR) من خلال إيجاد نسبة عدد المضادات الحيوية التي تقاوم العزلة لها إلى إجمالي عدد المضادات الحيوية التي تتعرض لها العزل.
ملاحظة: لمراقبة الجودة ، E. coli ATCC 25922 و S. الذهبيه يتم استخدام ATCC 29213 كسلالات مرجعية تتبع البروتوكول كما في الخطوة 4.
- قم بقياس قطر ZOI بالمليمترات (مم) ، وفسر وفقا لقيم نقطة التوقف التي قدمتها EUCAST11. انظر المثالين الواردين أدناه.
5. الكشف القائم على تفاعل البوليميراز المتسلسل للجينات المقاومة للمضادات الحيوية في العزلات
- استخدم بروتوكول PCR قياسي لتحديد ARGs في العزلات. تحضير قالب الحمض النووي (DNA) باستخدام البروتوكول المعطى في الخطوة 3.1.
ملاحظة: كانت ظروف دورة تفاعل البوليميراز المتسلسل المستخدمة في هذه الدراسة 94 درجة مئوية لمدة 10 دقائق ، تليها 35 دورة من 94 درجة مئوية لمدة 30 ثانية ، والتلدين لمدة 30 ثانية عند درجة الحرارة المناسبة (كما هو موحد لكل مجموعة أولية) ، والتمديد عند 72 درجة مئوية لمدة 40 ثانية ، والتمديد النهائي عند 72 درجة مئوية لمدة 5 دقائق. يوصف خليط التفاعل في الجدول 4. ترد قائمة ARGs والبادئات ودرجات حرارة التلدين في الجدول 5. - لحل وتصور والتحقق من نقاء مكبرات الصوت ، اتبع الخطوات 3.2.3-3.2.10.
6. تحليل الحمض النووي الميتاجينومي الكامل لتحديد التنوع البكتيري الكلي والكشف عن ARGs في metagenome
- استخراج الحمض النووي الكلي (metagenome) من عينة الماء
- استخراج الحمض النووي metagenomic من عينات المياه المفلترة.
ملاحظة: في الدراسة الحالية ، تم استخراج الحمض النووي الميتاجينومي (الحمض النووي الكلي) من عينات المياه المفلترة باستخدام مجموعة عزل الحمض النووي المشار إليها باتباع بروتوكول الشركة المصنعة (انظر جدول المواد). - تحقق من جودة الحمض النووي عن طريق تحميل 3 ميكرولتر من الحمض النووي الميتاجينومي المستخرج على هلام أغاروز 0.8٪ ، وقم بتشغيل الجل عند 80-110 فولت لمدة 30 دقيقة تقريبا.
- تحقق من وجود شريط واحد سليم.
- تحقق من تركيز الحمض النووي باستخدام مقياس الفلور.
- استخراج الحمض النووي metagenomic من عينات المياه المفلترة.
- تحديد التنوع البكتيري والكشف عن ARGs باستخدام تسلسل الحمض النووي الميتاجينومي الكامل
- إعداد المكتبة وتضخيم تفاعل البوليميراز المتسلسل:
- قم بإعداد مكتبة تسلسل نهاية مزدوجة باستخدام مجموعة إعداد مكتبة الحمض النووي المشار إليها (انظر جدول المواد).
- جهز الحمض النووي لربط المحول عن طريق أخذ 200 نانوغرام من الحمض النووي وقصه ميكانيكيا إلى أجزاء أصغر ، تليها خطوة مستمرة من الإصلاح النهائي حيث تتم إضافة "A" إلى الأطراف 3.
- اعتمادا على النظام الأساسي المستخدم للتسلسل ، قم بتوصيل محولات محددة إلى طرفي شظايا الحمض النووي.
ملاحظة: توجد تسلسلات ضرورية لربط مكتبات الباركود المزدوج بخلية تدفق للتسلسل في هذه المحولات. يسمح ذلك بتضخيم تفاعل البوليميراز المتسلسل للأجزاء المربوطة بالمحول وربط بادئات التسلسل القياسية. - للتحقق من الجودة والكمية ، قم بتحليل المكتبة المضخمة باستخدام شريحة DNA عالية الحساسية وفقا لتعليمات الشركة المصنعة.
- توليد العنقود والتسلسل:
- قم بتحميل المكتبة المضخمة على منصة التسلسل المناسبة لإنشاء الكتلة والتسلسل اللاحق.
ملاحظة: ترتبط جزيئات المكتبة بالمحول التكميلي oligos على خلية التدفق ذات النهاية المقترنة. أثناء التسلسل ، يتم شق الخيوط الأمامية بشكل انتقائي بعد إعادة تكوين الشريط العكسي. ثم يتم تسلسل هذا الشريط العكسي المنسوخ من الطرف المقابل للجزء.
- قم بتحميل المكتبة المضخمة على منصة التسلسل المناسبة لإنشاء الكتلة والتسلسل اللاحق.
- تحليل المعلوماتية الحيوية:
- قم بإنشاء سقالات من البيانات عالية الجودة باستخدام النظام الأساسي المناسب.
- إخضاع هذه السقالات لتحليل المعلوماتية الحيوية للتصنيف التصنيفي وتحديد ARGs.
ملاحظة: يرد في الشكل 1 سير العمل لتحليل الحمض النووي الميتاجينومي بأكمله لتحديد التنوع البكتيري الكلي والكشف عن ARGs في metagenome. ويرد في الشكل 2 جدول سير للمنهجية الكاملة الموضحة في المخطوطة.
- إعداد المكتبة وتضخيم تفاعل البوليميراز المتسلسل:
النتائج
إجمالي الحمل البكتيري وعدد البكتيريا المقاومة للمضادات الحيوية (AR)
تم إجراء تعداد الحمل البكتيري الكلي عن طريق نشر 10−4 إلى 10−6 أضعاف التخفيفات لعينات المياه على R2A Agar ، الوسط المعدل. لتعداد عدد بكتيريا AR ، تم نشر 10−3 إلى 10−6 أضعاف التخفيفات على لوحات الوس?...
Discussion
يلعب جمع العينات ومعالجتها دورا مهما وقد يؤثر على نتائج الدراسة وتفسيرها. ومن ثم ، لاستبعاد التباين في العينات ، من المهم إجراء أخذ العينات في مواقع متعددة من جسم المياه العذبة قيد الدراسة. يمكن أن يؤدي الحفاظ على الظروف البيئية المعقمة المناسبة عند التعامل مع هذه العينات إلى منع التلوث. ع...
Disclosures
ليس لدى المؤلفين مصالح متضاربة للكشف عنها.
Acknowledgements
تم دعم هذا العمل جزئيا من خلال المنح المالية من قسم العلوم والتكنولوجيا - تعزيز البحث الجامعي والتميز العلمي (DST-PURSE) مخطط جامعة مومباي. عملت ديفيكا غاديغاونكار كزميلة مشروع في إطار المخطط. تم الاعتراف بالمساعدة الفنية التي قدمها Harshali Shinde ، زميل باحث أول في إطار مشروع مجلس أبحاث العلوم والتكنولوجيا والعلوم والهندسة (DST-SERB): CRG / 2018 / 003624.
Materials
| Name | Company | Catalog Number | Comments |
| 100 bp DNA ladder | Himedia | MBT049-50LN | For estimation of size of the amplicons |
| 2x PCR Taq mastermix | HiMedia | MBT061-50R | For making PCR reaction mixture |
| 37 °C Incubator | GS-192, Gayatri Scientific | NA | For incubation of bacteria |
| 6x Gel Loading Buffer | HiMedia | ML015-1ML | Loading and Tracking dye which helps to weigh down the DNA sample and track the progress of electrophoresis |
| Agarose powder | Himedia | MB229-50G | For resolving amplicons during Agarose Gel Electrophoresis (AGE) |
| Ampicillin antibiotic disc | HiMedia | SD002 | For performing AST |
| Autoclave | Equitron | NA | Required for sterilization of media, glass plates, test tubes, etc |
| Bioanalyzer 2100 | Agilent Technologies | NA | To check the quality and quantity of the amplified library |
| Bisafety B2 Cabinet | IMSET | IMSET BSC-Class II Type B2 | Used for microbiological work like bacterial culturing, AST etc. |
| Cefotaxime antibiotic disc | HiMedia | SD295E-5VL | For performing AST |
| Cefotaxime antibiotic powder | HiMedia | TC352-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Ceftriaxone antibiotic disc | HiMedia | SD065 | For performing AST |
| Centrifuge Minispin | Eppendorf | Minispin Plus-5453 | Used to pellet the debris during crude DNA preparation |
| Chloramphenicol antibiotic disc | HiMedia | SD006-5x50DS | For performing AST |
| Ciprofloxacin antibiotic disc | HiMedia | SD060-5x50DS | For performing AST |
| Ciprofloxacin antibiotic powder | HiMedia | TC447-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Colorimeter | Quest | NA | For checking the OD of culture suspensions |
| Comprehensive Antibiotic Resistance Database (CARD) database | functional annotation of ARGs; https://card.mcmaster.ca/ | ||
| Cooling Shaker Incubator | BTL41 Allied Scientific | NA | For incubation of media plates for culturing bacteria |
| Deep Freezer (-40 °C) | Haier | DW40L, Haier Biomedicals | For storage of glycerol stocks |
| DNA Library Prep Kit | NEB Next Ultra DNA Library Prep Kit for Illumina | NA | Paired-end sequencing library preparation |
| EDTA | HiMedia | GRM1195-100G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Electrophoresis Apparatus | TechResource | 15 cm gel casting tray | For making the agarose gel and carrying out electrophoresis |
| Electrophoresis Power pack with electrodes | Genei | NA | For running the AGE |
| Erythromycin antibiotic disc | HiMedia | SD222-5VL | For performing AST |
| Erythromycin antibiotic powder | HiMedia | CMS528-1G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Erythromycin antibiotic powder | HiMedia | TC024-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Escherichia coli ATCC 25922 | HiMedia | 0335X-1 | Used as a control while performing AST |
| Ethidium Bromide | HiMedia | MB071-1G | Intercalating agent and visualizaion of DNA after electrophoresis under Gel Documentation System |
| Fluorometer | Qubit 2.0 | NA | For determining concentration of extracted metagenomic DNA |
| Gel Documentation System | BioRad | Used for visualizing PCR amplicons after electrophoresis | |
| Gentamicin antibiotic disc | HiMedia | SD170-5x50DS | For performing AST |
| Glacial Acetic Acid | HiMedia | AS119-500ML | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Glycerol | HiMedia | GRM1027-500ML | For making glycerol stocks |
| Imipenem antibiotic disc | HiMedia | SD073 | For performing AST |
| Kaiju Database | NA | NA | For taxonomical classification of reads; https://kaiju.binf.ku.dk/ |
| Kanamycin antibiotic disc | HiMedia | SD017-5x50DS | For performing AST |
| Kanamycin antibiotic powder | HiMedia | MB105-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Levofloxacin antibiotic disc | HiMedia | SD216-5VL | For performing AST |
| Luria Bertani broth | Himedia | M1245-500G | For enrichment of cultures |
| McFarland Standards | Himedia | R092-1No | To compare density of culture suspension |
| Molecular Biology water | HiMedia | TCL018-500ML | For making PCR reaction mixture |
| Mueller-Hinton Agar (MHA) | HiMedia | M173-500G | For performing Antibiotc Susceptibility Testing (AST) |
| Neomycin antibiotic disc | HiMedia | SD731-5x50DS | For performing AST |
| PCR Gradient Thermal Cycler | Eppendorf | Mastercycler Nexus Gradient 230V/50-60 Hz | Used for performing PCR for amplification of 16S rRNA region and various Antibiotic Resistance genes |
| Primers | Xcelris | NA | For PCR amplication |
| R2A Agar, Modified | HiMedia | M1743 | For preparation of media plates for isolation of total and antibiotic resistant (AR) bacterial load |
| Scaffold generation | CLC Genomics Workbench 6.0 | NA | For generation of scaffolds |
| Sequencer | Illumina platform (2 x 150 bp chemistry) | NA | Sequencing of amplified library |
| Sodium Chloride | HiMedia | TC046-500G | For preparation of 0.85% saline for serially diluting the water sample |
| Soil DNA isolation Kit | Xcelgen | NA | For extraction of whole metagenomic DNA from the filtered water sample |
| Staphylococcus aureus subsp. aureus ATCC 29213 | HiMedia | 0365P | Used as a control while performing AST |
| Taxonomical Classification | Kaiju ioinformatics tool | NA | For classification of reads into different taxonomic groups from phylum to genus level |
| The Comprehensive Antibiotic Resistance Database (CARD) | NA | NA | For functional annotation of ARGs |
| Tigecycline antibiotic disc | HiMedia | SD278 | For performing AST |
| Trimethoprim antibiotic disc | HiMedia | SD039-5x50DS | For performing AST |
| Tris base | HiMedia | TC072-500G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Vancomycin antibiotic powder | HiMedia | CMS217 | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Weighing Balance | Mettler Toledo | ME204 Mettler Toledo | Used for weighing media powders, reagent powders etc. |
| NA - Not Applicable |
References
- Prestinaci, F., Pezzotti, P., Pantosti, A. Antimicrobial resistance: A global multifaceted phenomenon. Pathogens and Global Health. 109 (7), 309-318 (2015).
- Knight, G., et al. Antimicrobial resistance and COVID-19: Intersections and implications. Elife. 10, 64139 (2021).
- Ventola, C. L. The antibiotic resistance crisis: Part 1: Causes and threats. Pharmacy and Therapeutics. 40 (4), 277-283 (2015).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J. R., Rath, A. Metagenomic analysis of total microbial diversity and antibiotic resistance of culturable microorganisms in raw chicken meat and mung sprouts (Phaseolus aureus) sold in retail markets of Mumbai. India. Current Science. 113 (1), 71-79 (2017).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J., Rath, A. Characterization of multiple antibiotic resistance of culturable microorganisms and metagenomic analysis of total microbial diversity of marine fish sold in retail shops in Mumbai, India. Environmental Science and Pollution Research. 25 (7), 6228-6239 (2018).
- Czekalski, N., GascónDíez, E., Bürgmann, H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake. The ISME Journal. 8 (7), 1381-1390 (2014).
- Kraemer, S., Ramachandran, A., Perron, G. Antibiotic pollution in the environment: From microbial ecology to public policy. Microorganisms. 7 (6), 180 (2019).
- Edmonds-Wilson, S., Nurinova, N., Zapka, C., Fierer, N., Wilson, M. Review of human hand microbiome research. Journal of Dermatological Science. 80 (1), 3-12 (2015).
- de Abreu, V., Perdigão, J., Almeida, S. Metagenomic approaches to analyze antimicrobial resistance: An overview. Frontiers in Genetics. 11, 575592 (2021).
- Carlson, S., et al. Detection of multiresistant Salmonella typhimurium DT104 using multiplex and fluorogenic PCR. Molecular and Cellular Probes. 13 (3), 213-222 (1999).
- Breakpoint tables for interpretation of MICs and zone diameters, Version 12.0. European Committee on Antimicrobial Susceptibility Testing Available from: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_12.0_Breakpoint_Tables.pdf (2022)
- Bharti, R., Grimm, D. Current challenges and best-practice protocols for microbiome analysis. Briefings in Bioinformatics. 22 (1), 178-193 (2019).
- Choo, J., Leong, L., Rogers, G. Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports. 5, 16350 (2015).
- Clinical and Laboratory Standards Institute. . Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria that Grow Aerobically, 11th edition. , (2015).
- Bayot, M., Bragg, B. Antimicrobial Susceptibility Testing. StatPearls. , (2021).
- Joseph, A. A., Odimayo, M. S., Olokoba, L. B., Olokoba, A. B., Popoola, G. O. Multiple antibiotic resistance index of Escherichia coli isolates in a tertiary hospital in South-West Nigeria. Medical Journal of Zambia. 44 (4), 225-232 (2017).
- Lorenz, T. Polymerase chain reaction: Basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Rolin, J. Food and human gut as reservoirs of transferable antibiotic resistance encoding genes. Frontiers in Microbiology. 4, 173 (2013).
- Racewicz, P., et al. Prevalence and characterisation of antimicrobial resistance genes and class 1 and 2 integrons in multiresistant Escherichia coli isolated from poultry production. Scientific Reports. 12, 6062 (2022).
- Gebreyes, W., Thakur, S. Multidrug-resistant Salmonella enterica serovar Muenchen from pigs and humans and potential interserovar transfer of antimicrobial resistance. Antimicrobial Agents and Chemotherapy. 49 (2), 503-511 (2005).
- Li, L., et al. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui Province, China. PLoS One. 9 (8), 104356 (2014).
- Akers, K., et al. Aminoglycoside resistance and susceptibility testing errors in Acinetobacter baumannii-calcoaceticus complex. Journal Of Clinical Microbiology. 48 (4), 1132-1138 (2010).
- Ciesielczuk, H. . Extra-intestinal pathogenic Escherichia coli in the UK: The importance in bacteraemia versus urinary tract infection, colonisation of widespread clones and specific virulence factors. , (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved