É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Isolamento e Identificação de Bactérias Resistentes a Antibióticos Transmitidos pela Água e Caracterização Molecular de seus Genes de Resistência a Antibióticos
Neste Artigo
Resumo
Aqui, apresentamos um protocolo detalhado para o isolamento e identificação de bactérias resistentes a antibióticos da água e a caracterização molecular de seus genes de resistência a antibióticos (ARGs). O uso de técnicas baseadas em cultura e não baseadas em cultura (análise metagenômica) fornece informações completas sobre a diversidade bacteriana total e o pool total de diferentes ARGs presentes em águas doces de Mumbai, Índia.
Resumo
O desenvolvimento e disseminação da resistência aos antibióticos (RA) através da microbiota associada a corpos de água doce é uma grande preocupação de saúde global. No presente estudo, amostras de água doce foram coletadas e analisadas com relação à diversidade bacteriana total e aos genes AR (ARGs) usando técnicas convencionais baseadas em cultura e uma abordagem metagenômica independente de cultura de alto rendimento. Este trabalho apresenta um protocolo sistemático para a enumeração das bactérias cultiváveis totais e resistentes a antibióticos de amostras de água doce e a determinação da resistência fenotípica e genotípica em isolados cultiváveis. Além disso, relatamos o uso de análise metagenômica completa do DNA metagenômico total extraído da amostra de água doce para a identificação da diversidade bacteriana geral, incluindo bactérias não cultiváveis, e a identificação do pool total de diferentes ARGs (resistoma) no corpo d'água. Seguindo esses protocolos detalhados, observamos uma alta carga bacteriana resistente a antibióticos na faixa de 9,6 × 10 5-1,2 × 109 UFC/mL. A maioria dos isolados foi resistente aos múltiplos antibióticos testados, incluindo cefotaxima, ampicilina, levofloxacina, cloranfenicol, ceftriaxona, gentamicina, neomicina, trimetoprima e ciprofloxacina, com múltiplos índices de resistência a antibióticos (MAR) de ≥0,2, indicando altos níveis de resistência nos isolados. O sequenciamento do RNAr 16S identificou potenciais patógenos humanos, como Klebsiella pneumoniae, e bactérias oportunistas, como Comamonas spp., Micrococcus spp., Arthrobacter spp. e Aeromonas spp. A caracterização molecular dos isolados mostrou a presença de vários ARGs, como blaTEM, blaCTX-M (β-lactâmicos), aadA, aac (6')-Ib (aminoglicosídeos) e dfr1 (trimetoprims), o que também foi confirmado por toda a análise metagenômica de DNA. Uma alta prevalência de outros ARGs que codificam bombas de efluxo de antibióticos - mtrA, macB, mdtA, acrD, β-lactamases-SMB-1, VIM-20, ccrA, ampC, blaZ, o gene da cloranfenicol acetiltransferase catB10 e o gene de resistência à rifampicina rphB - também foi detectada no DNA metagenômico. Com a ajuda dos protocolos discutidos neste estudo, confirmamos a presença de bactérias MAR transmitidas pela água com diversas características fenotípicas e genotípicas da RA. Assim, a análise metagenômica completa do DNA pode ser usada como uma técnica complementar às técnicas convencionais baseadas em cultura para determinar o status geral de RA de um corpo d'água.
Introdução
A resistência antimicrobiana (RAM) tem sido identificada como um dos problemas globais mais prementes. A rápida evolução da RAM e a sua propagação mundial são uma das maiores ameaças à saúde humana e à economia global em termos dos custos de saúde que lhe estão associados1. O uso excessivo e indevido de antibióticos levaram a um aumento da RA. Isso foi destacado pela pandemia de COVID-19, durante a qual o tratamento de infecções secundárias associadas, em muitos casos, foi extremamente comprometido devido à RAM nos pacientes afetados2. Além do uso/uso indevido direto de antibióticos por seres humanos, o uso excessivo e indevido de antibióticos na agricultura e pecuária e sua descarga inadequada no meio ambiente, incluindo corpos d'água, são uma grande preocupação3. O surgimento de novas características de resistência e multirresistência em bactérias destaca urgentemente a necessidade de uma melhor compreensão dos fatores que levam ao desenvolvimento da RA e sua disseminação. Múltiplas bactérias resistentes a antibióticos, que muitas vezes carregam múltiplos genes AR (ARGs) em elementos genéticos móveis, como plasmídeos, podem transferir esses genes de resistência para microrganismos não resistentes, incluindo potenciais patógenos humanos, levando ao surgimento de superbactérias que são intratáveis mesmo com antibióticos de último recurso4. Essas múltiplas bactérias resistentes a antibióticos, se presentes nos ecossistemas aquáticos, podem entrar diretamente no intestino humano através do consumo de alimentos contaminados à base de água, como peixes, caranguejos e moluscos. Estudos anteriores mostraram que a disseminação de bactérias RA em sistemas de água que ocorrem naturalmente também pode atingir outros suprimentos de água, incluindo água potável, e, assim, pode entrar na cadeia alimentar humana 5,6,7.
O objetivo do presente estudo é fornecer um protocolo abrangente usando uma combinação de técnicas baseadas em cultura e não baseadas em cultura (análise metagenômica completa) para obter informações completas sobre a diversidade bacteriana total e o pool total de diferentes ARGs presentes em um corpo d'água em Mumbai, Índia. Convencionalmente, técnicas baseadas em cultura têm sido usadas para estudar a diversidade bacteriana em corpos d'água. Como os microrganismos cultiváveis constituem apenas uma pequena porcentagem da microbiota total em qualquer nicho, para ter uma melhor compreensão do status geral da diversidade bacteriana e das várias características resistentes prevalentes em qualquer amostra, várias técnicas baseadas em cultura e independentes da cultura devem ser usadas em conjunto. Uma dessas técnicas robustas e confiáveis independentes de cultura é a análise metagenômica completa do DNA. Esse método de alto rendimento tem sido utilizado com sucesso em diversos estudos sobre diversidade bacteriana ou anotações funcionais de vários ARGs 8,9. Esta técnica utiliza o metagenoma (o material genético total de uma amostra) como matéria-prima para várias análises e, por isso, é independente da cultura. Os protocolos do presente estudo podem ser utilizados para análise metagenômica completa de DNA para obtenção de informações sobre a diversidade bacteriana total e vários ARGs (resistoma) em amostras de água.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Recolha e processamento de amostras
- Coleta de amostras
- Recolher o volume adequado da amostra de água no(s) recipiente(s) de amostra estéril(s), assegurando que não mais de 3/4 do recipiente estão cheios.
- Transportar as amostras para o laboratório em condições assépticas o mais rapidamente possível após a colheita e processá-las imediatamente.
- Processamento de amostras
- Filtre assepticamente a amostra de água através de um pano de musselina estéril para remover qualquer material particulado.
- Efectuar diluições em série adequadas da água filtrada para uma análise mais aprofundada.
2. Estimativa da carga bacteriana total e da contagem de bactérias resistentes a antibióticos
- Determinação da carga bacteriana total
- Suspender 18,12 g de Agar R2A, pó modificado em 1.000 mL de água duplamente destilada e dissolver a mistura por aquecimento. Autoclave a mistura dissolvida a 121 °C, 15 psi por 20 min. Prepare o ágar R2A, Placas modificadas derramando a quantidade apropriada da mistura autoclavada em placas de Petri estéreis (por exemplo, adicione aproximadamente 20 mL de meio estéril autoclavado a uma placa de Petri estéril de 90 mm).
- Espalhar uniformemente 100 μL das diluições apropriadas da amostra de água filtrada no ágar R2A, placa modificada uma vez que o meio se solidifica. Execute o experimento em duplicata.
- Incubar todas as placas acima a 35-37 °C durante 48 h (variar a temperatura e o tempo de incubação dependendo do meio utilizado para o isolamento).
- Exprimir a carga bacteriana total em termos de unidades formadoras de colónias por mililitro (UFC/ml) utilizando a equação (1):
 (1)
(1)
- Determinação da contagem bacteriana de RA
- Siga as etapas 2.1.1-2.1.4. No entanto, em vez de Agar R2A, placas modificadas, use ágar R2A, placas modificadas suplementadas individualmente com cinco antibióticos diferentes, a saber, cefotaxima (3 μg/mL), ciprofloxacina (0,5 μg/mL), eritromicina (20 μg/mL), canamicina (15 μg/mL) e vancomicina (3 μg/mL).
- Adicionar os antibióticos separadamente em tubos contendo 20 ml de ágar R2A fundido estéril, modificado (com a temperatura do ágar R2A fundido, modificado a ≤40 °C) para atingir a concentração final do antibiótico, tal como mencionado na etapa 2.2.1.
- Redemoinho para misturar uniformemente e despeje em placas de Petri estéreis antes que o ágar se solidifique. Execute o experimento em duplicata.
- Incubar todas as placas acima a 35-37 °C durante 48 h (se utilizar um meio diferente, a temperatura e o tempo de incubação podem variar).
- Para controle de qualidade e verificação da eficácia dos antibióticos, espalhe 100 μL de suspensões bacterianas das cepas Escherichia coli ATCC 25922 e Staphylococcus aureus ATCC 29213 em suas respectivas placas R2A contendo antibióticos, Placas modificadas (certifique-se de que a densidade da cultura fresca usada para a inoculação seja OD = 0,5 a 600 nm).
- Determinar a contagem bacteriana resistente aos antibióticos em termos de UFC/ml, conforme descrito no passo 2.1.4.
- Existências de glicerol dos isolados
- Selecione colônias de RA morfologicamente distintas.
- Suspender uma única colônia isolada em 2 mL de caldo estéril de Luria-Bertani contendo o respectivo antibiótico (por exemplo, se uma colônia foi selecionada de uma placa contendo cefotaxima, inocular a colônia a partir da etapa 2.3.1 em caldo estéril de Luria-Bertani contendo cefotaxima em sua respectiva concentração).
- Incubar os tubos inoculados a 37 °C a 80 rpm até que o OD600 atinja 0,5.
- Preparar as reservas de glicerol dos isolados misturando 750 μL da suspensão de cultura da fase 2.3.3 em 250 μL de glicerol estéril a 100% em condições assépticas.
- Conservar as existências de glicerol a -80 °C até uma análise mais aprofundada.
NOTA: Para o renascimento das culturas das unidades populacionais de glicerol, descongelar as reservas de glicerol a 4 °C. Inocular uma alça deste estoque em 2 mL de caldo estéril de Luria-Bertani contendo o respectivo antibiótico e deixe crescer.
3. Identificação de bactérias cultiváveis por sequenciamento do gene 16S rRNA
- Preparação do molde de DNA dos isolados para PCR
NOTA: O protocolo descrito para a preparação do molde de DNA para PCR para o isolamento do DNA bruto das bactérias é dado por Carlson et al.10.- Usando um palito de dente estéril, pegue uma única colônia isolada e pura do isolado que cresce em uma placa de Petri. Suspender a colônia bacteriana em 100 μL de água estéril duplamente destilada em um tubo de microcentrífuga estéril e ferver por 10 min.
- Centrifugar a suspensão a 10.000 × g durante 2 minutos para peletizar os detritos e transferir o sobrenadante para um tubo de microcentrífuga estéril fresco para utilização como molde de ADN bruto.
- Amplificação por PCR direcionada da região V3 do gene 16S rRNA e sequenciamento
- Preparar 40 μL da mistura de reação em um tubo de PCR para amplificação por PCR, conforme mencionado na Tabela 1.
NOTA: A preparação do DNA deve ser realizada em um bloco de gelo, minimizando a chance de contaminação (use luvas ao manusear os reagentes e limpe a superfície de trabalho completamente com etanol a 70%). - Coloque o tubo no bloco térmico e execute o programa apropriado no termociclador de PCR. Veja a Tabela 2 para as condições padronizadas de ciclo de PCR e as informações do primer para a amplificação das regiões V3 dos genes 16S rRNA.
- Para resolver os amplificadores e visualização, realizar eletroforese em gel de agarose (AGE). Misture 10 μL do produto de PCR amplificado e 2 μL de tampão de carga de gel 6x (Tabela 3) e carregue essa mistura em poços em gel de agarose a 1,5% (dissolver 1,5 g de pó de agarose em 100 mL de tampão TAE 1x [Tabela 3]) contendo 5 μL de brometo de etídio de 10 mg/mL (EtBr) até uma concentração final de 0,5 μg/mL de EtBr em 100 mL do gel de agarose.
CUIDADO: EtBr é um potente carcinógeno. As luvas devem ser usadas em todos os momentos durante o manuseio de EtBr e géis contendo EtBr. - Adicione a escada de DNA para a estimativa do tamanho dos amplificadores.
- Realize a eletroforese do gel em um tampão de tanque TAE a 80-100 V.
- Uma vez que o corante de rastreamento executa 3/4 do gel, pare a eletroforese e visualize as bandas de amplificador sob um transiluminador UV.
- Use o produto de PCR (amplicon) para sequenciamento do gene 16S rRNA para identificar o isolado.
- Quantificar o amplicon submetendo-o à análise espectrofotométrica usando a equação (2).
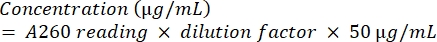 (2)
(2) - Para verificar a pureza do ADN, calcule a relação A260/A280.
NOTA: Idealmente, esse número deve estar acima de 1,5 e, de preferência, entre 1,8 e 2,0. - Para identificar os isolados, compare as sequências obtidas com bancos de dados de sequência usando uma ferramenta de pesquisa de alinhamento apropriada.
- Preparar 40 μL da mistura de reação em um tubo de PCR para amplificação por PCR, conforme mencionado na Tabela 1.
4. Detecção de resistência a antibióticos nos isolados utilizando testes de sensibilidade a antibióticos
NOTA: Este protocolo descreve o método para o teste de suscetibilidade a antibióticos (AST) por difusão em disco. Foram utilizados os seguintes discos antibióticos: cefotaxima (5 μg), ampicilina (10 μg), levofloxacina (5 μg), cloranfenicol (30 μg), tigeciclina (15 μg), ceftriaxona (30 μg), imipenem (10 μg), gentamicina (10 μg), neomicina (10 μg), trimetoprim (5 μg) e ciprofloxacina (5 μg).
- Preparação do inóculo para a AST
- Inocular assepticamente uma única colônia de RA purificada isolada usando uma alça estéril em 2 mL de um meio estéril não seletivo, como o caldo de Luria-Bertani (sem qualquer antibiótico), e incubar a 37 °C a 80 rpm durante a noite.
- Ressuspender tomando 100-150 μL da cultura cultivada durante a noite (aproximadamente, OD 600 = 1,8-2,0) em 2 mL de caldo fresco não seletivo de Luria-Bertani e incubar por 2-4 h (até que a OD600 atinja 0,4-0,5).
- Diluir esta suspensão de cultura recém-cultivada usando solução salina estéril a 0,85%, de modo que a densidade da cultura seja igual a 0,5 padrão de McFarland (aproximadamente, OD600 = 0,1), o que corresponde aproximadamente a 1-2 × 108 células/mL.
- Misture suavemente a suspensão bacteriana para uma distribuição celular uniforme.
- Utilize a suspensão acima referida no prazo de 15 minutos após a diluição.
- Inoculação das placas de ágar
- Preparar placas de ágar Mueller-Hinton (MHA) para a realização do AST misturando 38 g de MHA em 1.000 mL de água duplamente destilada e dissolver a mistura por aquecimento. Autoclave a mistura dissolvida a 121 °C, 15 psi durante 15 min.
- Certifique-se de que a profundidade do MHA nas placas seja de 4 mm (25 mL de meio por placa).
- Simultaneamente, retire os discos antibióticos do congelador e aqueça-os à temperatura ambiente.
NOTA: Os discos de antibióticos devem ser gradualmente descongelados descongelando inicialmente os discos a 4 °C e posteriormente à temperatura ambiente para reduzir qualquer perigo potencial de condensação nos discos, o que pode subsequentemente afectar a zona de inibição (ZOI). - Em condições assépticas, mergulhe um cotonete estéril no inóculo preparado na etapa 4.1 e remova o excesso de suspensão para evitar a inoculação excessiva das placas.
- Espalhe a cultura uniformemente nas placas, começando do topo da placa MHA e indo e voltando de ponta a ponta. Gire a placa em 60° enquanto balança.
- Aplicação dos discos antibióticos
- Com a ajuda de pinças esterilizadas por chama, transfira assepticamente os discos antibióticos para as placas MHA inoculadas e pressione os discos suavemente para garantir o contato completo do nível com o ágar.
NOTA: Este procedimento deve ser feito dentro de 15 min da inoculação da cultura nas placas. - Coloque o número apropriado de discos de antibióticos na placa de ágar, levando em conta o organismo, o antibiótico usado e o tamanho da placa para evitar a sobreposição das zonas de inibição.
NOTA: Quatro a cinco discos podem ser acomodados em uma placa circular de 90 mm.
- Com a ajuda de pinças esterilizadas por chama, transfira assepticamente os discos antibióticos para as placas MHA inoculadas e pressione os discos suavemente para garantir o contato completo do nível com o ágar.
- Incubação das placas
- No prazo de 15 minutos após a aplicação dos discos antibióticos, inverter as placas e incubar a 37 °C durante a noite.
- Interpretação dos resultados
- Medir o diâmetro do ZOI em milímetros (mm) e interpretar de acordo com os valores do ponto de interrupção indicados pelo EUCAST11. Veja os dois exemplos dados abaixo.
- O ponto de ruptura do diâmetro da zona (mm) para um disco antibiótico ciprofloxacina (5 μg) para Enterobacterales é S ≥ 25 e R < 22, o que significa que é considerado sensível (S) se o ZOI ≥ 25 mm, enquanto é resistente (R) se o ZOI < 22 mm. Se o diâmetro do ZOI cair entre 22 e 25, o isolado é considerado intermediário (I).
- O ponto de ruptura do diâmetro da zona (mm) para um disco antibiótico cloranfenicol (30 μg) para Staphylococcus spp. é S ≥ 18 e R < 18, o que significa que é considerado sensível se o ZOI ≥ 18 mm, enquanto é resistente se o ZOI < 18 mm.
- Determine o índice de resistência múltipla a antibióticos (MAR) encontrando a proporção do número de antibióticos aos quais o isolado é resistente ao número total de antibióticos aos quais o isolado está exposto.
NOTA: Para o controle de qualidade, E. coli ATCC 25922 e S. áureo ATCC 29213 são usados como cepas de referência seguindo o protocolo como na etapa 4.
- Medir o diâmetro do ZOI em milímetros (mm) e interpretar de acordo com os valores do ponto de interrupção indicados pelo EUCAST11. Veja os dois exemplos dados abaixo.
5. Detecção baseada em PCR de genes de resistência a antibióticos nos isolados
- Utilizar um protocolo de PCR padrão para a identificação de ARGs nos isolados. Preparar o molde de ADN utilizando o protocolo indicado no passo 3.1.
NOTA: As condições de ciclagem por PCR utilizadas neste estudo foram de 94 °C por 10 min, seguidas por 35 ciclos de 94 °C por 30 s, recozimento por 30 s na temperatura apropriada (conforme padronizado para cada conjunto de primer), extensão a 72 °C por 40 s e extensão final a 72 °C por 5 min. A mistura de reação está descrita na Tabela 4. A lista de ARGs, primers e temperaturas de recozimento é apresentada na Tabela 5. - Para resolver, visualizar e verificar a pureza dos amplificadores, siga as etapas 3.2.3-3.2.10.
6. Análise metagenômica completa do DNA para a identificação da diversidade bacteriana total e a detecção de ARGs no metagenoma
- Extração do DNA total (metagenoma) da amostra de água
- Extraia o DNA metagenômico das amostras de água filtrada.
NOTA: No presente estudo, o DNA metagenômico (DNA total) foi extraído das amostras de água filtrada usando o kit de isolamento de DNA referenciado seguindo o protocolo do fabricante (consulte a Tabela de Materiais). - Verifique a qualidade do DNA carregando 3 μL do DNA metagenômico extraído em um gel de agarose a 0,8% e execute o gel a 80-110 V por aproximadamente 30 min.
- Verifique a presença de uma única banda intacta.
- Verifique a concentração de DNA usando um fluorômetro.
- Extraia o DNA metagenômico das amostras de água filtrada.
- Determinação da diversidade bacteriana e detecção de ARGs usando sequenciamento de DNA metagenômico completo
- Preparação da biblioteca e amplificação por PCR:
- Prepare uma biblioteca de sequenciamento final emparelhada usando o kit de preparação da biblioteca de DNA referenciada (consulte a Tabela de Materiais).
- Prepare o DNA para a ligadura do adaptador, pegando 200 ng de DNA e cortando-o mecanicamente em fragmentos menores, seguido por uma etapa contínua de reparo final na qual um "A" é adicionado às extremidades de 3'.
- Dependendo da plataforma usada para sequenciamento, ligue adaptadores específicos para ambas as extremidades dos fragmentos de DNA.
Observação : sequências cruciais para vincular bibliotecas de código de barras duplas a uma célula de fluxo para sequenciamento estão presentes nesses adaptadores. Isso permite a amplificação por PCR dos fragmentos ligados ao adaptador e a ligação dos primers de sequenciamento padrão. - Para verificar a qualidade e a quantidade, analise a biblioteca amplificada usando um chip de DNA de alta sensibilidade, de acordo com as instruções do fabricante.
- Geração e sequenciamento de clusters:
- Carregue a biblioteca amplificada na plataforma de sequenciamento apropriada para geração de cluster e sequenciamento subsequente.
NOTA: As moléculas da biblioteca ligam-se aos oligos do adaptador complementar na célula de fluxo final emparelhada. Durante o sequenciamento, os fios dianteiros são seletivamente clivados após a ressíntese do fio reverso. Essa fita reversa copiada é então sequenciada a partir da extremidade oposta do fragmento.
- Carregue a biblioteca amplificada na plataforma de sequenciamento apropriada para geração de cluster e sequenciamento subsequente.
- Análise bioinformática:
- Gere andaimes a partir de dados de alta qualidade usando a plataforma apropriada.
- Submeter estes andaimes à análise bioinformática para a classificação taxonómica e identificação dos ARGs.
NOTA: O fluxo de trabalho para toda a análise metagenômica de DNA para a identificação da diversidade bacteriana total e a detecção de ARGs no metagenoma é dado na Figura 1. Um fluxograma da metodologia completa descrita no manuscrito é apresentado na Figura 2.
- Preparação da biblioteca e amplificação por PCR:
Access restricted. Please log in or start a trial to view this content.
Resultados
Carga bacteriana total e contagem de bactérias resistentes a antibióticos (AR)
A enumeração da carga bacteriana total foi realizada espalhando diluições de 10−4 a 10−6 vezes das amostras de água em Agar R2A, meio modificado. Para a enumeração da contagem de bactérias AR, diluições de 10−3 a 10−6 vezes foram espalhadas em placas de meio contendo antibióticos (Figura 3). As contagens de bactérias totais e de ...
Access restricted. Please log in or start a trial to view this content.
Discussão
A coleta e o processamento da amostra desempenham um papel significativo e podem afetar os resultados e a interpretação do estudo. Assim, para descartar a variabilidade nas amostras, é importante realizar a amostragem em vários locais do corpo de água doce em estudo. Manter condições ambientais assépticas adequadas ao manusear essas amostras pode evitar a contaminação. Além disso, para evitar alterações na composição bacteriana que possam influenciar a qualidade e a quantidade de ácidos nucleicos extraíd...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm interesses conflitantes a divulgar.
Agradecimentos
Este trabalho foi parcialmente apoiado por doações financeiras do Departamento de Ciência e Tecnologia-Promoção da Pesquisa Universitária e Excelência Científica (DST-PURSE) Esquema da Universidade de Mumbai. Devika Ghadigaonkar trabalhou como bolsista do projeto sob o esquema. É reconhecida a ajuda técnica prestada por Harshali Shinde, investigador sénior do Departamento de Ciência e Tecnologia-Conselho de Investigação em Ciência e Engenharia (DST-SERB): CRG/2018/003624.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 100 bp DNA ladder | Himedia | MBT049-50LN | For estimation of size of the amplicons |
| 2x PCR Taq mastermix | HiMedia | MBT061-50R | For making PCR reaction mixture |
| 37 °C Incubator | GS-192, Gayatri Scientific | NA | For incubation of bacteria |
| 6x Gel Loading Buffer | HiMedia | ML015-1ML | Loading and Tracking dye which helps to weigh down the DNA sample and track the progress of electrophoresis |
| Agarose powder | Himedia | MB229-50G | For resolving amplicons during Agarose Gel Electrophoresis (AGE) |
| Ampicillin antibiotic disc | HiMedia | SD002 | For performing AST |
| Autoclave | Equitron | NA | Required for sterilization of media, glass plates, test tubes, etc |
| Bioanalyzer 2100 | Agilent Technologies | NA | To check the quality and quantity of the amplified library |
| Bisafety B2 Cabinet | IMSET | IMSET BSC-Class II Type B2 | Used for microbiological work like bacterial culturing, AST etc. |
| Cefotaxime antibiotic disc | HiMedia | SD295E-5VL | For performing AST |
| Cefotaxime antibiotic powder | HiMedia | TC352-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Ceftriaxone antibiotic disc | HiMedia | SD065 | For performing AST |
| Centrifuge Minispin | Eppendorf | Minispin Plus-5453 | Used to pellet the debris during crude DNA preparation |
| Chloramphenicol antibiotic disc | HiMedia | SD006-5x50DS | For performing AST |
| Ciprofloxacin antibiotic disc | HiMedia | SD060-5x50DS | For performing AST |
| Ciprofloxacin antibiotic powder | HiMedia | TC447-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Colorimeter | Quest | NA | For checking the OD of culture suspensions |
| Comprehensive Antibiotic Resistance Database (CARD) database | functional annotation of ARGs; https://card.mcmaster.ca/ | ||
| Cooling Shaker Incubator | BTL41 Allied Scientific | NA | For incubation of media plates for culturing bacteria |
| Deep Freezer (-40 °C) | Haier | DW40L, Haier Biomedicals | For storage of glycerol stocks |
| DNA Library Prep Kit | NEB Next Ultra DNA Library Prep Kit for Illumina | NA | Paired-end sequencing library preparation |
| EDTA | HiMedia | GRM1195-100G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Electrophoresis Apparatus | TechResource | 15 cm gel casting tray | For making the agarose gel and carrying out electrophoresis |
| Electrophoresis Power pack with electrodes | Genei | NA | For running the AGE |
| Erythromycin antibiotic disc | HiMedia | SD222-5VL | For performing AST |
| Erythromycin antibiotic powder | HiMedia | CMS528-1G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Erythromycin antibiotic powder | HiMedia | TC024-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Escherichia coli ATCC 25922 | HiMedia | 0335X-1 | Used as a control while performing AST |
| Ethidium Bromide | HiMedia | MB071-1G | Intercalating agent and visualizaion of DNA after electrophoresis under Gel Documentation System |
| Fluorometer | Qubit 2.0 | NA | For determining concentration of extracted metagenomic DNA |
| Gel Documentation System | BioRad | Used for visualizing PCR amplicons after electrophoresis | |
| Gentamicin antibiotic disc | HiMedia | SD170-5x50DS | For performing AST |
| Glacial Acetic Acid | HiMedia | AS119-500ML | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Glycerol | HiMedia | GRM1027-500ML | For making glycerol stocks |
| Imipenem antibiotic disc | HiMedia | SD073 | For performing AST |
| Kaiju Database | NA | NA | For taxonomical classification of reads; https://kaiju.binf.ku.dk/ |
| Kanamycin antibiotic disc | HiMedia | SD017-5x50DS | For performing AST |
| Kanamycin antibiotic powder | HiMedia | MB105-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Levofloxacin antibiotic disc | HiMedia | SD216-5VL | For performing AST |
| Luria Bertani broth | Himedia | M1245-500G | For enrichment of cultures |
| McFarland Standards | Himedia | R092-1No | To compare density of culture suspension |
| Molecular Biology water | HiMedia | TCL018-500ML | For making PCR reaction mixture |
| Mueller-Hinton Agar (MHA) | HiMedia | M173-500G | For performing Antibiotc Susceptibility Testing (AST) |
| Neomycin antibiotic disc | HiMedia | SD731-5x50DS | For performing AST |
| PCR Gradient Thermal Cycler | Eppendorf | Mastercycler Nexus Gradient 230V/50-60 Hz | Used for performing PCR for amplification of 16S rRNA region and various Antibiotic Resistance genes |
| Primers | Xcelris | NA | For PCR amplication |
| R2A Agar, Modified | HiMedia | M1743 | For preparation of media plates for isolation of total and antibiotic resistant (AR) bacterial load |
| Scaffold generation | CLC Genomics Workbench 6.0 | NA | For generation of scaffolds |
| Sequencer | Illumina platform (2 x 150 bp chemistry) | NA | Sequencing of amplified library |
| Sodium Chloride | HiMedia | TC046-500G | For preparation of 0.85% saline for serially diluting the water sample |
| Soil DNA isolation Kit | Xcelgen | NA | For extraction of whole metagenomic DNA from the filtered water sample |
| Staphylococcus aureus subsp. aureus ATCC 29213 | HiMedia | 0365P | Used as a control while performing AST |
| Taxonomical Classification | Kaiju ioinformatics tool | NA | For classification of reads into different taxonomic groups from phylum to genus level |
| The Comprehensive Antibiotic Resistance Database (CARD) | NA | NA | For functional annotation of ARGs |
| Tigecycline antibiotic disc | HiMedia | SD278 | For performing AST |
| Trimethoprim antibiotic disc | HiMedia | SD039-5x50DS | For performing AST |
| Tris base | HiMedia | TC072-500G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Vancomycin antibiotic powder | HiMedia | CMS217 | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Weighing Balance | Mettler Toledo | ME204 Mettler Toledo | Used for weighing media powders, reagent powders etc. |
| NA - Not Applicable |
Referências
- Prestinaci, F., Pezzotti, P., Pantosti, A. Antimicrobial resistance: A global multifaceted phenomenon. Pathogens and Global Health. 109 (7), 309-318 (2015).
- Knight, G., et al. Antimicrobial resistance and COVID-19: Intersections and implications. Elife. 10, 64139(2021).
- Ventola, C. L. The antibiotic resistance crisis: Part 1: Causes and threats. Pharmacy and Therapeutics. 40 (4), 277-283 (2015).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J. R., Rath, A. Metagenomic analysis of total microbial diversity and antibiotic resistance of culturable microorganisms in raw chicken meat and mung sprouts (Phaseolus aureus) sold in retail markets of Mumbai. India. Current Science. 113 (1), 71-79 (2017).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J., Rath, A. Characterization of multiple antibiotic resistance of culturable microorganisms and metagenomic analysis of total microbial diversity of marine fish sold in retail shops in Mumbai, India. Environmental Science and Pollution Research. 25 (7), 6228-6239 (2018).
- Czekalski, N., GascónDíez, E., Bürgmann, H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake. The ISME Journal. 8 (7), 1381-1390 (2014).
- Kraemer, S., Ramachandran, A., Perron, G. Antibiotic pollution in the environment: From microbial ecology to public policy. Microorganisms. 7 (6), 180(2019).
- Edmonds-Wilson, S., Nurinova, N., Zapka, C., Fierer, N., Wilson, M. Review of human hand microbiome research. Journal of Dermatological Science. 80 (1), 3-12 (2015).
- de Abreu, V., Perdigão, J., Almeida, S. Metagenomic approaches to analyze antimicrobial resistance: An overview. Frontiers in Genetics. 11, 575592(2021).
- Carlson, S., et al. Detection of multiresistant Salmonella typhimurium DT104 using multiplex and fluorogenic PCR. Molecular and Cellular Probes. 13 (3), 213-222 (1999).
- Breakpoint tables for interpretation of MICs and zone diameters, Version 12.0. European Committee on Antimicrobial Susceptibility Testing. , Available from: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_12.0_Breakpoint_Tables.pdf (2022).
- Bharti, R., Grimm, D. Current challenges and best-practice protocols for microbiome analysis. Briefings in Bioinformatics. 22 (1), 178-193 (2019).
- Choo, J., Leong, L., Rogers, G. Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports. 5, 16350(2015).
- Clinical and Laboratory Standards Institute. Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria that Grow Aerobically, 11th edition. , Clinical and Laboratory Standards Institute. Wayne, PA. (2015).
- Bayot, M., Bragg, B. Antimicrobial Susceptibility Testing. StatPearls. , StatPearls Publishing. Treasure Island, FL. (2021).
- Joseph, A. A., Odimayo, M. S., Olokoba, L. B., Olokoba, A. B., Popoola, G. O. Multiple antibiotic resistance index of Escherichia coli isolates in a tertiary hospital in South-West Nigeria. Medical Journal of Zambia. 44 (4), 225-232 (2017).
- Lorenz, T. Polymerase chain reaction: Basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998(2012).
- Rolin, J. Food and human gut as reservoirs of transferable antibiotic resistance encoding genes. Frontiers in Microbiology. 4, 173(2013).
- Racewicz, P., et al. Prevalence and characterisation of antimicrobial resistance genes and class 1 and 2 integrons in multiresistant Escherichia coli isolated from poultry production. Scientific Reports. 12, 6062(2022).
- Gebreyes, W., Thakur, S. Multidrug-resistant Salmonella enterica serovar Muenchen from pigs and humans and potential interserovar transfer of antimicrobial resistance. Antimicrobial Agents and Chemotherapy. 49 (2), 503-511 (2005).
- Li, L., et al. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui Province, China. PLoS One. 9 (8), 104356(2014).
- Akers, K., et al. Aminoglycoside resistance and susceptibility testing errors in Acinetobacter baumannii-calcoaceticus complex. Journal Of Clinical Microbiology. 48 (4), 1132-1138 (2010).
- Ciesielczuk, H. Extra-intestinal pathogenic Escherichia coli in the UK: The importance in bacteraemia versus urinary tract infection, colonisation of widespread clones and specific virulence factors. , Queen Mary University of London. PhD thesis (2015).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados