このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
水系抗生物質耐性菌の単離・同定と抗生物質耐性遺伝子の分子特性評価
要約
ここでは、水からの抗生物質耐性菌の分離と同定、およびそれらの抗生物質耐性遺伝子(ARG)の分子特性評価のための詳細なプロトコルを紹介します。培養ベースおよび非培養ベース(メタゲノム解析)技術の使用は、インドのムンバイからの淡水に存在する細菌の多様性全体とさまざまなARGの総プールに関する完全な情報を提供します。
要約
淡水域に関連する微生物叢を介した抗生物質耐性(AR)の発生と拡散は、世界的な健康上の大きな懸念事項です。本研究では、淡水サンプルを収集し、従来の培養ベースの技術とハイスループット培養に依存しないメタゲノムアプローチの両方を使用して、細菌の多様性とAR遺伝子(ARG)全体に関して分析しました。この論文は、淡水サンプルから総および抗生物質耐性の培養可能な細菌を列挙し、培養可能な分離株の表現型および遺伝子型耐性を決定するための体系的なプロトコルを提示します。さらに、淡水サンプルから抽出された全メタゲノムDNAの全メタゲノム解析を使用して、非培養細菌を含む全体的な細菌の多様性を特定し、水域内のさまざまなARG(レジストーム)の総プールを同定したことを報告します。これらの詳細なプロトコルに従って、9.6 × 10 5-1.2 × 109 CFU / mLの範囲で高い抗生物質耐性菌負荷が観察されました。ほとんどの分離株は、セフォタキシム、アンピシリン、レボフロキサシン、クロラムフェニコール、セフトリアキソン、ゲンタマイシン、ネオマイシン、トリメトプリム、シプロフロキサシンなどの複数の試験済み抗生物質に耐性があり、複数の抗生物質耐性(MAR)指数は≥0.2であり、分離株の耐性が高いことを示しています。16S rRNAシーケンシングにより、肺炎桿菌などの潜在的なヒト病原体、およびコマモナス属、ミクロコッカス属、アルスロバクター属、アエロモナス属などの日和見細菌が特定されました。 分離株の分子特性評価は、blaTEM、blaCTX-M(β-ラクタム)、aadA、aac(6')-Ib(アミノグリコシド)、およびdfr1(トリメトプリム)などのさまざまなARGの存在を示し、これもメタゲノムDNA分析全体で確認されました。抗生物質排出ポンプをコードする他のARGの高い有病率-mtrA、macB、mdtA、acrD、β-ラクタマーゼ-SMB-1、VIM-20、ccrA、ampC、blaZ、クロラムフェニコールアセチルトランスフェラーゼ遺伝子catB10、およびリファンピシン耐性遺伝子rphB-もメタゲノムDNAで検出されました。この研究で議論されたプロトコルの助けを借りて、多様なAR表現型および遺伝子型形質を有する水系MAR細菌の存在を確認しました。したがって、全メタゲノムDNA分析は、水域の全体的なAR状態を決定するための従来の培養ベースの技術を補完する技術として使用できます。
概要
薬剤耐性(AMR)は、最も差し迫った地球規模の問題の1つとして認識されています。AMRの急速な進化とその世界的な普及は、それに関連する医療費の観点から、人間の健康と世界経済にとって最大の脅威の1つです1。抗生物質の過剰使用と誤用は、ARの増加につながっています。これはCOVID-19のパンデミックによって強調されており、その間、多くの場合、影響を受けた患者のAMRにより、関連する二次感染症の治療が大幅に損なわれました2。人間による抗生物質の直接使用/誤用に加えて、農業および畜産における抗生物質の過剰使用および誤用、および水域を含む環境への不適切な排出が大きな懸念事項です3。細菌における新しい耐性形質と多剤耐性の台頭は、ARの開発とその普及につながる要因のより良い理解の必要性を緊急に浮き彫りにしています。プラスミドなどの移動性遺伝要素に複数のAR遺伝子(ARG)を運ぶことが多い複数の抗生物質耐性菌は、これらの耐性遺伝子を潜在的なヒト病原体を含む非耐性微生物に移す可能性があるため、最後の手段の抗生物質でさえ治療できないスーパーバグの出現につながります4。これらの複数の抗生物質耐性菌は、水生態系に存在する場合、魚、カニ、軟体動物などの汚染された水ベースの食品を消費することにより、人間の腸に直接侵入する可能性があります。以前の研究では、天然に存在する水系におけるAR細菌の拡散は、飲料水を含む他の水供給にも到達する可能性があり、したがって、人間の食物連鎖に入る可能性があることが示されています5,6,7。
本研究の目的は、培養ベースと非培養ベースの組み合わせを使用した包括的なプロトコルを提供することです(全メタゲノム分析)技術 完全な情報を取得する インドのムンバイの水域に存在する細菌の多様性とさまざまなARGの総プール。従来、培養ベースの技術は、水域における細菌の多様性を研究するために使用されてきた。培養可能な微生物は、どのニッチでも微生物叢全体のごく一部しか構成していないため、細菌の多様性の全体的な状態と、あらゆるサンプルに蔓延しているさまざまな耐性形質をよりよく理解するには、さまざまな培養ベースおよび培養に依存しない技術を並行して使用する必要があります。そのような堅牢で信頼性の高い培養に依存しない技術の1つは、全メタゲノムDNA分析です。このハイスループットメソッドは、細菌の多様性やさまざまなARGの機能アノテーションに関するさまざまな研究で成功裏に利用されています8,9。この手法は、メタゲノム(サンプル中の総遺伝物質)をさまざまな分析の出発材料として使用するため、培養に依存しません。本研究のプロトコルは、全メタゲノムDNA分析に使用して、水サンプル中の細菌全体の多様性とさまざまなARG(レジストーム)に関する情報を取得できます。
プロトコル
1.サンプルの収集と処理
- サンプル収集
- 適切な量の水サンプルを滅菌サンプル容器に収集し、容器の3/4以下が満たされていることを確認します。
- 採取後できるだけ早く無菌条件下でサンプルを実験室に輸送し、直ちに処理します。
- サンプル処理
- 滅菌モスリン布で水サンプルを無菌ろ過し、粒子状物質を除去します。
- さらなる分析のために、ろ過水の適切な段階希釈を実行します。
2. 総細菌量と抗生物質耐性菌数の推定
- 総細菌量の決定
- 18.12 gのR2A寒天、改質粉末を1,000 mLの二重蒸留水に懸濁し、加熱して溶解します。溶解した混合物を121°C、15psiで20分間オートクレーブします。 R2A寒天培地を調製し、 適量のオートクレーブ処理された混合物を滅菌ペトリプレートに注ぐことにより、プレートを変更します(たとえば、約20 mLのオートクレーブ滅菌培地を90 mmの滅菌ペトリプレートに加えます)。
- ろ過水サンプルの適切な希釈液100 μLをR2A寒天培地上に均一に広げ、培地が固まったら修正プレート。実験を二重に実行します。
- 上記のすべてのプレートを35〜37°Cで48時間インキュベートします(分離に使用する培地に応じて温度とインキュベーション時間を変えます)。
- 式 (1) を使用して、ミリリットルあたりのコロニー形成単位 (CFU/mL) で総細菌負荷を表します。
 (1)
(1)
- AR細菌数の決定
- 手順 2.1.1-2.1.4 に従います。ただし、R2A寒天培地の代わりに、R2A寒天培地を使用し、セフォタキシム(3μg/mL)、シプロフロキサシン(0.5μg/mL)、エリスロマイシン(20μg/mL)、カナマイシン(15μg/mL)、およびバンコマイシン(3μg/mL)の5つの異なる抗生物質を個別に補充した改変プレートを使用する。
- 手順2.2.1で述べたように、最終的な抗生物質濃度を達成するために、20 mLの滅菌溶融R2A寒天培地(溶融R2A寒天培地の温度、≤40°Cで修正)を含むチューブに抗生物質を別々に追加します。
- 均一に混合するために渦巻き、寒天が固まる前に滅菌ペトリプレートに注ぎます。実験を二重に実行します。
- 上記のすべてのプレートを35〜37°Cで48時間インキュベートします(異なる培地を使用する場合、温度とインキュベーション時間は異なる場合があります)。
- 抗生物質の品質管理と有効性の確認のために、大腸菌ATCC 25922および黄色ブドウ球菌ATCC 29213株の細菌懸濁液100 μLをそれぞれの抗生物質含有R2A寒天培地、修飾プレートに広げます(接種に使用される新鮮な培養物の密度が600 nmでOD = 0.5であることを確認してください)。
- ステップ2.1.4で説明されているように、CFU / mLの観点から抗生物質耐性菌数を決定します。
- 分離株のグリセロールストック
- 形態学的に異なるARコロニーを選択します。
- 単一の単離されたコロニーを、それぞれの抗生物質を含有する滅菌ルリア・ベルタニブロス2mLに懸濁する(例えば、コロニーがセフォタキシムを含有するプレートから選択される場合、ステップ2.3.1のコロニーを、それぞれの濃度のセフォタキシムを含有する滅菌ルリア・ベルタニブロスに接種する)。
- 接種したチューブを37°C、80rpmでOD600 が0.5に達するまでインキュベートします。
- ステップ2.3.3の培養懸濁液750 μLを無菌条件下で滅菌100%グリセロール250 μLに混合することにより、分離株のグリセロールストックを調製します。
- グリセロールストックは、さらに分析されるまで-80°Cで保存してください。
注:グリセロールストックから培養物を復活させるには、グリセロールストックを4°Cで解凍します。 このストックを、それぞれの抗生物質を含む2 mLの滅菌ルリアベルターニブロスにループで接種し、増殖させます。.
3. 16S rRNA遺伝子シーケンシングによる培養可能菌の同定
- PCR用の分離株からのDNAテンプレートの調製
注:細菌からの粗DNAの単離のためのPCR用のDNAテンプレートの調製について記載されているプロトコルは、Carlsonらによって与えられています10。- 滅菌つまようじを使用して、ペトリプレート上で成長している分離物の単一の分離された純粋なコロニーを取ります。細菌コロニーを滅菌マイクロ遠心チューブ内の滅菌二重蒸留水100 μLに懸濁し、10分間煮沸します。
- 懸濁液を10,000 × g で2分間遠心分離して破片をペレット化し、上清を新しい滅菌マイクロ遠心チューブに移して粗DNAテンプレートとして使用します。
- 16S rRNA遺伝子のV3領域の標的PCR増幅とシーケンシング
- 表1に記載されているように、PCR増幅用のPCRチューブで40 μLの反応混合物を調製します。
注:DNA調製は、汚染の可能性を最小限に抑えながら、氷のブロックで実行する必要があります(試薬を取り扱うときは手袋を着用し、70%エタノールで作業面を完全に洗浄してください)。 - チューブをサーマルブロックに入れ、PCRサーマルサイクラーで適切なプログラムを実行します。標準化されたPCRサイクリング条件および16S rRNA遺伝子のV3領域の増幅のためのプライマー情報については 、表2 を参照されたい。
- アンプリコンの分解と可視化のために、アガロースゲル電気泳動(AGE)を実施してください。増幅PCR産物10 μLと6xゲルローディングバッファー2 μLを混合し(表3)、この混合物を10 mg/mLのエチジウムブロマイド(EtBr)を含む1.5 μLの1.5%アガロースゲル(100 mLの1x TAEバッファーに1.5 gのアガロース粉末を溶解[表3])のウェルにロードします。
注意: EtBrは強力な発がん性物質です。EtBrおよびEtBrを含むジェルを取り扱うときは、常に手袋を着用する必要があります。 - アンプリコンのサイズを推定するためのDNAラダーを追加します。
- 80〜100 VのTAEタンクバッファー中でゲルの電気泳動を実行します。
- トラッキング色素がゲルの3/4を走ったら、電気泳動を停止し、UVトランスイルミネーターの下でアンプリコンバンドを可視化します。
- 16S rRNA遺伝子シーケンシング用のPCR産物(アンプリコン)を使用して、分離株を特定します。
- アンプリコンを式(2)を用いた分光光度分析に供して定量する。
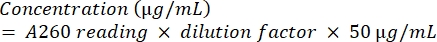 (2)
(2) - DNAの純度を確認するには、A260 / A280の比率を計算します。
注: 理想的には、この数値は 1.5 より大きく、できれば 1.8 から 2.0 の間である必要があります。 - 分離株を特定するには、適切なアライメント検索ツールを使用して、取得した配列を配列データベースと比較します。
- 表1に記載されているように、PCR増幅用のPCRチューブで40 μLの反応混合物を調製します。
4.抗生物質感受性試験を用いた分離株中の抗生物質耐性の検出
注:このプロトコルは、椎間板拡散による抗生物質感受性試験(AST)の方法を説明しています。以下の抗生物質ディスクを使用した:セフォタキシム(5μg)、アンピシリン(10μg)、レボフロキサシン(5μg)、クロラムフェニコール(30μg)、チゲサイクリン(15μg)、セフトリアキソン(30μg)、イミペネム(10μg)、ゲンタマイシン(10μg)、ネオマイシン(10μg)、トリメトプリム(5μg)、およびシプロフロキサシン(5μg)。
- ASTのための接種材料の調製
- Luria-Bertaniブロス(抗生物質なし)などの滅菌非選択培地2 mL中の滅菌ループを使用して、単一の単離精製ARコロニーを無菌的に接種し、37°Cで80rpmで一晩インキュベートします。
- 100〜150 μLの一晩培養培養物(約OD600 = 1.8-2.0)を2 mLの新鮮な非選択的Luria-Bertaniブロス培地に取り、2〜4時間インキュベートします(OD600 が0.4〜0.5に達するまで)。
- 培養液の密度が0.5マクファーランド標準(約OD600 = 0.1)に等しくなるように、滅菌0.85%生理食塩水を使用してこの新たに成長した培養懸濁液を希釈し、これはおよそ1〜2 ×10 8 細胞/ mLに相当します。
- 均一な細胞分布のために細菌懸濁液を穏やかに混合する。
- 上記の懸濁液は、希釈から15分以内に使用してください。.
- 寒天プレートの接種
- 1,000 mLの二重蒸留水にMHA38 gを混合してASTを行うためのミューラーヒントン寒天培地(MHA)プレートを準備し、加熱して溶解します。溶解した混合物を121°C、15psiで15分間オートクレーブします。
- プレート内のMHAの深さが4 mm(プレートあたり25 mLの培地)であることを確認します。
- 同時に、抗生物質ディスクを冷凍庫から取り出し、室温まで温めます。
注意: 抗生物質ディスクは、最初に4°Cでディスクを解凍し、その後室温で解凍して徐々に解凍し、ディスク上の結露の潜在的な危険性を減らす必要があります。 - 無菌条件下で、滅菌綿棒をステップ4.1で調製した接種材料に浸し、プレートの過剰接種を避けるために余分な懸濁液を取り除きます。
- MHAプレートの上部から始めて、端から端まで前後に培養液をプレートに均等に広げます。拭き取りながらプレートを60°回転させます。
- 抗生物質ディスクの応用
- 火炎滅菌された鉗子の助けを借りて、抗生物質ディスクを接種されたMHAプレートに無菌的に移し、ディスクを軽く押して寒天との完全な水平接触を確保します。
注:この手順は、プレート上の培養物の接種から15分以内に行わなければなりません。 - 阻害ゾーンの重なりを避けるために、生物、使用する抗生物質、およびプレートのサイズを考慮して、適切な数の抗生物質ディスクを寒天プレートに配置します。
注意: 90mmの円形プレートに4〜5枚のディスクを収容できます。
- 火炎滅菌された鉗子の助けを借りて、抗生物質ディスクを接種されたMHAプレートに無菌的に移し、ディスクを軽く押して寒天との完全な水平接触を確保します。
- プレートのインキュベーション
- 抗生物質ディスクの適用から15分以内に、プレートを反転させ、37°Cで一晩インキュベートします。
- 結果の解釈
- ZOI の直径をミリメートル (mm) 単位で測定し、EUCAST11 で指定されたブレークポイント値に従って解釈します。以下の2つの例を参照してください。
- エンテロバクテラレスのシプロフロキサシン系抗生物質ディスク(5μg)のゾーン直径ブレークポイント(mm)はS ≥ 25およびR < 22であり、これはZOIが25mm≥場合は感度(S)であると見なされ、ZOIが22mmの場合は耐性(R)である<であることを意味します。ZOI直径が22〜25の場合、分離物は中間(I)と見なされます。
- ブドウ球菌属のクロラムフェニコール系抗生物質ディスク(30μg)のゾーン直径ブレークポイント(mm)はS ≥ 18およびR < 18であり、ZOIが18 mmであれば感度がある≥、ZOIが18 mm<場合は耐性があると考えられます。
- 分離株が曝露される抗生物質の総数に対する分離株が耐性である抗生物質の数の比率を見つけることにより、複数の抗生物質耐性(MAR)指数を決定します。
注:品質管理のために、 大腸菌 ATCC 25922および S。 黄色ブドウ球菌 ATCC 29213は、ステップ4のようなプロトコルに従って参照株として使用されます。
- ZOI の直径をミリメートル (mm) 単位で測定し、EUCAST11 で指定されたブレークポイント値に従って解釈します。以下の2つの例を参照してください。
5. PCRによる分離株中の抗生物質耐性遺伝子の検出
- 分離株中のARGの同定には、標準的なPCRプロトコルを使用してください。ステップ3.1で与えられたプロトコルを使用してDNAテンプレートを準備します。
注:この研究で使用されたPCRサイクル条件は、94°Cで10分間、続いて94°Cで30秒間の35サイクル、適切な温度で30秒間のアニーリング(各プライマーセットで標準化)、72°Cで40秒間の伸長、および72°Cで5分間の最終延長でした。反応混合物を 表4に記載する。ARG、プライマー、およびアニーリング温度のリストを 表5に示します。 - アンプリコンの純度を分解、視覚化、および確認するには、手順3.2.3〜3.2.10に従います。
6.細菌全体の多様性の同定とメタゲノム中のARGの検出のための全メタゲノムDNA分析
- 水サンプルからの全DNA(メタゲノム)の抽出
- ろ過された水サンプルからメタゲノムDNAを抽出します。
注:現在の研究では、メタゲノムDNA(総DNA)は、製造元のプロトコルに従って参照されたDNA分離キットを使用して、ろ過された水サンプルから抽出されました( 材料の表を参照)。 - 抽出したメタゲノムDNA3 μLを0.8%アガロースゲルにロードしてDNAの品質を確認し、ゲルを80-110 Vで約30分間実行します。
- 単一の無傷のバンドの存在を確認します。
- 蛍光光度計を使用してDNA濃度を確認します。
- ろ過された水サンプルからメタゲノムDNAを抽出します。
- 全メタゲノムDNAシーケンシングを用いた細菌多様性の決定とARGの検出
- ライブラリ調製とPCR増幅:
- 参照されているDNAライブラリ調製キットを使用して、ペアエンドシーケンシングライブラリを調製します(材料表を参照)。
- 200 ngのDNAを採取し、それを機械的に小さな断片にせん断した後、3'末端に「A」を追加するエンドリペアの連続ステップを行うことにより、アダプターライゲーション用のDNAを準備します。
- シーケンシングに使用するプラットフォームに応じて、DNA断片の両端に特定のアダプターをライゲーションします。
注:シーケンスのためにデュアルバーコードライブラリをフローセルにバインドするために重要なシーケンスは、これらのアダプターに存在します。これにより、アダプターライゲーションフラグメントのPCR増幅と標準シーケンシングプライマーの結合が可能になります。 - 品質と量を確認するには、製造元の指示に従って、高感度DNAチップを使用して増幅ライブラリを分析します。
- クラスターの生成と順序付け:
- 増幅されたライブラリを適切なシーケンシングプラットフォームにロードして、クラスター生成とその後のシーケンシングを行います。
注:ライブラリ分子は、ペアエンドフローセル上の相補的なアダプターオリゴに結合します。シーケンシング中、順鎖は、逆鎖の再合成後に選択的に切断される。次いで、このコピーされた逆鎖は、フラグメントの反対側の端部から配列決定される。
- 増幅されたライブラリを適切なシーケンシングプラットフォームにロードして、クラスター生成とその後のシーケンシングを行います。
- バイオインフォマティクス解析:
- 適切なプラットフォームを使用して、高品質のデータからスキャフォールドを生成します。
- これらの足場を、分類学的分類とARGの同定のためのバイオインフォマティクス分析にかけます。
注:細菌全体の多様性の同定とメタゲノム中のARGの検出のためのメタゲノムDNA分析全体のワークフローを 図1に示します。原稿に記載されている完全な方法論のフローシートを 図2に示します。
- ライブラリ調製とPCR増幅:
結果
総細菌量と抗生物質耐性(AR)細菌数
総細菌負荷の列挙は、水サンプルの10−4〜10−6倍希釈をR2A寒天培地上に広げることによって行った。AR細菌数の列挙のために、抗生物質を含む培地プレート上に10−3〜10−6倍希釈を広げました(図3)。総菌数とAR菌数をCFU/mLとして計算し、すべてのめっき実験を重複して実施した。上記のプ...
ディスカッション
サンプルの収集と処理は重要な役割を果たし、研究の結果と解釈に影響を与える可能性があります。したがって、サンプルの変動性を排除するには、調査対象の淡水域の複数の場所でサンプリングを実行することが重要です。このようなサンプルを取り扱う際に適切な無菌環境条件を維持することで、汚染を防ぐことができます。さらに、抽出された核酸の質と量に影響を与える可能性のあ?...
開示事項
著者は、開示する相反する利益を持っていません。
謝辞
この作業は、ムンバイ大学の科学技術省-大学研究および科学的卓越性促進(DST-PURSE)スキームからの財政的助成金によって部分的に支援されました。Devika Ghadigaonkarは、このスキームの下でプロジェクトフェローとして働いていました。科学技術省科学技術研究委員会(DST-SERB)プロジェクト番号:CRG/2018/003624の上級研究員であるHarshali Shindeによる技術的支援が認められています。
資料
| Name | Company | Catalog Number | Comments |
| 100 bp DNA ladder | Himedia | MBT049-50LN | For estimation of size of the amplicons |
| 2x PCR Taq mastermix | HiMedia | MBT061-50R | For making PCR reaction mixture |
| 37 °C Incubator | GS-192, Gayatri Scientific | NA | For incubation of bacteria |
| 6x Gel Loading Buffer | HiMedia | ML015-1ML | Loading and Tracking dye which helps to weigh down the DNA sample and track the progress of electrophoresis |
| Agarose powder | Himedia | MB229-50G | For resolving amplicons during Agarose Gel Electrophoresis (AGE) |
| Ampicillin antibiotic disc | HiMedia | SD002 | For performing AST |
| Autoclave | Equitron | NA | Required for sterilization of media, glass plates, test tubes, etc |
| Bioanalyzer 2100 | Agilent Technologies | NA | To check the quality and quantity of the amplified library |
| Bisafety B2 Cabinet | IMSET | IMSET BSC-Class II Type B2 | Used for microbiological work like bacterial culturing, AST etc. |
| Cefotaxime antibiotic disc | HiMedia | SD295E-5VL | For performing AST |
| Cefotaxime antibiotic powder | HiMedia | TC352-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Ceftriaxone antibiotic disc | HiMedia | SD065 | For performing AST |
| Centrifuge Minispin | Eppendorf | Minispin Plus-5453 | Used to pellet the debris during crude DNA preparation |
| Chloramphenicol antibiotic disc | HiMedia | SD006-5x50DS | For performing AST |
| Ciprofloxacin antibiotic disc | HiMedia | SD060-5x50DS | For performing AST |
| Ciprofloxacin antibiotic powder | HiMedia | TC447-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Colorimeter | Quest | NA | For checking the OD of culture suspensions |
| Comprehensive Antibiotic Resistance Database (CARD) database | functional annotation of ARGs; https://card.mcmaster.ca/ | ||
| Cooling Shaker Incubator | BTL41 Allied Scientific | NA | For incubation of media plates for culturing bacteria |
| Deep Freezer (-40 °C) | Haier | DW40L, Haier Biomedicals | For storage of glycerol stocks |
| DNA Library Prep Kit | NEB Next Ultra DNA Library Prep Kit for Illumina | NA | Paired-end sequencing library preparation |
| EDTA | HiMedia | GRM1195-100G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Electrophoresis Apparatus | TechResource | 15 cm gel casting tray | For making the agarose gel and carrying out electrophoresis |
| Electrophoresis Power pack with electrodes | Genei | NA | For running the AGE |
| Erythromycin antibiotic disc | HiMedia | SD222-5VL | For performing AST |
| Erythromycin antibiotic powder | HiMedia | CMS528-1G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Erythromycin antibiotic powder | HiMedia | TC024-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Escherichia coli ATCC 25922 | HiMedia | 0335X-1 | Used as a control while performing AST |
| Ethidium Bromide | HiMedia | MB071-1G | Intercalating agent and visualizaion of DNA after electrophoresis under Gel Documentation System |
| Fluorometer | Qubit 2.0 | NA | For determining concentration of extracted metagenomic DNA |
| Gel Documentation System | BioRad | Used for visualizing PCR amplicons after electrophoresis | |
| Gentamicin antibiotic disc | HiMedia | SD170-5x50DS | For performing AST |
| Glacial Acetic Acid | HiMedia | AS119-500ML | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Glycerol | HiMedia | GRM1027-500ML | For making glycerol stocks |
| Imipenem antibiotic disc | HiMedia | SD073 | For performing AST |
| Kaiju Database | NA | NA | For taxonomical classification of reads; https://kaiju.binf.ku.dk/ |
| Kanamycin antibiotic disc | HiMedia | SD017-5x50DS | For performing AST |
| Kanamycin antibiotic powder | HiMedia | MB105-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Levofloxacin antibiotic disc | HiMedia | SD216-5VL | For performing AST |
| Luria Bertani broth | Himedia | M1245-500G | For enrichment of cultures |
| McFarland Standards | Himedia | R092-1No | To compare density of culture suspension |
| Molecular Biology water | HiMedia | TCL018-500ML | For making PCR reaction mixture |
| Mueller-Hinton Agar (MHA) | HiMedia | M173-500G | For performing Antibiotc Susceptibility Testing (AST) |
| Neomycin antibiotic disc | HiMedia | SD731-5x50DS | For performing AST |
| PCR Gradient Thermal Cycler | Eppendorf | Mastercycler Nexus Gradient 230V/50-60 Hz | Used for performing PCR for amplification of 16S rRNA region and various Antibiotic Resistance genes |
| Primers | Xcelris | NA | For PCR amplication |
| R2A Agar, Modified | HiMedia | M1743 | For preparation of media plates for isolation of total and antibiotic resistant (AR) bacterial load |
| Scaffold generation | CLC Genomics Workbench 6.0 | NA | For generation of scaffolds |
| Sequencer | Illumina platform (2 x 150 bp chemistry) | NA | Sequencing of amplified library |
| Sodium Chloride | HiMedia | TC046-500G | For preparation of 0.85% saline for serially diluting the water sample |
| Soil DNA isolation Kit | Xcelgen | NA | For extraction of whole metagenomic DNA from the filtered water sample |
| Staphylococcus aureus subsp. aureus ATCC 29213 | HiMedia | 0365P | Used as a control while performing AST |
| Taxonomical Classification | Kaiju ioinformatics tool | NA | For classification of reads into different taxonomic groups from phylum to genus level |
| The Comprehensive Antibiotic Resistance Database (CARD) | NA | NA | For functional annotation of ARGs |
| Tigecycline antibiotic disc | HiMedia | SD278 | For performing AST |
| Trimethoprim antibiotic disc | HiMedia | SD039-5x50DS | For performing AST |
| Tris base | HiMedia | TC072-500G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Vancomycin antibiotic powder | HiMedia | CMS217 | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Weighing Balance | Mettler Toledo | ME204 Mettler Toledo | Used for weighing media powders, reagent powders etc. |
| NA - Not Applicable |
参考文献
- Prestinaci, F., Pezzotti, P., Pantosti, A. Antimicrobial resistance: A global multifaceted phenomenon. Pathogens and Global Health. 109 (7), 309-318 (2015).
- Knight, G., et al. Antimicrobial resistance and COVID-19: Intersections and implications. Elife. 10, 64139 (2021).
- Ventola, C. L. The antibiotic resistance crisis: Part 1: Causes and threats. Pharmacy and Therapeutics. 40 (4), 277-283 (2015).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J. R., Rath, A. Metagenomic analysis of total microbial diversity and antibiotic resistance of culturable microorganisms in raw chicken meat and mung sprouts (Phaseolus aureus) sold in retail markets of Mumbai. India. Current Science. 113 (1), 71-79 (2017).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J., Rath, A. Characterization of multiple antibiotic resistance of culturable microorganisms and metagenomic analysis of total microbial diversity of marine fish sold in retail shops in Mumbai, India. Environmental Science and Pollution Research. 25 (7), 6228-6239 (2018).
- Czekalski, N., GascónDíez, E., Bürgmann, H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake. The ISME Journal. 8 (7), 1381-1390 (2014).
- Kraemer, S., Ramachandran, A., Perron, G. Antibiotic pollution in the environment: From microbial ecology to public policy. Microorganisms. 7 (6), 180 (2019).
- Edmonds-Wilson, S., Nurinova, N., Zapka, C., Fierer, N., Wilson, M. Review of human hand microbiome research. Journal of Dermatological Science. 80 (1), 3-12 (2015).
- de Abreu, V., Perdigão, J., Almeida, S. Metagenomic approaches to analyze antimicrobial resistance: An overview. Frontiers in Genetics. 11, 575592 (2021).
- Carlson, S., et al. Detection of multiresistant Salmonella typhimurium DT104 using multiplex and fluorogenic PCR. Molecular and Cellular Probes. 13 (3), 213-222 (1999).
- Breakpoint tables for interpretation of MICs and zone diameters, Version 12.0. European Committee on Antimicrobial Susceptibility Testing Available from: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_12.0_Breakpoint_Tables.pdf (2022)
- Bharti, R., Grimm, D. Current challenges and best-practice protocols for microbiome analysis. Briefings in Bioinformatics. 22 (1), 178-193 (2019).
- Choo, J., Leong, L., Rogers, G. Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports. 5, 16350 (2015).
- Clinical and Laboratory Standards Institute. . Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria that Grow Aerobically, 11th edition. , (2015).
- Bayot, M., Bragg, B. Antimicrobial Susceptibility Testing. StatPearls. , (2021).
- Joseph, A. A., Odimayo, M. S., Olokoba, L. B., Olokoba, A. B., Popoola, G. O. Multiple antibiotic resistance index of Escherichia coli isolates in a tertiary hospital in South-West Nigeria. Medical Journal of Zambia. 44 (4), 225-232 (2017).
- Lorenz, T. Polymerase chain reaction: Basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Rolin, J. Food and human gut as reservoirs of transferable antibiotic resistance encoding genes. Frontiers in Microbiology. 4, 173 (2013).
- Racewicz, P., et al. Prevalence and characterisation of antimicrobial resistance genes and class 1 and 2 integrons in multiresistant Escherichia coli isolated from poultry production. Scientific Reports. 12, 6062 (2022).
- Gebreyes, W., Thakur, S. Multidrug-resistant Salmonella enterica serovar Muenchen from pigs and humans and potential interserovar transfer of antimicrobial resistance. Antimicrobial Agents and Chemotherapy. 49 (2), 503-511 (2005).
- Li, L., et al. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui Province, China. PLoS One. 9 (8), 104356 (2014).
- Akers, K., et al. Aminoglycoside resistance and susceptibility testing errors in Acinetobacter baumannii-calcoaceticus complex. Journal Of Clinical Microbiology. 48 (4), 1132-1138 (2010).
- Ciesielczuk, H. . Extra-intestinal pathogenic Escherichia coli in the UK: The importance in bacteraemia versus urinary tract infection, colonisation of widespread clones and specific virulence factors. , (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved