需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
水性抗生素耐药细菌的分离和鉴定及其抗生素耐药基因的分子表征
摘要
在这里,我们提出了一个详细的方案,用于从水中分离和鉴定抗生素耐药细菌及其抗生素抗性基因(ARG)的分子特征。使用基于培养物和非基于培养物(宏基因组分析)技术提供了有关印度孟买淡水中存在的细菌多样性和不同ARG的总库的完整信息。
摘要
抗生素耐药性(AR)通过与淡水水体相关的微生物群的发展和传播是一个主要的全球健康问题。在本研究中,使用传统的基于培养的技术和高通量非培养物的宏基因组方法收集和分析淡水样品的总细菌多样性和AR基因(ARG)。本文提出了一种系统方案,用于从淡水样品中计数总细菌和抗生素耐药菌,并测定可培养分离株中的表型和基因型耐药性。此外,我们报告了使用从淡水样品中提取的总宏基因组DNA的全宏基因组分析来鉴定整体细菌多样性,包括不可培养的细菌,以及鉴定水体中不同ARG(抗性组)的总库。按照这些详细的方案,我们观察到抗生素耐药细菌负荷在9.6×10 5-1.2×109 CFU/mL范围内。大多数分离株对多种测试的抗生素耐药,包括头孢噻肟、氨苄西林、左氧氟沙星、氯霉素、头孢曲松、庆大霉素、新霉素、甲氧苄啶和环丙沙星,多种抗生素耐药性(MAR)指数为≥0.2,表明分离株的耐药性水平很高。16S rRNA测序鉴定了潜在的人类病原体,如肺炎克雷伯菌,以及机会性细菌,如Comamonas spp.、Micrococcus spp.、Arthrobacter spp.和Aeromonas spp.。 分离株的分子表征表明存在各种ARG,例如blaTEM,blaCTX-M(β-内酰胺),aadA,aac(6')-Ib(氨基糖苷类)和dfr1(甲氧苄啶),这也得到了全宏基因组DNA分析的证实。在宏基因组DNA中还检测到编码抗生素外排泵的其他ARGs-mtrA,macB,mdtA,acrD,β-内酰胺酶-SMB-1,VIM-20,ccrA,ampC,blaZ,氯霉素乙酰转移酶基因catB10和利福平耐药基因rphB-的高患病率。在本研究中讨论的方案的帮助下,我们确认了具有不同AR表型和基因型性状的水性MAR细菌的存在。因此,全宏基因组DNA分析可以用作传统基于培养的技术的补充技术,以确定水体的整体AR状态。
引言
抗菌素耐药性(AMR)已被确定为最紧迫的全球问题之一。抗微生物药物耐药性的快速演变及其在世界范围内的传播是人类健康和全球经济面临的最大威胁之一,就与之相关的健康成本而言1.抗生素的过度使用和滥用导致AR增加。COVID-19大流行突出了这一点,在此期间,在许多情况下,由于受影响患者的AMR,相关继发感染的治疗受到巨大损害2。除了人类直接使用/滥用抗生素外,在农业和畜牧业中过度使用和滥用抗生素以及不适当地排放到环境(包括水体)中也是一个主要问题3。细菌中新的耐药性状和多重耐药性的兴起迫切需要更好地了解导致AR发展及其传播的因素。多种抗生素耐药细菌,通常在质粒等移动遗传元件上携带多个AR基因(ARG),可以将这些耐药基因转移到非耐药微生物,包括潜在的人类病原体,从而导致超级细菌的出现,即使是最后的抗生素也无法治愈4。这些多重抗生素耐药细菌,如果存在于水生态系统中,可以通过食用受污染的水基食物(如鱼、螃蟹和软体动物)直接进入人体肠道。先前的研究表明,AR细菌在天然水系统中的传播也可以到达其他供水,包括饮用水,从而可以进入人类食物链5,6,7。
本研究的目的是使用基于培养物和非基于培养物(全宏基因组分析)技术的组合来提供全面的方案,以获得有关印度孟买水体中存在的细菌多样性和不同ARG的总库的完整信息。传统上,基于培养的技术已被用于研究水体中的细菌多样性。由于可培养微生物仅占任何生态位中总微生物群的一小部分,为了更好地了解细菌多样性的整体状况以及任何样品中普遍存在的各种耐药性状,必须同时使用各种基于培养和独立于培养的技术。一种强大而可靠的非培养技术是全宏基因组DNA分析。这种高通量方法已成功用于细菌多样性的各种研究或各种ARG的功能注释8,9。该技术使用宏基因组(样品中的总遗传物质)作为各种分析的起始材料,因此与培养无关。本研究中的方案可用于全宏基因组DNA分析,以获取有关水样中总细菌多样性和各种ARG(抗性组)的信息。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 样品采集和处理
- 样品采集
- 在无菌样品容器中收集适当体积的水样,确保填充不超过容器的 3/4。
- 收集后尽快将样品在无菌条件下运输到实验室,并立即进行处理。
- 来样加工
- 通过无菌平纹细布无菌过滤水样,以去除任何颗粒物质。
- 对过滤水进行适当的连续稀释以进行进一步分析。
2. 估计细菌总载量和抗生素耐药细菌计数
- 测定总细菌负荷
- 将 18.12 g R2A 琼脂、改性粉末悬浮在 1,000 mL 双蒸水中,并通过加热溶解混合物。将溶解的混合物在121°C,15psi高压灭菌20分钟。 通过将适量的高压灭菌混合物倒入无菌培养皿中来制备R2A琼脂,改性板(例如,将约20mL高压灭菌的无菌培养基加入90mm无菌培养皿中)。
- 将 100 μL 过滤水样品的适当稀释液均匀地铺在 R2A 琼脂上,一旦培养基凝固,就改性板。执行实验一式两份。
- 将上述所有板在35-37°C孵育48小时(根据用于分离的培养基改变温度和孵育时间)。
- 使用公式(1)以每毫升菌落形成单位(CFU/mL)表示总细菌负荷:
 (1)
(1)
- AR 细菌计数的测定
- 按照步骤 2.1.1-2.1.4 操作。然而,代替R2A琼脂,改良板,使用R2A琼脂,改性平板单独补充五种不同的抗生素,即头孢噻肟(3μg/ mL),环丙沙星(0.5μg/ mL),红霉素(20μg/ mL),卡那霉素(15μg/ mL)和万古霉素(3μg/ mL)。
- 将抗生素分别加入含有20mL无菌熔融R2A琼脂的管中,改性(用熔融R2A琼脂的温度,在≤40°C下改性)以达到步骤2.2.1中提到的最终抗生素浓度。
- 漩涡均匀混合,并在琼脂凝固之前倒入无菌培养皿上。执行实验一式两份。
- 将上述所有板在35-37°C孵育48小时(如果使用不同的培养基,温度和孵育时间可能会有所不同)。
- 为了质量控制和检查抗生素的功效,将100μL 大肠杆菌 ATCC 25922和 金黄色葡萄球菌 ATCC 29213菌株的细菌悬浮液散布到各自含有抗生素的R2A琼脂,修饰的平板上(确保用于接种的新鲜培养物的密度在600nm处为OD = 0.5)。
- 如步骤 2.1.4 中所述,以 CFU/mL 为单位确定抗生素耐药细菌计数。
- 分离物的甘油库存
- 选择形态不同的 AR 菌落。
- 将单个分离菌落悬浮在含有相应抗生素的 2 mL 无菌 Luria-Bertani 肉汤中(例如,如果从含有头孢噻肟的平板中选择菌落,则将步骤 2.3.1 中的菌落接种在含有相应浓度的头孢噻肟的无菌 Luria-Bertani 肉汤中)。
- 将接种的试管在37°C以80rpm孵育,直到OD600 达到0.5。
- 通过在无菌条件下将步骤 2.3.3 中的 750 μL 培养悬浮液混合到 250 μL 无菌 100% 甘油中来制备分离物的甘油储备液。
- 将甘油储备液储存在-80°C直至进一步分析。
注意:为了从甘油原液中恢复培养物,请在4°C下解冻甘油原液。 将该原液的循环接种到 2 mL 含有相应抗生素的无菌 Luria-Bertani 肉汤中并使其生长。
3. 通过16S rRNA基因测序鉴定可培养细菌
- 从分离株制备用于PCR的DNA模板
注意:Carlson等人给出了用于PCR制备用于从细菌中分离粗DNA的DNA模板的协议10。- 使用无菌牙签,取在培养皿上生长的单个分离的纯分离菌落。将细菌菌落悬浮在无菌微量离心管中的 100 μL 无菌双蒸水中并煮沸 10 分钟。
- 将悬浮液以10,000 ×g 离心2分钟以沉淀碎片,并将上清液转移到新鲜的无菌微量离心管中用作粗DNA模板。
- 16S rRNA基因V3区域的靶向PCR扩增和测序
- 如 表1所示,在PCR管中制备40μL反应混合物进行PCR扩增。
注意:DNA制备应在冰块上进行,同时尽量减少污染的机会(处理试剂时戴手套,并用70%乙醇彻底清洁工作表面)。 - 将试管放入热模块中,然后在PCR热循环仪中运行适当的程序。有关标准化PCR循环条件和16S rRNA基因V3区域扩增的引物信息,请参见 表2 。
- 为了分离扩增子和可视化,进行琼脂糖凝胶电泳(AGE)。将 10 μL 扩增的 PCR 产物和 2 μL 6x 凝胶上样缓冲液(表 3)混合,并将该混合物上样到含有 5 μL 10 mg/mL 溴化乙锭 (EtBr) 的 1.5% 琼脂糖凝胶上的孔中(将 1.5 g 琼脂糖粉末溶解在 100 mL 的 1x TAE 缓冲液 [表 3]) 中,终浓度为 0.5 μg/mL EtBr。
注意:EtBr是一种强致癌物质。处理 EtBr 和含有 EtBr 的凝胶时,应始终佩戴手套。 - 添加DNA分子量标准以估计扩增子的大小。
- 在80-100 V的TAE罐缓冲液中进行凝胶电泳。
- 一旦示踪染料运行3/4的凝胶,停止电泳,并在紫外透射仪下观察扩增子条带。
- 使用 PCR 产物(扩增子)进行 16S rRNA 基因测序以鉴定分离株。
- 通过使用公式(2)对其进行分光光度分析来量化扩增子。
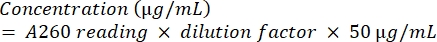 (二)
(二) - 要检查DNA的纯度,请计算A260 / A280的比率。
注意:理想情况下,此数字应高于 1.5,最好介于 1.8 和 2.0 之间。 - 要识别分离株,请使用适当的比对搜索工具比较与序列数据库获得的序列。
- 如 表1所示,在PCR管中制备40μL反应混合物进行PCR扩增。
4. 使用抗生素敏感性测试检测分离株中的抗生素耐药性
注意:本协议描述了通过椎间盘扩散进行抗生素敏感性测试(AST)的方法。使用以下抗生素盘:头孢噻肟(5μg)、氨苄西林(10μg)、左氧氟沙星(5μg)、氯霉素(30μg)、替加环素(15μg)、头孢曲松(30μg)、亚胺培南(10μg)、庆大霉素(10μg)、新霉素(10μg)、甲氧苄啶(5μg)和环丙沙星(5μg)。
- 制备 AST 接种物
- 使用无菌环在2mL无菌非选择性培养基(例如Luria-Bertani肉汤(不含任何抗生素)中无菌接种单个分离的纯化的AR菌落,并在37°C以80rpm孵育过夜。
- 通过将 100-150 μL 过夜生长的培养物(大约,OD 600 = 1.8-2.0)重悬于 2 mL 新鲜的非选择性 Luria-Bertani 肉汤培养基中并孵育 2-4 小时(直到 OD600 达到 0.4-0.5)。
- 使用无菌0.85%盐水溶液稀释这种新鲜生长的培养悬浮液,使培养物的密度等于0.5麦克法兰标准品(大约OD600 = 0.1),大致相当于1-2×108 个细胞/ mL。
- 轻轻混合细菌悬浮液,使细胞分布均匀。
- 在稀释后15分钟内使用上述悬浮液。
- 琼脂平板的接种
- 通过将 38 g MHA 混合在 1,000 mL 双蒸水中,准备用于执行 AST 的 Mueller-Hinton 琼脂 (MHA) 板,并通过加热溶解混合物。将溶解的混合物在121°C,15psi下高压灭菌15分钟。
- 确保板中MHA的深度为4 mm(每块板25 mL培养基)。
- 同时,从冰箱中取出抗生素盘并加热至室温。
注意:抗生素盘片应通过最初在4°C下解冻,然后在室温下解冻来逐渐解冻,以减少盘上冷凝的任何潜在危险,这随后可能会影响抑制区(ZOI)。 - 在无菌条件下,将无菌棉签浸入步骤4.1中制备的接种物中,并去除多余的悬浮液以避免板过度接种。
- 将培养物均匀地铺在平板上,从MHA板的顶部开始,从边缘到边缘来回。擦拭时将板旋转 60°。
- 抗生素盘的应用
- 在火焰灭菌镊子的帮助下,无菌地将抗生素盘转移到接种的MHA平板上,然后轻轻按压盘以确保与琼脂完全水平接触。
注意:此过程必须在将培养物接种到平板上的15分钟内完成。 - 考虑到微生物、使用的抗生素和平板的大小,将适当数量的抗生素盘放在琼脂平板上,以避免抑制区域的重叠。
注意: 90 毫米圆板上可容纳四到五个圆盘。
- 在火焰灭菌镊子的帮助下,无菌地将抗生素盘转移到接种的MHA平板上,然后轻轻按压盘以确保与琼脂完全水平接触。
- 培养板的孵育
- 在施用抗生素盘后15分钟内,将板倒置并在37°C孵育过夜。
- 结果解读
- 以毫米 (mm) 为单位测量 ZOI 直径,并根据 EUCAST11 给出的断点值进行解释。请参阅下面给出的两个示例。
- 用于 肠杆菌 的环丙沙星抗生素盘(5μg)的区域直径断点(mm)为S ≥ 25和R < 22,这意味着如果ZOI≥25 mm,则认为它是敏感的(S),而如果ZOI<22 mm,则被认为是耐药的(R)。如果 ZOI 直径介于 22 和 25 之间,则分离物被认为是中间 (I)。
- 氯霉素抗生素盘(30 μg)用于葡萄球菌 属的区域直径 断点(mm)为S ≥ 18和R < 18,这意味着如果ZOI≥18 mm,则认为它是敏感的,而如果ZOI<18 mm,则被认为是耐药的。
- 通过查找分离株耐药的抗生素数量与分离株暴露的抗生素总数的比率来确定多种抗生素耐药性 (MAR) 指数。
注意:对于质量控制, 大肠杆菌 ATCC 25922和 S。 葡萄球菌 ATCC 29213 按照步骤 4 中的协议用作参考菌株。
- 以毫米 (mm) 为单位测量 ZOI 直径,并根据 EUCAST11 给出的断点值进行解释。请参阅下面给出的两个示例。
5. 基于PCR检测分离株中的抗生素耐药基因
- 使用标准 PCR 方案鉴定分离株中的 ARG。使用步骤3.1中给出的方案制备DNA模板。
注意:本研究中使用的PCR循环条件为94°C10分钟,然后35个94°C循环30秒,在适当温度下退火30秒(每个引物组的标准化),在72°C下延伸40秒,并在72°C下最终延伸5分钟。反应混合物描述见 表4。ARG、引物和退火温度的列表见 表5。 - 要分离、可视化和检查扩增子的纯度,请执行步骤3.2.3-3.2.10。
6. 全宏基因组DNA分析,用于鉴定总细菌多样性和检测宏基因组中的ARG
- 从水样中提取总DNA(宏基因组)
- 从过滤的水样中提取宏基因组DNA。
注意:在当前的研究中,按照制造商的方案,使用参考的DNA分离试剂盒从过滤后的水样中提取宏基因组DNA(总DNA)(参见 材料表)。 - 通过将 3 μL 提取的宏基因组 DNA 加载到 0.8% 琼脂糖凝胶上来检查 DNA 的质量,并在 80-110 V 下运行凝胶约 30 分钟。
- 检查是否存在单个完整条带。
- 使用荧光计检查DNA浓度。
- 从过滤的水样中提取宏基因组DNA。
- 使用全宏基因组DNA测序测定细菌多样性并检测ARG
- 文库制备和PCR扩增:
- 使用参考的DNA文库制备试剂盒制备配对末端测序文库(参见 材料表)。
- 通过取 200 ng 的 DNA 并将其机械剪切成更小的片段,准备 DNA 进行接头连接,然后进行连续的末端修复步骤,其中在 3' 端添加一个"A"。
- 根据用于测序的平台,将特异性接头连接到DNA片段的两端。
注意:这些适配器中存在将双条形码文库与流通池结合以进行测序至关重要的序列。这允许对接头连接的片段进行PCR扩增并结合标准测序引物。 - 要检查质量和数量,请按照制造商的说明使用高灵敏度DNA芯片分析扩增的文库。
- 集群生成和排序:
- 将扩增的文库加载到适当的测序平台上,用于簇生成和后续测序。
注意:文库分子与配对端流通池上的互补适配器寡核苷酸结合。在测序过程中,在反向链的再合成后选择性地裂解前链。然后从片段的另一端对复制的反向链进行测序。
- 将扩增的文库加载到适当的测序平台上,用于簇生成和后续测序。
- 生物信息学分析:
- 使用适当的平台从高质量数据生成脚手架。
- 对这些支架进行生物信息学分析,以进行ARG的分类和鉴定。
注意: 图1给出了用于鉴定总细菌多样性和检测宏基因组中ARG的整个宏基因组DNA分析的工作流程。 图 2 给出了手稿中描述的完整方法的流程图。
- 文库制备和PCR扩增:
Access restricted. Please log in or start a trial to view this content.
结果
细菌总载量和抗生素耐药 (AR) 细菌计数
通过在改良培养基R2A琼脂上散布10-4至10-6倍稀释的水样品来计数总细菌负荷。为了计数AR细菌计数,将10-3至10-6倍稀释液散布在含有抗生素的培养基板上(图3)。总细菌计数和AR细菌计数计算为CFU/mL,所有电镀实验一式两份。按照上述方案,本研究表明细菌总数为3.0×109CFU / mL。?...
Access restricted. Please log in or start a trial to view this content.
讨论
样本收集和处理起着重要作用,可能会影响研究结果和解释。因此,为了排除样品的变化,重要的是在所研究的淡水体的多个位置进行采样。在处理此类样品时保持适当的无菌环境条件可以防止污染。此外,为了防止细菌组成的变化可能影响提取核酸的质量和数量,运输条件应保持在4°C,从样品收集点到后续处理的时间最短。一些研究强调,如果不仔细进行,样品收集和处理之间的过渡期可能会...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有需要披露的利益冲突。
致谢
这项工作得到了孟买大学科技部-大学研究和科学卓越促进(DST-PURSE)计划的财政资助的部分支持。Devika Ghadigaonkar在该计划下担任项目研究员。科学和技术部科学与工程研究委员会(DST-SERB)项目编号:CRG/2018/003624高级研究员Harshali Shinde提供的技术支持得到了认可。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 100 bp DNA ladder | Himedia | MBT049-50LN | For estimation of size of the amplicons |
| 2x PCR Taq mastermix | HiMedia | MBT061-50R | For making PCR reaction mixture |
| 37 °C Incubator | GS-192, Gayatri Scientific | NA | For incubation of bacteria |
| 6x Gel Loading Buffer | HiMedia | ML015-1ML | Loading and Tracking dye which helps to weigh down the DNA sample and track the progress of electrophoresis |
| Agarose powder | Himedia | MB229-50G | For resolving amplicons during Agarose Gel Electrophoresis (AGE) |
| Ampicillin antibiotic disc | HiMedia | SD002 | For performing AST |
| Autoclave | Equitron | NA | Required for sterilization of media, glass plates, test tubes, etc |
| Bioanalyzer 2100 | Agilent Technologies | NA | To check the quality and quantity of the amplified library |
| Bisafety B2 Cabinet | IMSET | IMSET BSC-Class II Type B2 | Used for microbiological work like bacterial culturing, AST etc. |
| Cefotaxime antibiotic disc | HiMedia | SD295E-5VL | For performing AST |
| Cefotaxime antibiotic powder | HiMedia | TC352-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Ceftriaxone antibiotic disc | HiMedia | SD065 | For performing AST |
| Centrifuge Minispin | Eppendorf | Minispin Plus-5453 | Used to pellet the debris during crude DNA preparation |
| Chloramphenicol antibiotic disc | HiMedia | SD006-5x50DS | For performing AST |
| Ciprofloxacin antibiotic disc | HiMedia | SD060-5x50DS | For performing AST |
| Ciprofloxacin antibiotic powder | HiMedia | TC447-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Colorimeter | Quest | NA | For checking the OD of culture suspensions |
| Comprehensive Antibiotic Resistance Database (CARD) database | functional annotation of ARGs; https://card.mcmaster.ca/ | ||
| Cooling Shaker Incubator | BTL41 Allied Scientific | NA | For incubation of media plates for culturing bacteria |
| Deep Freezer (-40 °C) | Haier | DW40L, Haier Biomedicals | For storage of glycerol stocks |
| DNA Library Prep Kit | NEB Next Ultra DNA Library Prep Kit for Illumina | NA | Paired-end sequencing library preparation |
| EDTA | HiMedia | GRM1195-100G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Electrophoresis Apparatus | TechResource | 15 cm gel casting tray | For making the agarose gel and carrying out electrophoresis |
| Electrophoresis Power pack with electrodes | Genei | NA | For running the AGE |
| Erythromycin antibiotic disc | HiMedia | SD222-5VL | For performing AST |
| Erythromycin antibiotic powder | HiMedia | CMS528-1G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Erythromycin antibiotic powder | HiMedia | TC024-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Escherichia coli ATCC 25922 | HiMedia | 0335X-1 | Used as a control while performing AST |
| Ethidium Bromide | HiMedia | MB071-1G | Intercalating agent and visualizaion of DNA after electrophoresis under Gel Documentation System |
| Fluorometer | Qubit 2.0 | NA | For determining concentration of extracted metagenomic DNA |
| Gel Documentation System | BioRad | Used for visualizing PCR amplicons after electrophoresis | |
| Gentamicin antibiotic disc | HiMedia | SD170-5x50DS | For performing AST |
| Glacial Acetic Acid | HiMedia | AS119-500ML | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Glycerol | HiMedia | GRM1027-500ML | For making glycerol stocks |
| Imipenem antibiotic disc | HiMedia | SD073 | For performing AST |
| Kaiju Database | NA | NA | For taxonomical classification of reads; https://kaiju.binf.ku.dk/ |
| Kanamycin antibiotic disc | HiMedia | SD017-5x50DS | For performing AST |
| Kanamycin antibiotic powder | HiMedia | MB105-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Levofloxacin antibiotic disc | HiMedia | SD216-5VL | For performing AST |
| Luria Bertani broth | Himedia | M1245-500G | For enrichment of cultures |
| McFarland Standards | Himedia | R092-1No | To compare density of culture suspension |
| Molecular Biology water | HiMedia | TCL018-500ML | For making PCR reaction mixture |
| Mueller-Hinton Agar (MHA) | HiMedia | M173-500G | For performing Antibiotc Susceptibility Testing (AST) |
| Neomycin antibiotic disc | HiMedia | SD731-5x50DS | For performing AST |
| PCR Gradient Thermal Cycler | Eppendorf | Mastercycler Nexus Gradient 230V/50-60 Hz | Used for performing PCR for amplification of 16S rRNA region and various Antibiotic Resistance genes |
| Primers | Xcelris | NA | For PCR amplication |
| R2A Agar, Modified | HiMedia | M1743 | For preparation of media plates for isolation of total and antibiotic resistant (AR) bacterial load |
| Scaffold generation | CLC Genomics Workbench 6.0 | NA | For generation of scaffolds |
| Sequencer | Illumina platform (2 x 150 bp chemistry) | NA | Sequencing of amplified library |
| Sodium Chloride | HiMedia | TC046-500G | For preparation of 0.85% saline for serially diluting the water sample |
| Soil DNA isolation Kit | Xcelgen | NA | For extraction of whole metagenomic DNA from the filtered water sample |
| Staphylococcus aureus subsp. aureus ATCC 29213 | HiMedia | 0365P | Used as a control while performing AST |
| Taxonomical Classification | Kaiju ioinformatics tool | NA | For classification of reads into different taxonomic groups from phylum to genus level |
| The Comprehensive Antibiotic Resistance Database (CARD) | NA | NA | For functional annotation of ARGs |
| Tigecycline antibiotic disc | HiMedia | SD278 | For performing AST |
| Trimethoprim antibiotic disc | HiMedia | SD039-5x50DS | For performing AST |
| Tris base | HiMedia | TC072-500G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Vancomycin antibiotic powder | HiMedia | CMS217 | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Weighing Balance | Mettler Toledo | ME204 Mettler Toledo | Used for weighing media powders, reagent powders etc. |
| NA - Not Applicable |
参考文献
- Prestinaci, F., Pezzotti, P., Pantosti, A. Antimicrobial resistance: A global multifaceted phenomenon. Pathogens and Global Health. 109 (7), 309-318 (2015).
- Knight, G., et al. Antimicrobial resistance and COVID-19: Intersections and implications. Elife. 10, 64139(2021).
- Ventola, C. L. The antibiotic resistance crisis: Part 1: Causes and threats. Pharmacy and Therapeutics. 40 (4), 277-283 (2015).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J. R., Rath, A. Metagenomic analysis of total microbial diversity and antibiotic resistance of culturable microorganisms in raw chicken meat and mung sprouts (Phaseolus aureus) sold in retail markets of Mumbai. India. Current Science. 113 (1), 71-79 (2017).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J., Rath, A. Characterization of multiple antibiotic resistance of culturable microorganisms and metagenomic analysis of total microbial diversity of marine fish sold in retail shops in Mumbai, India. Environmental Science and Pollution Research. 25 (7), 6228-6239 (2018).
- Czekalski, N., GascónDíez, E., Bürgmann, H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake. The ISME Journal. 8 (7), 1381-1390 (2014).
- Kraemer, S., Ramachandran, A., Perron, G. Antibiotic pollution in the environment: From microbial ecology to public policy. Microorganisms. 7 (6), 180(2019).
- Edmonds-Wilson, S., Nurinova, N., Zapka, C., Fierer, N., Wilson, M. Review of human hand microbiome research. Journal of Dermatological Science. 80 (1), 3-12 (2015).
- de Abreu, V., Perdigão, J., Almeida, S. Metagenomic approaches to analyze antimicrobial resistance: An overview. Frontiers in Genetics. 11, 575592(2021).
- Carlson, S., et al. Detection of multiresistant Salmonella typhimurium DT104 using multiplex and fluorogenic PCR. Molecular and Cellular Probes. 13 (3), 213-222 (1999).
- Breakpoint tables for interpretation of MICs and zone diameters, Version 12.0. European Committee on Antimicrobial Susceptibility Testing. , Available from: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_12.0_Breakpoint_Tables.pdf (2022).
- Bharti, R., Grimm, D. Current challenges and best-practice protocols for microbiome analysis. Briefings in Bioinformatics. 22 (1), 178-193 (2019).
- Choo, J., Leong, L., Rogers, G. Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports. 5, 16350(2015).
- Clinical and Laboratory Standards Institute. Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria that Grow Aerobically, 11th edition. , Clinical and Laboratory Standards Institute. Wayne, PA. (2015).
- Bayot, M., Bragg, B. Antimicrobial Susceptibility Testing. StatPearls. , StatPearls Publishing. Treasure Island, FL. (2021).
- Joseph, A. A., Odimayo, M. S., Olokoba, L. B., Olokoba, A. B., Popoola, G. O. Multiple antibiotic resistance index of Escherichia coli isolates in a tertiary hospital in South-West Nigeria. Medical Journal of Zambia. 44 (4), 225-232 (2017).
- Lorenz, T. Polymerase chain reaction: Basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998(2012).
- Rolin, J. Food and human gut as reservoirs of transferable antibiotic resistance encoding genes. Frontiers in Microbiology. 4, 173(2013).
- Racewicz, P., et al. Prevalence and characterisation of antimicrobial resistance genes and class 1 and 2 integrons in multiresistant Escherichia coli isolated from poultry production. Scientific Reports. 12, 6062(2022).
- Gebreyes, W., Thakur, S. Multidrug-resistant Salmonella enterica serovar Muenchen from pigs and humans and potential interserovar transfer of antimicrobial resistance. Antimicrobial Agents and Chemotherapy. 49 (2), 503-511 (2005).
- Li, L., et al. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui Province, China. PLoS One. 9 (8), 104356(2014).
- Akers, K., et al. Aminoglycoside resistance and susceptibility testing errors in Acinetobacter baumannii-calcoaceticus complex. Journal Of Clinical Microbiology. 48 (4), 1132-1138 (2010).
- Ciesielczuk, H. Extra-intestinal pathogenic Escherichia coli in the UK: The importance in bacteraemia versus urinary tract infection, colonisation of widespread clones and specific virulence factors. , Queen Mary University of London. PhD thesis (2015).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。