È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento e identificazione di batteri resistenti agli antibiotici trasportati dall'acqua e caratterizzazione molecolare dei loro geni di resistenza agli antibiotici
In questo articolo
Riepilogo
Qui, presentiamo un protocollo dettagliato per l'isolamento e l'identificazione di batteri resistenti agli antibiotici dall'acqua e la caratterizzazione molecolare dei loro geni di resistenza agli antibiotici (ARG). L'uso di tecniche basate su colture e non basate su colture (analisi metagenomica) fornisce informazioni complete sulla diversità batterica totale e sul pool totale di diversi ARG presenti nelle acque dolci di Mumbai, in India.
Abstract
Lo sviluppo e la diffusione della resistenza agli antibiotici (AR) attraverso il microbiota associato ai corpi d'acqua dolce è una delle principali preoccupazioni per la salute globale. Nel presente studio, sono stati raccolti e analizzati campioni di acqua dolce rispetto alla diversità batterica totale e ai geni AR (ARG) utilizzando sia tecniche convenzionali basate sulla coltura che un approccio metagenomico indipendente dalla coltura ad alto rendimento. Questo articolo presenta un protocollo sistematico per l'enumerazione dei batteri coltivabili totali e resistenti agli antibiotici da campioni di acqua dolce e la determinazione della resistenza fenotipica e genotipica negli isolati coltivabili. Inoltre, riportiamo l'uso dell'intera analisi metagenomica del DNA metagenomico totale estratto dal campione di acqua dolce per l'identificazione della diversità batterica complessiva, compresi i batteri non coltivabili, e l'identificazione del pool totale di diversi ARG (resistoma) nel corpo idrico. Seguendo questi protocolli dettagliati, abbiamo osservato un elevato carico di batteri resistenti agli antibiotici nell'intervallo di 9,6 × 10 5-1,2 × 109 CFU / ml. La maggior parte degli isolati erano resistenti ai molteplici antibiotici testati, tra cui cefotaxime, ampicillina, levofloxacina, cloramfenicolo, ceftriaxone, gentamicina, neomicina, trimetoprim e ciprofloxacina, con indici multipli di resistenza agli antibiotici (MAR) di ≥0,2, indicando alti livelli di resistenza negli isolati. Il sequenziamento dell'rRNA 16S ha identificato potenziali patogeni umani, come Klebsiella pneumoniae, e batteri opportunisti, come Comamonas spp., Micrococcus spp., Arthrobacter spp. e Aeromonas spp. La caratterizzazione molecolare degli isolati ha mostrato la presenza di vari ARG, quali blaTEM, blaCTX-M (β-lattamici), aadA, aac (6')-Ib (aminoglicosidi) e dfr1 (trimetoprims), confermata anche dall'analisi metagenomica del DNA. Nel DNA metagenomico è stata rilevata anche un'elevata prevalenza di altri ARG che codificano per le pompe di efflusso antibiotico - mtrA, macB, mdtA, acrD, β-lattamasi - SMB-1, VIM-20, ccrA, ampC, blaZ, il gene cloramfenicolo acetiltransferasi catB10 e il gene di resistenza alla rifampicina rphB - . Con l'aiuto dei protocolli discussi in questo studio, abbiamo confermato la presenza di batteri MAR trasportati dall'acqua con diversi tratti fenotipici e genotipici dell'AR. Pertanto, l'analisi metagenomica del DNA completo può essere utilizzata come tecnica complementare alle tecniche convenzionali basate sulla coltura per determinare lo stato complessivo di AR di un corpo idrico.
Introduzione
La resistenza antimicrobica (AMR) è stata identificata come uno dei problemi globali più urgenti. La rapida evoluzione della resistenza antimicrobica e la sua diffusione a livello mondiale rappresentano una delle maggiori minacce per la salute umana e l'economia globale in termini di costi sanitari ad essa associati1. L'uso eccessivo e l'abuso di antibiotici hanno portato ad un aumento dell'AR. Ciò è stato evidenziato dalla pandemia di COVID-19, durante la quale il trattamento delle infezioni secondarie associate, in molti casi, è stato enormemente compromesso a causa della resistenza antimicrobica nei pazienti colpiti2. Oltre all'uso/abuso diretto di antibiotici da parte dell'uomo, l'uso eccessivo e improprio di antibiotici in agricoltura e zootecnia e il loro scarico improprio nell'ambiente, compresi i corpi idrici, costituiscono una delle principali preoccupazioni3. L'aumento di nuovi tratti di resistenza e resistenza multifarmaco nei batteri evidenzia urgentemente la necessità di una migliore comprensione dei fattori che portano allo sviluppo dell'AR e alla sua diffusione. Più batteri resistenti agli antibiotici, che spesso trasportano più geni AR (ARG) su elementi genetici mobili come i plasmidi, possono trasferire questi geni di resistenza a microrganismi non resistenti, compresi potenziali agenti patogeni umani, portando così alla comparsa di superbatteri che non sono trattabili anche con antibiotici di ultima istanza4. Questi molteplici batteri resistenti agli antibiotici, se presenti negli ecosistemi acquatici, possono entrare direttamente nell'intestino umano attraverso il consumo di alimenti contaminati a base d'acqua come pesci, granchi e molluschi. Studi precedenti hanno dimostrato che la diffusione dei batteri AR nei sistemi idrici presenti in natura può raggiungere anche altre riserve idriche, compresa l'acqua potabile, e, quindi, può entrare nella catena alimentare umana 5,6,7.
Lo scopo del presente studio è quello di fornire un protocollo completo utilizzando una combinazione di tecniche basate su colture e non basate su colture (analisi metagenomica completa) per ottenere informazioni complete sulla diversità batterica totale e sul pool totale di diversi ARG presenti in un corpo idrico a Mumbai, in India. Convenzionalmente, sono state utilizzate tecniche basate sulla coltura per studiare la diversità batterica nei corpi idrici. Poiché i microrganismi coltivabili costituiscono solo una piccola percentuale del microbiota totale in qualsiasi nicchia, per avere una migliore comprensione dello stato generale della diversità batterica e dei vari tratti resistenti prevalenti in qualsiasi campione, devono essere utilizzate in tandem varie tecniche basate sulla coltura e indipendenti dalla coltura. Una di queste tecniche robuste e affidabili indipendenti dalla coltura è l'analisi del DNA metagenomico completo. Questo metodo ad alto rendimento è stato utilizzato con successo in vari studi sulla diversità batterica o nelle annotazioni funzionali di vari ARG 8,9. Questa tecnica utilizza il metagenoma (il materiale genetico totale in un campione) come materiale di partenza per varie analisi e, quindi, è indipendente dalla coltura. I protocolli del presente studio possono essere utilizzati per l'analisi dell'intero DNA metagenomico per ottenere informazioni sulla diversità batterica totale e vari ARG (resistoma) in campioni di acqua.
Protocollo
1. Raccolta ed elaborazione dei campioni
- Raccolta di campioni
- Raccogliere il volume appropriato del campione d'acqua in contenitori sterili, assicurandosi che non siano riempiti più di 3/4 del contenitore.
- Trasportare i campioni in laboratorio in condizioni asettiche il più presto possibile dopo la raccolta e trattarli immediatamente.
- Elaborazione dei campioni
- Filtrare asetticamente il campione d'acqua attraverso un panno di mussola sterile per rimuovere qualsiasi particella.
- Effettuare adeguate diluizioni seriali dell'acqua filtrata per ulteriori analisi.
2. Stima della carica batterica totale e del conteggio dei batteri resistenti agli antibiotici
- Determinazione della carica batterica totale
- Sospendere 18,12 g di Agar R2A, polvere modificata in 1.000 ml di acqua bidistillata e sciogliere la miscela riscaldando. Autoclavare la miscela disciolta a 121 °C, 15 psi per 20 min. Preparare le piastre R2A Agar, Modified versando la quantità appropriata della miscela autoclavata in piastre di Petri sterili (ad esempio, aggiungere circa 20 ml di terreno sterile autoclavato a una piastra di Petri sterile di 90 mm).
- Distribuire uniformemente 100 μL delle diluizioni appropriate del campione di acqua filtrata sulla piastra R2A Agar, modificata una volta che il mezzo si solidifica. Esegui l'esperimento in duplicato.
- Incubare tutte le piastre di cui sopra a 35-37 °C per 48 h (variare la temperatura e il tempo di incubazione a seconda del mezzo utilizzato per l'isolamento).
- Esprimere la carica batterica totale in termini di unità formanti colonie per millilitro (CFU/ml) usando l'equazione (1):
 (1)
(1)
- Determinazione della carica batterica AR
- Seguire i passaggi 2.1.1-2.1.4. Tuttavia, invece di R2A Agar, piastre modificate, utilizzare R2A Agar, piastre modificate integrate individualmente con cinque diversi antibiotici, vale a dire cefotaxime (3 μg / mL), ciprofloxacina (0,5 μg / mL), eritromicina (20 μg / mL), kanamicina (15 μg / mL) e vancomicina (3 μg / mL).
- Aggiungere gli antibiotici separatamente in provette contenenti 20 mL di agar R2A fuso sterile, modificato (con la temperatura dell'agar R2A fuso, modificato a ≤40 °C) per ottenere la concentrazione finale di antibiotico come indicato al punto 2.2.1.
- Agitare per una miscelazione uniforme e versare su piastre di Petri sterili prima che l'agar si solidifichi. Esegui l'esperimento in duplicato.
- Incubare tutte le piastre di cui sopra a 35-37 °C per 48 h (se si utilizza un mezzo diverso, la temperatura e il tempo di incubazione possono variare).
- Per il controllo di qualità e la verifica dell'efficacia degli antibiotici, distribuire 100 μL di sospensioni batteriche dei ceppi di Escherichia coli ATCC 25922 e Staphylococcus aureus ATCC 29213 sui rispettivi ceppi di Agar R2A contenente antibiotici, piastre modificate (assicurarsi che la densità della coltura fresca utilizzata per l'inoculazione sia OD = 0,5 a 600 nm).
- Determinare la conta batterica resistente agli antibiotici in termini di CFU/ml come descritto al punto 2.1.4.
- Scorte di glicerolo degli isolati
- Selezionare colonie AR morfologicamente distinte.
- Sospendere una singola colonia isolata in 2 ml di brodo Luria-Bertani sterile contenente il rispettivo antibiotico (ad esempio, se una colonia è stata selezionata da una piastra contenente cefotaxime, inoculare la colonia dal punto 2.3.1 in brodo sterile di Luria-Bertani contenente cefotaxime alla rispettiva concentrazione).
- Incubare le provette inoculate a 37 °C a 80 giri/min fino a quando l'OD600 raggiunge 0,5.
- Preparare le scorte di glicerolo degli isolati mescolando 750 μL della sospensione di coltura dalla fase 2.3.3 in 250 μL di glicerolo sterile al 100% in condizioni asettiche.
- Conservare le scorte di glicerolo a -80 °C fino a ulteriori analisi.
NOTA: Per la rinascita delle colture dalle scorte di glicerolo, scongelare le scorte di glicerolo a 4 °C. Inoculare un ciclo di questo stock in 2 ml di brodo Luria-Bertani sterile contenente il rispettivo antibiotico e lasciare crescere.
3. Identificazione di batteri coltivabili mediante sequenziamento del gene 16S rRNA
- Preparazione del modello di DNA dagli isolati per PCR
NOTA: Il protocollo descritto per la preparazione del modello di DNA per la PCR per l'isolamento del DNA grezzo dai batteri è dato da Carlson et al.10.- Usando uno stuzzicadenti sterile, prendi una singola colonia isolata e pura dell'isolato che cresce su una piastra di Petri. Sospendere la colonia batterica in 100 μL di acqua sterile bidistillata in una provetta sterile per microcentrifuga e far bollire per 10 minuti.
- Centrifugare la sospensione a 10.000 × g per 2 minuti per pellettare i detriti e trasferire il surnatante in una provetta di microcentrifuga sterile fresca da utilizzare come modello di DNA grezzo.
- Amplificazione PCR mirata della regione V3 del gene rRNA 16S e sequenziamento
- Preparare 40 μL della miscela di reazione in una provetta PCR per l'amplificazione della PCR, come indicato nella Tabella 1.
NOTA: La preparazione del DNA deve essere effettuata su un blocco di ghiaccio riducendo al minimo la possibilità di contaminazione (indossare guanti durante la manipolazione dei reagenti e pulire accuratamente la superficie di lavoro con etanolo al 70%). - Posizionare il tubo nel blocco termico ed eseguire il programma appropriato nel termociclatore PCR. Vedere la Tabella 2 per le condizioni di ciclo standardizzate della PCR e le informazioni di primer per l'amplificazione delle regioni V3 dei geni rRNA 16S.
- Per risolvere gli ampliconi e la visualizzazione, eseguire l'elettroforesi su gel di agarosio (AGE). Mescolare 10 μL del prodotto PCR amplificato e 2 μL di tampone caricante gel 6x (Tabella 3) e caricare questa miscela in pozzetti su gel di agarosio all'1,5% (sciogliere 1,5 g di polvere di agarosio in 100 mL di 1x tampone TAE [Tabella 3]) contenente 5 μL di 10 mg/mL di bromuro di etidio (EtBr) ad una concentrazione finale di 0,5 μg/mL di EtBr in 100 mL di gel di agarosio.
ATTENZIONE: EtBr è un potente cancerogeno. I guanti devono essere indossati in ogni momento durante la manipolazione di EtBr e gel contenenti EtBr. - Aggiungi la scala del DNA per la stima della dimensione degli ampliconi.
- Effettuare l'elettroforesi del gel in un tampone serbatoio TAE a 80-100 V.
- Una volta che il colorante tracciante esegue 3/4 del gel, interrompere l'elettroforesi e visualizzare le bande di amplicone sotto un transilluminatore UV.
- Utilizzare il prodotto PCR (amplicone) per il sequenziamento del gene rRNA 16S per identificare l'isolato.
- Quantificare l'amplicone sottoponendolo ad analisi spettrofotometrica utilizzando l'equazione (2).
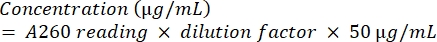 (2)
(2) - Per verificare la purezza del DNA, calcolare il rapporto A260/A280.
NOTA: Idealmente, questo numero dovrebbe essere superiore a 1,5 e, preferibilmente, compreso tra 1,8 e 2,0. - Per identificare gli isolati, confrontare le sequenze ottenute con i database di sequenze utilizzando uno strumento di ricerca di allineamento appropriato.
- Preparare 40 μL della miscela di reazione in una provetta PCR per l'amplificazione della PCR, come indicato nella Tabella 1.
4. Rilevazione della resistenza agli antibiotici negli isolati mediante test di sensibilità agli antibiotici
NOTA: Questo protocollo descrive il metodo per i test di sensibilità agli antibiotici (AST) mediante diffusione del disco. Sono stati utilizzati i seguenti dischi antibiotici: cefotaxime (5 μg), ampicillina (10 μg), levofloxacina (5 μg), cloramfenicolo (30 μg), tigeciclina (15 μg), ceftriaxone (30 μg), imipenem (10 μg), gentamicina (10 μg), neomicina (10 μg), trimetoprim (5 μg) e ciprofloxacina (5 μg).
- Preparazione dell'inoculo per l'AST
- Inocula asetticamente una singola colonia di AR purificata isolata utilizzando un'ansa sterile in 2 ml di un mezzo sterile non selettivo, come il brodo di Luria-Bertani (senza alcun antibiotico), e incubare a 37 °C a 80 giri/min durante la notte.
- Risospendere prelevando 100-150 μL della coltura coltivata durante la notte (approssimativamente, OD 600 = 1,8-2,0) in 2 ml di terreno di brodo fresco non selettivo Luria-Bertani e incubare per 2-4 ore (fino a quando l'OD600 raggiunge 0,4-0,5).
- Diluire questa sospensione di coltura appena coltivata utilizzando una soluzione salina sterile allo 0,85% in modo tale che la densità della coltura sia pari allo standard di 0,5 McFarland (approssimativamente, OD600 = 0,1), che corrisponde approssimativamente a 1-2 × 108 cellule / ml.
- Mescolare delicatamente la sospensione batterica per una distribuzione uniforme delle cellule.
- Utilizzare la sospensione di cui sopra entro 15 minuti dalla diluizione.
- Inoculo delle piastre di agar
- Preparare le piastre Mueller-Hinton Agar (MHA) per eseguire l'AST mescolando 38 g di MHA in 1.000 ml di acqua bidistillata e sciogliere la miscela riscaldando. Autoclavare la miscela disciolta a 121 °C, 15 psi per 15 min.
- Assicurarsi che la profondità di MHA nelle piastre sia di 4 mm (25 ml di terreno per piastra).
- Contemporaneamente, rimuovere i dischi di antibiotico dal congelatore e riscaldarli a temperatura ambiente.
NOTA: I dischi antibiotici devono essere gradualmente scongelati scongelando inizialmente i dischi a 4 °C e successivamente a temperatura ambiente per ridurre qualsiasi potenziale pericolo di condensa sui dischi, che può successivamente influenzare la zona di inibizione (ZOI). - In condizioni asettiche, immergere un batuffolo di cotone sterile nell'inoculo preparato al punto 4.1 e rimuovere la sospensione in eccesso per evitare un'eccessiva inoculazione delle piastre.
- Distribuire uniformemente la coltura sui piatti, partendo dalla parte superiore della piastra MHA e andando avanti e indietro da un bordo all'altro. Ruotare la piastra di 60° durante il tampone.
- Applicazione dei dischi antibiotici
- Con l'aiuto di una pinza sterilizzata a fiamma, trasferire asetticamente i dischi antibiotici sulle piastre MHA inoculate e premere delicatamente i dischi per garantire un contatto completo con l'agar.
NOTA: Questa procedura deve essere eseguita entro 15 minuti dall'inoculazione della coltura sulle piastre. - Posizionare il numero appropriato di dischi di antibiotico sulla piastra di agar tenendo conto dell'organismo, dell'antibiotico utilizzato e delle dimensioni della piastra per evitare sovrapposizioni delle zone di inibizione.
NOTA: da quattro a cinque dischi possono essere alloggiati su una piastra circolare da 90 mm.
- Con l'aiuto di una pinza sterilizzata a fiamma, trasferire asetticamente i dischi antibiotici sulle piastre MHA inoculate e premere delicatamente i dischi per garantire un contatto completo con l'agar.
- Incubazione delle piastre
- Entro 15 minuti dall'applicazione dei dischi antibiotici, capovolgere le piastre e incubare a 37 °C durante la notte.
- Interpretazione dei risultati
- Misurare il diametro ZOI in millimetri (mm) e interpretare in base ai valori di breakpoint forniti da EUCAST11. Vedi i due esempi riportati di seguito.
- Il punto di rottura del diametro della zona (mm) per un disco antibiotico di ciprofloxacina (5 μg) per Enterobacterales è S ≥ 25 e R < 22, il che significa che è considerato sensibile (S) se ZOI ≥ 25 mm, mentre è resistente (R) se ZOI < 22 mm. Se il diametro ZOI scende tra 22 e 25, l'isolato è considerato intermedio (I).
- Il punto di rottura del diametro della zona (mm) per un disco antibiotico cloramfenicolo (30 μg) per Staphylococcus spp. è S ≥ 18 e R < 18, il che significa che è considerato sensibile se ZOI ≥ 18 mm, mentre è resistente se ZOI < 18 mm.
- Determinare l'indice di resistenza agli antibiotici multipli (MAR) trovando il rapporto tra il numero di antibiotici a cui l'isolato è resistente e il numero totale di antibiotici a cui l'isolato è esposto.
NOTA: Per il controllo di qualità, E. coli ATCC 25922 e S. aureo Gli ATCC 29213 sono utilizzati come ceppi di riferimento seguendo il protocollo come nella fase 4.
- Misurare il diametro ZOI in millimetri (mm) e interpretare in base ai valori di breakpoint forniti da EUCAST11. Vedi i due esempi riportati di seguito.
5. Rilevazione basata sulla PCR di geni di resistenza agli antibiotici negli isolati
- Utilizzare un protocollo PCR standard per l'identificazione degli ARG negli isolati. Preparare il modello di DNA utilizzando il protocollo indicato al punto 3.1.
NOTA: Le condizioni di ciclo della PCR utilizzate in questo studio erano 94 °C per 10 minuti, seguite da 35 cicli di 94 °C per 30 s, ricottura per 30 s alla temperatura appropriata (come standardizzato per ciascun set di primer), estensione a 72 °C per 40 s e estensione finale a 72 °C per 5 minuti. La miscela di reazione è descritta nella tabella 4. L'elenco degli ARG, dei primer e delle temperature di ricottura è riportato nella tabella 5. - Per risolvere, visualizzare e verificare la purezza degli ampliconi, seguire i passaggi 3.2.3-3.2.10.
6. Analisi del DNA metagenomico integrale per l'identificazione della diversità batterica totale e la rilevazione di ARG nel metagenoma
- Estrazione del DNA totale (metagenoma) dal campione d'acqua
- Estrarre il DNA metagenomico dai campioni di acqua filtrata.
NOTA: Nel presente studio, il DNA metagenomico (DNA totale) è stato estratto dai campioni di acqua filtrata utilizzando il kit di isolamento del DNA di riferimento seguendo il protocollo del produttore (vedere la tabella dei materiali). - Controllare la qualità del DNA caricando 3 μL del DNA metagenomico estratto su un gel di agarosio allo 0,8% e far scorrere il gel a 80-110 V per circa 30 minuti.
- Verificare la presenza di una singola banda intatta.
- Controllare la concentrazione di DNA utilizzando un fluorometro.
- Estrarre il DNA metagenomico dai campioni di acqua filtrata.
- Determinazione della diversità batterica e rilevazione di ARG mediante sequenziamento dell'intero DNA metagenomico
- Preparazione della libreria e amplificazione PCR:
- Preparare una libreria di sequenziamento con estremità accoppiate utilizzando il kit di preparazione della libreria del DNA di riferimento (vedere la tabella dei materiali).
- Preparare il DNA per la legatura dell'adattatore prendendo 200 ng di DNA e tagliandolo meccanicamente in frammenti più piccoli, seguito da una fase continua di riparazione finale in cui viene aggiunta una "A" alle estremità 3'.
- A seconda della piattaforma utilizzata per il sequenziamento, applicare adattatori specifici a entrambe le estremità dei frammenti di DNA.
NOTA: in questi adattatori sono presenti sequenze cruciali per l'associazione di librerie con doppio codice a barre a una cella di flusso per il sequenziamento. Ciò consente l'amplificazione PCR dei frammenti legati all'adattatore e il legame dei primer di sequenziamento standard. - Per verificare la qualità e la quantità, analizzare la libreria amplificata utilizzando un chip DNA ad alta sensibilità secondo le istruzioni del produttore.
- Generazione e sequenziamento dei cluster:
- Caricare la libreria amplificata sulla piattaforma di sequenziazione appropriata per la generazione e la successiva sequenziazione dei cluster.
NOTA: Le molecole di libreria si legano agli oligo adattatori complementari sulla cella di flusso accoppiata. Durante il sequenziamento, i filamenti anteriori vengono selettivamente scissi dopo la risintesi del filamento inverso. Questo filamento inverso copiato viene quindi sequenziato dall'estremità opposta del frammento.
- Caricare la libreria amplificata sulla piattaforma di sequenziazione appropriata per la generazione e la successiva sequenziazione dei cluster.
- Analisi bioinformatica:
- Genera scaffold dai dati di alta qualità utilizzando la piattaforma appropriata.
- Sottoporre questi scaffold ad analisi bioinformatica per la classificazione tassonomica e l'identificazione degli ARG.
NOTA: Il flusso di lavoro per l'analisi dell'intero DNA metagenomico per l'identificazione della diversità batterica totale e la rilevazione di ARG nel metagenoma è riportato nella Figura 1. Un foglio di flusso della metodologia completa descritta nel manoscritto è riportato nella Figura 2.
- Preparazione della libreria e amplificazione PCR:
Risultati
Carica batterica totale e conteggio dei batteri resistenti agli antibiotici (AR)
Il calcolo della carica batterica totale è stato effettuato diffondendo da 10−4 a 10−6 diluizioni dei campioni d'acqua su R2A Agar, mezzo modificato. Per l'enumerazione della conta dei batteri AR, sono state distribuite diluizioni da 10−3 a 10−6 volte su piastre contenenti antibiotici (Figura 3). I conteggi totali e dei batteri AR sono stat...
Discussione
La raccolta e l'elaborazione dei campioni svolgono un ruolo significativo e potrebbero influenzare i risultati e l'interpretazione dello studio. Pertanto, per escludere la variabilità nei campioni, è importante effettuare campionamenti in più punti del corpo d'acqua dolce studiato. Mantenere condizioni ambientali asettiche adeguate durante la manipolazione di tali campioni può prevenire la contaminazione. Inoltre, per evitare cambiamenti nella composizione batterica che possono influenzare la qualità e la quantità ...
Divulgazioni
Gli autori non hanno interessi contrastanti da rivelare.
Riconoscimenti
Questo lavoro è stato parzialmente sostenuto da sovvenzioni finanziarie del Department of Science and Technology-Promotion of University Research and Scientific Excellence (DST-PURSE) Scheme dell'Università di Mumbai. Devika Ghadigaonkar ha lavorato come Project Fellow nell'ambito del programma. Si riconosce l'aiuto tecnico fornito da Harshali Shinde, Senior Research Fellow presso il progetto n. del Department of Science and Technology-Science and Engineering Research Board (DST-SERB): CRG/2018/003624.
Materiali
| Name | Company | Catalog Number | Comments |
| 100 bp DNA ladder | Himedia | MBT049-50LN | For estimation of size of the amplicons |
| 2x PCR Taq mastermix | HiMedia | MBT061-50R | For making PCR reaction mixture |
| 37 °C Incubator | GS-192, Gayatri Scientific | NA | For incubation of bacteria |
| 6x Gel Loading Buffer | HiMedia | ML015-1ML | Loading and Tracking dye which helps to weigh down the DNA sample and track the progress of electrophoresis |
| Agarose powder | Himedia | MB229-50G | For resolving amplicons during Agarose Gel Electrophoresis (AGE) |
| Ampicillin antibiotic disc | HiMedia | SD002 | For performing AST |
| Autoclave | Equitron | NA | Required for sterilization of media, glass plates, test tubes, etc |
| Bioanalyzer 2100 | Agilent Technologies | NA | To check the quality and quantity of the amplified library |
| Bisafety B2 Cabinet | IMSET | IMSET BSC-Class II Type B2 | Used for microbiological work like bacterial culturing, AST etc. |
| Cefotaxime antibiotic disc | HiMedia | SD295E-5VL | For performing AST |
| Cefotaxime antibiotic powder | HiMedia | TC352-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Ceftriaxone antibiotic disc | HiMedia | SD065 | For performing AST |
| Centrifuge Minispin | Eppendorf | Minispin Plus-5453 | Used to pellet the debris during crude DNA preparation |
| Chloramphenicol antibiotic disc | HiMedia | SD006-5x50DS | For performing AST |
| Ciprofloxacin antibiotic disc | HiMedia | SD060-5x50DS | For performing AST |
| Ciprofloxacin antibiotic powder | HiMedia | TC447-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Colorimeter | Quest | NA | For checking the OD of culture suspensions |
| Comprehensive Antibiotic Resistance Database (CARD) database | functional annotation of ARGs; https://card.mcmaster.ca/ | ||
| Cooling Shaker Incubator | BTL41 Allied Scientific | NA | For incubation of media plates for culturing bacteria |
| Deep Freezer (-40 °C) | Haier | DW40L, Haier Biomedicals | For storage of glycerol stocks |
| DNA Library Prep Kit | NEB Next Ultra DNA Library Prep Kit for Illumina | NA | Paired-end sequencing library preparation |
| EDTA | HiMedia | GRM1195-100G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Electrophoresis Apparatus | TechResource | 15 cm gel casting tray | For making the agarose gel and carrying out electrophoresis |
| Electrophoresis Power pack with electrodes | Genei | NA | For running the AGE |
| Erythromycin antibiotic disc | HiMedia | SD222-5VL | For performing AST |
| Erythromycin antibiotic powder | HiMedia | CMS528-1G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Erythromycin antibiotic powder | HiMedia | TC024-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Escherichia coli ATCC 25922 | HiMedia | 0335X-1 | Used as a control while performing AST |
| Ethidium Bromide | HiMedia | MB071-1G | Intercalating agent and visualizaion of DNA after electrophoresis under Gel Documentation System |
| Fluorometer | Qubit 2.0 | NA | For determining concentration of extracted metagenomic DNA |
| Gel Documentation System | BioRad | Used for visualizing PCR amplicons after electrophoresis | |
| Gentamicin antibiotic disc | HiMedia | SD170-5x50DS | For performing AST |
| Glacial Acetic Acid | HiMedia | AS119-500ML | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Glycerol | HiMedia | GRM1027-500ML | For making glycerol stocks |
| Imipenem antibiotic disc | HiMedia | SD073 | For performing AST |
| Kaiju Database | NA | NA | For taxonomical classification of reads; https://kaiju.binf.ku.dk/ |
| Kanamycin antibiotic disc | HiMedia | SD017-5x50DS | For performing AST |
| Kanamycin antibiotic powder | HiMedia | MB105-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Levofloxacin antibiotic disc | HiMedia | SD216-5VL | For performing AST |
| Luria Bertani broth | Himedia | M1245-500G | For enrichment of cultures |
| McFarland Standards | Himedia | R092-1No | To compare density of culture suspension |
| Molecular Biology water | HiMedia | TCL018-500ML | For making PCR reaction mixture |
| Mueller-Hinton Agar (MHA) | HiMedia | M173-500G | For performing Antibiotc Susceptibility Testing (AST) |
| Neomycin antibiotic disc | HiMedia | SD731-5x50DS | For performing AST |
| PCR Gradient Thermal Cycler | Eppendorf | Mastercycler Nexus Gradient 230V/50-60 Hz | Used for performing PCR for amplification of 16S rRNA region and various Antibiotic Resistance genes |
| Primers | Xcelris | NA | For PCR amplication |
| R2A Agar, Modified | HiMedia | M1743 | For preparation of media plates for isolation of total and antibiotic resistant (AR) bacterial load |
| Scaffold generation | CLC Genomics Workbench 6.0 | NA | For generation of scaffolds |
| Sequencer | Illumina platform (2 x 150 bp chemistry) | NA | Sequencing of amplified library |
| Sodium Chloride | HiMedia | TC046-500G | For preparation of 0.85% saline for serially diluting the water sample |
| Soil DNA isolation Kit | Xcelgen | NA | For extraction of whole metagenomic DNA from the filtered water sample |
| Staphylococcus aureus subsp. aureus ATCC 29213 | HiMedia | 0365P | Used as a control while performing AST |
| Taxonomical Classification | Kaiju ioinformatics tool | NA | For classification of reads into different taxonomic groups from phylum to genus level |
| The Comprehensive Antibiotic Resistance Database (CARD) | NA | NA | For functional annotation of ARGs |
| Tigecycline antibiotic disc | HiMedia | SD278 | For performing AST |
| Trimethoprim antibiotic disc | HiMedia | SD039-5x50DS | For performing AST |
| Tris base | HiMedia | TC072-500G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Vancomycin antibiotic powder | HiMedia | CMS217 | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Weighing Balance | Mettler Toledo | ME204 Mettler Toledo | Used for weighing media powders, reagent powders etc. |
| NA - Not Applicable |
Riferimenti
- Prestinaci, F., Pezzotti, P., Pantosti, A. Antimicrobial resistance: A global multifaceted phenomenon. Pathogens and Global Health. 109 (7), 309-318 (2015).
- Knight, G., et al. Antimicrobial resistance and COVID-19: Intersections and implications. Elife. 10, 64139 (2021).
- Ventola, C. L. The antibiotic resistance crisis: Part 1: Causes and threats. Pharmacy and Therapeutics. 40 (4), 277-283 (2015).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J. R., Rath, A. Metagenomic analysis of total microbial diversity and antibiotic resistance of culturable microorganisms in raw chicken meat and mung sprouts (Phaseolus aureus) sold in retail markets of Mumbai. India. Current Science. 113 (1), 71-79 (2017).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J., Rath, A. Characterization of multiple antibiotic resistance of culturable microorganisms and metagenomic analysis of total microbial diversity of marine fish sold in retail shops in Mumbai, India. Environmental Science and Pollution Research. 25 (7), 6228-6239 (2018).
- Czekalski, N., GascónDíez, E., Bürgmann, H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake. The ISME Journal. 8 (7), 1381-1390 (2014).
- Kraemer, S., Ramachandran, A., Perron, G. Antibiotic pollution in the environment: From microbial ecology to public policy. Microorganisms. 7 (6), 180 (2019).
- Edmonds-Wilson, S., Nurinova, N., Zapka, C., Fierer, N., Wilson, M. Review of human hand microbiome research. Journal of Dermatological Science. 80 (1), 3-12 (2015).
- de Abreu, V., Perdigão, J., Almeida, S. Metagenomic approaches to analyze antimicrobial resistance: An overview. Frontiers in Genetics. 11, 575592 (2021).
- Carlson, S., et al. Detection of multiresistant Salmonella typhimurium DT104 using multiplex and fluorogenic PCR. Molecular and Cellular Probes. 13 (3), 213-222 (1999).
- Breakpoint tables for interpretation of MICs and zone diameters, Version 12.0. European Committee on Antimicrobial Susceptibility Testing Available from: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_12.0_Breakpoint_Tables.pdf (2022)
- Bharti, R., Grimm, D. Current challenges and best-practice protocols for microbiome analysis. Briefings in Bioinformatics. 22 (1), 178-193 (2019).
- Choo, J., Leong, L., Rogers, G. Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports. 5, 16350 (2015).
- Clinical and Laboratory Standards Institute. . Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria that Grow Aerobically, 11th edition. , (2015).
- Bayot, M., Bragg, B. Antimicrobial Susceptibility Testing. StatPearls. , (2021).
- Joseph, A. A., Odimayo, M. S., Olokoba, L. B., Olokoba, A. B., Popoola, G. O. Multiple antibiotic resistance index of Escherichia coli isolates in a tertiary hospital in South-West Nigeria. Medical Journal of Zambia. 44 (4), 225-232 (2017).
- Lorenz, T. Polymerase chain reaction: Basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Rolin, J. Food and human gut as reservoirs of transferable antibiotic resistance encoding genes. Frontiers in Microbiology. 4, 173 (2013).
- Racewicz, P., et al. Prevalence and characterisation of antimicrobial resistance genes and class 1 and 2 integrons in multiresistant Escherichia coli isolated from poultry production. Scientific Reports. 12, 6062 (2022).
- Gebreyes, W., Thakur, S. Multidrug-resistant Salmonella enterica serovar Muenchen from pigs and humans and potential interserovar transfer of antimicrobial resistance. Antimicrobial Agents and Chemotherapy. 49 (2), 503-511 (2005).
- Li, L., et al. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui Province, China. PLoS One. 9 (8), 104356 (2014).
- Akers, K., et al. Aminoglycoside resistance and susceptibility testing errors in Acinetobacter baumannii-calcoaceticus complex. Journal Of Clinical Microbiology. 48 (4), 1132-1138 (2010).
- Ciesielczuk, H. . Extra-intestinal pathogenic Escherichia coli in the UK: The importance in bacteraemia versus urinary tract infection, colonisation of widespread clones and specific virulence factors. , (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon