Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Isolierung und Identifizierung wässriger antibiotikaresistenter Bakterien und molekulare Charakterisierung ihrer Antibiotikaresistenzgene
In diesem Artikel
Zusammenfassung
Hier stellen wir ein detailliertes Protokoll für die Isolierung und Identifizierung von antibiotikaresistenten Bakterien aus Wasser und die molekulare Charakterisierung ihrer Antibiotikaresistenzgene (ARGs) vor. Die Verwendung von kulturbasierten und nicht-kulturbasierten (metagenomischen Analysen) Techniken liefert vollständige Informationen über die gesamte bakterielle Vielfalt und den Gesamtpool verschiedener ARGs, die in Süßgewässern aus Mumbai, Indien, vorhanden sind.
Zusammenfassung
Die Entwicklung und Ausbreitung von Antibiotikaresistenzen (AR) durch Mikrobiota in Verbindung mit Süßwasserkörpern ist ein großes globales Gesundheitsproblem. In der vorliegenden Studie wurden Süßwasserproben gesammelt und in Bezug auf die gesamte bakterielle Diversität und AR-Gene (ARGs) analysiert, wobei sowohl konventionelle kulturbasierte Techniken als auch ein kulturunabhängiger metagenomischer Ansatz mit hohem Durchsatz verwendet wurden. Dieser Artikel stellt ein systematisches Protokoll für die Zählung der gesamten und antibiotikaresistenten kultivierbaren Bakterien aus Süßwasserproben und die Bestimmung der phänotypischen und genotypischen Resistenz in den kultivierbaren Isolaten vor. Darüber hinaus berichten wir über die Verwendung der gesamten metagenomischen Analyse der gesamten metagenomischen DNA, die aus der Süßwasserprobe extrahiert wurde, zur Identifizierung der gesamten bakteriellen Vielfalt, einschließlich nicht kultivierbarer Bakterien, und zur Identifizierung des gesamten Pools verschiedener ARGs (Resistome) im Gewässer. Nach diesen detaillierten Protokollen beobachteten wir eine hohe Antibiotika-resistente Bakterienlast im Bereich von 9,6 × 10 5-1,2 × 109 KBE/ml. Die meisten Isolate waren resistent gegen die mehrfach getesteten Antibiotika, darunter Cefotaxim, Ampicillin, Levofloxacin, Chloramphenicol, Ceftriaxon, Gentamicin, Neomycin, Trimethoprim und Ciprofloxacin, mit multiplen Antibiotikaresistenzindizes (MAR) von ≥0,2, was auf eine hohe Resistenz der Isolate hinweist. Die 16S-rRNA-Sequenzierung identifizierte potenzielle humanpathogene wie Klebsiella pneumoniae und opportunistische Bakterien wie Comamonas spp., Micrococcus spp., Arthrobacter spp. und Aeromonas spp. Die molekulare Charakterisierung der Isolate zeigte das Vorhandensein verschiedener ARGs, wie blaTEM, blaCTX-M (β-Lactame), aadA, aac (6')-Ib (Aminoglykoside) und dfr1 (Trimethoprims), was auch durch die gesamte metagenomische DNA-Analyse bestätigt wurde. Eine hohe Prävalenz anderer ARGs, die für antibiotische Ausflusspumpen kodieren - mtrA, macB, mdtA, acrD, β-Lactamasen-SMB-1, VIM-20, ccrA, ampC, blaZ, das Chloramphenicol-Acetyltransferase-Gen catB10 und das Rifampicin-Resistenzgen rphB- wurde ebenfalls in der metagenomischen DNA nachgewiesen. Mit Hilfe der in dieser Studie diskutierten Protokolle bestätigten wir das Vorhandensein von wasserbürtigen MAR-Bakterien mit verschiedenen phänotypischen und genotypischen Merkmalen von AR. Daher kann die gesamte metagenomische DNA-Analyse als komplementäre Technik zu herkömmlichen kulturbasierten Techniken verwendet werden, um den gesamten AR-Status eines Gewässers zu bestimmen.
Einleitung
Antimikrobielle Resistenzen (AMR) wurden als eines der drängendsten globalen Probleme identifiziert. Die rasche Entwicklung antimikrobieller Resistenzen und ihre weltweite Ausbreitung sind eine der größten Bedrohungen für die menschliche Gesundheit und die Weltwirtschaft in Bezug auf die damit verbundenen Gesundheitskosten1. Der übermäßige Gebrauch und Missbrauch von Antibiotika haben zu einer Zunahme von AR geführt. Dies wurde durch die COVID-19-Pandemie unterstrichen, bei der die Behandlung von assoziierten Sekundärinfektionen in vielen Fällen aufgrund von AMR bei den betroffenen Patienten stark beeinträchtigt war2. Neben der direkten Verwendung/dem Missbrauch von Antibiotika durch den Menschen sind der übermäßige Einsatz und Missbrauch von Antibiotika in der Landwirtschaft und Tierhaltung und ihre unsachgemäße Einleitung in die Umwelt, einschließlich der Gewässer, eingroßes Problem 3. Das Aufkommen neuer Resistenzmerkmale und Multiresistenzen bei Bakterien unterstreicht dringend die Notwendigkeit eines besseren Verständnisses der Faktoren, die zur Entwicklung von AR und ihrer Verbreitung führen. Mehrere antibiotikaresistente Bakterien, die oft mehrere AR-Gene (ARGs) auf mobilen genetischen Elementen wie Plasmiden tragen, können diese Resistenzgene auf nicht-resistente Mikroorganismen, einschließlich potenzieller humaner Krankheitserreger, übertragen, was zur Entstehung von Superbugs führt, die selbst mit Antibiotika als letztes Mittel nicht behandelbar sind4. Diese multiplen antibiotikaresistenten Bakterien können, wenn sie in Wasserökosystemen vorkommen, direkt über den Verzehr von kontaminierten wasserbasierten Lebensmitteln wie Fischen, Krabben und Weichtieren in den menschlichen Darm gelangen. Frühere Studien haben gezeigt, dass die Ausbreitung von AR-Bakterien in natürlich vorkommenden Wassersystemen auch andere Wasservorräte, einschließlich Trinkwasser, erreichen und so in die menschliche Nahrungskette gelangen kann 5,6,7.
Das Ziel der vorliegenden Studie ist es, ein umfassendes Protokoll zu erstellen, das eine Kombination aus kulturbasierten und nicht-kulturbasierten (gesamte metagenomische Analyse) Techniken verwendet, um vollständige Informationen über die gesamte bakterielle Vielfalt und den Gesamtpool verschiedener ARGs in einem Gewässer in Mumbai, Indien, zu erhalten. Herkömmlicherweise wurden kulturbasierte Techniken verwendet, um die bakterielle Vielfalt in Gewässern zu untersuchen. Da kultivierbare Mikroorganismen nur einen kleinen Prozentsatz der gesamten Mikrobiota in jeder Nische ausmachen, müssen verschiedene kulturbasierte und kulturunabhängige Techniken gleichzeitig eingesetzt werden, um den Gesamtstatus der bakteriellen Vielfalt und die verschiedenen Resistenzmerkmale in jeder Probe besser zu verstehen. Eine solche robuste und zuverlässige kulturunabhängige Technik ist die gesamte metagenomische DNA-Analyse. Diese Hochdurchsatzmethode wurde erfolgreich in verschiedenen Studien zur bakteriellen Diversität oder den funktionellen Annotationen verschiedener ARGs 8,9 eingesetzt. Diese Technik nutzt das Metagenom (das gesamte Erbgut einer Probe) als Ausgangsmaterial für verschiedene Analysen und ist somit kulturunabhängig. Die Protokolle in der vorliegenden Studie können für die gesamte metagenomische DNA-Analyse verwendet werden, um Informationen über die gesamte bakterielle Diversität und verschiedene ARGs (Resistome) in Wasserproben zu erhalten.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Probenentnahme und -verarbeitung
- Musterkollektion
- Sammeln Sie das entsprechende Volumen der Wasserprobe in sterilen Probenbehältern und stellen Sie sicher, dass nicht mehr als 3/4 des Behälters gefüllt ist.
- Transportieren Sie die Proben so schnell wie möglich nach der Entnahme unter aseptischen Bedingungen ins Labor und verarbeiten Sie sie sofort.
- Probenbearbeitung
- Filtern Sie die Wasserprobe aseptisch durch ein steriles Musselintuch, um Partikel zu entfernen.
- Führen Sie geeignete serielle Verdünnungen des gefilterten Wassers zur weiteren Analyse durch.
2. Abschätzung der Gesamtkeimbelastung und der Keimzahl der Antibiotikaresistenzen
- Bestimmung der Gesamtkeimbelastung
- 18,12 g R2A-Agar, modifiziertes Pulver, in 1.000 ml doppelt destilliertem Wasser suspendieren und die Mischung durch Erhitzen auflösen. Das gelöste Gemisch wird bei 121 °C, 15 psi für 20 Minuten autoklaviert. Bereiten Sie R2A-Agar, modifizierte Platten vor, indem Sie die entsprechende Menge des autoklavierten Gemisches in sterile Petriplatten gießen (z. B. etwa 20 ml autoklaviertes steriles Medium zu einer 90 mm sterilen Petriplatte).
- Verteilen Sie gleichmäßig 100 μL der entsprechenden Verdünnungen der gefilterten Wasserprobe auf die R2A-Agar-modifizierte Platte, sobald das Medium erstarrt ist. Führen Sie das Experiment doppelt durch.
- Alle oben genannten Platten werden 48 Stunden lang bei 35-37 °C inkubiert (je nach verwendetem Medium variieren Temperatur und Inkubationszeit).
- Drücken Sie die Gesamtbakterienbelastung in koloniebildenden Einheiten pro Milliliter (KBE/ml) unter Verwendung von Gleichung (1) aus:
 (1)
(1)
- Bestimmung der AR-Keimzahl
- Befolgen Sie die Schritte 2.1.1-2.1.4. Anstelle von R2A-Agar verwenden modifizierte Platten jedoch R2A-Agar, modifizierte Platten, die einzeln mit fünf verschiedenen Antibiotika ergänzt werden, nämlich Cefotaxim (3 μg / ml), Ciprofloxacin (0,5 μg / ml), Erythromycin (20 μg / ml), Kanamycin (15 μg / ml) und Vancomycin (3 μg / ml).
- Die Antibiotika werden separat in Röhrchen gegeben, die 20 ml steriles geschmolzenes R2A-Agar, modifiziert (mit der Temperatur des geschmolzenen R2A-Agars, modifiziert bei ≤40 °C), enthalten, um die endgültige Antibiotikakonzentration gemäß Schritt 2.2.1 zu erreichen.
- Zum gleichmäßigen Mischen schwenken und auf sterile Petriplatten gießen, bevor der Agar erstarrt. Führen Sie das Experiment doppelt durch.
- Inkubieren Sie alle oben genannten Platten bei 35-37 °C für 48 h (bei Verwendung eines anderen Mediums können Temperatur und Inkubationszeit variieren).
- Zur Qualitätskontrolle und Überprüfung der Wirksamkeit der Antibiotika verteilen Sie 100 μL Bakteriensuspensionen der Stämme Escherichia coli ATCC 25922 und Staphylococcus aureus ATCC 29213 auf ihre jeweiligen antibiotikahaltigen R2A-Agar, modifizierte Platten (stellen Sie sicher, dass die Dichte der für die Impfung verwendeten Frischkultur OD = 0,5 bei 600 nm beträgt).
- Die Anzahl der antibiotikaresistenten Keime in KBE/ml wird bestimmt, wie in Schritt 2.1.4 beschrieben.
- Glycerinvorräte der Isolate
- Wählen Sie morphologisch unterschiedliche AR-Kolonien aus.
- Eine einzelne isolierte Kolonie wird in 2 ml steriler Luria-Bertani-Bouillon suspendiert, die das entsprechende Antibiotikum enthält (z. B. wenn eine Kolonie aus einer Platte ausgewählt wurde, die Cefotaxim enthält, wird die Kolonie aus Schritt 2.3.1 in steriler Luria-Bertani-Bouillon, die Cefotaxim in ihrer jeweiligen Konzentration enthält, beimpft).
- Die beimpften Röhrchen werden bei 37 °C bei 80 U/min inkubiert, bis der OD600 0,5 erreicht.
- Glycerinvorräte der Isolate werden hergestellt, indem 750 μL der Kultursuspension aus Schritt 2.3.3 unter aseptischen Bedingungen in 250 μL steriles 100%iges Glycerin gemischt werden.
- Lagern Sie die Glycerinvorräte bis zur weiteren Analyse bei −80 °C.
HINWEIS: Zur Wiederbelebung der Kulturen aus den Glycerinvorräten die Glycerinvorräte bei 4 °C auftauen. Eine Schleife voll dieser Brühe in 2 ml sterile Luria-Bertani-Bouillon mit dem jeweiligen Antibiotikum einimpfen und wachsen lassen.
3. Identifizierung kultivierbarer Bakterien durch 16S-rRNA-Gensequenzierung
- Herstellung eines DNA-Templates aus den Isolaten für die PCR
ANMERKUNG: Das beschriebene Protokoll für die Herstellung einer DNA-Vorlage für die PCR zur Isolierung von Roh-DNA aus den Bakterien wird von Carlson et al.10 gegeben.- Nehmen Sie mit einem sterilen Zahnstocher eine einzelne, isolierte, reine Kolonie des Isolats, das auf einer Petriplatte wächst. Die Bakterienkolonie in 100 μL sterilem, doppelt destilliertem Wasser in einem sterilen Mikrozentrifugenröhrchen suspendieren und 10 min kochen lassen.
- Zentrifugieren Sie die Suspension bei 10.000 × g für 2 Minuten, um die Trümmer zu pelletieren, und überführen Sie den Überstand in ein frisches steriles Mikrozentrifugenröhrchen, das als rohe DNA-Vorlage verwendet wird.
- Gezielte PCR-Amplifikation der V3-Region des 16S-rRNA-Gens und Sequenzierung
- 40 μL des Reaktionsgemisches werden in einem PCR-Röhrchen für die PCR-Amplifikation hergestellt, wie in Tabelle 1 beschrieben.
HINWEIS: Die DNA-Präparation sollte auf einem Eisblock durchgeführt werden, wobei die Gefahr einer Kontamination minimiert werden sollte (tragen Sie Handschuhe bei der Handhabung der Reagenzien und reinigen Sie die Arbeitsfläche gründlich mit 70% Ethanol). - Legen Sie das Röhrchen in den Thermoblock und führen Sie das entsprechende Programm im PCR-Thermocycler aus. Siehe Tabelle 2 für die standardisierten PCR-Zyklusbedingungen und die Primer-Informationen für die Amplifikation der V3-Regionen der 16S-rRNA-Gene.
- Zur Auflösung der Amplikone und Visualisierung wird eine Agarose-Gelelektrophorese (AGE) durchgeführt. Mischen Sie 10 μL des amplifizierten PCR-Produkts und 2 μL 6x Gelbeladungspuffer (Tabelle 3) und laden Sie dieses Gemisch in Vertiefungen auf 1,5%igem Agarosegel (lösen Sie 1,5 g Agarosepulver in 100 mL 1x TAE-Puffer [Tabelle 3]), das 5 μL 10 mg/ml Ethidiumbromid (EtBr) enthält, bis zu einer Endkonzentration von 0,5 μg/ml EtBr in 100 ml des Agarosegels.
VORSICHT: EtBr ist ein starkes Karzinogen. Beim Umgang mit EtBr und Gelen, die EtBr enthalten, sollten immer Handschuhe getragen werden. - Fügen Sie eine DNA-Leiter hinzu, um die Größe der Amplikonen abzuschätzen.
- Elektrophorese des Gels in einem TAE-Tankpuffer bei 80-100 V durchführen.
- Sobald der Tracking-Farbstoff 3/4 des Gels verläuft, stoppen Sie die Elektrophorese und visualisieren Sie die Amplikonbänder unter einem UV-Transilluminator.
- Verwenden Sie das PCR-Produkt (Amplicon) für die 16S-rRNA-Gensequenzierung, um das Isolat zu identifizieren.
- Quantifizieren Sie das Amplikon, indem Sie es einer spektralphotometrischen Analyse mit Gleichung (2) unterziehen.
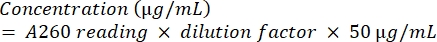 (2)
(2) - Um die Reinheit der DNA zu überprüfen, berechnen Sie das Verhältnis von A260 zu A280.
HINWEIS: Idealerweise sollte diese Zahl über 1,5 und vorzugsweise zwischen 1,8 und 2,0 liegen. - Um die Isolate zu identifizieren, vergleichen Sie die erhaltenen Sequenzen mit Sequenzdatenbanken mithilfe eines geeigneten Alignment-Suchwerkzeugs.
- 40 μL des Reaktionsgemisches werden in einem PCR-Röhrchen für die PCR-Amplifikation hergestellt, wie in Tabelle 1 beschrieben.
4. Nachweis von Antibiotikaresistenzen in den Isolaten mittels Antibiotika-Empfindlichkeitstests
HINWEIS: Dieses Protokoll beschreibt die Methode für die Antibiotika-Empfindlichkeitsprüfung (AST) durch Bandscheibendiffusion. Folgende antibiotische Bandscheiben wurden verwendet: Cefotaxim (5 μg), Ampicillin (10 μg), Levofloxacin (5 μg), Chloramphenicol (30 μg), Tigecyclin (15 μg), Ceftriaxon (30 μg), Imipenem (10 μg), Gentamicin (10 μg), Neomycin (10 μg), Trimethoprim (5 μg) und Ciprofloxacin (5 μg).
- Vorbereitung des Inokulums für die AST
- Eine einzelne, isolierte, gereinigte AR-Kolonie wird unter Verwendung einer sterilen Schleife in 2 ml eines sterilen, nicht selektiven Mediums, wie z. B. Luria-Bertani-Bouillon (ohne Antibiotikum), aseptisch beimpft und über Nacht bei 37 °C bei 80 U/min inkubiert.
- Resuspendieren, indem 100-150 μL der über Nacht gewachsenen Kultur (ca. OD 600 = 1,8-2,0) in 2 ml frischem, nicht selektivem Luria-Bertani-Brühmedium entnommen und 2-4 h inkubiert werden (bis der OD600 0,4-0,5 erreicht).
- Diese frisch gezüchtete Kultursuspension wird mit steriler 0,85%iger Kochsalzlösung so verdünnt, dass die Dichte der Kultur dem McFarland-Standard von 0,5 entspricht (ungefähr OD600 = 0,1), was ungefähr 1-2 × 108 Zellen/ml entspricht.
- Mischen Sie die Bakteriensuspension vorsichtig für eine gleichmäßige Zellverteilung.
- Verwenden Sie die obige Suspension innerhalb von 15 Minuten nach der Verdünnung.
- Beimpfung der Agarplatten
- Bereiten Sie Müller-Hinton-Agarplatten (MHA) für die Durchführung der AST vor, indem Sie 38 g MHA in 1.000 ml doppelt destilliertem Wasser mischen und die Mischung durch Erhitzen auflösen. Das gelöste Gemisch wird bei 121 °C, 15 psi für 15 min autoklaviert.
- Stellen Sie sicher, dass die MHA-Tiefe in den Platten 4 mm beträgt (25 ml Medium pro Platte).
- Nehmen Sie gleichzeitig die antibiotischen Scheiben aus dem Gefrierschrank und erwärmen Sie sie auf Raumtemperatur.
HINWEIS: Die antibiotischen Scheiben sollten allmählich aufgetaut werden, indem die Scheiben zunächst bei 4 °C und später bei Raumtemperatur aufgetaut werden, um eine mögliche Kondensationsgefahr auf den Scheiben zu verringern, die sich anschließend auf die Hemmzone (ZOI) auswirken kann. - Unter aseptischen Bedingungen wird ein steriles Wattestäbchen in das in Schritt 4.1 vorbereitete Inokulum getaucht und überschüssige Suspension entfernt, um eine Überbeimpfung der Platten zu vermeiden.
- Verteilen Sie die Kultur gleichmäßig auf den Tellern, beginnend mit der Oberseite der MHA-Platte und von Rand zu Rand hin und her. Drehen Sie die Platte beim Abtupfen um 60°.
- Anwendung der antibiotischen Scheiben
- Übertragen Sie die antibiotischen Scheiben mit Hilfe einer flammsterilisierten Pinzette aseptisch auf die inokulierten MHA-Platten und drücken Sie die Scheiben vorsichtig an, um einen vollständigen Kontakt mit dem Agar zu gewährleisten.
HINWEIS: Dieser Vorgang muss innerhalb von 15 Minuten nach der Beimpfung der Kultur auf den Platten durchgeführt werden. - Legen Sie die entsprechende Anzahl von Antibiotikascheiben auf die Agarplatte unter Berücksichtigung des Organismus, des verwendeten Antibiotikums und der Größe der Platte, um eine Überlappung der Hemmzonen zu vermeiden.
HINWEIS: Vier bis fünf Scheiben können auf einer 90 mm Rundplatte untergebracht werden.
- Übertragen Sie die antibiotischen Scheiben mit Hilfe einer flammsterilisierten Pinzette aseptisch auf die inokulierten MHA-Platten und drücken Sie die Scheiben vorsichtig an, um einen vollständigen Kontakt mit dem Agar zu gewährleisten.
- Inkubation der Platten
- Innerhalb von 15 min nach dem Auftragen der antibiotischen Scheiben die Platten umdrehen und über Nacht bei 37 °C inkubieren.
- Interpretation der Ergebnisse
- Messen Sie den ZOI-Durchmesser in Millimetern (mm) und interpretieren Sie gemäß den Haltepunktwerten von EUCAST11. Sehen Sie sich die beiden unten aufgeführten Beispiele an.
- Der Bruchpunkt des Zonendurchmessers (mm) für eine Ciprofloxacin-Antibiotikascheibe (5 μg) für Enterobacterales beträgt S ≥ 25 und R < 22, was bedeutet, dass sie als empfindlich (S) gilt, wenn ZOI 25 mm ≥, während sie resistent ist (R), wenn ZOI 22 mm <. Liegt der ZOI-Durchmesser zwischen 22 und 25, gilt das Isolat als intermediär (I).
- Der Bruchpunkt des Zonendurchmessers (mm) für eine Chloramphenicol-Antibiotikascheibe (30 μg) für Staphylococcus spp. ist S ≥ 18 und R < 18, was bedeutet, dass er als empfindlich gilt, wenn ZOI 18 mm ≥, während er resistent ist, wenn ZOI 18 mm <.
- Bestimmen Sie den Index der multiplen Antibiotikaresistenz (MAR), indem Sie das Verhältnis der Anzahl der Antibiotika, gegen die das Isolat resistent ist, zur Gesamtzahl der Antibiotika, denen das Isolat ausgesetzt ist, ermitteln.
HINWEIS: Für die Qualitätskontrolle E. coli ATCC 25922 und S. aureus ATCC 29213 werden als Referenzstämme gemäß dem Protokoll in Schritt 4 verwendet.
- Messen Sie den ZOI-Durchmesser in Millimetern (mm) und interpretieren Sie gemäß den Haltepunktwerten von EUCAST11. Sehen Sie sich die beiden unten aufgeführten Beispiele an.
5. PCR-basierter Nachweis von Antibiotikaresistenzgenen in den Isolaten
- Verwenden Sie ein Standard-PCR-Protokoll für die Identifizierung von ARGs in den Isolaten. Bereiten Sie die DNA-Vorlage unter Verwendung des in Schritt 3.1 angegebenen Protokolls vor.
ANMERKUNG: Die in dieser Studie verwendeten PCR-Zyklusbedingungen betrugen 94 °C für 10 Minuten, gefolgt von 35 Zyklen von 94 °C für 30 s, Glühen für 30 s bei der entsprechenden Temperatur (wie für jeden Primer-Satz standardisiert), Dehnung bei 72 °C für 40 s und eine abschließende Verlängerung bei 72 °C für 5 Minuten. Das Reaktionsgemisch ist in Tabelle 4 beschrieben. Die Liste der ARGs, Primer und Glühtemperaturen ist in Tabelle 5 aufgeführt. - Führen Sie die Schritte 3.2.3 bis 3.2.10 aus, um die Reinheit der Amplikonen aufzulösen, zu visualisieren und zu überprüfen.
6. Metagenomische DNA-Analyse zur Identifizierung der gesamten bakteriellen Diversität und zum Nachweis von ARGs im Metagenom
- Extraktion der gesamten DNA (Metagenom) aus der Wasserprobe
- Extrahieren Sie die metagenomische DNA aus den gefilterten Wasserproben.
HINWEIS: In der aktuellen Studie wurde die metagenomische DNA (Gesamt-DNA) aus den gefilterten Wasserproben unter Verwendung des referenzierten DNA-Isolationskits gemäß dem Protokoll des Herstellers extrahiert (siehe Materialtabelle). - Überprüfen Sie die Qualität der DNA, indem Sie 3 μL der extrahierten metagenomischen DNA auf ein 0,8%iges Agarose-Gel laden und das Gel bei 80-110 V für ca. 30 Minuten laufen lassen.
- Prüfen Sie, ob ein einzelnes intaktes Band vorhanden ist.
- Überprüfen Sie die DNA-Konzentration mit einem Fluorometer.
- Extrahieren Sie die metagenomische DNA aus den gefilterten Wasserproben.
- Bestimmung der bakteriellen Diversität und Detektion von ARGs mittels metagenomischer DNA-Sequenzierung
- Bibliotheksvorbereitung und PCR-Amplifikation:
- Bereiten Sie eine Paired-End-Sequenzierungsbibliothek mit dem referenzierten DNA-Bibliotheksvorbereitungskit vor (siehe Materialtabelle).
- Bereiten Sie die DNA für die Adapterligatur vor, indem Sie 200 ng DNA nehmen und mechanisch in kleinere Fragmente scheren, gefolgt von einem kontinuierlichen Schritt der Endreparatur, bei dem ein "A" an die 3'-Enden angehängt wird.
- Abhängig von der Plattform, die für die Sequenzierung verwendet wird, ligieren Sie spezifische Adapter an beiden Enden der DNA-Fragmente.
HINWEIS: Sequenzen, die für die Bindung von Dual-Barcode-Bibliotheken an eine Flusszelle für die Sequenzierung entscheidend sind, sind in diesen Adaptern vorhanden. Dies ermöglicht die PCR-Amplifikation der adapterligierten Fragmente und die Bindung der Standard-Sequenzierungsprimer. - Um die Qualität und Quantität zu überprüfen, analysieren Sie die amplifizierte Bibliothek mit einem hochempfindlichen DNA-Chip gemäß den Anweisungen des Herstellers.
- Cluster-Generierung und -Sequenzierung:
- Laden Sie die erweiterte Bibliothek auf die entsprechende Sequenzierungsplattform für die Clustergenerierung und die anschließende Sequenzierung.
HINWEIS: Die Bibliotheksmoleküle binden an die komplementären Adapter-Oligos auf der Paired-End-Durchflusszelle. Bei der Sequenzierung werden die Vorwärtsstränge nach der Resynthese des Reversestrangs selektiv gespalten. Dieser kopierte umgekehrte Strang wird dann vom gegenüberliegenden Ende des Fragments sequenziert.
- Laden Sie die erweiterte Bibliothek auf die entsprechende Sequenzierungsplattform für die Clustergenerierung und die anschließende Sequenzierung.
- Bioinformatische Analyse:
- Generieren Sie aus den hochwertigen Daten Scaffolds mit der entsprechenden Plattform.
- Unterziehen Sie diese Gerüste einer bioinformatischen Analyse für die taxonomische Klassifizierung und Identifizierung der ARGs.
HINWEIS: Der Arbeitsablauf für die gesamte metagenomische DNA-Analyse zur Identifizierung der gesamten bakteriellen Diversität und zum Nachweis von ARGs im Metagenom ist in Abbildung 1 dargestellt. Abbildung 2 zeigt ein Flussdiagramm der vollständigen Methodik, die im Manuskript beschrieben ist.
- Bibliotheksvorbereitung und PCR-Amplifikation:
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Gesamtkeimbelastung und Anzahl der antibiotikaresistenten (AR) Bakterien
Die Zählung der Gesamtbakterienbelastung erfolgte durch Verteilen von 10−4- bis 10−6-fachen Verdünnungen der Wasserproben auf R2A-Agar, modifiziertes Medium. Für die Zählung der AR-Bakterienzahl wurden 10−3- bis 10−6-fache Verdünnungen auf antibiotikahaltige Medienplatten aufgetragen (Abbildung 3). Die Gesamt- und AR-Bakterienzahlen wurden als ...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die Probenentnahme und -verarbeitung spielt eine wichtige Rolle und kann die Ergebnisse und die Interpretation der Studie beeinflussen. Um eine Variabilität der Proben auszuschließen, ist es daher wichtig, die Probenahme an mehreren Stellen des untersuchten Süßwasserkörpers durchzuführen. Die Aufrechterhaltung geeigneter aseptischer Umgebungsbedingungen bei der Handhabung solcher Proben kann eine Kontamination verhindern. Um Veränderungen in der Bakterienzusammensetzung zu vermeiden, die die Qualität und Quantit?...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine widersprüchlichen Interessen offenzulegen.
Danksagungen
Diese Arbeit wurde teilweise durch finanzielle Zuschüsse des Department of Science and Technology-Promotion of University Research and Scientific Excellence (DST-PURSE) Scheme der University of Mumbai unterstützt. Devika Ghadigaonkar arbeitete als Projektstipendiatin im Rahmen des Programms. Die technische Hilfe von Harshali Shinde, Senior Research Fellow unter der Projektnummer CRG/2018/003624 des Department of Science and Technology-Science and Engineering Research Board (DST-SERB), wird gewürdigt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 100 bp DNA ladder | Himedia | MBT049-50LN | For estimation of size of the amplicons |
| 2x PCR Taq mastermix | HiMedia | MBT061-50R | For making PCR reaction mixture |
| 37 °C Incubator | GS-192, Gayatri Scientific | NA | For incubation of bacteria |
| 6x Gel Loading Buffer | HiMedia | ML015-1ML | Loading and Tracking dye which helps to weigh down the DNA sample and track the progress of electrophoresis |
| Agarose powder | Himedia | MB229-50G | For resolving amplicons during Agarose Gel Electrophoresis (AGE) |
| Ampicillin antibiotic disc | HiMedia | SD002 | For performing AST |
| Autoclave | Equitron | NA | Required for sterilization of media, glass plates, test tubes, etc |
| Bioanalyzer 2100 | Agilent Technologies | NA | To check the quality and quantity of the amplified library |
| Bisafety B2 Cabinet | IMSET | IMSET BSC-Class II Type B2 | Used for microbiological work like bacterial culturing, AST etc. |
| Cefotaxime antibiotic disc | HiMedia | SD295E-5VL | For performing AST |
| Cefotaxime antibiotic powder | HiMedia | TC352-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Ceftriaxone antibiotic disc | HiMedia | SD065 | For performing AST |
| Centrifuge Minispin | Eppendorf | Minispin Plus-5453 | Used to pellet the debris during crude DNA preparation |
| Chloramphenicol antibiotic disc | HiMedia | SD006-5x50DS | For performing AST |
| Ciprofloxacin antibiotic disc | HiMedia | SD060-5x50DS | For performing AST |
| Ciprofloxacin antibiotic powder | HiMedia | TC447-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Colorimeter | Quest | NA | For checking the OD of culture suspensions |
| Comprehensive Antibiotic Resistance Database (CARD) database | functional annotation of ARGs; https://card.mcmaster.ca/ | ||
| Cooling Shaker Incubator | BTL41 Allied Scientific | NA | For incubation of media plates for culturing bacteria |
| Deep Freezer (-40 °C) | Haier | DW40L, Haier Biomedicals | For storage of glycerol stocks |
| DNA Library Prep Kit | NEB Next Ultra DNA Library Prep Kit for Illumina | NA | Paired-end sequencing library preparation |
| EDTA | HiMedia | GRM1195-100G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Electrophoresis Apparatus | TechResource | 15 cm gel casting tray | For making the agarose gel and carrying out electrophoresis |
| Electrophoresis Power pack with electrodes | Genei | NA | For running the AGE |
| Erythromycin antibiotic disc | HiMedia | SD222-5VL | For performing AST |
| Erythromycin antibiotic powder | HiMedia | CMS528-1G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Erythromycin antibiotic powder | HiMedia | TC024-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Escherichia coli ATCC 25922 | HiMedia | 0335X-1 | Used as a control while performing AST |
| Ethidium Bromide | HiMedia | MB071-1G | Intercalating agent and visualizaion of DNA after electrophoresis under Gel Documentation System |
| Fluorometer | Qubit 2.0 | NA | For determining concentration of extracted metagenomic DNA |
| Gel Documentation System | BioRad | Used for visualizing PCR amplicons after electrophoresis | |
| Gentamicin antibiotic disc | HiMedia | SD170-5x50DS | For performing AST |
| Glacial Acetic Acid | HiMedia | AS119-500ML | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Glycerol | HiMedia | GRM1027-500ML | For making glycerol stocks |
| Imipenem antibiotic disc | HiMedia | SD073 | For performing AST |
| Kaiju Database | NA | NA | For taxonomical classification of reads; https://kaiju.binf.ku.dk/ |
| Kanamycin antibiotic disc | HiMedia | SD017-5x50DS | For performing AST |
| Kanamycin antibiotic powder | HiMedia | MB105-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Levofloxacin antibiotic disc | HiMedia | SD216-5VL | For performing AST |
| Luria Bertani broth | Himedia | M1245-500G | For enrichment of cultures |
| McFarland Standards | Himedia | R092-1No | To compare density of culture suspension |
| Molecular Biology water | HiMedia | TCL018-500ML | For making PCR reaction mixture |
| Mueller-Hinton Agar (MHA) | HiMedia | M173-500G | For performing Antibiotc Susceptibility Testing (AST) |
| Neomycin antibiotic disc | HiMedia | SD731-5x50DS | For performing AST |
| PCR Gradient Thermal Cycler | Eppendorf | Mastercycler Nexus Gradient 230V/50-60 Hz | Used for performing PCR for amplification of 16S rRNA region and various Antibiotic Resistance genes |
| Primers | Xcelris | NA | For PCR amplication |
| R2A Agar, Modified | HiMedia | M1743 | For preparation of media plates for isolation of total and antibiotic resistant (AR) bacterial load |
| Scaffold generation | CLC Genomics Workbench 6.0 | NA | For generation of scaffolds |
| Sequencer | Illumina platform (2 x 150 bp chemistry) | NA | Sequencing of amplified library |
| Sodium Chloride | HiMedia | TC046-500G | For preparation of 0.85% saline for serially diluting the water sample |
| Soil DNA isolation Kit | Xcelgen | NA | For extraction of whole metagenomic DNA from the filtered water sample |
| Staphylococcus aureus subsp. aureus ATCC 29213 | HiMedia | 0365P | Used as a control while performing AST |
| Taxonomical Classification | Kaiju ioinformatics tool | NA | For classification of reads into different taxonomic groups from phylum to genus level |
| The Comprehensive Antibiotic Resistance Database (CARD) | NA | NA | For functional annotation of ARGs |
| Tigecycline antibiotic disc | HiMedia | SD278 | For performing AST |
| Trimethoprim antibiotic disc | HiMedia | SD039-5x50DS | For performing AST |
| Tris base | HiMedia | TC072-500G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Vancomycin antibiotic powder | HiMedia | CMS217 | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Weighing Balance | Mettler Toledo | ME204 Mettler Toledo | Used for weighing media powders, reagent powders etc. |
| NA - Not Applicable |
Referenzen
- Prestinaci, F., Pezzotti, P., Pantosti, A. Antimicrobial resistance: A global multifaceted phenomenon. Pathogens and Global Health. 109 (7), 309-318 (2015).
- Knight, G., et al. Antimicrobial resistance and COVID-19: Intersections and implications. Elife. 10, 64139(2021).
- Ventola, C. L. The antibiotic resistance crisis: Part 1: Causes and threats. Pharmacy and Therapeutics. 40 (4), 277-283 (2015).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J. R., Rath, A. Metagenomic analysis of total microbial diversity and antibiotic resistance of culturable microorganisms in raw chicken meat and mung sprouts (Phaseolus aureus) sold in retail markets of Mumbai. India. Current Science. 113 (1), 71-79 (2017).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J., Rath, A. Characterization of multiple antibiotic resistance of culturable microorganisms and metagenomic analysis of total microbial diversity of marine fish sold in retail shops in Mumbai, India. Environmental Science and Pollution Research. 25 (7), 6228-6239 (2018).
- Czekalski, N., GascónDíez, E., Bürgmann, H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake. The ISME Journal. 8 (7), 1381-1390 (2014).
- Kraemer, S., Ramachandran, A., Perron, G. Antibiotic pollution in the environment: From microbial ecology to public policy. Microorganisms. 7 (6), 180(2019).
- Edmonds-Wilson, S., Nurinova, N., Zapka, C., Fierer, N., Wilson, M. Review of human hand microbiome research. Journal of Dermatological Science. 80 (1), 3-12 (2015).
- de Abreu, V., Perdigão, J., Almeida, S. Metagenomic approaches to analyze antimicrobial resistance: An overview. Frontiers in Genetics. 11, 575592(2021).
- Carlson, S., et al. Detection of multiresistant Salmonella typhimurium DT104 using multiplex and fluorogenic PCR. Molecular and Cellular Probes. 13 (3), 213-222 (1999).
- Breakpoint tables for interpretation of MICs and zone diameters, Version 12.0. European Committee on Antimicrobial Susceptibility Testing. , Available from: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_12.0_Breakpoint_Tables.pdf (2022).
- Bharti, R., Grimm, D. Current challenges and best-practice protocols for microbiome analysis. Briefings in Bioinformatics. 22 (1), 178-193 (2019).
- Choo, J., Leong, L., Rogers, G. Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports. 5, 16350(2015).
- Clinical and Laboratory Standards Institute. Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria that Grow Aerobically, 11th edition. , Clinical and Laboratory Standards Institute. Wayne, PA. (2015).
- Bayot, M., Bragg, B. Antimicrobial Susceptibility Testing. StatPearls. , StatPearls Publishing. Treasure Island, FL. (2021).
- Joseph, A. A., Odimayo, M. S., Olokoba, L. B., Olokoba, A. B., Popoola, G. O. Multiple antibiotic resistance index of Escherichia coli isolates in a tertiary hospital in South-West Nigeria. Medical Journal of Zambia. 44 (4), 225-232 (2017).
- Lorenz, T. Polymerase chain reaction: Basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998(2012).
- Rolin, J. Food and human gut as reservoirs of transferable antibiotic resistance encoding genes. Frontiers in Microbiology. 4, 173(2013).
- Racewicz, P., et al. Prevalence and characterisation of antimicrobial resistance genes and class 1 and 2 integrons in multiresistant Escherichia coli isolated from poultry production. Scientific Reports. 12, 6062(2022).
- Gebreyes, W., Thakur, S. Multidrug-resistant Salmonella enterica serovar Muenchen from pigs and humans and potential interserovar transfer of antimicrobial resistance. Antimicrobial Agents and Chemotherapy. 49 (2), 503-511 (2005).
- Li, L., et al. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui Province, China. PLoS One. 9 (8), 104356(2014).
- Akers, K., et al. Aminoglycoside resistance and susceptibility testing errors in Acinetobacter baumannii-calcoaceticus complex. Journal Of Clinical Microbiology. 48 (4), 1132-1138 (2010).
- Ciesielczuk, H. Extra-intestinal pathogenic Escherichia coli in the UK: The importance in bacteraemia versus urinary tract infection, colonisation of widespread clones and specific virulence factors. , Queen Mary University of London. PhD thesis (2015).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten