Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Isolement et identification de bactéries résistantes aux antibiotiques d’origine hydrique et caractérisation moléculaire de leurs gènes de résistance aux antibiotiques
Dans cet article
Résumé
Nous présentons ici un protocole détaillé pour l’isolement et l’identification de bactéries résistantes aux antibiotiques à partir de l’eau et la caractérisation moléculaire de leurs gènes de résistance aux antibiotiques (ARG). L’utilisation de techniques basées sur la culture et non basées sur la culture (analyse métagénomique) fournit des informations complètes sur la diversité bactérienne totale et le pool total de différents ARG présents dans les eaux douces de Mumbai, en Inde.
Résumé
Le développement et la propagation de la résistance aux antibiotiques (RA) par le microbiote associé aux masses d’eau douce est un problème de santé mondial majeur. Dans la présente étude, des échantillons d’eau douce ont été prélevés et analysés en fonction de la diversité bactérienne totale et des gènes AR (ARG) à l’aide de techniques conventionnelles basées sur la culture et d’une approche métagénomique indépendante de la culture à haut débit. Cet article présente un protocole systématique pour le dénombrement des bactéries cultivables totales et résistantes aux antibiotiques à partir d’échantillons d’eau douce et la détermination de la résistance phénotypique et génotypique dans les isolats cultivables. En outre, nous signalons l’utilisation de l’analyse métagénomique complète de l’ADN métagénomique total extrait de l’échantillon d’eau douce pour l’identification de la diversité bactérienne globale, y compris les bactéries non cultivables, et l’identification du pool total de différents ARG (résistome) dans le plan d’eau. En suivant ces protocoles détaillés, nous avons observé une charge bactérienne élevée résistante aux antibiotiques de l’ordre de 9,6 × 10 5-1,2 × 109 UFC / mL. La plupart des isolats étaient résistants aux multiples antibiotiques testés, y compris la céfotaxime, l’ampicilline, la lévofloxacine, le chloramphénicol, la ceftriaxone, la gentamicine, la néomycine, le triméthoprime et la ciprofloxacine, avec des indices de résistance multiple aux antibiotiques (PMA) de ≥0,2, indiquant des niveaux élevés de résistance dans les isolats. Le séquençage de l’ARNr 16S a identifié des agents pathogènes humains potentiels, tels que Klebsiella pneumoniae, et des bactéries opportunistes, telles que Comamonas spp., Micrococcus spp., Arthrobacter spp. et Aeromonas spp. La caractérisation moléculaire des isolats a montré la présence de divers ARG, tels que blaTEM, blaCTX-M (β-lactamines), aadA, aac (6')-Ib (aminoglycosides) et dfr1 (triméthoprimes), ce qui a également été confirmé par l’analyse de l’ADN métagénomique complet. Une prévalence élevée d’autres ARG codant pour les pompes à efflux d’antibiotiques - mtrA, macB, mdtA, acrD, β-lactamases-SMB-1, VIM-20, ccrA, ampC, blaZ, le gène de la chloramphénicol acétyltransférase catB10 et le gène de résistance à la rifampicine rphB - a également été détectée dans l’ADN métagénomique. À l’aide des protocoles discutés dans cette étude, nous avons confirmé la présence de bactéries MAR d’origine hydrique présentant divers traits phénotypiques et génotypiques de l’AR. Ainsi, l’analyse de l’ADN métagénomique entier peut être utilisée comme technique complémentaire aux techniques conventionnelles basées sur la culture pour déterminer l’état global de la RA d’un plan d’eau.
Introduction
La résistance aux antimicrobiens (RAM) a été identifiée comme l’un des problèmes mondiaux les plus urgents. L’évolution rapide de la RAM et sa propagation mondiale constituent l’une des plus grandes menaces pour la santé humaine et l’économie mondiale en termes de coûts sanitaires qui y sont associés1. La surutilisation et la mauvaise utilisation des antibiotiques ont entraîné une augmentation de l’AR. Cela a été mis en évidence par la pandémie de COVID-19, au cours de laquelle le traitement des infections secondaires associées, dans de nombreux cas, a été considérablement compromis en raison de la RAM chez les patients touchés2. Outre l’utilisation directe ou abusive d’antibiotiques par l’homme, la surutilisation et la mauvaise utilisation des antibiotiques dans l’agriculture et l’élevage et leur rejet inapproprié dans l’environnement, y compris les masses d’eau, constituent une préoccupation majeure3. L’augmentation de nouveaux caractères de résistance et de multirésistance chez les bactéries souligne de toute urgence la nécessité de mieux comprendre les facteurs conduisant au développement de l’AR et à sa dissémination. De multiples bactéries résistantes aux antibiotiques, qui portent souvent plusieurs gènes AR (ARG) sur des éléments génétiques mobiles tels que les plasmides, peuvent transférer ces gènes de résistance à des microorganismes non résistants, y compris des agents pathogènes humains potentiels, conduisant ainsi à l’émergence de superbactéries qui ne peuvent être traitées même avec des antibiotiques de dernier recours4. Ces multiples bactéries résistantes aux antibiotiques, si elles sont présentes dans les écosystèmes aquatiques, peuvent pénétrer directement dans l’intestin humain via la consommation d’aliments contaminés à base d’eau tels que le poisson, les crabes et les mollusques. Des études antérieures ont montré que la propagation des bactéries AR dans les systèmes d’eau naturels peut également atteindre d’autres sources d’approvisionnement en eau, y compris l’eau potable, et, par conséquent, peut entrer dans la chaîne alimentaire humaine 5,6,7.
L’objectif de la présente étude est de fournir un protocole complet utilisant une combinaison de techniques basées sur la culture et non basées sur la culture (analyse métagénomique complète) pour obtenir des informations complètes sur la diversité bactérienne totale et le pool total de différents ARG présents dans un plan d’eau à Mumbai, en Inde. Classiquement, des techniques basées sur la culture ont été utilisées pour étudier la diversité bactérienne dans les plans d’eau. Étant donné que les micro-organismes cultivables ne constituent qu’un faible pourcentage du microbiote total dans n’importe quelle niche, pour mieux comprendre l’état général de la diversité bactérienne et les divers caractères de résistance prévalent dans tout échantillon, diverses techniques basées sur la culture et indépendantes de la culture doivent être utilisées en tandem. L’une de ces techniques robustes et fiables indépendantes de la culture est l’analyse de l’ADN métagénomique entier. Cette méthode à haut débit a été utilisée avec succès dans diverses études sur la diversité bactérienne ou les annotations fonctionnelles de divers ARG 8,9. Cette technique utilise le métagénome (le matériel génétique total d’un échantillon) comme matériau de départ pour diverses analyses et, par conséquent, est indépendante de la culture. Les protocoles de la présente étude peuvent être utilisés pour l’analyse de l’ADN métagénomique entier afin d’obtenir des informations sur la diversité bactérienne totale et divers ARG (résistomes) dans les échantillons d’eau.
Protocole
1. Prélèvement et traitement des échantillons
- Prélèvement d’échantillons
- Prélever le volume approprié de l’échantillon d’eau dans un ou plusieurs récipients d’échantillon stériles, en veillant à ce que les 3/4 au plus du contenant ne soient pas remplis.
- Transporter les échantillons au laboratoire dans des conditions aseptiques dès que possible après le prélèvement et les traiter immédiatement.
- Traitement des échantillons
- Filtrer de manière aseptique l’échantillon d’eau à travers un chiffon de mousseline stérile pour éliminer toute particule.
- Effectuer des dilutions en série appropriées de l’eau filtrée pour une analyse plus approfondie.
2. Estimation de la charge bactérienne totale et du nombre de bactéries résistantes aux antibiotiques
- Détermination de la charge bactérienne totale
- Suspendre 18,12 g de gélose R2A, poudre modifiée dans 1 000 mL d’eau bidistillée et dissoudre le mélange par chauffage. Autoclaver le mélange dissous à 121 °C, 15 psi pendant 20 min. Préparer la gélose R2A, Plaques modifiées en versant la quantité appropriée du mélange autoclavé dans des plaques de Petri stériles (p. ex., ajouter environ 20 mL de milieu stérile autoclavé sur une plaque de Petri stérile de 90 mm).
- Répartir uniformément 100 μL des dilutions appropriées de l’échantillon d’eau filtrée sur la gélose R2A, Plaque modifiée une fois que le milieu s’est solidifié. Effectuez l’expérience en double.
- Incuber toutes les plaques ci-dessus à 35-37 °C pendant 48 h (faire varier la température et le temps d’incubation en fonction du milieu utilisé pour l’isolement).
- Exprimez la charge bactérienne totale en unités formant colonies par millilitre (UFC/mL) à l’aide de l’équation (1) :
 (1)
(1)
- Détermination du nombre de bactéries AR
- Suivez les étapes 2.1.1-2.1.4. Cependant, au lieu de la gélose R2A, plaques modifiées, utilisez la gélose R2A, les plaques modifiées complétées individuellement avec cinq antibiotiques différents, à savoir le céfotaxime (3 μg/mL), la ciprofloxacine (0,5 μg/mL), l’érythromycine (20 μg/mL), la kanamycine (15 μg/mL) et la vancomycine (3 μg/mL).
- Ajouter les antibiotiques séparément dans des tubes contenant 20 ml de gélose R2A fondue stérile, modifiée (avec la température de la gélose R2A fondue, modifiée à ≤40 °C) pour atteindre la concentration finale d’antibiotique mentionnée à l’étape 2.2.1.
- Remuer pour un mélange uniforme et verser sur des plaques de Petri stériles avant que la gélose ne se solidifie. Effectuez l’expérience en double.
- Incuber toutes les plaques ci-dessus à 35-37 °C pendant 48 h (si vous utilisez un milieu différent, la température et le temps d’incubation peuvent varier).
- Pour le contrôle de la qualité et la vérification de l’efficacité des antibiotiques, étaler 100 μL de suspensions bactériennes des souches ATCC 25922 et Staphylococcus aureus ATCC 29213 d’Escherichia coli sur leur gélose R2A modifiée contenant des antibiotiques respectifs (s’assurer que la densité de la culture fraîche utilisée pour l’inoculation est OD = 0,5 à 600 nm).
- Déterminer le nombre de bactéries résistantes aux antibiotiques en termes d’UFC/ml comme décrit à l’étape 2.1.4.
- Stocks de glycérol des isolats
- Sélectionnez des colonies de RA morphologiquement distinctes.
- Suspendre une seule colonie isolée dans 2 mL de bouillon stérile Luria-Bertani contenant l’antibiotique correspondant (p. ex., si une colonie a été choisie dans une plaque contenant du céfotaxime, inoculer la colonie de l’étape 2.3.1 dans un bouillon Luria-Bertani stérile contenant du céfotaxime à sa concentration respective).
- Incuber les tubes inoculés à 37 °C à 80 tr/min jusqu’à ce que le DO600 atteigne 0,5.
- Préparer les stocks de glycérol des isolats en mélangeant 750 μL de la suspension de culture de l’étape 2.3.3 dans 250 μL de glycérol stérile à 100 % dans des conditions aseptiques.
- Conservez les stocks de glycérol à -80 °C jusqu’à une analyse plus approfondie.
NOTE: Pour la relance des cultures à partir des stocks de glycérol, décongeler les stocks de glycérol à 4 ° C. Inoculer une boucle de ce bouillon dans 2 ml de bouillon stérile Luria-Bertani contenant l’antibiotique respectif et laisser pousser.
3. Identification de bactéries cultivables par séquençage du gène de l’ARNr 16S
- Préparation d’un modèle d’ADN à partir des isolats pour la PCR
NOTE: Le protocole décrit pour la préparation du modèle d’ADN pour la PCR pour l’isolement de l’ADN brut de la bactérie est donné par Carlson et al.10.- À l’aide d’un cure-dent stérile, prélever une seule colonie isolée et pure de l’isolat qui pousse sur une plaque de Petri. Suspendre la colonie bactérienne dans 100 μL d’eau stérile bidistillée dans un tube microcentrifuge stérile et faire bouillir pendant 10 min.
- Centrifuger la suspension à 10 000 × g pendant 2 minutes pour enrober les débris et transférer le surnageant dans un tube microcentrifuge frais et stérile pour l’utiliser comme matrice d’ADN brut.
- Amplification PCR ciblée de la région V3 du gène de l’ARNr 16S et séquençage
- Préparer 40 μL du mélange réactionnel dans un tube de PCR pour l’amplification par PCR, comme mentionné dans le tableau 1.
NOTE: La préparation de l’ADN doit être effectuée sur un bloc de glace tout en minimisant les risques de contamination (porter des gants lors de la manipulation des réactifs et nettoyer soigneusement la surface de travail avec de l’éthanol à 70%). - Placez le tube dans le bloc thermique et exécutez le programme approprié dans le cycleur thermique PCR. Voir le tableau 2 pour les conditions de cycle PCR normalisées et les informations d’amorce pour l’amplification des régions V3 des gènes de l’ARNr 16S.
- Pour résoudre les amplicons et visualiser, effectuez une électrophorèse sur gel d’agarose (AGE). Mélanger 10 μL du produit de PCR amplifié et 2 μL du tampon de chargement de gel 6x (tableau 3), et charger ce mélange dans des puits sur du gel d’agarose à 1,5 % (dissoudre 1,5 g de poudre d’agarose dans 100 mL de 1x tampon TAE [Tableau 3]) contenant 5 μL de bromure d’éthidium (EtBr) à une concentration finale de 0,5 μg/mL d’EtBr dans 100 mL du gel d’agarose.
ATTENTION : L’EtBr est un cancérogène puissant. Les gants doivent être portés en tout temps lors de la manipulation de l’EtBr et des gels contenant de l’EtBr. - Ajouter une échelle d’ADN pour l’estimation de la taille des amplicons.
- Effectuer l’électrophorèse du gel dans un tampon de réservoir TAE à 80-100 V.
- Une fois que le colorant de suivi exécute les 3/4 du gel, arrêtez l’électrophorèse et visualisez les bandes d’amplicon sous un transilluminateur UV.
- Utilisez le produit PCR (amplicon) pour le séquençage du gène de l’ARNr 16S afin d’identifier l’isolat.
- Quantifier l’amplicon en le soumettant à une analyse spectrophotométrique à l’aide de l’équation (2).
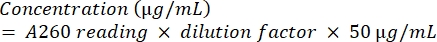 (2)
(2) - Pour vérifier la pureté de l’ADN, calculez le rapport A260/A280.
REMARQUE: Idéalement, ce nombre devrait être supérieur à 1,5 et, de préférence, compris entre 1,8 et 2,0. - Pour identifier les isolats, comparez les séquences obtenues avec les bases de données de séquences à l’aide d’un outil de recherche d’alignement approprié.
- Préparer 40 μL du mélange réactionnel dans un tube de PCR pour l’amplification par PCR, comme mentionné dans le tableau 1.
4. Détection de la résistance aux antibiotiques dans les isolats à l’aide de tests de sensibilité aux antibiotiques
REMARQUE : Ce protocole décrit la méthode de test de sensibilité aux antibiotiques (AST) par diffusion discale. Les disques antibiotiques suivants ont été utilisés : céfotaxime (5 μg), ampicilline (10 μg), lévofloxacine (5 μg), chloramphénicol (30 μg), tigécycline (15 μg), ceftriaxone (30 μg), imipénème (10 μg), gentamicine (10 μg), néomycine (10 μg), triméthoprime (5 μg) et ciprofloxacine (5 μg).
- Préparation de l’inoculum pour l’AST
- Inoculer de manière aseptique une seule colonie de RA purifiée isolée à l’aide d’une boucle stérile dans 2 mL d’un milieu stérile non sélectif, tel que le bouillon Luria-Bertani (sans antibiotique), et incuber à 37 °C à 80 tr/min pendant la nuit.
- Resuspendre en prenant 100-150 μL de la culture cultivée pendant la nuit (environ, DO 600 = 1,8-2,0) dans 2 mL de milieu de bouillon frais non sélectif Luria-Bertani et incuber pendant 2-4 h (jusqu’à ce que la DO600 atteigne 0,4-0,5).
- Diluer cette suspension de culture fraîchement cultivée en utilisant une solution saline stérile à 0,85 % de telle sorte que la densité de la culture soit égale à 0,5 McFarland standard (approximativement,OD 600 = 0,1), ce qui correspond à peu près à 1-2 × 108 cellules/mL.
- Mélangez délicatement la suspension bactérienne pour une répartition uniforme des cellules.
- Utiliser la suspension ci-dessus dans les 15 minutes suivant la dilution.
- Inoculation des plaques de gélose
- Préparer les plaques de gélose Mueller-Hinton (MHA) pour effectuer l’AST en mélangeant 38 g de MHA dans 1 000 ml d’eau bidistillée et dissoudre le mélange par chauffage. Autoclaver le mélange dissous à 121 °C, 15 psi pendant 15 min.
- S’assurer que la profondeur de MHA dans les plaques est de 4 mm (25 mL de milieu par plaque).
- Simultanément, retirez les disques antibiotiques du congélateur et réchauffez-les à température ambiante.
NOTE: Les disques antibiotiques doivent être décongelés progressivement en décongelant d’abord les disques à 4 ° C et plus tard à température ambiante pour réduire tout risque potentiel de condensation sur les disques, ce qui peut affecter ultérieurement la zone d’inhibition (ZOI). - Dans des conditions aseptiques, tremper un coton-tige stérile dans l’inoculum préparé à l’étape 4.1 et retirer l’excès de suspension pour éviter la surinoculation des plaques.
- Répartir la culture uniformément sur les plaques, en commençant par le haut de la plaque MHA et en allant et venant d’un bord à l’autre. Tournez la plaque de 60° pendant l’écouvillonnage.
- Application des disques antibiotiques
- À l’aide de pinces stérilisées à la flamme, transférer de manière aseptique les disques antibiotiques sur les plaques de MHA inoculées et appuyer doucement sur les disques pour assurer un contact complet avec l’agar-agar.
NOTE: Cette procédure doit être effectuée dans les 15 minutes suivant l’inoculation de la culture sur les plaques. - Placer le nombre approprié de disques antibiotiques sur la plaque de gélose en tenant compte de l’organisme, de l’antibiotique utilisé et de la taille de la plaque pour éviter le chevauchement des zones d’inhibition.
NOTE: Quatre à cinq disques peuvent être logés sur une plaque circulaire de 90 mm.
- À l’aide de pinces stérilisées à la flamme, transférer de manière aseptique les disques antibiotiques sur les plaques de MHA inoculées et appuyer doucement sur les disques pour assurer un contact complet avec l’agar-agar.
- Incubation des plaques
- Dans les 15 minutes suivant l’application des disques antibiotiques, inverser les plaques et incuber à 37 °C pendant la nuit.
- Interprétation des résultats
- Mesurer le diamètre ZOI en millimètres (mm) et interpréter en fonction des valeurs de point d’arrêt données par EUCAST11. Voir les deux exemples donnés ci-dessous.
- Le point de rupture du diamètre de la zone (mm) pour un disque antibiotique à base de ciprofloxacine (5 μg) pour les entérobactéries est S ≥ 25 et R < 22, ce qui signifie qu’il est considéré comme sensible (S) si ZOI ≥ 25 mm, alors qu’il est résistant (R) si ZOI < 22 mm. Si le diamètre du ZOI se situe entre 22 et 25, l’isolat est considéré comme intermédiaire (I).
- Le point de rupture du diamètre de la zone (mm) pour un disque antibiotique chloramphénicol (30 μg) pour Staphylococcus spp. est S ≥ 18 et R < 18, ce qui signifie qu’il est considéré comme sensible si ZOI ≥ 18 mm, alors qu’il est résistant si ZOI < 18 mm.
- Déterminer l’indice de résistance multiple aux antibiotiques (PMA) en trouvant le rapport entre le nombre d’antibiotiques auxquels l’isolat est résistant et le nombre total d’antibiotiques auxquels l’isolat est exposé.
NOTA : Pour le contrôle de la qualité, E. coli ATCC 25922 et S. Aureus Les souches ATCC 29213 sont utilisées comme souches de référence conformément au protocole de l’étape 4.
- Mesurer le diamètre ZOI en millimètres (mm) et interpréter en fonction des valeurs de point d’arrêt données par EUCAST11. Voir les deux exemples donnés ci-dessous.
5. Détection par PCR des gènes de résistance aux antibiotiques dans les isolats
- Utiliser un protocole PCR standard pour l’identification des ARG dans les isolats. Préparez le modèle d’ADN en utilisant le protocole donné à l’étape 3.1.
NOTE: Les conditions de cycle PCR utilisées dans cette étude étaient de 94 °C pendant 10 min, suivies de 35 cycles de 94 °C pendant 30 s, d’un recuit pendant 30 s à la température appropriée (normalisée pour chaque jeu d’amorces), d’une extension à 72 °C pendant 40 s et d’une extension finale à 72 °C pendant 5 min. Le mélange réactionnel est décrit dans le tableau 4. La liste des ARG, des amorces et des températures de recuit est donnée dans le tableau 5. - Pour résoudre, visualiser et vérifier la pureté des amplicons, suivez les étapes 3.2.3-3.2.10.
6. Analyse de l’ADN métagénomique entier pour l’identification de la diversité bactérienne totale et la détection des ARG dans le métagénome
- Extraction de l’ADN total (métagénome) de l’échantillon d’eau
- Extraire l’ADN métagénomique des échantillons d’eau filtrée.
REMARQUE : Dans la présente étude, l’ADN métagénomique (ADN total) a été extrait des échantillons d’eau filtrée à l’aide de la trousse d’isolement de l’ADN référencée conformément au protocole du fabricant (voir le tableau des matériaux). - Vérifiez la qualité de l’ADN en chargeant 3 μL de l’ADN métagénomique extrait sur un gel d’agarose à 0,8% et faites fonctionner le gel à 80-110 V pendant environ 30 minutes.
- Vérifiez la présence d’une seule bande intacte.
- Vérifiez la concentration d’ADN à l’aide d’un fluoromètre.
- Extraire l’ADN métagénomique des échantillons d’eau filtrée.
- Détermination de la diversité bactérienne et détection des ARG par séquençage de l’ADN métagénomique entier
- Préparation de la bibliothèque et amplification PCR :
- Préparez une bibliothèque de séquençage d’extrémités appariées à l’aide du kit de préparation de la bibliothèque d’ADN référencé (voir le tableau des matériaux).
- Préparez l’ADN pour la ligature de l’adaptateur en prenant 200 ng d’ADN et en le cisaillant mécaniquement en fragments plus petits, suivi d’une étape continue de réparation finale dans laquelle un « A » est ajouté aux extrémités 3'.
- Selon la plate-forme utilisée pour le séquençage, ligaturez des adaptateurs spécifiques aux deux extrémités des fragments d’ADN.
REMARQUE : des séquences cruciales pour lier des bibliothèques à double code-barres à une cellule de flux pour le séquençage sont présentes dans ces cartes. Cela permet l’amplification par PCR des fragments ligaturés par adaptateur et la liaison des amorces de séquençage standard. - Pour vérifier la qualité et la quantité, analysez la bibliothèque amplifiée à l’aide d’une puce à ADN haute sensibilité selon les instructions du fabricant.
- Génération et séquençage des clusters :
- Chargez la bibliothèque amplifiée sur la plate-forme de séquençage appropriée pour la génération de cluster et le séquençage ultérieur.
REMARQUE: Les molécules de la bibliothèque se lient aux oligos adaptateurs complémentaires sur la cellule d’écoulement d’extrémité appariée. Pendant le séquençage, les brins avant sont sélectivement clivés après la resynthèse du brin inverse. Ce brin inverse copié est ensuite séquencé à partir de l’extrémité opposée du fragment.
- Chargez la bibliothèque amplifiée sur la plate-forme de séquençage appropriée pour la génération de cluster et le séquençage ultérieur.
- Analyse bioinformatique :
- Générez des échafaudages à partir des données de haute qualité à l’aide de la plate-forme appropriée.
- Soumettre ces échafaudages à une analyse bioinformatique pour la classification taxonomique et l’identification des ARG.
REMARQUE: Le flux de travail pour l’analyse de l’ADN métagénomique complet pour l’identification de la diversité bactérienne totale et la détection des ARG dans le métagénome est donné à la figure 1. Un schéma de traitement de la méthodologie complète décrite dans le manuscrit est donné à la figure 2.
- Préparation de la bibliothèque et amplification PCR :
Résultats
Charge bactérienne totale et nombre de bactéries résistantes aux antibiotiques (EI)
Le dénombrement de la charge bactérienne totale a été effectué en étalant des dilutions de 10−4 à 10−6 fois des échantillons d’eau sur gélose R2A, milieu modifié. Pour le dénombrement du nombre de bactéries AR, des dilutions de 10−3 à 10−6 fois ont été réparties sur des plaques de milieu contenant des antibiotiques (figure 3
Discussion
La collecte et le traitement des échantillons jouent un rôle important et peuvent avoir une incidence sur les résultats et l’interprétation de l’étude. Par conséquent, pour exclure la variabilité des échantillons, il est important d’effectuer un échantillonnage à plusieurs endroits de la masse d’eau douce étudiée. Le maintien de conditions environnementales aseptiques appropriées lors de la manipulation de tels échantillons peut prévenir la contamination. En outre, pour éviter toute modification d...
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts contradictoires à divulguer.
Remerciements
Ce travail a été partiellement soutenu par des subventions financières du programme DST-PURSE (Department of Science and Technology-Promotion of University Research and Scientific Excellence) de l’Université de Mumbai. Devika Ghadigaonkar a travaillé en tant que Project Fellow dans le cadre de ce programme. L’aide technique fournie par Harshali Shinde, chercheur principal au sein du Conseil de recherche en sciences et en génie du Département des sciences et de la technologie (DST-SERB) Projet no: CRG/2018/003624, est reconnue.
matériels
| Name | Company | Catalog Number | Comments |
| 100 bp DNA ladder | Himedia | MBT049-50LN | For estimation of size of the amplicons |
| 2x PCR Taq mastermix | HiMedia | MBT061-50R | For making PCR reaction mixture |
| 37 °C Incubator | GS-192, Gayatri Scientific | NA | For incubation of bacteria |
| 6x Gel Loading Buffer | HiMedia | ML015-1ML | Loading and Tracking dye which helps to weigh down the DNA sample and track the progress of electrophoresis |
| Agarose powder | Himedia | MB229-50G | For resolving amplicons during Agarose Gel Electrophoresis (AGE) |
| Ampicillin antibiotic disc | HiMedia | SD002 | For performing AST |
| Autoclave | Equitron | NA | Required for sterilization of media, glass plates, test tubes, etc |
| Bioanalyzer 2100 | Agilent Technologies | NA | To check the quality and quantity of the amplified library |
| Bisafety B2 Cabinet | IMSET | IMSET BSC-Class II Type B2 | Used for microbiological work like bacterial culturing, AST etc. |
| Cefotaxime antibiotic disc | HiMedia | SD295E-5VL | For performing AST |
| Cefotaxime antibiotic powder | HiMedia | TC352-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Ceftriaxone antibiotic disc | HiMedia | SD065 | For performing AST |
| Centrifuge Minispin | Eppendorf | Minispin Plus-5453 | Used to pellet the debris during crude DNA preparation |
| Chloramphenicol antibiotic disc | HiMedia | SD006-5x50DS | For performing AST |
| Ciprofloxacin antibiotic disc | HiMedia | SD060-5x50DS | For performing AST |
| Ciprofloxacin antibiotic powder | HiMedia | TC447-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Colorimeter | Quest | NA | For checking the OD of culture suspensions |
| Comprehensive Antibiotic Resistance Database (CARD) database | functional annotation of ARGs; https://card.mcmaster.ca/ | ||
| Cooling Shaker Incubator | BTL41 Allied Scientific | NA | For incubation of media plates for culturing bacteria |
| Deep Freezer (-40 °C) | Haier | DW40L, Haier Biomedicals | For storage of glycerol stocks |
| DNA Library Prep Kit | NEB Next Ultra DNA Library Prep Kit for Illumina | NA | Paired-end sequencing library preparation |
| EDTA | HiMedia | GRM1195-100G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Electrophoresis Apparatus | TechResource | 15 cm gel casting tray | For making the agarose gel and carrying out electrophoresis |
| Electrophoresis Power pack with electrodes | Genei | NA | For running the AGE |
| Erythromycin antibiotic disc | HiMedia | SD222-5VL | For performing AST |
| Erythromycin antibiotic powder | HiMedia | CMS528-1G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Erythromycin antibiotic powder | HiMedia | TC024-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Escherichia coli ATCC 25922 | HiMedia | 0335X-1 | Used as a control while performing AST |
| Ethidium Bromide | HiMedia | MB071-1G | Intercalating agent and visualizaion of DNA after electrophoresis under Gel Documentation System |
| Fluorometer | Qubit 2.0 | NA | For determining concentration of extracted metagenomic DNA |
| Gel Documentation System | BioRad | Used for visualizing PCR amplicons after electrophoresis | |
| Gentamicin antibiotic disc | HiMedia | SD170-5x50DS | For performing AST |
| Glacial Acetic Acid | HiMedia | AS119-500ML | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Glycerol | HiMedia | GRM1027-500ML | For making glycerol stocks |
| Imipenem antibiotic disc | HiMedia | SD073 | For performing AST |
| Kaiju Database | NA | NA | For taxonomical classification of reads; https://kaiju.binf.ku.dk/ |
| Kanamycin antibiotic disc | HiMedia | SD017-5x50DS | For performing AST |
| Kanamycin antibiotic powder | HiMedia | MB105-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Levofloxacin antibiotic disc | HiMedia | SD216-5VL | For performing AST |
| Luria Bertani broth | Himedia | M1245-500G | For enrichment of cultures |
| McFarland Standards | Himedia | R092-1No | To compare density of culture suspension |
| Molecular Biology water | HiMedia | TCL018-500ML | For making PCR reaction mixture |
| Mueller-Hinton Agar (MHA) | HiMedia | M173-500G | For performing Antibiotc Susceptibility Testing (AST) |
| Neomycin antibiotic disc | HiMedia | SD731-5x50DS | For performing AST |
| PCR Gradient Thermal Cycler | Eppendorf | Mastercycler Nexus Gradient 230V/50-60 Hz | Used for performing PCR for amplification of 16S rRNA region and various Antibiotic Resistance genes |
| Primers | Xcelris | NA | For PCR amplication |
| R2A Agar, Modified | HiMedia | M1743 | For preparation of media plates for isolation of total and antibiotic resistant (AR) bacterial load |
| Scaffold generation | CLC Genomics Workbench 6.0 | NA | For generation of scaffolds |
| Sequencer | Illumina platform (2 x 150 bp chemistry) | NA | Sequencing of amplified library |
| Sodium Chloride | HiMedia | TC046-500G | For preparation of 0.85% saline for serially diluting the water sample |
| Soil DNA isolation Kit | Xcelgen | NA | For extraction of whole metagenomic DNA from the filtered water sample |
| Staphylococcus aureus subsp. aureus ATCC 29213 | HiMedia | 0365P | Used as a control while performing AST |
| Taxonomical Classification | Kaiju ioinformatics tool | NA | For classification of reads into different taxonomic groups from phylum to genus level |
| The Comprehensive Antibiotic Resistance Database (CARD) | NA | NA | For functional annotation of ARGs |
| Tigecycline antibiotic disc | HiMedia | SD278 | For performing AST |
| Trimethoprim antibiotic disc | HiMedia | SD039-5x50DS | For performing AST |
| Tris base | HiMedia | TC072-500G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Vancomycin antibiotic powder | HiMedia | CMS217 | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Weighing Balance | Mettler Toledo | ME204 Mettler Toledo | Used for weighing media powders, reagent powders etc. |
| NA - Not Applicable |
Références
- Prestinaci, F., Pezzotti, P., Pantosti, A. Antimicrobial resistance: A global multifaceted phenomenon. Pathogens and Global Health. 109 (7), 309-318 (2015).
- Knight, G., et al. Antimicrobial resistance and COVID-19: Intersections and implications. Elife. 10, 64139 (2021).
- Ventola, C. L. The antibiotic resistance crisis: Part 1: Causes and threats. Pharmacy and Therapeutics. 40 (4), 277-283 (2015).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J. R., Rath, A. Metagenomic analysis of total microbial diversity and antibiotic resistance of culturable microorganisms in raw chicken meat and mung sprouts (Phaseolus aureus) sold in retail markets of Mumbai. India. Current Science. 113 (1), 71-79 (2017).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J., Rath, A. Characterization of multiple antibiotic resistance of culturable microorganisms and metagenomic analysis of total microbial diversity of marine fish sold in retail shops in Mumbai, India. Environmental Science and Pollution Research. 25 (7), 6228-6239 (2018).
- Czekalski, N., GascónDíez, E., Bürgmann, H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake. The ISME Journal. 8 (7), 1381-1390 (2014).
- Kraemer, S., Ramachandran, A., Perron, G. Antibiotic pollution in the environment: From microbial ecology to public policy. Microorganisms. 7 (6), 180 (2019).
- Edmonds-Wilson, S., Nurinova, N., Zapka, C., Fierer, N., Wilson, M. Review of human hand microbiome research. Journal of Dermatological Science. 80 (1), 3-12 (2015).
- de Abreu, V., Perdigão, J., Almeida, S. Metagenomic approaches to analyze antimicrobial resistance: An overview. Frontiers in Genetics. 11, 575592 (2021).
- Carlson, S., et al. Detection of multiresistant Salmonella typhimurium DT104 using multiplex and fluorogenic PCR. Molecular and Cellular Probes. 13 (3), 213-222 (1999).
- Breakpoint tables for interpretation of MICs and zone diameters, Version 12.0. European Committee on Antimicrobial Susceptibility Testing Available from: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_12.0_Breakpoint_Tables.pdf (2022)
- Bharti, R., Grimm, D. Current challenges and best-practice protocols for microbiome analysis. Briefings in Bioinformatics. 22 (1), 178-193 (2019).
- Choo, J., Leong, L., Rogers, G. Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports. 5, 16350 (2015).
- Clinical and Laboratory Standards Institute. . Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria that Grow Aerobically, 11th edition. , (2015).
- Bayot, M., Bragg, B. Antimicrobial Susceptibility Testing. StatPearls. , (2021).
- Joseph, A. A., Odimayo, M. S., Olokoba, L. B., Olokoba, A. B., Popoola, G. O. Multiple antibiotic resistance index of Escherichia coli isolates in a tertiary hospital in South-West Nigeria. Medical Journal of Zambia. 44 (4), 225-232 (2017).
- Lorenz, T. Polymerase chain reaction: Basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Rolin, J. Food and human gut as reservoirs of transferable antibiotic resistance encoding genes. Frontiers in Microbiology. 4, 173 (2013).
- Racewicz, P., et al. Prevalence and characterisation of antimicrobial resistance genes and class 1 and 2 integrons in multiresistant Escherichia coli isolated from poultry production. Scientific Reports. 12, 6062 (2022).
- Gebreyes, W., Thakur, S. Multidrug-resistant Salmonella enterica serovar Muenchen from pigs and humans and potential interserovar transfer of antimicrobial resistance. Antimicrobial Agents and Chemotherapy. 49 (2), 503-511 (2005).
- Li, L., et al. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui Province, China. PLoS One. 9 (8), 104356 (2014).
- Akers, K., et al. Aminoglycoside resistance and susceptibility testing errors in Acinetobacter baumannii-calcoaceticus complex. Journal Of Clinical Microbiology. 48 (4), 1132-1138 (2010).
- Ciesielczuk, H. . Extra-intestinal pathogenic Escherichia coli in the UK: The importance in bacteraemia versus urinary tract infection, colonisation of widespread clones and specific virulence factors. , (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon