Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Aislamiento e identificación de bacterias resistentes a antibióticos transmitidas por el agua y caracterización molecular de sus genes de resistencia a antibióticos
En este artículo
Resumen
Aquí, presentamos un protocolo detallado para el aislamiento e identificación de bacterias resistentes a antibióticos del agua y la caracterización molecular de sus genes de resistencia a antibióticos (ARG). El uso de técnicas basadas en cultivos y no basadas en cultivos (análisis metagenómico) proporciona información completa sobre la diversidad bacteriana total y el conjunto total de diferentes ARG presentes en aguas dulces de Mumbai, India.
Resumen
El desarrollo y la propagación de la resistencia a los antibióticos (RA) a través de la microbiota asociada con cuerpos de agua dulce es un importante problema de salud mundial. En el presente estudio, se recolectaron muestras de agua dulce y se analizaron con respecto a la diversidad bacteriana total y los genes AR (ARG) utilizando técnicas convencionales basadas en cultivos y un enfoque metagenómico independiente del cultivo de alto rendimiento. Este artículo presenta un protocolo sistemático para la enumeración de las bacterias cultivables totales y resistentes a antibióticos a partir de muestras de agua dulce y la determinación de la resistencia fenotípica y genotípica en los aislados cultivables. Además, informamos el uso de un análisis metagenómico completo del ADN metagenómico total extraído de la muestra de agua dulce para la identificación de la diversidad bacteriana general, incluidas las bacterias no cultivables, y la identificación del grupo total de diferentes ARG (resistoma) en el cuerpo de agua. Siguiendo estos protocolos detallados, observamos una alta carga de bacterias resistentes a los antibióticos en el rango de 9.6 × 10 5-1.2 × 109 UFC/mL. La mayoría de los aislamientos fueron resistentes a los múltiples antibióticos probados, incluyendo cefotaxima, ampicilina, levofloxacina, cloranfenicol, ceftriaxona, gentamicina, neomicina, trimetoprima y ciprofloxacina, con índices de resistencia a antibióticos múltiples (MAR) de ≥0,2, lo que indica altos niveles de resistencia en los aislados. La secuenciación del ARNr 16S identificó patógenos humanos potenciales, como Klebsiella pneumoniae, y bacterias oportunistas, como Comamonas spp., Micrococcus spp., Arthrobacter spp. y Aeromonas spp. La caracterización molecular de los aislados mostró la presencia de varios ARGs, como blaTEM, blaCTX-M (β-lactámicos), aadA, aac (6')-Ib (aminoglucósidos) y dfr1 (trimetoprimas), lo que también fue confirmado por todo el análisis metagenómico de ADN. También se detectó una alta prevalencia de otros ARG que codifican para bombas de eflujo de antibióticos-mtrA, macB, mdtA, acrD, β-lactamasas-SMB-1, VIM-20, ccrA, ampC, blaZ, el gen de la cloranfenicol acetiltransferasa catB10 y el gen de resistencia a la rifampicina rphB-. Con la ayuda de los protocolos discutidos en este estudio, confirmamos la presencia de bacterias SAM transmitidas por el agua con diversos rasgos fenotípicos y genotípicos de AR. Por lo tanto, el análisis de ADN metagenómico completo se puede utilizar como una técnica complementaria a las técnicas convencionales basadas en cultivos para determinar el estado general de AR de un cuerpo de agua.
Introducción
La resistencia a los antimicrobianos (RAM) se ha identificado como uno de los problemas mundiales más acuciantes. La rápida evolución de la RAM y su propagación mundial son una de las mayores amenazas para la salud humana y la economía mundial en términos de los costos de salud asociados con ella1. El uso excesivo y el mal uso de antibióticos han llevado a un aumento en AR. Esto ha sido destacado por la pandemia de COVID-19, durante la cual el tratamiento de infecciones secundarias asociadas, en muchos casos, se vio enormemente comprometido debido a la RAM en los pacientes afectados2. Además del uso directo / mal uso de antibióticos por parte de los seres humanos, el uso excesivo y el mal uso de antibióticos en la agricultura y la ganadería y su descarga inadecuada en el medio ambiente, incluidas las masas de agua, son una preocupación importante3. El aumento de nuevos rasgos de resistencia y resistencia a múltiples fármacos en bacterias resalta urgentemente la necesidad de una mejor comprensión de los factores que conducen al desarrollo de AR y su diseminación. Múltiples bacterias resistentes a los antibióticos, que a menudo portan múltiples genes AR (ARG) en elementos genéticos móviles como los plásmidos, pueden transferir estos genes de resistencia a microorganismos no resistentes, incluidos los patógenos humanos potenciales, lo que lleva a la aparición de superbacterias que son intratables incluso con antibióticos de último recurso4. Estas múltiples bacterias resistentes a los antibióticos, si están presentes en los ecosistemas acuáticos, pueden ingresar directamente al intestino humano a través del consumo de alimentos contaminados a base de agua como peces, cangrejos y moluscos. Estudios previos han demostrado que la propagación de bacterias AR en sistemas de agua naturales también puede llegar a otros suministros de agua, incluida el agua potable, y, por lo tanto, puede ingresar a la cadena alimentaria humana 5,6,7.
El objetivo del presente estudio es proporcionar un protocolo integral utilizando una combinación de técnicas basadas en cultivos y no basadas en cultivos (análisis metagenómico completo) para obtener información completa sobre la diversidad bacteriana total y el conjunto total de diferentes ARG presentes en un cuerpo de agua en Mumbai, India. Convencionalmente, se han utilizado técnicas basadas en el cultivo para estudiar la diversidad bacteriana en cuerpos de agua. Como los microorganismos cultivables constituyen solo un pequeño porcentaje de la microbiota total en cualquier nicho, para tener una mejor comprensión del estado general de la diversidad bacteriana y los diversos rasgos resistentes que prevalecen en cualquier muestra, se deben utilizar varias técnicas basadas en el cultivo e independientes del cultivo en conjunto. Una de esas técnicas robustas y confiables independientes del cultivo es el análisis de ADN metagenómico completo. Este método de alto rendimiento ha sido utilizado con éxito en diversos estudios sobre diversidad bacteriana o anotaciones funcionales de varios ARG 8,9. Esta técnica utiliza el metagenoma (el material genético total en una muestra) como material de partida para varios análisis y, por lo tanto, es independiente del cultivo. Los protocolos del presente estudio se pueden utilizar para el análisis de ADN metagenómico completo para obtener información sobre la diversidad bacteriana total y varios ARG (resistoma) en muestras de agua.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Recogida y procesamiento de muestras
- Recogida de muestras
- Recoja el volumen apropiado de la muestra de agua en recipientes de muestras estériles, asegurándose de que no se llenen más de 3/4 del recipiente.
- Transportar las muestras al laboratorio en condiciones asépticas tan pronto como sea posible después de la recolección y procesarlas inmediatamente.
- Procesamiento de muestras
- Filtrar asépticamente la muestra de agua a través de un paño de muselina estéril para eliminar cualquier partícula.
- Llevar a cabo diluciones seriadas apropiadas del agua filtrada para su posterior análisis.
2. Estimación de la carga bacteriana total y del recuento de bacterias resistentes a los antibióticos
- Determinación de la carga bacteriana total
- Suspender 18,12 g de agar R2A, polvo modificado en 1.000 ml de agua doble destilada, y disolver la mezcla por calentamiento. Autoclave la mezcla disuelta a 121 °C, 15 psi durante 20 min. Prepare agar R2A, placas modificadas vertiendo la cantidad apropiada de la mezcla esterilizada en autoclave en placas de Petri estériles (por ejemplo, agregue aproximadamente 20 ml de medio estéril esterilizado en autoclave a una placa de Petri estéril de 90 mm).
- Distribuir uniformemente 100 μL de las diluciones apropiadas de la muestra de agua filtrada en el agar R2A, placa modificada una vez que el medio se solidifique. Realice el experimento por duplicado.
- Incubar todas las placas anteriores a 35-37 °C durante 48 h (variar la temperatura y el tiempo de incubación dependiendo del medio utilizado para el aislamiento).
- Expresar la carga bacteriana total en términos de unidades formadoras de colonias por mililitro (UFC/ml) utilizando la ecuación (1):
 (1)
(1)
- Determinación del recuento bacteriano de AR
- Siga los pasos 2.1.1-2.1.4. Sin embargo, en lugar de agar R2A, las placas modificadas, use agar R2A, placas modificadas complementadas individualmente con cinco antibióticos diferentes, a saber, cefotaxima (3 μg / ml), ciprofloxacina (0.5 μg / ml), eritromicina (20 μg / ml), kanamicina (15 μg / ml) y vancomicina (3 μg / ml).
- Añadir los antibióticos por separado en tubos que contengan 20 ml de agar R2A fundido estéril, modificado (con la temperatura del agar R2A fundido, modificado a ≤40 °C) para alcanzar la concentración final de antibiótico como se menciona en el paso 2.2.1.
- Agite para mezclar uniformemente y vierta en placas de Petri estériles antes de que el agar se solidifique. Realice el experimento por duplicado.
- Incubar todas las placas anteriores a 35-37 °C durante 48 h (si se utiliza un medio diferente, la temperatura y el tiempo de incubación pueden variar).
- Para el control de calidad y la comprobación de la eficacia de los antibióticos, esparcir 100 μL de suspensiones bacterianas de las cepas de Agar R2A de Escherichia coli ATCC 25922 y Staphylococcus aureus ATCC 29213 en sus respectivas placas modificadas de Agar R2A que contienen antibióticos (asegúrese de que la densidad del cultivo fresco utilizado para la inoculación sea OD = 0,5 a 600 nm).
- Determinar el recuento bacteriano resistente a los antibióticos en términos de UFC/ml como se describe en el paso 2.1.4.
- Existencias de glicerol de las cepas aisladas
- Seleccionar colonias de AR morfológicamente distintas.
- Suspender una sola colonia aislada en 2 ml de caldo estéril de Luria-Bertani que contenga el antibiótico respectivo (por ejemplo, si se seleccionó una colonia de una placa que contiene cefotaxima, inocular la colonia del paso 2.3.1 en caldo estéril de Luria-Bertani que contenga cefotaxima en su concentración respectiva).
- Incubar los tubos inoculados a 37 °C a 80 rpm hasta que el OD600 alcance 0,5.
- Preparar las existencias de glicerol de los aislados mezclando 750 μL de la suspensión de cultivo del paso 2.3.3 en 250 μL de glicerol estéril al 100% en condiciones asépticas.
- Almacenar las existencias de glicerol a -80 °C hasta un análisis adicional.
NOTA: Para la reactivación de los cultivos de las reservas de glicerol, descongelar las reservas de glicerol a 4 °C. Inocular un bucle de este caldo en 2 ml de caldo estéril Luria-Bertani que contiene el antibiótico respectivo y dejar crecer.
3. Identificación de bacterias cultivables mediante secuenciación del gen 16S rRNA
- Preparación de la plantilla de ADN a partir de los aislados para PCR
NOTA: El protocolo descrito para la preparación de la plantilla de ADN para PCR para el aislamiento de ADN crudo de la bacteria es dado por Carlson et al.10.- Usando un palillo de dientes estéril, tome una colonia única, aislada y pura del aislado que crece en una placa de Petri. Suspender la colonia bacteriana en 100 μL de agua estéril doble destilada en un tubo de microcentrífuga estéril y hervir durante 10 min.
- Centrifugar la suspensión a 10.000 × g durante 2 minutos para granular los residuos y transferir el sobrenadante a un tubo de microcentrífuga estéril nuevo para su uso como plantilla de ADN crudo.
- Amplificación por PCR dirigida de la región V3 del gen 16S rRNA y secuenciación
- Preparar 40 μL de la mezcla de reacción en un tubo de PCR para la amplificación por PCR, como se menciona en la Tabla 1.
NOTA: La preparación de ADN debe llevarse a cabo en un bloque de hielo minimizando la posibilidad de contaminación (use guantes mientras manipula los reactivos y limpie la superficie de trabajo a fondo con etanol al 70%). - Coloque el tubo en el bloque térmico y ejecute el programa apropiado en el termociclador de PCR. Consulte la Tabla 2 para las condiciones estandarizadas de ciclo de PCR y la información del manual para la amplificación de las regiones V3 de los genes 16S rRNA.
- Para resolver los amplicones y la visualización, realice electroforesis en gel de agarosa (AGE). Mezclar 10 μL del producto de PCR amplificado y 2 μL de tampón de carga de gel 6x (Tabla 3), y cargar esta mezcla en pocillos en gel de agarosa al 1,5% (disolver 1,5 g de polvo de agarosa en 100 ml de 1x tampón TAE [Tabla 3]) que contiene 5 μL de bromuro de etidio (EtBr) a una concentración final de 0,5 μg/ml de EtBr en 100 ml del gel de agarosa.
PRECAUCIÓN: EtBr es un potente carcinógeno. Se deben usar guantes en todo momento mientras se manipula EtBr y geles que contengan EtBr. - Agregue una escalera de ADN para la estimación del tamaño de los amplicones.
- Realice la electroforesis del gel en un tampón de tanque TAE a 80-100 V.
- Una vez que el tinte de seguimiento ejecute 3/4 del gel, detenga la electroforesis y visualice las bandas de amplicón bajo un transiluminador UV.
- Utilice el producto de PCR (amplicón) para la secuenciación del gen 16S rRNA para identificar el aislado.
- Cuantificar el amplicón sometiéndolo a análisis espectrofotométrico usando la ecuación (2).
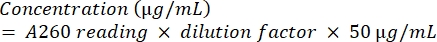 (2)
(2) - Para verificar la pureza del ADN, calcule la proporción de A260 / A280.
NOTA: Idealmente, este número debe estar por encima de 1.5 y, preferiblemente, entre 1.8 y 2.0. - Para identificar los aislamientos, compare las secuencias que se obtienen con las bases de datos de secuencias utilizando una herramienta de búsqueda de alineación adecuada.
- Preparar 40 μL de la mezcla de reacción en un tubo de PCR para la amplificación por PCR, como se menciona en la Tabla 1.
4. Detección de resistencia a antibióticos en los aislados mediante pruebas de sensibilidad a antibióticos
NOTA: Este protocolo describe el método para la prueba de susceptibilidad a los antibióticos (AST) por difusión de disco. Se utilizaron los siguientes discos antibióticos: cefotaxima (5 μg), ampicilina (10 μg), levofloxacino (5 μg), cloranfenicol (30 μg), tigeciclina (15 μg), ceftriaxona (30 μg), imipenem (10 μg), gentamicina (10 μg), neomicina (10 μg), trimetoprima (5 μg) y ciprofloxacino (5 μg).
- Preparación del inóculo para el AST
- Inocular asépticamente una colonia de RA única, aislada y purificada utilizando un asa estéril en 2 ml de un medio estéril no selectivo, como el caldo Luria-Bertani (sin ningún antibiótico), e incubar a 37 °C a 80 rpm durante la noche.
- Resuspender tomando 100-150 μL del cultivo cultivado durante la noche (aproximadamente, OD 600 = 1.8-2.0) en 2 ml de medio de caldo fresco no selectivo Luria-Bertani e incubar durante 2-4 h (hasta que el OD600 alcance 0.4-0.5).
- Diluir esta suspensión de cultivo recién cultivada utilizando solución salina estéril al 0,85% de tal manera que la densidad del cultivo sea igual a 0,5 McFarland estándar (aproximadamente, OD600 = 0,1), que corresponde aproximadamente a 1-2 × 108 células/ml.
- Mezcle suavemente la suspensión bacteriana para una distribución celular uniforme.
- Use la suspensión anterior dentro de los 15 minutos posteriores a la dilución.
- Inoculación de las placas de agar
- Prepare placas de agar Mueller-Hinton (MHA) para realizar el AST mezclando 38 g de MHA en 1,000 ml de agua doble destilada y disuelva la mezcla calentando. Autoclave la mezcla disuelta a 121 °C, 15 psi durante 15 min.
- Asegúrese de que la profundidad de MHA en las placas sea de 4 mm (25 ml de medio por placa).
- Simultáneamente, retire los discos antibióticos del congelador y caliéntelos a temperatura ambiente.
NOTA: Los discos antibióticos deben descongelarse gradualmente descongelándolos inicialmente a 4 °C y posteriormente a temperatura ambiente para reducir cualquier peligro potencial de condensación en los discos, que posteriormente puede afectar a la zona de inhibición (ZOI). - En condiciones asépticas, sumergir un hisopo de algodón estéril en el inóculo preparado en el paso 4.1 y eliminar el exceso de suspensión para evitar la sobreinoculación de las placas.
- Extienda el cultivo uniformemente en las placas, comenzando desde la parte superior de la placa MHA y yendo y viniendo de borde a borde. Gire la placa 60° mientras frota.
- Aplicación de los discos antibióticos
- Con la ayuda de fórceps esterilizados por llama, transfiera asépticamente los discos antibióticos a las placas MHA inoculadas y presione los discos suavemente para asegurar un contacto nivelado completo con el agar.
NOTA: Este procedimiento debe realizarse dentro de los 15 minutos posteriores a la inoculación del cultivo en las placas. - Coloque el número adecuado de discos antibióticos en la placa de agar teniendo en cuenta el organismo, el antibiótico utilizado y el tamaño de la placa para evitar la superposición de las zonas de inhibición.
NOTA: Se pueden acomodar de cuatro a cinco discos en una placa circular de 90 mm.
- Con la ayuda de fórceps esterilizados por llama, transfiera asépticamente los discos antibióticos a las placas MHA inoculadas y presione los discos suavemente para asegurar un contacto nivelado completo con el agar.
- Incubación de las placas
- Dentro de los 15 minutos posteriores a la aplicación de los discos antibióticos, invertir las placas e incubar a 37 °C durante la noche.
- Interpretación de los resultados
- Mida el diámetro ZOI en milímetros (mm) e infórmelo de acuerdo con los valores de punto de ruptura dados por EUCAST11. Véanse los dos ejemplos que figuran a continuación.
- El punto de ruptura del diámetro de la zona (mm) para un disco antibiótico ciprofloxacino (5 μg) para Enterobacterales es S ≥ 25 y R < 22, lo que significa que se considera sensible (S) si ZOI ≥ 25 mm, mientras que es resistente (R) si ZOI < 22 mm. Si el diámetro de ZOI cae entre 22 y 25, el aislado se considera intermedio (I).
- El punto de ruptura del diámetro de la zona (mm) para un disco antibiótico cloranfenicol (30 μg) para Staphylococcus spp. es S ≥ 18 y R < 18, lo que significa que se considera sensible si ZOI ≥ 18 mm, mientras que es resistente si ZOI < 18 mm.
- Determine el índice de resistencia a antibióticos múltiples (SAM) encontrando la relación entre el número de antibióticos a los que el aislado es resistente y el número total de antibióticos a los que está expuesto el aislado.
NOTA: Para el control de calidad, E. coli ATCC 25922 y S. áureo ATCC 29213 se utilizan como cepas de referencia siguiendo el protocolo como en el paso 4.
- Mida el diámetro ZOI en milímetros (mm) e infórmelo de acuerdo con los valores de punto de ruptura dados por EUCAST11. Véanse los dos ejemplos que figuran a continuación.
5. Detección basada en PCR de genes de resistencia a antibióticos en los aislados
- Utilice un protocolo de PCR estándar para la identificación de ARG en los aislados. Prepare la plantilla de ADN utilizando el protocolo indicado en el paso 3.1.
NOTA: Las condiciones de ciclo de PCR utilizadas en este estudio fueron 94 °C durante 10 min, seguidas de 35 ciclos de 94 °C durante 30 s, recocido durante 30 s a la temperatura apropiada (estandarizado para cada juego de cebadores), extensión a 72 °C durante 40 s y una extensión final a 72 °C durante 5 min. La mezcla de reacción se describe en la Tabla 4. La lista de ARG, cebadores y temperaturas de recocido se da en la Tabla 5. - Para resolver, visualizar y comprobar la pureza de los amplicones, siga los pasos 3.2.3-3.2.10.
6. Análisis de ADN metagenómico completo para la identificación de la diversidad bacteriana total y la detección de ARG en el metagenoma
- Extracción del ADN total (metagenoma) de la muestra de agua
- Extraer el ADN metagenómico de las muestras de agua filtrada.
NOTA: En el presente estudio, el ADN metagenómico (ADN total) se extrajo de las muestras de agua filtrada utilizando el kit de aislamiento de ADN referenciado siguiendo el protocolo del fabricante (ver la Tabla de materiales). - Verifique la calidad del ADN cargando 3 μL del ADN metagenómico extraído en un gel de agarosa al 0,8% y ejecute el gel a 80-110 V durante aproximadamente 30 minutos.
- Compruebe la presencia de una sola banda intacta.
- Verifique la concentración de ADN con un fluorómetro.
- Extraer el ADN metagenómico de las muestras de agua filtrada.
- Determinación de la diversidad bacteriana y detección de ARG mediante secuenciación de ADN metagenómico completo
- Preparación de bibliotecas y amplificación por PCR:
- Prepare una biblioteca de secuenciación de extremo emparejado utilizando el kit de preparación de la biblioteca de ADN al que se hace referencia (consulte la Tabla de materiales).
- Prepare el ADN para la ligadura del adaptador tomando 200 ng de ADN y cortándolo mecánicamente en fragmentos más pequeños, seguido de un paso continuo de reparación final en el que se agrega una "A" a los extremos 3'.
- Dependiendo de la plataforma utilizada para la secuenciación, ligar adaptadores específicos a ambos extremos de los fragmentos de ADN.
NOTA: Las secuencias cruciales para enlazar bibliotecas con doble código de barras a una celda de flujo para la secuenciación están presentes en estos adaptadores. Esto permite la amplificación por PCR de los fragmentos ligados por adaptador y la unión de los cebadores de secuenciación estándar. - Para verificar la calidad y la cantidad, analice la biblioteca amplificada utilizando un chip de ADN de alta sensibilidad según las instrucciones del fabricante.
- Generación y secuenciación de clústeres:
- Cargue la biblioteca amplificada en la plataforma de secuenciación adecuada para la generación de clústeres y la secuenciación posterior.
NOTA: Las moléculas de biblioteca se unen a los oligos adaptadores complementarios en la celda de flujo de extremo pareado. Durante la secuenciación, las hebras hacia adelante se escinden selectivamente después de la resíntesis de la cadena inversa. Esta hebra inversa copiada se secuencia desde el extremo opuesto del fragmento.
- Cargue la biblioteca amplificada en la plataforma de secuenciación adecuada para la generación de clústeres y la secuenciación posterior.
- Análisis bioinformático:
- Genere andamios a partir de los datos de alta calidad utilizando la plataforma adecuada.
- Someter estos andamios a análisis bioinformáticos para la clasificación taxonómica e identificación de los ARGs.
NOTA: El flujo de trabajo para todo el análisis de ADN metagenómico para la identificación de la diversidad bacteriana total y la detección de ARG en el metagenoma se da en la Figura 1. Un diagrama de flujo de la metodología completa descrita en el manuscrito se da en la Figura 2.
- Preparación de bibliotecas y amplificación por PCR:
Access restricted. Please log in or start a trial to view this content.
Resultados
Carga bacteriana total y recuento de bacterias resistentes a los antibióticos (AR)
La enumeración de la carga bacteriana total se llevó a cabo mediante la difusión de diluciones de 10−4 a 10−6 veces de las muestras de agua en agar R2A, medio modificado. Para la enumeración del recuento de bacterias AR, se distribuyeron diluciones de 10-3 a 10-6 veces en placas de medios que contenían antibióticos (Figura 3). Los recue...
Access restricted. Please log in or start a trial to view this content.
Discusión
La recolección y el procesamiento de la muestra juegan un papel importante y pueden afectar los resultados y la interpretación del estudio. Por lo tanto, para descartar la variabilidad en las muestras, es importante llevar a cabo el muestreo en múltiples ubicaciones de la masa de agua dulce que se está estudiando. Mantener condiciones ambientales asépticas adecuadas al manipular dichas muestras puede prevenir la contaminación. Además, para evitar cambios en la composición bacteriana que puedan influir en la calid...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen intereses contradictorios que revelar.
Agradecimientos
Este trabajo fue parcialmente apoyado por subvenciones financieras del Departamento de Ciencia y Tecnología-Promoción de la Investigación Universitaria y el Esquema de Excelencia Científica (DST-PURSE) de la Universidad de Mumbai. Devika Ghadigaonkar trabajó como becaria de proyecto bajo el esquema. Se reconoce la ayuda técnica proporcionada por Harshali Shinde, investigador principal del Proyecto n.º de investigación del Departamento de Ciencia y Tecnología-Junta de Investigación de Ciencia e Ingeniería (DST-SERB): CRG / 2018 / 003624.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 100 bp DNA ladder | Himedia | MBT049-50LN | For estimation of size of the amplicons |
| 2x PCR Taq mastermix | HiMedia | MBT061-50R | For making PCR reaction mixture |
| 37 °C Incubator | GS-192, Gayatri Scientific | NA | For incubation of bacteria |
| 6x Gel Loading Buffer | HiMedia | ML015-1ML | Loading and Tracking dye which helps to weigh down the DNA sample and track the progress of electrophoresis |
| Agarose powder | Himedia | MB229-50G | For resolving amplicons during Agarose Gel Electrophoresis (AGE) |
| Ampicillin antibiotic disc | HiMedia | SD002 | For performing AST |
| Autoclave | Equitron | NA | Required for sterilization of media, glass plates, test tubes, etc |
| Bioanalyzer 2100 | Agilent Technologies | NA | To check the quality and quantity of the amplified library |
| Bisafety B2 Cabinet | IMSET | IMSET BSC-Class II Type B2 | Used for microbiological work like bacterial culturing, AST etc. |
| Cefotaxime antibiotic disc | HiMedia | SD295E-5VL | For performing AST |
| Cefotaxime antibiotic powder | HiMedia | TC352-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Ceftriaxone antibiotic disc | HiMedia | SD065 | For performing AST |
| Centrifuge Minispin | Eppendorf | Minispin Plus-5453 | Used to pellet the debris during crude DNA preparation |
| Chloramphenicol antibiotic disc | HiMedia | SD006-5x50DS | For performing AST |
| Ciprofloxacin antibiotic disc | HiMedia | SD060-5x50DS | For performing AST |
| Ciprofloxacin antibiotic powder | HiMedia | TC447-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Colorimeter | Quest | NA | For checking the OD of culture suspensions |
| Comprehensive Antibiotic Resistance Database (CARD) database | functional annotation of ARGs; https://card.mcmaster.ca/ | ||
| Cooling Shaker Incubator | BTL41 Allied Scientific | NA | For incubation of media plates for culturing bacteria |
| Deep Freezer (-40 °C) | Haier | DW40L, Haier Biomedicals | For storage of glycerol stocks |
| DNA Library Prep Kit | NEB Next Ultra DNA Library Prep Kit for Illumina | NA | Paired-end sequencing library preparation |
| EDTA | HiMedia | GRM1195-100G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Electrophoresis Apparatus | TechResource | 15 cm gel casting tray | For making the agarose gel and carrying out electrophoresis |
| Electrophoresis Power pack with electrodes | Genei | NA | For running the AGE |
| Erythromycin antibiotic disc | HiMedia | SD222-5VL | For performing AST |
| Erythromycin antibiotic powder | HiMedia | CMS528-1G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Erythromycin antibiotic powder | HiMedia | TC024-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Escherichia coli ATCC 25922 | HiMedia | 0335X-1 | Used as a control while performing AST |
| Ethidium Bromide | HiMedia | MB071-1G | Intercalating agent and visualizaion of DNA after electrophoresis under Gel Documentation System |
| Fluorometer | Qubit 2.0 | NA | For determining concentration of extracted metagenomic DNA |
| Gel Documentation System | BioRad | Used for visualizing PCR amplicons after electrophoresis | |
| Gentamicin antibiotic disc | HiMedia | SD170-5x50DS | For performing AST |
| Glacial Acetic Acid | HiMedia | AS119-500ML | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Glycerol | HiMedia | GRM1027-500ML | For making glycerol stocks |
| Imipenem antibiotic disc | HiMedia | SD073 | For performing AST |
| Kaiju Database | NA | NA | For taxonomical classification of reads; https://kaiju.binf.ku.dk/ |
| Kanamycin antibiotic disc | HiMedia | SD017-5x50DS | For performing AST |
| Kanamycin antibiotic powder | HiMedia | MB105-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Levofloxacin antibiotic disc | HiMedia | SD216-5VL | For performing AST |
| Luria Bertani broth | Himedia | M1245-500G | For enrichment of cultures |
| McFarland Standards | Himedia | R092-1No | To compare density of culture suspension |
| Molecular Biology water | HiMedia | TCL018-500ML | For making PCR reaction mixture |
| Mueller-Hinton Agar (MHA) | HiMedia | M173-500G | For performing Antibiotc Susceptibility Testing (AST) |
| Neomycin antibiotic disc | HiMedia | SD731-5x50DS | For performing AST |
| PCR Gradient Thermal Cycler | Eppendorf | Mastercycler Nexus Gradient 230V/50-60 Hz | Used for performing PCR for amplification of 16S rRNA region and various Antibiotic Resistance genes |
| Primers | Xcelris | NA | For PCR amplication |
| R2A Agar, Modified | HiMedia | M1743 | For preparation of media plates for isolation of total and antibiotic resistant (AR) bacterial load |
| Scaffold generation | CLC Genomics Workbench 6.0 | NA | For generation of scaffolds |
| Sequencer | Illumina platform (2 x 150 bp chemistry) | NA | Sequencing of amplified library |
| Sodium Chloride | HiMedia | TC046-500G | For preparation of 0.85% saline for serially diluting the water sample |
| Soil DNA isolation Kit | Xcelgen | NA | For extraction of whole metagenomic DNA from the filtered water sample |
| Staphylococcus aureus subsp. aureus ATCC 29213 | HiMedia | 0365P | Used as a control while performing AST |
| Taxonomical Classification | Kaiju ioinformatics tool | NA | For classification of reads into different taxonomic groups from phylum to genus level |
| The Comprehensive Antibiotic Resistance Database (CARD) | NA | NA | For functional annotation of ARGs |
| Tigecycline antibiotic disc | HiMedia | SD278 | For performing AST |
| Trimethoprim antibiotic disc | HiMedia | SD039-5x50DS | For performing AST |
| Tris base | HiMedia | TC072-500G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Vancomycin antibiotic powder | HiMedia | CMS217 | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Weighing Balance | Mettler Toledo | ME204 Mettler Toledo | Used for weighing media powders, reagent powders etc. |
| NA - Not Applicable |
Referencias
- Prestinaci, F., Pezzotti, P., Pantosti, A. Antimicrobial resistance: A global multifaceted phenomenon. Pathogens and Global Health. 109 (7), 309-318 (2015).
- Knight, G., et al. Antimicrobial resistance and COVID-19: Intersections and implications. Elife. 10, 64139(2021).
- Ventola, C. L. The antibiotic resistance crisis: Part 1: Causes and threats. Pharmacy and Therapeutics. 40 (4), 277-283 (2015).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J. R., Rath, A. Metagenomic analysis of total microbial diversity and antibiotic resistance of culturable microorganisms in raw chicken meat and mung sprouts (Phaseolus aureus) sold in retail markets of Mumbai. India. Current Science. 113 (1), 71-79 (2017).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J., Rath, A. Characterization of multiple antibiotic resistance of culturable microorganisms and metagenomic analysis of total microbial diversity of marine fish sold in retail shops in Mumbai, India. Environmental Science and Pollution Research. 25 (7), 6228-6239 (2018).
- Czekalski, N., GascónDíez, E., Bürgmann, H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake. The ISME Journal. 8 (7), 1381-1390 (2014).
- Kraemer, S., Ramachandran, A., Perron, G. Antibiotic pollution in the environment: From microbial ecology to public policy. Microorganisms. 7 (6), 180(2019).
- Edmonds-Wilson, S., Nurinova, N., Zapka, C., Fierer, N., Wilson, M. Review of human hand microbiome research. Journal of Dermatological Science. 80 (1), 3-12 (2015).
- de Abreu, V., Perdigão, J., Almeida, S. Metagenomic approaches to analyze antimicrobial resistance: An overview. Frontiers in Genetics. 11, 575592(2021).
- Carlson, S., et al. Detection of multiresistant Salmonella typhimurium DT104 using multiplex and fluorogenic PCR. Molecular and Cellular Probes. 13 (3), 213-222 (1999).
- Breakpoint tables for interpretation of MICs and zone diameters, Version 12.0. European Committee on Antimicrobial Susceptibility Testing. , Available from: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_12.0_Breakpoint_Tables.pdf (2022).
- Bharti, R., Grimm, D. Current challenges and best-practice protocols for microbiome analysis. Briefings in Bioinformatics. 22 (1), 178-193 (2019).
- Choo, J., Leong, L., Rogers, G. Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports. 5, 16350(2015).
- Clinical and Laboratory Standards Institute. Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria that Grow Aerobically, 11th edition. , Clinical and Laboratory Standards Institute. Wayne, PA. (2015).
- Bayot, M., Bragg, B. Antimicrobial Susceptibility Testing. StatPearls. , StatPearls Publishing. Treasure Island, FL. (2021).
- Joseph, A. A., Odimayo, M. S., Olokoba, L. B., Olokoba, A. B., Popoola, G. O. Multiple antibiotic resistance index of Escherichia coli isolates in a tertiary hospital in South-West Nigeria. Medical Journal of Zambia. 44 (4), 225-232 (2017).
- Lorenz, T. Polymerase chain reaction: Basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998(2012).
- Rolin, J. Food and human gut as reservoirs of transferable antibiotic resistance encoding genes. Frontiers in Microbiology. 4, 173(2013).
- Racewicz, P., et al. Prevalence and characterisation of antimicrobial resistance genes and class 1 and 2 integrons in multiresistant Escherichia coli isolated from poultry production. Scientific Reports. 12, 6062(2022).
- Gebreyes, W., Thakur, S. Multidrug-resistant Salmonella enterica serovar Muenchen from pigs and humans and potential interserovar transfer of antimicrobial resistance. Antimicrobial Agents and Chemotherapy. 49 (2), 503-511 (2005).
- Li, L., et al. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui Province, China. PLoS One. 9 (8), 104356(2014).
- Akers, K., et al. Aminoglycoside resistance and susceptibility testing errors in Acinetobacter baumannii-calcoaceticus complex. Journal Of Clinical Microbiology. 48 (4), 1132-1138 (2010).
- Ciesielczuk, H. Extra-intestinal pathogenic Escherichia coli in the UK: The importance in bacteraemia versus urinary tract infection, colonisation of widespread clones and specific virulence factors. , Queen Mary University of London. PhD thesis (2015).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados