A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ملاقط مغناطيسية عالية السرعة للقياسات الميكانيكية النانوية على العناصر الحساسة للقوة
* These authors contributed equally
In This Article
Summary
هنا ، نصف إعداد ملقط مغناطيسي عالي السرعة يقوم بإجراء قياسات ميكانيكية نانوية على جزيئات حيوية حساسة للقوة بمعدل أقصى يبلغ 1.2 كيلو هرتز. نقدم تطبيقه على دبابيس شعر الحمض النووي ومجمعات SNARE كأنظمة نموذجية ، ولكنه سيكون قابلا للتطبيق أيضا على الجزيئات الأخرى المشاركة في الأحداث الميكانيكية البيولوجية.
Abstract
كانت الملقط المغناطيسي أحادي الجزيء (MTs) بمثابة أدوات قوية لاستجواب الجزيئات الحيوية بقوة ، مثل الأحماض النووية والبروتينات ، وبالتالي فهي مستعدة لتكون مفيدة في مجال البيولوجيا الميكانيكية. نظرا لأن الطريقة تعتمد عادة على التتبع القائم على الصور للخرز المغناطيسي ، فإن الحد الأقصى للسرعة في تسجيل الصور وتحليلها ، وكذلك التقلبات الحرارية للخرز ، قد أعاق تطبيقه منذ فترة طويلة في مراقبة التغيرات الهيكلية الصغيرة والسريعة في الجزيئات المستهدفة. توضح هذه المقالة طرقا مفصلة لبناء وتشغيل إعداد MT عالي الدقة يمكنه حل ديناميكيات النانو بالمللي ثانية للجزيئات الحيوية ومجمعاتها. كأمثلة تطبيقية ، يتم عرض التجارب مع دبابيس شعر الحمض النووي ومجمعات SNARE (آلية الاندماج الغشائي) ، مع التركيز على كيفية اكتشاف حالاتها العابرة وانتقالاتها في وجود قوى على نطاق piconewton. نتوقع أن تستمر MTs عالية السرعة في تمكين القياسات الميكانيكية النانوية عالية الدقة على الجزيئات التي تستشعر القوى وتنقلها وتوليدها في الخلايا ، وبالتالي تعميق فهمنا على المستوى الجزيئي لعلم الأحياء الميكانيكي.
Introduction
تستشعر الخلايا بنشاط وتستجيب للمنبهات الميكانيكية. عند القيام بذلك ، تعرض العديد من الجزيئات الحيوية خصائص تعتمد على القوة تمكن من إجراء تغييرات هيكلية ديناميكية. تشمل الأمثلة التي تحظى بتقدير جيد القنوات الأيونية الحساسة للميكانيكا والعناصر الهيكلية الخلوية التي تزود الخلايا بالمعلومات الميكانيكية الرئيسية من البيئة المحيطة بها.
بالإضافة إلى ذلك ، يمكن أيضا اعتبار الجزيئات التي تظهر طبيعة فريدة تحمل القوة حساسة ميكانيكيا بمعنى أوسع. على سبيل المثال ، يلعب التكوين المحلي وذوبان دوبلكس الحمض النووي ، بالإضافة إلى الهياكل ذات الترتيب الأعلى مثل G-quadruplexes ، أدوارا حاسمة في النسخ المتماثل والنسخ وإعادة التركيب ، ومؤخرا تحرير الجينوم. علاوة على ذلك ، تؤدي بعض البروتينات العصبية المشاركة في الاتصالات المشبكية وظائفها عن طريق توليد قوى فيزيائية تتجاوز مستويات التفاعلات النموذجية بين الجزيئات. بغض النظر عن المثال الذي يدرسه المرء ، فإن التحقيق في الميكانيكا النانوية للجزيئات الحيوية المعنية بدقة مكانية زمانية عالية سيثبت أنه مفيد للغاية في الكشف عن الآليات الجزيئية للعمليات الميكانيكية البيولوجية المرتبطة1،2،3.
عملت طرق التحليل الطيفي للقوة أحادية الجزيء كأدوات قوية لفحص الخواص الميكانيكية للجزيئات الحيوية2،4،5،6. يمكنهم مراقبة التغيرات الهيكلية في الأحماض النووية والبروتينات بالتزامن مع تطبيق القوة ، وبالتالي فحص الخصائص المعتمدة على القوة. هناك إعدادان معروفان هما الملقط البصري والملاقط المغناطيسية (MTs) ، والتي تستخدم حبات بحجم ميكرون لمعالجة الجزيئات5،6،7،8. في هذه المنصات ، يتم ربط البوليسترين (للملاقط البصرية) أو الخرز المغناطيسي (ل MTs) للجزيئات المستهدفة (مثل الأحماض النووية والبروتينات) عبر "مقابض" جزيئية ، مصنوعة عادة من شظايا قصيرة من الحمض النووي المزدوج (dsDNA). ثم يتم نقل الخرزات لممارسة القوة وتصويرها لتتبع مواقعها التي تبلغ عن التغيرات الهيكلية في الجزيئات المستهدفة. الملقط البصري والمغنطيسي قابل للتبديل إلى حد كبير في تطبيقاته ، ولكن توجد اختلافات مهمة في مناهجها للتحكم في القوة. الملقط البصري هي في جوهرها أدوات تثبيت موضعية تحبس الخرز في موضعها ، والتي بسببها تتقلب القوة المطبقة عندما يتغير شكل بناء الهدف ؛ زيادة التمديد ، مثل من تتكشف ، يخفف الحبل ويقلل من التوتر ، والعكس صحيح. على الرغم من أنه يمكن تنفيذ التغذية المرتدة النشطة للتحكم في القوة في الملقط البصري ، إلا أن MTs على النقيض من ذلك تعمل بشكل طبيعي كجهاز مشبك للقوة ، مستفيدة من القوى المغناطيسية المستقرة بعيدة المدى بواسطة مغناطيس دائم ، والذي يمكنه أيضا تحمل الاضطرابات البيئية.
على الرغم من تاريخها الطويل وتصميمها البسيط ، فقد تخلفت MTs عن الملقط البصري في تطبيقاتها للقياسات عالية الدقة ، ويرجع ذلك إلى حد كبير إلى التحديات التقنية في تتبع الخرز السريع. ومع ذلك ، في الآونة الأخيرة ، قادت عدة مجموعات بشكل مشترك تحسينا متعدد الأوجه لكل من الأجهزة والبرامج لأدوات MT2،9،10،11،12،13،14،15،16،17،18،19. في هذا العمل ، نقدم مثالا على مثل هذا الإعداد الذي يعمل عند 1.2 كيلو هرتز ونصف كيفية استخدامه لإجراء قياسات ميكانيكية نانوية على الجزيئات الحيوية الحساسة للقوة. كأنظمة نموذجية ، نستخدم دبابيس شعر الحمض النووي ومجمعات SNARE العصبية ونفحص تغييراتها الهيكلية السريعة في نظام piconewton. تظهر دبابيس شعر الحمض النووي انتقالات بسيطة من حالتين في نطاق قوة محدد جيدا20,21 ، وبالتالي فهي بمثابة نماذج ألعاب للتحقق من أداء إعداد الملقط. عندما تتجمع بروتينات SNARE في مركب حساس للقوة يدفع اندماج الغشاء 22 ، فقد تمت دراستها أيضا على نطاق واسع بواسطة التحليل الطيفي للقوة أحادية الجزيء 14،23،24،25. يتم تقديم الأساليب القياسية لتحليل البيانات واستخراج معلومات مفيدة عن الديناميكا الحرارية والحركية. نأمل أن تسهل هذه المقالة اعتماد MTs عالية الدقة في الدراسات الميكانيكية البيولوجية وتحفيز القراء على استكشاف أنظمة اهتمامهم الحساسة للقوة.
Protocol
يتم سرد جميع المواد والمعدات الموضحة في هذا البروتوكول في جدول المواد. يتم إيداع برنامج LabVIEW لتشغيل إعداد MT عالي السرعة الموضح أدناه ، بالإضافة إلى البرامج النصية MATLAB لتحليل بيانات العينة ، على GitHub (https://github.com/ShonLab/Magnetic-Tweezers) ومتاح للجمهور.
1. بناء الجهاز
ملاحظة: يشبه المبدأ العام لبناء MT عالي السرعة أنظمة MT التقليدية القياسية ، باستثناء استخدام كاميرا أشباه موصلات أكسيد المعادن التكميلية عالية السرعة (CMOS) ومصدر ضوء متماسك عالي الطاقة (الشكل 1). ارجع إلى مصادر أخرى لمزيد من الأوصاف لأدوات MT القياسية5،26،27.
- قم بإعداد مجهر مقلوب على طاولة بصرية مضادة للاهتزاز. قم بتثبيت كاميرا CMOS عالية السرعة وملتقط الإطار.
- بناء مرحلة ترجمة لمعالجة المغناطيس في 3D. قم بتركيب مرحلة خطية آلية (مسافة انتقال > 20 مم) عموديا أعلى مرحلة XY اليدوية.
ملاحظة: تتحكم الحركة الرأسية في القوة ، في حين أن المرحلة XY مخصصة للمحاذاة اليدوية للمغناطيس إلى المحور الأصلي للبناء الأولي للإعداد. - قم بتركيب محرك متدرج دوار ونظام حزام وبكرة للمغناطيس الدوار.
ملاحظة: ينقل الحزام الحركة الدورانية بين عمود المحرك والمغناطيس الذي يفصل بينهما بضعة سنتيمترات. دوران المغناطيس داخلي للتلاعب الانتقالي. - جبل المغناطيس. استخدم حامل أكريليك (مطلوب من شركة تصنيع ؛ انظر الشكل التكميلي S1) يمكنه إيواء مغناطيسين متطابقين بإحكام بالتوازي ، مع وجود فجوة محددة جيدا تبلغ 1 مم بين المغناطيس (الشكل 1 ب). للاستفادة من أقصى قوة يمكن الحصول عليها مع زوج معين من المغناطيسات ، اضبط الموضع الرأسي لمرحلة الترجمة بحيث يحاذي السطح السفلي للمغناطيس مستوى العينة عند نقله إلى أدنى موضع.
ملاحظة: ارجع إلى Lipfert et al. لمزيد من المعلومات حول تصميم الحامل وتكوين المغناطيس28. يتم التحكم في ارتفاع واتجاه المغناطيس بواسطة برنامج LabVIEW بالتزامن مع الحصول على البيانات. - المشاهدة باستخدام عدسة موضوعية منخفضة التكبير ، قم بمحاذاة المغناطيس إلى مركز مجال الرؤية. تأكد من أن تدوير المغناطيس لا يسبب إزاحة كبيرة لمركز زوج المغناطيس.
ملاحظة: إذا كانت نقطة المنتصف بين المغناطيس تدور حول محور الدوران ، فمن المحتمل أن يكون المغناطيس بعيدا عن المركز بسبب حامل غير كامل. يمكن قبول مستوى صغير من عدم المحاذاة بالنسبة لحجم الفجوة ، حيث أن دوران المغناطيس مخصص فقط لفحص الحبال وتطبيق عزم الدوران في تطبيقات محددة. - قم بتركيب صمام ثنائي فائق الإضاءة (SLD) لإضاءة الخرز. مرر الحزمة عبر فجوة 1 مم بين المغناطيسين. تأكد من أن الحزمة موازية بشكل صحيح لتناسب الفجوة وأن الإضاءة ليست مظللة بالمغناطيس.
- قم بتثبيت ماسح ضوئي لعدسة بيزو على قطعة الأنف وقم بتركيب عدسة موضوعية للغمر بالزيت 100x (الفتحة العددية [NA]: 1.45) لتتبع الخرز. لتجنب القطع الأثرية المحتملة في تتبع النتائج ، تأكد من الحفاظ على الإضاءة بشكل موحد عند تحريك المغناطيس. أخيرا ، اضبط مستوى الإضاءة على أقصى سطوع دون تشبع وحدات البكسل.
ملاحظة: لمقارنة مصادر الضوء المختلفة لتتبع الخرز عالي السرعة ، راجع Dulin et al.29.
2. معايرة القوة المغناطيسية
- باستخدام تفاعل البلمرة المتسلسل (PCR ؛ انظر الجدول 1) ، قم بإعداد شظايا dsDNA 5 kbp (باستخدام Primer B و Primer Z_5k و λ-DNA) التي تم تمييزها بالبيوتين في أحد طرفيها (لربط السطح) و azide على الطرف الآخر (لتعلق الخرزة).
- بعد القسم 6 ، قم بإعداد خلية تدفق بجزيئات 5 kbp.
- بعد القسم 7 ، حدد بنية حبال حبة جيدة من خلال التحقق من امتدادها ودورانها. على وجه الخصوص ، تأكد من اختيار حبة ذات مسار دوران ضئيل (أي بنصف قطر <200 نانومتر) لتقليل إزاحة ارتفاع الخرزة بسبب المرفق خارج المركز30,31. بمجرد تحديد الحبل الجيد ، ابدأ في تتبع الخرز ، بالإشارة إلى القسم 9.
- إذا كان الإعداد جديدا ، فقم بتمييز الضوضاء والثبات للحصول على قياسات موثوقة عالية الدقة. ضع المغناطيس ~ 3 مم من سطح خلية التدفق (لتطبيق >10 pN وقمع الحركة البراونية للخرزة) ، وتتبع موضع z للحبة عند 1.2 كيلو هرتز ، واحسب انحراف ألان (AD) من السلسلة الزمنية z-إحداثيات 32,33 (الشكل 2C). تحقق من أن قيم AD لبضعة نانومترات قابلة للتحقيق في نظام السرعة العالية (<0.1 ثانية) ، وأن التتبع التفاضلي (موضع الخرزة المغناطيسية بالنسبة إلى حبة مرجعية) يقلل من AD في النطاق الزمني الأطول.
ملاحظة: نحصل عادة على AD يبلغ <3 نانومتر بأقصى معدل (دقة 1.2 كيلو هرتز أو 0.83 مللي ثانية) ، ويستمر AD في الانخفاض حتى 10 ثوان على الأقل ، مما يعني الحد الأدنى من الانجراف. أبلغ آخرون عن قيم مماثلة في إعدادات مماثلة9،10،11،12،34. - مع وجود مغناطيس في وضع الراحة (F ~ 0 pN) ، قم بتسجيل إحداثيات x و y للحبة المربوطة عند 1.2 كيلو هرتز. سجل الموضع لفترة طويلة بما فيه الكفاية (أي أطول بما فيه الكفاية من وقت الاسترخاء المميز للتذبذب35) بحيث يتم أخذ عينات كافية من الحركة البراونية.
ملاحظة: هنا ، يكون الاتجاه x على طول اتجاه المجال المغناطيسي ، في حين أن الحركة في y تمثل الحركة المستعرضة العمودية على المجال. - حرك المغناطيس بالقرب من خلية التدفق وكرر قياسات موضع الخرزة حتى يلمس المغناطيس الجزء العلوي من خلية التدفق برفق. تحرك بخطوات كبيرة (على سبيل المثال ، 1-2 مم) عندما يكون المغناطيس على بعد أكثر من 7 مم من مستوى العينة (نظرا لأن القوة المطبقة تزداد ببطء في المجال البعيد للمغناطيس) ، ولكن قلل حجم الخطوة تدريجيا (على سبيل المثال ، 0.1-0.5 مم) كلما اقتربت من الاقتراب من أجل معايرة أدق عند مستويات قوة أعلى (الشكل 2 ب).
- احسب القوة عند كل موضع مغناطيسي ، d ، باستخدام أي من الطريقتين البديلتين (يتم توفير برنامج نصي MATLAB "force calibration.m" بما في ذلك كلتا الطريقتين ؛ انظر الملف التكميلي 1).
- قم بقياس تباين إحداثيات الخرزة y ، (الشكل 2D) ومتوسط موضع z للحبة بالنسبة إلى أدنى موضع
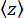 (الشكل 2B ،
(الشكل 2B ، 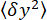 أسفل). ثم استخدم المعادلة (1) 7،27،36 لتقدير القوة (مع نصف قطر حبة ثابتة R = 1,400 نانومتر والطاقة الحرارية kRT = 4.11 pN∙nm):
أسفل). ثم استخدم المعادلة (1) 7،27،36 لتقدير القوة (مع نصف قطر حبة ثابتة R = 1,400 نانومتر والطاقة الحرارية kRT = 4.11 pN∙nm):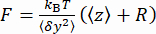 (1)
(1) - بدلا من ذلك ، احسب الكثافة الطيفية للقدرة (PSD) للإحداثيات y ، Sy (الشكل 2E). أوجد القوة المطبقة F عن طريق تركيب نموذج لورنتزيان مزدوج37 مع Sy المقاس باستخدام المعادلة (2).
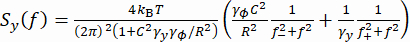 (2)
(2)
هنا ، R هو نصف قطر حبة ، γ yو γφ هما معاملات السحب الانتقالية والدورانية ، على التوالي (مقدرة من معادلة ستوكس أينشتاين) ، kRT هي الطاقة الحرارية ،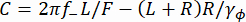 f + و f- هما ترددان مميزان تم الحصول عليهما باستخدام المعادلة (3).
f + و f- هما ترددان مميزان تم الحصول عليهما باستخدام المعادلة (3).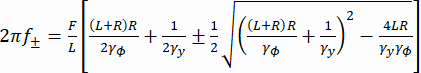 (3)
(3)
ملاحظة: نظرا لأن امتداد الحبل L هو دالة للقوة تتبع نموذج السلسلة الشبيهة بالديدان (WLC) الراسخ ، فإن التعبيرات أعلاه تترك F كمعلمة مناسبة وحيدة (نصلح R لتكون 1,400 نانومتر للبساطة لأنها مشتركة عبر جميع مستويات القوة ولا تؤثر القيمة الدقيقة على النتائج بشكل ملحوظ). عند الضرورة ، يجب اعتبار ضبابية الحركة والتعرج من الحصول على الصور المستندة إلى الكاميرا38,39 ، ولكن هذا التأثير لا يكاد يذكر في قياساتنا عالية السرعة فوق 1 كيلو هرتز مع حبال 5 كيلو بكسل.
- قم بقياس تباين إحداثيات الخرزة y ، (الشكل 2D) ومتوسط موضع z للحبة بالنسبة إلى أدنى موضع
- كرر الخطوات 2.4-2.7 للحصول على بعض البنيات الأخرى. دقق في ثلاث إلى خمس حبات مختلفة لحساب متوسط تباين القوة بين الخرزات المغناطيسية.
ملاحظة: يجب مراعاة تباين القوة بين الخرزات المغناطيسية المستخدمة لتحديد العدد المناسب من التركيبات للمتوسط. هذا التباين صغير ولكن يمكن أن يؤدي إلى أكثر من 1 pN من الخطأ في القوة المقاسة ، حتى بالنسبة للمنتجات التجارية31. بالنسبة لمعظم التطبيقات ، حيث لا يكون التحديد المطلق للقوى المعنية أمرا بالغ الأهمية ، فإن متوسط نتائج المعايرة من ثلاث إلى خمس حبات يكفي بشكل عام. هناك طريقة بديلة لتفسير هذا الاختلاف وهي قياس القوة باستخدام الحبال الفردية في بداية التجربة ، والتي يمكن أن تستغرق وقتا طويلا. خيار آخر هو تضمين هياكل دبوس الشعر التي يتم فك ضغطها عند مستويات القوة المعروفة في كل بناء31. - ارسم القوة المقاسة كدالة لمسافة المغناطيس وقم بملاءمة دالة أسية مزدوجة للبيانات (الشكل 2F) باستخدام المعادلة (4).
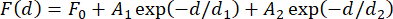 (4)
(4)
هنا ، F0 (خط الأساس) ، A 1 و A 2 (السعة) ، و d 1 و d2 (ثوابت الاضمحلال) هي معلمات مناسبة. تأكد من أن قيم القوة من الطريقتين ، بالإضافة إلى النوبات الأسية المزدوجة الناتجة ، تتفق إلى حد كبير (الشكل 2F ، G).
ملاحظة: للتأكد من إجراء معايرة القوة بشكل صحيح ، تحقق من علاقة امتداد القوة للتركيبات التي تم فحصها عن طريق رسم الامتداد مقابل القوة المقاسة. - لتصحيح إزاحة ارتفاع الخرزة z الناتجة عن الإمالة المعتمدة على القوة للخرز المغناطيسي30,31 ، قم بتقديرz من الإزاحة الجانبية x ، مع الأخذ في الاعتبار هندسة الحبل غير المركزي مع نصف قطر حبة باستخدام المعادلة (5) ، وتطبيق القيم على قيم الامتداد المقاسة. يتم تنفيذ هذه الخطوة في البرنامج النصي MATLAB "force calibration.m" (الأسطر 252-254).
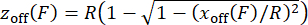 (5)
(5)
ملاحظة: على الرغم من أن هذا التصحيح يحدث تغييرات صغيرة على التمديد ، خاصة بالنسبة للخرزات ذات نصف قطر دوران صغير (<200 نانومتر) ، فإن هذا الإزاحة غالبا ما يؤثر بشكل خطير على الاستجابة المرنة ، كما هو موضح في التغيير من الشكل 2H إلى الشكل 2I 30,31. - تحقق من طول الثبات Lp عن طريق تركيب نموذج WLC قابل للتوسيع للبيانات باستخدام المعادلة (6).
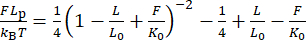 (6)
(6)
هنا ، L 0 هو طول الكفاف (1.7 ميكرومتر ل 5 kbp) و K0 هو معامل تمدد المحتوى الحراري.
ملاحظة: على الرغم من أن L p من dsDNA مقبول جيدا ليكون 40-50 نانومتر في مخزن مؤقت نموذجي مثل محلول ملحي مخزن بالفوسفات (PBS) ، فإن صيغة WLC المطبقة على الجزيئات القصيرة (<5 kbp) تقلل بشكل منهجي من L p حيث ينخفض L0 31,40. وذلك لأن نموذج WLC الكلاسيكي يفترض وجود بوليمر طول سلسلته أطول بما فيه الكفاية من طول ثباته. هنا ، حصلنا على L p = 40 ± 3 نانومتر لبناء 5 kbp (الشكل 2H) ، وأسفر تصحيح التمديد عن K 0 متجانس من 1100 ± 200 pN (الشكل 2I). سيؤدي تطبيق نموذج WLC المحدود 31,40 ، بالإضافة إلى تصحيح غير Gaussianity في توزيع الامتداد 41 ، إلى زيادة طفيفة Lp. - بمجرد التحقق من معايرة القوة ، قم بتطبيق معلمات التركيب التي تم الحصول عليها للنموذج الأسي المزدوج على برنامج LabVIEW المقدم (الملف التكميلي 2) وانتظر حتى يقوم البرنامج بحساب القوة الحالية في الوقت الفعلي من قراءات المحرك (أي موضع المغناطيس). نظرا لعدم توفر تعبير تحليلي للدالة العكسية d (F) ، قم بإعداد جدول بحث ل d مقابل F بخطوات 0.1 pN عن طريق التقدير العددي لمستويات القوة المستهدفة d. قم بتخزين هذا الجدول في البرنامج أيضا لقيادة التحكم في القوة.
3. تخليق دبابيس الشعر الحمض النووي
ملاحظة: يتم تحضير تركيبات دبوس شعر الحمض النووي لتجارب MT عن طريق تضخيم تفاعل البوليميراز المتسلسل لمنطقة 510 bp في λ-DNA مع اثنين من البادئات المخصصة ، أحدهما يحتوي على بنية دبوس الشعر على نهايته 5 (الشكل 3 أ). بهذه الطريقة ، يتم وضع شكل دبوس الشعر في أحد طرفي منتج PCR.
- تحضير الاشعال.
- التمهيدي الأمامي: التمهيدي B_hp الذي يحمل علامة 5 ′ بيوتين لربط سطح الزجاج ويرتبط ب λ-DNA. يحتوي هذا التمهيدي على شكل دبوس شعر بساق 8 bp وحلقة 6 nt ، 5 ′ إلى منطقة الارتباط λ.
- التمهيدي العكسي: التمهيدي Z_hp المسمى 5 ′ azide لمرفق حبة مغناطيسية ويرتبط ب λ-DNA 1 kbp بعيدا عن التمهيدي الأمامي.
- قم بإعداد وتشغيل تفاعل البوليميراز المتسلسل باستخدام λ-DNA (قالب) ، وبوليميراز nTaq ، وظروف تفاعل البوليميراز المتسلسل القياسية (انظر الجدول 1). نظف المنتج باستخدام مجموعة تنقية تجارية.
- قم بقياس تركيز الحمض النووي عن طريق امتصاص الأشعة فوق البنفسجية عند 260 نانومتر (A260) وقم بإجراء الرحلان الكهربائي لهلام الأغاروز (2٪ هلام) (انظر الجدول 2) للتحقق من حجم المنتج. العائد النموذجي هو ~ 35 ميكرولتر من محلول ~ 600 نانومتر.
4. إعداد بروتينات SNARE
ملاحظة: يتم تجميع معقدات SNARE العصبية من خلال الجمع بين ثلاثة بروتينات فئران منقاة يتم التعبير عنها من الإشريكية القولونية: VAMP2 / synaptobrevin-2 و syntaxin-1A و SNAP-25 (الشكل 3B). لتسهيل تجميعها ، يتم التعبير عن بناء الجملة و SNAP-25 مع جزء VAMP2 (يفتقر إلى منطقة N-terminal ؛ تسمى "ΔN-VAMP2") في بنية تسمى "ΔN-complex" ، ثم يتم خلطها مع VAMP2 كامل الطول بعد ارتباط مقبض الحمض النووي لتشكيل مجمعات كاملة.
- تحضير البلازميدات التي تحتوي على cDNA للتعبير عن بروتينات SNARE (يتم إعطاء تسلسل الحمض النووي لجميع البلازميدات في جدول المواد).
- تحضير 6×His-Taged VAMP2 الذي يفتقر إلى المجال عبر الغشاء (2-97; L32C/I97C لوصلات ثاني كبريتيد) المستنسخة في ناقل pET28a.
- تحضير syntaxin-1A الذي يفتقر إلى Habc والمجال عبر الغشاء (191-267 ، بدائل I202C / I266C لروابط ثاني كبريتيد) المستنسخة مع 6×His-Taged ΔN-VAMP2 (49-96) في متجه pETDuet-1.
- قم بإعداد الشكل المتماثل SNAP-25 كامل الطول b (2-206 ، كل C إلى A) المستنسخ في متجه pET28a. سيتم استخدام هذا لإعداد مجمعات ΔN.
- قم بإعداد الشكل المتماثل SNAP-25 كامل الطول 6× له (1-206 ، كل C إلى A) المستنسخ في متجه pET28a للإضافة المباشرة إلى المخزن المؤقت لمقايسة MT لإعادة تجميع مجمعات SNARE بعد الكشف.
- تحضير أنبوبين من خلايا Rosetta (DE3) E. coli. قم بتحويل مجموعة واحدة باستخدام بلازميدات VAMP2 (من الخطوة 4.1.1) ، واحدة مع كل من syntaxin-1A / ΔN-VAMP2 وبلازميدات SNAP-25 غير الموسومة (من الخطوتين 4.1.2 و 4.1.3) للتعبير عن مركب ΔN ، والأخرى مع بلازميدات SNAP-25 الموسومة به (من الخطوة 4.1.4).
- انقل الخلايا المحولة إلى مرق لوريا بيرتاني (LB) بالمضادات الحيوية المناسبة (هنا ، كاناميسين وكلورامفينيكول ل VAMP2 و SNAP-25 الموسوم به ؛ كاناميسين ، كلورامفينيكول ، وأمبيسيلين لمركب ΔN). قم بزراعتها عند 37 درجة مئوية في حاضنة اهتزاز (220 دورة في الدقيقة) حتى تصل الكثافة البصرية (OD) للمرق إلى 0.7-0.9.
- أضف 1 mM isopropyl β-d-1-thiogalactopyranoside (IPTG) للحث على التعبير عن البروتين واحتضان الخلايا لمدة 3-4 ساعات عند 37 درجة مئوية في حاضنة اهتزاز (220 دورة في الدقيقة).
- قم بتكسير الخلايا عن طريق الطرد المركزي للمزرعة عند 4500 × جم لمدة 15 دقيقة عند 4 درجات مئوية.
- تحضير المخازن المؤقتة لتنقية البروتين (انظر الجدول 2).
- تعليق كريات الخلايا المعبرة عن SNARE في 40 مل من محلول التحلل المثلج البارد وتحلل الخلايا عن طريق الصوتنة على الجليد (سعة 15٪ ، 5 ثوان و 5 ثوان ، إجمالي 30 دقيقة).
- أجهزة الطرد المركزي المحللة عند 15000 × جم لمدة 30 دقيقة عند 4 درجات مئوية لإزالة المواد غير القابلة للذوبان.
- مرر المادة الطافية من خلال عمود جاذبية مملوء ب 1 مل من راتنج Ni-NTA. اغسل الراتنج بمحلول الغسيل A ، ثم باستخدام المخزن المؤقت للغسيل B ، وقم بإزالة البروتينات ب 10 مل من محلول الشطف.
- قم بإزالة tris (2-carboxyethyl) phosphine (TCEP) والإيميدازول من المحلول باستخدام عمود تحلية (اتبع تعليمات الشركة المصنعة). قم بسحب العينة باستخدام برنامج تلفزيوني.
- ركز البروتينات باستخدام مرشحات الطرد المركزي (قطع 10 كيلو دالتون) إلى ~ 70 ميكرومتر مع الحفاظ على البروتينات في PBS (عادة ما تنتج 2 مل). قم بقياس تركيز البروتين إما عن طريق امتصاص الأشعة فوق البنفسجية (UV) عند 280 نانومتر (A280) أو عن طريق فحص برادفورد.
- تحضير الحصصات الصغيرة، وتجميدها في النيتروجين السائل، وتخزينها في -80 درجة مئوية حتى الاستخدام.
ملاحظة: سيتم تجميع مجمعات SNARE الكاملة بعد اقتران مركب ΔN على مقبض الحمض النووي (انظر أدناه).
5. ربط مقابض الحمض النووي
ملاحظة: يتم أولا تحضير مقبضين dsDNA 510 bp يحتويان على مجموعات أمينية أولية في أحد طرفيه بواسطة تفاعل البوليميراز المتسلسل ، ثم يتم تحويل مجموعات الأمين إلى مجموعات ماليميد باستخدام رابط متشابك ثنائي الوظيفة ، SM (PEG) 2. ثم يتم ربط المقبضين تساهميا بمعقدات SNARE عبر مجموعات السيستين الخاصة بهم للاقتران الخاص بالموقع (الشكل 3B).
- تحضير الاشعال.
- تحضير الاشعال الأمامية: التمهيدي B (لتضخيم المقبض B) الذي يحمل علامة 5 ′ البيوتين لربط سطح الزجاج ويرتبط ب λ-DNA ؛ التمهيدي Z (لتضخيم المقبض Z) المسمى 5 ′ azide لمرفق حبة مغناطيسية وله نفس تسلسل التمهيدي B.
- قم بإعداد التمهيدي العكسي: التمهيدي N (مشترك للمقبض B والمقبض Z) المسمى 5 ′ أمين لاقتران البروتين ويرتبط ب λ-DNA 510 bp بعيدا عن التمهيدي الأمامي.
- قم بإعداد وتشغيل مجموعتين من تفاعلات تفاعل البوليميراز المتسلسل (18 أنبوبا من تفاعل 200 ميكرولتر لكل مقبض) باستخدام λ-DNA (قالب) ، وبوليميراز nTaq ، وظروف تفاعل البوليميراز المتسلسل القياسية (انظر الجدول 1). نظف المنتج باستخدام مجموعة تنظيف PCR وقم بإزالة كل مقبض ب 45 ميكرولتر من الماء عالي النقاء. استخدم كمية صغيرة من الماء للحصول على تركيزات عالية من المقابض للحصول على تفاعل فعال في خطوات لاحقة.
- قم بقياس تركيز الحمض النووي بواسطة A260. العائد النموذجي هو ~ 650 ميكرولتر من محلول ~ 2 ميكرومتر لكل مقبض. احتفظ بالعينات الصغيرة بعيدا عن بعضها البعض للتحقق لاحقا في الرحلان الكهربائي لهلام الأغاروز.
- تفاعل مع كل مقبض (1 ميكرومتر في PBS) مع 5 مللي متر SM (PEG) 2. احتضان في درجة حرارة الغرفة مع دوران لطيف. بعد 1 ساعة ، استخدم مجموعة تنقية الحمض النووي لإزالة SM (PEG) 2 غير المتفاعل. قم بسحب كل مقبض ب 250 ميكرولتر من PBS للحصول على حلول ~ 2 ميكرومتر.
- امزج محاليل المقبض B ومركب ΔN بنسبة مولية تبلغ 1:16 (على سبيل المثال ، 1 ميكرومتر مقبض B و 16 ميكرومتر مركب ΔN) في PBS واحتضانها لمدة 2 ساعة في درجة حرارة الغرفة مع الإثارة. احفظ عينة صغيرة عن الرحلان الكهربائي لهلام الأغاروز.
- أضف محلول VAMP2 في فائض مولي 2.5 أضعاف فوق مركب ΔN المستخدم في الخطوة السابقة. احتضان الخليط لمدة 1 ساعة أخرى في درجة حرارة الغرفة مع الإثارة. يتم تجميع مجمعات SNARE الكاملة في هذه الخطوة.
- قم بإزالة البروتينات الحرة عن طريق التبادل العازل مع برنامج تلفزيوني جديد ومرشح طرد مركزي (قطع 100 كيلو دالتون): جهاز طرد مركزي عند 14000 × جم لمدة 5 دقائق عند 4 درجات مئوية ، كرر 6x على الأقل ، وقم بتشغيله لمدة 15 دقيقة لآخر دورة. قم بقياس الزيادة في نسبة A260 / A280 لمراقبة إزالة البروتينات الحرة. احفظ عينة صغيرة عن الرحلان الكهربائي لهلام الأغاروز.
- أضف المقبض Z إلى المحلول في فائض مولي بمقدار 15 ضعفا على المقبض B. حافظ على تركيز المقبض Z أعلى من 1 ميكرومتر على الأقل لتسهيل التفاعل. احتضان الخليط طوال الليل على حرارة 4 درجات مئوية مع التقليب.
- تحقق من المواد الوسيطة (المقبض B واتحادات البروتين) والمنتج النهائي (مركب SNARE بمقبضين) عن طريق الرحلان الكهربائي لهلام الأغاروز (الشكل 3B ، أقحم) (انظر الجدول 2).
ملاحظة: إذا تم ربط البروتينات بنجاح بالمقبض B ، اكتشاف تحول في الحركة. على وجه الخصوص ، يمكن تأكيد تكوين معقدات SNARE الكاملة على مقابض الحمض النووي من خلال مقاومتها لكبريتات دوديسيل الصوديوم (SDS) ، على عكس مجمعات ΔN ، التي يتم تفكيكها في SDS وتترك فقط بناء الجملة مرتبطا بالحمض النووي (قارن b و c في الشكل 3B). - تحضير الحصصات الصغيرة، وتجميدها في النيتروجين السائل، وتخزينها في -80 درجة مئوية حتى الاستخدام.
ملاحظة: على الرغم من أن الحل النهائي يحتوي على مقابض غير متفاعلة ، إلا أنه سيتم اختيار البنية المطلوبة التي تحمل علامة مزدوجة بالبيوتين والأزيد فقط أثناء تجميع العينة في خلية التدفق.
6. تصنيع خلايا التدفق
ملاحظة: يتم إنشاء خلايا التدفق لقياسات MT من خطتين زجاجيتين مرتبطتين معا بشريط على الوجهين (الشكل 3C). يتم طلاء غطاء واحد بمزيج من PEG والبولي إيثيلين جلايكول البيوتينيل (PEG) لتجنب الارتباط غير المحدد ولتمكين الربط المحدد للجزيئات المستهدفة عبر ارتباط البيوتين والنيوترافيدين (الشكل 3D). بعد ذلك ، يتم غرس محاليل المواد الخاصة بتجارب MT بالتتابع في خلية تدفق باستخدام مضخة حقنة (الشكل 3C ، D).
- قم بإعداد غطاءين زجاجيين ، أحدهما لكل من السطح العلوي (24 مم × 50 مم ، رقم 1.5 سمك) والسطح السفلي (24 مم × 60 مم ، رقم 1.5 سمك). قم بتنظيف أغطية الغطاء عن طريق الصوتنة في 1 M KOH لمدة 30 دقيقة. بعد صوتنة ، شطف الغطاءزلات بالماء المقطر والاحتفاظ بها في الماء حتى الخطوة التالية.
- PEGylate الغطاء السفلي باتباع البروتوكولات المنشورة42,43. استخدم N- [3- (تريميثوكسي سيليل) بروبيل] إيثيلين ديامين للسيلانا وخليط 1: 100 (ww) من البيوتين-PEG-SVA و mPEG-SVA في مخزن مؤقت بيكربونات 100 مللي متر. حافظ على أغطية PEGylated جافة عند -20 درجة مئوية وقم بتخزينها لبضعة أسابيع.
- في يوم التجارب ، أخرج أغطية PEGylated وجففها بمسدس النيتروجين. افحصها بصريا بحثا عن الأوساخ للتأكد من نظافتها.
- لعمل قنوات العينة ، قم بإعداد شرائط بعرض ~ 2 مم من الشريط على الوجهين ووضع أربعة شرائط على غطاء سفلي (سطح PEGylated لأعلى) ، بالتوازي مع بعضها البعض ومفصولة عن بعضها البعض بواسطة ~ 5 مم (الشكل 3C).
ملاحظة: بهذه الطريقة ، يمكن إنشاء ثلاث قنوات عينة بعرض 5 مم في خلية تدفق واحدة. - ضع غطاء علوي في وسط قسيمة الغطاء السفلية ، مع ترك ~ 5 مم من المساحة على الحواف القصيرة لمداخل ومنافذ القناة. اضغط برفق على الجزء الخلفي من الغطاء العلوي باستخدام ملاقط لإغلاق القنوات بإحكام.
- لعمل خزان مدخل ، قم بقص حافة طرف ماصة 200 ميكرولتر. اقطع ~ 10 مم من الفتحة الأوسع للسماح بالاحتفاظ ~ 200 ميكرولتر من المحلول. اصنع ثلاثة منهم لقنوات التدفق الثلاثة. لتكوين المنافذ ، قم بإعداد ثلاث إبر حقنة تناسب الأنبوب لمضخة المحقنة.
- باستخدام 5 دقائق من الإيبوكسي ، قم بلصق الخزانات ومحاور الإبرة بخلية التدفق. تأكد من تشكيل ختم كامل لتجنب التسرب ، وأن القنوات ليست مسدودة بالغراء الزائد. اتركه يجف لمدة 30 دقيقة على الأقل.
7. تجميع بنى حبة الحبل
ملاحظة: يتم إدخال حلول المواد لتجارب MT ، بما في ذلك تلك الخاصة بتركيبات حبال الخرز ، بالتتابع في خلايا التدفق باستخدام مضخة حقنة (الشكل 3C ، D).
- تحضير الخرز المغناطيسي. خذ 5 ملغ من حبات الإيبوكسي M270 من محلول مخزون (~ 3.3 × 108 حبات في 167.5 ميكرولتر من ثنائي ميثيل فورماميد) واستبدل المذيب بمحلول فوسفات (انظر الجدول 2) عن طريق الفصل المغناطيسي للخرز.
- تحضير الخرز في ~ 1.1 × 109 حبات mL−1 في مخزن فوسفات مع 1 M كبريتات الأمونيوم وتفاعلها مع 2 mM dibenzocyclooctyne (DBCO) -NH2. احتضان الخليط لمدة 3 ساعات على خلاط دوار في درجة حرارة الغرفة. بعد التفاعل ، اغسل الخرزات 3x بمحلول فوسفات طازج لإزالة الجزيئات غير المتفاعلة.
ملاحظة: يمكن تخزين الخرزات المغسولة دون دوران إضافي عند 4 درجات مئوية لعدة أسابيع قبل الاستخدام. - قم بتوصيل إبرة على مخرج قناة خلية التدفق بمضخة المحقنة باستخدام أنابيب البولي إيثيلين. موازنة القنوات مع برنامج تلفزيوني.
- أدخل الحلول التالية بالتتابع في القناة عن طريق الشفط بالمضخة: NeutrAvidin ، التركيبات المستهدفة (دبابيس شعر الحمض النووي أو مجمعات SNARE بمقابض الحمض النووي) ، حبات البوليسترين المرجعية ، والخرز المغناطيسي المطلي ب DBCO. قبل الاستخدام ، دوامة محاليل الخرزة تماما لتفريق مجاميع الخرز المحتملة.
- اغسل الخرزات غير المنضمة أثناء تطبيق 0.1 pN من القوة.
ملاحظة: يسهل تطبيق قوة تصاعدية صغيرة إزالة الخرز غير المنضم ويساعد على تجنب تمزق تركيبات حبال الخرز المربوطة على وجه التحديد. - للتجارب مع مجمعات SNARE ، قم بتضمين 1.5 ميكرومتر SNAP-25 في المخزن المؤقت النهائي.
ملاحظة: يمكن لجزيئات SNAP-25 الحرة إعادة ربط مجمعات SNARE بعد الكشف عنها والسماح بإجراء قياسات متكررة على مركب واحد.
8. تحديد التركيبات المستهدفة
- على سطح قناة خلية التدفق ، ابحث عن الخرزات المغناطيسية المربوطة بجزيئات مفردة من البنية المستهدفة. تأكد من وجود حبة مرجعية في مكان قريب.
- قم بتدوير حبة مرشحة وتأكد من أنها تدور بحرية. إذا كانت الخرزة مربوطة بجزيئات متعددة ، فإنها تظهر حركة مقيدة.
- قم بتدوير الخرزة لبضع لفات كاملة واكتشف نصف قطر الدوران (يتم تنفيذ هذه الوظيفة في البرنامج المقدم). على نحو مفضل ، اختر حبة ذات نصف قطر دوران صغير.
ملاحظة: يشير نصف القطر هذا إلى مقدار انحراف الخرزة عن محور الحبل ، والذي يتم تحديده عشوائيا أثناء تجميع حبل الخرز30,31. في جميع التجارب ، يخفف الحد الأدنى من التمركز للخرزة من العديد من القطع الأثرية المرتبطة بنصف قطر الخرزة العالي إلى نسبة تمديد الحبل التي نستخدمها. - قم بزيادة القوة من 0 إلى 5 pN لتحديد حبات جيدة مربوطة واحدة. ابحث عن تغيير كبير في نمط الحيود للحبة الناتجة عن تمدد حبل 1 كيلو بايت (أو ما يعادل مقبضين 510 bp). إذا لم يتغير نمط الحيود بشكل كبير ، فقم بخفض القوة إلى الصفر وابحث عن حبة مرشح أخرى.
ملاحظة: يمكن ملاحظة رفع حبة ~ 300 نانومتر بسهولة من الصور الأولية دون البدء فعليا في عملية التتبع.
9. تتبع حبة لقياسات التمديد
ملاحظة: يتم إجراء تتبع الخرز عن طريق تحليل صور الخرز في الوقت الفعلي في برنامج LabVIEW المزود مع هذه المقالة. تم استخدام طريقة التتبع ومتغيراتها في معظم أنظمة MT التقليدية وتم شرحها في الأدبيات السابقة2،5،7،26. من خلال قياس موضع حبة مغناطيسية بالنسبة إلى حبة مرجعية ثابتة (أي التتبع التفاضلي) ، تصبح قياسات الموضع قوية للغاية لاضطراب خارجي.
- بمجرد وضع حبة مغناطيسية مناسبة مع حبة مرجعية ، انقر فوق الزر معايرة لبدء التحضير لتتبع الخرزة.
- انقر على الخرز في الصورة لتحديد مواقع الخرز. سيتم بعد ذلك اقتصاص الصور إلى مناطق الاهتمام (ROIs) (على سبيل المثال ، 150 × 150 بكسل لحبة 3 ميكرومتر) حول الخرز ثم تحليلها بشكل أكبر لاستخراج إحداثيات الخرزة الدقيقة.
- انتظر حتى يكتمل دوران المغناطيس. تسجل هذه العملية إحداثيات x و y للخرزة (عن طريق حساب الارتباط المتقاطع 2D44 أو باستخدام التماثل الشعاعي45 لصور الخرز ، مع أداء مماثل) أثناء تدوير المغناطيس لتوثيق المرفق خارج المركز للحبة31.
- للتتبع في الاتجاه z ، انتظر حتى يقوم البرنامج بإنشاء جدول بحث لصور حيود الخرز على مسافات مختلفة من المستوى البؤري. يتم ذلك عن طريق تحريك العدسة الموضوعية باستخدام ماسح بيزو في خطوات متساوية البعد وتسجيل صور حبة متوسط التذبذب في كل موضع. بعد ذلك ، يتم تحديد إحداثيات z للخرز في التجارب الفعلية من خلال مقارنة صور الخرز في الوقت الفعلي بجدول البحث مع الاستيفاء7.
- عند الانتهاء من إنشاء جدول البحث، قم بتمكين التتبع والتركيز التلقائي (اضغط على الزر تعقب ؟ والتركيز البؤري التلقائي؟ الأزرار) وانقر على زر الاستحواذ لبدء تسجيل مواضع الخرز.
ملاحظة: التركيز البؤري التلقائي اختياري ولكن يوصى بتصحيحه لانحراف المرحلة في z أثناء الاستحواذ.
10. فرض مخططات التطبيق
- تجارب منحدر القوة: للتحقق من علاقة امتداد القوة للبناء ، قم بتطبيق منحدر القوة لأعلى ولأسفل بمعدل تحميل ثابت (± 1.0 pN s−1) (الشكل 4A). على سبيل المثال ، قم بتطبيق ثلاث جولات من دورة 0-20-0 pN للتحقق من الطول الإجمالي للبنية ومنحنى تمديد القوة للمقابض.
- من خلال تحديد معلمات الحبل في البرنامج ، قم بتراكب منحنى تمديد قوة WLC أعلى البيانات المقاسة ، وحدد ما إذا كانت حبة الهدف مربوطة ببناء عينة حقيقية بمقابض الحمض النووي المناسبة. استخدم طول الكفاف المعروف (على سبيل المثال ، ~ 340 نانومتر ل 1 كيلو بايت dsDNA) وطول ثبات WLC (30-45 نانومتر ل dsDNA31 القصير) للبناء كنقطة بداية. قم بتطبيق طريقة تصحيح التمديد الموضحة في الخطوة 2.11 إذا لزم الأمر.
- إذا تم التحقق من البنية ، فقم بفحص استجابة امتداد القوة بالتفصيل للبحث عن امتداد إضافي ناتج عن دبابيس الشعر للجزيئات المستهدفة أو مجمعات SNARE.
- تجارب القوة الثابتة: قم بتغيير القوة المطبقة تدريجيا في خطوات منفصلة للتحقق من حساسية قوة الجزيئات المستهدفة (الشكل 4B).
ملاحظة: تتيح MTs إجراء تجارب بسيطة وفعالة للقوة الثابتة لأن القوة المطبقة تظل ثابتة عندما يظل المغناطيس ثابتا.- بالنسبة لدبابيس شعر الحمض النووي ، قم بتطبيق 4-8 pN من القوة بخطوات 0.2-0.5 pN ، وقم بقياس موضع الخرزة ل ~ 10 s عند كل مستوى قوة.
- بالنسبة لمجمعات SNARE ، قم بتطبيق 14-16 pN من القوة بخطوات 0.1-0.2 pN ، وقم بقياس موضع الخرزة ل ~ 10 s عند كل مستوى قوة.
- تجارب القفز بالقوة: مراقبة الأحداث الانتقالية لمجمعات SNARE.
ملاحظة: تتضمن تجارب القفز بالقوة، مثل تجارب القوة الثابتة، تغييرات في مستويات القوة. ومع ذلك ، فإن قفزات القوة تستخدم المزيد من التغييرات المفاجئة في القوة المطبقة ، مما يسمح بمراقبة الأحداث التي تسببها القوة في الجزيئات التي تم فحصها ، مثل التمزق المفاجئ لمجمعات البروتين. على سبيل المثال ، نظرا لأن مجمعات SNARE تظهر تباطؤا هيكليا في دورة القوة23 ، فمن المفيد إجراء تجارب القفز بالقوة وقياس زمن الانتقال إلى الانتقال (الشكل 4C).- حيويي: تقشير جزيء VAMP2 من مركب SNARE ثلاثي سليم ، تاركا معقدا ثنائيا من syntaxin-1A و SNAP-25.
- إعادة الضغط: ضغط جزيء VAMP2 غير المضغوط لتجديد مركب SNARE سليم.
- تتكشف: التفكيك الكامل لمجمع SNARE مصحوبا بتفكك كامل ل SNAP-25. تبقى جزيئات VAMP2 و syntaxin فقط في البناء بعد أن تتكشف.
- إعادة الطي: تجديد مركب SNARE عند ربط جزيء SNAP-25 الحر من المخزن المؤقت.
- عند 2 pN ، قم بتحفيز تجميع مجمع SNARE سليم عن طريق الانتظار (~ 30 ثانية) لارتباط جزيء SNAP25 الحر. لوحظ انخفاض مفاجئ في التمديد عند تكوين مجمع SNARE.
- لمراقبة أحداث فك الضغط ، انتظر بضع ثوان عند 10-12 pN ، ثم انتقل إلى 14-15 pN فجأة بأقصى سرعة ممكنة للمحرك. اعتمادا على القوة المستهدفة ، سيظهر مجمع SNARE إما انتقالا عكسيا بين المواد الوسيطة غير المضغوطة جزئيا (كما هو الحال في تجارب القوة الثابتة) أو قفزة ~ 25 نانومتر إلى حالة أعلى غير مضغوطة بعد وقت انتظار عشوائي (أو زمن انتقال).
- لمراقبة أحداث إعادة الضغط ، قم بخفض القوة إلى 10-12 pN فور ملاحظة فك الضغط. مرة أخرى ، يظهر مجمع SNARE انتقالا عشوائيا إلى الحالة السفلية بسحاب بعد بعض الكمون العشوائي. إذا حدث التكشف بعد فك الضغط ، فسوف يفشل المجمع في إعادة الضغط ، حيث سيكون جزيء SNAP-25 مفقودا.
- لمراقبة الأحداث التي تتكشف ، انتظر لفترة أطول بعد ملاحظة فك الضغط لاكتشاف زيادة أخرى في الامتداد (~ 2 نانومتر).
11. تحليل البيانات
ملاحظة: تعتمد أنواع التحليل التي يمكن للمرء إجراؤها باستخدام بيانات الترجمة الآلية على النظام المستهدف. ومع ذلك ، هناك طرق شائعة لاستخراج معلومات مفيدة من التجارب المعنية الموضحة في الشكل 4. يتم إجراء جميع التحليلات باستخدام MATLAB (R2021a) باستخدام الرموز المخصصة المتوفرة مع هذه المقالة. تقوم هذه الرموز بإنشاء مؤامرات باستخدام نفس البيانات الواردة في هذه المقالة. لاحظ أنه بينما تم أخذ البيانات الأولية من تتبع 100 هرتز مباشرة للتحليل ، فإن البيانات من تتبع 1.2 كيلو هرتز كانت عادة ما تتم تصفيتها بشكل متوسط (مع نافذة منزلقة من خمس نقاط) قبل التحليل لتقليل الضوضاء (باستثناء تحليل الضوضاء).
- تجارب منحدر القوة: تحليل العلاقة بين القوة والتوسع (على سبيل المثال ، مرونة البوليمرات) ونقل القوة لاستخراج المعلومات حول الخواص الميكانيكية النانوية.
- تجارب القوة الثابتة: تحليل سكان الولاية ووقت السكون (أو معدل الانتقال) كدالة للقوة لاستخراج المعلمات الهيكلية (على سبيل المثال ، المناطق المشاركة في المرحلة الانتقالية) ، والديناميكا الحرارية (على سبيل المثال ، فرق الطاقة الحرة) ، والمعلمات الحركية (على سبيل المثال ، حاجز الطاقة) للتغيرات المطابقة.
- تجارب القفز بالقوة: تحليل حركية التمزق (على سبيل المثال ، تفاعلات البروتين والبروتين وربط مستقبلات اليجند) أو عمر الوسيطات العابرة (على سبيل المثال ، تكشف الجزيئات الحيوية) لاستخراج استقرار الجزيئات المستهدفة وحالاتها.
- كتطبيقات تمثيلية ، قم بتحليل بيانات العينة لدبابيس شعر الحمض النووي ومجمعات SNARE:
- انتقالات الدولتين لدبوس شعر الحمض النووي: قوة فك الضغط ، ومسافة الفتح ، واعتماد القوة على التحول السكاني ، وتعيين الحالة وقياسات معدل الانتقال باستخدام نموذج ماركوف المخفي (HMM) (رموز MATLAB المقدمة).
- التغييرات التوافقية لمجمعات SNARE: قوة فك الضغط ، واعتماد القوة على الحالات الوسيطة وزمن انتقال فك الضغط ، والتباطؤ في إعادة الضغط ، وسلوك التكشف / إعادة الطي.
ملاحظة: تم إعطاء نماذج تمديد القوة لمقابض الحمض النووي ودبابيس شعر الحمض النووي والمطابقات المعقدة SNARE في المراجع السابقة14،31.
النتائج
معايرة القوة
اختلفت النتائج من طريقتي قياس القوة (تباين الإزاحة الجانبية للخرز وتحليل طيف القدرة) بمقدار 0-2 pN (الشكل 2G). وفقا للنتائج الواردة في الشكل 2F ، يمكننا الوصول بشكل موثوق إلى 30 pN باستخدام مغناطيس النيوديميوم العادي.
ان?...
Discussion
في هذا العمل ، قدمنا إعداد التحليل الطيفي للقوة أحادية الجزيء الذي يمكنه مراقبة التغيرات الهيكلية للجزيئات الحيوية بدقة مكانية زمانية عالية. تكتسب كاميرا CMOS عالية السرعة المستخدمة 1200 إطار s−1 بدقة 1280 × 1024 ، مما يتيح تتبع حبة 1.2 كيلو هرتز. ومع ذلك ، فإن سرعة القياسات محدودة حاليا بواسط...
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح للإعلان عنه.
Acknowledgements
تم دعم هذا العمل من خلال منحة المؤسسة الوطنية للبحوث الكورية (NRF) الممولة من الحكومة الكورية (MSIT) (NRF-2022R1C1C1012176 و NRF-2021R1A4A1031754 و NRF- 2021R1A6A1A10042944). تم دعم S.-H.R. من خلال منحة NRF (2021R1C1C2009717).
Materials
| Name | Company | Catalog Number | Comments |
| Materials for construct synthesis | |||
| Agarose gel electrophoresis system | Advance | Mupid-2plus | |
| DNA ladder | Bioneer | D-1037 | |
| nTaq polymerase | Enzynomics | P050A | |
| PCR purification kit | LaboPass | CMR0112 | |
| PEGylated SMCC crosslinker / SM(PEG)2 | ThermoFisher Scientific | 22102 | For SNARE–DNA coupling |
| Primer B | Bioneer | 5'-Biotin/TCGCCACCATCATTTCCA-3' | For 5-kbp force calibration construct and DNA handles |
| Primer B_hp | IDT | 5'-Biotin/TTTTTTTTTTGTTCTCTATTT TTTTAGAGAAC /AP site/ /AP site/ TCGCCACCATCATTTCCA-3' | For hairpin construct |
| Primer N | Bioneer | 5'-C6Amine/CATGTGGGTGACGCGAAA-3' | For DNA handles |
| Primer Z | Bioneer | 5'-Azide/TCGCCACCATCATTTCCA-3' | For DNA handles |
| Primer Z_5k | Bioneer | 5'-Azide/TTAGAGAGTATGGGTATATGACA TCG-3' | For 5-kbp force calibration construct |
| Primer Z_hp | Bioneer | 5'-Azide/GTGGCAGCATGACACC-3' | For hairpin construct |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| λ-DNA | Bioneer | D-2510 | Template strand for PCR |
| DNA sequences for SNARE proteins | |||
| 6×His-tagged SNAP-25b (2-206; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctat gagaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagcgg aagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCACA GCAGCGGCCTGGTGCCGCGC GGCAGCCATACTAGCGGAGAT ATCGCCGAGGACGCAGACAT GCGCAATGAGCTGGAGGAGA TGCAGAGGAGGGCTGACCAG CTGGCTGATGAGTCCCTGGA AAGCACCCGTCGCATGCTGC AGCTGGTTGAAGAGAGTAAA GATGCTGGCATCAGGACTTT GGTTATGTTGGATGAGCAAG GCGAACAACTGGAACGCATT GAGGAAGGGATGGACCAAAT CAATAAGGACATGAAAGAAG CAGAAAAGAATTTGACGGAC CTAGGAAAATTCGCCGGCCT TGCCGTGGCCCCCGCCAAC AAGCTTAAATCCAGTGATGC TTACAAAAAAGCCTGGGGC AATAATCAGGATGGAGTAGT GGCCAGCCAGCCTGCCCG TGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTG GCTTCATCCGCAGGGTAAC AAATGATGCCCGGGAAAAT GAGATGGATGAGAACCTG GAGCAGGTGAGCGGCATC ATCGGAAACCTCCGCCAC ATGGCTCTAGACATGGGCA ATGAGATTGACACCCAGA ATCGCCAGATCGACAGGA TCATGGAGAAGGCTGATT CCAACAAAACCAGAATTG ATGAAGCCAACCAACGTG CAACAAAGATGCTGGGAA GTGGTTAAggatccgaattcgag ctccgtcgacaagcttgcggccgcactc gagcaccaccaccaccaccactgagat ccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgag caataactagcataaccccttggggcct ctaaacgggtcttgaggggttttttgctga aaggaggaactatatccggat | |
| 6×His-tagged VAMP2 (2-97, L32C/I97C; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCAC AGCAGCGGCCTGGTGCCGC GCGGCAGCCATATGGCAGAT CTCTCGGCTACCGCTGCCAC CGTCCCGCCTGCCGCCCCG GCCGGCGAGGGTGGCCCCC CTGCACCTCCTCCAAATCTTA CCAGTAACAGGAGATGCCAG CAGACCCAGGCCCAGGTGG ATGAGGTGGTGGACATCATG AGGGTGAATGTGGACAAGGT CCTGGAGCGAGACCAGAAG CTATCGGAACTGGATGATCG CGCAGATGCCCTCCAGGCA GGGGCCTCCCAGTTTGAAA CAAGTGCAGCCAAGCTCAA GCGCAAATACTGGTGGAAA AACCTCAAGATGATGTGCTA Aggatccgaattcgagctccgtcg acaagcttgcggccgcactcgagcaccacca ccaccaccactgagatccggctgctaacaaa gcccgaaaggaagctgagttggctgctgcca ccgctgagcaataactagcataaccccttgg ggcctctaaacgggtcttgaggggttttttg ctgaaaggaggaactatatccggat | |
| 6×His-tagged ΔN-VAMP2 (49–96; capitalized) and Syntaxin-1A (191–267, I202C/I266C; capitalized) in pETDuet-1 | homemade | ggggaattgtgagcggataacaattcccctc tagaaataattttgtttaactttaagaagga gatataccATGGGCAGCAGCCATCA TCATCATCATCACAGCAGCGG CCTGGAAGTTCTGTTCCAGGG GCCCGGTAATGTGGACAAGGT CCTGGAGCGAGACCAGAAGCT ATCGGAACTGGATGATCGCGC AGATGCCCTCCAGGCAGGGGC CTCCCAGTTTGAAACAAGTGC AGCCAAGCTCAAGCGCAAATAC TGGTGGAAAAACCTCAAGATGAT GTAAgcggccgcataatgcttaagtcgaaca gaaagtaatcgtattgtacacggccgcataa tcgaaattaatacgactcactataggggaat tgtgagcggataacaattccccatcttagta tattagttaagtataagaaggagatatacat ATGGCCCTCAGTGAGATCGAGA CCAGGCACAGTGAGTGCATC AAGTTGGAGAACAGCATCCG GGAGCTACACGATATGTTCAT GGACATGGCCATGCTGGTGG AGAGCCAGGGGGAGATGATT GACAGGATCGAGTACAATGTG GAACACGCTGTGGACTACGTG GAGAGGGCCGTGTCTGACACC AAGAAGGCCGTCAAGTACCAG AGCAAGGCACGCAGGAAGAA GTGCATGATCTAActcgagtc tggtaaagaaaccgctgctgcgaaatttgaa cgccagcacatggactcgtctactagcgcag cttaattaacctaggctgctgccaccgctga gcaataactagcataaccccttggggcctct aaacgggtcttgaggggttttttgctgaaag gaggaactatatccggattggcgaatgggac gcgccctgtagcggcgcattaagcgcggcgg gtgtggtggttacgcgcagcgtgaccgctac acttgccagcgccctagcgcccgctcctttc gctttcttcccttcctttctcgccacgttcg ccggctttccccgtcaagctctaaatcgggg gctccctttagggttccgatttagtgcttta cggcacctcgaccccaaaaaacttgattagg gtgatggttcacgtagtgggccatcgccctg atagacggtttttcgccctttgacgttggag tccacgttctttaatagtggactcttgttcc aaactggaacaacactcaaccctatctcggt ctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctga tttaacaaaaatttaacgcgaattttaacaa aatattaacgtttacaatttctggcggcacg atggcatgagattatcaaaaaggatcttcac ctagatccttttaaattaaaaatgaagtttt aaatcaatctaaagtatatatgagtaaactt ggtctgacagttaccaatgcttaatcagtga ggcacctatctcagcgatctgtctatttcgt tcatccatagttgcctgactccccgtcgtgt agataactacgatacgggagggcttaccatc tggccccagtgctgcaatgataccgcgagac ccacgctcaccggctccagatttatcagcaa taaaccagccagccggaagggccgagcgca gaagtggtcctgcaactttatccgcctccatc cagtctattaattgttgccgggaagctagag taagtagttcgccagttaatagtttgcgcaa cgttgttgccattgctacaggcatcgtggtg tcacgctcgtcgtttggtatggcttcattca gctccggttcccaacgatcaaggcgagttac atgatcccccatgttgtgcaaaaaagcggtt agctccttcggtcctccgatcgttgtcagaa gtaagttggccgcagtgttatcactcatggt tatggcagcactgcataattctcttactgtc atgccatccgtaagatgcttttctgtgactg gtgagtactcaaccaagtcattctgagaata gtgtatgcggcgaccgagttgctcttgcccg gcgtcaatacgggataataccgcgccacata gcagaactttaaaagtgctcatcattggaaa acgttcttcggggcgaaaactctcaaggatc ttaccgctgttgagatccagttcgatgtaac ccactcgtgcacccaactgatcttcagcatc ttttactttcaccagcgtttctgggtgagcaaa aacaggaaggcaaaatgccgcaaaaaagg gaataagggcgacacggaaatgttgaatact catactcttcctttttcaatcatgattgaag catttatcagggttattgtctcatgagcgga tacatatttgaatgtatttagaaaaataaac aaataggtcatgaccaaaatcccttaacgtg agttttcgttccactgagcgtcagaccccgt agaaaagatcaaaggatcttcttgagatcct ttttttctgcgcgtaatctgctgcttgcaaa caaaaaaaccaccgctaccagcggtggtttg tttgccggatcaagagctaccaactcttttt ccgaaggtaactggcttcagcagagcgcaga taccaaatactgtccttctagtgtagccgta gttaggccaccacttcaagaactctgtagca ccgcctacatacctcgctctgctaatcctgt taccagtggctgctgccagtggcgataagtc gtgtcttaccgggttggactcaagacgatag ttaccggataaggcgcagcggtcgggctgaa cggggggttcgtgcacacagcccagcttgga gcgaacgacctacaccgaactgagataccta cagcgtgagctatgagaaagcgccacgcttccc gaagggagaaaggcggacaggtatccggta agcggcagggtcggaacaggagagcgcac gagggagcttccagggggaaacgcctggtatc tttatagtcctgtcgggtttcgccacctctg acttgagcgtcgatttttgtgatgctcgtca ggggggcggagcctatggaaaaacgccagc aacgcggcctttttacggttcctggccttttg ctggccttttgctcacatgttctttcctgcg ttatcccctgattctgtggataaccgtatta ccgcctttgagtgagctgataccgctcgccgc agccgaacgaccgagcgcagcgagtcagtg agcgaggaagcggaagagcgcctgatgcgg tattttctccttacgcatctgtgcggtatttc acaccgcatatatggtgcactctcagtacaa tctgctctgatgccgcatagttaagccagta tacactccgctatcgctacgtgactgggtca tggctgcgccccgacacccgccaacacccgc tgacgcgccctgacgggcttgtctgctcccg gcatccgcttacagacaagctgtgaccgtct ccgggagctgcatgtgtcagaggttttcacc gtcatcaccgaaacgcgcgaggcagctgcgg taaagctcatcagcgtggtcgtgaagcgatt cacagatgtctgcctgttcatccgcgtccag ctcgttgagtttctccagaagcgttaatgtc tggcttctgataaagcgggccatgttaaggg cggttttttcctgtttggtcactgatgcctc cgtgtaagggggatttctgttcatgggggta atgataccgatgaaacgagagaggatgctca cgatacgggttactgatgatgaacatgcccg gttactggaacgttgtgagggtaaacaactg gcggtatggatgcggcgggaccagagaaaaa tcactcagggtcaatgccagcgcttcgttaa tacagatgtaggtgttccacagggtagccag cagcatcctgcgatgcagatccggaacataa tggtgcagggcgctgacttccgcgtttccag actttacgaaacacggaaaccgaagaccatt catgttgttgctcaggtcgcagacgttttgc agcagcagtcgcttcacgttcgctcgcgtat cggtgattcattctgctaaccagtaaggcaa ccccgccagcctagccgggtcctcaacgaca ggagcacgatcatgctagtcatgccccgcgc ccaccggaaggagctgactgggttgaaggct ctcaagggcatcggtcgagatcccggtgcct aatgagtgagctaacttacattaattgcgtt gcgctcactgcccgctttccagtcgggaaac ctgtcgtgccagctgcattaatgaatcggcc aacgcgcggggagaggcggtttgcgtattgg gcgccagggtggtttttcttttcaccagtga gacgggcaacagctgattgcccttcaccgcc tggccctgagagagttgcagcaagcggtcca cgctggtttgccccagcaggcgaaaatcctg tttgatggtggttaacggcgggatataacat gagctgtcttcggtatcgtcgtatcccacta ccgagatgtccgcaccaacgcgcagcccgga ctcggtaatggcgcgcattgcgcccagcgcc atctgatcgttggcaaccagcatcgcagtgg gaacgatgccctcattcagcatttgcatggt ttgttgaaaaccggacatggcactccagtcg ccttcccgttccgctatcggctgaatttgat tgcgagtgagatatttatgccagccagccag acgcagacgcgccgagacagaacttaatggg cccgctaacagcgcgatttgctggtgaccca atgcgaccagatgctccacgcccagtcgcgt accgtcttcatgggagaaaataatactgttg atgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccac agcaatggcatcctggtcatccagcggatag ttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttc gacgccgcttcgttctaccatcgacaccacc acgctggcacccagttgatcggcgcgagatt taatcgccgcgacaatttgcgacggcgcgtg cagggccagactggaggtggcaacgccaatc agcaacgactgtttgcccgccagttgttgtg ccacgcggttgggaatgtaattcagctccgc catcgccgcttccactttttcccgcgttttc gcagaaacgtggctggcctggttcaccacgc gggaaacggtctgataagagacaccggcata ctctgcgacatcgtataacgttactggtttc acattcaccaccctgaattgactctcttccg ggcgctatcatgccataccgcgaaaggtttt gcgccattcgatggtgtccgggatctcgacg ctctcccttatgcgactcctgcattaggaag cagcccagtagtaggttgaggccgttgagca ccgccgccgcaaggaatggtgcatgcaagga gatggcgcccaacagtcccccggccacgggg cctgccaccatacccacgccgaaacaagcgc tcatgagcccgaagtggcgagcccgatcttc cccatcggtgatgtcggcgatataggcgcca gcaaccgcacctgtggcgccggtgatgccgg ccacgatgcgtccggcgtagaggatcgagat cgatctcgatcccgcgaaattaatacgactc actata | |
| SNAP-25b (1–206, all C to A; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcc agggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGCCGA GGACGCAGACATGCGCAATG AGCTGGAGGAGATGCAGAGG AGGGCTGACCAGCTGGCTGA TGAGTCCCTGGAAAGCACCC GTCGCATGCTGCAGCTGGTT GAAGAGAGTAAAGATGCTGG CATCAGGACTTTGGTTATGTT GGATGAGCAAGGCGAACAAC TGGAACGCATTGAGGAAGGG ATGGACCAAATCAATAAGGAC ATGAAAGAAGCAGAAAAGAAT TTGACGGACCTAGGAAAATTC GCCGGCCTTGCCGTGGCCCC CGCCAACAAGCTTAAATCCAG TGATGCTTACAAAAAAGCCTG GGGCAATAATCAGGATGGAGT AGTGGCCAGCCAGCCTGCCC GTGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTGGC TTCATCCGCAGGGTAACAAAT GATGCCCGGGAAAATGAGATG GATGAGAACCTGGAGCAGGT GAGCGGCATCATCGGAAACCT CCGCCACATGGCTCTAGACAT GGGCAATGAGATTGACACCCA GAATCGCCAGATCGACAGGAT CATGGAGAAGGCTGATTCCAA CAAAACCAGAATTGATGAAGC CAACCAACGTGCAACAAAGAT GCTGGGAAGTGGTTAA ctcgagcaccaccaccaccaccactgag atccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgagc aataactagcataaccccttggggcctc taaacgggtcttgaggggttttttgctgaa aggaggaactatatccggat | |
| Materials for protein purificaiton | |||
| 2-Mercaptoethanol | SIGMA | M3148-25ML | |
| Agar | LPS Solution | AGA500 | |
| Ampicillin, Sodium salt | PLS | AC1043-005-00 | |
| Chloramphenicol | PLS | CR1023-050-00 | |
| Competent cells (E. coli) | Novagen | 70956 | Rosetta(DE3)pLysS |
| Glycerol | SIGMA | G5516-500ML | |
| HEPES | SIGMA | H4034-100G | |
| Hydrochloric acid / HCl | SIGMA | 320331-500ML | |
| Imidazole | SIGMA | I2399-100G | |
| Isopropyl β-D-1-thiogalactopyranoside / IPTG | SIGMA | 10724815001 | |
| Kanamycin Sulfate | PLS | KC1001-005-02 | |
| Luria-Bertani (LB) Broth | LPS Solution | LB-05 | |
| Ni-NTA resin | Qiagen | 30210 | |
| PD MiniTrap G-25 (desalting column) | Cytiva | GE28-9180-07 | For instructions, see: https://www.cytivalifesciences.com/en/us/shop/chromatography/prepacked-columns/desalting-and-buffer-exchange/pd-minitrap-desalting-columns-with-sephadex-g-25-resin-p-06174 |
| Phenylmethylsulfonyl fluoride / PMSF | ThermoFisher Scientific | 36978 | |
| Plasmids for SNARE proteins | cloned in house | N/A | Available upon request |
| Protease inhibitor cocktail | genDEPOT | P3100 | |
| Sodium chloride | SIGMA | S5886-500G | |
| Sodium phosphate dibasic / Na2HPO4 | SIGMA | S7907-100G | |
| Sodium phosphate monobasic / NaH2PO4 | SIGMA | S3139-250G | |
| Tris(2-carboxyethyl)phosphine / TCEP | SIGMA | C4706-2G | |
| Trizma base | SIGMA | T1503-250G | |
| Materials for sample assembly | |||
| Biotin-PEG-SVA | LAYSAN BIO | BIO-PEG-SVA-5K-100MG & MPEG-SVA-5K-1g | For PEGylation |
| Dibenzocyclooctyne-amine / DBCO-NH2 | SIGMA | 761540-10MG | For bead coating |
| Double-sided tape | 3M | 136 | For flow cell assembly |
| Epoxy glue | DEVCON | S-208 | For flow cell assembly |
| Glass coverslip for bottom surface | VWR | 48393-251 | Rectangular, 60×24 mm, #1.5 |
| Glass coverslip for top surface | VWR | 48393-241 | Rectangular, 50×24 mm, #1.5 |
| Magnetic bead | ThermoFisher Scientific | 14301 | Dynabeads M-270 Epoxy, 2.8 μm |
| mPEG-SVA | LAYSAN BIO | mPEG-SVA 1g | For PEGylation |
| N,N-Dimethylformamide / DMF | SIGMA | D4551-250ML | For bead coating |
| N-[3-(trimethoxysilyl)propyl]ethylenediamine | SIGMA | 104884-100ML | For PEGylation |
| Neutravidin | ThermoFisher Scientific | 31000 | For sample tethering |
| Phosphate buffered saline / PBS, pH 7.2 | PLS | PR2007-100-00 | |
| Plastic syringe | Norm-ject | A5 | 5 ml, luer tip |
| Polyethylene Tubing | SCI | BB31695-PE/4 | PE-60 |
| Reference bead | SPHEROTECH | SVP-30-5 | Streptavidin-coated Polystyrene Particles; 3.0-3.4 µm |
| Syringe needle | Kovax | 21G-1 1/4'' | 21 G |
| Syringe pump | KD SCIENTIFIC | 788210 | |
| Equipment for magnetic tweezer instrument | |||
| 1-axis motorized microtranslation stage | PI | M-126.PD1 | For vertical positioning of magnets |
| 2-axis manual translation stage | ST1 | LEE400 | For alignment of magnets to the optical axis |
| Acrylic holder for magnets | DaiKwang Precision | custum order | Drawing available upon request |
| Frame grabber | Active Silicon | AS-FBD-4XCXP6-2PE8 | |
| High-speed CMOS camera | Mikrotron | EoSens 3CXP | |
| Inverted microscope | Olympus | IX73P2F-1-2 | |
| Neodymium magnets | LG magnet | ND 10x10x12t | Dimension: 10 mm × 10 mm × 12 mm; two needed |
| Objective lens | Olympus | UPLXAPO100XO | Oil-immersion, NA 1.45 |
| Objective lens nanopositioner | Mad City Labs | Nano-F100S | |
| Rotation stepper motor | AUTONICS | A3K-S545W | For rotating magnets |
| Superluminescent diode | QPHOTONICS | QSDM-680-2 | 680 nm |
| Software | |||
| LabVIEW | National Instruments | v20.0f1 | |
| MATLAB | MathWorks | v2021a |
References
- Le, S., Liu, R., Lim, C. T., Yan, J. Uncovering mechanosensing mechanisms at the single protein level using magnetic tweezers. Methods. 94, 13-18 (2016).
- Choi, H. -. K., Kim, H. G., Shon, M. J., Yoon, T. -. Y. High-resolution single-molecule magnetic tweezers. Annual Review of Biochemistry. 91 (1), 33-59 (2022).
- Yang, T., Park, C., Rah, S. -. H., Shon, M. J. Nano-precision tweezers for mechanosensitive proteins and beyond. Molecules and Cells. 45 (1), 16-25 (2022).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- De Vlaminck, I., Dekker, C. Recent advances in magnetic tweezers. Annual Review of Biophysics. 41 (1), 453-472 (2012).
- Bustamante, C. J., Chemla, Y. R., Liu, S., Wang, M. D. Optical tweezers in single-molecule biophysics. Nature Reviews Methods Primers. 1, 25 (2021).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical Journal. 82 (6), 3314-3329 (2002).
- Smith, S. B., Finzi, L., Bustamante, C. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258 (5085), 1122-1126 (1992).
- Lansdorp, B. M., Tabrizi, S. J., Dittmore, A., Saleh, O. A. A high-speed magnetic tweezer beyond 10,000 frames per second. Review of Scientific Instruments. 84 (4), 044301 (2013).
- Cnossen, J. P., Dulin, D., Dekker, N. H. An optimized software framework for real-time, high-throughput tracking of spherical beads. Review of Scientific Instruments. 85 (10), 103712 (2014).
- Dulin, D., et al. High spatiotemporal-resolution magnetic tweezers: calibration and applications for DNA dynamics. Biophysical Journal. 109 (10), 2113-2125 (2015).
- Huhle, A., et al. Camera-based three-dimensional real-time particle tracking at kHz rates and Ångström accuracy. Nature Communications. 6 (1), 5885 (2015).
- Popa, I., et al. A HaloTag anchored ruler for week-long studies of protein dynamics. Journal of the American Chemical Society. 138 (33), 10546-10553 (2016).
- Shon, M. J., Kim, H., Yoon, T. -. Y. Focused clamping of a single neuronal SNARE complex by complexin under high mechanical tension. Nature Communications. 9 (1), 3639 (2018).
- Tapia-Rojo, R., Eckels, E. C., Fernández, J. M. Ephemeral states in protein folding under force captured with a magnetic tweezers design. Proceedings of the National Academy of Sciences. 116 (16), 7873-7878 (2019).
- Löf, A., et al. Multiplexed protein force spectroscopy reveals equilibrium protein folding dynamics and the low-force response of von Willebrand factor. Proceedings of the National Academy of Sciences. 116 (38), 18798-18807 (2019).
- Tapia-Rojo, R., Alonso-Caballero, A., Fernandez, J. M. Direct observation of a coil-to-helix contraction triggered by vinculin binding to talin. Science Advances. 6 (21), (2020).
- Rieu, M., et al. Parallel, linear, and subnanometric 3D tracking of microparticles with Stereo Darkfield Interferometry. Science Advances. 7 (6), (2021).
- Rieu, M., Valle-Orero, J., Ducos, B., Allemand, J. -. F., Croquette, V. Single-molecule kinetic locking allows fluorescence-free quantification of protein/nucleic-acid binding. Communications Biology. 4 (1), 1083 (2021).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences. 103 (16), 6190-6195 (2006).
- Camunas-Soler, J., Ribezzi-Crivellari, M., Ritort, F. Elastic properties of nucleic acids by single-molecule force spectroscopy. Annual Review of Biophysics. 45 (1), 65-84 (2016).
- Südhof, T. C., Rothman, J. E. Membrane fusion: grappling with SNARE and SM proteins. Science. 323 (5913), 474-477 (2009).
- Gao, Y., et al. Single reconstituted neuronal SNARE complexes zipper in three distinct stages. Science. 337 (6100), 1340-1343 (2012).
- Zorman, S., et al. Common intermediates and kinetics, but different energetics, in the assembly of SNARE proteins. eLife. 3, e03348 (2014).
- Zhang, Y., Hughson, F. M. Chaperoning SNARE folding and assembly. Annual Review of Biochemistry. 90 (1), 581-603 (2021).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H. Magnetic tweezers for single-molecule experiments. Handbook of Single-Molecule Biophysics. , 371-395 (2009).
- You, H., Le, S., Chen, H., Qin, L., Yan, J. Single-molecule manipulation of G-quadruplexes by magnetic tweezers. Journal of Visualized Experiments. (127), e56328 (2017).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical Journal. 96 (12), 5040-5049 (2009).
- Dulin, D., Barland, S., Hachair, X., Pedaci, F. Efficient illumination for microsecond tracking microscopy. PLoS One. 9 (9), e107335 (2014).
- Klaue, D., Seidel, R. Torsional stiffness of single superparamagnetic microspheres in an external magnetic field. Physical Review Letters. 102 (2), 028302 (2009).
- Shon, M. J., Rah, S. -. H., Yoon, T. -. Y. Submicrometer elasticity of double-stranded DNA revealed by precision force-extension measurements with magnetic tweezers. Science Advances. 5 (6), 1697 (2019).
- Czerwinski, F., Richardson, A. C., Oddershede, L. B. Quantifying noise in optical tweezers by Allan variance. Optics Express. 17 (15), 13255-13269 (2009).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. Review of Scientific Instruments. 83 (2), 025115 (2012).
- Ostrofet, E., Papini, F. S., Dulin, D. High spatiotemporal resolution data from a custom magnetic tweezers instrument. Data in Brief. 30, 105397 (2020).
- Yu, Z., et al. A force calibration standard for magnetic tweezers. Review of Scientific Instruments. 85 (12), 123114 (2014).
- Strick, T. R., Allemand, J. -. F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271 (5257), 1835-1837 (1996).
- Daldrop, P., Brutzer, H., Huhle, A., Kauert, D. J., Seidel, R. Extending the range for force calibration in magnetic tweezers. Biophysical Journal. 108 (10), 2550-2561 (2015).
- te Velthuis, A. J. W., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative guidelines for force calibration through spectral analysis of magnetic tweezers data. Biophysical Journal. 99 (4), 1292-1302 (2010).
- Ostrofet, E., Papini, F. S., Dulin, D. Correction-free force calibration for magnetic tweezers experiments. Scientific Reports. 8 (1), 15920 (2018).
- Seol, Y., Li, J., Nelson, P. C., Perkins, T. T., Betterton, M. D. Elasticity of short DNA molecules: theory and experiment for contour lengths of 0.6-7 µm. Biophysical Journal. 93 (12), 4360-4373 (2007).
- Burnham, D. R., Vlaminck, I. D., Henighan, T., Dekker, C. Skewed Brownian fluctuations in single-molecule magnetic tweezers. PLoS One. 9 (9), 108271 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protocols. 3 (1), 101152 (2022).
- Lee, H. -. W., et al. Profiling of protein-protein interactions via single-molecule techniques predicts the dependence of cancers on growth-factor receptors. Nature Biomedical Engineering. 2 (4), 239-253 (2018).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Parthasarathy, R. Rapid, accurate particle tracking by calculation of radial symmetry centers. Nature Methods. 9 (7), 724-726 (2012).
- Woodside, M. T., Block, S. M. Reconstructing folding energy landscapes by single-molecule force spectroscopy. Annual Review of Biophysics. 43 (1), 19-39 (2014).
- Evans, E., Ritchie, K. Dynamic strength of molecular adhesion bonds. Biophysical Journal. 72 (4), 1541-1555 (1997).
- Zhang, Y. Energetics, kinetics, and pathway of SNARE folding and assembly revealed by optical tweezers. Protein Science. 26 (7), 1252-1265 (2017).
- Chen, H., et al. Improved high-force magnetic tweezers for stretching and refolding of proteins and short DNA. Biophysical Journal. 100 (2), 517-523 (2011).
- Cho, S., et al. Tension exerted on cells by magnetic nanoparticles regulates differentiation of human mesenchymal stem cells. Biomaterials Advances. 139, 213028 (2022).
- Shon, M. J., Cohen, A. E. Nano-mechanical measurements of protein-DNA interactions with a silicon nitride pulley. Nucleic Acids Research. 44 (1), 7 (2016).
- Cheng, Y. Single-particle cryo-EM-How did it get here and where will it go. Science. 361 (6405), 876-880 (2018).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Neupane, K., et al. Direct observation of transition paths during the folding of proteins and nucleic acids. Science. 352 (6282), 239-242 (2016).
- Choi, H. -. K., et al. Watching helical membrane proteins fold reveals a common N-to-C-terminal folding pathway. Science. 366 (6469), 1150-1156 (2019).
- Kim, C., et al. Extreme parsimony in ATP consumption by 20S complexes in the global disassembly of single SNARE complexes. Nature Communications. 12 (1), 3206 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved