需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
高速磁镊,用于力敏元件的纳米力学测量
* 这些作者具有相同的贡献
摘要
在这里,我们描述了一种高速磁镊装置,该装置以最大1.2 kHz的速率对力敏生物分子进行纳米力学测量。我们将它作为模型系统介绍其在DNA发夹和SNARE复合物中的应用,但它也适用于参与机械生物学事件的其他分子。
摘要
单分子磁镊(MTs)已成为有力询问生物分子(如核酸和蛋白质)的强大工具,因此有望在机械生物学领域发挥作用。由于该方法通常依赖于基于图像的磁珠跟踪,因此记录和分析图像的速度限制以及磁珠的热波动长期以来一直阻碍其在观察目标分子中微小而快速的结构变化中的应用。本文介绍了高分辨率MT装置的构建和操作的详细方法,该装置可以解析生物分子及其复合物的纳米级毫秒动力学。作为应用示例,展示了DNA发夹和SNARE复合物(膜融合机制)的实验,重点是如何在存在皮克顿级力的情况下检测它们的瞬态和转变。我们预计高速MT将继续对在细胞中感知,传输和产生力的分子进行高精度纳米力学测量,从而加深我们对机械生物学的分子水平的理解。
引言
细胞主动感知并响应机械刺激。在这样做的过程中,许多生物分子显示出力依赖性特性,从而实现动态结构变化。广为人知的例子包括机械敏感离子通道和细胞骨架元件,它们为细胞提供来自周围环境的关键机械信息。
此外,具有独特承载力性质的分子也可以被认为是更广泛意义上的机械敏感分子。例如,核酸双链体的局部形成和熔解,以及G-四链体等高阶结构,在复制、转录、重组以及最近的基因组编辑中起着至关重要的作用。此外,参与突触通讯的一些神经元蛋白质通过产生超过典型分子间相互作用水平的物理力来执行其功能。无论研究哪个例子,以高时空精度研究所涉及的生物分子的纳米力学将被证明在揭示相关机械生物学过程的分子机制方面非常有用1,2,3。
单分子力谱方法已成为检查生物分子2,4,5,6力学性能的有力工具。它们可以在施加力的同时监测核酸和蛋白质的结构变化,从而检查力依赖性。两种众所周知的装置是光学镊子和磁镊(MTs),它们使用微米大小的珠子来操纵分子5,6,7,8。在这些平台中,聚苯乙烯(用于光学镊子)或磁珠(用于MTs)通过分子"手柄"拴在靶分子(例如核酸和蛋白质)上,通常由双链DNA(dsDNA)的短片段组成。然后移动磁珠以施加力并成像以跟踪其位置,从而报告目标分子的结构变化。光学和磁性镊子在其应用中在很大程度上可以互换,但它们控制力的方法存在重要差异。光镊本质上是将磁珠捕获到位的位置夹具,因此当目标构建体发生形状变化时,施加的力会波动;伸展增加,例如展开,松开系绳并降低张力,反之亦然。虽然可以实现主动反馈来控制光镊中的力,但相比之下,MT自然地作为力钳装置运行,利用永磁体稳定的远场磁力,也可以承受环境扰动。
尽管MT历史悠久,设计简单,但在高精度测量方面的应用仍落后于光镊,这主要是因为快速磁珠跟踪的技术挑战。然而,最近,几个小组共同领导了MT仪器2,9,10,11,12,13,14,15,16,17,18,19的硬件和软件的多方面改进。.在这项工作中,我们介绍了一个以1.2 kHz运行的这种设置的示例,并描述了如何使用它对力敏感的生物分子进行纳米力学测量。作为模型系统,我们使用DNA发夹和神经元SNARE复合物,并检查它们在皮科顿体系中的快速结构变化。DNA发夹在明确定义的力范围内表现出简单的双态跃迁20,21,因此可作为玩具模型来验证镊子设置的性能。由于SNARE蛋白组装成驱动膜融合的力敏感复合物22,因此单分子力谱14,23,24,25也对它们进行了广泛的研究。介绍了分析数据和提取热力学和动力学有用信息的标准方法。我们希望本文能够促进高精度MT在机械生物学研究中的采用,并激励读者探索自己感兴趣的力敏感系统。
研究方案
本协议中描述的所有材料和设备都列在 材料表中。用于 操作 下面 所述 的 高速 机器 翻译 设置 的 LabVIEW 软件, 以及 用于 分析 样本 数据 的 MATLAB 脚本 存放 在 GitHub (https://github.com/ShonLab/Magnetic-Tweezers) 上 并 公开 提供。
1. 设备建设
注意:高速 MT 结构的一般原理与标准的传统 MT 系统相似,但使用高速互补金属氧化物半导体 (CMOS) 相机和高功率相干光源除外(图 1)。有关标准MT仪器5,26,27的更多说明,请参阅其他来源。
- 在防振光学台上设置倒置显微镜。安装高速CMOS相机和图像采集卡。
- 构建一个用于在 3D 中操纵磁铁的平移阶段。将电动线性载物台(行程>20 mm)垂直安装在手动XY载物台的顶部。
注意: 垂直运动控制力,而XY平台用于手动将磁铁对准光轴,以进行初始设置构建。 - 安装旋转步进电机以及用于旋转磁铁的皮带和皮带轮系统。
注意: 皮带在电机轴和相距几厘米的磁铁之间传递旋转运动。磁铁的旋转是平移操作的内部。 - 安装磁铁。使用亚克力支架(从制造公司订购;见 补充图S1),它可以并联两个相同的磁铁,磁体之间有明确的1毫米间隙(图1B)。为了利用给定的一对磁体可获得的最大力,请调整平移台的垂直位置,以便在将磁体移动到最低位置时磁体的底面与样品平面对齐。
注:有关磁铁28的支架设计和配置的更多信息,请参阅Lipfert等人。磁体 的 高度 和 方向 由 LabVIEW 软件 结合 数据 采集 进行 控制。 - 使用低放大倍率物镜观察,将磁体对准视场中心。检查旋转磁体是否不会导致磁体对中心的大位移。
注意:如果磁铁之间的中点绕旋转轴旋转,则由于支架不完美,磁铁很可能偏离中心。相对于间隙尺寸的较小程度的不对中是可以容忍的,因为磁体旋转仅用于检查系绳和在特定应用中施加扭矩。 - 安装超发光二极管(SLD)用于照亮珠子。将光束穿过两个磁铁之间的 1 mm 间隙。确保光束正确准直以适合间隙,并且照明不会被磁铁遮挡。
- 在物镜转盘上安装压电透镜扫描仪,并安装100倍油浸物镜(数值孔径[NA]:1.45)进行磁珠跟踪。为避免跟踪结果中出现潜在的伪影,请确保在移动磁体时保持均匀的照明。最后,在不饱和像素的情况下将光照水平调整到最大亮度。
注意:有关用于高速跟踪珠子的不同光源的比较,请参阅Dulin等人29。
2. 磁力校准
- 使用聚合酶链反应(PCR;见 表1),制备5 kbp dsDNA片段(使用引物B、引物Z_5k和λ-DNA),一端用生物素标记(用于表面附着),另一端用叠氮化物标记(用于磁珠附着)。
- 在第 6 节之后,用 5 kbp 分子制备流通池。
- 在第 7 节之后,通过验证其延伸和旋转来确定良好的珠系结构。特别是,确保选择旋转轨迹最小(即半径为<200 nm)的磁珠,以最大程度地减少由于偏心附件30,31引起的磁珠高度偏移。一旦确定了良好的系绳,就开始磁珠跟踪,请参阅第 9 节。
- 如果设置是新的,请表征其噪声和稳定性,以实现可靠的高分辨率测量。将磁体放置在距离流通池表面~3 mm的位置(施加>10 pN并抑制磁珠的布朗运动),在1.2 kHz处跟踪磁珠的 z位置,并计算来自 z坐标时间序列32,33 的艾伦偏差(AD)(图2C)。检查在高速状态(<0.1 s)下是否可以达到几纳米的AD值,以及差分跟踪(磁珠相对于参考磁珠的位置)在较长的时间尺度上降低AD。
注意:我们通常以最大速率(1.2 kHz或0.83 ms分辨率)获得<3 nm的AD,并且AD至少持续下降10 s,这意味着漂移最小。其他人在类似的设置9,10,11,12,34上报告了类似的值。 - 当磁铁处于静止位置 (F ~ 0 pN) 时,以 1.2 kHz 记录系留磁珠的 x 和 y 坐标。记录足够长的时间段(即,足够长于波动35 的特征弛豫时间)的位置,以便对布朗运动进行充分采样。
注意:这里, x方向是沿着磁场的方向,而 y 中的运动表示垂直于磁场的横向运动。 - 将磁体移近流通池并重复磁珠位置测量,直到磁体轻轻接触流通池的顶部。当磁体距离样品平面超过 7 毫米时(因为施加的力在磁体的远场中缓慢增加),以较大的步长(例如,1-2 mm)移动,但逐渐减小步长(例如,0.1-0.5 mm),因为它们接近更近,以便在更高的力水平下进行更精细的校准(图 2B)。
- 使用两种替代方法中的任何一种计算每个磁体位置 d 处的力(提供了包含两种方法的 MATLAB 脚本"力校准.m";请参阅 补充文件 1)。
- 测量磁珠的y坐标
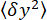 (图2D)和磁珠相对于最低位置的平均z位置
(图2D)和磁珠相对于最低位置的平均z位置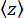 (图2B,底部)的方差。然后,使用公式(1)7,27,36估计力(固定磁珠半径R = 1,400 nm,热能kRT = 4.11 pN∙nm):
(图2B,底部)的方差。然后,使用公式(1)7,27,36估计力(固定磁珠半径R = 1,400 nm,热能kRT = 4.11 pN∙nm):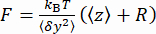 (1)
(1) - 或者,计算y坐标Sy的功率谱密度(PSD)(图2E)。通过使用方程 (2) 将双洛伦兹模型37 拟合到测量的 Sy 来确定施加的力 F。
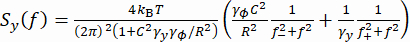 (二)
(二)
这里,R是磁珠半径,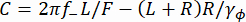 γy和γφ分别是平移和旋转阻力系数(根据斯托克斯-爱因斯坦方程估计),kRT是热能,f+和f-是使用方程(3)获得的两个特征频率。
γy和γφ分别是平移和旋转阻力系数(根据斯托克斯-爱因斯坦方程估计),kRT是热能,f+和f-是使用方程(3)获得的两个特征频率。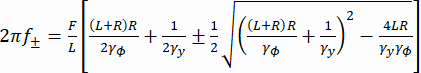 (三)
(三)
注意:由于系绳扩展 L 是遵循完善的蠕虫状链 (WLC) 模型的力函数,因此上述表达式将 F 作为唯一的拟合参数(为简单起见,我们将 R 固定为 1,400 nm,因为它在所有力水平上共享,并且确切的值不会明显影响结果)。必要时,必须考虑基于相机的图像采集产生的运动模糊和混叠38,39,但在我们使用 5 kbp 系绳进行 1 kHz 以上的高速测量时,这种影响可以忽略不计。
- 测量磁珠的y坐标
- 重复步骤 2.4-2.7 以进行更多构造。探测三到五个不同的磁珠,以平均磁珠之间的力变化。
注意:应考虑使用中的磁珠之间的力变化,以确定用于平均的适当结构数量。这种可变性很小,但可能导致测量力的误差超过1 pN,即使对于商业产品也是如此31。对于大多数应用,其中所涉及的力的绝对测定并不重要,平均三到五个磁珠的校准结果通常就足够了。解释这种变化的另一种方法是在实验开始时用单个系绳测量力,这可能很耗时。另一种选择是嵌入发夹结构,在每个构造31 中以已知的力水平解压缩。 - 将测量的力绘制为磁体距离的函数,并使用公式(4)将双指数函数拟合到数据(图2F)。
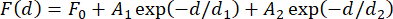 (四)
(四)
这里,F0(基线)、A 1 和 A 2(振幅)以及 d 1 和 d2(衰减常数)是拟合参数。确保两种方法的力值以及由此产生的双指数拟合基本一致(图 2F,G)。
注意: 要确认力校准是否正确进行,请通过绘制延伸与测量力的曲线来验证探测结构的力-伸展关系。 - 为了校正磁珠30,31 的力相关倾斜导致的磁珠高度偏移 z关闭,使用公式 (5) 考虑具有磁珠半径的偏心系绳的几何形状,估计横向偏移 x off 的 zoff,并将这些值应用于测量的延伸值。此步骤在 MATLAB 脚本"force calibration.m"(第 252-254 行)中实现。
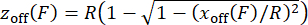 (五)
(五)
注意:尽管此校正对延伸进行了微小的更改,特别是对于旋转半径小(<200 nm)的磁珠,但这种偏移通常会严重影响弹性响应,如图 2H 到 图2I30,31的变化所示。 - 通过使用公式 (6) 将可扩展 WLC 模型拟合到数据来检查持久性长度 Lp。
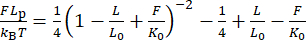 (六)
(六)
这里,L 0 是轮廓长度(5 kbp 为 1.7 μm),K0 是焓拉伸的模量。
注意:尽管在磷酸盐缓冲盐水(PBS)等典型缓冲液中,dsDNA的L p被普遍认为为40-50 nm,但应用于短分子(<5 kbp)的WLC公式系统地低估了L p,因为L0降低31,40。这是因为经典的WLC模型假定聚合物的链长足以长于其持久性长度。在这里,我们获得了 5 kbp 构建体的 Lp = 40 ± 3 nm(图 2H),并且延伸校正进一步产生了 1,100 ± 200 pN 的均匀 K0(图 2I)。应用有限 WLC 模型 31,40,以及对扩展分布41 中的非高斯性进行校正,将略微增加 Lp。 - 验证力校准后,将双指数模型获得的拟合参数应用于提供的LabVIEW软件(补充文件2),并等待软件根据电机读数(即磁体位置)实时计算电流。由于没有反函数 d(F) 的分析表达式,因此通过对 d 目标力水平的数值估计,以 0.1 pN 步长准备 d 与 F 的查找表。将此表也存储在软件中以命令力控制。
3. DNA发夹的合成
注意:用于MT实验的DNA发夹构建体是通过用两个定制引物对λ-DNA中的510 bp区域进行PCR扩增来制备的,其中一个引物在其5'末端包含发夹结构(图3A)。通过这种方式,将发夹基序放置在PCR产物的一端。
- 准备引物。
- 正向引物:引物B_hp,5′-生物素标记,用于玻璃表面附着并与λ-DNA结合。该引物包含一个发夹基序,具有 8 bp 茎和一个 6 nt 环,5′ 到 λ 结合区。
- 反向引物:引物Z_hp,5′-叠氮化物标记用于磁珠附着,并与距离正向引物1 kbp的λ-DNA结合。
- 使用λ-DNA(模板)、nTaq聚合酶和标准PCR条件设置并运行PCR(见 表1)。使用商业纯化试剂盒清理产品。
- 通过260nm(A260)的紫外吸收测量DNA浓度,并进行琼脂糖凝胶电泳(2%凝胶)(见 表2)以验证产品大小。典型产率为 ~35 μL ~600 nM 溶液。
4. SNARE蛋白的制备
注意:神经元SNARE复合物通过结合从大 肠杆菌表达的三种纯化大鼠蛋白来组装:VAMP2 / synaptobrevin-2,syntaxin-1A和SNAP-25(图3B)。为了促进它们的组装,将句法蛋白和SNAP-25与VAMP2片段(缺乏N末端区域;称为"ΔN-VAMP2")共表达成称为"ΔN-复合物"的结构,然后在DNA处理附着后与全长VAMP2混合以形成完整的复合物。
- 制备含有cDNA的质粒,用于表达SNARE蛋白(所有质粒的DNA序列在 材料表中给出)。
- 制备缺乏跨膜结构域的6×His标记的VAMP2(2-97;L32C/I97C用于二硫键)克隆到pET28a载体中。
- 制备缺乏Habc和跨膜结构域(191-267,I202C / I266C取代二硫键)的语法素-1A与6×His标记的ΔN-VAMP2(49-96)克隆到pETDuet-1载体中。
- 准备克隆到 pET28a 载体中的全长 SNAP-25 亚型 b(2-206,所有 C 到 A)。这将用于制备ΔN配合物。
- 准备克隆到pET28a载体中的6×His标记的全长SNAP-25亚型b(1-206,所有C至A),以直接添加到MT测定缓冲液中,以在展开后重新组装SNARE复合物。
- 准备两管罗塞塔(DE3) 大肠杆菌 细胞。用VAMP2质粒(来自步骤4.1.1)转化一组,一组同时使用语法蛋白-1A / ΔN-VAMP2和未标记的SNAP-25质粒(来自步骤4.1.2和4.1.3)进行转化以表达ΔN复合物,另一组使用His标记的SNAP-25质粒(来自步骤4.1.4)。
- 将转化的细胞转移到含有适当抗生素的Luria-Bertani肉汤(LB)中(此处为卡那霉素和氯霉素用于VAMP2和His标记的SNAP-25;卡那霉素,氯霉素和氨苄西林用于ΔN复合物)。在振荡培养箱(220rpm)中在37°C下培养它们,直到肉汤的光密度(OD)达到0.7-0.9。
- 加入1mM异丙基β-d-1-硫代吡喃半乳糖苷(IPTG)诱导蛋白表达,并在振荡培养箱(220rpm)中于37°C孵育细胞3-4小时。
- 通过在4°C下以4,500× g 离心培养物15分钟来沉淀细胞。
- 制备用于蛋白质纯化的缓冲液(见 表2)。
- 将表达SNARE的细胞沉淀悬浮在40mL冰冷的裂解缓冲液中,并通过在冰上超声处理(15%振幅,5秒开启和5秒关闭,总共30分钟)裂解细胞。
- 将裂解物在4°C下以15,000× g 离心30分钟以除去不溶性物质。
- 将上清液通过装有 1 mL Ni-NTA 树脂的重力柱。先用洗涤缓冲液A洗涤树脂,然后用洗涤缓冲液B洗涤树脂,并用10 mL洗脱缓冲液洗脱蛋白质。
- 使用脱盐柱从洗脱液中去除三(2-羧乙基)膦(TCEP)和咪唑(按照制造商的说明)。用PBS洗脱样品。
- 用离心过滤器(10 kDa截止值)将蛋白质浓缩至~70μM,同时将蛋白质保持在PBS中(通常产生2mL)。通过 280 nm (A280) 处的紫外线 (UV) 吸收或通过布拉德福德测定法测量蛋白质浓度。
- 准备小等分试样,在液氮中快速冷冻,并在-80°C下储存直至使用。
注意:完整的SNARE复合物将在DNA手柄上偶联ΔN复合物后组装(见下文)。
5. DNA手柄的附着
注意:首先通过PCR制备一端含有伯胺基团的两个510 bp dsDNA手柄,然后使用双功能交联剂SM(PEG)2将胺基团转化为马来酰亚胺基团。然后,两个手柄通过其半胱氨酸基团 共 价连接到SNARE复合物,以进行位点特异性偶联(图3B)。
- 准备引物。
- 准备正向引物: 引物B(用于扩增手柄B),其5′-生物素标记用于玻璃表面附着并与λ-DNA结合;引物Z(用于扩增手柄Z),5′-叠氮化物标记,用于磁珠附着,与引物B具有相同的序列。
- 准备反向引物:引物N(与手柄B和手柄Z共享),其5′-胺标记用于蛋白质偶联,并与远离正向 引物的λ-DNA 510 bp结合。
- 使用λ-DNA(模板)、nTaq聚合酶和标准PCR条件设置并运行两组PCR反应(每个处理18管200μL反应)(见 表1)。用PCR净化试剂盒净化产物,并用45 μL超纯水洗脱每个手柄。使用最小体积的水来获得高浓度的手柄,以便在后续步骤中进行有效反应。
- 通过A260测量DNA浓度。每个手柄的典型产量为 ~650 μL ~2 μM 溶液。将小样品分开,以便以后在琼脂糖凝胶电泳中验证。
- 用 5 mM SM(PEG)反应每个手柄(PBS 中的 1 μM)2。在室温下轻轻旋转孵育。1小时后,使用DNA纯化试剂盒去除未反应的SM(PEG)2。用 250 μL PBS 洗脱每个手柄以获得 ~2 μM 溶液。
- 在PBS中以1:16的摩尔比混合处理B和ΔN复合物的溶液(例如,1μM手柄B和16μM ΔN复合物),并在室温下搅拌孵育2小时。分开一个小样品进行琼脂糖凝胶电泳。
- 在上一步中使用的ΔN复合物上添加2.5倍摩尔过量的VAMP2溶液。将混合物在室温下搅拌再孵育1小时。在此步骤中组装完整的SNARE复合体。
- 通过用新鲜PBS和离心过滤器(100kDa截止值)交换缓冲液去除游离蛋白:在4°C下以14,000× g 离心5分钟,重复至少6x,并运行15分钟进行最后一次旋转。测量 A260/A280 比率的增加以监测游离蛋白的去除。分开一个小样品进行琼脂糖凝胶电泳。
- 将手柄Z以超过手柄B的15倍摩尔浓度添加到溶液中,保持手柄Z的浓度至少高于1μM以促进反应。搅拌将混合物在4°C下孵育过夜。
- 通过琼脂糖凝胶电泳验证中间体(处理B及其蛋白质偶联物)和最终产物(具有两个手柄的SNARE复合物)(图3B,插图)(见 表2)。
注意:如果蛋白质成功附着到手柄B上,将检测到迁移率偏移。特别是,DNA手柄上完整SNARE复合物的形成可以通过它们对十二烷基硫酸钠(SDS)的抗性来确认,这与ΔN复合物不同,后者在SDS中被分解并仅留下与DNA结合的合成蛋白(比较 图3B中的b和c)。 - 准备小等分试样,在液氮中快速冷冻,并在-80°C下储存直至使用。
注意:尽管最终溶液包含未反应的手柄,但在流通池中的样品组装过程中,只会选择用生物素和叠氮化物双重标记的所需构建体。
6. 流通池的制造
注意:用于MT测量的流通池由两个玻璃盖玻片制成,通过双面胶带粘合在一起(图3C)。一个盖玻片涂有PEG和生物素化聚乙二醇(PEG)的混合物,以避免非特异性结合,并通过生物素-中性亲和素键 实现 目标分子的特异性拴系(图3D)。然后,使用注射泵将MT实验材料的溶液依次注入流通池中(图3C,D)。
- 准备两个玻璃盖玻片,顶部(24 毫米× 50 毫米,1.5 号厚度)和底部(24 毫米× 60 毫米,1.5 号厚度)表面各一张。通过在1M KOH中超声处理30分钟来清洁盖玻片。超声处理后,用蒸馏水冲洗盖玻片并保持在水中直至下一步。
- 按照公布的协议42,43对底部盖玻片进行聚乙二醇。使用 N-[3-(三甲氧基硅基)丙基]乙二胺进行硅烷化,并在 100 mM 碳酸氢盐缓冲液中使用生物素-PEG-SVA 和 mPEG-SVA 的 1:100 (ww) 混合物。将聚乙二醇化盖玻片在-20°C下保持干燥并储存数周。
- 在实验当天,取出聚乙二醇盖玻片并用氮气枪吹干。目视检查它们是否有污垢,以确保它们干净。
- 要制作样品通道,请准备~2毫米宽的双面胶带,并在底部盖玻片(聚乙二醇化表面向上)上放置四条条,彼此平行并彼此分开~5毫米(图3C)。
注意:这样,可以在单个流通池中创建三个 5 mm 宽的样品通道。 - 将顶部盖玻片放在底部盖玻片的中心,在短边缘上留出~5毫米的空间,用于通道入口和出口。用镊子轻轻按压顶部盖玻片的背面,以牢固地密封通道。
- 要制作入口储液槽,请修剪 200 μL 移液器吸头的边缘。从较宽的开口切出~10 mm,以容纳~200 μL溶液。为三个流道制作三个。要配置出口,请准备三个适合注射泵管道的注射器针头。
- 使用5分钟环氧树脂,将储液槽和针轮毂粘在流通池上。确保形成完整的密封以避免泄漏,并且通道不会被多余的胶水堵塞。让它干燥至少30分钟。
7. 珠系结构的组装
注意:使用注射泵将用于MT实验的材料溶液(包括用于珠系构建体的材料)按顺序引入流通池(图3C,D)。
- 准备磁珠。从储备溶液中取 5 mg M270-环氧珠(167.5 μL 二甲基甲酰胺中的 ~3.3 × 108 个珠子),并通过磁分离磁珠用磷酸盐缓冲液替换溶剂(参见 表 2)。
- 在磷酸盐缓冲液中制备~1.1×109 珠mL−1 的珠子,并用2 mM二苯并环辛炔(DBCO)-NH2反应。将混合物在室温下在旋转混合器上孵育3小时。反应后,用新鲜磷酸盐缓冲液洗涤珠子3倍,以除去未反应的分子。
注意:洗过的珠子可以在使用前在4°C下储存数周,无需额外旋转。 - 用聚乙烯管将流通池通道出口上的针头连接到注射泵。用PBS平衡通道。
- 通过泵抽吸,将以下溶液依次引入通道:NeutrAvidin,靶标构建体(DNA发夹或带有DNA手柄的SNARE复合物),参考聚苯乙烯珠和DBCO包被的磁珠。使用前,彻底涡旋磁珠溶液以分散潜在的珠状聚集体。
- 洗掉未结合的珠子,同时施加 0.1 pN 的力。
注意:施加较小的向上力有助于去除未结合的磁珠,并有助于避免特定结合的珠系结构破裂。 - 对于SNARE复合物的实验,在最终缓冲液中加入1.5μM SNAP-25。
注意:游离的SNAP-25分子可以在展开后重新结合SNARE复合物,并允许对单个复合物进行重复测量。
8. 目标结构的识别
- 在流通池通道的表面上,搜索由目标构建体的单个分子束缚的磁珠。确保参考珠位于附近。
- 旋转候选珠子并检查其是否自由旋转。如果珠子被多个分子束缚,它就会表现出约束运动。
- 旋转珠子几圈并找出旋转半径(此功能在提供的软件中实现)。优选地,选择旋转半径小的珠子。
注意:此半径表示磁珠偏离系绳轴的程度,这是在磁珠-系绳组件30,31 期间随机确定的。在所有实验中,磁珠的最小偏心减轻了与我们使用的高磁珠半径与系绳延伸比相关的许多伪影。 - 将力从 0 增加到 5 pN,以识别良好的单系磁珠。寻找由于拉伸 1 kbp 系线(或等效的两个 510 bp 手柄)而导致的磁珠衍射图样的巨大变化。如果衍射图案没有显著变化,请将力降低到零并扫描另一个候选珠。
注意:磁珠的~300 nm提升可以从原始图像中轻松注意到,而无需实际开始跟踪过程。
9. 用于延伸测量的磁珠跟踪
注意:通过 本文 提供的 LabVIEW 软件 中 实 时 分析 磁珠 图像 来 跟踪 磁珠。跟踪方法及其变体已用于大多数常规MT系统,并在以前的文献2,5,7,26中进行了解释。通过测量磁珠相对于固定参考磁珠的位置(即差分跟踪),位置测量对外部扰动变得非常可靠。
- 将合适的磁珠与参考磁珠一起找到后,单击" 校准 "按钮开始准备磁珠跟踪。
- 单击图像中的珠子以定义珠子的位置。然后将图像裁剪到磁珠周围的感兴趣区域(ROI)(例如,3μm磁珠为150 x 150像素),然后进一步分析以提取精确的磁珠坐标。
- 等待磁铁旋转完成。该过程记录磁珠的 x和 y坐标(通过计算2D互相关44 或使用磁珠图像的径向对称性45 ,具有相当的性能),同时旋转磁体以记录磁珠31的偏心附着。
- 对于z方向的跟踪,请等待软件生成距焦平面不同距离的珠子衍射图像的查找表。这是通过用压电扫描仪以等距步进物镜并记录每个位置的波动平均磁珠图像来实现的。然后,通过将实时磁珠图像与插值7的查找表进行比较来确定实际实验中磁珠的z坐标。
- 查找表生成完成后,启用跟踪和自动对焦(按 跟踪 ? 和 自动对焦? 按钮),然后单击" 获取 "按钮开始记录磁珠位置。
注意:自动对焦是可选的,但建议校正 采集过程中z 轴中的级漂移。
10. 强制应用方案
- 力斜坡实验: 为了验证结构的力-延伸关系,请在恒定加载速率(± 1.0 pN s−1)下施加力斜坡(图4A)。例如,以0-20-0 pN循环进行三轮,以验证结构的总长度和手柄的力伸展曲线。
- 通过在软件中指定系绳参数,在测量数据之上叠加WLC力延伸曲线,并确定目标磁珠是否被具有适当DNA手柄的真实样品构建体拴住。使用构建体的已知轮廓长度(例如,1 kbp dsDNA 的 ~340 nm)和 WLC 持久性长度(短 dsDNA31 为 30-45 nm)作为起点。如有必要,应用步骤 2.11 中所述的扩展更正方法。
- 如果构建体经过验证,请详细检查力-延伸响应,以寻找由目标分子-发夹或SNARE复合物产生的其他延伸。
- 恒力实验: 以离散步骤逐渐改变施加的力,以探测目标分子的力灵敏度(图4B)。
注意:MT可以实现简单有效的恒力实验,因为当磁铁保持静止时,施加的力保持恒定。- 对于 DNA 发夹,以 0.2-0.5 pN 步长施加 4-8 pN 的力,并在每个力水平上测量磁珠位置 ~10 秒。
- 对于SNARE复合物,以0.1-0.2 pN的步长施加14-16 pN的力,并在每个力水平上测量珠子位置~10秒。
- 力跳实验: 观察SNARE复合体的过渡事件。
注意:力跳跃实验与恒定力实验一样,涉及力水平的变化。然而,力跳跃在施加的力中采用更突然的变化,允许监测探测分子中的力触发事件,例如蛋白质复合物的突然破裂。例如,由于SNARE复合物在力循环23中表现出结构滞后,因此进行力跳跃实验并测量过渡延迟是有益的(图4C)。- 解压缩: 从完整的三元SNARE复合物中剥离VAMP2分子,留下合成蛋白-1A和SNAP-25的二元复合物。
- 重新压缩: 解压缩的VAMP2分子以再生完整的SNARE复合物。
- 展开: SNARE复合物的完全解离伴随着SNAP-25的完全解离。展开后,只有VAMP2和合成蛋白分子留在构建体中。
- 折叠: 从缓冲液中结合游离 SNAP-25 分子后再生 SNARE 复合物。
- 在 2 pN 处,通过等待(~30 秒)游离 SNAP25 分子的结合来诱导完整 SNARE 复合物的组装。在SNARE复合物形成时观察到延伸的突然减少。
- 要观察解压缩事件,请在 10-12 pN 处等待几秒钟,然后以可能的最大电机速度突然移动到 14-15 pN。根据目标力,SNARE复合物将在部分解压缩的中间体之间表现出可逆的过渡(如在恒定力实验中),或者在随机等待时间(或延迟)后~25nm跳跃到更高的解压缩状态。
- 要观察重新压缩事件,请在观察到解压缩后立即将力降低到 10-12 pN。同样,SNARE复合体在经过一些随机延迟后表现出向较低拉链状态的随机过渡。如果在解压缩后展开,复合物将无法重新压缩,因为 SNAP-25 分子将丢失。
- 要观察展开事件,请在观察到解压缩后等待更长的时间以检测延伸的进一步增加(~2 nm)。
11. 数据分析
注意:可以使用MT数据执行的分析类型取决于目标系统。但是,有一些常用的方法可以从 图4中描述的相应实验中提取有用的信息。所有分析均使用 MATLAB (R2021a) 使用本文提供的自定义代码执行。这些代码使用本文中提供的相同数据生成绘图。请注意,虽然直接获取来自100 Hz跟踪的原始数据进行分析,但来自1.2 kHz跟踪的数据通常在分析之前进行中值滤波(具有五点滑动窗口),以减少噪声(噪声分析除外)。
- 力斜坡实验: 分析力延伸关系(例如,聚合物的弹性)并转换力以提取有关纳米力学性能的信息。
- 恒力实验: 分析状态种群和停留时间(或跃迁率)作为力的函数,以提取构象变化的结构(例如,涉及过渡的区域),热力学(例如,自由能差)和动力学(例如,能量势垒)参数。
- 力跳实验: 分析断裂动力学(例如,蛋白质-蛋白质相互作用和受体-配体结合)或瞬时中间体的寿命(例如,生物分子的展开),以提取靶分子的稳定性及其状态。
- 作为代表性应用,分析DNA发夹和SNARE复合物的样品数据:
- DNA发夹的双态跃迁: 解压缩力,开口距离,群体移位的力依赖性,以及使用隐马尔可夫模型(HMM)(提供MATLAB代码)进行状态分配和转移速率测量。
- SNARE复合物的构象变化: 解压缩力,中间状态和解压缩潜伏期的力依赖性,重新压缩中的滞后以及展开/重新折叠行为。
注意:DNA手柄,DNA发夹和SNARE复合体构象的力延伸模型在之前的参考文献14,31中给出。
结果
力校准
两种力测量方法(磁珠横向位移方差和功率谱分析)的结果相差0-2 pN(图2G)。根据 图2F中的结果,使用常规钕磁铁可以可靠地达到30 pN。
8 bp DNA 发夹的双态转换
我们首先研究了短DNA发夹的纳米力学(图5)。DNA发夹已经通过传统的单分子力谱进行了广泛的表征,因此有大...
讨论
在这项工作中,我们引入了一种单分子力谱装置,可以以高时空精度观察生物分子的结构变化。所使用的高速CMOS相机以1,280 x 1,024分辨率采集1,200帧s−1 ,可实现1.2 kHz磁珠跟踪。然而,测量速度目前受到磁珠跟踪软件的限制,因此在高速测量中,ROI通常会降低到较小的区域。SLD的高功率提供明亮的照明,这对于高达几kHz带宽的快速磁珠成像至关重要。此外,光束的空间相干性在磁珠周...
披露声明
作者没有利益冲突需要声明。
致谢
这项工作得到了韩国政府(MSIT)资助的韩国国家研究基金会(NRF)资助(NRF-2022R1C1C1012176,NRF-2021R1A4A1031754和NRF-2021R1A6A1A10042944)的支持。S.-H.R. 得到了 NRF 拨款 (2021R1C1C2009717) 的支持。
材料
| Name | Company | Catalog Number | Comments |
| Materials for construct synthesis | |||
| Agarose gel electrophoresis system | Advance | Mupid-2plus | |
| DNA ladder | Bioneer | D-1037 | |
| nTaq polymerase | Enzynomics | P050A | |
| PCR purification kit | LaboPass | CMR0112 | |
| PEGylated SMCC crosslinker / SM(PEG)2 | ThermoFisher Scientific | 22102 | For SNARE–DNA coupling |
| Primer B | Bioneer | 5'-Biotin/TCGCCACCATCATTTCCA-3' | For 5-kbp force calibration construct and DNA handles |
| Primer B_hp | IDT | 5'-Biotin/TTTTTTTTTTGTTCTCTATTT TTTTAGAGAAC /AP site/ /AP site/ TCGCCACCATCATTTCCA-3' | For hairpin construct |
| Primer N | Bioneer | 5'-C6Amine/CATGTGGGTGACGCGAAA-3' | For DNA handles |
| Primer Z | Bioneer | 5'-Azide/TCGCCACCATCATTTCCA-3' | For DNA handles |
| Primer Z_5k | Bioneer | 5'-Azide/TTAGAGAGTATGGGTATATGACA TCG-3' | For 5-kbp force calibration construct |
| Primer Z_hp | Bioneer | 5'-Azide/GTGGCAGCATGACACC-3' | For hairpin construct |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| λ-DNA | Bioneer | D-2510 | Template strand for PCR |
| DNA sequences for SNARE proteins | |||
| 6×His-tagged SNAP-25b (2-206; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctat gagaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagcgg aagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCACA GCAGCGGCCTGGTGCCGCGC GGCAGCCATACTAGCGGAGAT ATCGCCGAGGACGCAGACAT GCGCAATGAGCTGGAGGAGA TGCAGAGGAGGGCTGACCAG CTGGCTGATGAGTCCCTGGA AAGCACCCGTCGCATGCTGC AGCTGGTTGAAGAGAGTAAA GATGCTGGCATCAGGACTTT GGTTATGTTGGATGAGCAAG GCGAACAACTGGAACGCATT GAGGAAGGGATGGACCAAAT CAATAAGGACATGAAAGAAG CAGAAAAGAATTTGACGGAC CTAGGAAAATTCGCCGGCCT TGCCGTGGCCCCCGCCAAC AAGCTTAAATCCAGTGATGC TTACAAAAAAGCCTGGGGC AATAATCAGGATGGAGTAGT GGCCAGCCAGCCTGCCCG TGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTG GCTTCATCCGCAGGGTAAC AAATGATGCCCGGGAAAAT GAGATGGATGAGAACCTG GAGCAGGTGAGCGGCATC ATCGGAAACCTCCGCCAC ATGGCTCTAGACATGGGCA ATGAGATTGACACCCAGA ATCGCCAGATCGACAGGA TCATGGAGAAGGCTGATT CCAACAAAACCAGAATTG ATGAAGCCAACCAACGTG CAACAAAGATGCTGGGAA GTGGTTAAggatccgaattcgag ctccgtcgacaagcttgcggccgcactc gagcaccaccaccaccaccactgagat ccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgag caataactagcataaccccttggggcct ctaaacgggtcttgaggggttttttgctga aaggaggaactatatccggat | |
| 6×His-tagged VAMP2 (2-97, L32C/I97C; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCAC AGCAGCGGCCTGGTGCCGC GCGGCAGCCATATGGCAGAT CTCTCGGCTACCGCTGCCAC CGTCCCGCCTGCCGCCCCG GCCGGCGAGGGTGGCCCCC CTGCACCTCCTCCAAATCTTA CCAGTAACAGGAGATGCCAG CAGACCCAGGCCCAGGTGG ATGAGGTGGTGGACATCATG AGGGTGAATGTGGACAAGGT CCTGGAGCGAGACCAGAAG CTATCGGAACTGGATGATCG CGCAGATGCCCTCCAGGCA GGGGCCTCCCAGTTTGAAA CAAGTGCAGCCAAGCTCAA GCGCAAATACTGGTGGAAA AACCTCAAGATGATGTGCTA Aggatccgaattcgagctccgtcg acaagcttgcggccgcactcgagcaccacca ccaccaccactgagatccggctgctaacaaa gcccgaaaggaagctgagttggctgctgcca ccgctgagcaataactagcataaccccttgg ggcctctaaacgggtcttgaggggttttttg ctgaaaggaggaactatatccggat | |
| 6×His-tagged ΔN-VAMP2 (49–96; capitalized) and Syntaxin-1A (191–267, I202C/I266C; capitalized) in pETDuet-1 | homemade | ggggaattgtgagcggataacaattcccctc tagaaataattttgtttaactttaagaagga gatataccATGGGCAGCAGCCATCA TCATCATCATCACAGCAGCGG CCTGGAAGTTCTGTTCCAGGG GCCCGGTAATGTGGACAAGGT CCTGGAGCGAGACCAGAAGCT ATCGGAACTGGATGATCGCGC AGATGCCCTCCAGGCAGGGGC CTCCCAGTTTGAAACAAGTGC AGCCAAGCTCAAGCGCAAATAC TGGTGGAAAAACCTCAAGATGAT GTAAgcggccgcataatgcttaagtcgaaca gaaagtaatcgtattgtacacggccgcataa tcgaaattaatacgactcactataggggaat tgtgagcggataacaattccccatcttagta tattagttaagtataagaaggagatatacat ATGGCCCTCAGTGAGATCGAGA CCAGGCACAGTGAGTGCATC AAGTTGGAGAACAGCATCCG GGAGCTACACGATATGTTCAT GGACATGGCCATGCTGGTGG AGAGCCAGGGGGAGATGATT GACAGGATCGAGTACAATGTG GAACACGCTGTGGACTACGTG GAGAGGGCCGTGTCTGACACC AAGAAGGCCGTCAAGTACCAG AGCAAGGCACGCAGGAAGAA GTGCATGATCTAActcgagtc tggtaaagaaaccgctgctgcgaaatttgaa cgccagcacatggactcgtctactagcgcag cttaattaacctaggctgctgccaccgctga gcaataactagcataaccccttggggcctct aaacgggtcttgaggggttttttgctgaaag gaggaactatatccggattggcgaatgggac gcgccctgtagcggcgcattaagcgcggcgg gtgtggtggttacgcgcagcgtgaccgctac acttgccagcgccctagcgcccgctcctttc gctttcttcccttcctttctcgccacgttcg ccggctttccccgtcaagctctaaatcgggg gctccctttagggttccgatttagtgcttta cggcacctcgaccccaaaaaacttgattagg gtgatggttcacgtagtgggccatcgccctg atagacggtttttcgccctttgacgttggag tccacgttctttaatagtggactcttgttcc aaactggaacaacactcaaccctatctcggt ctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctga tttaacaaaaatttaacgcgaattttaacaa aatattaacgtttacaatttctggcggcacg atggcatgagattatcaaaaaggatcttcac ctagatccttttaaattaaaaatgaagtttt aaatcaatctaaagtatatatgagtaaactt ggtctgacagttaccaatgcttaatcagtga ggcacctatctcagcgatctgtctatttcgt tcatccatagttgcctgactccccgtcgtgt agataactacgatacgggagggcttaccatc tggccccagtgctgcaatgataccgcgagac ccacgctcaccggctccagatttatcagcaa taaaccagccagccggaagggccgagcgca gaagtggtcctgcaactttatccgcctccatc cagtctattaattgttgccgggaagctagag taagtagttcgccagttaatagtttgcgcaa cgttgttgccattgctacaggcatcgtggtg tcacgctcgtcgtttggtatggcttcattca gctccggttcccaacgatcaaggcgagttac atgatcccccatgttgtgcaaaaaagcggtt agctccttcggtcctccgatcgttgtcagaa gtaagttggccgcagtgttatcactcatggt tatggcagcactgcataattctcttactgtc atgccatccgtaagatgcttttctgtgactg gtgagtactcaaccaagtcattctgagaata gtgtatgcggcgaccgagttgctcttgcccg gcgtcaatacgggataataccgcgccacata gcagaactttaaaagtgctcatcattggaaa acgttcttcggggcgaaaactctcaaggatc ttaccgctgttgagatccagttcgatgtaac ccactcgtgcacccaactgatcttcagcatc ttttactttcaccagcgtttctgggtgagcaaa aacaggaaggcaaaatgccgcaaaaaagg gaataagggcgacacggaaatgttgaatact catactcttcctttttcaatcatgattgaag catttatcagggttattgtctcatgagcgga tacatatttgaatgtatttagaaaaataaac aaataggtcatgaccaaaatcccttaacgtg agttttcgttccactgagcgtcagaccccgt agaaaagatcaaaggatcttcttgagatcct ttttttctgcgcgtaatctgctgcttgcaaa caaaaaaaccaccgctaccagcggtggtttg tttgccggatcaagagctaccaactcttttt ccgaaggtaactggcttcagcagagcgcaga taccaaatactgtccttctagtgtagccgta gttaggccaccacttcaagaactctgtagca ccgcctacatacctcgctctgctaatcctgt taccagtggctgctgccagtggcgataagtc gtgtcttaccgggttggactcaagacgatag ttaccggataaggcgcagcggtcgggctgaa cggggggttcgtgcacacagcccagcttgga gcgaacgacctacaccgaactgagataccta cagcgtgagctatgagaaagcgccacgcttccc gaagggagaaaggcggacaggtatccggta agcggcagggtcggaacaggagagcgcac gagggagcttccagggggaaacgcctggtatc tttatagtcctgtcgggtttcgccacctctg acttgagcgtcgatttttgtgatgctcgtca ggggggcggagcctatggaaaaacgccagc aacgcggcctttttacggttcctggccttttg ctggccttttgctcacatgttctttcctgcg ttatcccctgattctgtggataaccgtatta ccgcctttgagtgagctgataccgctcgccgc agccgaacgaccgagcgcagcgagtcagtg agcgaggaagcggaagagcgcctgatgcgg tattttctccttacgcatctgtgcggtatttc acaccgcatatatggtgcactctcagtacaa tctgctctgatgccgcatagttaagccagta tacactccgctatcgctacgtgactgggtca tggctgcgccccgacacccgccaacacccgc tgacgcgccctgacgggcttgtctgctcccg gcatccgcttacagacaagctgtgaccgtct ccgggagctgcatgtgtcagaggttttcacc gtcatcaccgaaacgcgcgaggcagctgcgg taaagctcatcagcgtggtcgtgaagcgatt cacagatgtctgcctgttcatccgcgtccag ctcgttgagtttctccagaagcgttaatgtc tggcttctgataaagcgggccatgttaaggg cggttttttcctgtttggtcactgatgcctc cgtgtaagggggatttctgttcatgggggta atgataccgatgaaacgagagaggatgctca cgatacgggttactgatgatgaacatgcccg gttactggaacgttgtgagggtaaacaactg gcggtatggatgcggcgggaccagagaaaaa tcactcagggtcaatgccagcgcttcgttaa tacagatgtaggtgttccacagggtagccag cagcatcctgcgatgcagatccggaacataa tggtgcagggcgctgacttccgcgtttccag actttacgaaacacggaaaccgaagaccatt catgttgttgctcaggtcgcagacgttttgc agcagcagtcgcttcacgttcgctcgcgtat cggtgattcattctgctaaccagtaaggcaa ccccgccagcctagccgggtcctcaacgaca ggagcacgatcatgctagtcatgccccgcgc ccaccggaaggagctgactgggttgaaggct ctcaagggcatcggtcgagatcccggtgcct aatgagtgagctaacttacattaattgcgtt gcgctcactgcccgctttccagtcgggaaac ctgtcgtgccagctgcattaatgaatcggcc aacgcgcggggagaggcggtttgcgtattgg gcgccagggtggtttttcttttcaccagtga gacgggcaacagctgattgcccttcaccgcc tggccctgagagagttgcagcaagcggtcca cgctggtttgccccagcaggcgaaaatcctg tttgatggtggttaacggcgggatataacat gagctgtcttcggtatcgtcgtatcccacta ccgagatgtccgcaccaacgcgcagcccgga ctcggtaatggcgcgcattgcgcccagcgcc atctgatcgttggcaaccagcatcgcagtgg gaacgatgccctcattcagcatttgcatggt ttgttgaaaaccggacatggcactccagtcg ccttcccgttccgctatcggctgaatttgat tgcgagtgagatatttatgccagccagccag acgcagacgcgccgagacagaacttaatggg cccgctaacagcgcgatttgctggtgaccca atgcgaccagatgctccacgcccagtcgcgt accgtcttcatgggagaaaataatactgttg atgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccac agcaatggcatcctggtcatccagcggatag ttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttc gacgccgcttcgttctaccatcgacaccacc acgctggcacccagttgatcggcgcgagatt taatcgccgcgacaatttgcgacggcgcgtg cagggccagactggaggtggcaacgccaatc agcaacgactgtttgcccgccagttgttgtg ccacgcggttgggaatgtaattcagctccgc catcgccgcttccactttttcccgcgttttc gcagaaacgtggctggcctggttcaccacgc gggaaacggtctgataagagacaccggcata ctctgcgacatcgtataacgttactggtttc acattcaccaccctgaattgactctcttccg ggcgctatcatgccataccgcgaaaggtttt gcgccattcgatggtgtccgggatctcgacg ctctcccttatgcgactcctgcattaggaag cagcccagtagtaggttgaggccgttgagca ccgccgccgcaaggaatggtgcatgcaagga gatggcgcccaacagtcccccggccacgggg cctgccaccatacccacgccgaaacaagcgc tcatgagcccgaagtggcgagcccgatcttc cccatcggtgatgtcggcgatataggcgcca gcaaccgcacctgtggcgccggtgatgccgg ccacgatgcgtccggcgtagaggatcgagat cgatctcgatcccgcgaaattaatacgactc actata | |
| SNAP-25b (1–206, all C to A; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcc agggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGCCGA GGACGCAGACATGCGCAATG AGCTGGAGGAGATGCAGAGG AGGGCTGACCAGCTGGCTGA TGAGTCCCTGGAAAGCACCC GTCGCATGCTGCAGCTGGTT GAAGAGAGTAAAGATGCTGG CATCAGGACTTTGGTTATGTT GGATGAGCAAGGCGAACAAC TGGAACGCATTGAGGAAGGG ATGGACCAAATCAATAAGGAC ATGAAAGAAGCAGAAAAGAAT TTGACGGACCTAGGAAAATTC GCCGGCCTTGCCGTGGCCCC CGCCAACAAGCTTAAATCCAG TGATGCTTACAAAAAAGCCTG GGGCAATAATCAGGATGGAGT AGTGGCCAGCCAGCCTGCCC GTGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTGGC TTCATCCGCAGGGTAACAAAT GATGCCCGGGAAAATGAGATG GATGAGAACCTGGAGCAGGT GAGCGGCATCATCGGAAACCT CCGCCACATGGCTCTAGACAT GGGCAATGAGATTGACACCCA GAATCGCCAGATCGACAGGAT CATGGAGAAGGCTGATTCCAA CAAAACCAGAATTGATGAAGC CAACCAACGTGCAACAAAGAT GCTGGGAAGTGGTTAA ctcgagcaccaccaccaccaccactgag atccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgagc aataactagcataaccccttggggcctc taaacgggtcttgaggggttttttgctgaa aggaggaactatatccggat | |
| Materials for protein purificaiton | |||
| 2-Mercaptoethanol | SIGMA | M3148-25ML | |
| Agar | LPS Solution | AGA500 | |
| Ampicillin, Sodium salt | PLS | AC1043-005-00 | |
| Chloramphenicol | PLS | CR1023-050-00 | |
| Competent cells (E. coli) | Novagen | 70956 | Rosetta(DE3)pLysS |
| Glycerol | SIGMA | G5516-500ML | |
| HEPES | SIGMA | H4034-100G | |
| Hydrochloric acid / HCl | SIGMA | 320331-500ML | |
| Imidazole | SIGMA | I2399-100G | |
| Isopropyl β-D-1-thiogalactopyranoside / IPTG | SIGMA | 10724815001 | |
| Kanamycin Sulfate | PLS | KC1001-005-02 | |
| Luria-Bertani (LB) Broth | LPS Solution | LB-05 | |
| Ni-NTA resin | Qiagen | 30210 | |
| PD MiniTrap G-25 (desalting column) | Cytiva | GE28-9180-07 | For instructions, see: https://www.cytivalifesciences.com/en/us/shop/chromatography/prepacked-columns/desalting-and-buffer-exchange/pd-minitrap-desalting-columns-with-sephadex-g-25-resin-p-06174 |
| Phenylmethylsulfonyl fluoride / PMSF | ThermoFisher Scientific | 36978 | |
| Plasmids for SNARE proteins | cloned in house | N/A | Available upon request |
| Protease inhibitor cocktail | genDEPOT | P3100 | |
| Sodium chloride | SIGMA | S5886-500G | |
| Sodium phosphate dibasic / Na2HPO4 | SIGMA | S7907-100G | |
| Sodium phosphate monobasic / NaH2PO4 | SIGMA | S3139-250G | |
| Tris(2-carboxyethyl)phosphine / TCEP | SIGMA | C4706-2G | |
| Trizma base | SIGMA | T1503-250G | |
| Materials for sample assembly | |||
| Biotin-PEG-SVA | LAYSAN BIO | BIO-PEG-SVA-5K-100MG & MPEG-SVA-5K-1g | For PEGylation |
| Dibenzocyclooctyne-amine / DBCO-NH2 | SIGMA | 761540-10MG | For bead coating |
| Double-sided tape | 3M | 136 | For flow cell assembly |
| Epoxy glue | DEVCON | S-208 | For flow cell assembly |
| Glass coverslip for bottom surface | VWR | 48393-251 | Rectangular, 60×24 mm, #1.5 |
| Glass coverslip for top surface | VWR | 48393-241 | Rectangular, 50×24 mm, #1.5 |
| Magnetic bead | ThermoFisher Scientific | 14301 | Dynabeads M-270 Epoxy, 2.8 μm |
| mPEG-SVA | LAYSAN BIO | mPEG-SVA 1g | For PEGylation |
| N,N-Dimethylformamide / DMF | SIGMA | D4551-250ML | For bead coating |
| N-[3-(trimethoxysilyl)propyl]ethylenediamine | SIGMA | 104884-100ML | For PEGylation |
| Neutravidin | ThermoFisher Scientific | 31000 | For sample tethering |
| Phosphate buffered saline / PBS, pH 7.2 | PLS | PR2007-100-00 | |
| Plastic syringe | Norm-ject | A5 | 5 ml, luer tip |
| Polyethylene Tubing | SCI | BB31695-PE/4 | PE-60 |
| Reference bead | SPHEROTECH | SVP-30-5 | Streptavidin-coated Polystyrene Particles; 3.0-3.4 µm |
| Syringe needle | Kovax | 21G-1 1/4'' | 21 G |
| Syringe pump | KD SCIENTIFIC | 788210 | |
| Equipment for magnetic tweezer instrument | |||
| 1-axis motorized microtranslation stage | PI | M-126.PD1 | For vertical positioning of magnets |
| 2-axis manual translation stage | ST1 | LEE400 | For alignment of magnets to the optical axis |
| Acrylic holder for magnets | DaiKwang Precision | custum order | Drawing available upon request |
| Frame grabber | Active Silicon | AS-FBD-4XCXP6-2PE8 | |
| High-speed CMOS camera | Mikrotron | EoSens 3CXP | |
| Inverted microscope | Olympus | IX73P2F-1-2 | |
| Neodymium magnets | LG magnet | ND 10x10x12t | Dimension: 10 mm × 10 mm × 12 mm; two needed |
| Objective lens | Olympus | UPLXAPO100XO | Oil-immersion, NA 1.45 |
| Objective lens nanopositioner | Mad City Labs | Nano-F100S | |
| Rotation stepper motor | AUTONICS | A3K-S545W | For rotating magnets |
| Superluminescent diode | QPHOTONICS | QSDM-680-2 | 680 nm |
| Software | |||
| LabVIEW | National Instruments | v20.0f1 | |
| MATLAB | MathWorks | v2021a |
参考文献
- Le, S., Liu, R., Lim, C. T., Yan, J. Uncovering mechanosensing mechanisms at the single protein level using magnetic tweezers. Methods. 94, 13-18 (2016).
- Choi, H. -. K., Kim, H. G., Shon, M. J., Yoon, T. -. Y. High-resolution single-molecule magnetic tweezers. Annual Review of Biochemistry. 91 (1), 33-59 (2022).
- Yang, T., Park, C., Rah, S. -. H., Shon, M. J. Nano-precision tweezers for mechanosensitive proteins and beyond. Molecules and Cells. 45 (1), 16-25 (2022).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- De Vlaminck, I., Dekker, C. Recent advances in magnetic tweezers. Annual Review of Biophysics. 41 (1), 453-472 (2012).
- Bustamante, C. J., Chemla, Y. R., Liu, S., Wang, M. D. Optical tweezers in single-molecule biophysics. Nature Reviews Methods Primers. 1, 25 (2021).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical Journal. 82 (6), 3314-3329 (2002).
- Smith, S. B., Finzi, L., Bustamante, C. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258 (5085), 1122-1126 (1992).
- Lansdorp, B. M., Tabrizi, S. J., Dittmore, A., Saleh, O. A. A high-speed magnetic tweezer beyond 10,000 frames per second. Review of Scientific Instruments. 84 (4), 044301 (2013).
- Cnossen, J. P., Dulin, D., Dekker, N. H. An optimized software framework for real-time, high-throughput tracking of spherical beads. Review of Scientific Instruments. 85 (10), 103712 (2014).
- Dulin, D., et al. High spatiotemporal-resolution magnetic tweezers: calibration and applications for DNA dynamics. Biophysical Journal. 109 (10), 2113-2125 (2015).
- Huhle, A., et al. Camera-based three-dimensional real-time particle tracking at kHz rates and Ångström accuracy. Nature Communications. 6 (1), 5885 (2015).
- Popa, I., et al. A HaloTag anchored ruler for week-long studies of protein dynamics. Journal of the American Chemical Society. 138 (33), 10546-10553 (2016).
- Shon, M. J., Kim, H., Yoon, T. -. Y. Focused clamping of a single neuronal SNARE complex by complexin under high mechanical tension. Nature Communications. 9 (1), 3639 (2018).
- Tapia-Rojo, R., Eckels, E. C., Fernández, J. M. Ephemeral states in protein folding under force captured with a magnetic tweezers design. Proceedings of the National Academy of Sciences. 116 (16), 7873-7878 (2019).
- Löf, A., et al. Multiplexed protein force spectroscopy reveals equilibrium protein folding dynamics and the low-force response of von Willebrand factor. Proceedings of the National Academy of Sciences. 116 (38), 18798-18807 (2019).
- Tapia-Rojo, R., Alonso-Caballero, A., Fernandez, J. M. Direct observation of a coil-to-helix contraction triggered by vinculin binding to talin. Science Advances. 6 (21), (2020).
- Rieu, M., et al. Parallel, linear, and subnanometric 3D tracking of microparticles with Stereo Darkfield Interferometry. Science Advances. 7 (6), (2021).
- Rieu, M., Valle-Orero, J., Ducos, B., Allemand, J. -. F., Croquette, V. Single-molecule kinetic locking allows fluorescence-free quantification of protein/nucleic-acid binding. Communications Biology. 4 (1), 1083 (2021).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences. 103 (16), 6190-6195 (2006).
- Camunas-Soler, J., Ribezzi-Crivellari, M., Ritort, F. Elastic properties of nucleic acids by single-molecule force spectroscopy. Annual Review of Biophysics. 45 (1), 65-84 (2016).
- Südhof, T. C., Rothman, J. E. Membrane fusion: grappling with SNARE and SM proteins. Science. 323 (5913), 474-477 (2009).
- Gao, Y., et al. Single reconstituted neuronal SNARE complexes zipper in three distinct stages. Science. 337 (6100), 1340-1343 (2012).
- Zorman, S., et al. Common intermediates and kinetics, but different energetics, in the assembly of SNARE proteins. eLife. 3, e03348 (2014).
- Zhang, Y., Hughson, F. M. Chaperoning SNARE folding and assembly. Annual Review of Biochemistry. 90 (1), 581-603 (2021).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H. Magnetic tweezers for single-molecule experiments. Handbook of Single-Molecule Biophysics. , 371-395 (2009).
- You, H., Le, S., Chen, H., Qin, L., Yan, J. Single-molecule manipulation of G-quadruplexes by magnetic tweezers. Journal of Visualized Experiments. (127), e56328 (2017).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical Journal. 96 (12), 5040-5049 (2009).
- Dulin, D., Barland, S., Hachair, X., Pedaci, F. Efficient illumination for microsecond tracking microscopy. PLoS One. 9 (9), e107335 (2014).
- Klaue, D., Seidel, R. Torsional stiffness of single superparamagnetic microspheres in an external magnetic field. Physical Review Letters. 102 (2), 028302 (2009).
- Shon, M. J., Rah, S. -. H., Yoon, T. -. Y. Submicrometer elasticity of double-stranded DNA revealed by precision force-extension measurements with magnetic tweezers. Science Advances. 5 (6), 1697 (2019).
- Czerwinski, F., Richardson, A. C., Oddershede, L. B. Quantifying noise in optical tweezers by Allan variance. Optics Express. 17 (15), 13255-13269 (2009).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. Review of Scientific Instruments. 83 (2), 025115 (2012).
- Ostrofet, E., Papini, F. S., Dulin, D. High spatiotemporal resolution data from a custom magnetic tweezers instrument. Data in Brief. 30, 105397 (2020).
- Yu, Z., et al. A force calibration standard for magnetic tweezers. Review of Scientific Instruments. 85 (12), 123114 (2014).
- Strick, T. R., Allemand, J. -. F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271 (5257), 1835-1837 (1996).
- Daldrop, P., Brutzer, H., Huhle, A., Kauert, D. J., Seidel, R. Extending the range for force calibration in magnetic tweezers. Biophysical Journal. 108 (10), 2550-2561 (2015).
- te Velthuis, A. J. W., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative guidelines for force calibration through spectral analysis of magnetic tweezers data. Biophysical Journal. 99 (4), 1292-1302 (2010).
- Ostrofet, E., Papini, F. S., Dulin, D. Correction-free force calibration for magnetic tweezers experiments. Scientific Reports. 8 (1), 15920 (2018).
- Seol, Y., Li, J., Nelson, P. C., Perkins, T. T., Betterton, M. D. Elasticity of short DNA molecules: theory and experiment for contour lengths of 0.6-7 µm. Biophysical Journal. 93 (12), 4360-4373 (2007).
- Burnham, D. R., Vlaminck, I. D., Henighan, T., Dekker, C. Skewed Brownian fluctuations in single-molecule magnetic tweezers. PLoS One. 9 (9), 108271 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protocols. 3 (1), 101152 (2022).
- Lee, H. -. W., et al. Profiling of protein-protein interactions via single-molecule techniques predicts the dependence of cancers on growth-factor receptors. Nature Biomedical Engineering. 2 (4), 239-253 (2018).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Parthasarathy, R. Rapid, accurate particle tracking by calculation of radial symmetry centers. Nature Methods. 9 (7), 724-726 (2012).
- Woodside, M. T., Block, S. M. Reconstructing folding energy landscapes by single-molecule force spectroscopy. Annual Review of Biophysics. 43 (1), 19-39 (2014).
- Evans, E., Ritchie, K. Dynamic strength of molecular adhesion bonds. Biophysical Journal. 72 (4), 1541-1555 (1997).
- Zhang, Y. Energetics, kinetics, and pathway of SNARE folding and assembly revealed by optical tweezers. Protein Science. 26 (7), 1252-1265 (2017).
- Chen, H., et al. Improved high-force magnetic tweezers for stretching and refolding of proteins and short DNA. Biophysical Journal. 100 (2), 517-523 (2011).
- Cho, S., et al. Tension exerted on cells by magnetic nanoparticles regulates differentiation of human mesenchymal stem cells. Biomaterials Advances. 139, 213028 (2022).
- Shon, M. J., Cohen, A. E. Nano-mechanical measurements of protein-DNA interactions with a silicon nitride pulley. Nucleic Acids Research. 44 (1), 7 (2016).
- Cheng, Y. Single-particle cryo-EM-How did it get here and where will it go. Science. 361 (6405), 876-880 (2018).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Neupane, K., et al. Direct observation of transition paths during the folding of proteins and nucleic acids. Science. 352 (6282), 239-242 (2016).
- Choi, H. -. K., et al. Watching helical membrane proteins fold reveals a common N-to-C-terminal folding pathway. Science. 366 (6469), 1150-1156 (2019).
- Kim, C., et al. Extreme parsimony in ATP consumption by 20S complexes in the global disassembly of single SNARE complexes. Nature Communications. 12 (1), 3206 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。