A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
פינצטה מגנטית במהירות גבוהה למדידות ננומכניות על אלמנטים רגישים לכוח
* These authors contributed equally
In This Article
Summary
במאמר זה אנו מתארים מערך פינצטה מגנטית במהירות גבוהה המבצע מדידות ננו-מכניות על ביומולקולות רגישות לכוח בקצב מרבי של 1.2 קילוהרץ. אנו מציגים את היישום שלה על סיכות ראש DNA וקומפלקסים SNARE כמערכות מודל, אבל זה יהיה ישים גם על מולקולות אחרות המעורבות באירועים מכנוביולוגיים.
Abstract
פינצטה מגנטית של מולקולה בודדת (MTs) שימשה ככלי רב עוצמה לחקירה מאולצת של ביומולקולות, כגון חומצות גרעין וחלבונים, ולכן הן צפויות להיות שימושיות בתחום המכנוביולוגיה. מכיוון שהשיטה מסתמכת בדרך כלל על מעקב מבוסס תמונה של חרוזים מגנטיים, המהירות המותרת בהקלטה וניתוח של תמונות, כמו גם התנודות התרמיות של החרוזים, הקשו זה מכבר על יישומה בצפייה בשינויים מבניים קטנים ומהירים במולקולות המטרה. מאמר זה מתאר שיטות מפורטות לבנייה ותפעול של מערך MT ברזולוציה גבוהה שיכול לפתור דינמיקה ננומטרית, אלפית שנייה של ביומולקולות והקומפלקסים שלהן. כדוגמאות ליישום, מודגמים ניסויים עם סיכות ראש של DNA וקומפלקסים של SNARE (מכונות איחוי ממברנה), תוך התמקדות באופן שבו ניתן לזהות את המצבים והמעברים הארעיים שלהם בנוכחות כוחות בקנה מידה פיקוניוטון. אנו מצפים כי MTs במהירות גבוהה ימשיכו לאפשר מדידות ננומכניות בדיוק גבוה על מולקולות החשות, מעבירות ומייצרות כוחות בתאים, ובכך יעמיקו את ההבנה שלנו ברמה המולקולרית של מכנוביולוגיה.
Introduction
תאים חשים באופן פעיל ומגיבים לגירויים מכניים. בעשותן כך, ביומולקולות רבות מציגות תכונות תלויות כוח המאפשרות שינויים מבניים דינמיים. דוגמאות מוערכות כוללות תעלות יונים רגישות למכנו ואלמנטים ציטו-שלדיים המספקים לתאים מידע מכני חשוב מסביבתם.
בנוסף, מולקולות בעלות אופי ייחודי של נשיאת כוח יכולות להיחשב גם רגישות למכנו במובן רחב יותר. לדוגמה, היווצרות מקומית והתכה של דופלקסים של חומצות גרעין, כמו גם מבנים מסדר גבוה יותר כגון G-quadruplexes, ממלאים תפקידים מכריעים בשכפול, שעתוק, רקומבינציה, ולאחרונה, עריכת גנום. יתר על כן, כמה חלבונים עצביים המעורבים בתקשורת סינפטית לבצע את תפקידיהם על ידי יצירת כוחות פיזיים העולים על רמות של אינטראקציות בין-מולקולריות טיפוסיות. לא משנה איזו דוגמה חוקרים, חקירת ננומכניקה של הביומולקולות המעורבות בדיוק מרחבי-זמני גבוה תוכיח את עצמה כשימושית ביותר בחשיפת מנגנונים מולקולריים של התהליכים המכנוביולוגיים הקשורים 1,2,3.
שיטות ספקטרוסקופיית כוח של מולקולה בודדת שימשו ככלים רבי עוצמה לבחינת התכונות המכניות של ביומולקולות 2,4,5,6. הם יכולים לעקוב אחר שינויים מבניים בחומצות גרעין ובחלבונים במקביל להפעלת כוח, ובכך לבחון תכונות תלויות כוח. שני מערכים ידועים הם פינצטה אופטית ופינצטה מגנטית (MTs), המשתמשים בחרוזים בגודל מיקרון כדי לתפעל מולקולות 5,6,7,8. בפלטפורמות אלה, פוליסטירן (עבור פינצטה אופטית) או חרוזים מגנטיים (עבור MTs) קשורים למולקולות מטרה (למשל, חומצות גרעין וחלבונים) באמצעות "ידיות" מולקולריות, העשויות בדרך כלל מקטעים קצרים של DNA דו-גדילי (dsDNA). לאחר מכן החרוזים מועברים כדי להפעיל כוח ומצולמים כדי לעקוב אחר מיקומם המדווח על שינויים מבניים במולקולות המטרה. פינצטה אופטית ומגנטית ניתנות במידה רבה להחלפה ביישומים שלהן, אך קיימים הבדלים חשובים בגישותיהן לשליטה בכוח. פינצטה אופטית היא במהותה מכשירי מהדק מיקום הלוכדים חרוזים במקומם, שבגללם הכוח המופעל משתנה כאשר מבנה מטרה עובר שינויי צורה; הארכה מוגברת, כגון מפתיחה, משחררת את הקשירה ומפחיתה מתח, ולהיפך. למרות שניתן ליישם משוב אקטיבי כדי לשלוט בכוח בפינצטה אופטית, MTs לעומת זאת פועלים באופן טבעי כמכשיר מהדק כוח, תוך ניצול כוחות מגנטיים יציבים בשדה רחוק על ידי מגנטים קבועים, שיכולים לעמוד גם בהפרעות סביבתיות.
למרות ההיסטוריה הארוכה והעיצוב הפשוט שלהם, MTs פיגרו אחרי פינצטה אופטית ביישומים שלהם למדידות דיוק גבוה, בעיקר בגלל אתגרים טכניים במעקב חרוזים מהיר. לאחרונה, עם זאת, מספר קבוצות הובילו במשותף שיפור רב-גוני של חומרה ותוכנה עבור מכשירי MT 2,9,10,11,12,13,14,15,16,17,18,19 . בעבודה זו, אנו מציגים דוגמה של מערך כזה הפועל במהירות של 1.2 קילוהרץ ומתארים כיצד להשתמש בו לביצוע מדידות ננו-מכניות על ביומולקולות רגישות לכוח. כמערכות מודל, אנו משתמשים בסיכות ראש של DNA ובקומפלקסים של SNARE עצבי ובוחנים את השינויים המבניים המהירים שלהם במשטר הפיקוניוטון. סיכות ראש DNA מציגות מעברים פשוטים של שני מצבים בטווח כוח מוגדר היטב20,21, ולכן משמשות כמודלים צעצוע כדי לאמת את הביצועים של מערך פינצטה. כאשר חלבוני SNARE מתאספים לקומפלקס רגיש לכוח המניע היתוך ממברנה22, הם נחקרו בהרחבה גם על ידי ספקטרוסקופיית כוח של מולקולה בודדת 14,23,24,25. מוצגות גישות סטנדרטיות לניתוח נתונים וחילוץ מידע שימושי על תרמודינמיקה וקינטיקה. אנו מקווים שמאמר זה יוכל להקל על אימוץ MTs מדויקים במחקרים מכנוביולוגיים ולהניע את הקוראים לחקור את מערכות העניין הרגישות לכוח שלהם.
Protocol
כל החומרים והציוד המתוארים בפרוטוקול זה מפורטים בטבלת החומרים. תוכנת LabVIEW להפעלת הגדרת MT במהירות גבוהה המתוארת להלן, כמו גם סקריפטים של MATLAB לניתוח נתונים לדוגמה, מופקדים ב- GitHub (https://github.com/ShonLab/Magnetic-Tweezers) וזמינים לציבור.
1. בניית מנגנון
הערה: העיקרון הכללי של בניית MT במהירות גבוהה דומה למערכות MT סטנדרטיות קונבנציונליות, למעט השימוש במצלמת מוליכים למחצה (CMOS) משלימה במהירות גבוהה ומקור אור קוהרנטי בעוצמה גבוהה (איור 1). עיין במקורות אחרים לקבלת תיאורים נוספים של מכשירי MT סטנדרטיים 5,26,27.
- הגדר מיקרוסקופ הפוך על שולחן אופטי נגד רטט. התקן מצלמת CMOS במהירות גבוהה ותופס פריימים.
- בניית שלב תרגום למניפולציה של מגנטים בתלת מימד. הרכיבו שלב ליניארי ממונע (>20 מ"מ נסיעה) אנכית על גבי שלב XY ידני.
הערה: התנועה האנכית שולטת בכוח, ואילו שלב XY מיועד ליישור ידני של מגנטים לציר האופטי לצורך הבנייה הראשונית של ההתקנה. - התקן מנוע צעד סיבובי ומערכת חגורה וגלגלת למגנטים מסתובבים.
הערה: החגורה משדרת את התנועה הסיבובית בין מוט המנוע לבין מגנטים הנמצאים במרחק של כמה סנטימטרים זה מזה. סיבוב המגנטים הוא פנימי למניפולציה התרגומית. - הרכיבו את המגנטים. השתמשו במחזיק אקריליק (שהוזמן מחברה יצרנית; ראו איור משלים S1) שיכול לאחסן בחוזקה שני מגנטים זהים במקביל, עם רווח מוגדר היטב של 1 מ"מ בין המגנטים (איור 1B). כדי לנצל את הכוח המרבי שניתן להשיג עם זוג מגנטים נתון, התאימו את המיקום האנכי של שלב התרגום כך שהמשטח התחתון של המגנטים יתיישר עם מישור הדגימה כאשר הוא מועבר למיקום הנמוך ביותר.
הערה: עיין ב- Lipfert et al. לקבלת מידע נוסף על עיצוב המחזיק ותצורה של מגנטים28. הגובה והכיוון של מגנטים נשלטים על ידי תוכנת LabVIEW בשילוב עם איסוף נתונים. - צפייה בעדשה אובייקטיבית בהגדלה נמוכה, יישרו את המגנטים למרכז שדה הראייה. בדקו שסיבוב המגנטים אינו גורם לתזוזה גדולה של מרכז זוג המגנטים.
הערה: אם נקודת האמצע בין המגנטים מסתובבת סביב ציר הסיבוב, סביר להניח שהמגנטים אינם ממורכזים בגלל מחזיק לא מושלם. רמה קטנה של חוסר התאמה ביחס לגודל הפער היא נסבלת, מכיוון שסיבוב המגנט נועד רק לבדיקת קשירה והפעלת מומנטים ביישומים ספציפיים. - התקן דיודה סופרלומינסנטית (SLD) להארת חרוזים. מעבירים את הקרן דרך רווח של 1 מ"מ בין שני המגנטים. יש לוודא שהקרן מחוברת כראוי כדי להתאים לרווח והתאורה אינה מוצללת על ידי המגנטים.
- התקן סורק עדשות piezo על האף והרכיב עדשה אובייקטיבית 100x טבילת שמן (צמצם מספרי [NA]: 1.45) למעקב אחר חרוזים. כדי למנוע ממצאים פוטנציאליים במעקב אחר תוצאות, ודא שהתאורה נשמרת באופן אחיד בעת הזזת המגנטים. לבסוף, התאם את רמת האור לבהירות המרבית מבלי להרוות פיקסלים.
הערה: להשוואה בין מקורות אור שונים למעקב מהיר אחר חרוזים, עיין ב- Dulin et al.29.
2. כיול הכוח המגנטי
- באמצעות תגובת שרשרת פולימראז (PCR; ראו טבלה 1), הכינו מקטעי dsDNA של 5kbp (באמצעות Primer B, Primer Z_5k ו-λ-DNA) המסומנים בביוטין בקצה אחד (לחיבור פני השטח) ואזיד בקצה השני (לחיבור חרוזים).
- לאחר סעיף 6, הכינו תא זרימה עם מולקולות 5 kbp.
- לאחר סעיף 7, זהה מבנה קשירת חרוזים טוב על ידי אימות ההרחבה והסיבוב שלו. בפרט, הקפד לבחור חרוז עם מסלול סיבוב מינימלי (כלומר, עם רדיוס <200 ננומטר) כדי למזער את היסט גובה החרוז עקב חיבור לא ממורכז30,31. לאחר זיהוי קשירה טובה, התחל לעקוב אחר חרוזים, תוך התייחסות לסעיף 9.
- אם ההתקנה חדשה, אפיין את הרעש והיציבות שלה למדידות אמינות ברזולוציה גבוהה. מקמו את המגנט ~3 מ"מ מפני השטח של תא הזרימה (כדי להחיל >10 pN ולדכא את התנועה הבראונית של חרוז), עקבו אחר מיקום z של החרוז ב-1.2 קילוהרץ, וחשבו את סטיית אלן (AD) מסדרת הזמן קואורדינטות z 32,33 (איור 2C). בדקו כי ערכי אלצהיימר של ננומטרים בודדים ניתנים להשגה במשטר המהירות הגבוהה (<0.1 שניות), וכי מעקב דיפרנציאלי (מיקום חרוז מגנטי ביחס לחרוז ייחוס) מפחית את האלצהיימר בטווח הזמן הארוך יותר.
הערה: בדרך כלל אנו מקבלים AD של <3 ננומטר בקצב המרבי (רזולוציה של 1.2 קילו-הרץ או 0.83 אלפיות השנייה), וה-AD ממשיך לרדת לפחות עד 10 שניות, מה שמרמז על סחף מינימלי. אחרים דיווחו על ערכים דומים בהגדרות דומות 9,10,11,12,34. - עם מגנטים במצב מנוחה (F ~ 0 pN), רשום את קואורדינטות x ו - y של החרוז הקשור ב- 1.2 קילוהרץ. רשום את המיקום לתקופה ארוכה מספיק (כלומר, ארוכה מספיק מזמן ההרפיה האופייני של תנודות35) כך שהתנועה הבראונית נדגמת מספיק.
הערה: כאן, כיוון ה-x הוא לאורך כיוון השדה המגנטי, ואילו התנועה ב-y מייצגת את התנועה הרוחבית בניצב לשדה. - קרבו את המגנטים לתא הזרימה וחזרו על מדידות מיקום החרוזים עד שהמגנטים נוגעים בעדינות בחלק העליון של תא הזרימה. נעו בצעדים גדולים (למשל, 1-2 מ"מ) כאשר המגנטים נמצאים במרחק של יותר מ-7 מ"מ ממישור הדגימה (מאחר שהכוח המופעל גדל לאט בשדה הרחוק של מגנטים), אך צמצמו את גודל הצעד בהדרגה (למשל, 0.1-0.5 מ"מ) ככל שהם מתקרבים יותר לכיול עדין יותר ברמות כוח גבוהות יותר (איור 2B).
- חשב את הכוח בכל מיקום מגנט, d, באמצעות אחת משתי השיטות החלופיות (סקריפט MATLAB "force calibration.m" הכולל את שתי השיטות מסופק; ראה קובץ משלים 1).
- מדדו את השונות של קואורדינטות
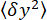 y של החרוז (איור 2D) ואת מיקום
y של החרוז (איור 2D) ואת מיקום 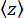 z הממוצע של החרוז ביחס למיקום הנמוך ביותר (איור 2B, למטה). לאחר מכן, השתמש במשוואה (1)7,27,36 כדי להעריך את הכוח (עם רדיוס חרוז קבוע R = 1,400 ננומטר ואנרגיה תרמית k RT = 4.11 pN∙nm):
z הממוצע של החרוז ביחס למיקום הנמוך ביותר (איור 2B, למטה). לאחר מכן, השתמש במשוואה (1)7,27,36 כדי להעריך את הכוח (עם רדיוס חרוז קבוע R = 1,400 ננומטר ואנרגיה תרמית k RT = 4.11 pN∙nm):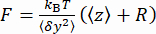 (1)
(1) - לחלופין, חשב את הצפיפות הספקטרלית של הספק (PSD) של קואורדינטות y, Sy (איור 2E). קבע את הכוח המופעל F על ידי התאמת מודל לורנציאני כפול37 ל- Sy הנמדד באמצעות משוואה (2).
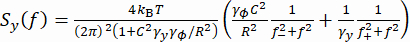 (2)
(2)
כאן, , R הוא רדיוס חרוזים, γ yו- γφ הם מקדמי הגרר התרגומי והסיבובי, בהתאמה (מוערך ממשוואת סטוקס-איינשטיין), kRT היא האנרגיה התרמית,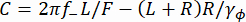 f+ ו- f- הם שני תדרים אופייניים המתקבלים באמצעות משוואה (3).
f+ ו- f- הם שני תדרים אופייניים המתקבלים באמצעות משוואה (3).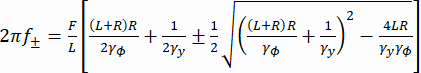 (3)
(3)
הערה: מכיוון שסיומת הקשירה L היא פונקציה של כוח העוקבת אחר מודל השרשרת דמוית התולעת (WLC) המבוסס היטב, הביטויים לעיל משאירים את F כפרמטר המתאים היחיד (אנו מתקנים את R להיות 1,400 ננומטר לשם הפשטות מכיוון שהוא משותף לכל רמות הכוח והערך המדויק אינו משפיע על התוצאות במידה ניכרת). במידת הצורך, טשטוש תנועה והחלקה מרכישת תמונה מבוססת מצלמה חייבים להיחשב38,39, אך השפעה זו זניחה במדידות המהירות הגבוהה שלנו מעל 1 kHz עם קשירה של 5 kbp.
- מדדו את השונות של קואורדינטות
- חזור על שלבים 2.4-2.7 לקבלת כמה מבנים נוספים. בדקו שלושה עד חמישה חרוזים שונים כדי לחשב את השתנות הכוח בין החרוזים המגנטיים.
הערה: יש לשקול שינוי כוח בין החרוזים המגנטיים הנמצאים בשימוש כדי לקבוע את המספר הנכון של מבנים לממוצע. שונות זו קטנה אך יכולה להוביל לטעות של יותר מ-1 pN בכוח הנמדד, אפילו עבור מוצרים מסחריים31. עבור רוב היישומים, שבהם הקביעה המוחלטת של הכוחות המעורבים אינה חיונית, ממוצע תוצאות הכיול של שלושה עד חמישה חרוזים הוא בדרך כלל מספיק. גישה חלופית להסביר שונות זו היא למדוד את הכוח עם קשירה בודדת בתחילת הניסוי, אשר יכול לקחת זמן. אפשרות נוספת היא להטמיע מבני סיכות ראש שנפתחים ברמות כוח ידועות בכל מבנה31. - התווה את הכוח הנמדד כפונקציה של מרחק מגנט והתאים פונקציה מעריכית כפולה לנתונים (איור 2F) באמצעות משוואה (4).
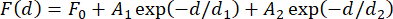 (4)
(4)
כאן, F0 (קו בסיס), A 1 ו- A 2 (אמפליטודות), ו- d 1 ו- d2 (קבועי דעיכה) הם פרמטרים מתאימים. ודא שערכי הכוח משתי השיטות, כמו גם ההתאמות המעריכיות הכפולות הנובעות מכך, מסכימים במידה רבה (איור 2F,G).
הערה: כדי לוודא שכיול הכוח מתבצע כראוי, ודא את יחסי הכוח-הארכה של המבנים הנבדקים על-ידי התוויית ההרחבה לעומת הכוח הנמדד. - כדי לתקן את היסט גובה החרוז z off כתוצאה מהטיה תלוית כוח של החרוזים המגנטיים30,31, הערך z off מההיסט הצידי x off, בהתחשב בגיאומטריה של קשירה לא ממורכזת עם רדיוס חרוז באמצעות משוואה (5), והחל את הערכים על ערכי ההרחבה הנמדדים. שלב זה מיושם בסקריפט MATLAB "force calibration.m" (שורות 252-254).
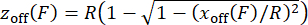 (5)
(5)
הערה: אף על פי שתיקון זה מבצע שינויים קטנים בהרחבה, במיוחד עבור חרוזים עם רדיוס סיבוב קטן (<200 ננומטר), היסט זה משפיע לעתים קרובות באופן קריטי על התגובה האלסטית, כפי שניתן לראות בשינוי מאיור 2H לאיור 2I 30,31. - בדוק את אורך ההתמדה Lp על-ידי התאמת מודל WLC ניתן להרחבה לנתונים באמצעות משוואה (6).
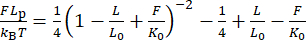 (6)
(6)
כאן, L 0 הוא אורך קווי המתאר (1.7 מיקרומטר עבור 5 kbp) ו- K0 הוא המודולוס למתיחה אנתלפית.
הערה: למרות ש-L p של dsDNA מקובל היטב להיות 40-50 ננומטר במאגר טיפוסי כגון מלח חוצץ פוספט (PBS), נוסחת WLC המיושמת על מולקולות קצרות (<5 kbp) מעריכה באופן שיטתי את L p כמו L0 פוחת31,40. הסיבה לכך היא שמודל WLC הקלאסי מניח פולימר שאורך השרשרת שלו ארוך מספיק מאורך ההתמדה שלו. כאן, קיבלנו Lp = 40 ± 3 ננומטר עבור מבנה 5 kbp (איור 2H), ותיקון ההרחבה הניב K0 הומוגני של 1,100 ± 200 pN (איור 2I). החלת מודל WLC סופי 31,40, כמו גם תיקון לאי-גאוסיות בהתפלגות הרחבה 41, תגדיל מעט את Lp. - לאחר אימות כיול הכוח, החל את פרמטרי ההתאמה המתקבלים של המודל המעריכי הכפול על תוכנת LabVIEW שסופקה (קובץ משלים 2) והמתן עד שהתוכנה תחשב את הכוח הנוכחי בזמן אמת מקריאות מוטוריות (כלומר, מיקום מגנט). מכיוון שביטוי אנליטי עבור הפונקציה ההופכית d(F) אינו זמין, הכינו טבלת בדיקת מידע של d לעומת F בשלבים של 0.1 pN לפי הערכה מספרית של רמות כוח היעד d. אחסן טבלה זו גם בתוכנה כדי לפקד על בקרת הכוח.
3. סינתזה של סיכות שיער DNA
הערה: מבנים של סיכות ראש של דנ"א עבור ניסויי MT מוכנים על-ידי הגברה PCR של אזור של 510 bp ב-λ-DNA עם שני פריימרים מותאמים אישית, שאחד מהם מכיל מבנה סיכת ראש בקצה 5′ שלו (איור 3A). בדרך זו, מוטיב סיכת ראש ממוקם בקצה אחד של מוצר PCR.
- מכינים את הפריימרים.
- פריימר קדמי: פריימר B_hp שמסומן ב-5′-ביוטין לחיבור משטח זכוכית ונקשר ל-λ-DNA. פריימר זה מכיל מוטיב סיכת ראש עם גבעול 8 bp ולולאה 6 nt, 5′ לאזור קשירת λ.
- פריימר הפוך: פריימר Z_hp המסומן בתווית 5′-אזיד לחיבור חרוזים מגנטיים ונקשר ל-λ-DNA במרחק של 1 kbp מהפריימר הקדמי.
- הגדר והפעל את ה- PCR עם λ-DNA (תבנית), nTaq פולימראז ותנאי PCR סטנדרטיים (ראה טבלה 1). נקו את המוצר עם ערכת טיהור מסחרית.
- למדוד את ריכוז הדנ"א על ידי ספיגת UV ב 260 ננומטר (A260) ולבצע אלקטרופורזה ג'ל אגרוז (2% ג'ל) (ראה טבלה 2) כדי לאמת את גודל המוצר. תפוקה אופיינית היא ~ 35 μL של ~ 600 ננומטר פתרון.
4. הכנת חלבוני SNARE
הערה: קומפלקסים עצביים של SNARE מורכבים על-ידי שילוב של שלושה חלבוני חולדה מטוהרים המבוטאים מ-E. coli: VAMP2/synaptobrevin-2, syntaxin-1A ו-SNAP-25 (איור 3B). כדי להקל על הרכבתם, תחביר ו-SNAP-25 מבוטאים יחד עם מקטע VAMP2 (חסר את אזור N-terminal; המכונה "ΔN-VAMP2") למבנה הנקרא "קומפלקס ΔN", ולאחר מכן מעורבבים עם VAMP2 באורך מלא לאחר חיבור DNA לטיפול ליצירת קומפלקסים מלאים.
- הכינו פלסמידים המכילים cDNA לביטוי חלבוני SNARE (רצפי DNA לכל הפלסמידים ניתנים בטבלת החומרים).
- הכינו VAMP2 6×His-tagged חסר את תחום הטרנסממברנה (2-97; L32C/I97C עבור קישורים דיסולפידים) משוכפל לתוך וקטור pET28a.
- הכינו תחביר-1A חסר את תחום Habc ואת תחום הטרנסממברנה (191-267, I202C/I266C החלפות לקישורים דיסולפידים) המשוכפלים יחד עם 6×His-tagged ΔN-VAMP2 (49-96) לווקטור pETDuet-1.
- הכינו את האיזופורם b באורך מלא מסוג SNAP-25 (2-206, כל C עד A) המשוכפל לווקטור pET28a. זה ישמש להכנת מתחמי ΔN.
- הכינו את האיזופורם b באורך מלא SNAP-25 באורך מלא באורך ×6 (1-206, כל C עד A) המשוכפל לווקטור pET28a להוספה ישירה למאגר בדיקת MT כדי להרכיב מחדש את מתחמי SNARE לאחר הפתיחה.
- הכינו שתי שפופרות של תאי E. coli של רוזטה (DE3). המר קבוצה אחת עם פלסמידים VAMP2 (משלב 4.1.1), קבוצה אחת עם תחביר-1A/ΔN-VAMP2 ופלסמידים SNAP-25 לא מתויגים (משלבים 4.1.2 ו-4.1.3) לביטוי קומפלקס ΔN, והשנייה עם פלסמידים SNAP-25 המתויגים שלו (משלב 4.1.4).
- העבירו את התאים שעברו טרנספורמציה למרק לוריא-ברטאני (LB) עם אנטיביוטיקה מתאימה (כאן, קנמיצין וכלוראמפניקול עבור VAMP2 ו-His-tagged SNAP-25; קנמיצין, כלוראמפניקול ואמפיצילין עבור קומפלקס ΔN). מגדלים אותם בטמפרטורה של 37 מעלות צלזיוס באינקובטור רועד (220 סל"ד) עד שהצפיפות האופטית (OD) של המרק מגיעה ל-0.7-0.9.
- הוסף 1 mM isopropyl β-d-1-thiogalactopyranoside (IPTG) כדי לגרום ביטוי חלבון לדגור את התאים במשך 3-4 שעות ב 37 ° C באינקובטור רועד (220 סל"ד).
- גלולה למטה את התאים על ידי צנטריפוגה התרבית ב 4,500 × גרם במשך 15 דקות ב 4 ° C.
- הכינו מאגרים לטיהור חלבונים (ראו טבלה 2).
- יש להשהות כדורי תאים המבטאים SNARE ב-40 מ"ל של חיץ ליזה קר כקרח וליז את התאים על ידי סוניקציה על קרח (15% משרעת, 5 שניות דולקות ו-5 שניות, 30 דקות בסך הכל).
- צנטריפוגה את הליזט ב-15,000 × גרם למשך 30 דקות ב-4°C כדי להסיר חומרים בלתי מסיסים.
- מעבירים את הסופרנאטנט דרך עמוד כבידה מלא ב-1 מ"ל של שרף ני-נת"ע. לשטוף את השרף עם לשטוף חיץ A, ולאחר מכן עם חיץ לשטוף B, ואת delute החלבונים עם 10 מ"ל של חיץ elution.
- הסר tris(2-carboxyethyl)phosphine (TCEP) ו imidazole מן eluent באמצעות עמודת התפלה (בצע את הוראות היצרן). יש להצדיע לדגימה באמצעות PBS.
- רכז את החלבונים עם מסננים צנטריפוגליים (חיתוך של 10 kDa ) ל~ 70 מיקרומטר תוך שמירה על החלבונים ב- PBS (בדרך כלל מניב 2 מ"ל). מדוד את ריכוז החלבון על ידי ספיגה אולטרה סגולה (UV) ב 280 ננומטר (A280) או על ידי מבחן ברדפורד.
- הכינו אליציטוטים קטנים, הקפיאו במהירות הבזק בחנקן נוזלי ואחסנו בטמפרטורה של -80°C עד לשימוש.
הערה: קומפלקסים מלאים של SNARE יורכבו לאחר הצמדת קומפלקס ΔN על ידית DNA (ראה להלן).
5. חיבור של ידיות DNA
הערה: שתי ידיות dsDNA של 510 bp המכילות קבוצות אמין ראשוניות בקצה אחד מוכנות תחילה על ידי PCR, ולאחר מכן קבוצות האמין מומרות לקבוצות מלימיד באמצעות crosslinker דו-תפקודי, SM(PEG)2. שתי הידיות מקושרות באופן קוולנטי לקומפלקסים של SNARE באמצעות קבוצות הציסטאין שלהן לצורך צימוד ספציפי לאתר (איור 3B).
- הכינו פריימרים.
- הכינו פריימרים קדמיים: פריימר B (להגברת ידית B) המסומן 5′-ביוטין לחיבור משטח זכוכית ונקשר ל-λ-DNA; פריימר Z (להגברת ידית Z) המסומן 5′-אזיד לחיבור חרוזים מגנטיים ובעל רצף זהה לפריימר B.
- הכינו פריימר הפוך: פריימר N (משותף לידית B ולידית Z) המסומן בתווית 5′-אמין להצמדת חלבונים ונקשר ל-λ-DNA במרחק של 510 bp מהפריימר הקדמי.
- הגדר והפעל שתי קבוצות של תגובות PCR (18 צינורות של תגובת 200 μL לכל ידית) עם λ-DNA (תבנית), nTaq פולימראז ותנאי PCR סטנדרטיים (ראה טבלה 1). נקו את המוצר עם ערכת ניקוי PCR ועטפו כל ידית ב-45 מיקרוליטר מים טהורים במיוחד. השתמש בנפח מינימלי של מים כדי להשיג ריכוזים גבוהים של ידיות לתגובה יעילה בשלבים מאוחרים יותר.
- מדדו את ריכוז הדנ"א ב-A260. התפוקה האופיינית היא ~ 650 μL של ~ 2 מיקרומטר תמיסה עבור כל ידית. יש לשמור דגימות קטנות בנפרד לצורך אימות מאוחר יותר באלקטרופורזה של ג'ל אגרוז.
- הגב כל ידית (1 מיקרומטר ב-PBS) עם 5 mm sm(peg)2. דוגרים בטמפרטורת החדר עם סיבוב עדין. לאחר שעה אחת, השתמש בערכת טיהור DNA כדי להסיר SM(PEG)2 שלא מגיב. הצביעו על כל ידית עם 250 μL של PBS כדי לקבל ~2 מיקרומטר תמיסות.
- ערבבו את התמיסות של קומפלקס ידית B ו-ΔN ביחס מולארי של 1:16 (למשל, 1 מיקרומטר ידית B ו-16 מיקרומטר ΔN קומפלקס) ב-PBS ודגרו במשך שעתיים בטמפרטורת החדר עם תסיסה. יש להפריד דגימה קטנה לאלקטרופורזה של ג'ל אגרוז.
- הוסף תמיסה של VAMP2 בעודף טוחנת פי 2.5 מעל קומפלקס ΔN ששימש בשלב הקודם. דוגרים על התערובת עוד שעה בטמפרטורת החדר עם תסיסה. מתחמי SNARE מלאים מורכבים בשלב זה.
- הסר חלבונים חופשיים על ידי החלפת חיץ עם PBS טרי ומסנן צנטריפוגלי (חיתוך של 100 kDa ): צנטריפוגה ב 14,000 × גרם במשך 5 דקות ב 4 ° C, חזור על לפחות 6x, ולרוץ במשך 15 דקות עבור הסחרור האחרון. מדוד את הגידול ביחס A260/A280 כדי לפקח על הסרת חלבונים חופשיים. יש להפריד דגימה קטנה לאלקטרופורזה של ג'ל אגרוז.
- הוסף את ידית Z לתמיסה בעודף טוחנת של פי 15 מעל ידית B. שמור על ריכוז ידית Z לפחות מעל 1 מיקרומטר כדי להקל על התגובה. לדגור על התערובת במשך הלילה ב 4 °C עם תסיסה.
- אמת את חומרי הביניים (ידית B והצמדות החלבונים שלה) ואת המוצר הסופי (קומפלקס SNARE עם שתי ידיות) באמצעות אלקטרופורזה של ג'ל אגרוז (איור 3B, inset) (ראה טבלה 2).
הערה: אם החלבונים מחוברים בהצלחה לידית ב', יזוהה שינוי תנועתי. בפרט, היווצרות קומפלקסים מלאים של SNARE על ידיות דנ"א יכולה להיות מאושרת על ידי עמידותם לנתרן דודציל סולפט (SDS), בניגוד לקומפלקסים של ΔN, שמפורקים ב-SDS ומשאירים רק תחביר קשור לדנ"א (השוו בין b ו-c באיור 3B). - הכינו אליציטוטים קטנים, הקפיאו במהירות הבזק בחנקן נוזלי ואחסנו בטמפרטורה של -80°C עד לשימוש.
הערה: למרות שהפתרון הסופי מכיל ידיות לא מגיבות, רק המבנה הרצוי המסומן פעמיים עם ביוטין ואזיד ייבחר במהלך הרכבת הדגימה בתא זרימה.
6. ייצור תאי זרימה
הערה: תאי זרימה למדידות MT בנויים משני כיסויי זכוכית המחוברים זה לזה באמצעות סרט דו-צדדי (איור 3C). כיסוי אחד מצופה בתערובת של PEG ופוליאתילן גליקול ביוטינילציה (PEG) כדי למנוע קשירה לא ספציפית ולאפשר קשירה ספציפית של מולקולות מטרה באמצעות קישור ביוטין-נויטראבידין (איור 3D). לאחר מכן, התמיסות של חומרים עבור ניסויי MT מוחדרות ברצף לתוך תא זרימה באמצעות משאבת מזרק (איור 3C,D).
- הכינו שני כיסויי זכוכית, אחד כל אחד למשטח העליון (24 מ"מ × 50 מ"מ, עובי מס' 1.5) והתחתון (24 מ"מ × 60 מ"מ, עובי מס' 1.5). נקו את הכיסויים על ידי סוניקציה ב-1 M KOH למשך 30 דקות. לאחר הסוניקציה, שטפו את הכיסויים במים מזוקקים ושמרו במים עד לשלב הבא.
- PEGylate את תלוש הכריכה התחתונה בעקבות פרוטוקולים שפורסמו42,43. השתמש N-[3-(trimethoxysilyl)propyl]ethylenediamine עבור silanization ותערובת 1:100 (ww) של ביוטין-PEG-SVA ו- mPEG-SVA במאגר ביקרבונט 100 mM. שמרו את הכיסויים PEGylated יבשים בטמפרטורה של -20°C ואחסנו אותם למשך מספר שבועות.
- ביום הניסויים, הוציאו את כיסויי ה-PEGylated וייבשו אותם באקדח חנקן. בדקו ויזואלית אם יש לכלוך כדי לוודא שהם נקיים.
- כדי ליצור את תעלות הדגימה, הכינו רצועות ברוחב ~2 מ"מ של סרט דו-צדדי והניחו ארבע רצועות על משטח כיסוי תחתון (משטח PEGylated למעלה), במקביל ומופרדות זו מזו על ידי ~5 מ"מ (איור 3C).
הערה: בדרך זו, ניתן ליצור שלושה ערוצי דגימה ברוחב 5 מ"מ בתא זרימה יחיד. - הניחו את החלקת הכיסוי העליונה במרכז הכיסוי התחתון, והותירו ~ 5 מ"מ של מקום בקצוות הקצרים עבור פתחי ערוצים ושקעים. לחץ בעדינות על החלק האחורי של הכיסוי העליון עם פינצטה כדי לאטום היטב את התעלות.
- כדי ליצור מאגר כניסה, קצצו את הקצה של קצה פיפטה של 200 מיקרוליטר. חותכים ~ 10 מ"מ מהפתח הרחב יותר כדי לאפשר אחיזה ~ 200 μL של תמיסה. הכינו שלושה מהם לשלושת ערוצי הזרימה. כדי להגדיר את השקעים, הכינו שלוש מחטי מזרק שמתאימות לצינור עבור משאבת המזרק.
- באמצעות אפוקסי של 5 דקות, מדביקים את המאגרים ורכזות המחטים לתא הזרימה. ודאו שנוצר איטום מלא למניעת דליפה, ושהתעלות אינן חסומות בדבק עודף. תן לו להתייבש לפחות 30 דקות.
7. הרכבה של מבני קשירת חרוזים
הערה: התמיסות של חומרים לניסויי MT, כולל אלה של מבני קשירת חרוזים, מוחדרות ברצף לתאי זרימה באמצעות משאבת מזרק (איור 3C,D).
- הכינו חרוזים מגנטיים. קח 5 מ"ג של חרוזי אפוקסי M270 מתמיסת מלאי (~ 3.3 × 108 חרוזים ב 167.5 מיקרוליטר של דימתילפורממיד) והחלף את הממס בחיץ פוספט (ראה טבלה 2) על ידי הפרדה מגנטית של החרוזים.
- הכינו את החרוזים ב~1.1 × 109 חרוזים מ"ל−1 בחיץ פוספט עם 1 M אמוניום סולפט והגיבו אותם עם 2 mM dibenzocyclooctyne (DBCO)-NH2. דוגרים על התערובת במשך 3 שעות על מיקסר מסתובב בטמפרטורת החדר. לאחר התגובה, שטפו את החרוזים 3x עם חיץ פוספט טרי כדי להסיר מולקולות שלא הגיבו.
הערה: ניתן לאחסן את החרוזים השטופים ללא סיבוב נוסף בטמפרטורה של 4°C למשך מספר שבועות לפני השימוש. - חבר מחט ביציאת תעלת תא הזרימה למשאבת המזרק באמצעות צינורות פוליאתילן. אזנו את הערוצים עם PBS.
- הכניסו את הפתרונות הבאים ברצף לתעלה על ידי שאיבה עם המשאבה: נויטראבידין, מבני מטרה (סיכות ראש DNA או קומפלקסים SNARE עם ידיות DNA), חרוזי פוליסטירן ייחוס וחרוזים מגנטיים מצופים DBCO. לפני השימוש, מערבלים היטב את תמיסות החרוזים כדי לפזר אגרגטים פוטנציאליים של חרוזים.
- שטפו חרוזים לא קשורים תוך הפעלת 0.1 pN של כוח.
הערה: הפעלת כוח קטן כלפי מעלה מקלה על הסרת חרוזים לא קשורים ומסייעת למנוע קרע של מבנים קשורים במיוחד של קשירת חרוזים. - עבור ניסויים עם מתחמי SNARE, כלול 1.5 מיקרומטר SNAP-25 במאגר הסופי.
הערה: מולקולות SNAP-25 החופשיות יכולות לקשור מחדש קומפלקסים של SNARE לאחר הפתיחה ולאפשר מדידות חוזרות ונשנות על קומפלקס יחיד.
8. זיהוי מבני מטרה
- על פני השטח של תעלת תאי זרימה, חפשו את החרוזים המגנטיים הקשורים על ידי מולקולות בודדות של מבנה המטרה. ודא שחרוז הפניה נמצא בקרבת מקום.
- סובבו חרוז מועמד ובדקו שהוא מסתובב בחופשיות. אם החרוז קשור במספר מולקולות, הוא מפגין תנועה מוגבלת.
- סובב את החרוז במשך כמה סיבובים שלמים וגלה את רדיוס הסיבוב (פונקציה זו מיושמת בתוכנה שסופקה). רצוי, לבחור חרוז עם רדיוס סיבוב קטן.
הערה: רדיוס זה מציין עד כמה החרוז מנותק מציר הקשירה, אשר נקבע באופן אקראי במהלך מכלול קשירת החרוזים30,31. בכל הניסויים, מרכוז מינימלי של חרוז מקל על ממצאים רבים הקשורים ליחס ההארכה הגבוה של רדיוס החרוז לקשירה שבו אנו משתמשים. - הגדל את הכוח מ- 0 ל- 5 pN כדי לזהות חרוזים קשורים יחידים טובים. חפש שינוי גדול בתבנית העקיפה של חרוז כתוצאה מתיחה של קשירה של 1 kbp (או שתי ידיות מקבילות של 510 bp). אם דפוס העקיפה אינו משתנה באופן משמעותי, הנמיכו את הכוח לאפס וסרקו חרוז מועמד אחר.
הערה: ניתן להבחין בקלות בהרמת ~300 ננומטר של חרוז מהתמונות הגולמיות מבלי להתחיל את תהליך המעקב בפועל.
9. מעקב חרוזים למדידות הרחבה
הערה: מעקב אחר חרוזים מתבצע על ידי ניתוח תמונות חרוזים בזמן אמת בתוכנת LabVIEW המצורפת למאמר זה. שיטת המעקב וגרסאותיה שימשו ברוב מערכות MT הקונבנציונליות ומוסברות בספרות קודמת 2,5,7,26. על ידי מדידת מיקומו של חרוז מגנטי ביחס לחרוז ייחוס קבוע (כלומר, מעקב דיפרנציאלי), מדידות המיקום הופכות חסינות ביותר להפרעה חיצונית.
- לאחר איתור חרוז מגנטי תקין יחד עם חרוז ייחוס, לחצו על כפתור הכיול כדי להתחיל להתכונן למעקב אחר חרוזים.
- לחץ על החרוזים בתמונה כדי להגדיר את מיקומי החרוזים. לאחר מכן התמונות ייחתכו לאזורי עניין (ROIs) (לדוגמה, 150 x 150 פיקסלים עבור חרוז של 3 מיקרומטר) סביב החרוזים ולאחר מכן ינותחו עוד יותר כדי לחלץ את קואורדינטות החרוזים המדויקות.
- המתן להשלמת סיבוב המגנט. תהליך זה מתעד את קואורדינטות x ו- y של החרוז (על ידי חישוב מתאם צולב דו-ממדי44 או באמצעות סימטריה רדיאלית45 של תמונות החרוז, עם ביצועים דומים) תוך סיבוב המגנטים כדי לתעד את החיבור הלא ממורכז של החרוז31.
- למעקב בכיוון z, המתן עד שהתוכנה תיצור טבלת חיפוש של תמונות עקיפה של החרוזים במרחקים שונים ממישור המוקד. זה מתבצע על ידי דריכת עדשת האובייקט עם סורק piezo בצעדים שווים ורישום תמונות חרוזים ממוצעים תנודתיות בכל מיקום. לאחר מכן, קואורדינטות z של החרוזים בניסויים אמיתיים נקבעות על ידי השוואת תמונות החרוזים בזמן אמת לטבלת החיפוש עם אינטרפולציה7.
- בסיום יצירת טבלת בדיקת המידע, הפעל מעקב ומיקוד אוטומטי (לחץ על Track ? and AF? כפתורים) ולחץ על לרכוש כפתור כדי להתחיל להקליט מיקומי חרוזים.
הערה: מיקוד אוטומטי הוא אופציונלי אך מומלץ לתקן את סטיית השלב ב - z במהלך הרכישה.
10. כפה תוכניות יישום
- ניסויי כבש כוח: כדי לאמת את יחסי הכוח-הארכה של המבנה, הפעילו כבש כוח למעלה ולמטה בקצב העמסה קבוע (± 1.0 pN s−1) (איור 4A). לדוגמה, החל שלושה סבבים של מחזור pN של 0-20-0 כדי לאמת את האורך הכולל של המבנה ואת עקומת הארכת הכוח של נקודות האחיזה.
- על ידי ציון פרמטרי הקשירה בתוכנה, שכבו עקומת הארכת כוח WLC על גבי הנתונים הנמדדים, וקבעו אם חרוז המטרה קשור על ידי מבנה דגימה אמיתי עם ידיות DNA מתאימות. השתמש באורך קווי המתאר הידוע (למשל, ~ 340 ננומטר עבור 1 kbp dsDNA) ובאורך התמדה WLC (30-45 ננומטר עבור dsDNA31 קצר) של המבנה כנקודת התחלה. החל את שיטת תיקון ההרחבה המתוארת בשלב 2.11 במידת הצורך.
- אם המבנה מאומת, בחן בפירוט את תגובת הארכת הכוח כדי לחפש הרחבה נוספת הנובעת ממולקולות המטרה - סיכות שיער או קומפלקסים של SNARE.
- ניסויי כוח קבוע: שנו בהדרגה את הכוח המופעל בצעדים בדידים כדי לבחון את רגישות הכוח של מולקולות המטרה (איור 4B).
הערה: MTs מאפשרים ניסויים פשוטים ויעילים בכוח קבוע מכיוון שהכוח המופעל נשמר קבוע כאשר המגנטים מוחזקים במקום.- עבור סיכות שיער DNA, יש להפעיל 4-8 pN של כוח עם צעדים של 0.2-0.5 pN, ולמדוד את מיקום החרוז עבור ~ 10 שניות בכל רמת כוח.
- עבור מתחמי SNARE, החל 14-16 pN של כוח עם צעדים 0.1-0.2 pN, ולמדוד את מיקום החרוז עבור ~ 10 s בכל רמת כוח.
- ניסויי קפיצת כוח: שימו לב לאירועי המעבר של מתחמי SNARE.
הערה: ניסויים בקפיצת כוח, כמו ניסויים בכוח קבוע, כרוכים בשינויים ברמות הכוח. עם זאת, קפיצות כוח משתמשות בשינויים פתאומיים יותר בכוח המופעל, ומאפשרות ניטור של אירועים המופעלים על ידי כוח במולקולות הנחקרות, כגון קרע פתאומי של קומפלקסים חלבוניים. לדוגמה, מאחר שקומפלקסים של SNARE מפגינים היסטרזה מבנית במחזורכוח 23, זה אינפורמטיבי לבצע ניסויים בקפיצת כוח ולמדוד את ההשהיה למעבר (איור 4C).- פתיחת רוכסן: קילוף מולקולת VAMP2 מקומפלקס SNARE טרינרי שלם, ומותיר קומפלקס בינארי של תחביר-1A ו-SNAP-25.
- רוכסן: רוכסן של מולקולת VAMP2 ללא רוכסן כדי ליצור קומפלקס SNARE שלם.
- מתגלגל: פירוק מלא של קומפלקס SNARE מלווה בדיסוציאציה מוחלטת של SNAP-25. רק מולקולות VAMP2 ותחביר נשארות במבנה לאחר ההתפתחות.
- קיפול מחדש: התחדשות של קומפלקס SNARE עם קשירה של מולקולת SNAP-25 חופשית מהחיץ.
- ב 2 pN, לגרום להרכבה של קומפלקס SNARE שלם על ידי המתנה (~ 30 שניות) עבור שיוך של מולקולת SNAP25 חופשית. ירידה פתאומית בהרחבה נצפתה עם היווצרות קומפלקס SNARE.
- כדי לצפות באירועי פתיחת רוכסן, המתן מספר שניות ב- 10-12 pN, ולאחר מכן עבור ל- 14-15 pN בפתאומיות עם מהירות המנוע המרבית האפשרית. בהתאם לכוח המטרה, קומפלקס SNARE יציג מעבר הפיך בין מתווכים לא מכווצים חלקית (כמו בניסויי כוח קבוע) או קפיצה ~25 ננומטר למצב גבוה יותר, ללא רוכסן לאחר זמן המתנה אקראי (או השהיה).
- כדי לצפות באירועי רוכסן, הנמיכו את הכוח ל 10-12 pN מיד לאחר פתיחת הרוכסן. שוב, קומפלקס SNARE מציג מעבר סטוכסטי למצב התחתון עם הרוכסן לאחר השהיה אקראית כלשהי. אם הפתיחה התרחשה לאחר פתיחת הרוכסן, המתחם לא יצליח לדחוס, מכיוון שמולקולת SNAP-25 תהיה חסרה.
- כדי לצפות באירועים מתפתחים, המתן פרק זמן ארוך יותר לאחר פתיחת הרוכסן נצפה כדי לזהות עלייה נוספת בהרחבה (~ 2 ננומטר).
11. ניתוח נתונים
הערה: סוגי הניתוח שניתן לבצע עם נתוני MT תלויים במערכת היעד. אולם ישנן גישות נפוצות לחילוץ מידע שימושי מהניסויים המתאימים המתוארים באיור 4. כל הניתוחים מבוצעים באמצעות MATLAB (R2021a) באמצעות הקודים המותאמים אישית שסופקו במאמר זה. קודים אלה יוצרים מגרשים באמצעות אותם נתונים המוצגים במאמר זה. שים לב שבעוד שנתונים גולמיים ממעקב של 100 הרץ נלקחו ישירות לניתוח, נתונים ממעקב של 1.2 קילוהרץ היו בדרך כלל מסוננים בחציון (עם חלון הזזה של חמש נקודות) לפני הניתוח כדי להפחית את הרעש (למעט ניתוח רעש).
- ניסויי כבש כוח: לנתח את יחסי הכוח-הארכה (למשל, גמישות של פולימרים) ולהעביר את הכוח כדי לחלץ מידע על תכונות ננומכניות.
- ניסויי כוח קבוע: לנתח אוכלוסיות מצב וזמן שהייה (או קצב מעבר) כפונקציה של כוח כדי לחלץ פרמטרים מבניים (למשל, אזורים המעורבים במעבר), תרמודינמיים (למשל, הפרשי אנרגיה חופשית) וקינטיים (למשל, מחסום אנרגיה) של השינויים הקונפורמטיביים.
- ניסויי קפיצת כוח: לנתח קינטיקה של קרע (למשל, אינטראקציות חלבון-חלבון וקישור קולטן-ליגנד) או את משך החיים של מתווכים חולפים (למשל, התפתחות של ביומולקולות) כדי לחלץ את היציבות של מולקולות המטרה ואת מצבן.
- כיישומים מייצגים, נתח את נתוני הדגימה עבור סיכות ראש DNA וקומפלקסים SNARE:
- מעברים דו-מדינתיים של סיכת ראש של DNA: פתיחת כוח, מרחק פתיחה, תלות בכוח של הסטת אוכלוסייה, ומדידות הקצאה וקצב מעבר של מצב עם מודל מרקוב נסתר (HMM) (קודי MATLAB מסופקים).
- שינויים קונפורמטיביים של קומפלקסים SNARE: כוח רוכסן, תלות בכוח של מצבי ביניים וחביון פתיחת רוכסן, היסטרזיס ב rezipping, והתנהגות פתיחה/קיפול מחדש.
הערה: מודלים של הארכת כוח עבור ידיות DNA, סיכות שיער של DNA וקונפורמציות מורכבות של SNARE ניתנים בהפניות קודמות14,31.
תוצאות
כיול כוח
התוצאות משתי שיטות מדידת הכוח (שונות התזוזה הצידית של חרוזים וניתוח ספקטרום הספקטרום) היו שונות ב-0-2 pN (איור 2G). לפי התוצאות באיור 2F, אנו יכולים להגיע באופן אמין עד 30 pN עם מגנטים רגילים של ניאודימיום.
מעברים דו-מדינתיים ש...
Discussion
בעבודה זו הצגנו מערך ספקטרוסקופיית כוח של מולקולה בודדת שיכול לצפות בשינויים מבניים של ביומולקולות בדיוק מרחבי-זמני גבוה. מצלמת ה-CMOS המהירה שבה נעשה שימוש מקבלת 1,200 פריימים s−1 ברזולוציה של 1,280 x 1,024, ומאפשרת מעקב חרוזים של 1.2 קילוהרץ. עם זאת, מהירות המדידות מוגבלת כיום על ידי תוכנת מעק?...
Disclosures
למחברים אין ניגודי עניינים להצהיר.
Acknowledgements
עבודה זו נתמכה על ידי מענק קרן המחקר הלאומית של קוריאה (NRF) במימון ממשלת קוריאה (MSIT) (NRF-2022R1C1C1012176, NRF-2021R1A4A1031754 ו-NRF- 2021R1A6A1A10042944). S.-H.R. נתמך על ידי מענק NRF (2021R1C1C2009717).
Materials
| Name | Company | Catalog Number | Comments |
| Materials for construct synthesis | |||
| Agarose gel electrophoresis system | Advance | Mupid-2plus | |
| DNA ladder | Bioneer | D-1037 | |
| nTaq polymerase | Enzynomics | P050A | |
| PCR purification kit | LaboPass | CMR0112 | |
| PEGylated SMCC crosslinker / SM(PEG)2 | ThermoFisher Scientific | 22102 | For SNARE–DNA coupling |
| Primer B | Bioneer | 5'-Biotin/TCGCCACCATCATTTCCA-3' | For 5-kbp force calibration construct and DNA handles |
| Primer B_hp | IDT | 5'-Biotin/TTTTTTTTTTGTTCTCTATTT TTTTAGAGAAC /AP site/ /AP site/ TCGCCACCATCATTTCCA-3' | For hairpin construct |
| Primer N | Bioneer | 5'-C6Amine/CATGTGGGTGACGCGAAA-3' | For DNA handles |
| Primer Z | Bioneer | 5'-Azide/TCGCCACCATCATTTCCA-3' | For DNA handles |
| Primer Z_5k | Bioneer | 5'-Azide/TTAGAGAGTATGGGTATATGACA TCG-3' | For 5-kbp force calibration construct |
| Primer Z_hp | Bioneer | 5'-Azide/GTGGCAGCATGACACC-3' | For hairpin construct |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| λ-DNA | Bioneer | D-2510 | Template strand for PCR |
| DNA sequences for SNARE proteins | |||
| 6×His-tagged SNAP-25b (2-206; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctat gagaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagcgg aagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCACA GCAGCGGCCTGGTGCCGCGC GGCAGCCATACTAGCGGAGAT ATCGCCGAGGACGCAGACAT GCGCAATGAGCTGGAGGAGA TGCAGAGGAGGGCTGACCAG CTGGCTGATGAGTCCCTGGA AAGCACCCGTCGCATGCTGC AGCTGGTTGAAGAGAGTAAA GATGCTGGCATCAGGACTTT GGTTATGTTGGATGAGCAAG GCGAACAACTGGAACGCATT GAGGAAGGGATGGACCAAAT CAATAAGGACATGAAAGAAG CAGAAAAGAATTTGACGGAC CTAGGAAAATTCGCCGGCCT TGCCGTGGCCCCCGCCAAC AAGCTTAAATCCAGTGATGC TTACAAAAAAGCCTGGGGC AATAATCAGGATGGAGTAGT GGCCAGCCAGCCTGCCCG TGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTG GCTTCATCCGCAGGGTAAC AAATGATGCCCGGGAAAAT GAGATGGATGAGAACCTG GAGCAGGTGAGCGGCATC ATCGGAAACCTCCGCCAC ATGGCTCTAGACATGGGCA ATGAGATTGACACCCAGA ATCGCCAGATCGACAGGA TCATGGAGAAGGCTGATT CCAACAAAACCAGAATTG ATGAAGCCAACCAACGTG CAACAAAGATGCTGGGAA GTGGTTAAggatccgaattcgag ctccgtcgacaagcttgcggccgcactc gagcaccaccaccaccaccactgagat ccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgag caataactagcataaccccttggggcct ctaaacgggtcttgaggggttttttgctga aaggaggaactatatccggat | |
| 6×His-tagged VAMP2 (2-97, L32C/I97C; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCAC AGCAGCGGCCTGGTGCCGC GCGGCAGCCATATGGCAGAT CTCTCGGCTACCGCTGCCAC CGTCCCGCCTGCCGCCCCG GCCGGCGAGGGTGGCCCCC CTGCACCTCCTCCAAATCTTA CCAGTAACAGGAGATGCCAG CAGACCCAGGCCCAGGTGG ATGAGGTGGTGGACATCATG AGGGTGAATGTGGACAAGGT CCTGGAGCGAGACCAGAAG CTATCGGAACTGGATGATCG CGCAGATGCCCTCCAGGCA GGGGCCTCCCAGTTTGAAA CAAGTGCAGCCAAGCTCAA GCGCAAATACTGGTGGAAA AACCTCAAGATGATGTGCTA Aggatccgaattcgagctccgtcg acaagcttgcggccgcactcgagcaccacca ccaccaccactgagatccggctgctaacaaa gcccgaaaggaagctgagttggctgctgcca ccgctgagcaataactagcataaccccttgg ggcctctaaacgggtcttgaggggttttttg ctgaaaggaggaactatatccggat | |
| 6×His-tagged ΔN-VAMP2 (49–96; capitalized) and Syntaxin-1A (191–267, I202C/I266C; capitalized) in pETDuet-1 | homemade | ggggaattgtgagcggataacaattcccctc tagaaataattttgtttaactttaagaagga gatataccATGGGCAGCAGCCATCA TCATCATCATCACAGCAGCGG CCTGGAAGTTCTGTTCCAGGG GCCCGGTAATGTGGACAAGGT CCTGGAGCGAGACCAGAAGCT ATCGGAACTGGATGATCGCGC AGATGCCCTCCAGGCAGGGGC CTCCCAGTTTGAAACAAGTGC AGCCAAGCTCAAGCGCAAATAC TGGTGGAAAAACCTCAAGATGAT GTAAgcggccgcataatgcttaagtcgaaca gaaagtaatcgtattgtacacggccgcataa tcgaaattaatacgactcactataggggaat tgtgagcggataacaattccccatcttagta tattagttaagtataagaaggagatatacat ATGGCCCTCAGTGAGATCGAGA CCAGGCACAGTGAGTGCATC AAGTTGGAGAACAGCATCCG GGAGCTACACGATATGTTCAT GGACATGGCCATGCTGGTGG AGAGCCAGGGGGAGATGATT GACAGGATCGAGTACAATGTG GAACACGCTGTGGACTACGTG GAGAGGGCCGTGTCTGACACC AAGAAGGCCGTCAAGTACCAG AGCAAGGCACGCAGGAAGAA GTGCATGATCTAActcgagtc tggtaaagaaaccgctgctgcgaaatttgaa cgccagcacatggactcgtctactagcgcag cttaattaacctaggctgctgccaccgctga gcaataactagcataaccccttggggcctct aaacgggtcttgaggggttttttgctgaaag gaggaactatatccggattggcgaatgggac gcgccctgtagcggcgcattaagcgcggcgg gtgtggtggttacgcgcagcgtgaccgctac acttgccagcgccctagcgcccgctcctttc gctttcttcccttcctttctcgccacgttcg ccggctttccccgtcaagctctaaatcgggg gctccctttagggttccgatttagtgcttta cggcacctcgaccccaaaaaacttgattagg gtgatggttcacgtagtgggccatcgccctg atagacggtttttcgccctttgacgttggag tccacgttctttaatagtggactcttgttcc aaactggaacaacactcaaccctatctcggt ctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctga tttaacaaaaatttaacgcgaattttaacaa aatattaacgtttacaatttctggcggcacg atggcatgagattatcaaaaaggatcttcac ctagatccttttaaattaaaaatgaagtttt aaatcaatctaaagtatatatgagtaaactt ggtctgacagttaccaatgcttaatcagtga ggcacctatctcagcgatctgtctatttcgt tcatccatagttgcctgactccccgtcgtgt agataactacgatacgggagggcttaccatc tggccccagtgctgcaatgataccgcgagac ccacgctcaccggctccagatttatcagcaa taaaccagccagccggaagggccgagcgca gaagtggtcctgcaactttatccgcctccatc cagtctattaattgttgccgggaagctagag taagtagttcgccagttaatagtttgcgcaa cgttgttgccattgctacaggcatcgtggtg tcacgctcgtcgtttggtatggcttcattca gctccggttcccaacgatcaaggcgagttac atgatcccccatgttgtgcaaaaaagcggtt agctccttcggtcctccgatcgttgtcagaa gtaagttggccgcagtgttatcactcatggt tatggcagcactgcataattctcttactgtc atgccatccgtaagatgcttttctgtgactg gtgagtactcaaccaagtcattctgagaata gtgtatgcggcgaccgagttgctcttgcccg gcgtcaatacgggataataccgcgccacata gcagaactttaaaagtgctcatcattggaaa acgttcttcggggcgaaaactctcaaggatc ttaccgctgttgagatccagttcgatgtaac ccactcgtgcacccaactgatcttcagcatc ttttactttcaccagcgtttctgggtgagcaaa aacaggaaggcaaaatgccgcaaaaaagg gaataagggcgacacggaaatgttgaatact catactcttcctttttcaatcatgattgaag catttatcagggttattgtctcatgagcgga tacatatttgaatgtatttagaaaaataaac aaataggtcatgaccaaaatcccttaacgtg agttttcgttccactgagcgtcagaccccgt agaaaagatcaaaggatcttcttgagatcct ttttttctgcgcgtaatctgctgcttgcaaa caaaaaaaccaccgctaccagcggtggtttg tttgccggatcaagagctaccaactcttttt ccgaaggtaactggcttcagcagagcgcaga taccaaatactgtccttctagtgtagccgta gttaggccaccacttcaagaactctgtagca ccgcctacatacctcgctctgctaatcctgt taccagtggctgctgccagtggcgataagtc gtgtcttaccgggttggactcaagacgatag ttaccggataaggcgcagcggtcgggctgaa cggggggttcgtgcacacagcccagcttgga gcgaacgacctacaccgaactgagataccta cagcgtgagctatgagaaagcgccacgcttccc gaagggagaaaggcggacaggtatccggta agcggcagggtcggaacaggagagcgcac gagggagcttccagggggaaacgcctggtatc tttatagtcctgtcgggtttcgccacctctg acttgagcgtcgatttttgtgatgctcgtca ggggggcggagcctatggaaaaacgccagc aacgcggcctttttacggttcctggccttttg ctggccttttgctcacatgttctttcctgcg ttatcccctgattctgtggataaccgtatta ccgcctttgagtgagctgataccgctcgccgc agccgaacgaccgagcgcagcgagtcagtg agcgaggaagcggaagagcgcctgatgcgg tattttctccttacgcatctgtgcggtatttc acaccgcatatatggtgcactctcagtacaa tctgctctgatgccgcatagttaagccagta tacactccgctatcgctacgtgactgggtca tggctgcgccccgacacccgccaacacccgc tgacgcgccctgacgggcttgtctgctcccg gcatccgcttacagacaagctgtgaccgtct ccgggagctgcatgtgtcagaggttttcacc gtcatcaccgaaacgcgcgaggcagctgcgg taaagctcatcagcgtggtcgtgaagcgatt cacagatgtctgcctgttcatccgcgtccag ctcgttgagtttctccagaagcgttaatgtc tggcttctgataaagcgggccatgttaaggg cggttttttcctgtttggtcactgatgcctc cgtgtaagggggatttctgttcatgggggta atgataccgatgaaacgagagaggatgctca cgatacgggttactgatgatgaacatgcccg gttactggaacgttgtgagggtaaacaactg gcggtatggatgcggcgggaccagagaaaaa tcactcagggtcaatgccagcgcttcgttaa tacagatgtaggtgttccacagggtagccag cagcatcctgcgatgcagatccggaacataa tggtgcagggcgctgacttccgcgtttccag actttacgaaacacggaaaccgaagaccatt catgttgttgctcaggtcgcagacgttttgc agcagcagtcgcttcacgttcgctcgcgtat cggtgattcattctgctaaccagtaaggcaa ccccgccagcctagccgggtcctcaacgaca ggagcacgatcatgctagtcatgccccgcgc ccaccggaaggagctgactgggttgaaggct ctcaagggcatcggtcgagatcccggtgcct aatgagtgagctaacttacattaattgcgtt gcgctcactgcccgctttccagtcgggaaac ctgtcgtgccagctgcattaatgaatcggcc aacgcgcggggagaggcggtttgcgtattgg gcgccagggtggtttttcttttcaccagtga gacgggcaacagctgattgcccttcaccgcc tggccctgagagagttgcagcaagcggtcca cgctggtttgccccagcaggcgaaaatcctg tttgatggtggttaacggcgggatataacat gagctgtcttcggtatcgtcgtatcccacta ccgagatgtccgcaccaacgcgcagcccgga ctcggtaatggcgcgcattgcgcccagcgcc atctgatcgttggcaaccagcatcgcagtgg gaacgatgccctcattcagcatttgcatggt ttgttgaaaaccggacatggcactccagtcg ccttcccgttccgctatcggctgaatttgat tgcgagtgagatatttatgccagccagccag acgcagacgcgccgagacagaacttaatggg cccgctaacagcgcgatttgctggtgaccca atgcgaccagatgctccacgcccagtcgcgt accgtcttcatgggagaaaataatactgttg atgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccac agcaatggcatcctggtcatccagcggatag ttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttc gacgccgcttcgttctaccatcgacaccacc acgctggcacccagttgatcggcgcgagatt taatcgccgcgacaatttgcgacggcgcgtg cagggccagactggaggtggcaacgccaatc agcaacgactgtttgcccgccagttgttgtg ccacgcggttgggaatgtaattcagctccgc catcgccgcttccactttttcccgcgttttc gcagaaacgtggctggcctggttcaccacgc gggaaacggtctgataagagacaccggcata ctctgcgacatcgtataacgttactggtttc acattcaccaccctgaattgactctcttccg ggcgctatcatgccataccgcgaaaggtttt gcgccattcgatggtgtccgggatctcgacg ctctcccttatgcgactcctgcattaggaag cagcccagtagtaggttgaggccgttgagca ccgccgccgcaaggaatggtgcatgcaagga gatggcgcccaacagtcccccggccacgggg cctgccaccatacccacgccgaaacaagcgc tcatgagcccgaagtggcgagcccgatcttc cccatcggtgatgtcggcgatataggcgcca gcaaccgcacctgtggcgccggtgatgccgg ccacgatgcgtccggcgtagaggatcgagat cgatctcgatcccgcgaaattaatacgactc actata | |
| SNAP-25b (1–206, all C to A; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcc agggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGCCGA GGACGCAGACATGCGCAATG AGCTGGAGGAGATGCAGAGG AGGGCTGACCAGCTGGCTGA TGAGTCCCTGGAAAGCACCC GTCGCATGCTGCAGCTGGTT GAAGAGAGTAAAGATGCTGG CATCAGGACTTTGGTTATGTT GGATGAGCAAGGCGAACAAC TGGAACGCATTGAGGAAGGG ATGGACCAAATCAATAAGGAC ATGAAAGAAGCAGAAAAGAAT TTGACGGACCTAGGAAAATTC GCCGGCCTTGCCGTGGCCCC CGCCAACAAGCTTAAATCCAG TGATGCTTACAAAAAAGCCTG GGGCAATAATCAGGATGGAGT AGTGGCCAGCCAGCCTGCCC GTGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTGGC TTCATCCGCAGGGTAACAAAT GATGCCCGGGAAAATGAGATG GATGAGAACCTGGAGCAGGT GAGCGGCATCATCGGAAACCT CCGCCACATGGCTCTAGACAT GGGCAATGAGATTGACACCCA GAATCGCCAGATCGACAGGAT CATGGAGAAGGCTGATTCCAA CAAAACCAGAATTGATGAAGC CAACCAACGTGCAACAAAGAT GCTGGGAAGTGGTTAA ctcgagcaccaccaccaccaccactgag atccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgagc aataactagcataaccccttggggcctc taaacgggtcttgaggggttttttgctgaa aggaggaactatatccggat | |
| Materials for protein purificaiton | |||
| 2-Mercaptoethanol | SIGMA | M3148-25ML | |
| Agar | LPS Solution | AGA500 | |
| Ampicillin, Sodium salt | PLS | AC1043-005-00 | |
| Chloramphenicol | PLS | CR1023-050-00 | |
| Competent cells (E. coli) | Novagen | 70956 | Rosetta(DE3)pLysS |
| Glycerol | SIGMA | G5516-500ML | |
| HEPES | SIGMA | H4034-100G | |
| Hydrochloric acid / HCl | SIGMA | 320331-500ML | |
| Imidazole | SIGMA | I2399-100G | |
| Isopropyl β-D-1-thiogalactopyranoside / IPTG | SIGMA | 10724815001 | |
| Kanamycin Sulfate | PLS | KC1001-005-02 | |
| Luria-Bertani (LB) Broth | LPS Solution | LB-05 | |
| Ni-NTA resin | Qiagen | 30210 | |
| PD MiniTrap G-25 (desalting column) | Cytiva | GE28-9180-07 | For instructions, see: https://www.cytivalifesciences.com/en/us/shop/chromatography/prepacked-columns/desalting-and-buffer-exchange/pd-minitrap-desalting-columns-with-sephadex-g-25-resin-p-06174 |
| Phenylmethylsulfonyl fluoride / PMSF | ThermoFisher Scientific | 36978 | |
| Plasmids for SNARE proteins | cloned in house | N/A | Available upon request |
| Protease inhibitor cocktail | genDEPOT | P3100 | |
| Sodium chloride | SIGMA | S5886-500G | |
| Sodium phosphate dibasic / Na2HPO4 | SIGMA | S7907-100G | |
| Sodium phosphate monobasic / NaH2PO4 | SIGMA | S3139-250G | |
| Tris(2-carboxyethyl)phosphine / TCEP | SIGMA | C4706-2G | |
| Trizma base | SIGMA | T1503-250G | |
| Materials for sample assembly | |||
| Biotin-PEG-SVA | LAYSAN BIO | BIO-PEG-SVA-5K-100MG & MPEG-SVA-5K-1g | For PEGylation |
| Dibenzocyclooctyne-amine / DBCO-NH2 | SIGMA | 761540-10MG | For bead coating |
| Double-sided tape | 3M | 136 | For flow cell assembly |
| Epoxy glue | DEVCON | S-208 | For flow cell assembly |
| Glass coverslip for bottom surface | VWR | 48393-251 | Rectangular, 60×24 mm, #1.5 |
| Glass coverslip for top surface | VWR | 48393-241 | Rectangular, 50×24 mm, #1.5 |
| Magnetic bead | ThermoFisher Scientific | 14301 | Dynabeads M-270 Epoxy, 2.8 μm |
| mPEG-SVA | LAYSAN BIO | mPEG-SVA 1g | For PEGylation |
| N,N-Dimethylformamide / DMF | SIGMA | D4551-250ML | For bead coating |
| N-[3-(trimethoxysilyl)propyl]ethylenediamine | SIGMA | 104884-100ML | For PEGylation |
| Neutravidin | ThermoFisher Scientific | 31000 | For sample tethering |
| Phosphate buffered saline / PBS, pH 7.2 | PLS | PR2007-100-00 | |
| Plastic syringe | Norm-ject | A5 | 5 ml, luer tip |
| Polyethylene Tubing | SCI | BB31695-PE/4 | PE-60 |
| Reference bead | SPHEROTECH | SVP-30-5 | Streptavidin-coated Polystyrene Particles; 3.0-3.4 µm |
| Syringe needle | Kovax | 21G-1 1/4'' | 21 G |
| Syringe pump | KD SCIENTIFIC | 788210 | |
| Equipment for magnetic tweezer instrument | |||
| 1-axis motorized microtranslation stage | PI | M-126.PD1 | For vertical positioning of magnets |
| 2-axis manual translation stage | ST1 | LEE400 | For alignment of magnets to the optical axis |
| Acrylic holder for magnets | DaiKwang Precision | custum order | Drawing available upon request |
| Frame grabber | Active Silicon | AS-FBD-4XCXP6-2PE8 | |
| High-speed CMOS camera | Mikrotron | EoSens 3CXP | |
| Inverted microscope | Olympus | IX73P2F-1-2 | |
| Neodymium magnets | LG magnet | ND 10x10x12t | Dimension: 10 mm × 10 mm × 12 mm; two needed |
| Objective lens | Olympus | UPLXAPO100XO | Oil-immersion, NA 1.45 |
| Objective lens nanopositioner | Mad City Labs | Nano-F100S | |
| Rotation stepper motor | AUTONICS | A3K-S545W | For rotating magnets |
| Superluminescent diode | QPHOTONICS | QSDM-680-2 | 680 nm |
| Software | |||
| LabVIEW | National Instruments | v20.0f1 | |
| MATLAB | MathWorks | v2021a |
References
- Le, S., Liu, R., Lim, C. T., Yan, J. Uncovering mechanosensing mechanisms at the single protein level using magnetic tweezers. Methods. 94, 13-18 (2016).
- Choi, H. -. K., Kim, H. G., Shon, M. J., Yoon, T. -. Y. High-resolution single-molecule magnetic tweezers. Annual Review of Biochemistry. 91 (1), 33-59 (2022).
- Yang, T., Park, C., Rah, S. -. H., Shon, M. J. Nano-precision tweezers for mechanosensitive proteins and beyond. Molecules and Cells. 45 (1), 16-25 (2022).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- De Vlaminck, I., Dekker, C. Recent advances in magnetic tweezers. Annual Review of Biophysics. 41 (1), 453-472 (2012).
- Bustamante, C. J., Chemla, Y. R., Liu, S., Wang, M. D. Optical tweezers in single-molecule biophysics. Nature Reviews Methods Primers. 1, 25 (2021).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical Journal. 82 (6), 3314-3329 (2002).
- Smith, S. B., Finzi, L., Bustamante, C. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258 (5085), 1122-1126 (1992).
- Lansdorp, B. M., Tabrizi, S. J., Dittmore, A., Saleh, O. A. A high-speed magnetic tweezer beyond 10,000 frames per second. Review of Scientific Instruments. 84 (4), 044301 (2013).
- Cnossen, J. P., Dulin, D., Dekker, N. H. An optimized software framework for real-time, high-throughput tracking of spherical beads. Review of Scientific Instruments. 85 (10), 103712 (2014).
- Dulin, D., et al. High spatiotemporal-resolution magnetic tweezers: calibration and applications for DNA dynamics. Biophysical Journal. 109 (10), 2113-2125 (2015).
- Huhle, A., et al. Camera-based three-dimensional real-time particle tracking at kHz rates and Ångström accuracy. Nature Communications. 6 (1), 5885 (2015).
- Popa, I., et al. A HaloTag anchored ruler for week-long studies of protein dynamics. Journal of the American Chemical Society. 138 (33), 10546-10553 (2016).
- Shon, M. J., Kim, H., Yoon, T. -. Y. Focused clamping of a single neuronal SNARE complex by complexin under high mechanical tension. Nature Communications. 9 (1), 3639 (2018).
- Tapia-Rojo, R., Eckels, E. C., Fernández, J. M. Ephemeral states in protein folding under force captured with a magnetic tweezers design. Proceedings of the National Academy of Sciences. 116 (16), 7873-7878 (2019).
- Löf, A., et al. Multiplexed protein force spectroscopy reveals equilibrium protein folding dynamics and the low-force response of von Willebrand factor. Proceedings of the National Academy of Sciences. 116 (38), 18798-18807 (2019).
- Tapia-Rojo, R., Alonso-Caballero, A., Fernandez, J. M. Direct observation of a coil-to-helix contraction triggered by vinculin binding to talin. Science Advances. 6 (21), (2020).
- Rieu, M., et al. Parallel, linear, and subnanometric 3D tracking of microparticles with Stereo Darkfield Interferometry. Science Advances. 7 (6), (2021).
- Rieu, M., Valle-Orero, J., Ducos, B., Allemand, J. -. F., Croquette, V. Single-molecule kinetic locking allows fluorescence-free quantification of protein/nucleic-acid binding. Communications Biology. 4 (1), 1083 (2021).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences. 103 (16), 6190-6195 (2006).
- Camunas-Soler, J., Ribezzi-Crivellari, M., Ritort, F. Elastic properties of nucleic acids by single-molecule force spectroscopy. Annual Review of Biophysics. 45 (1), 65-84 (2016).
- Südhof, T. C., Rothman, J. E. Membrane fusion: grappling with SNARE and SM proteins. Science. 323 (5913), 474-477 (2009).
- Gao, Y., et al. Single reconstituted neuronal SNARE complexes zipper in three distinct stages. Science. 337 (6100), 1340-1343 (2012).
- Zorman, S., et al. Common intermediates and kinetics, but different energetics, in the assembly of SNARE proteins. eLife. 3, e03348 (2014).
- Zhang, Y., Hughson, F. M. Chaperoning SNARE folding and assembly. Annual Review of Biochemistry. 90 (1), 581-603 (2021).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H. Magnetic tweezers for single-molecule experiments. Handbook of Single-Molecule Biophysics. , 371-395 (2009).
- You, H., Le, S., Chen, H., Qin, L., Yan, J. Single-molecule manipulation of G-quadruplexes by magnetic tweezers. Journal of Visualized Experiments. (127), e56328 (2017).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical Journal. 96 (12), 5040-5049 (2009).
- Dulin, D., Barland, S., Hachair, X., Pedaci, F. Efficient illumination for microsecond tracking microscopy. PLoS One. 9 (9), e107335 (2014).
- Klaue, D., Seidel, R. Torsional stiffness of single superparamagnetic microspheres in an external magnetic field. Physical Review Letters. 102 (2), 028302 (2009).
- Shon, M. J., Rah, S. -. H., Yoon, T. -. Y. Submicrometer elasticity of double-stranded DNA revealed by precision force-extension measurements with magnetic tweezers. Science Advances. 5 (6), 1697 (2019).
- Czerwinski, F., Richardson, A. C., Oddershede, L. B. Quantifying noise in optical tweezers by Allan variance. Optics Express. 17 (15), 13255-13269 (2009).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. Review of Scientific Instruments. 83 (2), 025115 (2012).
- Ostrofet, E., Papini, F. S., Dulin, D. High spatiotemporal resolution data from a custom magnetic tweezers instrument. Data in Brief. 30, 105397 (2020).
- Yu, Z., et al. A force calibration standard for magnetic tweezers. Review of Scientific Instruments. 85 (12), 123114 (2014).
- Strick, T. R., Allemand, J. -. F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271 (5257), 1835-1837 (1996).
- Daldrop, P., Brutzer, H., Huhle, A., Kauert, D. J., Seidel, R. Extending the range for force calibration in magnetic tweezers. Biophysical Journal. 108 (10), 2550-2561 (2015).
- te Velthuis, A. J. W., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative guidelines for force calibration through spectral analysis of magnetic tweezers data. Biophysical Journal. 99 (4), 1292-1302 (2010).
- Ostrofet, E., Papini, F. S., Dulin, D. Correction-free force calibration for magnetic tweezers experiments. Scientific Reports. 8 (1), 15920 (2018).
- Seol, Y., Li, J., Nelson, P. C., Perkins, T. T., Betterton, M. D. Elasticity of short DNA molecules: theory and experiment for contour lengths of 0.6-7 µm. Biophysical Journal. 93 (12), 4360-4373 (2007).
- Burnham, D. R., Vlaminck, I. D., Henighan, T., Dekker, C. Skewed Brownian fluctuations in single-molecule magnetic tweezers. PLoS One. 9 (9), 108271 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protocols. 3 (1), 101152 (2022).
- Lee, H. -. W., et al. Profiling of protein-protein interactions via single-molecule techniques predicts the dependence of cancers on growth-factor receptors. Nature Biomedical Engineering. 2 (4), 239-253 (2018).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Parthasarathy, R. Rapid, accurate particle tracking by calculation of radial symmetry centers. Nature Methods. 9 (7), 724-726 (2012).
- Woodside, M. T., Block, S. M. Reconstructing folding energy landscapes by single-molecule force spectroscopy. Annual Review of Biophysics. 43 (1), 19-39 (2014).
- Evans, E., Ritchie, K. Dynamic strength of molecular adhesion bonds. Biophysical Journal. 72 (4), 1541-1555 (1997).
- Zhang, Y. Energetics, kinetics, and pathway of SNARE folding and assembly revealed by optical tweezers. Protein Science. 26 (7), 1252-1265 (2017).
- Chen, H., et al. Improved high-force magnetic tweezers for stretching and refolding of proteins and short DNA. Biophysical Journal. 100 (2), 517-523 (2011).
- Cho, S., et al. Tension exerted on cells by magnetic nanoparticles regulates differentiation of human mesenchymal stem cells. Biomaterials Advances. 139, 213028 (2022).
- Shon, M. J., Cohen, A. E. Nano-mechanical measurements of protein-DNA interactions with a silicon nitride pulley. Nucleic Acids Research. 44 (1), 7 (2016).
- Cheng, Y. Single-particle cryo-EM-How did it get here and where will it go. Science. 361 (6405), 876-880 (2018).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Neupane, K., et al. Direct observation of transition paths during the folding of proteins and nucleic acids. Science. 352 (6282), 239-242 (2016).
- Choi, H. -. K., et al. Watching helical membrane proteins fold reveals a common N-to-C-terminal folding pathway. Science. 366 (6469), 1150-1156 (2019).
- Kim, C., et al. Extreme parsimony in ATP consumption by 20S complexes in the global disassembly of single SNARE complexes. Nature Communications. 12 (1), 3206 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved