É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Pinças magnéticas de alta velocidade para medições nanomecânicas em elementos sensíveis à força
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, descrevemos uma configuração de pinça magnética de alta velocidade que realiza medições nanomecânicas em biomoléculas sensíveis à força na taxa máxima de 1,2 kHz. Apresentamos sua aplicação a pinos de DNA e complexos SNARE como sistemas modelo, mas também será aplicável a outras moléculas envolvidas em eventos mecanobiológicos.
Resumo
Pinças magnéticas de molécula única (MTs) têm servido como ferramentas poderosas para interrogar com força biomoléculas, como ácidos nucléicos e proteínas, e, portanto, estão prontas para serem úteis no campo da mecanobiologia. Uma vez que o método geralmente se baseia no rastreamento baseado em imagens de esferas magnéticas, o limite de velocidade no registro e análise de imagens, bem como as flutuações térmicas das contas, há muito tempo dificultam sua aplicação na observação de pequenas e rápidas mudanças estruturais em moléculas-alvo. Este artigo descreve métodos detalhados para a construção e operação de uma configuração MT de alta resolução que pode resolver a dinâmica de nanoescala e milissegundos de biomoléculas e seus complexos. Como exemplos de aplicação, experimentos com pinos de cabelo de DNA e complexos SNARE (máquinas de fusão por membrana) são demonstrados, com foco em como seus estados transitórios e transições podem ser detectados na presença de forças em escala de piconewton. Esperamos que as MTs de alta velocidade continuem a permitir medições nanomecânicas de alta precisão em moléculas que detectam, transmitem e geram forças nas células e, assim, aprofundam nossa compreensão em nível molecular da mecanobiologia.
Introdução
As células sentem e respondem ativamente a estímulos mecânicos. Ao fazer isso, muitas biomoléculas exibem propriedades dependentes da força que permitem mudanças estruturais dinâmicas. Exemplos bem apreciados incluem canais iônicos mecanossensíveis e elementos citoesqueléticos que fornecem às células informações mecânicas importantes de seu ambiente circundante.
Além disso, moléculas que mostram uma natureza única de suporte de força também podem ser consideradas mecanossensíveis em um sentido mais amplo. Por exemplo, a formação local e o derretimento de duplexes de ácidos nucleicos, bem como estruturas de ordem superior, como G-quadruplexes, desempenham papéis cruciais na replicação, transcrição, recombinação e, mais recentemente, edição do genoma. Além disso, algumas proteínas neuronais envolvidas nas comunicações sinápticas desempenham suas funções gerando forças físicas que excedem os níveis de interações intermoleculares típicas. Não importa qual exemplo se estude, investigar a nanomecânica das biomoléculas envolvidas com alta precisão espaço-temporal será altamente útil na revelação de mecanismos moleculares dos processos mecanobiológicosassociados1,2,3.
Métodos de espectroscopia de força de molécula única têm servido como ferramentas poderosas para examinar as propriedades mecânicas de biomoléculas 2,4,5,6. Eles podem monitorar mudanças estruturais em ácidos nucleicos e proteínas simultaneamente com a aplicação de força, examinando assim propriedades dependentes da força. Duas configurações bem conhecidas são as pinças ópticas e as pinças magnéticas (MTs), que empregam esferas do tamanho de mícrons para manipular moléculas 5,6,7,8. Nessas plataformas, o poliestireno (para pinças ópticas) ou esferas magnéticas (para MTs) são amarrados a moléculas-alvo (por exemplo, ácidos nucléicos e proteínas) por meio de "alças" moleculares, tipicamente feitas de fragmentos curtos de DNA de fita dupla (dsDNA). As contas são então movidas para exercer força e fotografadas para rastrear suas localizações que relatam mudanças estruturais nas moléculas-alvo. Pinças ópticas e magnéticas são amplamente intercambiáveis em suas aplicações, mas existem diferenças importantes em suas abordagens para controlar a força. Pinças ópticas são instrumentos de fixação intrinsecamente posicionados que aprisionam contas na posição, por causa das quais a força aplicada flutua quando uma construção alvo sofre mudanças de forma; aumento de extensão, como de desdobramento, afrouxa a amarração e reduz a tensão, e vice-versa. Embora a realimentação ativa possa ser implementada para controlar a força em pinças ópticas, as MTs, em contraste, operam naturalmente como um dispositivo de fixação de força, aproveitando as forças magnéticas estáveis e de campo distante por ímãs permanentes, que também podem resistir à perturbação ambiental.
Apesar de sua longa história e design simples, as MTs ficaram atrás das pinças ópticas em suas aplicações para medições de alta precisão, em grande parte devido aos desafios técnicos no rastreamento rápido de contas. Recentemente, entretanto, vários grupos têm liderado conjuntamente um aprimoramento multifacetado tanto do hardware quanto do software para instrumentos de MT2,9,10,11,12,13,14,15,16,17,18,19 . Neste trabalho, apresentamos um exemplo de tal configuração rodando a 1,2 kHz e descrevemos como usá-lo para realizar medições nanomecânicas em biomoléculas sensíveis à força. Como sistemas modelo, empregamos pinos de cabelo de DNA e complexos SNARE neuronais e examinamos suas rápidas mudanças estruturais no regime de piconewton. Pinos de DNA exibem transições simples de dois estados em uma faixa de força bem definida20,21 e, portanto, servem como modelos de brinquedo para verificar o desempenho de uma pinça. À medida que as proteínas SNARE se agrupam em um complexo sensível à força que impulsiona a fusão da membrana22, elas também têm sido extensivamente estudadas por espectroscopia de força de molécula única 14,23,24,25. Abordagens padrão para analisar dados e extrair informações úteis sobre termodinâmica e cinética são apresentadas. Esperamos que este artigo possa facilitar a adoção de MTs de alta precisão em estudos mecanobiológicos e motivar os leitores a explorar seus próprios sistemas sensíveis à força de interesse.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos os materiais e equipamentos descritos neste protocolo estão listados na Tabela de Materiais. O software LabVIEW para operar a configuração MT de alta velocidade descrita abaixo, bem como os scripts MATLAB para analisar dados de amostra, são depositados no GitHub (https://github.com/ShonLab/Magnetic-Tweezers) e disponibilizados publicamente.
1. Construção de aparelhos
NOTA: O princípio geral da construção MT de alta velocidade é semelhante aos sistemas MT convencionais padrão, exceto pelo uso de uma câmera de semicondutor de óxido metálico complementar (CMOS) de alta velocidade e uma fonte de luz coerente e de alta potência (Figura 1). Consulte outras fontes para obter mais descrições de instrumentos padrão de TM 5,26,27.
- Configure um microscópio invertido em uma mesa óptica antivibração. Instale uma câmera CMOS de alta velocidade e um frame grabber.
- Construa um estágio de tradução para manipular ímãs em 3D. Monte um estágio linear motorizado (>20 mm de curso) verticalmente sobre um estágio XY manual.
NOTA: O movimento vertical controla a força, enquanto o estágio XY é para o alinhamento manual dos ímãs ao eixo óptico para a construção inicial do setup. - Instale um motor de passo rotativo e um sistema de correia e polia para ímãs rotativos.
NOTA: A correia transmite o movimento rotativo entre o eixo do motor e ímãs que estão a poucos centímetros de distância. A rotação dos ímãs é interna à manipulação translacional. - Monte os ímãs. Use um suporte de acrílico (encomendado a uma empresa de fabricação; veja Figura Suplementar S1) que possa abrigar firmemente dois ímãs idênticos em paralelo, com um espaço bem definido de 1 mm entre os ímãs (Figura 1B). Para utilizar a força máxima obtida com um determinado par de ímãs, ajuste a posição vertical do estágio de translação para que a superfície inferior dos ímãs se alinhe com o plano da amostra quando ele for movido para a posição mais baixa.
NOTA: Consulte Lipfert e colaboradores para obter mais informações sobre o projeto e a configuração do suporte de ímãs28. A altura e a orientação dos ímãs são controladas pelo software LabVIEW em conjunto com a aquisição de dados. - Visualizando com uma lente objetiva de baixa ampliação, alinhe os ímãs ao centro do campo de visão. Verifique se a rotação dos ímãs não causa um grande deslocamento do centro do par de ímãs.
NOTA: Se o ponto médio entre os ímãs girar em torno do eixo de rotação, é provável que os ímãs estejam descentrados devido a um suporte imperfeito. Um pequeno nível de desalinhamento em relação ao tamanho da folga é tolerável, já que a rotação do ímã é apenas para verificar amarras e aplicar torques em aplicações específicas. - Instale um diodo superluminescente (SLD) para a iluminação de contas. Passe o feixe através do espaço de 1 mm entre os dois ímãs. Certifique-se de que o feixe está devidamente colimado para caber na lacuna e a iluminação não é sombreada pelos ímãs.
- Instale um scanner de lente piezo na peça nasal e monte uma lente objetiva de imersão em óleo de 100x (abertura numérica [NA]: 1,45) para rastreamento de contas. Para evitar possíveis artefatos no rastreamento de resultados, certifique-se de que a iluminação seja mantida uniformemente quando os ímãs forem movidos. Finalmente, ajuste o nível de luz para o brilho máximo sem saturar pixels.
NOTA: Para a comparação de diferentes fontes de luz para o rastreamento de alta velocidade de contas, consulte Dulin et al.29.
2. Calibração da força magnética
- Usando a reação em cadeia da polimerase (PCR; ver Tabela 1), prepare fragmentos de dsDNA de 5 kbp (usando o Primer B, o Primer Z_5k e o λ-DNA) que são marcados com biotina em uma extremidade (para fixação à superfície) e azida na outra extremidade (para fixação do talão).
- Após a seção 6, prepare uma célula de fluxo com as moléculas de 5 kbp.
- Após a seção 7, identifique uma boa construção de cordão de contas verificando sua extensão e rotação. Em particular, certifique-se de escolher um talão com uma trajetória rotacional mínima (ou seja, com um raio <200 nm) para minimizar o deslocamento da altura do cordão devido à fixação descentralizada30,31. Uma vez que uma boa amarração é identificada, inicie o rastreamento de contas, referindo-se à seção 9.
- Se a configuração for nova, caracterize seu ruído e estabilidade para medições confiáveis de alta resolução. Coloque o ímã a ~3 mm da superfície da célula de fluxo (para aplicar >10 pN e suprimir o movimento browniano de um talão), rastreie a posição z do talão em 1,2 kHz e calcule o desvio de Allan (AD) da série temporal de coordenadas z 32,33 (Figura 2C). Verifique se os valores de AD de alguns nanômetros são alcançáveis no regime de alta velocidade (<0,1 s) e se o rastreamento diferencial (posição do cordão magnético em relação a um cordão de referência) reduz o AD na escala de tempo mais longa.
NOTA: Normalmente, obtemos um AD de <3 nm na taxa máxima (resolução de 1,2 kHz ou 0,83 ms), e o AD continua diminuindo pelo menos até 10 s, implicando uma deriva mínima. Outros relataram valores semelhantes em configurações semelhantes9,10,11,12,34. - Com ímãs na posição de repouso (F ~ 0 pN), registre as coordenadas x e y do talão amarrado em 1,2 kHz. Registrar a posição por um período suficientemente longo (isto é, suficientemente longo do que o tempo de relaxamento característico da flutuação35) para que o movimento browniano seja suficientemente amostrado.
NOTA: Aqui, a direção x é ao longo da direção do campo magnético, enquanto o movimento em y representa o movimento transversal perpendicular ao campo. - Aproxime os ímãs da célula de fluxo e repita as medições da posição do talão até que os ímãs toquem suavemente a parte superior da célula de fluxo. Mova-se em passos grandes (por exemplo, 1-2 mm) quando os ímãs estiverem a mais de 7 mm de distância do plano da amostra (já que a força aplicada aumenta lentamente no campo distante dos ímãs), mas reduza o tamanho do passo gradualmente (por exemplo, 0,1-0,5 mm) à medida que se aproximam para uma calibração mais fina em níveis de força mais altos (Figura 2B).
- Calcule a força em cada posição do ímã, d, usando um dos dois métodos alternativos (um script MATLAB "force calibration.m" incluindo ambos os métodos é fornecido; veja Arquivo Suplementar 1).
- Meça a variância das coordenadas
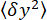 y do talão (Figura 2D) e a posição
y do talão (Figura 2D) e a posição 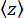 z média do talão em relação à posição mais baixa (Figura 2B, abaixo). Em seguida, use a equação (1)7,27,36 para estimar a força (com raio fixo do cordão R = 1.400 nm e energia térmica kR T = 4,11 pN∙nm):
z média do talão em relação à posição mais baixa (Figura 2B, abaixo). Em seguida, use a equação (1)7,27,36 para estimar a força (com raio fixo do cordão R = 1.400 nm e energia térmica kR T = 4,11 pN∙nm):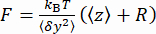 (1º)
(1º) - Alternativamente, calcule a densidade espectral de potência (PSD) das coordenadas y, Sy(Figura 2E). Determinar a força aplicada F ajustando um modelo duplo-lorentziano37 ao Sy medido usando a equação (2).
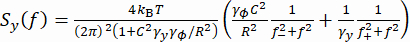 (2º)
(2º)
Aqui, , R é o raio do talão, γye γ φ são os coeficientes de arrasto translacional e rotacional, respectivamente (estimados a partir da equação de Stokes-Einstein), kRT é a energia térmica,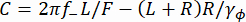 f+ e f- são duas frequências características obtidas pela equação (3).
f+ e f- são duas frequências características obtidas pela equação (3).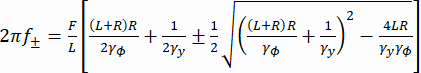 (3º)
(3º)
NOTA: Como a extensão de amarração L é uma função de força que segue o modelo de cadeia semelhante a worm (WLC) bem estabelecido, as expressões acima deixam F como o único parâmetro de ajuste (fixamos R como sendo 1.400 nm para simplificar, porque ele é compartilhado entre todos os níveis de força e o valor exato não influencia os resultados sensivelmente). Quando necessário, o desfoque de movimento e o aliasing da aquisição de imagens baseadas em câmera devem ser considerados38,39, mas esse efeito é desprezível em nossas medições de alta velocidade acima de 1 kHz com amarras de 5 kbp.
- Meça a variância das coordenadas
- Repita as etapas 2.4-2.7 para mais algumas construções. Sonda de três a cinco contas diferentes para calcular a média da variabilidade de força entre as esferas magnéticas.
NOTA: A variação de força entre as esferas magnéticas em uso deve ser considerada para determinar o número adequado de construtos para a média. Essa variabilidade é pequena, mas pode levar a mais de 1 pN de erro na força medida, mesmo para produtos comerciais31. Para a maioria das aplicações, onde a determinação absoluta das forças envolvidas não é crucial, a média dos resultados de calibração de três a cinco esferas é geralmente suficiente. Uma abordagem alternativa para explicar essa variação é medir a força com amarras individuais no início do experimento, o que pode ser demorado. Outra opção é incorporar estruturas hairpin que se descompactam em níveis de força conhecidos em cada construto31. - Plotar a força medida em função da distância do ímã e ajustar uma função exponencial dupla aos dados (Figura 2F) usando a equação (4).
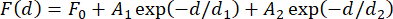 (4º)
(4º)
Aqui, F0 (linha de base), A 1 e A 2 (amplitudes) e d 1 e d 2 (constantes de decaimento) são parâmetros de ajuste. Certifique-se de que os valores de força dos dois métodos, bem como os ajustes duplo-exponenciais resultantes, estejam amplamente de acordo (Figura 2F,G).
NOTA: Para confirmar se a calibração da força é conduzida corretamente, verifique a relação força-extensão das construções sondadas plotando a extensão versus a força medida. - Para corrigir o deslocamento z off da altura do cordão resultante da inclinação dependente da força das esferas magnéticas30,31, estime z off a partir do offset lateral x off, considerando a geometria de uma corda descentralizada com raio do cordão usando a equação (5), e aplique os valores aos valores de extensão medidos. Esta etapa é implementada no script MATLAB "force calibration.m" (linhas 252-254).
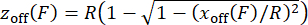 (5º)
(5º)
NOTA: Embora essa correção faça pequenas alterações na extensão, especialmente para as esferas com um pequeno raio de rotação (<200 nm), esse deslocamento geralmente afeta criticamente a resposta elástica, como visto na mudança da Figura 2H para a Figura 2I30,31. - Verifique o comprimento de persistência Lp ajustando um modelo WLC extensível aos dados usando a equação (6).
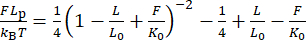 (6)
(6)
Aqui, L 0 é o comprimento do contorno (1,7 μm por 5 kbp) e K0 é o módulo para o alongamento entálico.
NOTA: Embora o L p do dsDNA seja bem aceito como sendo 40-50 nm em um tampão típico como o fosfato tamponada salino (PBS), a fórmula WLC aplicada a moléculas curtas (<5 kbp) subestima sistematicamente L p à medida que L0 diminui31,40. Isso ocorre porque o modelo WLC clássico presume um polímero cujo comprimento de cadeia é suficientemente maior do que seu comprimento de persistência. Aqui, obtivemos L p = 40 ± 3 nm para a construção de 5 kbp (Figura 2H), e a correção da extensão resultou em um K0 homogêneo de 1.100 ± 200 pN (Figura 2I). A aplicação de um modelo WLC finito 31,40, bem como uma correção para não-Gaussianidade na distribuição de extensão 41, aumentará ligeiramente Lp. - Uma vez verificada a calibração da força, aplique os parâmetros de ajuste obtidos do modelo duplo-exponencial ao software LabVIEW fornecido (Supplemental File 2) e aguarde que o software calcule a força atual em tempo real a partir das leituras do motor (ou seja, posição do ímã). Como uma expressão analítica para a função inversa d(F) não está disponível, prepare uma tabela de pesquisa de d versus F em passos de 0,1 pN por estimativa numérica dos níveis de força alvo d. Armazene esta tabela no software também para comandar o controle de força.
3. Síntese de pinos de DNA
NOTA: As construções hairpin de DNA para experimentos de MT são preparadas por amplificação por PCR de uma região de 510 pb em λ-DNA com dois primers personalizados, um dos quais contém uma estrutura hairpin em sua extremidade 5′ (Figura 3A). Desta forma, um motivo hairpin é colocado em uma extremidade do produto PCR.
- Prepare os primers.
- Primer para frente: O primer B_hp que é marcado com 5′-biotina para fixação da superfície de vidro e se liga ao λ-DNA. Este primer contém um motivo hairpin com uma haste de 8 pb e um laço de 6 nt, 5′ para a região de ligação λ.
- Primer reverso: O primer Z_hp marcado com 5′-azida para fixação do cordão magnético e liga-se ao λ-DNA a 1 kbp de distância do primer dianteiro.
- Configure e execute a PCR com λ-DNA (modelo), nTaq polimerase e condições padrão de PCR (consulte a Tabela 1). Limpe o produto com um kit de purificação comercial.
- Medir a concentração de ADN por absorção de UV a 260 nm (A260) e realizar electroforese em gel de agarose (gel a 2%) (ver Tabela 2) para verificar o tamanho do produto. Um rendimento típico é de ~35 μL de ~600 nM solução.
4. Preparação de proteínas SNARE
NOTA: Os complexos SNARE neuronais são montados pela combinação de três proteínas purificadas de rato expressas de E. coli: VAMP2/synaptobrevin-2, syntaxin-1A e SNAP-25 (Figura 3B). Para facilitar sua montagem, a sintaxe e o SNAP-25 são co-expressos com um fragmento VAMP2 (sem a região N-terminal; denominado "ΔN-VAMP2") em uma estrutura chamada "complexo ΔN", e então misturados com VAMP2 de comprimento total após a ligação do DNA para formar complexos completos.
- Preparar plasmídeos contendo cDNA para a expressão de proteínas SNARE (sequências de DNA para todos os plasmídeos são dadas na Tabela de Materiais).
- Preparar 6×His-tagged VAMP2 sem o domínio transmembrana (2-97; L32C/I97C para ligações dissulfeto) clonado em um vetor pET28a.
- Prepare a sintaxina-1A sem o domínio Habc e transmembrana (substituições 191-267, I202C/I266C para ligações dissulfeto) clonadas juntamente com 6×His-tagged ΔN-VAMP2 (49-96) em um vetor pETDuet-1.
- Prepare a isoforma b SNAP-25 de comprimento total (2-206, todos C a A) clonada em um vetor pET28a. Isso será usado para preparar complexos ΔN.
- Prepare a isoforma b (1-206, todos C a A) de 6×His-tagged SNAP-25 clonada em um vetor pET28a para adição direta ao buffer de ensaio MT para remontar os complexos SNARE após o desdobramento.
- Preparar dois tubos de células de E. coli Rosetta (DE3). Transformar um grupo com plasmídeos VAMP2 (da etapa 4.1.1), um com plasmídeos SNAP-25 marcados com sintaxe 1A/ΔN-VAMP2 e não marcados (da etapa 4.1.2 e 4.1.3) para expressar o complexo ΔN, e o outro com plasmídeos SNAP-25 marcados com His (da etapa 4.1.4).
- Transferir as células transformadas para o caldo Luria-Bertani (LB) com antibióticos apropriados (aqui, canamicina e cloranfenicol para VAMP2 e SNAP-25; canamicina, cloranfenicol e ampicilina para o complexo ΔN). Cultivá-los a 37 °C em incubadora de agitação (220 rpm) até que a densidade óptica (DO) do caldo atinja 0,7-0,9.
- Adicionar 1 mM de isopropil β-d-1-thiogalactopyranoside (IPTG) para induzir a expressão proteica e incubar as células por 3-4 h a 37 °C em uma incubadora de agitação (220 rpm).
- Pellet down das células centrifugando a cultura a 4.500 × g por 15 min a 4 °C.
- Preparar tampões para a purificação de proteínas (ver Tabela 2).
- Suspender os pellets de células que expressam SNARE em 40 mL de tampão de lise gelado e lisar as células por sonicação no gelo (15% de amplitude, 5 s on e 5 s off, 30 min total).
- Centrifugar o lisado a 15.000 × g durante 30 min a 4 °C para remover materiais insolúveis.
- Passar o sobrenadante através de uma coluna de gravidade preenchida com 1 mL de resina Ni-NTA. Lavar a resina com tampão de lavagem A, depois com tampão de lavagem B e eluir as proteínas com 10 mL de tampão de eluição.
- Remova tris(2-carboxietil)fosfina (TCEP) e imidazol do eluente usando uma coluna de dessalinização (siga as instruções do fabricante). Eluir a amostra com PBS.
- Concentrar as proteínas com filtros centrífugos (ponto de corte de 10 kDa) até ~70 μM, mantendo as proteínas em PBS (tipicamente produzindo 2 mL). Medir a concentração de proteínas por absorção ultravioleta (UV) a 280 nm (A280) ou pelo ensaio de Bradford.
- Prepare alíquotas pequenas, congele em nitrogênio líquido e armazene a -80 °C até o uso.
NOTA: Complexos SNARE completos serão montados após a conjugação do complexo ΔN em um identificador de DNA (veja abaixo).
5. Fixação de alças de DNA
NOTA: Duas alças de dsDNA de 510 pb contendo grupos amina primária em uma extremidade são primeiramente preparadas por PCR, e os grupos amina são então convertidos em grupos maleimida usando um reticulador bifuncional, SM(PEG)2. As duas alças são então ligadas covalentemente aos complexos SNARE através de seus grupos cisteína para conjugação sítio-específica (Figura 3B).
- Prepare primers.
- Preparar primers para a frente: Primer B (para amplificar o punho B) que é marcado com 5′-biotina para fixação da superfície de vidro e se liga ao λ-DNA; Primer Z (para amplificar o Handle Z) que é marcado com 5′-azida para fixação de esferas magnéticas e tem a mesma sequência do Primer B.
- Prepare um primer reverso: Primer N (compartilhado para Handle B e Handle Z) que é marcado com 5′-amina para conjugação de proteínas e se liga ao λ-DNA 510 pb longe do primer dianteiro.
- Configurar e executar dois conjuntos de reações de PCR (18 tubos de reação de 200 μL para cada alça) com λ-DNA (modelo), nTaq polimerase e condições padrão de PCR (ver Tabela 1). Limpe o produto com um kit de limpeza PCR e elimine cada cabo com 45 μL de água ultrapura. Use um volume mínimo de água para obter altas concentrações de alças para uma reação eficaz em etapas posteriores.
- Medir a concentração de ADN por A260. O rendimento típico é de ~650 μL de ~2 μM de solução para cada alça. Manter pequenas amostras separadas para posterior verificação em eletroforese em gel de agarose.
- Reaja cada alça (1 μM em PBS) com 5 mM SM(PEG)2. Incubar à temperatura ambiente com rotação suave. Após 1 h, use um kit de purificação de DNA para remover SM(PEG)2 não reagido. Eluir cada cabo com 250 μL de PBS para obter soluções de ~2 μM.
- Misturar as soluções do Handle B e do complexo ΔN a uma razão molar de 1:16 (por exemplo, 1 μM Handle B e 16 μM ΔN-complex) em PBS e incubar por 2 h à temperatura ambiente com agitação. Separe uma pequena amostra para eletroforese em gel de agarose.
- Adicionar uma solução de VAMP2 em um excesso molar de 2,5 vezes sobre o complexo ΔN usado na etapa anterior. Incubar a mistura por mais 1 h à temperatura ambiente com agitação. Complexos SNARE completos são montados nesta etapa.
- Remover as proteínas livres por troca de tampão com PBS fresco e um filtro centrífugo (ponto de corte de 100 kDa): centrifugar a 14.000 × g por 5 min a 4 °C, repetir pelo menos 6x e executar por 15 min para o último giro. Medir o aumento da relação A260/A280 para monitorar a remoção de proteínas livres. Separe uma pequena amostra para eletroforese em gel de agarose.
- Adicione a Alça Z à solução em um excesso molar de 15 vezes sobre a Alça B. Mantenha a concentração da Alça Z pelo menos acima de 1 μM para facilitar a reação. Incubar a mistura durante a noite a 4 °C com agitação.
- Verificar os intermediários (alça B e seus conjugados proteicos) e o produto final (complexo SNARE com duas alças) por eletroforese em gel de agarose (Figura 3B, inset) (ver Tabela 2).
NOTA: Se as proteínas forem conectadas com êxito à alça B, uma mudança de mobilidade será detectada. Em particular, a formação de complexos SNARE completos em manipuladores de DNA pode ser confirmada por sua resistência ao dodecil sulfato de sódio (SDS), ao contrário dos complexos ΔN, que são desmontados em SDS e deixam apenas a sintaxe ligada ao DNA (compare b e c na Figura 3B). - Prepare alíquotas pequenas, congele em nitrogênio líquido e armazene a -80 °C até o uso.
NOTA: Embora a solução final contenha alças não reagidas, somente a construção desejada que é duplamente marcada com biotina e azida será selecionada durante a montagem da amostra em uma célula de fluxo.
6. Fabricação de células de fluxo
NOTA: As células de fluxo para medições de MT são construídas a partir de duas lamínulas de vidro unidas por fita dupla face (Figura 3C). Uma lamínula é revestida com uma mistura de PEG e polietilenoglicol biotinilado (PEG) para evitar a ligação inespecífica e permitir a ligação específica de moléculas-alvo via ligação biotina-NeutrAvidin (Figura 3D). Em seguida, as soluções dos materiais para experimentos de MT são infundidas sequencialmente em uma célula de fluxo usando uma bomba de seringa (Figura 3C,D).
- Prepare duas lamínulas de vidro, uma para cada uma das superfícies superior (24 mm × 50 mm, espessura nº 1,5) e inferior (24 mm × 60 mm, espessura nº 1,5). Limpe as lamínulas por sonicação em 1 M KOH por 30 min. Após a sonicação, enxágue as lamínulas com água destilada e mantenha em água até o passo seguinte.
- PEGilato a lamínula inferior seguindo protocolos publicados42,43. Use N-[3-(trimetoxisilil)propil]etilenodiamina para silanização e uma mistura 1:100 (ww) de biotina-PEG-SVA e mPEG-SVA em tampão bicarbonato 100 mM. Mantenha as lamínulas PEGylated secas a -20 °C e guarde-as por algumas semanas.
- No dia dos experimentos, retire as tampas PEGylated e seque-as com uma pistola de nitrogênio. Inspecione-os visualmente em busca de sujeira para se certificar de que estão limpos.
- Para fazer os canais de amostra, prepare tiras de fita dupla face de ~2 mm de largura e coloque quatro tiras em uma lamínula inferior (superfície PEGylated para cima), paralelas e separadas umas das outras por ~5 mm (Figura 3C).
NOTA: Dessa forma, três canais de amostra de 5 mm de largura podem ser criados em uma única célula de fluxo. - Coloque uma tampa superior no centro da tampa inferior, deixando ~5 mm de espaço nas bordas curtas para entradas e saídas de canal. Pressione suavemente a parte de trás da tampa superior com uma pinça para selar firmemente os canais.
- Para fazer um reservatório de entrada, apague a borda de uma ponta de pipeta de 200 μL. Recorte ~10 mm da abertura mais larga para permitir a retenção de ~200 μL de solução. Faça três deles para os três canais de fluxo. Para configurar as tomadas, prepare três agulhas de seringa que se encaixam na tubulação para a bomba de seringa.
- Usando 5 min de epóxi, cole os reservatórios e cubos da agulha na célula de fluxo. Certifique-se de que uma vedação completa seja formada para evitar vazamentos e que os canais não sejam bloqueados com excesso de cola. Deixe secar por pelo menos 30 min.
7. Montagem de construções de talão-cordão
NOTA: As soluções de materiais para experimentos de MT, incluindo as de construção de cordas, são introduzidas sequencialmente em células de fluxo usando uma bomba de seringa (Figura 3C,D).
- Prepare contas magnéticas. Tome 5 mg de contas M270-epóxi de uma solução-mãe (~3,3 × 108 contas em 167,5 μL de dimetilformamida) e substitua o solvente por tampão fosfato (ver Tabela 2) por separação magnética das contas.
- Preparar as contas a ~1,1 × 109 contas mL−1 em tampão fosfato com sulfato de amônio 1 M e reagir com 2 mM de dibenzociclooctyne (DBCO)-NH2. Incubar a mistura durante 3 h num misturador rotativo à temperatura ambiente. Após a reação, lave as esferas 3x com tampão fosfato fresco para remover moléculas não reagidas.
NOTA: As contas lavadas podem ser armazenadas sem rotação extra a 4 °C durante várias semanas antes da utilização. - Conecte uma agulha na saída do canal da célula de fluxo à bomba de seringa com tubulação de polietileno. Equilibre os canais com PBS.
- Introduza as seguintes soluções sequencialmente no canal por sucção com a bomba: NeutrAvidin, construções-alvo (pinos de cabelo de DNA ou complexos SNARE com alças de DNA), contas de poliestireno de referência e contas magnéticas revestidas com DBCO. Antes de usar, vórtice as soluções de contas completamente para dispersar potenciais agregados de contas.
- Lave as contas não acopladas enquanto aplica 0,1 pN de força.
NOTA: A aplicação de uma pequena força ascendente facilita a remoção de contas não ligadas e ajuda a evitar a ruptura de construções de cordão especificamente ligadas. - Para experimentos com complexos SNARE, incluir 1,5 μM SNAP-25 no buffer final.
NOTA: As moléculas SNAP-25 livres podem religar complexos SNARE após o desdobramento e permitir medições repetidas em um único complexo.
8. Identificação dos construtos-alvo
- Na superfície de um canal de célula de fluxo, procure as esferas magnéticas que são amarradas por moléculas únicas da construção alvo. Certifique-se de que um talão de referência esteja localizado nas proximidades.
- Gire uma conta candidata e verifique se ela gira livremente. Se o talão é amarrado por várias moléculas, ele exibe um movimento restrito.
- Gire o talão por algumas voltas completas e descubra o raio de rotação (esta função é implementada no software fornecido). De preferência, escolha um talão com um pequeno raio de rotação.
NOTA: Este raio indica quanto o talão está descentralizado do eixo de amarração, que é determinado aleatoriamente durante a montagem do talão30,31. Em todos os experimentos, a descentralização mínima de um talão alivia muitos artefatos associados à alta relação raio do talão para a extensão do cordão que usamos. - Aumente a força de 0 para 5 pN para identificar boas contas de amarração única. Procure uma grande mudança no padrão de difração de um talão resultante do alongamento de um cabo de 1 kbp (ou o equivalente a duas alças de 510 pb). Se o padrão de difração não mudar significativamente, reduza a força para zero e procure outro talão candidato.
NOTA: O levantamento de ~300 nm de um talão pode ser facilmente notado a partir das imagens brutas sem realmente iniciar o processo de rastreamento.
9. Rastreamento de contas para medições de extensão
NOTA: O rastreamento de contas é realizado através da análise de imagens de contas em tempo real no software LabVIEW fornecido com este artigo. O método de rastreamento e suas variantes têm sido utilizados na maioria dos sistemas convencionais de MT e são explicados na literatura anterior2,5,7,26. Ao medir a posição de um cordão magnético em relação a um cordão de referência fixo (isto é, rastreamento diferencial), as medições de posição tornam-se extremamente robustas a uma perturbação externa.
- Uma vez que um talão magnético adequado esteja localizado junto com um talão de referência, clique no botão Calibrar para começar a se preparar para o rastreamento de contas.
- Clique nas contas na imagem para definir a localização das contas. As imagens serão então cortadas para regiões de interesse (ROIs) (por exemplo, 150 x 150 pixels para um talão de 3 μm) ao redor das contas e, em seguida, analisadas para extrair as coordenadas precisas das contas.
- Aguarde a rotação do ímã ser concluída. Esse processo registra as coordenadas x e y do talão (calculando a correlação cruzada 2D44 ou usando simetria radial45 das imagens do talão, com desempenho comparável) enquanto gira os ímãs para documentar a fixação descentralizada do talão31.
- Para rastreamento na direção z, aguarde até que o software gere uma tabela de pesquisa de imagens de difração das contas a diferentes distâncias do plano focal. Isso é realizado pisando a lente objetiva com um scanner piezo em passos equidistantes e registrando imagens de contas de flutuação média em cada posição. Em seguida, as coordenadas z das contas em experimentos reais são determinadas comparando-se as imagens de contas em tempo real com a tabela de pesquisa com interpolação7.
- Quando a geração da tabela de pesquisa terminar, habilite o rastreamento e o foco automático (pressione a tecla Track ? e AF? Buttons) e clique no botão Adquirir para começar a gravar as posições das contas.
NOTA: O foco automático é opcional, mas recomendado para corrigir o desvio de estágio em z durante a aquisição.
10. Forçar esquemas de aplicação
- Experimentos de rampa de força: Para verificar a relação força-extensão do construto, aplique uma rampa de força para cima e para baixo a uma taxa de carregamento constante (± 1,0 pN s−1) (Figura 4A). Por exemplo, aplique três rodadas de um ciclo 0-20-0 pN para verificar o comprimento total da construção e a curva de extensão de força das alças.
- Especificando os parâmetros de amarração no software, sobreponha uma curva de extensão de força WLC sobre os dados medidos e determine se o talão alvo está preso por uma construção de amostra genuína com alças de DNA adequadas. Use o comprimento de contorno conhecido (por exemplo, ~340 nm para dsDNA de 1 kbp) e o comprimento de persistência WLC (30-45 nm para dsDNA31 curto) da construção como ponto de partida. Se necessário, aplique o método de correção de extensão descrito na etapa 2.11.
- Se a construção for verificada, examine a resposta de extensão de força em detalhes para procurar extensão adicional resultante das moléculas-pinos de cabelo alvo ou complexos SNARE.
- Experimentos de força constante: Variar gradualmente a força aplicada em passos discretos para sondar a sensibilidade à força das moléculas alvo (Figura 4B).
NOTA: As MTs permitem experimentos de força constante simples e eficazes porque a força aplicada é mantida constante quando os ímãs são mantidos imóveis.- Para pinos de DNA, aplique 4-8 pN de força com passos de 0,2-0,5 pN e meça a posição do talão por ~10 s em cada nível de força.
- Para complexos SNARE, aplique 14-16 pN de força com passos de 0,1-0,2 pN e meça a posição do talão para ~10 s em cada nível de força.
- Experimentos de salto de força: Observe os eventos de transição dos complexos SNARE.
NOTA: Experimentos de salto de força, como experimentos de força constante, envolvem mudanças nos níveis de força. No entanto, os saltos de força empregam mudanças mais abruptas na força aplicada, permitindo o monitoramento de eventos desencadeados por força nas moléculas sondadas, como uma ruptura súbita de complexos proteicos. Por exemplo, como os complexos SNARE exibem histerese estrutural em ciclagem de força23, é informativo realizar experimentos de salto de força e medir a latência para transição (Figura 4C).- Descompactar: Descascamento de uma molécula VAMP2 de um complexo SNARE ternário intacto, deixando um complexo binário de sintaxina-1A e SNAP-25.
- Recompactando: Zipping da molécula VAMP2 descompactada para regenerar um complexo SNARE intacto.
- Desdobramento: Desmontagem completa de um complexo SNARE acompanhada por dissociação completa do SNAP-25. Apenas as moléculas VAMP2 e sintaxina permanecem na construção após o desdobramento.
- Redobramento: Regeneração de um complexo SNARE após a ligação de uma molécula SNAP-25 livre do tampão.
- Em 2 pN, induzir a montagem de um complexo SNARE intacto aguardando (~30 s) a associação de uma molécula livre de SNAP25. Uma diminuição súbita na extensão é observada na formação de um complexo SNARE.
- Para observar eventos de descompactação, aguarde alguns segundos em 10-12 pN e, em seguida, passe para 14-15 pN abruptamente com a velocidade máxima do motor possível. Dependendo da força alvo, o complexo SNARE exibirá uma transição reversível entre intermediários parcialmente descompactados (como em experimentos de força constante) ou um salto de ~25 nm para um estado mais alto e descompactado após um tempo de espera aleatório (ou latência).
- Para observar eventos de rezipping, diminua a força para 10-12 pN imediatamente após o descompactamento. Novamente, o complexo SNARE exibe uma transição estocástica para o estado inferior com zíper após alguma latência aleatória. Se o desdobramento tiver ocorrido após o descompactamento, o complexo não conseguirá recompactar, pois uma molécula SNAP-25 estará faltando.
- Para observar o desdobramento dos eventos, aguarde um período mais longo após o descompactamento para detectar um novo aumento na extensão (~2 nm).
11. Análise dos dados
NOTA: Os tipos de análise que se pode realizar com dados MT dependem do sistema de destino. No entanto, existem abordagens comuns para extrair informações úteis dos respectivos experimentos descritos na Figura 4. Todas as análises são realizadas com o MATLAB (R2021a) usando os códigos personalizados fornecidos com este artigo. Esses códigos geram gráficos usando os mesmos dados apresentados neste artigo. Observe que, enquanto os dados brutos do rastreamento de 100 Hz foram levados diretamente para análise, os dados do rastreamento de 1,2 kHz foram tipicamente filtrados por mediana (com uma janela deslizante de cinco pontos) antes da análise para reduzir o ruído (exceto para análise de ruído).
- Experimentos de rampa de força: Analisar a relação força-extensão (por exemplo, elasticidade de polímeros) e fazer a transição da força para extrair informações sobre propriedades nanomecânicas.
- Experimentos de força constante: Analise as populações de estado e o tempo de permanência (ou taxa de transição) em função da força para extrair parâmetros estruturais (por exemplo, regiões envolvidas na transição), termodinâmicos (por exemplo, diferença de energia livre) e cinéticos (por exemplo, barreira de energia) das mudanças conformacionais.
- Experimentos de salto de força: Analise a cinética de ruptura (por exemplo, interações proteína-proteína e ligação receptor-ligante) ou o tempo de vida de intermediários transitórios (por exemplo, desdobramento de biomoléculas) para extrair a estabilidade das moléculas alvo e seus estados.
- Como aplicações representativas, analise os dados de amostra para pinos de cabelo de DNA e complexos SNARE:
- Transições de dois estados de um pino de cabelo de DNA: força de descompactação, distância de abertura, dependência de força de deslocamento populacional e atribuição de estado e medidas de taxa de transição com um modelo de Markov oculto (HMM) (códigos MATLAB fornecidos).
- Mudanças conformacionais dos complexos SNARE: força de descompactação, dependência de força de estados intermediários e latência de descompactação, histerese no rezipping e comportamento de desdobramento/redobramento.
NOTA: Modelos de força-extensão para alças de DNA, pinos de cabelo de DNA e conformações do complexo SNARE são dados em referências anteriores14,31.
Access restricted. Please log in or start a trial to view this content.
Resultados
Calibração da força
Os resultados dos dois métodos de medição de força (variância do deslocamento lateral das contas e análise do espectro de potência) diferiram em 0-2 pN (Figura 2G). De acordo com os resultados da Figura 2F, podemos alcançar de forma confiável até 30 pN com ímãs regulares de neodímio.
Transições de dois estados de um hairpin de DNA de 8 pb
Primeiramente, inve...
Access restricted. Please log in or start a trial to view this content.
Discussão
Neste trabalho, introduzimos uma configuração de espectroscopia de força de molécula única que pode observar mudanças estruturais de biomoléculas com alta precisão espaço-temporal. A câmera CMOS de alta velocidade usada adquire 1.200 quadros s−1 com resolução de 1.280 x 1.024, permitindo o rastreamento de contas de 1,2 kHz. No entanto, a velocidade das medições é atualmente limitada pelo software de rastreamento de contas, de modo que o ROI é normalmente reduzido a áreas menores em mediçõe...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado pela bolsa da National Research Foundation of Korea (NRF) financiada pelo governo coreano (MSIT) (NRF-2022R1C1C1012176, NRF-2021R1A4A1031754 e NRF- 2021R1A6A1A10042944). S.-H.R. foi apoiado pela subvenção NRF (2021R1C1C2009717).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Materials for construct synthesis | |||
| Agarose gel electrophoresis system | Advance | Mupid-2plus | |
| DNA ladder | Bioneer | D-1037 | |
| nTaq polymerase | Enzynomics | P050A | |
| PCR purification kit | LaboPass | CMR0112 | |
| PEGylated SMCC crosslinker / SM(PEG)2 | ThermoFisher Scientific | 22102 | For SNARE–DNA coupling |
| Primer B | Bioneer | 5'-Biotin/TCGCCACCATCATTTCCA-3' | For 5-kbp force calibration construct and DNA handles |
| Primer B_hp | IDT | 5'-Biotin/TTTTTTTTTTGTTCTCTATTT TTTTAGAGAAC /AP site/ /AP site/ TCGCCACCATCATTTCCA-3' | For hairpin construct |
| Primer N | Bioneer | 5'-C6Amine/CATGTGGGTGACGCGAAA-3' | For DNA handles |
| Primer Z | Bioneer | 5'-Azide/TCGCCACCATCATTTCCA-3' | For DNA handles |
| Primer Z_5k | Bioneer | 5'-Azide/TTAGAGAGTATGGGTATATGACA TCG-3' | For 5-kbp force calibration construct |
| Primer Z_hp | Bioneer | 5'-Azide/GTGGCAGCATGACACC-3' | For hairpin construct |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| λ-DNA | Bioneer | D-2510 | Template strand for PCR |
| DNA sequences for SNARE proteins | |||
| 6×His-tagged SNAP-25b (2-206; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctat gagaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagcgg aagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCACA GCAGCGGCCTGGTGCCGCGC GGCAGCCATACTAGCGGAGAT ATCGCCGAGGACGCAGACAT GCGCAATGAGCTGGAGGAGA TGCAGAGGAGGGCTGACCAG CTGGCTGATGAGTCCCTGGA AAGCACCCGTCGCATGCTGC AGCTGGTTGAAGAGAGTAAA GATGCTGGCATCAGGACTTT GGTTATGTTGGATGAGCAAG GCGAACAACTGGAACGCATT GAGGAAGGGATGGACCAAAT CAATAAGGACATGAAAGAAG CAGAAAAGAATTTGACGGAC CTAGGAAAATTCGCCGGCCT TGCCGTGGCCCCCGCCAAC AAGCTTAAATCCAGTGATGC TTACAAAAAAGCCTGGGGC AATAATCAGGATGGAGTAGT GGCCAGCCAGCCTGCCCG TGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTG GCTTCATCCGCAGGGTAAC AAATGATGCCCGGGAAAAT GAGATGGATGAGAACCTG GAGCAGGTGAGCGGCATC ATCGGAAACCTCCGCCAC ATGGCTCTAGACATGGGCA ATGAGATTGACACCCAGA ATCGCCAGATCGACAGGA TCATGGAGAAGGCTGATT CCAACAAAACCAGAATTG ATGAAGCCAACCAACGTG CAACAAAGATGCTGGGAA GTGGTTAAggatccgaattcgag ctccgtcgacaagcttgcggccgcactc gagcaccaccaccaccaccactgagat ccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgag caataactagcataaccccttggggcct ctaaacgggtcttgaggggttttttgctga aaggaggaactatatccggat | |
| 6×His-tagged VAMP2 (2-97, L32C/I97C; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCAC AGCAGCGGCCTGGTGCCGC GCGGCAGCCATATGGCAGAT CTCTCGGCTACCGCTGCCAC CGTCCCGCCTGCCGCCCCG GCCGGCGAGGGTGGCCCCC CTGCACCTCCTCCAAATCTTA CCAGTAACAGGAGATGCCAG CAGACCCAGGCCCAGGTGG ATGAGGTGGTGGACATCATG AGGGTGAATGTGGACAAGGT CCTGGAGCGAGACCAGAAG CTATCGGAACTGGATGATCG CGCAGATGCCCTCCAGGCA GGGGCCTCCCAGTTTGAAA CAAGTGCAGCCAAGCTCAA GCGCAAATACTGGTGGAAA AACCTCAAGATGATGTGCTA Aggatccgaattcgagctccgtcg acaagcttgcggccgcactcgagcaccacca ccaccaccactgagatccggctgctaacaaa gcccgaaaggaagctgagttggctgctgcca ccgctgagcaataactagcataaccccttgg ggcctctaaacgggtcttgaggggttttttg ctgaaaggaggaactatatccggat | |
| 6×His-tagged ΔN-VAMP2 (49–96; capitalized) and Syntaxin-1A (191–267, I202C/I266C; capitalized) in pETDuet-1 | homemade | ggggaattgtgagcggataacaattcccctc tagaaataattttgtttaactttaagaagga gatataccATGGGCAGCAGCCATCA TCATCATCATCACAGCAGCGG CCTGGAAGTTCTGTTCCAGGG GCCCGGTAATGTGGACAAGGT CCTGGAGCGAGACCAGAAGCT ATCGGAACTGGATGATCGCGC AGATGCCCTCCAGGCAGGGGC CTCCCAGTTTGAAACAAGTGC AGCCAAGCTCAAGCGCAAATAC TGGTGGAAAAACCTCAAGATGAT GTAAgcggccgcataatgcttaagtcgaaca gaaagtaatcgtattgtacacggccgcataa tcgaaattaatacgactcactataggggaat tgtgagcggataacaattccccatcttagta tattagttaagtataagaaggagatatacat ATGGCCCTCAGTGAGATCGAGA CCAGGCACAGTGAGTGCATC AAGTTGGAGAACAGCATCCG GGAGCTACACGATATGTTCAT GGACATGGCCATGCTGGTGG AGAGCCAGGGGGAGATGATT GACAGGATCGAGTACAATGTG GAACACGCTGTGGACTACGTG GAGAGGGCCGTGTCTGACACC AAGAAGGCCGTCAAGTACCAG AGCAAGGCACGCAGGAAGAA GTGCATGATCTAActcgagtc tggtaaagaaaccgctgctgcgaaatttgaa cgccagcacatggactcgtctactagcgcag cttaattaacctaggctgctgccaccgctga gcaataactagcataaccccttggggcctct aaacgggtcttgaggggttttttgctgaaag gaggaactatatccggattggcgaatgggac gcgccctgtagcggcgcattaagcgcggcgg gtgtggtggttacgcgcagcgtgaccgctac acttgccagcgccctagcgcccgctcctttc gctttcttcccttcctttctcgccacgttcg ccggctttccccgtcaagctctaaatcgggg gctccctttagggttccgatttagtgcttta cggcacctcgaccccaaaaaacttgattagg gtgatggttcacgtagtgggccatcgccctg atagacggtttttcgccctttgacgttggag tccacgttctttaatagtggactcttgttcc aaactggaacaacactcaaccctatctcggt ctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctga tttaacaaaaatttaacgcgaattttaacaa aatattaacgtttacaatttctggcggcacg atggcatgagattatcaaaaaggatcttcac ctagatccttttaaattaaaaatgaagtttt aaatcaatctaaagtatatatgagtaaactt ggtctgacagttaccaatgcttaatcagtga ggcacctatctcagcgatctgtctatttcgt tcatccatagttgcctgactccccgtcgtgt agataactacgatacgggagggcttaccatc tggccccagtgctgcaatgataccgcgagac ccacgctcaccggctccagatttatcagcaa taaaccagccagccggaagggccgagcgca gaagtggtcctgcaactttatccgcctccatc cagtctattaattgttgccgggaagctagag taagtagttcgccagttaatagtttgcgcaa cgttgttgccattgctacaggcatcgtggtg tcacgctcgtcgtttggtatggcttcattca gctccggttcccaacgatcaaggcgagttac atgatcccccatgttgtgcaaaaaagcggtt agctccttcggtcctccgatcgttgtcagaa gtaagttggccgcagtgttatcactcatggt tatggcagcactgcataattctcttactgtc atgccatccgtaagatgcttttctgtgactg gtgagtactcaaccaagtcattctgagaata gtgtatgcggcgaccgagttgctcttgcccg gcgtcaatacgggataataccgcgccacata gcagaactttaaaagtgctcatcattggaaa acgttcttcggggcgaaaactctcaaggatc ttaccgctgttgagatccagttcgatgtaac ccactcgtgcacccaactgatcttcagcatc ttttactttcaccagcgtttctgggtgagcaaa aacaggaaggcaaaatgccgcaaaaaagg gaataagggcgacacggaaatgttgaatact catactcttcctttttcaatcatgattgaag catttatcagggttattgtctcatgagcgga tacatatttgaatgtatttagaaaaataaac aaataggtcatgaccaaaatcccttaacgtg agttttcgttccactgagcgtcagaccccgt agaaaagatcaaaggatcttcttgagatcct ttttttctgcgcgtaatctgctgcttgcaaa caaaaaaaccaccgctaccagcggtggtttg tttgccggatcaagagctaccaactcttttt ccgaaggtaactggcttcagcagagcgcaga taccaaatactgtccttctagtgtagccgta gttaggccaccacttcaagaactctgtagca ccgcctacatacctcgctctgctaatcctgt taccagtggctgctgccagtggcgataagtc gtgtcttaccgggttggactcaagacgatag ttaccggataaggcgcagcggtcgggctgaa cggggggttcgtgcacacagcccagcttgga gcgaacgacctacaccgaactgagataccta cagcgtgagctatgagaaagcgccacgcttccc gaagggagaaaggcggacaggtatccggta agcggcagggtcggaacaggagagcgcac gagggagcttccagggggaaacgcctggtatc tttatagtcctgtcgggtttcgccacctctg acttgagcgtcgatttttgtgatgctcgtca ggggggcggagcctatggaaaaacgccagc aacgcggcctttttacggttcctggccttttg ctggccttttgctcacatgttctttcctgcg ttatcccctgattctgtggataaccgtatta ccgcctttgagtgagctgataccgctcgccgc agccgaacgaccgagcgcagcgagtcagtg agcgaggaagcggaagagcgcctgatgcgg tattttctccttacgcatctgtgcggtatttc acaccgcatatatggtgcactctcagtacaa tctgctctgatgccgcatagttaagccagta tacactccgctatcgctacgtgactgggtca tggctgcgccccgacacccgccaacacccgc tgacgcgccctgacgggcttgtctgctcccg gcatccgcttacagacaagctgtgaccgtct ccgggagctgcatgtgtcagaggttttcacc gtcatcaccgaaacgcgcgaggcagctgcgg taaagctcatcagcgtggtcgtgaagcgatt cacagatgtctgcctgttcatccgcgtccag ctcgttgagtttctccagaagcgttaatgtc tggcttctgataaagcgggccatgttaaggg cggttttttcctgtttggtcactgatgcctc cgtgtaagggggatttctgttcatgggggta atgataccgatgaaacgagagaggatgctca cgatacgggttactgatgatgaacatgcccg gttactggaacgttgtgagggtaaacaactg gcggtatggatgcggcgggaccagagaaaaa tcactcagggtcaatgccagcgcttcgttaa tacagatgtaggtgttccacagggtagccag cagcatcctgcgatgcagatccggaacataa tggtgcagggcgctgacttccgcgtttccag actttacgaaacacggaaaccgaagaccatt catgttgttgctcaggtcgcagacgttttgc agcagcagtcgcttcacgttcgctcgcgtat cggtgattcattctgctaaccagtaaggcaa ccccgccagcctagccgggtcctcaacgaca ggagcacgatcatgctagtcatgccccgcgc ccaccggaaggagctgactgggttgaaggct ctcaagggcatcggtcgagatcccggtgcct aatgagtgagctaacttacattaattgcgtt gcgctcactgcccgctttccagtcgggaaac ctgtcgtgccagctgcattaatgaatcggcc aacgcgcggggagaggcggtttgcgtattgg gcgccagggtggtttttcttttcaccagtga gacgggcaacagctgattgcccttcaccgcc tggccctgagagagttgcagcaagcggtcca cgctggtttgccccagcaggcgaaaatcctg tttgatggtggttaacggcgggatataacat gagctgtcttcggtatcgtcgtatcccacta ccgagatgtccgcaccaacgcgcagcccgga ctcggtaatggcgcgcattgcgcccagcgcc atctgatcgttggcaaccagcatcgcagtgg gaacgatgccctcattcagcatttgcatggt ttgttgaaaaccggacatggcactccagtcg ccttcccgttccgctatcggctgaatttgat tgcgagtgagatatttatgccagccagccag acgcagacgcgccgagacagaacttaatggg cccgctaacagcgcgatttgctggtgaccca atgcgaccagatgctccacgcccagtcgcgt accgtcttcatgggagaaaataatactgttg atgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccac agcaatggcatcctggtcatccagcggatag ttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttc gacgccgcttcgttctaccatcgacaccacc acgctggcacccagttgatcggcgcgagatt taatcgccgcgacaatttgcgacggcgcgtg cagggccagactggaggtggcaacgccaatc agcaacgactgtttgcccgccagttgttgtg ccacgcggttgggaatgtaattcagctccgc catcgccgcttccactttttcccgcgttttc gcagaaacgtggctggcctggttcaccacgc gggaaacggtctgataagagacaccggcata ctctgcgacatcgtataacgttactggtttc acattcaccaccctgaattgactctcttccg ggcgctatcatgccataccgcgaaaggtttt gcgccattcgatggtgtccgggatctcgacg ctctcccttatgcgactcctgcattaggaag cagcccagtagtaggttgaggccgttgagca ccgccgccgcaaggaatggtgcatgcaagga gatggcgcccaacagtcccccggccacgggg cctgccaccatacccacgccgaaacaagcgc tcatgagcccgaagtggcgagcccgatcttc cccatcggtgatgtcggcgatataggcgcca gcaaccgcacctgtggcgccggtgatgccgg ccacgatgcgtccggcgtagaggatcgagat cgatctcgatcccgcgaaattaatacgactc actata | |
| SNAP-25b (1–206, all C to A; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcc agggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGCCGA GGACGCAGACATGCGCAATG AGCTGGAGGAGATGCAGAGG AGGGCTGACCAGCTGGCTGA TGAGTCCCTGGAAAGCACCC GTCGCATGCTGCAGCTGGTT GAAGAGAGTAAAGATGCTGG CATCAGGACTTTGGTTATGTT GGATGAGCAAGGCGAACAAC TGGAACGCATTGAGGAAGGG ATGGACCAAATCAATAAGGAC ATGAAAGAAGCAGAAAAGAAT TTGACGGACCTAGGAAAATTC GCCGGCCTTGCCGTGGCCCC CGCCAACAAGCTTAAATCCAG TGATGCTTACAAAAAAGCCTG GGGCAATAATCAGGATGGAGT AGTGGCCAGCCAGCCTGCCC GTGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTGGC TTCATCCGCAGGGTAACAAAT GATGCCCGGGAAAATGAGATG GATGAGAACCTGGAGCAGGT GAGCGGCATCATCGGAAACCT CCGCCACATGGCTCTAGACAT GGGCAATGAGATTGACACCCA GAATCGCCAGATCGACAGGAT CATGGAGAAGGCTGATTCCAA CAAAACCAGAATTGATGAAGC CAACCAACGTGCAACAAAGAT GCTGGGAAGTGGTTAA ctcgagcaccaccaccaccaccactgag atccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgagc aataactagcataaccccttggggcctc taaacgggtcttgaggggttttttgctgaa aggaggaactatatccggat | |
| Materials for protein purificaiton | |||
| 2-Mercaptoethanol | SIGMA | M3148-25ML | |
| Agar | LPS Solution | AGA500 | |
| Ampicillin, Sodium salt | PLS | AC1043-005-00 | |
| Chloramphenicol | PLS | CR1023-050-00 | |
| Competent cells (E. coli) | Novagen | 70956 | Rosetta(DE3)pLysS |
| Glycerol | SIGMA | G5516-500ML | |
| HEPES | SIGMA | H4034-100G | |
| Hydrochloric acid / HCl | SIGMA | 320331-500ML | |
| Imidazole | SIGMA | I2399-100G | |
| Isopropyl β-D-1-thiogalactopyranoside / IPTG | SIGMA | 10724815001 | |
| Kanamycin Sulfate | PLS | KC1001-005-02 | |
| Luria-Bertani (LB) Broth | LPS Solution | LB-05 | |
| Ni-NTA resin | Qiagen | 30210 | |
| PD MiniTrap G-25 (desalting column) | Cytiva | GE28-9180-07 | For instructions, see: https://www.cytivalifesciences.com/en/us/shop/chromatography/prepacked-columns/desalting-and-buffer-exchange/pd-minitrap-desalting-columns-with-sephadex-g-25-resin-p-06174 |
| Phenylmethylsulfonyl fluoride / PMSF | ThermoFisher Scientific | 36978 | |
| Plasmids for SNARE proteins | cloned in house | N/A | Available upon request |
| Protease inhibitor cocktail | genDEPOT | P3100 | |
| Sodium chloride | SIGMA | S5886-500G | |
| Sodium phosphate dibasic / Na2HPO4 | SIGMA | S7907-100G | |
| Sodium phosphate monobasic / NaH2PO4 | SIGMA | S3139-250G | |
| Tris(2-carboxyethyl)phosphine / TCEP | SIGMA | C4706-2G | |
| Trizma base | SIGMA | T1503-250G | |
| Materials for sample assembly | |||
| Biotin-PEG-SVA | LAYSAN BIO | BIO-PEG-SVA-5K-100MG & MPEG-SVA-5K-1g | For PEGylation |
| Dibenzocyclooctyne-amine / DBCO-NH2 | SIGMA | 761540-10MG | For bead coating |
| Double-sided tape | 3M | 136 | For flow cell assembly |
| Epoxy glue | DEVCON | S-208 | For flow cell assembly |
| Glass coverslip for bottom surface | VWR | 48393-251 | Rectangular, 60×24 mm, #1.5 |
| Glass coverslip for top surface | VWR | 48393-241 | Rectangular, 50×24 mm, #1.5 |
| Magnetic bead | ThermoFisher Scientific | 14301 | Dynabeads M-270 Epoxy, 2.8 μm |
| mPEG-SVA | LAYSAN BIO | mPEG-SVA 1g | For PEGylation |
| N,N-Dimethylformamide / DMF | SIGMA | D4551-250ML | For bead coating |
| N-[3-(trimethoxysilyl)propyl]ethylenediamine | SIGMA | 104884-100ML | For PEGylation |
| Neutravidin | ThermoFisher Scientific | 31000 | For sample tethering |
| Phosphate buffered saline / PBS, pH 7.2 | PLS | PR2007-100-00 | |
| Plastic syringe | Norm-ject | A5 | 5 ml, luer tip |
| Polyethylene Tubing | SCI | BB31695-PE/4 | PE-60 |
| Reference bead | SPHEROTECH | SVP-30-5 | Streptavidin-coated Polystyrene Particles; 3.0-3.4 µm |
| Syringe needle | Kovax | 21G-1 1/4'' | 21 G |
| Syringe pump | KD SCIENTIFIC | 788210 | |
| Equipment for magnetic tweezer instrument | |||
| 1-axis motorized microtranslation stage | PI | M-126.PD1 | For vertical positioning of magnets |
| 2-axis manual translation stage | ST1 | LEE400 | For alignment of magnets to the optical axis |
| Acrylic holder for magnets | DaiKwang Precision | custum order | Drawing available upon request |
| Frame grabber | Active Silicon | AS-FBD-4XCXP6-2PE8 | |
| High-speed CMOS camera | Mikrotron | EoSens 3CXP | |
| Inverted microscope | Olympus | IX73P2F-1-2 | |
| Neodymium magnets | LG magnet | ND 10x10x12t | Dimension: 10 mm × 10 mm × 12 mm; two needed |
| Objective lens | Olympus | UPLXAPO100XO | Oil-immersion, NA 1.45 |
| Objective lens nanopositioner | Mad City Labs | Nano-F100S | |
| Rotation stepper motor | AUTONICS | A3K-S545W | For rotating magnets |
| Superluminescent diode | QPHOTONICS | QSDM-680-2 | 680 nm |
| Software | |||
| LabVIEW | National Instruments | v20.0f1 | |
| MATLAB | MathWorks | v2021a |
Referências
- Le, S., Liu, R., Lim, C. T., Yan, J. Uncovering mechanosensing mechanisms at the single protein level using magnetic tweezers. Methods. 94, 13-18 (2016).
- Choi, H. -K., Kim, H. G., Shon, M. J., Yoon, T. -Y. High-resolution single-molecule magnetic tweezers. Annual Review of Biochemistry. 91 (1), 33-59 (2022).
- Yang, T., Park, C., Rah, S. -H., Shon, M. J. Nano-precision tweezers for mechanosensitive proteins and beyond. Molecules and Cells. 45 (1), 16-25 (2022).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- De Vlaminck, I., Dekker, C. Recent advances in magnetic tweezers. Annual Review of Biophysics. 41 (1), 453-472 (2012).
- Bustamante, C. J., Chemla, Y. R., Liu, S., Wang, M. D. Optical tweezers in single-molecule biophysics. Nature Reviews Methods Primers. 1, 25(2021).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical Journal. 82 (6), 3314-3329 (2002).
- Smith, S. B., Finzi, L., Bustamante, C. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258 (5085), 1122-1126 (1992).
- Lansdorp, B. M., Tabrizi, S. J., Dittmore, A., Saleh, O. A. A high-speed magnetic tweezer beyond 10,000 frames per second. Review of Scientific Instruments. 84 (4), 044301(2013).
- Cnossen, J. P., Dulin, D., Dekker, N. H. An optimized software framework for real-time, high-throughput tracking of spherical beads. Review of Scientific Instruments. 85 (10), 103712(2014).
- Dulin, D., et al. High spatiotemporal-resolution magnetic tweezers: calibration and applications for DNA dynamics. Biophysical Journal. 109 (10), 2113-2125 (2015).
- Huhle, A., et al. Camera-based three-dimensional real-time particle tracking at kHz rates and Ångström accuracy. Nature Communications. 6 (1), 5885(2015).
- Popa, I., et al. A HaloTag anchored ruler for week-long studies of protein dynamics. Journal of the American Chemical Society. 138 (33), 10546-10553 (2016).
- Shon, M. J., Kim, H., Yoon, T. -Y. Focused clamping of a single neuronal SNARE complex by complexin under high mechanical tension. Nature Communications. 9 (1), 3639(2018).
- Tapia-Rojo, R., Eckels, E. C., Fernández, J. M. Ephemeral states in protein folding under force captured with a magnetic tweezers design. Proceedings of the National Academy of Sciences. 116 (16), 7873-7878 (2019).
- Löf, A., et al. Multiplexed protein force spectroscopy reveals equilibrium protein folding dynamics and the low-force response of von Willebrand factor. Proceedings of the National Academy of Sciences. 116 (38), 18798-18807 (2019).
- Tapia-Rojo, R., Alonso-Caballero, A., Fernandez, J. M. Direct observation of a coil-to-helix contraction triggered by vinculin binding to talin. Science Advances. 6 (21), (2020).
- Rieu, M., et al. Parallel, linear, and subnanometric 3D tracking of microparticles with Stereo Darkfield Interferometry. Science Advances. 7 (6), (2021).
- Rieu, M., Valle-Orero, J., Ducos, B., Allemand, J. -F., Croquette, V. Single-molecule kinetic locking allows fluorescence-free quantification of protein/nucleic-acid binding. Communications Biology. 4 (1), 1083(2021).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences. 103 (16), 6190-6195 (2006).
- Camunas-Soler, J., Ribezzi-Crivellari, M., Ritort, F. Elastic properties of nucleic acids by single-molecule force spectroscopy. Annual Review of Biophysics. 45 (1), 65-84 (2016).
- Südhof, T. C., Rothman, J. E. Membrane fusion: grappling with SNARE and SM proteins. Science. 323 (5913), 474-477 (2009).
- Gao, Y., et al. Single reconstituted neuronal SNARE complexes zipper in three distinct stages. Science. 337 (6100), 1340-1343 (2012).
- Zorman, S., et al. Common intermediates and kinetics, but different energetics, in the assembly of SNARE proteins. eLife. 3, e03348(2014).
- Zhang, Y., Hughson, F. M. Chaperoning SNARE folding and assembly. Annual Review of Biochemistry. 90 (1), 581-603 (2021).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H. Magnetic tweezers for single-molecule experiments. Handbook of Single-Molecule Biophysics. , Springer. New York, NY. 371-395 (2009).
- You, H., Le, S., Chen, H., Qin, L., Yan, J. Single-molecule manipulation of G-quadruplexes by magnetic tweezers. Journal of Visualized Experiments. (127), e56328(2017).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical Journal. 96 (12), 5040-5049 (2009).
- Dulin, D., Barland, S., Hachair, X., Pedaci, F. Efficient illumination for microsecond tracking microscopy. PLoS One. 9 (9), e107335(2014).
- Klaue, D., Seidel, R. Torsional stiffness of single superparamagnetic microspheres in an external magnetic field. Physical Review Letters. 102 (2), 028302(2009).
- Shon, M. J., Rah, S. -H., Yoon, T. -Y. Submicrometer elasticity of double-stranded DNA revealed by precision force-extension measurements with magnetic tweezers. Science Advances. 5 (6), 1697(2019).
- Czerwinski, F., Richardson, A. C., Oddershede, L. B. Quantifying noise in optical tweezers by Allan variance. Optics Express. 17 (15), 13255-13269 (2009).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. Review of Scientific Instruments. 83 (2), 025115(2012).
- Ostrofet, E., Papini, F. S., Dulin, D. High spatiotemporal resolution data from a custom magnetic tweezers instrument. Data in Brief. 30, 105397(2020).
- Yu, Z., et al. A force calibration standard for magnetic tweezers. Review of Scientific Instruments. 85 (12), 123114(2014).
- Strick, T. R., Allemand, J. -F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271 (5257), 1835-1837 (1996).
- Daldrop, P., Brutzer, H., Huhle, A., Kauert, D. J., Seidel, R. Extending the range for force calibration in magnetic tweezers. Biophysical Journal. 108 (10), 2550-2561 (2015).
- te Velthuis, A. J. W., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative guidelines for force calibration through spectral analysis of magnetic tweezers data. Biophysical Journal. 99 (4), 1292-1302 (2010).
- Ostrofet, E., Papini, F. S., Dulin, D. Correction-free force calibration for magnetic tweezers experiments. Scientific Reports. 8 (1), 15920(2018).
- Seol, Y., Li, J., Nelson, P. C., Perkins, T. T., Betterton, M. D. Elasticity of short DNA molecules: theory and experiment for contour lengths of 0.6-7 µm. Biophysical Journal. 93 (12), 4360-4373 (2007).
- Burnham, D. R., Vlaminck, I. D., Henighan, T., Dekker, C. Skewed Brownian fluctuations in single-molecule magnetic tweezers. PLoS One. 9 (9), 108271(2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protocols. 3 (1), 101152(2022).
- Lee, H. -W., et al. Profiling of protein-protein interactions via single-molecule techniques predicts the dependence of cancers on growth-factor receptors. Nature Biomedical Engineering. 2 (4), 239-253 (2018).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Parthasarathy, R. Rapid, accurate particle tracking by calculation of radial symmetry centers. Nature Methods. 9 (7), 724-726 (2012).
- Woodside, M. T., Block, S. M. Reconstructing folding energy landscapes by single-molecule force spectroscopy. Annual Review of Biophysics. 43 (1), 19-39 (2014).
- Evans, E., Ritchie, K. Dynamic strength of molecular adhesion bonds. Biophysical Journal. 72 (4), 1541-1555 (1997).
- Zhang, Y. Energetics, kinetics, and pathway of SNARE folding and assembly revealed by optical tweezers. Protein Science. 26 (7), 1252-1265 (2017).
- Chen, H., et al. Improved high-force magnetic tweezers for stretching and refolding of proteins and short DNA. Biophysical Journal. 100 (2), 517-523 (2011).
- Cho, S., et al. Tension exerted on cells by magnetic nanoparticles regulates differentiation of human mesenchymal stem cells. Biomaterials Advances. 139, 213028(2022).
- Shon, M. J., Cohen, A. E. Nano-mechanical measurements of protein-DNA interactions with a silicon nitride pulley. Nucleic Acids Research. 44 (1), 7(2016).
- Cheng, Y. Single-particle cryo-EM-How did it get here and where will it go. Science. 361 (6405), 876-880 (2018).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Neupane, K., et al. Direct observation of transition paths during the folding of proteins and nucleic acids. Science. 352 (6282), 239-242 (2016).
- Choi, H. -K., et al. Watching helical membrane proteins fold reveals a common N-to-C-terminal folding pathway. Science. 366 (6469), 1150-1156 (2019).
- Kim, C., et al. Extreme parsimony in ATP consumption by 20S complexes in the global disassembly of single SNARE complexes. Nature Communications. 12 (1), 3206(2021).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados