Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Kuvvete duyarlı elemanlarda nanomekanik ölçümler için yüksek hızlı manyetik cımbız
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Burada, kuvvete duyarlı biyomoleküller üzerinde maksimum 1.2 kHz hızında nanomekanik ölçümler yapan yüksek hızlı bir manyetik cımbız kurulumunu açıklıyoruz. DNA saç tokalarına ve SNARE komplekslerine model sistemler olarak uygulanmasını tanıtıyoruz, ancak mekanobiyolojik olaylarda yer alan diğer moleküllere de uygulanabilecektir.
Özet
Tek moleküllü manyetik cımbızlar (MT'ler), nükleik asitler ve proteinler gibi biyomolekülleri zorla sorgulamak için güçlü araçlar olarak hizmet etmiştir ve bu nedenle mekanobiyoloji alanında yararlı olmaya hazırdır. Yöntem genellikle manyetik boncukların görüntü tabanlı izlenmesine dayandığından, görüntülerin kaydedilmesi ve analiz edilmesindeki hız sınırının yanı sıra boncukların termal dalgalanmaları, hedef moleküllerdeki küçük ve hızlı yapısal değişiklikleri gözlemlemede uygulanmasını uzun zamandır engellemiştir. Bu makalede, biyomoleküllerin ve komplekslerinin nano ölçekli, milisaniyelik dinamiklerini çözebilen yüksek çözünürlüklü bir MT kurulumunun yapımı ve işletilmesi için ayrıntılı yöntemler açıklanmaktadır. Uygulama örnekleri olarak, DNA saç tokaları ve SNARE kompleksleri (membran-füzyon makineleri) ile yapılan deneyler, geçici durumlarının ve geçişlerinin pikonewton ölçekli kuvvetlerin varlığında nasıl tespit edilebileceğine odaklanarak gösterilmiştir. Yüksek hızlı MT'lerin, hücrelerdeki kuvvetleri algılayan, ileten ve üreten moleküller üzerinde yüksek hassasiyetli nanomekanik ölçümler yapmaya devam edeceğini ve böylece moleküler düzeyde mekanobiyoloji anlayışımızı derinleştireceğini umuyoruz.
Giriş
Hücreler mekanik uyaranları aktif olarak algılar ve bunlara yanıt verir. Bunu yaparken, birçok biyomolekül dinamik yapısal değişiklikleri mümkün kılan kuvvete bağımlı özellikler gösterir. İyi takdir edilen örnekler arasında, hücrelere çevrelerinden önemli mekanik bilgiler sağlayan mekanosensitivite iyon kanalları ve sitoiskelet elemanları bulunur.
Ek olarak, benzersiz bir kuvvet taşıyan nitelik gösteren moleküller, daha geniş anlamda mekanosensitif olarak da kabul edilebilir. Örneğin, nükleik asit dublekslerinin lokal oluşumu ve erimesi, ayrıca G-dörtlüleri gibi daha yüksek dereceli yapılar, replikasyon, transkripsiyon, rekombinasyon ve daha yakın zamanda genom düzenlemede çok önemli roller oynamaktadır. Dahası, sinaptik iletişimde yer alan bazı nöronal proteinler, tipik moleküller arası etkileşimlerin seviyelerini aşan fiziksel kuvvetler üreterek işlevlerini yerine getirirler. Hangi örnek üzerinde çalışılırsa çalışılsın, ilgili biyomoleküllerin nanomekaniğini yüksek uzaysal zamansal hassasiyetle araştırmak, ilişkili mekanobiyolojik süreçlerin moleküler mekanizmalarını ortaya çıkarmada oldukça yararlı olacaktır 1,2,3.
Tek moleküllü kuvvet spektroskopisi yöntemleri, biyomoleküllerin mekanik özelliklerini incelemek için güçlü araçlar olarak hizmet etmiştir 2,4,5,6. Nükleik asit ve proteinlerdeki yapısal değişiklikleri kuvvet uygulamasıyla eşzamanlı olarak izleyebilir, böylece kuvvete bağlı özellikleri inceleyebilirler. İyi bilinen iki kurulum, moleküllerimanipüle etmek için mikron boyutunda boncuklar kullanan optik cımbız ve manyetik cımbızdır (MT'ler), 5,6,7,8 moleküllerini manipüle eder. Bu platformlarda, polistiren (optik cımbız için) veya manyetik boncuklar (MT'ler için), tipik olarak çift sarmallı DNA'nın (dsDNA) kısa parçalarından yapılmış moleküler "tutamaklar" aracılığıyla hedef moleküllere (örneğin, nükleik asitler ve proteinler) bağlanır. Boncuklar daha sonra kuvvet uygulamak için hareket ettirilir ve hedef moleküllerdeki yapısal değişiklikleri bildiren konumlarını izlemek için görüntülenir. Optik ve manyetik cımbızlar uygulamalarında büyük ölçüde birbirinin yerine geçebilir, ancak kuvveti kontrol etme yaklaşımlarında önemli farklılıklar vardır. Optik cımbızlar, boncukları pozisyonda tutan doğal olarak pozisyon kelepçeli aletlerdir, çünkü bir hedef yapı şekil değişikliklerine uğradığında uygulanan kuvvet dalgalanır; Açılma gibi uzatma artışı, bağlantıyı gevşetir ve gerginliği azaltır ve bunun tersi de geçerlidir. Optik cımbızdaki kuvveti kontrol etmek için aktif geri bildirim uygulanabilse de, MT'ler aksine, doğal olarak çevresel bozulmaya da dayanabilen kalıcı mıknatısların kararlı, uzak alan manyetik kuvvetlerinden yararlanan bir kuvvet kelepçeleme cihazı olarak çalışır.
Uzun geçmişlerine ve basit tasarımlarına rağmen, MT'ler, büyük ölçüde hızlı boncuk izlemedeki teknik zorluklar nedeniyle, yüksek hassasiyetli ölçümlere uygulamalarında optik cımbızların gerisinde kalmıştır. Bununla birlikte, son zamanlarda, birkaç grup MT enstrümanları için hem donanım hem de yazılımın çok yönlü bir şekilde geliştirilmesine öncülük etmiştir 2,9,10,11,12,13,14,15,16,17,18,19 . Bu çalışmada, 1.2 kHz'de çalışan böyle bir kurulumun bir örneğini tanıtıyoruz ve kuvvete duyarlı biyomoleküller üzerinde nanomekanik ölçümler yapmak için nasıl kullanılacağını açıklıyoruz. Model sistemler olarak, DNA saç tokalarını ve nöronal SNARE komplekslerini kullanıyoruz ve pikonewton rejimindeki hızlı, yapısal değişikliklerini inceliyoruz. DNA saç tokaları, iyi tanımlanmış bir kuvvet aralığı20,21'de basit iki durumlu geçişler sergiler ve bu nedenle cımbız kurulumunun performansını doğrulamak için oyuncak modeller olarak hizmet eder. SNARE proteinleri, membran füzyonu22'yi yönlendiren kuvvete duyarlı bir kompleks halinde bir araya geldikçe, tek moleküllü kuvvet spektroskopisi 14,23,24,25 ile de kapsamlı bir şekilde incelenmiştir. Verileri analiz etmek ve termodinamik ve kinetik hakkında yararlı bilgiler çıkarmak için standart yaklaşımlar sunulmaktadır. Bu makalenin, mekanobiyolojik çalışmalarda yüksek hassasiyetli MT'lerin benimsenmesini kolaylaştırabileceğini ve okuyucuları kendi kuvvete duyarlı ilgi sistemlerini keşfetmeye motive edebileceğini umuyoruz.
Protokol
Bu protokolde açıklanan tüm malzeme ve ekipmanlar Malzeme Tablosunda listelenmiştir. Aşağıda açıklanan yüksek hızlı MT kurulumunu çalıştırmak için LabVIEW yazılımı ve örnek verileri analiz etmek için MATLAB betikleri GitHub'da (https://github.com/ShonLab/Magnetic-Tweezers) depolanır ve genel kullanıma sunulur.
1. Aparat yapımı
NOT: Yüksek hızlı MT yapısının genel prensibi, yüksek hızlı tamamlayıcı metal oksit yarı iletken (CMOS) kamera ve yüksek güçlü, tutarlı bir ışık kaynağının kullanılması dışında, standart, geleneksel MT sistemlerine benzerdir (Şekil 1). Standart MT cihazlarının daha fazla açıklaması için diğer kaynaklara bakın 5,26,27.
- Titreşim önleyici optik masaya ters çevrilmiş bir mikroskop kurun. Yüksek hızlı bir CMOS kamera ve bir çerçeve tutucu takın.
- Mıknatısları 3D olarak manipüle etmek için bir çeviri aşaması oluşturun. Motorlu bir lineer sahneyi (>20 mm hareket) manuel bir XY aşamasının üzerine dikey olarak monte edin.
NOT: Dikey hareket kuvveti kontrol ederken, XY aşaması kurulumun ilk yapımı için mıknatısların optik eksene manuel olarak hizalanması içindir. - Mıknatısları döndürmek için bir döner step motor ve bir kayış ve kasnak sistemi takın.
NOT: Kayış, motor mili ile birbirinden birkaç santimetre uzaklıktaki mıknatıslar arasındaki döner hareketi iletir. Mıknatısların dönüşü, translasyonel manipülasyonun içindedir. - Mıknatısları monte edin. İki özdeş mıknatısı paralel olarak sıkıca barındırabilen, mıknatıslar arasında iyi tanımlanmış 1 mm'lik bir boşluk bulunan bir akrilik tutucu kullanın (Şekil 1B). Belirli bir mıknatıs çifti ile elde edilebilecek maksimum kuvveti kullanmak için, çeviri aşamasının dikey konumunu, mıknatısların alt yüzeyi en düşük konuma taşındığında örnek düzlemle hizalanacak şekilde ayarlayın.
NOT: Mıknatıslar28'in tutucu tasarımı ve konfigürasyonu hakkında daha fazla bilgi için Lipfert ve ark.'ya bakınız. Mıknatısların yüksekliği ve yönü, veri toplama ile birlikte LabVIEW yazılımı tarafından kontrol edilir. - Düşük büyütmeli objektif lensle görüntüleme yaparak, mıknatısları görüş alanının merkezine hizalayın. Mıknatısların döndürülmesinin, mıknatıs çiftinin merkezinin büyük bir yer değiştirmesine neden olmadığını kontrol edin.
NOT: Mıknatıslar arasındaki orta nokta dönme ekseni etrafında dönerse, kusurlu bir tutucu nedeniyle mıknatısların merkezden uzak olması muhtemeldir. Mıknatıs dönüşü sadece bağları kontrol etmek ve belirli uygulamalarda tork uygulamak için olduğundan, boşluk boyutuna göre küçük bir yanlış hizalama seviyesi tolere edilebilir. - Boncukların aydınlatılması için bir süper lüminesan diyot (SLD) takın. Kirişi iki mıknatıs arasındaki 1 mm'lik boşluktan geçirin. Kirişin boşluğa sığacak şekilde düzgün bir şekilde harmanlandığından ve aydınlatmanın mıknatıslar tarafından gölgelenmediğinden emin olun.
- Burun parçasına bir piezo lens tarayıcı takın ve boncuk izleme için 100x yağa daldırma objektif lens (sayısal diyafram [NA]: 1.45) takın. İzleme sonuçlarında olası yapıları önlemek için, mıknatıslar hareket ettirildiğinde aydınlatmanın eşit şekilde korunduğundan emin olun. Son olarak, pikselleri doyurmadan ışık seviyesini maksimum parlaklığa ayarlayın.
NOT: Boncukların yüksek hızlı takibi için farklı ışık kaynaklarının karşılaştırılması için, Dulin ve ark.29'a bakınız.
2. Manyetik kuvvetin kalibrasyonu
- Polimeraz zincir reaksiyonu kullanarak (PCR; bakınız Tablo 1), bir ucunda biyotin (yüzey bağlantısı için) ve diğer ucunda azid (boncuk bağlantısı için) ile etiketlenmiş 5 kbp dsDNA fragmanları (Primer B, Primer Z_5k ve λ-DNA kullanarak) hazırlayın.
- Bölüm 6'yı takiben, 5 kbp molekülleri ile bir akış hücresi hazırlayın.
- Bölüm 7'yi takiben, uzantısını ve dönüşünü doğrulayarak iyi bir boncuk bağı yapısını tanımlayın. Özellikle, merkezsiz bağlantı 30,31 nedeniyle boncuk yüksekliği ofsetini en aza indirmek için minimum dönme yörüngesine sahip (yani, yarıçap <200 nm) bir boncuk seçtiğinizden emin olun. İyi bir tether tanımlandıktan sonra, bölüm 9'a atıfta bulunarak boncuk izlemeye başlayın.
- Kurulum yeniyse, güvenilir yüksek çözünürlüklü ölçümler için gürültüsünü ve kararlılığını karakterize edin. Mıknatısı akış hücresi yüzeyinden ~3 mm uzağa yerleştirin (>10 pN uygulamak ve bir boncuğun Brownian hareketini bastırmak için), boncuğun z-konumunu 1,2 kHz'de izleyin ve z-koordinat zaman serisi32,33'ten Allan sapmasını (AD) hesaplayın (Şekil 2C). Birkaç nanometrenin AD değerlerinin yüksek hızlı rejimde (<0.1 s) elde edilebildiğini ve diferansiyel izlemenin (referans boncuğa göre manyetik boncuk konumu) AD'yi daha uzun zaman ölçeğinde azalttığını kontrol edin.
NOT: Genellikle maksimum hızda (1,2 kHz veya 0,83 ms çözünürlük) <3 nm'lik bir AD elde ederiz ve AD en az 10 sn'ye kadar azalmaya devam eder, bu da minimum sapma anlamına gelir. Diğerleri benzer kurulumlarda benzer değerler bildirmiştir 9,10,11,12,34. - Dinlenme konumundaki mıknatıslarla (F ~ 0 pN), bağlı boncuğun x ve y koordinatlarını 1,2 kHz'de kaydedin. Brownian hareketinin yeterince örneklenmesi için pozisyonu yeterince uzun bir süre (yani, dalgalanma35'in karakteristik gevşeme süresinden yeterince uzun) kaydedin.
NOT: Burada, x yönü manyetik alanın yönü boyuncadır, oysa y'deki hareket alana dik olan enine hareketi temsil eder. - Mıknatısları akış hücresine yaklaştırın ve mıknatıslar akış hücresinin üst kısmına hafifçe dokunana kadar boncuk pozisyonu ölçümlerini tekrarlayın. Mıknatıslar numune düzleminden 7 mm'den daha uzaktayken (uygulanan kuvvet mıknatısların uzak alanında yavaşça arttığından) büyük adımlarla (örneğin, 1-2 mm) hareket edin, ancak daha yüksek kuvvet seviyelerinde daha ince kalibrasyon için yaklaştıkça adım boyutunu kademeli olarak azaltın (örneğin, 0,1-0,5 mm).
- İki alternatif yöntemden birini kullanarak her mıknatıs konumundaki kuvveti hesaplayın, d) (her iki yöntemi de içeren bir MATLAB betiği "force calibration.m" sağlanır; Ek Dosya 1'e bakınız).
- Boncuğun y-koordinatlarının
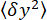 varyansını (Şekil 2D) ve boncuğun ortalama z-konumunu
varyansını (Şekil 2D) ve boncuğun ortalama z-konumunu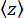 en düşük konuma göre ölçün (Şekil 2B, alt). Ardından, kuvveti tahmin etmek için (1)7,27,36 denklemini kullanın (sabit boncuk yarıçapı R = 1,400 nm ve termal enerji kR T = 4,11 pN∙nm ile):
en düşük konuma göre ölçün (Şekil 2B, alt). Ardından, kuvveti tahmin etmek için (1)7,27,36 denklemini kullanın (sabit boncuk yarıçapı R = 1,400 nm ve termal enerji kR T = 4,11 pN∙nm ile):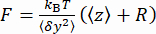 (1)
(1) - Alternatif olarak, y koordinatlarının güç spektral yoğunluğunu (PSD) hesaplayın, Sy (Şekil 2E). Denklemi (2) kullanarak ölçülen Sy'ye bir çift-Lorentzian model37 takarak uygulanan kuvvet F'yi belirleyin.
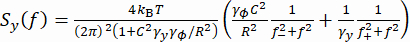 (2)
(2)
Burada, R boncuk yarıçapıdır, γ yve γφ sırasıyla translasyonel ve rotasyonel sürükleme katsayılarıdır (Stokes-Einstein denkleminden tahmin edilmiştir), kRT termal enerjidir,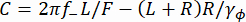 f + ve f - denklem kullanılarak elde edilen iki karakteristik frekanstır (3).
f + ve f - denklem kullanılarak elde edilen iki karakteristik frekanstır (3).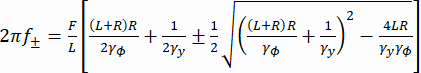 (3)
(3)
NOT: Tether uzantısı L, iyi kurulmuş solucan benzeri zincir (WLC) modelini izleyen bir kuvvet fonksiyonu olduğundan, yukarıdaki ifadeler F'yi tek uygun parametre olarak bırakır (R'yi basitlik için 1.400 nm olarak sabitleriz, çünkü tüm kuvvet seviyelerinde paylaşılır ve kesin değer sonuçları önemli ölçüde etkilemez). Gerektiğinde, kamera tabanlı görüntü alımından kaynaklanan hareket bulanıklığı ve örtüşme38,39 olarak düşünülmelidir, ancak bu etki, 5 kbp bağlamalarla 1 kHz'in üzerindeki yüksek hızlı ölçümlerimizde ihmal edilebilir.
- Boncuğun y-koordinatlarının
- Birkaç yapı daha için 2.4-2.7 arasındaki adımları yineleyin. Manyetik boncuklar arasındaki kuvvet değişkenliğinin ortalamasını almak için üç ila beş farklı boncuğu araştırın.
NOT: Ortalama için uygun yapı sayısını belirlemek için kullanılan manyetik boncuklar arasındaki kuvvet değişimi dikkate alınmalıdır. Bu değişkenlik küçüktür, ancak ticari ürünler için bile ölçülen kuvvette 1 pN'den fazla hataya yol açabilir31. İlgili kuvvetlerin mutlak olarak belirlenmesinin çok önemli olmadığı çoğu uygulama için, üç ila beş boncuğun kalibrasyon sonuçlarının ortalamasının alınması genellikle yeterlidir. Bu varyasyonu açıklamak için alternatif bir yaklaşım, deneyin başlangıcında kuvveti bireysel bağlarla ölçmektir, bu da zaman alıcı olabilir. Diğer bir seçenek, her yapı31'de bilinen kuvvet seviyelerinde açılan saç tokası yapılarını gömmektir. - Ölçülen kuvveti mıknatıs mesafesinin bir fonksiyonu olarak çizin ve denklemi (4) kullanarak verilere çift üstel bir fonksiyon sığdırın (Şekil 2F).
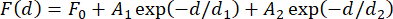 (4)
(4)
Burada, F0 (taban çizgisi), A 1 ve A2 (genlikler) ve d 1 ve d 2 (bozunma sabitleri) uygun parametrelerdir. İki yöntemden gelen kuvvet değerlerinin ve bunun sonucunda ortaya çıkan çift üstel uyumların büyük ölçüde aynı fikirde olduğundan emin olun (Şekil 2F, G).
NOT: Kuvvet kalibrasyonunun düzgün bir şekilde yapıldığını doğrulamak için, ölçülen kuvvete karşı uzantıyı çizerek problanan yapıların kuvvet-uzatma ilişkisini doğrulayın. - Manyetik boncukların30,31 kuvvete bağlı eğilmesindenkaynaklanan boncuk yüksekliği ofseti z'yi düzeltmek için, denklemi (5) kullanarak boncuk yarıçapına sahip merkezsiz bir bağlantının geometrisini göz önünde bulundurarak, yanal ofset xkapalıdan z'yi tahmin edin ve değerleri ölçülen uzatma değerlerine uygulayın. Bu adım, "force calibration.m" (satır 252-254) MATLAB betiğinde uygulanır.
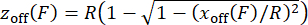 (5)
(5)
NOT: Bu düzeltme, özellikle küçük bir dönme yarıçapına (<200 nm) sahip boncuklar için uzantıda küçük değişiklikler yapsa da, bu ofset genellikle Şekil 2H'den Şekil 2I 30,31'e yapılan değişiklikte görüldüğü gibi elastik yanıtı kritik olarak etkiler. - Denklemi (6) kullanarak verilere genişletilebilir bir WLC modeli takarak L p kalıcılık uzunluğunu kontrol edin.
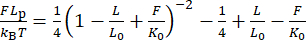 (6)
(6)
Burada, L 0 kontur uzunluğudur (5 kbp için 1.7 μm) ve K0 entalpik germe modülüdür.
NOT: Her ne kadar dsDNA'nın L p'sinin fosfat tamponlu salin (PBS) gibi tipik bir tamponda 40-50 nm olduğu kabul edilse de, kısa moleküllere (<5 kbp) uygulanan WLC formülü, L0 31,40'ı azalttığı için Lp'yi sistematik olarak hafife almaktadır. Bunun nedeni, klasik WLC modelinin, zincir uzunluğu kalıcılık uzunluğundan yeterince uzun olan bir polimeri varsaymasıdır. Burada, 5 kbp yapısı için Lp = 40 ± 3 nm elde ettik (Şekil 2H) ve uzatma düzeltmesi ayrıca 1.100 ± 200 pN'lik homojen bir K0 verdi (Şekil 2I). Sonlu bir WLC modeli 31,40'ın yanı sıra uzatma dağılımı 41'de Gaussiyanlık olmayan bir düzeltme uygulanması, Lp'yi biraz artıracaktır. - Kuvvet kalibrasyonu doğrulandıktan sonra, çift üstel modelin elde edilen uydurma parametrelerini sağlanan LabVIEW yazılımına (Ek Dosya 2) uygulayın ve yazılımın mevcut kuvveti motor okumalarından (yani mıknatıs konumu) gerçek zamanlı olarak hesaplamasını bekleyin. d(F) ters fonksiyonu için analitik bir ifade mevcut olmadığından, d hedef kuvvet düzeylerinin sayısal tahminini yaparak 0,1 pN adımlarında d'ye karşı F'nin bir arama tablosunu hazırlayın. Kuvvet kontrolünü komuta etmek için bu tabloyu yazılımda da saklayın.
3. DNA saç tokalarının sentezi
NOT: MT deneyleri için DNA saç tokası yapıları, λ-DNA'daki 510 bp'lik bir bölgenin PCR amplifikasyonu ile iki özel primer ile hazırlanır, bunlardan biri 5'-ucunda bir saç tokası yapısı içerir (Şekil 3A). Bu sayede PCR ürününün bir ucuna saç tokası motifi yerleştirilmiş olur.
- Astarları hazırlayın.
- İleri astar: Cam yüzey bağlantısı için 5′-biotin etiketli ve λ-DNA'ya bağlanan astar B_hp. Bu astar, λ-bağlama bölgesine 5′ 8 bp saplı ve 6 nt ilmekli bir saç tokası motifi içerir.
- Ters astar: Manyetik boncuk bağlantısı için 5′-azid etiketli olan ve ileri astardan 1 kbp uzakta λ-DNA'ya bağlanan astar Z_hp.
- PCR'yi λ-DNA (şablon), nTaq polimeraz ve standart PCR koşullarıyla ayarlayın ve çalıştırın (bkz. Tablo 1). Ürünü ticari bir arıtma kiti ile temizleyin.
- DNA konsantrasyonunu 260 nm'de (A260) UV absorpsiyonu ile ölçün ve ürün boyutunu doğrulamak için agaroz jeli elektroforezi (% 2 jel) uygulayın (bakınız Tablo 2). Tipik bir verim ~ 35 μL ~ 600 nM çözeltidir.
4. SNARE proteinlerinin hazırlanması
NOT: Nöronal SNARE kompleksleri, E. coli'den eksprese edilen üç saflaştırılmış sıçan proteininin birleştirilmesiyle birleştirilir: VAMP2 / synaptobrevin-2, syntaxin-1A ve SNAP-25 (Şekil 3B). Montajlarını kolaylaştırmak için, sözdizimi ve SNAP-25, bir VAMP2 parçası (N-terminal bölgesinden yoksun; "ΔN-VAMP2" olarak adlandırılır) ile "ΔN-kompleksi" adı verilen bir yapıya birlikte ifade edilir ve daha sonra tam kompleksler oluşturmak için DNA kolu bağlantısından sonra tam uzunlukta VAMP2 ile karıştırılır.
- SNARE proteinlerinin ekspresyonu için cDNA içeren plazmidler hazırlayın (tüm plazmidler için DNA dizileri Malzeme Tablosunda verilmiştir).
- Transmembran etki alanından yoksun 6×His-etiketli VAMP2'yi hazırlayın (2-97; Disülfür bağlantıları için L32C/I97C) bir pET28a vektörüne klonlanmıştır.
- Habc ve transmembran alanından (191-267, disülfit bağları için I202C / I266C ikameleri) yoksun olan sözdizimi-1A'yı, 6×His-etiketli ΔN-VAMP2 (49-96) ile birlikte klonlanmış bir pETDuet-1 vektörüne hazırlayın.
- Tam uzunluktaki SNAP-25 izoform b'yi (2-206, tüm C'den A'ya) bir pET28a vektörüne klonlanmış olarak hazırlayın. Bu, ΔN komplekslerini hazırlamak için kullanılacaktır.
- SNARE komplekslerini açıldıktan sonra yeniden birleştirmek üzere MT tahlil tamponuna doğrudan eklenmek üzere bir pET28a vektörüne klonlanmış 6×His-etiketli tam uzunlukta SNAP-25 izoform b'yi (1-206, tüm C'den A'ya) hazırlayın.
- Rosetta (DE3) E. coli hücrelerinin iki tüpünü hazırlayın. ΔN kompleksini ifade etmek için bir grubu VAMP2 plazmidleriyle (adım 4.1.1'den), biri hem sözdizimi-1A/ΔN-VAMP2 hem de etiketsiz SNAP-25 plazmidleriyle (adım 4.1.2 ve 4.1.3'ten) ve diğeri His etiketli SNAP-25 plazmidleriyle (adım 4.1.4'ten) dönüştürün.
- Dönüştürülmüş hücreleri uygun antibiyotiklerle Luria-Bertani suyuna (LB) aktarın (burada, VAMP2 için kanamisin ve kloramfenikol ve His-etiketli SNAP-25; kanamisin, kloramfenikol ve ΔN kompleksi için ampisilin). Et suyunun optik yoğunluğu (OD) 0.7-0.9'a ulaşana kadar onları bir sallama inkübatöründe (220 rpm) 37 ° C'de büyütün.
- Protein ekspresyonunu indüklemek için 1 mM izopropil β-d-1-tiyogalaktopiranosid (IPTG) ekleyin ve hücreleri 37 ° C'de 3-4 saat boyunca çalkalayan bir inkübatörde (220 rpm) inkübe edin.
- Kültürü 4.500 × g'da 4 ° C'de 15 dakika boyunca santrifüj ederek hücreleri pelet haline getirin.
- Protein saflaştırma için tamponlar hazırlayın (bakınız Tablo 2).
- SNARE eksprese eden hücre peletlerini 40 mL buz gibi soğuk lizis tamponunda askıya alın ve hücreleri buz üzerinde sonikasyonla lize edin (% 15 genlik, 5 s açık ve 5 s kapalı, toplam 30 dakika).
- Çözünmeyen malzemeleri gidermek için lizatı 15.000 × g'da 4 ° C'de 30 dakika boyunca santrifüj edin.
- Süpernatantı 1 mL Ni-NTA reçinesi ile doldurulmuş bir yerçekimi sütunundan geçirin. Reçineyi yıkama tamponu A, ardından yıkama tamponu B ile yıkayın ve proteinleri 10 mL elüsyon tamponu ile boşaltın.
- Bir tuzdan arındırma sütunu kullanarak tris (2-karboksiyetil) fosfin (TCEP) ve imidazolün elüentten çıkarılması (üreticinin talimatlarını izleyin). PBS ile numuneyi boşaltın.
- Proteinleri PBS'de tutarken (tipik olarak 2 mL üreten) proteinleri santrifüj filtrelerle (10 kDa kesme) ~ 70 μM'ye kadar konsantre edin. Protein konsantrasyonunu 280 nm'de (A280) ultraviyole (UV) absorpsiyonu veya Bradford testi ile ölçün.
- Küçük alikotlar hazırlayın, sıvı azotta flaş dondurun ve kullanıma kadar -80 ° C'de saklayın.
NOT: Tam SNARE kompleksleri, ΔN kompleksini bir DNA tutamağı üzerinde konjuge ettikten sonra birleştirilecektir (aşağıya bakınız).
5. DNA tutamaçlarının bağlanması
NOT: Bir ucunda primer amin grupları içeren iki adet 510 bp dsDNA tutamacı önce PCR ile hazırlanır ve amin grupları daha sonra iki işlevli bir çapraz bağlayıcı olan SM (PEG)2 kullanılarak maleimid gruplarına dönüştürülür. İki tutamak daha sonra bölgeye özgü konjugasyon için sistein grupları aracılığıyla SNARE komplekslerine kovalent olarak bağlanır (Şekil 3B).
- Astarları hazırlayın.
- İleriye doğru astarları hazırlayın: Cam yüzey bağlantısı için 5′-biotin etiketli olan ve λ-DNA'ya bağlanan Astar B (Sap B'yi yükseltmek için); Primer Z (Handle Z'yi yükseltmek için), manyetik boncuk bağlantısı için 5'-azid etiketlidir ve Primer B ile aynı sıraya sahiptir.
- Bir ters astar hazırlayın: Primer N (Handle B ve Handle Z için paylaşılan), protein konjugasyonu için 5′-amin etiketlidir ve ileri astardan uzakta λ-DNA 510 bp'ye bağlanır.
- λ-DNA (şablon), nTaq polimeraz ve standart PCR koşulları ile iki set PCR reaksiyonu (her tutamak için 200 μL reaksiyonun 18 tüpü) ayarlayın ve çalıştırın (bkz. Tablo 1). Ürünü bir PCR temizleme kiti ile temizleyin ve her bir kolu 45 μL ultra saf su ile temizleyin. Sonraki adımlarda etkili bir reaksiyon için yüksek konsantrasyonlarda tutamak elde etmek için minimum miktarda su kullanın.
- DNA konsantrasyonunu A260 ile ölçün. Tipik verim, her sap için ~650 μL ~2 μM çözeltidir. Agaroz jeli elektroforezinde daha sonra doğrulamak için küçük numuneleri ayrı tutun.
- Her tutamağı (PBS'de 1 μM) 5 mM SM(PEG)2 ile reaksiyona sokun. Nazik rotasyonla oda sıcaklığında inkübe edin. 1 saat sonra, reaksiyona girmemiş SM (PEG)2'yi çıkarmak için bir DNA saflaştırma kiti kullanın. ~2 μM çözeltileri elde etmek için her bir kolu 250 μL PBS ile aşın.
- PBS'de B sapı ve ΔN kompleksi çözeltilerini 1:16 molar oranında (örneğin, 1 μM Sap B ve 16 μM ΔN kompleksi) karıştırın ve oda sıcaklığında ajitasyonla 2 saat inkübe edin. Agaroz jeli elektroforezi için küçük bir örneği ayrı tutun.
- Önceki adımda kullanılan ΔN kompleksi üzerine 2,5 kat azı dişi fazlalığına bir VAMP2 çözeltisi ekleyin. Karışımı oda sıcaklığında ajitasyonla 1 saat daha inkübe edin. Tam SNARE kompleksleri bu adımda monte edilir.
- Taze PBS ve santrifüj filtre (100 kDa kesme) ile tampon değişimi yoluyla serbest proteinleri çıkarın: 4 ° C'de 5 dakika boyunca 14.000 × g'da santrifüj, en az 6x tekrarlayın ve son dönüş için 15 dakika çalıştırın. Serbest proteinlerin uzaklaştırılmasını izlemek için A260 / A280 oranındaki artışı ölçün. Agaroz jeli elektroforezi için küçük bir örneği ayrı tutun.
- Çözeltiye Sap Z'yi Sap B'nin üzerinde 15 kat azı dişi fazlalığında ekleyin. reaksiyonu kolaylaştırmak için Z Sapının konsantrasyonunu en az 1 μM'nin üzerinde tutun. Karışımı gece boyunca 4 ° C'de çalkalama ile inkübe edin.
- Ara ürünleri (Handle B ve protein konjugatları) ve nihai ürünü (iki kulplu SNARE kompleksi) agaroz jel elektroforezi (Şekil 3B, iç kısım) ile doğrulayın (bakınız Tablo 2).
NOT: Proteinler Sap B'ye başarıyla bağlanırsa, bir hareketlilik kayması tespit edilecektir. Özellikle, DNA saplarında tam SNARE komplekslerinin oluşumu, SDS'de demonte edilen ve DNA'ya bağlı sadece sözdizimi bırakan ΔN komplekslerinin aksine, sodyum dodesil sülfata (SDS) karşı dirençleri ile doğrulanabilir ( Şekil 3B'deki b ve c'yi karşılaştırın). - Küçük alikotlar hazırlayın, sıvı azotta flaş dondurun ve kullanıma kadar -80 ° C'de saklayın.
NOT: Nihai çözelti reaksiyona girmemiş tutamaklar içermesine rağmen, bir akış hücresindeki numune montajı sırasında yalnızca biyotin ve azid ile iki kez etiketlenmiş istenen yapı seçilecektir.
6. Akış hücrelerinin imalatı
NOT: MT ölçümleri için akış hücreleri, çift taraflı bantla birbirine bağlanmış iki cam kapaktan yapılmıştır (Şekil 3C). Bir kapak kayması, spesifik olmayan bağlanmayı önlemek ve hedef moleküllerin biyotin-NeutrAvidin bağlantısı yoluyla spesifik olarak bağlanmasını sağlamak için PEG ve biyotinile polietilen glikol (PEG) karışımı ile kaplanmıştır (Şekil 3D). Daha sonra, MT deneyleri için malzemelerin çözeltileri, bir şırınga pompası kullanılarak sırayla bir akış hücresine enjekte edilir (Şekil 3C, D).
- Her biri üst (24 mm × 50 mm, No. 1.5 kalınlık) ve alt (24 mm × 60 mm, No. 1.5 kalınlık) yüzey için birer tane olmak üzere iki cam kapak fişi hazırlayın. Kapak fişlerini 30 dakika boyunca 1 M KOH'da sonikasyonla temizleyin. Sonikasyondan sonra, kapakları damıtılmış suyla durulayın ve bir sonraki adıma kadar suda tutun.
- Yayınlanan protokoller42,43'ü izleyerek alt kapak kapağını PEGylate edin. Silanizasyon için N-[3- (trimetoksisilil)propil]etilendiamin ve 100 mM bikarbonat tamponunda 1:100 (ww) biotin-PEG-SVA ve mPEG-SVA karışımı kullanın. PEGile edilmiş kapak fişlerini -20 °C'de kuru tutun ve birkaç hafta saklayın.
- Deneylerin yapıldığı gün, PEGylated kapak fişlerini çıkarın ve bir azot tabancasıyla kurutun. Temiz olduklarından emin olmak için kir olup olmadığını görsel olarak kontrol edin.
- Numune kanallarını yapmak için, ~2 mm genişliğinde çift taraflı bant şeritleri hazırlayın ve birbirine paralel ve birbirinden ~5 mm ile ayrılmış bir alt kapak kayması (PEGylated yüzey yukarı) üzerine dört şerit yerleştirin (Şekil 3C).
NOT: Bu şekilde, tek bir akış hücresinde 5 mm genişliğinde üç numune kanalı oluşturulabilir. - Alt kapak kapağının ortasına bir üst kapak kayması yerleştirin ve kanal giriş ve çıkışları için kısa kenarlarda ~5 mm boşluk bırakın. Kanalları sıkıca kapatmak için üst kapak kapağının arkasına cımbızla hafifçe bastırın.
- Giriş haznesi yapmak için 200 μL pipet ucunun kenarını kesin. ~200 μL çözeltinin tutulmasına izin vermek için daha geniş açıklıktan ~10 mm kesin. Üç akış kanalı için bunlardan üçünü yapın. Çıkışları yapılandırmak için, şırınga pompasının borusuna uyan üç şırınga iğnesi hazırlayın.
- 5 dakikalık epoksi kullanarak, rezervuarları ve iğne göbeklerini akış hücresine yapıştırın. Sızıntıyı önlemek için tam bir conta oluşturulduğundan ve kanalların fazla yapıştırıcı ile tıkanmadığından emin olun. En az 30 dakika kurumaya bırakın.
7. Boncuk-bağ yapılarının montajı
NOT: MT deneyleri için malzemelerin çözeltileri, boncuk bağı yapıları için olanlar da dahil olmak üzere, bir şırınga pompası kullanılarak akış hücrelerine sırayla verilir (Şekil 3C, D).
- Manyetik boncuklar hazırlayın. Bir stok çözeltisinden 5 mg M270-epoksi boncuk alın (167.5 μL dimetilformamid içinde ~ 3.3 ×10 8 boncuk) ve çözücüyü boncukların manyetik olarak ayrılmasıyla fosfat tamponu ile değiştirin ( bakınız Tablo 2).
- Boncukları ~ 1.1 × 109 boncuk mL-1'de 1 M amonyum sülfat içeren bir fosfat tamponunda hazırlayın ve 2 mM dibenzosiklooktin (DBCO)- NH2 ile reaksiyona sürün. Karışımı oda sıcaklığında dönen bir karıştırıcı üzerinde 3 saat inkübe edin. Reaksiyondan sonra, reaksiyona girmemiş molekülleri çıkarmak için boncukları taze fosfat tamponu ile 3 kat yıkayın.
NOT: Yıkanan boncuklar, kullanımdan önce birkaç hafta boyunca 4 °C'de ekstra rotasyon olmaksızın saklanabilir. - Akış hücresi kanal çıkışındaki bir iğneyi polietilen borulu şırınga pompasına bağlayın. PBS ile kanalları dengeleyin.
- Aşağıdaki çözeltileri pompa ile emerek kanala sırayla uygulayın: NeutrAvidin, hedef yapılar (DNA saç tokaları veya DNA kulplu SNARE kompleksleri), referans polistiren boncuklar ve DBCO kaplı manyetik boncuklar. Kullanmadan önce, potansiyel boncuk agregalarını dağıtmak için boncuk çözeltilerini iyice vorteksleyin.
- 0,1 pN kuvvet uygularken bağlanmamış boncukları yıkayın.
NOT: Küçük bir yukarı doğru kuvvet uygulanması, bağlanmamış boncukların çıkarılmasını kolaylaştırır ve özel olarak bağlanmış boncuk bağı yapılarının kopmasını önlemeye yardımcı olur. - SNARE kompleksleri ile yapılan deneyler için, son tampona 1,5 μM SNAP-25 ekleyin.
NOT: Serbest SNAP-25 molekülleri, açıldıktan sonra SNARE komplekslerini yeniden bağlayabilir ve tek bir kompleks üzerinde tekrarlanan ölçümlere izin verebilir.
8. Hedef yapıların belirlenmesi
- Bir akış hücresi kanalının yüzeyinde, hedef yapının tek molekülleri tarafından bağlanan manyetik boncukları arayın. Yakınlarda bir referans boncuk bulunduğundan emin olun.
- Bir aday boncuğu döndürün ve serbestçe dönüp dönmediğini kontrol edin. Boncuk birden fazla molekül tarafından bağlanırsa, kısıtlı bir hareket sergiler.
- Boncuğu birkaç tam tur için döndürün ve dönme yarıçapını bulun (bu işlev sağlanan yazılımda uygulanır). Tercihen, küçük bir dönme yarıçapına sahip bir boncuk seçin.
NOT: Bu yarıçap, boncuğun bağ ekseninden ne kadar merkezsiz olduğunu gösterir ve bu, boncuk-bağ montajı30,31 sırasında rastgele belirlenir. Tüm deneylerde, bir boncuğun minimum merkezden ayrılması, kullandığımız yüksek boncuk yarıçapı - tether uzatma oranı ile ilişkili birçok eseri hafifletir. - İyi tek bağlı boncukları tanımlamak için kuvveti 0'dan 5 pN'ye yükseltin. 1 kbp'lik bir bağın (veya eşdeğer iki 510 bp tutamacının) gerilmesinden kaynaklanan bir boncuğun kırınım düzeninde büyük bir değişiklik olup olmadığına bakın. Kırınım deseni önemli ölçüde değişmezse, kuvveti sıfıra indirin ve başka bir aday boncuk için tarama yapın.
NOT: Bir boncuğun ~ 300 nm kaldırılması, izleme işlemine gerçekten başlamadan ham görüntülerden kolayca fark edilebilir.
9. Uzatma ölçümleri için boncuk izleme
NOT: Boncukların izlenmesi, bu makaleyle birlikte verilen LabVIEW yazılımında boncuk görüntüleri gerçek zamanlı olarak analiz edilerek gerçekleştirilir. İzleme yöntemi ve varyantları konvansiyonel MT sistemlerinin çoğunda kullanılmıştır ve önceki literatürdeaçıklanmıştır 2,5,7,26. Manyetik bir boncuğun sabit bir referans boncuğa göre konumunu ölçerek (yani, diferansiyel izleme), konum ölçümleri harici bir bozulmaya karşı son derece sağlam hale gelir.
- Uygun bir manyetik boncuk bir referans boncukla birlikte yerleştirildikten sonra, boncuk izlemeye hazırlanmaya başlamak için Kalibre Et düğmesine tıklayın.
- Boncukların yerlerini tanımlamak için resimdeki boncuklara tıklayın. Görüntüler daha sonra boncukların etrafındaki ilgi çekici bölgelere (ROI'ler) (örneğin, 3 μm boncuk için 150 x 150 piksel) kırpılır ve daha sonra kesin boncuk koordinatlarını çıkarmak için daha fazla analiz edilir.
- Mıknatıs dönüşünün tamamlanmasını bekleyin. Bu işlem, boncuk31'in merkezsiz bağlantısını belgelemek için mıknatısları döndürürken boncuğun x ve y koordinatlarını kaydeder (2D çapraz korelasyon44'ü hesaplayarak veya boncuk görüntülerinin radyal simetri45'ini kullanarak, karşılaştırılabilir performansla).
- Z yönünde izleme için, yazılımın odak düzleminden farklı mesafelerdeki boncukların kırınım görüntülerinin bir arama tablosunu oluşturmasını bekleyin. Bu, objektif lensi eşit mesafeli adımlarla bir piezo tarayıcı ile basamaklandırarak ve her konumda dalgalanma ortalamalı boncuk görüntüleri kaydederek gerçekleştirilir. Daha sonra, gerçek deneylerdeki boncukların z-koordinatları, gerçek zamanlı boncuk görüntüleri interpolasyon7 ile arama tablosuyla karşılaştırılarak belirlenir.
- Arama tablosu oluşturma işlemi tamamlandığında, izleme ve otomatik odaklamayı etkinleştirin ( İzle ? ve AF? Düğmeler) tıklayın ve boncuk konumlarını kaydetmeye başlamak için Al düğmesine tıklayın.
NOT: Otomatik netleme isteğe bağlıdır ancak alma sırasında z cinsinden sahne kayması için düzeltilmesi önerilir.
10. Kuvvet uygulama şemaları
- Kuvvet rampası deneyleri: Yapının kuvvet-uzatma ilişkisini doğrulamak için, sabit bir yükleme hızında (± 1,0 pN s−1) yukarı ve aşağı bir kuvvet artışı uygulayın (Şekil 4A). Örneğin, yapının toplam uzunluğunu ve tutamaçların kuvvet uzatma eğrisini doğrulamak için 0-20-0 pN döngüsünün üç turunu uygulayın.
- Yazılımdaki tether parametrelerini belirleyerek, ölçülen verilerin üzerine bir WLC kuvvet uzatma eğrisi yerleştirin ve hedef boncuğun uygun DNA tutamaklarına sahip gerçek bir numune yapısı ile bağlanıp bağlanmadığını belirleyin. Yapının bilinen kontur uzunluğunu (örneğin, 1 kbp dsDNA için ~ 340 nm) ve WLC kalıcılık uzunluğunu (kısa dsDNA31 için 30-45 nm) başlangıç noktası olarak kullanın. Gerekirse adım 2.11'de açıklanan uzantı düzeltme yöntemini uygulayın.
- Yapı doğrulanırsa, hedef moleküller-saç tokaları veya SNARE komplekslerinden kaynaklanan ek uzantıyı aramak için kuvvet uzatma tepkisini ayrıntılı olarak inceleyin.
- Sabit kuvvet deneyleri: Hedef moleküllerin kuvvet duyarlılığını araştırmak için uygulanan kuvveti ayrı adımlarda kademeli olarak değiştirin (Şekil 4B).
NOT: MT'ler basit ve etkili sabit kuvvet deneylerine olanak tanır, çünkü mıknatıslar sabit tutulduğunda uygulanan kuvvet sabit tutulur.- DNA saç tokaları için, 0.2-0.5 pN adımlarla 4-8 pN kuvvet uygulayın ve her kuvvet seviyesinde ~ 10 s için boncuk pozisyonunu ölçün.
- SNARE kompleksleri için, 0.1-0.2 pN adımlarla 14-16 pN kuvvet uygulayın ve her kuvvet seviyesinde ~ 10 s için boncuk pozisyonunu ölçün.
- Kuvvet atlama deneyleri: SNARE komplekslerinin geçiş olaylarını gözlemleyin.
NOT: Sabit kuvvet deneyleri gibi kuvvet atlama deneyleri, kuvvet seviyelerindeki değişiklikleri içerir. Bununla birlikte, kuvvet sıçramaları, uygulanan kuvvette daha ani değişiklikler kullanır ve protein komplekslerinin ani bir kopması gibi araştırılan moleküllerde kuvvetle tetiklenen olayların izlenmesine izin verir. Örneğin, SNARE kompleksleri kuvvet döngüsü23'te yapısal histerezis sergilediğinden, kuvvet atlama deneyleri yapmak ve geçişe gecikmeyi ölçmek bilgilendiricidir (Şekil 4C).- Sıkıştırmayı açma: Bir VAMP2 molekülünün sağlam, üçlü bir SNARE kompleksinden soyulması, ikili bir sözdizimi-1A ve SNAP-25 kompleksi bırakılması.
- Rezipleme: Bozulmamış bir SNARE kompleksini yeniden oluşturmak için sıkıştırılmamış VAMP2 molekülünün sıkıştırılması.
- Açılım: SNAP-25'in tamamen ayrışması ile birlikte bir SNARE kompleksinin tamamen sökülmesi. Sadece VAMP2 ve sözdizimi molekülleri açıldıktan sonra yapıda kalır.
- Yeniden katlama: Tampondan serbest bir SNAP-25 molekülünün bağlanması üzerine bir SNARE kompleksinin rejenerasyonu.
- 2 pN'de, serbest bir SNAP25 molekülünün birleşmesini bekleyerek (~ 30 sn) bozulmamış bir SNARE kompleksinin montajını indükleyin. Bir SNARE kompleksinin oluşumu üzerine genişlemede ani bir azalma gözlenir.
- Sıkıştırma açma olaylarını gözlemlemek için, 10-12 pN'de birkaç saniye bekleyin ve ardından mümkün olan maksimum motor hızıyla aniden 14-15 pN'ye geçin. Hedef kuvvete bağlı olarak, SNARE kompleksi ya kısmen sıkıştırılmamış ara ürünler arasında (sabit kuvvet deneylerinde olduğu gibi) tersine çevrilebilir bir geçiş sergileyecek ya da rastgele bir bekleme süresinden (veya gecikmeden) sonra daha yüksek, sıkıştırılmamış bir duruma ~ 25 nm'lik bir sıçrama gösterecektir.
- Rezipleme olaylarını gözlemlemek için, sıkıştırma gözlendikten hemen sonra kuvveti 10-12 pN'ye düşürün. Yine, SNARE kompleksi, bazı rastgele gecikmelerden sonra daha düşük, fermuarlı duruma stokastik bir geçiş sergiler. Sıkıştırmanın açılmasından sonra açılma meydana gelirse, bir SNAP-25 molekülü eksik olacağından, kompleks yeniden sıkıştırılamaz.
- Ortaya çıkan olayları gözlemlemek için, uzantıda (~ 2 nm) daha fazla bir artış tespit etmek için sıkıştırmanın açılması gözlendikten sonra daha uzun bir süre bekleyin.
11. Veri analizi
NOT: MT verileriyle yapılabilecek analiz türleri hedef sisteme bağlıdır. Bununla birlikte, Şekil 4'te açıklanan ilgili deneylerden yararlı bilgiler çıkarmak için yaygın yaklaşımlar vardır. Tüm analizler, bu makalede sağlanan özel kodlar kullanılarak MATLAB (R2021a) ile gerçekleştirilir. Bu kodlar, bu makalede sunulan verilerin aynısını kullanarak grafikler oluşturur. 100 Hz izlemeden elde edilen ham veriler doğrudan analiz için alınırken, 1,2 kHz izlemeden elde edilen verilerin, gürültüyü azaltmak için analizden önce (gürültü analizi hariç) tipik olarak medyan filtreli (beş noktalı bir kaydırma penceresiyle) olduğunu unutmayın.
- Kuvvet rampası deneyleri: Kuvvet-uzatma ilişkisini (örneğin, polimerlerin elastikiyeti) analiz edin ve nanomekanik özellikler hakkında bilgi elde etmek için kuvveti değiştirin.
- Sabit kuvvet deneyleri: Konformasyonel değişikliklerin yapısal (örneğin, geçişte yer alan bölgeler), termodinamik (örneğin, serbest enerji farkı) ve kinetik (örneğin, enerji bariyeri) parametrelerini çıkarmak için kuvvetin bir fonksiyonu olarak durum popülasyonlarını ve bekleme süresini (veya geçiş oranını) analiz edin.
- Kuvvet atlama deneyleri: Hedef moleküllerin ve durumlarının stabilitesini elde etmek için kopma kinetiğini (örneğin, protein-protein etkileşimleri ve reseptör-ligand bağlanması) veya geçici ara ürünlerin ömrünü (örneğin, biyomoleküllerin açılması) analiz edin.
- Temsili uygulamalar olarak, DNA saç tokaları ve SNARE kompleksleri için örnek verileri analiz edin:
- Bir DNA saç tokasının iki durumlu geçişleri: sıkıştırma kuvveti, açılma mesafesi, popülasyon kaymasının kuvvet bağımlılığı ve gizli bir Markov modeli (HMM) ile durum ataması ve geçiş hızı ölçümleri (MATLAB kodları sağlanır).
- SNARE komplekslerinin konformasyonel değişiklikleri: sıkıştırma kuvveti, ara durumların kuvvet bağımlılığı ve sıkıştırma gecikmesini açma, yeniden sıkıştırmada histerezis ve açılma / yeniden katlama davranışı.
NOT: DNA tutamaçları, DNA saç tokaları ve SNARE kompleks konformasyonları için kuvvet uzatma modelleri önceki referanslardaverilmiştir 14,31.
Sonuçlar
Kuvvet kalibrasyonu
İki kuvvet ölçüm yönteminden (boncukların yanal yer değiştirme varyansı ve güç spektrumu analizi) elde edilen sonuçlar 0-2 pN arasında farklılık göstermiştir (Şekil 2G). Şekil 2F'deki sonuçlara göre, normal neodimyum mıknatıslarla 30 pN'ye kadar güvenilir bir şekilde ulaşabiliriz.
8 bp DNA saç tokasının iki durumlu geçişleri
İlk önce kısa bir...
Tartışmalar
Bu çalışmada, biyomoleküllerin yapısal değişikliklerini yüksek uzaysal zamansal hassasiyette gözlemleyebilen tek moleküllü bir kuvvet spektroskopisi kurulumunu tanıttık. Kullanılan yüksek hızlı CMOS kamera, 1.280 x 1.024 çözünürlükte 1.200 kare s−1 elde ederek 1,2 kHz boncuk izleme sağlar. Bununla birlikte, ölçümlerin hızı şu anda boncuk izleme yazılımı ile sınırlıdır, bu nedenle yatırım getirisi genellikle yüksek hızlı ölçümlerde daha küçük alanlara indirgenir....
Açıklamalar
Yazarların beyan edecekleri çıkar çatışmaları yoktur.
Teşekkürler
Bu çalışma, Kore hükümeti (MSIT) tarafından finanse edilen Kore Ulusal Araştırma Vakfı (NRF) hibesi tarafından desteklenmiştir (NRF-2022R1C1C1012176, NRF-2021R1A4A1031754 ve NRF- 2021R1A6A1A10042944). S.-H.R. NMG hibesi (2021R1C1C2009717) tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Materials for construct synthesis | |||
| Agarose gel electrophoresis system | Advance | Mupid-2plus | |
| DNA ladder | Bioneer | D-1037 | |
| nTaq polymerase | Enzynomics | P050A | |
| PCR purification kit | LaboPass | CMR0112 | |
| PEGylated SMCC crosslinker / SM(PEG)2 | ThermoFisher Scientific | 22102 | For SNARE–DNA coupling |
| Primer B | Bioneer | 5'-Biotin/TCGCCACCATCATTTCCA-3' | For 5-kbp force calibration construct and DNA handles |
| Primer B_hp | IDT | 5'-Biotin/TTTTTTTTTTGTTCTCTATTT TTTTAGAGAAC /AP site/ /AP site/ TCGCCACCATCATTTCCA-3' | For hairpin construct |
| Primer N | Bioneer | 5'-C6Amine/CATGTGGGTGACGCGAAA-3' | For DNA handles |
| Primer Z | Bioneer | 5'-Azide/TCGCCACCATCATTTCCA-3' | For DNA handles |
| Primer Z_5k | Bioneer | 5'-Azide/TTAGAGAGTATGGGTATATGACA TCG-3' | For 5-kbp force calibration construct |
| Primer Z_hp | Bioneer | 5'-Azide/GTGGCAGCATGACACC-3' | For hairpin construct |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| λ-DNA | Bioneer | D-2510 | Template strand for PCR |
| DNA sequences for SNARE proteins | |||
| 6×His-tagged SNAP-25b (2-206; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctat gagaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagcgg aagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCACA GCAGCGGCCTGGTGCCGCGC GGCAGCCATACTAGCGGAGAT ATCGCCGAGGACGCAGACAT GCGCAATGAGCTGGAGGAGA TGCAGAGGAGGGCTGACCAG CTGGCTGATGAGTCCCTGGA AAGCACCCGTCGCATGCTGC AGCTGGTTGAAGAGAGTAAA GATGCTGGCATCAGGACTTT GGTTATGTTGGATGAGCAAG GCGAACAACTGGAACGCATT GAGGAAGGGATGGACCAAAT CAATAAGGACATGAAAGAAG CAGAAAAGAATTTGACGGAC CTAGGAAAATTCGCCGGCCT TGCCGTGGCCCCCGCCAAC AAGCTTAAATCCAGTGATGC TTACAAAAAAGCCTGGGGC AATAATCAGGATGGAGTAGT GGCCAGCCAGCCTGCCCG TGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTG GCTTCATCCGCAGGGTAAC AAATGATGCCCGGGAAAAT GAGATGGATGAGAACCTG GAGCAGGTGAGCGGCATC ATCGGAAACCTCCGCCAC ATGGCTCTAGACATGGGCA ATGAGATTGACACCCAGA ATCGCCAGATCGACAGGA TCATGGAGAAGGCTGATT CCAACAAAACCAGAATTG ATGAAGCCAACCAACGTG CAACAAAGATGCTGGGAA GTGGTTAAggatccgaattcgag ctccgtcgacaagcttgcggccgcactc gagcaccaccaccaccaccactgagat ccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgag caataactagcataaccccttggggcct ctaaacgggtcttgaggggttttttgctga aaggaggaactatatccggat | |
| 6×His-tagged VAMP2 (2-97, L32C/I97C; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCAC AGCAGCGGCCTGGTGCCGC GCGGCAGCCATATGGCAGAT CTCTCGGCTACCGCTGCCAC CGTCCCGCCTGCCGCCCCG GCCGGCGAGGGTGGCCCCC CTGCACCTCCTCCAAATCTTA CCAGTAACAGGAGATGCCAG CAGACCCAGGCCCAGGTGG ATGAGGTGGTGGACATCATG AGGGTGAATGTGGACAAGGT CCTGGAGCGAGACCAGAAG CTATCGGAACTGGATGATCG CGCAGATGCCCTCCAGGCA GGGGCCTCCCAGTTTGAAA CAAGTGCAGCCAAGCTCAA GCGCAAATACTGGTGGAAA AACCTCAAGATGATGTGCTA Aggatccgaattcgagctccgtcg acaagcttgcggccgcactcgagcaccacca ccaccaccactgagatccggctgctaacaaa gcccgaaaggaagctgagttggctgctgcca ccgctgagcaataactagcataaccccttgg ggcctctaaacgggtcttgaggggttttttg ctgaaaggaggaactatatccggat | |
| 6×His-tagged ΔN-VAMP2 (49–96; capitalized) and Syntaxin-1A (191–267, I202C/I266C; capitalized) in pETDuet-1 | homemade | ggggaattgtgagcggataacaattcccctc tagaaataattttgtttaactttaagaagga gatataccATGGGCAGCAGCCATCA TCATCATCATCACAGCAGCGG CCTGGAAGTTCTGTTCCAGGG GCCCGGTAATGTGGACAAGGT CCTGGAGCGAGACCAGAAGCT ATCGGAACTGGATGATCGCGC AGATGCCCTCCAGGCAGGGGC CTCCCAGTTTGAAACAAGTGC AGCCAAGCTCAAGCGCAAATAC TGGTGGAAAAACCTCAAGATGAT GTAAgcggccgcataatgcttaagtcgaaca gaaagtaatcgtattgtacacggccgcataa tcgaaattaatacgactcactataggggaat tgtgagcggataacaattccccatcttagta tattagttaagtataagaaggagatatacat ATGGCCCTCAGTGAGATCGAGA CCAGGCACAGTGAGTGCATC AAGTTGGAGAACAGCATCCG GGAGCTACACGATATGTTCAT GGACATGGCCATGCTGGTGG AGAGCCAGGGGGAGATGATT GACAGGATCGAGTACAATGTG GAACACGCTGTGGACTACGTG GAGAGGGCCGTGTCTGACACC AAGAAGGCCGTCAAGTACCAG AGCAAGGCACGCAGGAAGAA GTGCATGATCTAActcgagtc tggtaaagaaaccgctgctgcgaaatttgaa cgccagcacatggactcgtctactagcgcag cttaattaacctaggctgctgccaccgctga gcaataactagcataaccccttggggcctct aaacgggtcttgaggggttttttgctgaaag gaggaactatatccggattggcgaatgggac gcgccctgtagcggcgcattaagcgcggcgg gtgtggtggttacgcgcagcgtgaccgctac acttgccagcgccctagcgcccgctcctttc gctttcttcccttcctttctcgccacgttcg ccggctttccccgtcaagctctaaatcgggg gctccctttagggttccgatttagtgcttta cggcacctcgaccccaaaaaacttgattagg gtgatggttcacgtagtgggccatcgccctg atagacggtttttcgccctttgacgttggag tccacgttctttaatagtggactcttgttcc aaactggaacaacactcaaccctatctcggt ctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctga tttaacaaaaatttaacgcgaattttaacaa aatattaacgtttacaatttctggcggcacg atggcatgagattatcaaaaaggatcttcac ctagatccttttaaattaaaaatgaagtttt aaatcaatctaaagtatatatgagtaaactt ggtctgacagttaccaatgcttaatcagtga ggcacctatctcagcgatctgtctatttcgt tcatccatagttgcctgactccccgtcgtgt agataactacgatacgggagggcttaccatc tggccccagtgctgcaatgataccgcgagac ccacgctcaccggctccagatttatcagcaa taaaccagccagccggaagggccgagcgca gaagtggtcctgcaactttatccgcctccatc cagtctattaattgttgccgggaagctagag taagtagttcgccagttaatagtttgcgcaa cgttgttgccattgctacaggcatcgtggtg tcacgctcgtcgtttggtatggcttcattca gctccggttcccaacgatcaaggcgagttac atgatcccccatgttgtgcaaaaaagcggtt agctccttcggtcctccgatcgttgtcagaa gtaagttggccgcagtgttatcactcatggt tatggcagcactgcataattctcttactgtc atgccatccgtaagatgcttttctgtgactg gtgagtactcaaccaagtcattctgagaata gtgtatgcggcgaccgagttgctcttgcccg gcgtcaatacgggataataccgcgccacata gcagaactttaaaagtgctcatcattggaaa acgttcttcggggcgaaaactctcaaggatc ttaccgctgttgagatccagttcgatgtaac ccactcgtgcacccaactgatcttcagcatc ttttactttcaccagcgtttctgggtgagcaaa aacaggaaggcaaaatgccgcaaaaaagg gaataagggcgacacggaaatgttgaatact catactcttcctttttcaatcatgattgaag catttatcagggttattgtctcatgagcgga tacatatttgaatgtatttagaaaaataaac aaataggtcatgaccaaaatcccttaacgtg agttttcgttccactgagcgtcagaccccgt agaaaagatcaaaggatcttcttgagatcct ttttttctgcgcgtaatctgctgcttgcaaa caaaaaaaccaccgctaccagcggtggtttg tttgccggatcaagagctaccaactcttttt ccgaaggtaactggcttcagcagagcgcaga taccaaatactgtccttctagtgtagccgta gttaggccaccacttcaagaactctgtagca ccgcctacatacctcgctctgctaatcctgt taccagtggctgctgccagtggcgataagtc gtgtcttaccgggttggactcaagacgatag ttaccggataaggcgcagcggtcgggctgaa cggggggttcgtgcacacagcccagcttgga gcgaacgacctacaccgaactgagataccta cagcgtgagctatgagaaagcgccacgcttccc gaagggagaaaggcggacaggtatccggta agcggcagggtcggaacaggagagcgcac gagggagcttccagggggaaacgcctggtatc tttatagtcctgtcgggtttcgccacctctg acttgagcgtcgatttttgtgatgctcgtca ggggggcggagcctatggaaaaacgccagc aacgcggcctttttacggttcctggccttttg ctggccttttgctcacatgttctttcctgcg ttatcccctgattctgtggataaccgtatta ccgcctttgagtgagctgataccgctcgccgc agccgaacgaccgagcgcagcgagtcagtg agcgaggaagcggaagagcgcctgatgcgg tattttctccttacgcatctgtgcggtatttc acaccgcatatatggtgcactctcagtacaa tctgctctgatgccgcatagttaagccagta tacactccgctatcgctacgtgactgggtca tggctgcgccccgacacccgccaacacccgc tgacgcgccctgacgggcttgtctgctcccg gcatccgcttacagacaagctgtgaccgtct ccgggagctgcatgtgtcagaggttttcacc gtcatcaccgaaacgcgcgaggcagctgcgg taaagctcatcagcgtggtcgtgaagcgatt cacagatgtctgcctgttcatccgcgtccag ctcgttgagtttctccagaagcgttaatgtc tggcttctgataaagcgggccatgttaaggg cggttttttcctgtttggtcactgatgcctc cgtgtaagggggatttctgttcatgggggta atgataccgatgaaacgagagaggatgctca cgatacgggttactgatgatgaacatgcccg gttactggaacgttgtgagggtaaacaactg gcggtatggatgcggcgggaccagagaaaaa tcactcagggtcaatgccagcgcttcgttaa tacagatgtaggtgttccacagggtagccag cagcatcctgcgatgcagatccggaacataa tggtgcagggcgctgacttccgcgtttccag actttacgaaacacggaaaccgaagaccatt catgttgttgctcaggtcgcagacgttttgc agcagcagtcgcttcacgttcgctcgcgtat cggtgattcattctgctaaccagtaaggcaa ccccgccagcctagccgggtcctcaacgaca ggagcacgatcatgctagtcatgccccgcgc ccaccggaaggagctgactgggttgaaggct ctcaagggcatcggtcgagatcccggtgcct aatgagtgagctaacttacattaattgcgtt gcgctcactgcccgctttccagtcgggaaac ctgtcgtgccagctgcattaatgaatcggcc aacgcgcggggagaggcggtttgcgtattgg gcgccagggtggtttttcttttcaccagtga gacgggcaacagctgattgcccttcaccgcc tggccctgagagagttgcagcaagcggtcca cgctggtttgccccagcaggcgaaaatcctg tttgatggtggttaacggcgggatataacat gagctgtcttcggtatcgtcgtatcccacta ccgagatgtccgcaccaacgcgcagcccgga ctcggtaatggcgcgcattgcgcccagcgcc atctgatcgttggcaaccagcatcgcagtgg gaacgatgccctcattcagcatttgcatggt ttgttgaaaaccggacatggcactccagtcg ccttcccgttccgctatcggctgaatttgat tgcgagtgagatatttatgccagccagccag acgcagacgcgccgagacagaacttaatggg cccgctaacagcgcgatttgctggtgaccca atgcgaccagatgctccacgcccagtcgcgt accgtcttcatgggagaaaataatactgttg atgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccac agcaatggcatcctggtcatccagcggatag ttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttc gacgccgcttcgttctaccatcgacaccacc acgctggcacccagttgatcggcgcgagatt taatcgccgcgacaatttgcgacggcgcgtg cagggccagactggaggtggcaacgccaatc agcaacgactgtttgcccgccagttgttgtg ccacgcggttgggaatgtaattcagctccgc catcgccgcttccactttttcccgcgttttc gcagaaacgtggctggcctggttcaccacgc gggaaacggtctgataagagacaccggcata ctctgcgacatcgtataacgttactggtttc acattcaccaccctgaattgactctcttccg ggcgctatcatgccataccgcgaaaggtttt gcgccattcgatggtgtccgggatctcgacg ctctcccttatgcgactcctgcattaggaag cagcccagtagtaggttgaggccgttgagca ccgccgccgcaaggaatggtgcatgcaagga gatggcgcccaacagtcccccggccacgggg cctgccaccatacccacgccgaaacaagcgc tcatgagcccgaagtggcgagcccgatcttc cccatcggtgatgtcggcgatataggcgcca gcaaccgcacctgtggcgccggtgatgccgg ccacgatgcgtccggcgtagaggatcgagat cgatctcgatcccgcgaaattaatacgactc actata | |
| SNAP-25b (1–206, all C to A; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcc agggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGCCGA GGACGCAGACATGCGCAATG AGCTGGAGGAGATGCAGAGG AGGGCTGACCAGCTGGCTGA TGAGTCCCTGGAAAGCACCC GTCGCATGCTGCAGCTGGTT GAAGAGAGTAAAGATGCTGG CATCAGGACTTTGGTTATGTT GGATGAGCAAGGCGAACAAC TGGAACGCATTGAGGAAGGG ATGGACCAAATCAATAAGGAC ATGAAAGAAGCAGAAAAGAAT TTGACGGACCTAGGAAAATTC GCCGGCCTTGCCGTGGCCCC CGCCAACAAGCTTAAATCCAG TGATGCTTACAAAAAAGCCTG GGGCAATAATCAGGATGGAGT AGTGGCCAGCCAGCCTGCCC GTGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTGGC TTCATCCGCAGGGTAACAAAT GATGCCCGGGAAAATGAGATG GATGAGAACCTGGAGCAGGT GAGCGGCATCATCGGAAACCT CCGCCACATGGCTCTAGACAT GGGCAATGAGATTGACACCCA GAATCGCCAGATCGACAGGAT CATGGAGAAGGCTGATTCCAA CAAAACCAGAATTGATGAAGC CAACCAACGTGCAACAAAGAT GCTGGGAAGTGGTTAA ctcgagcaccaccaccaccaccactgag atccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgagc aataactagcataaccccttggggcctc taaacgggtcttgaggggttttttgctgaa aggaggaactatatccggat | |
| Materials for protein purificaiton | |||
| 2-Mercaptoethanol | SIGMA | M3148-25ML | |
| Agar | LPS Solution | AGA500 | |
| Ampicillin, Sodium salt | PLS | AC1043-005-00 | |
| Chloramphenicol | PLS | CR1023-050-00 | |
| Competent cells (E. coli) | Novagen | 70956 | Rosetta(DE3)pLysS |
| Glycerol | SIGMA | G5516-500ML | |
| HEPES | SIGMA | H4034-100G | |
| Hydrochloric acid / HCl | SIGMA | 320331-500ML | |
| Imidazole | SIGMA | I2399-100G | |
| Isopropyl β-D-1-thiogalactopyranoside / IPTG | SIGMA | 10724815001 | |
| Kanamycin Sulfate | PLS | KC1001-005-02 | |
| Luria-Bertani (LB) Broth | LPS Solution | LB-05 | |
| Ni-NTA resin | Qiagen | 30210 | |
| PD MiniTrap G-25 (desalting column) | Cytiva | GE28-9180-07 | For instructions, see: https://www.cytivalifesciences.com/en/us/shop/chromatography/prepacked-columns/desalting-and-buffer-exchange/pd-minitrap-desalting-columns-with-sephadex-g-25-resin-p-06174 |
| Phenylmethylsulfonyl fluoride / PMSF | ThermoFisher Scientific | 36978 | |
| Plasmids for SNARE proteins | cloned in house | N/A | Available upon request |
| Protease inhibitor cocktail | genDEPOT | P3100 | |
| Sodium chloride | SIGMA | S5886-500G | |
| Sodium phosphate dibasic / Na2HPO4 | SIGMA | S7907-100G | |
| Sodium phosphate monobasic / NaH2PO4 | SIGMA | S3139-250G | |
| Tris(2-carboxyethyl)phosphine / TCEP | SIGMA | C4706-2G | |
| Trizma base | SIGMA | T1503-250G | |
| Materials for sample assembly | |||
| Biotin-PEG-SVA | LAYSAN BIO | BIO-PEG-SVA-5K-100MG & MPEG-SVA-5K-1g | For PEGylation |
| Dibenzocyclooctyne-amine / DBCO-NH2 | SIGMA | 761540-10MG | For bead coating |
| Double-sided tape | 3M | 136 | For flow cell assembly |
| Epoxy glue | DEVCON | S-208 | For flow cell assembly |
| Glass coverslip for bottom surface | VWR | 48393-251 | Rectangular, 60×24 mm, #1.5 |
| Glass coverslip for top surface | VWR | 48393-241 | Rectangular, 50×24 mm, #1.5 |
| Magnetic bead | ThermoFisher Scientific | 14301 | Dynabeads M-270 Epoxy, 2.8 μm |
| mPEG-SVA | LAYSAN BIO | mPEG-SVA 1g | For PEGylation |
| N,N-Dimethylformamide / DMF | SIGMA | D4551-250ML | For bead coating |
| N-[3-(trimethoxysilyl)propyl]ethylenediamine | SIGMA | 104884-100ML | For PEGylation |
| Neutravidin | ThermoFisher Scientific | 31000 | For sample tethering |
| Phosphate buffered saline / PBS, pH 7.2 | PLS | PR2007-100-00 | |
| Plastic syringe | Norm-ject | A5 | 5 ml, luer tip |
| Polyethylene Tubing | SCI | BB31695-PE/4 | PE-60 |
| Reference bead | SPHEROTECH | SVP-30-5 | Streptavidin-coated Polystyrene Particles; 3.0-3.4 µm |
| Syringe needle | Kovax | 21G-1 1/4'' | 21 G |
| Syringe pump | KD SCIENTIFIC | 788210 | |
| Equipment for magnetic tweezer instrument | |||
| 1-axis motorized microtranslation stage | PI | M-126.PD1 | For vertical positioning of magnets |
| 2-axis manual translation stage | ST1 | LEE400 | For alignment of magnets to the optical axis |
| Acrylic holder for magnets | DaiKwang Precision | custum order | Drawing available upon request |
| Frame grabber | Active Silicon | AS-FBD-4XCXP6-2PE8 | |
| High-speed CMOS camera | Mikrotron | EoSens 3CXP | |
| Inverted microscope | Olympus | IX73P2F-1-2 | |
| Neodymium magnets | LG magnet | ND 10x10x12t | Dimension: 10 mm × 10 mm × 12 mm; two needed |
| Objective lens | Olympus | UPLXAPO100XO | Oil-immersion, NA 1.45 |
| Objective lens nanopositioner | Mad City Labs | Nano-F100S | |
| Rotation stepper motor | AUTONICS | A3K-S545W | For rotating magnets |
| Superluminescent diode | QPHOTONICS | QSDM-680-2 | 680 nm |
| Software | |||
| LabVIEW | National Instruments | v20.0f1 | |
| MATLAB | MathWorks | v2021a |
Referanslar
- Le, S., Liu, R., Lim, C. T., Yan, J. Uncovering mechanosensing mechanisms at the single protein level using magnetic tweezers. Methods. 94, 13-18 (2016).
- Choi, H. -. K., Kim, H. G., Shon, M. J., Yoon, T. -. Y. High-resolution single-molecule magnetic tweezers. Annual Review of Biochemistry. 91 (1), 33-59 (2022).
- Yang, T., Park, C., Rah, S. -. H., Shon, M. J. Nano-precision tweezers for mechanosensitive proteins and beyond. Molecules and Cells. 45 (1), 16-25 (2022).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- De Vlaminck, I., Dekker, C. Recent advances in magnetic tweezers. Annual Review of Biophysics. 41 (1), 453-472 (2012).
- Bustamante, C. J., Chemla, Y. R., Liu, S., Wang, M. D. Optical tweezers in single-molecule biophysics. Nature Reviews Methods Primers. 1, 25 (2021).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical Journal. 82 (6), 3314-3329 (2002).
- Smith, S. B., Finzi, L., Bustamante, C. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258 (5085), 1122-1126 (1992).
- Lansdorp, B. M., Tabrizi, S. J., Dittmore, A., Saleh, O. A. A high-speed magnetic tweezer beyond 10,000 frames per second. Review of Scientific Instruments. 84 (4), 044301 (2013).
- Cnossen, J. P., Dulin, D., Dekker, N. H. An optimized software framework for real-time, high-throughput tracking of spherical beads. Review of Scientific Instruments. 85 (10), 103712 (2014).
- Dulin, D., et al. High spatiotemporal-resolution magnetic tweezers: calibration and applications for DNA dynamics. Biophysical Journal. 109 (10), 2113-2125 (2015).
- Huhle, A., et al. Camera-based three-dimensional real-time particle tracking at kHz rates and Ångström accuracy. Nature Communications. 6 (1), 5885 (2015).
- Popa, I., et al. A HaloTag anchored ruler for week-long studies of protein dynamics. Journal of the American Chemical Society. 138 (33), 10546-10553 (2016).
- Shon, M. J., Kim, H., Yoon, T. -. Y. Focused clamping of a single neuronal SNARE complex by complexin under high mechanical tension. Nature Communications. 9 (1), 3639 (2018).
- Tapia-Rojo, R., Eckels, E. C., Fernández, J. M. Ephemeral states in protein folding under force captured with a magnetic tweezers design. Proceedings of the National Academy of Sciences. 116 (16), 7873-7878 (2019).
- Löf, A., et al. Multiplexed protein force spectroscopy reveals equilibrium protein folding dynamics and the low-force response of von Willebrand factor. Proceedings of the National Academy of Sciences. 116 (38), 18798-18807 (2019).
- Tapia-Rojo, R., Alonso-Caballero, A., Fernandez, J. M. Direct observation of a coil-to-helix contraction triggered by vinculin binding to talin. Science Advances. 6 (21), (2020).
- Rieu, M., et al. Parallel, linear, and subnanometric 3D tracking of microparticles with Stereo Darkfield Interferometry. Science Advances. 7 (6), (2021).
- Rieu, M., Valle-Orero, J., Ducos, B., Allemand, J. -. F., Croquette, V. Single-molecule kinetic locking allows fluorescence-free quantification of protein/nucleic-acid binding. Communications Biology. 4 (1), 1083 (2021).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences. 103 (16), 6190-6195 (2006).
- Camunas-Soler, J., Ribezzi-Crivellari, M., Ritort, F. Elastic properties of nucleic acids by single-molecule force spectroscopy. Annual Review of Biophysics. 45 (1), 65-84 (2016).
- Südhof, T. C., Rothman, J. E. Membrane fusion: grappling with SNARE and SM proteins. Science. 323 (5913), 474-477 (2009).
- Gao, Y., et al. Single reconstituted neuronal SNARE complexes zipper in three distinct stages. Science. 337 (6100), 1340-1343 (2012).
- Zorman, S., et al. Common intermediates and kinetics, but different energetics, in the assembly of SNARE proteins. eLife. 3, e03348 (2014).
- Zhang, Y., Hughson, F. M. Chaperoning SNARE folding and assembly. Annual Review of Biochemistry. 90 (1), 581-603 (2021).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H. Magnetic tweezers for single-molecule experiments. Handbook of Single-Molecule Biophysics. , 371-395 (2009).
- You, H., Le, S., Chen, H., Qin, L., Yan, J. Single-molecule manipulation of G-quadruplexes by magnetic tweezers. Journal of Visualized Experiments. (127), e56328 (2017).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical Journal. 96 (12), 5040-5049 (2009).
- Dulin, D., Barland, S., Hachair, X., Pedaci, F. Efficient illumination for microsecond tracking microscopy. PLoS One. 9 (9), e107335 (2014).
- Klaue, D., Seidel, R. Torsional stiffness of single superparamagnetic microspheres in an external magnetic field. Physical Review Letters. 102 (2), 028302 (2009).
- Shon, M. J., Rah, S. -. H., Yoon, T. -. Y. Submicrometer elasticity of double-stranded DNA revealed by precision force-extension measurements with magnetic tweezers. Science Advances. 5 (6), 1697 (2019).
- Czerwinski, F., Richardson, A. C., Oddershede, L. B. Quantifying noise in optical tweezers by Allan variance. Optics Express. 17 (15), 13255-13269 (2009).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. Review of Scientific Instruments. 83 (2), 025115 (2012).
- Ostrofet, E., Papini, F. S., Dulin, D. High spatiotemporal resolution data from a custom magnetic tweezers instrument. Data in Brief. 30, 105397 (2020).
- Yu, Z., et al. A force calibration standard for magnetic tweezers. Review of Scientific Instruments. 85 (12), 123114 (2014).
- Strick, T. R., Allemand, J. -. F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271 (5257), 1835-1837 (1996).
- Daldrop, P., Brutzer, H., Huhle, A., Kauert, D. J., Seidel, R. Extending the range for force calibration in magnetic tweezers. Biophysical Journal. 108 (10), 2550-2561 (2015).
- te Velthuis, A. J. W., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative guidelines for force calibration through spectral analysis of magnetic tweezers data. Biophysical Journal. 99 (4), 1292-1302 (2010).
- Ostrofet, E., Papini, F. S., Dulin, D. Correction-free force calibration for magnetic tweezers experiments. Scientific Reports. 8 (1), 15920 (2018).
- Seol, Y., Li, J., Nelson, P. C., Perkins, T. T., Betterton, M. D. Elasticity of short DNA molecules: theory and experiment for contour lengths of 0.6-7 µm. Biophysical Journal. 93 (12), 4360-4373 (2007).
- Burnham, D. R., Vlaminck, I. D., Henighan, T., Dekker, C. Skewed Brownian fluctuations in single-molecule magnetic tweezers. PLoS One. 9 (9), 108271 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protocols. 3 (1), 101152 (2022).
- Lee, H. -. W., et al. Profiling of protein-protein interactions via single-molecule techniques predicts the dependence of cancers on growth-factor receptors. Nature Biomedical Engineering. 2 (4), 239-253 (2018).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Parthasarathy, R. Rapid, accurate particle tracking by calculation of radial symmetry centers. Nature Methods. 9 (7), 724-726 (2012).
- Woodside, M. T., Block, S. M. Reconstructing folding energy landscapes by single-molecule force spectroscopy. Annual Review of Biophysics. 43 (1), 19-39 (2014).
- Evans, E., Ritchie, K. Dynamic strength of molecular adhesion bonds. Biophysical Journal. 72 (4), 1541-1555 (1997).
- Zhang, Y. Energetics, kinetics, and pathway of SNARE folding and assembly revealed by optical tweezers. Protein Science. 26 (7), 1252-1265 (2017).
- Chen, H., et al. Improved high-force magnetic tweezers for stretching and refolding of proteins and short DNA. Biophysical Journal. 100 (2), 517-523 (2011).
- Cho, S., et al. Tension exerted on cells by magnetic nanoparticles regulates differentiation of human mesenchymal stem cells. Biomaterials Advances. 139, 213028 (2022).
- Shon, M. J., Cohen, A. E. Nano-mechanical measurements of protein-DNA interactions with a silicon nitride pulley. Nucleic Acids Research. 44 (1), 7 (2016).
- Cheng, Y. Single-particle cryo-EM-How did it get here and where will it go. Science. 361 (6405), 876-880 (2018).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Neupane, K., et al. Direct observation of transition paths during the folding of proteins and nucleic acids. Science. 352 (6282), 239-242 (2016).
- Choi, H. -. K., et al. Watching helical membrane proteins fold reveals a common N-to-C-terminal folding pathway. Science. 366 (6469), 1150-1156 (2019).
- Kim, C., et al. Extreme parsimony in ATP consumption by 20S complexes in the global disassembly of single SNARE complexes. Nature Communications. 12 (1), 3206 (2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır