Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Pinces magnétiques à grande vitesse pour les mesures nanomécaniques sur des éléments sensibles à la force
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ici, nous décrivons une pince magnétique à haute vitesse qui effectue des mesures nanomécaniques sur des biomolécules sensibles à la force à la vitesse maximale de 1,2 kHz. Nous introduisons son application aux épingles à cheveux à ADN et aux complexes SNARE en tant que systèmes modèles, mais elle sera également applicable à d’autres molécules impliquées dans des événements mécanobiologiques.
Résumé
Les pincettes magnétiques à molécule unique (MT) ont servi d’outils puissants pour interroger avec force les biomolécules, telles que les acides nucléiques et les protéines, et sont donc sur le point d’être utiles dans le domaine de la mécanobiologie. Étant donné que la méthode repose généralement sur le suivi des billes magnétiques basé sur l’image, la limite de vitesse dans l’enregistrement et l’analyse des images, ainsi que les fluctuations thermiques des billes, ont longtemps entravé son application dans l’observation de changements structurels petits et rapides dans les molécules cibles. Cet article décrit en détail les méthodes de construction et d’exploitation d’une installation de traduction automatique à haute résolution capable de résoudre la dynamique nanométrique en millisecondes des biomolécules et de leurs complexes. À titre d’exemples d’application, des expériences avec des épingles à cheveux à ADN et des complexes SNARE (machinerie de fusion membranaire) sont démontrées, en se concentrant sur la façon dont leurs états transitoires et transitions peuvent être détectés en présence de forces à l’échelle du piconewton. Nous nous attendons à ce que les MT à grande vitesse continuent de permettre des mesures nanomécaniques de haute précision sur des molécules qui détectent, transmettent et génèrent des forces dans les cellules, approfondissant ainsi notre compréhension moléculaire de la mécanobiologie.
Introduction
Les cellules détectent activement les stimuli mécaniques et y répondent. Ce faisant, de nombreuses biomolécules présentent des propriétés dépendantes de la force qui permettent des changements structurels dynamiques. Des exemples très appréciés incluent les canaux ioniques mécanosensibles et les éléments cytosquelettiques qui fournissent aux cellules des informations mécaniques clés de leur environnement.
En outre, les molécules qui présentent une nature unique de force porteuse peuvent également être considérées comme mécanosensibles dans un sens plus large. Par exemple, la formation locale et la fusion des duplex d’acides nucléiques, ainsi que des structures d’ordre supérieur telles que les quadriplex G, jouent un rôle crucial dans la réplication, la transcription, la recombinaison et, plus récemment, l’édition du génome. De plus, certaines protéines neuronales impliquées dans les communications synaptiques remplissent leurs fonctions en générant des forces physiques qui dépassent les niveaux des interactions intermoléculaires typiques. Quel que soit l’exemple étudié, l’étude de la nanomécanique des biomolécules impliquées avec une grande précision spatio-temporelle s’avérera très utile pour révéler les mécanismes moléculaires des processus mécanobiologiques associés 1,2,3.
Les méthodes de spectroscopie de force à molécule unique ont servi d’outils puissants pour examiner les propriétés mécaniques des biomolécules 2,4,5,6. Ils peuvent surveiller les changements structurels dans les acides nucléiques et les protéines en même temps que l’application de la force, examinant ainsi les propriétés dépendantes de la force. Deux configurations bien connues sont les pinces optiques et les pinces magnétiques (MT), qui utilisent des billes de la taille d’un micron pour manipuler les molécules 5,6,7,8. Dans ces plateformes, le polystyrène (pour les pinces optiques) ou les billes magnétiques (pour les MT) sont attachés à des molécules cibles (p. ex. acides nucléiques et protéines) via des « poignées » moléculaires, généralement constituées de courts fragments d’ADN double brin (ADNds). Les billes sont ensuite déplacées pour exercer une force et imagées pour suivre leurs emplacements qui signalent les changements structurels dans les molécules cibles. Les pinces optiques et magnétiques sont largement interchangeables dans leurs applications, mais il existe des différences importantes dans leurs approches du contrôle de la force. Les pincettes optiques sont intrinsèquement des instruments de serrage de position qui emprisonnent les billes en position, à cause desquelles la force appliquée fluctue lorsqu’une construction cible subit des changements de forme; L’augmentation de l’extension, par exemple en se dépliant, desserre l’attache et réduit la tension, et vice versa. Bien que la rétroaction active puisse être mise en œuvre pour contrôler la force dans les pincettes optiques, les MT en revanche fonctionnent naturellement comme un dispositif de serrage de force, tirant parti des forces magnétiques stables et éloignées des aimants permanents, qui peuvent également résister à la perturbation de l’environnement.
Malgré leur longue histoire et leur conception simple, les MT ont pris du retard par rapport aux pincettes optiques dans leurs applications pour les mesures de haute précision, en grande partie en raison des défis techniques liés au suivi rapide des talons. Récemment, cependant, plusieurs groupes ont mené conjointement une amélioration multidimensionnelle du matériel et des logiciels pour les instruments de traduction automatique 2,9,10,11,12,13,14,15,16,17,18,19 . Dans ce travail, nous présentons un exemple d’une telle configuration fonctionnant à 1,2 kHz et décrivons comment l’utiliser pour effectuer des mesures nanomécaniques sur des biomolécules sensibles à la force. En tant que systèmes modèles, nous utilisons des épingles à cheveux à ADN et des complexes SNARE neuronaux et examinons leurs changements structurels rapides dans le régime piconewton. Les épingles à cheveux ADN présentent des transitions simples à deux états dans une plage de force bien définie20,21, et servent donc de modèles de jouets pour vérifier les performances d’une pince à épiler. Comme les protéines SNARE s’assemblent en un complexe sensible aux forces qui entraîne la fusion membranaire22, elles ont également été étudiées de manière approfondie par spectroscopie de force à molécule unique 14,23,24,25. Les approches standard pour analyser les données et extraire des informations utiles sur la thermodynamique et la cinétique sont présentées. Nous espérons que cet article facilitera l’adoption de MT de haute précision dans les études mécanobiologiques et motivera les lecteurs à explorer leurs propres systèmes d’intérêt sensibles à la force.
Protocole
Tous les matériaux et équipements décrits dans ce protocole sont énumérés dans le tableau des matériaux. Le logiciel LabVIEW permettant de faire fonctionner la configuration de traduction automatique à grande vitesse décrite ci-dessous, ainsi que les scripts MATLAB permettant d’analyser des exemples de données, sont déposés sur GitHub (https://github.com/ShonLab/Magnetic-Tweezers) et accessibles au public.
1. Construction de l’appareil
REMARQUE : Le principe général de la construction MT à grande vitesse est similaire à celui des systèmes de traduction automatique conventionnels standard, à l’exception de l’utilisation d’une caméra CMOS (Complementary Metal Oxide Semiconductor) à grande vitesse et d’une source lumineuse cohérente et de grande puissance (Figure 1). Reportez-vous à d’autres sources pour plus de descriptions des instruments de traduction automatique standard 5,26,27.
- Installez un microscope inversé sur une table optique antivibratoire. Installez une caméra CMOS haute vitesse et une carte d’acquisition.
- Construisez une étape de traduction pour manipuler des aimants en 3D. Montez une platine linéaire motorisée (>20 mm de débattement) verticalement sur une platine XY manuelle.
REMARQUE: Le mouvement vertical contrôle la force, tandis que l’étage XY est pour l’alignement manuel des aimants sur l’axe optique pour la construction initiale de la configuration. - Installez un moteur pas à pas rotatif et un système de courroie et de poulie pour les aimants rotatifs.
REMARQUE: La courroie transmet le mouvement rotatif entre l’arbre du moteur et les aimants distants de quelques centimètres. La rotation des aimants est interne à la manipulation translationnelle. - Montez les aimants. Utilisez un support en acrylique (commandé auprès d’une entreprise de fabrication; voir la figure supplémentaire S1) qui peut loger étroitement deux aimants identiques en parallèle, avec un espace bien défini de 1 mm entre les aimants (Figure 1B). Pour utiliser la force maximale pouvant être obtenue avec une paire d’aimants donnée, ajustez la position verticale de l’étage de translation de sorte que la surface inférieure des aimants s’aligne sur le plan de l’échantillon lorsqu’il est déplacé vers la position la plus basse.
NOTE: Se référer à Lipfert et al. pour plus d’informations sur la conception et la configuration du support magnétique28. La hauteur et l’orientation des aimants sont contrôlées par le logiciel LabVIEW en conjonction avec l’acquisition de données. - En regardant avec un objectif à faible grossissement, alignez les aimants au centre du champ de vision. Vérifiez que la rotation des aimants ne provoque pas un déplacement important du centre de la paire d’aimants.
REMARQUE: Si le point médian entre les aimants tourne autour de l’axe de rotation, il est probable que les aimants soient décentrés en raison d’un support imparfait. Un faible niveau de désalignement par rapport à la taille de l’espace est tolérable, car la rotation de l’aimant sert uniquement à vérifier les attaches et à appliquer des couples dans des applications spécifiques. - Installez une diode superluminescente (SLD) pour l’éclairage des billes. Passez le faisceau à travers l’espace de 1 mm entre les deux aimants. Assurez-vous que le faisceau est correctement collimaté pour tenir dans l’espace et que l’éclairage n’est pas ombragé par les aimants.
- Installez un scanner de lentille piézoélectrique sur le nez et montez un objectif d’objectif à immersion dans l’huile 100x (ouverture numérique [NA]: 1,45) pour le suivi du cordon. Pour éviter les artefacts potentiels dans le suivi des résultats, assurez-vous que l’éclairage est maintenu uniformément lorsque les aimants sont déplacés. Enfin, réglez le niveau de lumière à la luminosité maximale sans saturer les pixels.
NOTA : Pour la comparaison de différentes sources lumineuses pour le suivi à grande vitesse des billes, se reporter à Dulin et coll.29.
2. Calibrage de la force magnétique
- À l’aide de la réaction en chaîne de la polymérase (PCR; voir le tableau 1), préparer des fragments d’ADNds de 5 kbp (en utilisant l’amorce B, l’amorce Z_5k et l’ADNλ) marqués avec de la biotine à une extrémité (pour la fixation de surface) et de l’azoture à l’autre extrémité (pour la fixation des billes).
- Après la section 6, préparez une cellule d’écoulement avec les molécules de 5 kbp.
- À la suite de la section 7, identifier une bonne construction d’attache de perles en vérifiant son extension et sa rotation. En particulier, assurez-vous de choisir un cordon avec une trajectoire de rotation minimale (c.-à-d. avec un rayon <200 nm) pour minimiser le décalage de hauteur du cordon en raison de la fixation décentrée30,31. Une fois qu’une bonne attache est identifiée, commencez à suivre les perles, en vous référant à la section 9.
- Si la configuration est nouvelle, caractérisez son bruit et sa stabilité pour des mesures haute résolution fiables. Placez l’aimant à ~3 mm de la surface de la cellule d’écoulement (pour appliquer >10 pN et supprimer le mouvement brownien d’une bille), suivre la position z de la bille à 1,2 kHz et calculer la déviation d’Allan (AD) par rapport à la série chronologique de coordonnées z 32,33 (Figure 2C). Vérifier que des valeurs AD de quelques nanomètres sont réalisables dans le régime à grande vitesse (<0,1 s) et que le suivi différentiel (position du cordon magnétique par rapport à une bille de référence) réduit AD sur une échelle de temps plus longue.
REMARQUE: Nous obtenons généralement un AD de <3 nm à la vitesse maximale (résolution de 1,2 kHz ou 0,83 ms), et l’AD continue de diminuer au moins jusqu’à 10 s, ce qui implique une dérive minimale. D’autres ont signalé des valeurs similaires sur des configurations similaires 9,10,11,12,34. - Avec les aimants en position de repos (F ~ 0 pN), enregistrez les coordonnées x et y du cordon captif à 1,2 kHz. Enregistrer la position pendant une période suffisamment longue (c’est-à-dire suffisamment longue que le temps de relaxation caractéristique de la fluctuation35) pour que le mouvement brownien soit suffisamment échantillonné.
REMARQUE: Ici, la direction x est le long de la direction du champ magnétique, alors que le mouvement dans y représente le mouvement transversal perpendiculaire au champ. - Rapprochez les aimants de la cellule d’écoulement et répétez les mesures de position des billes jusqu’à ce que les aimants touchent doucement le haut de la cellule d’écoulement. Déplacez-vous par grands pas (p. ex., 1 à 2 mm) lorsque les aimants sont à plus de 7 mm du plan d’échantillonnage (puisque la force appliquée augmente lentement dans le champ éloigné des aimants), mais réduisez progressivement la taille des pas (p. ex., 0,1-0,5 mm) à mesure qu’ils se rapprochent pour un étalonnage plus fin à des niveaux de force plus élevés (figure 2B).
- Calculez la force à chaque position magnétique, d, en utilisant l’une des deux méthodes alternatives (un script MATLAB « force calibration.m » incluant les deux méthodes est fourni; voir Fichier supplémentaire 1).
- Mesurer la variance des coordonnées
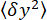 y du cordon (figure 2D) et la position z moyenne du cordon par rapport à la position
y du cordon (figure 2D) et la position z moyenne du cordon par rapport à la position 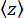 la plus basse (figure 2B, en bas). Ensuite, utilisez l’équation (1)7,27,36 pour estimer la force (avec un rayon de bille fixe R = 1 400 nm et une énergie thermique kRT = 4,11 pN∙nm):
la plus basse (figure 2B, en bas). Ensuite, utilisez l’équation (1)7,27,36 pour estimer la force (avec un rayon de bille fixe R = 1 400 nm et une énergie thermique kRT = 4,11 pN∙nm):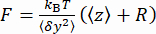 (1)
(1) - Vous pouvez également calculer la densité spectrale de puissance (PSD) des coordonnées y, Sy (Figure 2E). Déterminer la force appliquée F en ajustant un modèle de Lorentzien double37 au Sy mesuré à l’aide de l’équation (2).
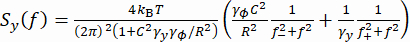 (2)
(2)
Ici, , R est le rayon de bille, γ yet γφ sont les coefficients de traînée translationnel et rotationnel, respectivement (estimés à partir de l’équation de Stokes-Einstein), kRT est l’énergie thermique,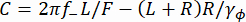 f+ et f- sont deux fréquences caractéristiques obtenues à l’aide de l’équation (3).
f+ et f- sont deux fréquences caractéristiques obtenues à l’aide de l’équation (3).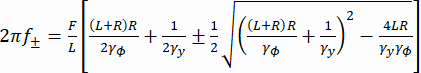 (3)
(3)
REMARQUE: Étant donné que l’extension d’attache L est une fonction de force qui suit le modèle bien établi de chaîne ressemblant à un ver (WLC), les expressions ci-dessus laissent F comme seul paramètre d’ajustement (nous fixons R à 1 400 nm pour plus de simplicité car il est partagé entre tous les niveaux de force et la valeur exacte n’influence pas sensiblement les résultats). Si nécessaire, le flou de mouvement et l’aliasing provenant de l’acquisition d’images par caméra doivent être pris en compte38,39, mais cet effet est négligeable dans nos mesures à grande vitesse au-dessus de 1 kHz avec des attaches de 5 kbp.
- Mesurer la variance des coordonnées
- Répétez les étapes 2.4 à 2.7 pour quelques constructions supplémentaires. Sondez trois à cinq billes différentes pour faire la moyenne de la variabilité de la force entre les billes magnétiques.
REMARQUE : La variation de force entre les billes magnétiques utilisées doit être prise en compte pour déterminer le nombre approprié de constructions pour la moyenne. Cette variabilité est faible mais peut entraîner une erreur de plus de 1 pN dans la force mesurée, même pour les produits commerciaux31. Pour la plupart des applications, où la détermination absolue des forces en jeu n’est pas cruciale, la moyenne des résultats d’étalonnage de trois à cinq billes est généralement suffisante. Une autre approche pour tenir compte de cette variation consiste à mesurer la force avec des attaches individuelles au début de l’expérience, ce qui peut prendre beaucoup de temps. Une autre option consiste à intégrer des structures en épingle à cheveux qui se décompressent à des niveaux de force connus dans chaque construction31. - Tracer la force mesurée en fonction de la distance de l’aimant et ajuster une fonction exponentielle double aux données (Figure 2F) à l’aide de l’équation (4).
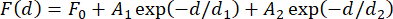 (4)
(4)
Ici, F0 (ligne de base), A 1 et A 2 (amplitudes) et d 1 et d 2 (constantes de désintégration) sont des paramètres d’ajustement. Assurez-vous que les valeurs de force des deux méthodes, ainsi que les ajustements à double exponentielle qui en résultent, concordent largement (Figure 2F,G).
REMARQUE : Pour confirmer que l’étalonnage de la force est effectué correctement, vérifiez la relation force-extension des constructions sondées en traçant l’extension par rapport à la force mesurée. - Pour corriger le décalage de hauteur du cordon z off résultant de l’inclinaison dépendante de la force des billes magnétiques30,31, estimer zoff à partir du décalage latéral xoff, en tenant compte de la géométrie d’une attache décentrée avec un rayon de cordon en utilisant l’équation (5), et appliquer les valeurs aux valeurs d’extension mesurées. Cette étape est implémentée dans le script MATLAB « force calibration.m » (lignes 252-254).
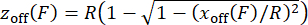 (5)
(5)
NOTE: Bien que cette correction apporte de petits changements à l’extension, en particulier pour les billes avec un petit rayon de rotation (<200 nm), ce décalage affecte souvent de manière critique la réponse élastique, comme on le voit dans le passage de la figure 2H à la figure 2I 30,31. - Vérifiez la longueur de persistance Lp en ajustant un modèle WLC extensible aux données à l’aide de l’équation (6).
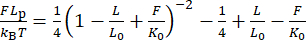 (6)
(6)
Ici, L 0 est la longueur du contour (1,7 μm pour 5 kbp) et K0 est le module pour l’étirement enthalpique.
REMARQUE: Bien que la L p de l’ADNds soit bien acceptée pour être de 40-50 nm dans un tampon typique tel que la solution saline tamponnée au phosphate (PBS), la formule WLC appliquée aux molécules courtes (<5 kbp) sous-estime systématiquement L p car L0 diminue de31,40. En effet, le modèle WLC classique suppose un polymère dont la longueur de chaîne est suffisamment plus longue que sa longueur de persistance. Ici, nous avons obtenu L p = 40 ± 3 nm pour la construction de 5 kbp (Figure 2H), et la correction d’extension a en outre donné un K0 homogène de 1 100 ± 200 pN (Figure 2I). L’application d’un modèle WLC fini 31,40, ainsi qu’une correction pour non-Gaussianité dans la distribution d’extension 41, augmentera légèrement L p. - Une fois l’étalonnage de la force vérifié, appliquez les paramètres d’ajustement obtenus du modèle à double exponentielle au logiciel LabVIEW fourni (fichier supplémentaire 2) et attendez que le logiciel calcule la force de courant en temps réel à partir des lectures du moteur (c’est-à-dire la position de l’aimant). Comme une expression analytique pour la fonction inverse d(F) n’est pas disponible, préparer une table de correspondance de d par rapport à F par pas de 0,1 pN par estimation numérique des niveaux de force cibles d. Stockez également cette table dans le logiciel pour commander le contrôle de force.
3. Synthèse d’épingles à cheveux ADN
NOTE: Les constructions en épingle à cheveux de l’ADN pour les expériences de TA sont préparées par amplification par PCR d’une région de 510 pb dans l’ADNλ avec deux amorces personnalisées, dont l’une contient une structure en épingle à cheveux à son extrémité 5' (Figure 3A). De cette façon, un motif en épingle à cheveux est placé à une extrémité du produit PCR.
- Préparez les amorces.
- Amorce avant : L’apprêt B_hp marqué à la 5′-biotine pour la fixation de la surface du verre et se lie à l’ADNλ. Cet apprêt contient un motif en épingle à cheveux avec une tige de 8 pb et une boucle de 6 nt, 5' à la région de liaison λ.
- Amorce inversée : L’amorce Z_hp marquée au 5′-azoture pour la fixation des billes magnétiques et se lie à l’ADNλ à 1 kbp de l’amorce avant.
- Configurez et exécutez la PCR avec λ-ADN (modèle), nTaq polymérase et des conditions PCR standard (voir Tableau 1). Nettoyez le produit avec un kit de purification commercial.
- Mesurer la concentration d’ADN par absorption UV à 260 nm (A260) et effectuer une électrophorèse sur gel d’agarose (gel à 2 %) (voir Tableau 2) pour vérifier la taille du produit. Un rendement typique est de ~35 μL de solution de ~600 nM.
4. Préparation des protéines SNARE
REMARQUE : Les complexes SNARE neuronaux sont assemblés en combinant trois protéines de rat purifiées exprimées à partir d’E. coli : VAMP2/synaptobrévine-2, syntaxine-1A et SNAP-25 (Figure 3B). Pour faciliter leur assemblage, la syntaxine et SNAP-25 sont co-exprimées avec un fragment VAMP2 (dépourvu de la région N-terminale; appelé « ΔN-VAMP2 ») dans une structure appelée « complexe ΔN », puis mélangées avec VAMP2 pleine longueur après fixation de la poignée d’ADN pour former des complexes complets.
- Préparer les plasmides contenant de l’ADNc pour l’expression des protéines SNARE (les séquences d’ADN pour tous les plasmides sont données dans le tableau des matériaux).
- Préparer le VAMP2 marqué 6×His dépourvu du domaine transmembranaire (2-97; L32C/I97C pour les liaisons disulfure) cloné dans un vecteur pET28a.
- Préparer la syntaxine-1A dépourvue de Habc et du domaine transmembranaire (191-267, I202C/I266C substitutions pour les liaisons disulfure) clonée avec 6×His marqué ΔN-VAMP2 (49-96) dans un vecteur pETDuet-1.
- Préparer l’isoforme b pleine longueur de SNAP-25 (2-206, toutes C à A) clonée en vecteur pET28a. Celui-ci sera utilisé pour la préparation des complexes ΔN.
- Préparer l’isoforme b pleine longueur SNAP-25 marquée 6×His (1-206, toutes C à A) clonée en un vecteur pET28a pour l’ajout direct au tampon de dosage MT afin de réassembler les complexes SNARE après le dépliage.
- Préparez deux tubes de cellules d’E. coli de Rosetta (DE3). Transformez un groupe avec des plasmides VAMP2 (à partir de l’étape 4.1.1), un avec des plasmides syntaxine-1A/ΔN-VAMP2 et SNAP-25 non marqués (à partir des étapes 4.1.2 et 4.1.3) pour exprimer le complexe ΔN, et l’autre avec des plasmides SNAP-25 marqués par His (à partir de l’étape 4.1.4).
- Transférer les cellules transformées dans un bouillon Luria-Bertani (LB) avec les antibiotiques appropriés (ici, la kanamycine et le chloramphénicol pour VAMP2 et le SNAP-25 marqué His; kanamycine, chloramphénicol et ampicilline pour le complexe ΔN). Cultivez-les à 37 °C dans un incubateur à agitation (220 tr/min) jusqu’à ce que la densité optique (OD) du bouillon atteigne 0,7-0,9.
- Ajouter 1 mM d’isopropyle β-d-1-thiogalactopyranoside (IPTG) pour induire l’expression des protéines et incuber les cellules pendant 3-4 h à 37 °C dans un incubateur à agitation (220 rpm).
- Enduire les cellules en centrifugeant la culture à 4 500 × g pendant 15 min à 4 °C.
- Préparer des tampons pour la purification des protéines (voir tableau 2).
- Suspendre les pastilles cellulaires exprimant SNARE dans 40 mL de tampon de lyse glacé et lyser les cellules par sonication sur glace (amplitude de 15%, 5 s allumé et 5 s éteint, 30 min au total).
- Centrifuger le lysat à 15 000 × g pendant 30 min à 4 °C pour éliminer les matières insolubles.
- Passez le surnageant à travers une colonne de gravité remplie de 1 mL de résine Ni-NTA. Laver la résine avec le tampon de lavage A, puis avec le tampon de lavage B, et éluer les protéines avec 10 mL de tampon d’élution.
- Retirer la tris(2-carboxyéthyl)phosphine (PTCE) et l’imidazole de l’éluant à l’aide d’une colonne de dessalage (suivre les instructions du fabricant). Éluer l’échantillon avec PBS.
- Concentrer les protéines avec des filtres centrifuges (coupure de 10 kDa) à ~70 μM tout en maintenant les protéines dans du PBS (produisant généralement 2 mL). Mesurer la concentration de protéines soit par absorption ultraviolette (UV) à 280 nm (A280), soit par le test de Bradford.
- Préparer de petites aliquotes, les congeler dans de l’azote liquide et conserver à -80 °C jusqu’à utilisation.
REMARQUE: Les complexes SNARE complets seront assemblés après la conjugaison du complexe ΔN sur une poignée d’ADN (voir ci-dessous).
5. Fixation des poignées d’ADN
REMARQUE: Deux poignées d’ADNds de 510 bp contenant des groupes amines primaires à une extrémité sont d’abord préparées par PCR, puis les groupes amines sont ensuite convertis en groupes maléimide à l’aide d’un agent de réticulation bifonctionnel, SM (PEG) 2. Les deux poignées sont ensuite liées par covalence aux complexes SNARE via leurs groupes cystéine pour une conjugaison spécifique au site (Figure 3B).
- Préparez les amorces.
- Préparer les amorces avant : l’amorce B (pour amplifier la poignée B) qui est marquée à la 5′-biotine pour la fixation de la surface du verre et qui se lie à l’ADNλ; Primer Z (pour amplifier la poignée Z) qui est marqué au 5′-azoture pour la fixation du cordon magnétique et a la même séquence que l’apprêt B.
- Préparez une amorce inversée : l’amorce N (partagée pour la poignée B et la poignée Z) qui est marquée à la 5′-amine pour la conjugaison des protéines et se lie à l’ADNλ à 510 pb de l’amorce avant.
- Configurez et exécutez deux séries de réactions PCR (18 tubes de réaction de 200 μL pour chaque poignée) avec de l’ADN λ (modèle), de la nTaq polymérase et des conditions PCR standard (voir le tableau 1). Nettoyez le produit avec un kit de nettoyage PCR et éluez chaque poignée avec 45 μL d’eau ultrapure. Utilisez un volume minimal d’eau pour obtenir des concentrations élevées de poignées pour une réaction efficace dans les étapes ultérieures.
- Mesurer la concentration d’ADN par A260. Le rendement typique est de ~650 μL de solution de ~2 μM pour chaque poignée. Conserver les petits échantillons à part pour une vérification ultérieure dans l’électrophorèse sur gel d’agarose.
- Réagissez chaque poignée (1 μM dans PBS) avec 5 mM SM(PEG)2. Incuber à température ambiante avec une rotation douce. Après 1 h, utilisez un kit de purification de l’ADN pour éliminer le SM(PEG)2 qui n’a pas réagi. Éluer chaque manche avec 250 μL de PBS pour obtenir des solutions de ~2 μM.
- Mélanger les solutions de poignée B et de complexe ΔN à un rapport molaire de 1:16 (par exemple, 1 μM de poignée B et 16 μM de complexe ΔN) dans du PBS et incuber pendant 2 h à température ambiante en agitant. Conserver un petit échantillon pour l’électrophorèse sur gel d’agarose.
- Ajouter une solution de VAMP2 dans un excès molaire de 2,5 fois sur le complexe ΔN utilisé à l’étape précédente. Incuber le mélange pendant encore 1 h à température ambiante en agitant. Des complexes SNARE complets sont assemblés dans cette étape.
- Éliminer les protéines libres par échange tampon avec du PBS frais et un filtre centrifuge (coupure de 100 kDa) : centrifuger à 14 000 × g pendant 5 min à 4 °C, répéter au moins 6x et faire fonctionner pendant 15 min pour le dernier essorage. Mesurer l’augmentation du rapport A260/A280 pour surveiller l’élimination des protéines libres. Conserver un petit échantillon pour l’électrophorèse sur gel d’agarose.
- Ajouter la poignée Z à la solution dans un excès molaire de 15 fois sur la poignée B. Maintenir la concentration de la poignée Z au moins au-dessus de 1 μM pour faciliter la réaction. Incuber le mélange pendant une nuit à 4 °C en agitant.
- Vérifier les intermédiaires (poignée B et ses conjugués protéiques) et le produit final (complexe SNARE à deux poignées) par électrophorèse sur gel d’agarose (figure 3B, encadré) (voir tableau 2).
REMARQUE: Si les protéines sont fixées avec succès à la poignée B, un changement de mobilité sera détecté. En particulier, la formation de complexes SNARE complets sur les poignées d’ADN peut être confirmée par leur résistance au dodécylsulfate de sodium (SDS), contrairement aux complexes ΔN, qui sont désassemblés en SDS et ne laissent que la syntaxine liée à l’ADN (comparer b et c dans la figure 3B). - Préparer de petites aliquotes, les congeler dans de l’azote liquide et conserver à -80 °C jusqu’à utilisation.
REMARQUE: Bien que la solution finale contienne des poignées n’ayant pas réagi, seule la construction recherchée qui est doublement marquée avec de la biotine et de l’azoture sera sélectionnée lors de l’assemblage de l’échantillon dans une cellule d’écoulement.
6. Fabrication de cellules d’écoulement
REMARQUE : Les cellules d’écoulement pour les mesures de TA sont construites à partir de deux lamelles de verre collées ensemble par du ruban adhésif double face (Figure 3C). Une lamelle de couverture est recouverte d’un mélange de PEG et de polyéthylène glycol biotinylé (PEG) pour éviter la liaison non spécifique et permettre l’attache spécifique des molécules cibles via la liaison biotine-NeutrAvidine (Figure 3D). Ensuite, les solutions de matériaux pour les expériences de MT sont infusées séquentiellement dans une cellule d’écoulement à l’aide d’une pompe à seringue (Figure 3C,D).
- Préparez deux lamelles de couvercle en verre, une pour la surface supérieure (24 mm × 50 mm, épaisseur no 1,5) et une pour la surface inférieure (24 mm × 60 mm, épaisseur no 1,5). Nettoyer les lamelles de couverture par sonication dans 1 M KOH pendant 30 min. Après la sonication, rincer les lamelles de couverture avec de l’eau distillée et conserver dans l’eau jusqu’à l’étape suivante.
- PEGylate le capot inférieur suivant les protocoles publiés42,43. Utiliser de la N-[3-(triméthoxysilyl)propyl]éthylènediamine pour la silanisation et un mélange 1:100 (pw) de biotine-PEG-SVA et de mPEG-SVA dans un tampon de bicarbonate de 100 mM. Gardez les lamelles de couverture PEGylated sèches à -20 °C et conservez-les pendant quelques semaines.
- Le jour des expériences, sortez les lamelles de couverture PEGylated et séchez-les avec un pistolet à azote. Inspectez-les visuellement pour détecter la saleté pour vous assurer qu’ils sont propres.
- Pour fabriquer les canaux d’échantillonnage, préparer des bandes de ruban adhésif double face de ~2 mm de large et déposer quatre bandes sur une lame de couverture inférieure (surface PEGylée vers le haut), parallèles et séparées les unes des autres par ~5 mm (Figure 3C).
REMARQUE: De cette façon, trois canaux d’échantillonnage de 5 mm de large peuvent être créés dans une seule cellule d’écoulement. - Placez un couvercle supérieur au centre du couvercle inférieur, en laissant ~5 mm d’espace sur les bords courts pour les entrées et les sorties de canal. Appuyez doucement sur le dos de la housse supérieure avec une pince à épiler pour bien sceller les canaux.
- Pour créer un réservoir d’entrée, coupez le bord d’une pointe de pipette de 200 μL. Découpez ~10 mm de l’ouverture plus large pour permettre de retenir ~200 μL de solution. Faites-en trois pour les trois canaux d’écoulement. Pour configurer les sorties, préparez trois aiguilles de seringue qui s’adaptent au tube de la pompe à seringue.
- À l’aide d’époxy de 5 min, collez les réservoirs et les moyeux à aiguilles à la cellule d’écoulement. Assurez-vous qu’un joint complet est formé pour éviter les fuites et que les canaux ne sont pas bloqués par un excès de colle. Laissez sécher pendant au moins 30 min.
7. Assemblage de constructions d’attache de perles
NOTE: Les solutions de matériaux pour les expériences de TA, y compris celles pour les constructions d’attache de perles, sont introduites séquentiellement dans les cellules d’écoulement à l’aide d’une pompe à seringue (Figure 3C, D).
- Préparez des billes magnétiques. Prélever 5 mg de billes époxy M270 dans une solution mère (~3,3 × 108 billes dans 167,5 μL de diméthylformamide) et remplacer le solvant par un tampon phosphate (voir tableau 2) par séparation magnétique des billes.
- Préparer les billes à ~1,1 × 109 billes mL−1 dans un tampon phosphate avec 1 M sulfate d’ammonium et les faire réagir avec 2 mM de dibenzocyclooctyne (DBCO)-NH2. Incuber le mélange pendant 3 h sur un batteur rotatif à température ambiante. Après la réaction, laver les billes 3x avec un tampon phosphate frais pour éliminer les molécules qui n’ont pas réagi.
REMARQUE: Les billes lavées peuvent être conservées sans rotation supplémentaire à 4 °C pendant plusieurs semaines avant utilisation. - Connectez une aiguille sur la sortie du canal de la cellule d’écoulement à la pompe à seringue avec un tube en polyéthylène. Équilibrez les canaux avec PBS.
- Introduisez les solutions suivantes séquentiellement dans le canal en aspirant avec la pompe : NeutrAvidin, constructions cibles (épingles à cheveux ADN ou complexes SNARE avec poignées d’ADN), billes de polystyrène de référence et billes magnétiques revêtues de DBCO. Avant utilisation, vortex les solutions de billes à fond pour disperser les agrégats de billes potentiels.
- Laver les billes non liées tout en appliquant une force de 0,1 pN.
NOTE: L’application d’une petite force ascendante facilite l’élimination des perles non liées et aide à éviter la rupture des constructions d’attache de perles spécifiquement liées. - Pour les expériences avec des complexes SNARE, inclure 1,5 μM SNAP-25 dans le tampon final.
REMARQUE: Les molécules libres de SNAP-25 peuvent se relier aux complexes SNARE après le déploiement et permettre des mesures répétées sur un seul complexe.
8. Identification des constructions cibles
- À la surface d’un canal de cellule d’écoulement, recherchez les billes magnétiques qui sont attachées par des molécules uniques de la construction cible. Assurez-vous qu’une perle de référence est située à proximité.
- Faites pivoter une perle candidate et vérifiez qu’elle pivote librement. Si le cordon est attaché par plusieurs molécules, il présente un mouvement contraint.
- Faites pivoter le cordon pendant quelques tours complets et découvrez le rayon de rotation (cette fonction est implémentée dans le logiciel fourni). De préférence, choisissez un cordon avec un petit rayon de rotation.
REMARQUE: Ce rayon indique dans quelle mesure le cordon est décentré de l’axe de l’attache, ce qui est déterminé aléatoirement lors de l’assemblageperle-attache 30,31. Dans toutes les expériences, le décentrage minimal d’une perle atténue de nombreux artefacts associés au rapport rayon de perle / extension de l’attache élevé que nous utilisons. - Augmentez la force de 0 à 5 pN pour identifier les bonnes billes attachées à une seule épingle. Recherchez un changement important dans le diagramme de diffraction d’une perle résultant de l’étirement d’une attache de 1 kbp (ou des deux poignées équivalentes de 510 pb). Si le diagramme de diffraction ne change pas de manière significative, abaissez la force à zéro et recherchez un autre cordon candidat.
REMARQUE: Le levage ~300 nm d’une perle peut être facilement remarqué à partir des images brutes sans réellement démarrer le processus de suivi.
9. Suivi des billes pour les mesures d’extension
REMARQUE : Le suivi des perles est effectué en analysant les images des perles en temps réel dans le logiciel LabVIEW fourni avec cet article. La méthode de suivi et ses variantes ont été utilisées dans la plupart des systèmes de TA conventionnels et sont expliquées dans la littérature précédente 2,5,7,26. En mesurant la position d’un cordon magnétique par rapport à un cordon de référence fixe (c.-à-d. suivi différentiel), les mesures de position deviennent extrêmement robustes à une perturbation externe.
- Une fois qu’une perle magnétique appropriée est localisée avec une perle de référence, cliquez sur le bouton Calibrer pour commencer à préparer le suivi des perles.
- Cliquez sur les perles dans l’image pour définir les emplacements des perles. Les images seront ensuite recadrées en régions d’intérêt (ROI) (par exemple, 150 x 150 pixels pour une perle de 3 μm) autour des perles, puis analysées pour extraire les coordonnées précises des perles.
- Attendez la fin de la rotation de l’aimant. Ce processus enregistre les coordonnées x et y du cordon (en calculant la corrélation croisée 2D44 ou en utilisant la symétrie radiale45 des images de la perle, avec des performances comparables) tout en faisant tourner les aimants pour documenter la fixation décentrée de la perle31.
- Pour le suivi dans la direction z, attendez que le logiciel génère une table de correspondance d’images de diffraction des billes à différentes distances du plan focal. Ceci est réalisé en déplaçant l’objectif avec un scanner piézoélectrique par pas équidistants et en enregistrant des images de perles moyennes de fluctuation à chaque position. Ensuite, les coordonnées z des perles dans les expériences réelles sont déterminées en comparant les images de perles en temps réel à la table de correspondance avec interpolation7.
- Lorsque la génération de la table de correspondance est terminée, activez le suivi et la mise au point automatique (appuyez sur la touche Track ? et AF? Boutons) et cliquez sur le bouton Acquérir pour commencer à enregistrer les positions des perles.
REMARQUE: La mise au point automatique est facultative mais recommandée pour corriger la dérive de l’étage en z lors de l’acquisition.
10. Régimes d’application forcée
- Expériences de rampe de force: Pour vérifier la relation force-extension de la construction, appliquer une montée et une descente de la force à une vitesse de charge constante (± 1,0 pN s−1) (figure 4A). Par exemple, appliquez trois cycles d’un cycle 0-20-0 pN pour vérifier la longueur totale de la construction et la courbe d’extension de force des poignées.
- En spécifiant les paramètres d’attache dans le logiciel, superposez une courbe d’extension de force WLC sur les données mesurées et déterminez si la perle cible est attachée par une véritable construction d’échantillon avec des poignées d’ADN appropriées. Utilisez la longueur de contour connue (par exemple, ~340 nm pour 1 kbp dsDNA) et la longueur de persistance WLC (30-45 nm pour l’ADNds31 abrégé) de la construction comme point de départ. Appliquez la méthode de correction d’extension décrite à l’étape 2.11 si nécessaire.
- Si la construction est vérifiée, examinez en détail la réponse d’extension de force pour rechercher une extension supplémentaire résultant des molécules-épingles à cheveux cibles ou des complexes SNARE.
- Expériences à force constante : Faire varier progressivement la force appliquée par étapes discrètes pour sonder la sensibilité à la force des molécules cibles (Figure 4B).
REMARQUE: Les MT permettent des expériences simples et efficaces à force constante, car la force appliquée est maintenue constante lorsque les aimants sont maintenus immobiles.- Pour les épingles à cheveux à ADN, appliquez une force de 4 à 8 pN avec des pas de 0,2 à 0,5 pN et mesurez la position du cordon pendant ~10 s à chaque niveau de force.
- Pour les complexes SNARE, appliquez une force de 14 à 16 pN avec des pas de 0,1 à 0,2 pN et mesurez la position du cordon pendant ~10 s à chaque niveau de force.
- Expériences de saut de force: Observez les événements de transition des complexes SNARE.
REMARQUE: Les expériences de saut de force, comme les expériences de force constante, impliquent des changements dans les niveaux de force. Cependant, les sauts de force utilisent des changements plus brusques dans la force appliquée, ce qui permet de surveiller les événements déclenchés par la force dans les molécules sondées, tels qu’une rupture soudaine de complexes protéiques. Par exemple, étant donné que les complexes SNARE présentent une hystérésis structurelle dans le cycle de force23, il est instructif d’effectuer des expériences de saut de force et de mesurer la latence de transition (Figure 4C).- Décompression: Décollement d’une molécule VAMP2 d’un complexe SNARE ternaire intact, laissant un complexe binaire de syntaxine-1A et SNAP-25.
- Rezippation: Fermeture éclair de la molécule VAMP2 décompressée pour régénérer un complexe SNARE intact.
- Déroulement: Démontage complet d’un complexe SNARE accompagné d’une dissociation complète de SNAP-25. Seules les molécules VAMP2 et syntaxine restent dans la construction après le déploiement.
- Repliage : Régénération d’un complexe SNARE lors de la liaison d’une molécule SNAP-25 libre du tampon.
- A 2 pN, induire l’assemblage d’un complexe SNARE intact en attendant (~30 s) l’association d’une molécule libre de SNAP25. Une diminution soudaine de l’extension est observée lors de la formation d’un complexe SNARE.
- Pour observer les événements de décompression, attendez quelques secondes à 10-12 pN, puis passez brusquement à 14-15 pN avec la vitesse maximale possible. Selon la force cible, le complexe SNARE présentera soit une transition réversible entre des intermédiaires partiellement décompressés (comme dans les expériences à force constante), soit un saut de ~25 nm vers un état plus élevé, non compressé, après un temps d’attente aléatoire (ou latence).
- Pour observer les événements de rezipping, abaissez la force à 10-12 pN immédiatement après l’observation du dézippage. Encore une fois, le complexe SNARE présente une transition stochastique vers l’état inférieur, zippé après une certaine latence aléatoire. Si le dépliage s’est produit après le dézippage, le complexe ne pourra pas se rezipper, car une molécule SNAP-25 sera manquante.
- Pour observer les événements qui se déroulent, attendez une période plus longue après l’observation de la décompression pour détecter une nouvelle augmentation de l’extension (~ 2 nm).
11. Analyse des données
REMARQUE: Les types d’analyse que l’on peut effectuer avec des données de TA dépendent du système cible. Cependant, il existe des approches communes pour extraire des informations utiles des expériences respectives décrites à la figure 4. Toutes les analyses sont effectuées avec MATLAB (R2021a) à l’aide des codes personnalisés fournis avec cet article. Ces codes génèrent des graphiques en utilisant les mêmes données présentées dans cet article. Il est à noter que même si les données brutes du suivi de 100 Hz ont été directement prises pour analyse, les données du suivi de 1,2 kHz ont généralement été filtrées par médiane (avec une fenêtre coulissante à cinq points) avant l’analyse pour réduire le bruit (sauf pour l’analyse du bruit).
- Expériences de rampe de force: Analyser la relation force-extension (p. ex., élasticité des polymères) et transférer la force pour extraire de l’information sur les propriétés nanomécaniques.
- Expériences à force constante : Analyser les populations d’état et le temps de séjour (ou taux de transition) en fonction de la force pour extraire les paramètres structurels (p. ex., régions impliquées dans la transition), thermodynamiques (p. ex., différence d’énergie libre) et cinétiques (p. ex., barrière énergétique) des changements conformationnels.
- Expériences de saut de force: Analyser la cinétique de rupture (p. ex., interactions protéine-protéine et liaison récepteur-ligand) ou la durée de vie des intermédiaires transitoires (p. ex., déploiement de biomolécules) pour extraire la stabilité des molécules cibles et leurs états.
- En tant qu’applications représentatives, analysez les données d’échantillon pour les épingles à cheveux ADN et les complexes SNARE :
- Transitions à deux états d’une épingle à cheveux d’ADN: force de décompression, distance d’ouverture, dépendance à la force du déplacement de population et mesures d’assignation d’état et de taux de transition avec un modèle de Markov caché (HMM) (codes MATLAB fournis).
- Changements conformationnels des complexes SNARE: force de décompression, dépendance de la force des états intermédiaires et latence de décompression, hystérésis dans la refermeture et comportement de déploiement / repliement.
NOTE: Les modèles d’extension de force pour les poignées d’ADN, les épingles à cheveux ADN et les conformations complexes SNARE sont donnés dans les références précédentes14,31.
Résultats
Calibrage de la force
Les résultats des deux méthodes de mesure de la force (variance de déplacement latéral des billes et analyse du spectre de puissance) différaient de 0 à 2 pN (figure 2G). Selon les résultats de la figure 2F, nous pouvons atteindre de manière fiable jusqu’à 30 pN avec des aimants en néodyme réguliers.
Transitions à deux états d’une épingle à cheveux ADN de 8 pb
Discussion
Dans ce travail, nous avons introduit une configuration de spectroscopie de force à molécule unique qui peut observer les changements structurels des biomolécules avec une grande précision spatio-temporelle. La caméra CMOS haute vitesse utilisée acquiert 1 200 images s−1 à une résolution de 1 280 x 1 024, permettant un suivi des billes de 1,2 kHz. Cependant, la vitesse des mesures est actuellement limitée par le logiciel de suivi des perles, de sorte que le retour sur investissement est généraleme...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Ce travail a été soutenu par la subvention de la Fondation nationale de recherche de Corée (NRF) financée par le gouvernement coréen (MSIT) (NRF-2022R1C1C1012176, NRF-2021R1A4A1031754 et NRF- 2021R1A6A1A10042944). S.-H.R. a été soutenu par la subvention NRF (2021R1C1C2009717).
matériels
| Name | Company | Catalog Number | Comments |
| Materials for construct synthesis | |||
| Agarose gel electrophoresis system | Advance | Mupid-2plus | |
| DNA ladder | Bioneer | D-1037 | |
| nTaq polymerase | Enzynomics | P050A | |
| PCR purification kit | LaboPass | CMR0112 | |
| PEGylated SMCC crosslinker / SM(PEG)2 | ThermoFisher Scientific | 22102 | For SNARE–DNA coupling |
| Primer B | Bioneer | 5'-Biotin/TCGCCACCATCATTTCCA-3' | For 5-kbp force calibration construct and DNA handles |
| Primer B_hp | IDT | 5'-Biotin/TTTTTTTTTTGTTCTCTATTT TTTTAGAGAAC /AP site/ /AP site/ TCGCCACCATCATTTCCA-3' | For hairpin construct |
| Primer N | Bioneer | 5'-C6Amine/CATGTGGGTGACGCGAAA-3' | For DNA handles |
| Primer Z | Bioneer | 5'-Azide/TCGCCACCATCATTTCCA-3' | For DNA handles |
| Primer Z_5k | Bioneer | 5'-Azide/TTAGAGAGTATGGGTATATGACA TCG-3' | For 5-kbp force calibration construct |
| Primer Z_hp | Bioneer | 5'-Azide/GTGGCAGCATGACACC-3' | For hairpin construct |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| λ-DNA | Bioneer | D-2510 | Template strand for PCR |
| DNA sequences for SNARE proteins | |||
| 6×His-tagged SNAP-25b (2-206; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctat gagaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagcgg aagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCACA GCAGCGGCCTGGTGCCGCGC GGCAGCCATACTAGCGGAGAT ATCGCCGAGGACGCAGACAT GCGCAATGAGCTGGAGGAGA TGCAGAGGAGGGCTGACCAG CTGGCTGATGAGTCCCTGGA AAGCACCCGTCGCATGCTGC AGCTGGTTGAAGAGAGTAAA GATGCTGGCATCAGGACTTT GGTTATGTTGGATGAGCAAG GCGAACAACTGGAACGCATT GAGGAAGGGATGGACCAAAT CAATAAGGACATGAAAGAAG CAGAAAAGAATTTGACGGAC CTAGGAAAATTCGCCGGCCT TGCCGTGGCCCCCGCCAAC AAGCTTAAATCCAGTGATGC TTACAAAAAAGCCTGGGGC AATAATCAGGATGGAGTAGT GGCCAGCCAGCCTGCCCG TGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTG GCTTCATCCGCAGGGTAAC AAATGATGCCCGGGAAAAT GAGATGGATGAGAACCTG GAGCAGGTGAGCGGCATC ATCGGAAACCTCCGCCAC ATGGCTCTAGACATGGGCA ATGAGATTGACACCCAGA ATCGCCAGATCGACAGGA TCATGGAGAAGGCTGATT CCAACAAAACCAGAATTG ATGAAGCCAACCAACGTG CAACAAAGATGCTGGGAA GTGGTTAAggatccgaattcgag ctccgtcgacaagcttgcggccgcactc gagcaccaccaccaccaccactgagat ccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgag caataactagcataaccccttggggcct ctaaacgggtcttgaggggttttttgctga aaggaggaactatatccggat | |
| 6×His-tagged VAMP2 (2-97, L32C/I97C; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCAC AGCAGCGGCCTGGTGCCGC GCGGCAGCCATATGGCAGAT CTCTCGGCTACCGCTGCCAC CGTCCCGCCTGCCGCCCCG GCCGGCGAGGGTGGCCCCC CTGCACCTCCTCCAAATCTTA CCAGTAACAGGAGATGCCAG CAGACCCAGGCCCAGGTGG ATGAGGTGGTGGACATCATG AGGGTGAATGTGGACAAGGT CCTGGAGCGAGACCAGAAG CTATCGGAACTGGATGATCG CGCAGATGCCCTCCAGGCA GGGGCCTCCCAGTTTGAAA CAAGTGCAGCCAAGCTCAA GCGCAAATACTGGTGGAAA AACCTCAAGATGATGTGCTA Aggatccgaattcgagctccgtcg acaagcttgcggccgcactcgagcaccacca ccaccaccactgagatccggctgctaacaaa gcccgaaaggaagctgagttggctgctgcca ccgctgagcaataactagcataaccccttgg ggcctctaaacgggtcttgaggggttttttg ctgaaaggaggaactatatccggat | |
| 6×His-tagged ΔN-VAMP2 (49–96; capitalized) and Syntaxin-1A (191–267, I202C/I266C; capitalized) in pETDuet-1 | homemade | ggggaattgtgagcggataacaattcccctc tagaaataattttgtttaactttaagaagga gatataccATGGGCAGCAGCCATCA TCATCATCATCACAGCAGCGG CCTGGAAGTTCTGTTCCAGGG GCCCGGTAATGTGGACAAGGT CCTGGAGCGAGACCAGAAGCT ATCGGAACTGGATGATCGCGC AGATGCCCTCCAGGCAGGGGC CTCCCAGTTTGAAACAAGTGC AGCCAAGCTCAAGCGCAAATAC TGGTGGAAAAACCTCAAGATGAT GTAAgcggccgcataatgcttaagtcgaaca gaaagtaatcgtattgtacacggccgcataa tcgaaattaatacgactcactataggggaat tgtgagcggataacaattccccatcttagta tattagttaagtataagaaggagatatacat ATGGCCCTCAGTGAGATCGAGA CCAGGCACAGTGAGTGCATC AAGTTGGAGAACAGCATCCG GGAGCTACACGATATGTTCAT GGACATGGCCATGCTGGTGG AGAGCCAGGGGGAGATGATT GACAGGATCGAGTACAATGTG GAACACGCTGTGGACTACGTG GAGAGGGCCGTGTCTGACACC AAGAAGGCCGTCAAGTACCAG AGCAAGGCACGCAGGAAGAA GTGCATGATCTAActcgagtc tggtaaagaaaccgctgctgcgaaatttgaa cgccagcacatggactcgtctactagcgcag cttaattaacctaggctgctgccaccgctga gcaataactagcataaccccttggggcctct aaacgggtcttgaggggttttttgctgaaag gaggaactatatccggattggcgaatgggac gcgccctgtagcggcgcattaagcgcggcgg gtgtggtggttacgcgcagcgtgaccgctac acttgccagcgccctagcgcccgctcctttc gctttcttcccttcctttctcgccacgttcg ccggctttccccgtcaagctctaaatcgggg gctccctttagggttccgatttagtgcttta cggcacctcgaccccaaaaaacttgattagg gtgatggttcacgtagtgggccatcgccctg atagacggtttttcgccctttgacgttggag tccacgttctttaatagtggactcttgttcc aaactggaacaacactcaaccctatctcggt ctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctga tttaacaaaaatttaacgcgaattttaacaa aatattaacgtttacaatttctggcggcacg atggcatgagattatcaaaaaggatcttcac ctagatccttttaaattaaaaatgaagtttt aaatcaatctaaagtatatatgagtaaactt ggtctgacagttaccaatgcttaatcagtga ggcacctatctcagcgatctgtctatttcgt tcatccatagttgcctgactccccgtcgtgt agataactacgatacgggagggcttaccatc tggccccagtgctgcaatgataccgcgagac ccacgctcaccggctccagatttatcagcaa taaaccagccagccggaagggccgagcgca gaagtggtcctgcaactttatccgcctccatc cagtctattaattgttgccgggaagctagag taagtagttcgccagttaatagtttgcgcaa cgttgttgccattgctacaggcatcgtggtg tcacgctcgtcgtttggtatggcttcattca gctccggttcccaacgatcaaggcgagttac atgatcccccatgttgtgcaaaaaagcggtt agctccttcggtcctccgatcgttgtcagaa gtaagttggccgcagtgttatcactcatggt tatggcagcactgcataattctcttactgtc atgccatccgtaagatgcttttctgtgactg gtgagtactcaaccaagtcattctgagaata gtgtatgcggcgaccgagttgctcttgcccg gcgtcaatacgggataataccgcgccacata gcagaactttaaaagtgctcatcattggaaa acgttcttcggggcgaaaactctcaaggatc ttaccgctgttgagatccagttcgatgtaac ccactcgtgcacccaactgatcttcagcatc ttttactttcaccagcgtttctgggtgagcaaa aacaggaaggcaaaatgccgcaaaaaagg gaataagggcgacacggaaatgttgaatact catactcttcctttttcaatcatgattgaag catttatcagggttattgtctcatgagcgga tacatatttgaatgtatttagaaaaataaac aaataggtcatgaccaaaatcccttaacgtg agttttcgttccactgagcgtcagaccccgt agaaaagatcaaaggatcttcttgagatcct ttttttctgcgcgtaatctgctgcttgcaaa caaaaaaaccaccgctaccagcggtggtttg tttgccggatcaagagctaccaactcttttt ccgaaggtaactggcttcagcagagcgcaga taccaaatactgtccttctagtgtagccgta gttaggccaccacttcaagaactctgtagca ccgcctacatacctcgctctgctaatcctgt taccagtggctgctgccagtggcgataagtc gtgtcttaccgggttggactcaagacgatag ttaccggataaggcgcagcggtcgggctgaa cggggggttcgtgcacacagcccagcttgga gcgaacgacctacaccgaactgagataccta cagcgtgagctatgagaaagcgccacgcttccc gaagggagaaaggcggacaggtatccggta agcggcagggtcggaacaggagagcgcac gagggagcttccagggggaaacgcctggtatc tttatagtcctgtcgggtttcgccacctctg acttgagcgtcgatttttgtgatgctcgtca ggggggcggagcctatggaaaaacgccagc aacgcggcctttttacggttcctggccttttg ctggccttttgctcacatgttctttcctgcg ttatcccctgattctgtggataaccgtatta ccgcctttgagtgagctgataccgctcgccgc agccgaacgaccgagcgcagcgagtcagtg agcgaggaagcggaagagcgcctgatgcgg tattttctccttacgcatctgtgcggtatttc acaccgcatatatggtgcactctcagtacaa tctgctctgatgccgcatagttaagccagta tacactccgctatcgctacgtgactgggtca tggctgcgccccgacacccgccaacacccgc tgacgcgccctgacgggcttgtctgctcccg gcatccgcttacagacaagctgtgaccgtct ccgggagctgcatgtgtcagaggttttcacc gtcatcaccgaaacgcgcgaggcagctgcgg taaagctcatcagcgtggtcgtgaagcgatt cacagatgtctgcctgttcatccgcgtccag ctcgttgagtttctccagaagcgttaatgtc tggcttctgataaagcgggccatgttaaggg cggttttttcctgtttggtcactgatgcctc cgtgtaagggggatttctgttcatgggggta atgataccgatgaaacgagagaggatgctca cgatacgggttactgatgatgaacatgcccg gttactggaacgttgtgagggtaaacaactg gcggtatggatgcggcgggaccagagaaaaa tcactcagggtcaatgccagcgcttcgttaa tacagatgtaggtgttccacagggtagccag cagcatcctgcgatgcagatccggaacataa tggtgcagggcgctgacttccgcgtttccag actttacgaaacacggaaaccgaagaccatt catgttgttgctcaggtcgcagacgttttgc agcagcagtcgcttcacgttcgctcgcgtat cggtgattcattctgctaaccagtaaggcaa ccccgccagcctagccgggtcctcaacgaca ggagcacgatcatgctagtcatgccccgcgc ccaccggaaggagctgactgggttgaaggct ctcaagggcatcggtcgagatcccggtgcct aatgagtgagctaacttacattaattgcgtt gcgctcactgcccgctttccagtcgggaaac ctgtcgtgccagctgcattaatgaatcggcc aacgcgcggggagaggcggtttgcgtattgg gcgccagggtggtttttcttttcaccagtga gacgggcaacagctgattgcccttcaccgcc tggccctgagagagttgcagcaagcggtcca cgctggtttgccccagcaggcgaaaatcctg tttgatggtggttaacggcgggatataacat gagctgtcttcggtatcgtcgtatcccacta ccgagatgtccgcaccaacgcgcagcccgga ctcggtaatggcgcgcattgcgcccagcgcc atctgatcgttggcaaccagcatcgcagtgg gaacgatgccctcattcagcatttgcatggt ttgttgaaaaccggacatggcactccagtcg ccttcccgttccgctatcggctgaatttgat tgcgagtgagatatttatgccagccagccag acgcagacgcgccgagacagaacttaatggg cccgctaacagcgcgatttgctggtgaccca atgcgaccagatgctccacgcccagtcgcgt accgtcttcatgggagaaaataatactgttg atgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccac agcaatggcatcctggtcatccagcggatag ttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttc gacgccgcttcgttctaccatcgacaccacc acgctggcacccagttgatcggcgcgagatt taatcgccgcgacaatttgcgacggcgcgtg cagggccagactggaggtggcaacgccaatc agcaacgactgtttgcccgccagttgttgtg ccacgcggttgggaatgtaattcagctccgc catcgccgcttccactttttcccgcgttttc gcagaaacgtggctggcctggttcaccacgc gggaaacggtctgataagagacaccggcata ctctgcgacatcgtataacgttactggtttc acattcaccaccctgaattgactctcttccg ggcgctatcatgccataccgcgaaaggtttt gcgccattcgatggtgtccgggatctcgacg ctctcccttatgcgactcctgcattaggaag cagcccagtagtaggttgaggccgttgagca ccgccgccgcaaggaatggtgcatgcaagga gatggcgcccaacagtcccccggccacgggg cctgccaccatacccacgccgaaacaagcgc tcatgagcccgaagtggcgagcccgatcttc cccatcggtgatgtcggcgatataggcgcca gcaaccgcacctgtggcgccggtgatgccgg ccacgatgcgtccggcgtagaggatcgagat cgatctcgatcccgcgaaattaatacgactc actata | |
| SNAP-25b (1–206, all C to A; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcc agggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGCCGA GGACGCAGACATGCGCAATG AGCTGGAGGAGATGCAGAGG AGGGCTGACCAGCTGGCTGA TGAGTCCCTGGAAAGCACCC GTCGCATGCTGCAGCTGGTT GAAGAGAGTAAAGATGCTGG CATCAGGACTTTGGTTATGTT GGATGAGCAAGGCGAACAAC TGGAACGCATTGAGGAAGGG ATGGACCAAATCAATAAGGAC ATGAAAGAAGCAGAAAAGAAT TTGACGGACCTAGGAAAATTC GCCGGCCTTGCCGTGGCCCC CGCCAACAAGCTTAAATCCAG TGATGCTTACAAAAAAGCCTG GGGCAATAATCAGGATGGAGT AGTGGCCAGCCAGCCTGCCC GTGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTGGC TTCATCCGCAGGGTAACAAAT GATGCCCGGGAAAATGAGATG GATGAGAACCTGGAGCAGGT GAGCGGCATCATCGGAAACCT CCGCCACATGGCTCTAGACAT GGGCAATGAGATTGACACCCA GAATCGCCAGATCGACAGGAT CATGGAGAAGGCTGATTCCAA CAAAACCAGAATTGATGAAGC CAACCAACGTGCAACAAAGAT GCTGGGAAGTGGTTAA ctcgagcaccaccaccaccaccactgag atccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgagc aataactagcataaccccttggggcctc taaacgggtcttgaggggttttttgctgaa aggaggaactatatccggat | |
| Materials for protein purificaiton | |||
| 2-Mercaptoethanol | SIGMA | M3148-25ML | |
| Agar | LPS Solution | AGA500 | |
| Ampicillin, Sodium salt | PLS | AC1043-005-00 | |
| Chloramphenicol | PLS | CR1023-050-00 | |
| Competent cells (E. coli) | Novagen | 70956 | Rosetta(DE3)pLysS |
| Glycerol | SIGMA | G5516-500ML | |
| HEPES | SIGMA | H4034-100G | |
| Hydrochloric acid / HCl | SIGMA | 320331-500ML | |
| Imidazole | SIGMA | I2399-100G | |
| Isopropyl β-D-1-thiogalactopyranoside / IPTG | SIGMA | 10724815001 | |
| Kanamycin Sulfate | PLS | KC1001-005-02 | |
| Luria-Bertani (LB) Broth | LPS Solution | LB-05 | |
| Ni-NTA resin | Qiagen | 30210 | |
| PD MiniTrap G-25 (desalting column) | Cytiva | GE28-9180-07 | For instructions, see: https://www.cytivalifesciences.com/en/us/shop/chromatography/prepacked-columns/desalting-and-buffer-exchange/pd-minitrap-desalting-columns-with-sephadex-g-25-resin-p-06174 |
| Phenylmethylsulfonyl fluoride / PMSF | ThermoFisher Scientific | 36978 | |
| Plasmids for SNARE proteins | cloned in house | N/A | Available upon request |
| Protease inhibitor cocktail | genDEPOT | P3100 | |
| Sodium chloride | SIGMA | S5886-500G | |
| Sodium phosphate dibasic / Na2HPO4 | SIGMA | S7907-100G | |
| Sodium phosphate monobasic / NaH2PO4 | SIGMA | S3139-250G | |
| Tris(2-carboxyethyl)phosphine / TCEP | SIGMA | C4706-2G | |
| Trizma base | SIGMA | T1503-250G | |
| Materials for sample assembly | |||
| Biotin-PEG-SVA | LAYSAN BIO | BIO-PEG-SVA-5K-100MG & MPEG-SVA-5K-1g | For PEGylation |
| Dibenzocyclooctyne-amine / DBCO-NH2 | SIGMA | 761540-10MG | For bead coating |
| Double-sided tape | 3M | 136 | For flow cell assembly |
| Epoxy glue | DEVCON | S-208 | For flow cell assembly |
| Glass coverslip for bottom surface | VWR | 48393-251 | Rectangular, 60×24 mm, #1.5 |
| Glass coverslip for top surface | VWR | 48393-241 | Rectangular, 50×24 mm, #1.5 |
| Magnetic bead | ThermoFisher Scientific | 14301 | Dynabeads M-270 Epoxy, 2.8 μm |
| mPEG-SVA | LAYSAN BIO | mPEG-SVA 1g | For PEGylation |
| N,N-Dimethylformamide / DMF | SIGMA | D4551-250ML | For bead coating |
| N-[3-(trimethoxysilyl)propyl]ethylenediamine | SIGMA | 104884-100ML | For PEGylation |
| Neutravidin | ThermoFisher Scientific | 31000 | For sample tethering |
| Phosphate buffered saline / PBS, pH 7.2 | PLS | PR2007-100-00 | |
| Plastic syringe | Norm-ject | A5 | 5 ml, luer tip |
| Polyethylene Tubing | SCI | BB31695-PE/4 | PE-60 |
| Reference bead | SPHEROTECH | SVP-30-5 | Streptavidin-coated Polystyrene Particles; 3.0-3.4 µm |
| Syringe needle | Kovax | 21G-1 1/4'' | 21 G |
| Syringe pump | KD SCIENTIFIC | 788210 | |
| Equipment for magnetic tweezer instrument | |||
| 1-axis motorized microtranslation stage | PI | M-126.PD1 | For vertical positioning of magnets |
| 2-axis manual translation stage | ST1 | LEE400 | For alignment of magnets to the optical axis |
| Acrylic holder for magnets | DaiKwang Precision | custum order | Drawing available upon request |
| Frame grabber | Active Silicon | AS-FBD-4XCXP6-2PE8 | |
| High-speed CMOS camera | Mikrotron | EoSens 3CXP | |
| Inverted microscope | Olympus | IX73P2F-1-2 | |
| Neodymium magnets | LG magnet | ND 10x10x12t | Dimension: 10 mm × 10 mm × 12 mm; two needed |
| Objective lens | Olympus | UPLXAPO100XO | Oil-immersion, NA 1.45 |
| Objective lens nanopositioner | Mad City Labs | Nano-F100S | |
| Rotation stepper motor | AUTONICS | A3K-S545W | For rotating magnets |
| Superluminescent diode | QPHOTONICS | QSDM-680-2 | 680 nm |
| Software | |||
| LabVIEW | National Instruments | v20.0f1 | |
| MATLAB | MathWorks | v2021a |
Références
- Le, S., Liu, R., Lim, C. T., Yan, J. Uncovering mechanosensing mechanisms at the single protein level using magnetic tweezers. Methods. 94, 13-18 (2016).
- Choi, H. -. K., Kim, H. G., Shon, M. J., Yoon, T. -. Y. High-resolution single-molecule magnetic tweezers. Annual Review of Biochemistry. 91 (1), 33-59 (2022).
- Yang, T., Park, C., Rah, S. -. H., Shon, M. J. Nano-precision tweezers for mechanosensitive proteins and beyond. Molecules and Cells. 45 (1), 16-25 (2022).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- De Vlaminck, I., Dekker, C. Recent advances in magnetic tweezers. Annual Review of Biophysics. 41 (1), 453-472 (2012).
- Bustamante, C. J., Chemla, Y. R., Liu, S., Wang, M. D. Optical tweezers in single-molecule biophysics. Nature Reviews Methods Primers. 1, 25 (2021).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical Journal. 82 (6), 3314-3329 (2002).
- Smith, S. B., Finzi, L., Bustamante, C. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258 (5085), 1122-1126 (1992).
- Lansdorp, B. M., Tabrizi, S. J., Dittmore, A., Saleh, O. A. A high-speed magnetic tweezer beyond 10,000 frames per second. Review of Scientific Instruments. 84 (4), 044301 (2013).
- Cnossen, J. P., Dulin, D., Dekker, N. H. An optimized software framework for real-time, high-throughput tracking of spherical beads. Review of Scientific Instruments. 85 (10), 103712 (2014).
- Dulin, D., et al. High spatiotemporal-resolution magnetic tweezers: calibration and applications for DNA dynamics. Biophysical Journal. 109 (10), 2113-2125 (2015).
- Huhle, A., et al. Camera-based three-dimensional real-time particle tracking at kHz rates and Ångström accuracy. Nature Communications. 6 (1), 5885 (2015).
- Popa, I., et al. A HaloTag anchored ruler for week-long studies of protein dynamics. Journal of the American Chemical Society. 138 (33), 10546-10553 (2016).
- Shon, M. J., Kim, H., Yoon, T. -. Y. Focused clamping of a single neuronal SNARE complex by complexin under high mechanical tension. Nature Communications. 9 (1), 3639 (2018).
- Tapia-Rojo, R., Eckels, E. C., Fernández, J. M. Ephemeral states in protein folding under force captured with a magnetic tweezers design. Proceedings of the National Academy of Sciences. 116 (16), 7873-7878 (2019).
- Löf, A., et al. Multiplexed protein force spectroscopy reveals equilibrium protein folding dynamics and the low-force response of von Willebrand factor. Proceedings of the National Academy of Sciences. 116 (38), 18798-18807 (2019).
- Tapia-Rojo, R., Alonso-Caballero, A., Fernandez, J. M. Direct observation of a coil-to-helix contraction triggered by vinculin binding to talin. Science Advances. 6 (21), (2020).
- Rieu, M., et al. Parallel, linear, and subnanometric 3D tracking of microparticles with Stereo Darkfield Interferometry. Science Advances. 7 (6), (2021).
- Rieu, M., Valle-Orero, J., Ducos, B., Allemand, J. -. F., Croquette, V. Single-molecule kinetic locking allows fluorescence-free quantification of protein/nucleic-acid binding. Communications Biology. 4 (1), 1083 (2021).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences. 103 (16), 6190-6195 (2006).
- Camunas-Soler, J., Ribezzi-Crivellari, M., Ritort, F. Elastic properties of nucleic acids by single-molecule force spectroscopy. Annual Review of Biophysics. 45 (1), 65-84 (2016).
- Südhof, T. C., Rothman, J. E. Membrane fusion: grappling with SNARE and SM proteins. Science. 323 (5913), 474-477 (2009).
- Gao, Y., et al. Single reconstituted neuronal SNARE complexes zipper in three distinct stages. Science. 337 (6100), 1340-1343 (2012).
- Zorman, S., et al. Common intermediates and kinetics, but different energetics, in the assembly of SNARE proteins. eLife. 3, e03348 (2014).
- Zhang, Y., Hughson, F. M. Chaperoning SNARE folding and assembly. Annual Review of Biochemistry. 90 (1), 581-603 (2021).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H. Magnetic tweezers for single-molecule experiments. Handbook of Single-Molecule Biophysics. , 371-395 (2009).
- You, H., Le, S., Chen, H., Qin, L., Yan, J. Single-molecule manipulation of G-quadruplexes by magnetic tweezers. Journal of Visualized Experiments. (127), e56328 (2017).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical Journal. 96 (12), 5040-5049 (2009).
- Dulin, D., Barland, S., Hachair, X., Pedaci, F. Efficient illumination for microsecond tracking microscopy. PLoS One. 9 (9), e107335 (2014).
- Klaue, D., Seidel, R. Torsional stiffness of single superparamagnetic microspheres in an external magnetic field. Physical Review Letters. 102 (2), 028302 (2009).
- Shon, M. J., Rah, S. -. H., Yoon, T. -. Y. Submicrometer elasticity of double-stranded DNA revealed by precision force-extension measurements with magnetic tweezers. Science Advances. 5 (6), 1697 (2019).
- Czerwinski, F., Richardson, A. C., Oddershede, L. B. Quantifying noise in optical tweezers by Allan variance. Optics Express. 17 (15), 13255-13269 (2009).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. Review of Scientific Instruments. 83 (2), 025115 (2012).
- Ostrofet, E., Papini, F. S., Dulin, D. High spatiotemporal resolution data from a custom magnetic tweezers instrument. Data in Brief. 30, 105397 (2020).
- Yu, Z., et al. A force calibration standard for magnetic tweezers. Review of Scientific Instruments. 85 (12), 123114 (2014).
- Strick, T. R., Allemand, J. -. F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271 (5257), 1835-1837 (1996).
- Daldrop, P., Brutzer, H., Huhle, A., Kauert, D. J., Seidel, R. Extending the range for force calibration in magnetic tweezers. Biophysical Journal. 108 (10), 2550-2561 (2015).
- te Velthuis, A. J. W., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative guidelines for force calibration through spectral analysis of magnetic tweezers data. Biophysical Journal. 99 (4), 1292-1302 (2010).
- Ostrofet, E., Papini, F. S., Dulin, D. Correction-free force calibration for magnetic tweezers experiments. Scientific Reports. 8 (1), 15920 (2018).
- Seol, Y., Li, J., Nelson, P. C., Perkins, T. T., Betterton, M. D. Elasticity of short DNA molecules: theory and experiment for contour lengths of 0.6-7 µm. Biophysical Journal. 93 (12), 4360-4373 (2007).
- Burnham, D. R., Vlaminck, I. D., Henighan, T., Dekker, C. Skewed Brownian fluctuations in single-molecule magnetic tweezers. PLoS One. 9 (9), 108271 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protocols. 3 (1), 101152 (2022).
- Lee, H. -. W., et al. Profiling of protein-protein interactions via single-molecule techniques predicts the dependence of cancers on growth-factor receptors. Nature Biomedical Engineering. 2 (4), 239-253 (2018).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Parthasarathy, R. Rapid, accurate particle tracking by calculation of radial symmetry centers. Nature Methods. 9 (7), 724-726 (2012).
- Woodside, M. T., Block, S. M. Reconstructing folding energy landscapes by single-molecule force spectroscopy. Annual Review of Biophysics. 43 (1), 19-39 (2014).
- Evans, E., Ritchie, K. Dynamic strength of molecular adhesion bonds. Biophysical Journal. 72 (4), 1541-1555 (1997).
- Zhang, Y. Energetics, kinetics, and pathway of SNARE folding and assembly revealed by optical tweezers. Protein Science. 26 (7), 1252-1265 (2017).
- Chen, H., et al. Improved high-force magnetic tweezers for stretching and refolding of proteins and short DNA. Biophysical Journal. 100 (2), 517-523 (2011).
- Cho, S., et al. Tension exerted on cells by magnetic nanoparticles regulates differentiation of human mesenchymal stem cells. Biomaterials Advances. 139, 213028 (2022).
- Shon, M. J., Cohen, A. E. Nano-mechanical measurements of protein-DNA interactions with a silicon nitride pulley. Nucleic Acids Research. 44 (1), 7 (2016).
- Cheng, Y. Single-particle cryo-EM-How did it get here and where will it go. Science. 361 (6405), 876-880 (2018).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Neupane, K., et al. Direct observation of transition paths during the folding of proteins and nucleic acids. Science. 352 (6282), 239-242 (2016).
- Choi, H. -. K., et al. Watching helical membrane proteins fold reveals a common N-to-C-terminal folding pathway. Science. 366 (6469), 1150-1156 (2019).
- Kim, C., et al. Extreme parsimony in ATP consumption by 20S complexes in the global disassembly of single SNARE complexes. Nature Communications. 12 (1), 3206 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon