Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Pinzas magnéticas de alta velocidad para mediciones nanomecánicas en elementos sensibles a la fuerza
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí, describimos una configuración de pinza magnética de alta velocidad que realiza mediciones nanomecánicas en biomoléculas sensibles a la fuerza a la velocidad máxima de 1.2 kHz. Introducimos su aplicación a horquillas de ADN y complejos SNARE como sistemas modelo, pero también será aplicable a otras moléculas involucradas en eventos mecanobiológicos.
Resumen
Las pinzas magnéticas (MT) de una sola molécula han servido como herramientas poderosas para interrogar con fuerza a las biomoléculas, como los ácidos nucleicos y las proteínas, y por lo tanto están preparadas para ser útiles en el campo de la mecanobiología. Dado que el método comúnmente se basa en el seguimiento basado en imágenes de perlas magnéticas, el límite de velocidad en la grabación y análisis de imágenes, así como las fluctuaciones térmicas de las perlas, ha obstaculizado durante mucho tiempo su aplicación en la observación de cambios estructurales pequeños y rápidos en las moléculas objetivo. Este artículo describe métodos detallados para la construcción y operación de una configuración de MT de alta resolución que puede resolver la dinámica de milisegundos a nanoescala de biomoléculas y sus complejos. Como ejemplos de aplicación, se demuestran experimentos con horquillas de ADN y complejos SNARE (maquinaria de fusión de membranas), centrándose en cómo se pueden detectar sus estados transitorios y transiciones en presencia de fuerzas a escala de piconewton. Esperamos que las MT de alta velocidad continúen permitiendo mediciones nanomecánicas de alta precisión en moléculas que detectan, transmiten y generan fuerzas en las células y, por lo tanto, profundicen nuestra comprensión a nivel molecular de la mecanobiología.
Introducción
Las células detectan y responden activamente a estímulos mecánicos. Al hacerlo, muchas biomoléculas muestran propiedades dependientes de la fuerza que permiten cambios estructurales dinámicos. Ejemplos bien apreciados incluyen canales iónicos mecanosensibles y elementos citoesqueléticos que proporcionan a las células información mecánica clave de su entorno.
Además, las moléculas que muestran una naturaleza única de soporte de fuerza también pueden considerarse mecanosensibles en un sentido más amplio. Por ejemplo, la formación local y la fusión de dúplex de ácidos nucleicos, así como estructuras de orden superior como los cuádruples G, desempeñan un papel crucial en la replicación, transcripción, recombinación y, más recientemente, en la edición del genoma. Además, algunas proteínas neuronales implicadas en las comunicaciones sinápticas realizan sus funciones generando fuerzas físicas que superan los niveles de interacciones intermoleculares típicas. No importa qué ejemplo se estudie, la investigación de la nanomecánica de las biomoléculas involucradas con alta precisión espaciotemporal resultará muy útil para revelar los mecanismos moleculares de los procesos mecanobiológicos asociados 1,2,3.
Los métodos de espectroscopia de fuerza de molécula única han servido como herramientas poderosas para examinar las propiedades mecánicas de las biomoléculas 2,4,5,6. Pueden monitorear los cambios estructurales en ácidos nucleicos y proteínas simultáneamente con la aplicación de fuerza, examinando así las propiedades dependientes de la fuerza. Dos configuraciones bien conocidas son las pinzas ópticas y las pinzas magnéticas (MT), que emplean perlas de tamaño micrométrico para manipular moléculas 5,6,7,8. En estas plataformas, el poliestireno (para pinzas ópticas) o las perlas magnéticas (para MT) están atados a moléculas objetivo (por ejemplo, ácidos nucleicos y proteínas) a través de "mangos" moleculares, típicamente hechos de fragmentos cortos de ADN bicatenario (dsDNA). Luego, las perlas se mueven para ejercer fuerza y se obtienen imágenes para rastrear sus ubicaciones que informan sobre cambios estructurales en las moléculas objetivo. Las pinzas ópticas y magnéticas son en gran medida intercambiables en sus aplicaciones, pero existen diferencias importantes en sus enfoques para controlar la fuerza. Las pinzas ópticas son instrumentos intrínsecamente de abrazadera de posición que atrapan cuentas en posición, debido a lo cual la fuerza aplicada fluctúa cuando una construcción objetivo sufre cambios de forma; El aumento de extensión, como el despliegue, afloja la correa y reduce la tensión, y viceversa. Aunque se puede implementar retroalimentación activa para controlar la fuerza en las pinzas ópticas, las MT, en contraste, operan naturalmente como un dispositivo de abrazadera de fuerza, aprovechando las fuerzas magnéticas estables y de campo lejano mediante imanes permanentes, que también pueden soportar perturbaciones ambientales.
A pesar de su larga historia y diseño simple, los MT se han quedado atrás de las pinzas ópticas en sus aplicaciones a mediciones de alta precisión, en gran parte debido a los desafíos técnicos en el seguimiento rápido de cuentas. Recientemente, sin embargo, varios grupos han liderado conjuntamente una mejora multifacética de hardware y software para instrumentos de MT 2,9,10,11,12,13,14,15,16,17,18,19 . En este trabajo, presentamos un ejemplo de tal configuración que se ejecuta a 1.2 kHz y describimos cómo usarla para realizar mediciones nanomecánicas en biomoléculas sensibles a la fuerza. Como sistemas modelo, empleamos horquillas de ADN y complejos SNARE neuronales y examinamos sus rápidos cambios estructurales en el régimen de piconewton. Las horquillas de ADN exhiben transiciones simples de dos estados en un rango de fuerza bien definido20,21 y, por lo tanto, sirven como modelos de juguete para verificar el rendimiento de una configuración de pinzas. A medida que las proteínas SNARE se ensamblan en un complejo sensible a la fuerza que impulsa la fusión de membrana22, también han sido ampliamente estudiadas por espectroscopia de fuerza de molécula única 14,23,24,25. Se presentan enfoques estándar para analizar datos y extraer información útil sobre termodinámica y cinética. Esperamos que este artículo pueda facilitar la adopción de MT de alta precisión en estudios mecanobiológicos y motivar a los lectores a explorar sus propios sistemas de interés sensibles a la fuerza.
Protocolo
Todos los materiales y equipos descritos en este protocolo se enumeran en la Tabla de materiales. El software LabVIEW para operar la configuración de MT de alta velocidad que se describe a continuación, así como los scripts de MATLAB para analizar datos de muestra, se depositan en GitHub (https://github.com/ShonLab/Magnetic-Tweezers) y están disponibles públicamente.
1. Construcción de aparatos
NOTA: El principio general de la construcción MT de alta velocidad es similar a los sistemas MT convencionales estándar, excepto por el uso de una cámara semiconductora complementaria de óxido metálico (CMOS) de alta velocidad y una fuente de luz coherente de alta potencia (Figura 1). Consulte otras fuentes para obtener más descripciones de los instrumentos de MT estándar 5,26,27.
- Coloque un microscopio invertido en una mesa óptica antivibración. Instale una cámara CMOS de alta velocidad y un capturador de fotogramas.
- Construye un escenario de traslación para manipular imanes en 3D. Monte una platina lineal motorizada (>20 mm de recorrido) verticalmente sobre una platina XY manual.
NOTA: El movimiento vertical controla la fuerza, mientras que la etapa XY es para la alineación manual de imanes al eje óptico para la construcción inicial de la configuración. - Instale un motor paso a paso rotativo y un sistema de correa y polea para imanes giratorios.
NOTA: La correa transmite el movimiento giratorio entre el eje del motor y los imanes que están a unos centímetros de distancia. La rotación de los imanes es interna a la manipulación traslacional. - Monta los imanes. Utilice un soporte de acrílico (pedido a una empresa fabricante; consulte la Figura suplementaria S1) que pueda albergar firmemente dos imanes idénticos en paralelo, con un espacio bien definido de 1 mm entre los imanes (Figura 1B). Para utilizar la fuerza máxima que se puede obtener con un par de imanes determinados, ajuste la posición vertical de la etapa de traslación de modo que la superficie inferior de los imanes se alinee con el plano de muestra cuando se mueva a la posición más baja.
NOTA: Consulte Lipfert et al. para obtener más información sobre el diseño y la configuración del soporte de los imanes28. La altura y la orientación de los imanes son controladas por el software LabVIEW junto con la adquisición de datos. - Viendo con una lente de objetivo de bajo aumento, alinee los imanes al centro del campo de visión. Compruebe que la rotación de los imanes no provoca un gran desplazamiento del centro del par de imanes.
NOTA: Si el punto medio entre los imanes gira alrededor del eje de rotación, es probable que los imanes estén descentrados debido a un soporte imperfecto. Un pequeño nivel de desalineación en relación con el tamaño del espacio es tolerable, ya que la rotación del imán es solo para verificar las correas y aplicar pares en aplicaciones específicas. - Instale un diodo superluminiscente (SLD) para la iluminación de cuentas. Pase el haz a través del espacio de 1 mm entre los dos imanes. Asegúrese de que el haz esté correctamente colimado para encajar en el espacio y que la iluminación no esté ensombrecida por los imanes.
- Instale un escáner de lente piezoeléctrica en la pieza nasal y monte una lente objetivo de inmersión en aceite de 100x (apertura numérica [NA]: 1.45) para el seguimiento de cuentas. Para evitar posibles artefactos en el seguimiento de los resultados, asegúrese de que la iluminación se mantenga uniformemente cuando se muevan los imanes. Finalmente, ajuste el nivel de luz al brillo máximo sin saturar píxeles.
NOTA: Para la comparación de diferentes fuentes de luz para el seguimiento de alta velocidad de cuentas, consulte Dulin et al.29.
2. Calibración de la fuerza magnética
- Usando la reacción en cadena de la polimerasa (PCR; ver Tabla 1), prepare fragmentos de dsDNA de 5 kbp (usando Primer B, Primer Z_5k y λ-ADN) que se marquen con biotina en un extremo (para la fijación a la superficie) y azida en el otro extremo (para la fijación de perlas).
- Después de la sección 6, prepare una celda de flujo con las moléculas de 5 kbp.
- Después de la sección 7, identifique una buena construcción de correa de cuentas verificando su extensión y rotación. En particular, asegúrese de elegir un talón con una trayectoria de rotación mínima (es decir, con un radio <200 nm) para minimizar el desplazamiento de la altura del talón debido al accesorio descentrado30,31. Una vez que se identifique una buena correa, comience a rastrear cuentas, refiriéndose a la sección 9.
- Si la configuración es nueva, caracterice su ruido y estabilidad para mediciones confiables de alta resolución. Coloque el imán ~3 mm de la superficie de la celda de flujo (para aplicar >10 pN y suprimir el movimiento browniano de una cuenta), rastree la posición z del cordón a 1.2 kHz y calcule la desviación de Allan (AD) de la serie temporal de coordenadas z 32,33 (Figura 2C). Compruebe que los valores de AD de unos pocos nanómetros son alcanzables en el régimen de alta velocidad (<0,1 s), y que el seguimiento diferencial (posición del cordón magnético en relación con un cordón de referencia) reduce la AD en una escala de tiempo más larga.
NOTA: Normalmente obtenemos un AD de <3 nm a la velocidad máxima (resolución de 1,2 kHz o 0,83 ms), y el AD sigue disminuyendo al menos hasta 10 s, lo que implica una deriva mínima. Otros han reportado valores similares en configuraciones similares 9,10,11,12,34. - Con imanes en posición de reposo (F ~ 0 pN), registre las coordenadas x e y de la cuenta atada a 1,2 kHz. Registre la posición durante un período suficientemente largo (es decir, suficientemente largo que el tiempo de relajación característico de fluctuación35) para que el movimiento browniano sea suficientemente muestreado.
NOTA: Aquí, la dirección x está a lo largo de la dirección del campo magnético, mientras que el movimiento en y representa el movimiento transversal perpendicular al campo. - Mueva los imanes más cerca de la celda de flujo y repita las mediciones de posición de perlas hasta que los imanes toquen suavemente la parte superior de la celda de flujo. Muévase en pasos grandes (por ejemplo, 1-2 mm) cuando los imanes estén a más de 7 mm de distancia del plano de muestra (ya que la fuerza aplicada aumenta lentamente en el campo lejano de imanes), pero reduzca el tamaño del paso gradualmente (por ejemplo, 0.1-0.5 mm) a medida que se acercan para una calibración más fina a niveles de fuerza más altos (Figura 2B).
- Calcule la fuerza en cada posición del imán, d, utilizando cualquiera de los dos métodos alternativos (se proporciona un script de MATLAB "force calibration.m" que incluye ambos métodos; consulte el archivo complementario 1).
- Mida la varianza de las coordenadas
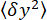 y de la cuenta (Figura 2D) y la posición z media de la cuenta en relación con la posición
y de la cuenta (Figura 2D) y la posición z media de la cuenta en relación con la posición 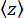 más baja (Figura 2B, abajo). Luego, use la ecuación (1)7,27,36 para estimar la fuerza (con un radio de perla fijo R = 1,400 nm y energía térmica k RT = 4.11 pN∙nm):
más baja (Figura 2B, abajo). Luego, use la ecuación (1)7,27,36 para estimar la fuerza (con un radio de perla fijo R = 1,400 nm y energía térmica k RT = 4.11 pN∙nm):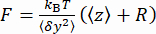 (1)
(1) - Alternativamente, calcule la densidad espectral de potencia (PSD) de las coordenadas y, Sy (Figura 2E). Determine la fuerza aplicada F ajustando un modelo de doble lorentziano37 a la medida Sy usando la ecuación (2).
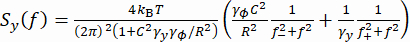 (2)
(2)
Aquí, , R es el radio de la cuenta, γ y y γφ son los coeficientes de arrastre traslacional y rotacional, respectivamente (estimado a partir de la ecuación de Stokes-Einstein), kRT es la energía térmica,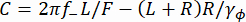 f + y f- son dos frecuencias características obtenidas usando la ecuación (3).
f + y f- son dos frecuencias características obtenidas usando la ecuación (3). 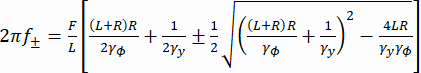 (3)
(3)
NOTA: Dado que la extensión de la correa L es una función de fuerza que sigue el modelo bien establecido de cadena helicoidal (WLC), las expresiones anteriores dejan F como el único parámetro de ajuste (fijamos R en 1.400 nm para simplificar porque se comparte en todos los niveles de fuerza y el valor exacto no influye apreciablemente en los resultados). Cuando es necesario, el desenfoque de movimiento y el aliasing de la adquisición de imágenes basadas en cámara deben considerarse38,39, pero este efecto es insignificante en nuestras mediciones de alta velocidad por encima de 1 kHz con ataduras de 5 kbp.
- Mida la varianza de las coordenadas
- Repita los pasos 2.4-2.7 para algunas construcciones más. Pruebe de tres a cinco cuentas diferentes para promediar la variabilidad de la fuerza entre las perlas magnéticas.
NOTA: La variación de fuerza entre las perlas magnéticas en uso debe considerarse para determinar el número adecuado de construcciones para el promedio. Esta variabilidad es pequeña, pero puede conducir a más de 1 pN de error en la fuerza medida, incluso para productos comerciales31. Para la mayoría de las aplicaciones, donde la determinación absoluta de las fuerzas involucradas no es crucial, promediar los resultados de calibración de tres a cinco perlas es generalmente suficiente. Un enfoque alternativo para explicar esta variación es medir la fuerza con ataduras individuales al comienzo del experimento, lo que puede llevar mucho tiempo. Otra opción es incrustar estructuras de horquilla que se descomprimen a niveles de fuerza conocidos en cada construcción31. - Trazar la fuerza medida en función de la distancia del imán y ajustar una función exponencial doble a los datos (Figura 2F) utilizando la ecuación (4).
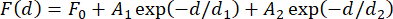 (4)
(4)
Aquí, F0 (línea de base), A 1 y A 2 (amplitudes), y d 1 y d2 (constantes de desintegración) son parámetros de ajuste. Asegúrese de que los valores de fuerza de los dos métodos, así como los ajustes doble-exponenciales resultantes, coincidan en gran medida (Figura 2F, G).
NOTA: Para confirmar que la calibración de la fuerza se realiza correctamente, verifique la relación fuerza-extensión de las construcciones probadas trazando la extensión frente a la fuerza medida. - Para corregir el desplazamiento z de la altura del talón resultante de la inclinación dependiente de la fuerza de las perlas magnéticas30,31, estime z off desde el desplazamiento lateral xoff, considerando la geometría de una correa descentrada con un radio de perla usando la ecuación (5), y aplique los valores a los valoresde extensión medidos. Este paso se implementa en el script de MATLAB "force calibration.m" (líneas 252-254).
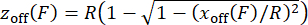 (5)
(5)
NOTA: Aunque esta corrección realiza pequeños cambios en la extensión, especialmente para las perlas con un radio de rotación pequeño (<200 nm), este desplazamiento a menudo afecta críticamente la respuesta elástica, como se ve en el cambio de la Figura 2H a la Figura 2I30,31. - Verifique la longitud de persistencia Lp ajustando un modelo WLC extensible a los datos utilizando la ecuación (6).
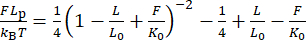 (6)
(6)
Aquí, L 0 es la longitud del contorno (1,7 μm para 5 kbp) y K0 es el módulo para el estiramiento entálpico.
NOTA: Aunque se acepta que el L p del dsDNA es de 40-50 nm en un tampón típico como la solución salina tamponada con fosfato (PBS), la fórmula WLC aplicada a moléculas cortas (<5 kbp) subestima sistemáticamente L p ya que L0 disminuye31,40. Esto se debe a que el modelo WLC clásico supone un polímero cuya longitud de cadena es suficientemente larga que su longitud de persistencia. Aquí, obtuvimos L p = 40 ± 3 nm para la construcción de 5 kbp (Figura 2H), y la corrección de extensión produjo además un K0 homogéneo de 1.100 ± 200 pN (Figura 2I). La aplicación de un modelo WLC finito 31,40, así como una corrección para la no gaussianidad en la distribución de extensión 41, aumentará ligeramente Lp. - Una vez que se verifique la calibración de fuerza, aplique los parámetros de ajuste obtenidos del modelo de doble exponencial al software LabVIEW proporcionado (Archivo Suplementario 2) y espere a que el software calcule la fuerza actual en tiempo real a partir de las lecturas del motor (es decir, la posición del imán). Dado que no se dispone de una expresión analítica para la función inversa d(F), prepare una tabla de búsqueda de d frente a F en pasos de 0,1 pN mediante una estimación numérica de para los niveles de fuerza objetivo d. Almacene esta tabla en el software también para comandar el control de fuerza.
3. Síntesis de horquillas de ADN
NOTA: Las construcciones de horquilla de ADN para experimentos de MT se preparan mediante amplificación por PCR de una región de 510 pb en λ-ADN con dos cebadores personalizados, uno de los cuales contiene una estructura de horquilla en su extremo 5' (Figura 3A). De esta manera, se coloca un motivo de horquilla en un extremo del producto de PCR.
- Prepara las imprimaciones.
- Imprimación directa: imprimación B_hp que está marcada con 5 '-biotina para la fijación de la superficie del vidrio y se une al λ-ADN. Este imprimador contiene un motivo de horquilla con un vástago de 8 pb y un bucle de 6 nt, 5' a la región de unión λ.
- Cebador inverso: El cebador Z_hp que está marcado con 5′-azida para la fijación de perlas magnéticas y se une al λ-ADN a 1 kbp del cebador delantero.
- Configure y ejecute la PCR con λ-ADN (plantilla), polimerasa nTaq y condiciones estándar de PCR (ver Tabla 1). Limpie el producto con un kit de purificación comercial.
- Mida la concentración de ADN por absorción UV a 260 nm (A260) y realice electroforesis en gel de agarosa (gel al 2%) (ver Tabla 2) para verificar el tamaño del producto. Un rendimiento típico es ~35 μL de ~600 nM solución.
4. Preparación de proteínas SNARE
NOTA: Los complejos SNARE neuronales se ensamblan combinando tres proteínas purificadas de rata expresadas a partir de E. coli: VAMP2/sinaptobrevin-2, sintaxina-1A y SNAP-25 (Figura 3B). Para facilitar su ensamblaje, la sintaxina y SNAP-25 se co-expresan con un fragmento VAMP2 (que carece de la región N-terminal; denominado "ΔN-VAMP2") en una estructura llamada "complejo ΔN", y luego se mezclan con VAMP2 de longitud completa después de la unión del mango de ADN para formar complejos completos.
- Preparar plásmidos que contengan ADNc para la expresión de proteínas SNARE (las secuencias de ADN para todos los plásmidos se dan en la Tabla de materiales).
- Preparar VAMP2 marcado con 6×His-His-que carece del dominio transmembrana (2-97; L32C/I97C para enlaces disulfuro) clonado en un vector pET28a.
- Preparar la sintaxina-1A que carece del Habc y el dominio transmembrana (191-267, sustituciones I202C/I266C para enlaces disulfuro) clonados junto con ΔN-VAMP2 (49-96) marcados con 6×His en un vector pETDuet-1.
- Prepare la isoforma b de SNAP-25 de longitud completa (2-206, todas C a A) clonada en un vector pET28a. Esto se utilizará para preparar complejos ΔN.
- Prepare la isoforma b SNAP-25 de longitud completa marcada con 6×His (1-206, todas C a A) clonada en un vector pET28a para su adición directa al tampón de ensayo MT para volver a ensamblar los complejos SNARE después del despliegue.
- Prepare dos tubos de células de E. coli Rosetta (DE3). Transformar un grupo con plásmidos VAMP2 (del paso 4.1.1), uno con sintaxina-1A/ΔN-VAMP2 y plásmidos SNAP-25 no marcados (de los pasos 4.1.2 y 4.1.3) para expresar el complejo ΔN, y el otro con plásmidos SNAP-25 marcados con His (desde el paso 4.1.4).
- Transfiera las células transformadas al caldo Luria-Bertani (LB) con antibióticos apropiados (aquí, kanamicina y cloranfenicol para VAMP2 y SNAP-25 marcado con His; kanamicina, cloranfenicol y ampicilina para el complejo ΔN). Cultivarlos a 37 °C en una incubadora agitadora (220 rpm) hasta que la densidad óptica (OD) del caldo alcance 0.7-0.9.
- Añadir 1 mM de isopropilo β-d-1-tiogalactopiranósido (IPTG) para inducir la expresión de proteínas e incubar las células durante 3-4 h a 37 °C en una incubadora agitadora (220 rpm).
- Granular las células centrifugando el cultivo a 4.500 × g durante 15 min a 4 °C.
- Prepare tampones para la purificación de proteínas (ver Tabla 2).
- Suspender los gránulos celulares que expresan SNARE en 40 ml de tampón de lisis helada y lisar las células por sonicación en hielo (15% de amplitud, 5 s encendido y 5 s apagado, 30 min en total).
- Centrifugar el lisado a 15.000 × g durante 30 min a 4 °C para eliminar los materiales insolubles.
- Pase el sobrenadante a través de una columna de gravedad llena con 1 ml de resina Ni-NTA. Lave la resina con tampón de lavado A, luego con tampón de lavado B, y eluya las proteínas con 10 ml de tampón de elución.
- Retire la tris(2-carboxietil)fosfina (TCEP) y el imidazol del eluyente usando una columna de desalinización (siga las instrucciones del fabricante). Eluya la muestra con PBS.
- Concentrar las proteínas con filtros centrífugos (corte de 10 kDa) a ~70 μM mientras se mantienen las proteínas en PBS (típicamente produciendo 2 mL). Mida la concentración de proteína por absorción ultravioleta (UV) a 280 nm (A280) o por el ensayo de Bradford.
- Preparar pequeñas alícuotas, congelar rápidamente en nitrógeno líquido y almacenar a -80 °C hasta su uso.
NOTA: Los complejos SNARE completos se ensamblarán después de conjugar el complejo ΔN en un mango de ADN (ver más abajo).
5. Fijación de mangos de ADN
NOTA: Dos mangos de dsDNA de 510 pb que contienen grupos amina primarios en un extremo se preparan primero mediante PCR, y los grupos amina se convierten en grupos de maleimida mediante el uso de un reticulante bifuncional, SM (PEG) 2. Los dos mangos se unen covalentemente a los complejos SNARE a través de sus grupos de cisteína para la conjugación específica del sitio (Figura 3B).
- Preparar imprimaciones.
- Prepare imprimaciones directas: imprimación B (para amplificar el mango B) que está marcado con 5′-biotina para la fijación de la superficie del vidrio y se une al λ-ADN; Primer Z (para amplificar el mango Z) que está marcado con 5′-azida para la fijación de perlas magnéticas y tiene la misma secuencia que el cebador B.
- Prepare un cebador inverso: Primer N (compartido para el mango B y el mango Z) que está marcado con 5′-amina para la conjugación de proteínas y se une al λ-ADN 510 pb lejos del cebador delantero.
- Configure y ejecute dos series de reacciones de PCR (18 tubos de reacción de 200 μL para cada mango) con λ-ADN (plantilla), polimerasa nTaq y condiciones de PCR estándar (ver Tabla 1). Limpie el producto con un kit de limpieza de PCR y eluya cada mango con 45 μL de agua ultrapura. Utilice un volumen mínimo de agua para obtener altas concentraciones de mangos para una reacción efectiva en pasos posteriores.
- Mida la concentración de ADN por A260. El rendimiento típico es ~650 μL de solución ~2 μM para cada mango. Mantenga las muestras pequeñas separadas para su posterior verificación en la electroforesis en gel de agarosa.
- Reaccione cada mango (1 μM en PBS) con 5 mM SM(PEG)2. Incubar a temperatura ambiente con una rotación suave. Después de 1 h, utilice un kit de purificación de ADN para eliminar SM(PEG)2 sin reaccionar. Eluya cada mango con 250 μL de PBS para obtener soluciones de ~2 μM.
- Mezclar las soluciones de mango B y complejo ΔN en una proporción molar de 1:16 (por ejemplo, 1 μM mango B y 16 μM ΔN-complejo) en PBS e incubar durante 2 h a temperatura ambiente con agitación. Mantenga separada una pequeña muestra para la electroforesis en gel de agarosa.
- Añadir una solución de VAMP2 en un exceso molar de 2,5 veces sobre el complejo ΔN utilizado en el paso anterior. Incubar la mezcla durante otras 1 h a temperatura ambiente con agitación. Los complejos SNARE completos se ensamblan en este paso.
- Eliminar las proteínas libres mediante intercambio amortiguador con PBS fresco y un filtro centrífugo (corte de 100 kDa): centrifugar a 14.000 × g durante 5 min a 4 °C, repetir al menos 6x y ejecutar durante 15 min para el último giro. Mida el aumento en la relación A260 / A280 para monitorear la eliminación de proteínas libres. Mantenga separada una pequeña muestra para la electroforesis en gel de agarosa.
- Agregue el mango Z a la solución en un exceso molar de 15 veces sobre el mango B. Mantenga la concentración del mango Z al menos por encima de 1 μM para facilitar la reacción. Incubar la mezcla durante la noche a 4 °C con agitación.
- Verificar los intermedios (Mango B y sus conjugados proteicos) y el producto final (complejo SNARE con dos asas) mediante electroforesis en gel de agarosa (Figura 3B, recuadro) (ver Tabla 2).
NOTA: Si las proteínas se unen correctamente al mango B, se detectará un cambio de movilidad. En particular, la formación de complejos SNARE completos en los mangos de ADN puede confirmarse por su resistencia al dodecil sulfato de sodio (SDS), a diferencia de los complejos ΔN, que se desmontan en SDS y dejan solo la sintaxina unida al ADN (comparar b y c en la Figura 3B). - Preparar pequeñas alícuotas, congelar rápidamente en nitrógeno líquido y almacenar a -80 °C hasta su uso.
NOTA: Aunque la solución final contiene mangos sin reaccionar, solo se seleccionará la construcción deseada que esté doblemente etiquetada con biotina y azida durante el ensamblaje de la muestra en una celda de flujo.
6. Fabricación de células de flujo
NOTA: Las celdas de flujo para mediciones de MT se construyen a partir de dos cubreobjetos de vidrio unidos entre sí por cinta de doble cara (Figura 3C). Un cubreobjetos está recubierto con una mezcla de PEG y polietilenglicol biotinilado (PEG) para evitar la unión inespecífica y para permitir el anclaje específico de las moléculas diana a través del enlace biotina-neutravidina (Figura 3D). Luego, las soluciones de materiales para experimentos de MT se infunden secuencialmente en una celda de flujo mediante el uso de una bomba de jeringa (Figura 3C, D).
- Prepare dos cubreobjetos de vidrio, uno para la superficie superior (24 mm × 50 mm, espesor nº 1,5) y otra inferior (24 mm × 60 mm, espesor nº 1,5). Limpie los cubreobjetos por sonicación en 1 M KOH durante 30 min. Después de la sonicación, enjuague los cubreobjetos con agua destilada y manténgalos en agua hasta el siguiente paso.
- PEGylate el cubreobjetos inferior siguiendo los protocolos publicados42,43. Use N-[3-(trimetoxisilil)propil]etilendiamina para la silanización y una mezcla 1:100 (ww) de biotina-PEG-SVA y mPEG-SVA en tampón bicarbonato de 100 mM. Mantener los cubreobjetos PEGylated secos a -20 °C y guárdelos durante algunas semanas.
- El día de los experimentos, saque los cubreobjetos PEGylated y séquelos con una pistola de nitrógeno. Inspecciónelos visualmente en busca de suciedad para asegurarse de que estén limpios.
- Para hacer los canales de muestra, prepare tiras de cinta de cinta de doble cara de ~2 mm de ancho y coloque cuatro tiras en un cubreobjetos inferior (superficie pegilada hacia arriba), paralelas y separadas entre sí por ~5 mm (Figura 3C).
NOTA: De esta manera, se pueden crear tres canales de muestra de 5 mm de ancho en una sola celda de flujo. - Coloque un cubreobjetos superior en el centro del cubreobjetos inferior, dejando ~5 mm de espacio en los bordes cortos para las entradas y salidas del canal. Presione suavemente la parte posterior del cubreobjetos superior con pinzas para sellar firmemente los canales.
- Para hacer un depósito de entrada, recorte el borde de una punta de pipeta de 200 μL. Corte ~10 mm de la abertura más ancha para permitir la retención de ~200 μL de solución. Haga tres de ellos para los tres canales de flujo. Para configurar las salidas, prepare tres agujas de jeringa que se ajusten al tubo de la bomba de la jeringa.
- Con un epoxi de 5 minutos, pegue los depósitos y los cubos de agujas a la celda de flujo. Asegúrese de que se forme un sello completo para evitar fugas y que los canales no estén bloqueados con exceso de pegamento. Dejar secar durante al menos 30 min.
7. Montaje de construcciones de correa de cuentas
NOTA: Las soluciones de materiales para experimentos de MT, incluidas las de construcciones de correa de cuentas, se introducen secuencialmente en celdas de flujo mediante el uso de una bomba de jeringa (Figura 3C, D).
- Prepare cuentas magnéticas. Tomar 5 mg de perlas M270-epoxi de una solución madre (~3,3 × 108 perlas en 167,5 μL de dimetilformamida) y sustituir el disolvente por tampón fosfato (ver Tabla 2) mediante separación magnética de las perlas.
- Preparar las perlas a ~1,1 × 109 perlas mL−1 en un tampón fosfato con 1 M de sulfato de amonio y reaccionarlas con 2 mM de dibenzociclooctino (DBCO)-NH2. Incubar la mezcla durante 3 h en una batidora giratoria a temperatura ambiente. Después de la reacción, lave las perlas 3 veces con tampón de fosfato fresco para eliminar las moléculas sin reaccionar.
NOTA: Las perlas lavadas se pueden almacenar sin rotación adicional a 4 °C durante varias semanas antes de su uso. - Conecte una aguja en la salida del canal de celda de flujo a la bomba de jeringa con tubo de polietileno. Equilibra los canales con PBS.
- Introduzca las siguientes soluciones secuencialmente en el canal por succión con la bomba: NeutrAvidin, construcciones objetivo (horquillas de ADN o complejos SNARE con mangos de ADN), perlas de poliestireno de referencia y perlas magnéticas recubiertas de DBCO. Antes de su uso, voltee las soluciones de perlas a fondo para dispersar los posibles agregados de perlas.
- Lave las perlas no unidas mientras aplica 0.1 pN de fuerza.
NOTA: La aplicación de una pequeña fuerza hacia arriba facilita la eliminación de las perlas no unidas y ayuda a evitar la ruptura de construcciones de amarre de cuentas específicamente unidas. - Para experimentos con complejos SNARE, incluya 1.5 μM SNAP-25 en el tampón final.
NOTA: Las moléculas libres de SNAP-25 pueden volver a unirse a los complejos SNARE después del despliegue y permiten mediciones repetidas en un solo complejo.
8. Identificación de constructos objetivo
- En la superficie de un canal de celda de flujo, busque las perlas magnéticas que están atadas por moléculas individuales de la construcción objetivo. Asegúrese de que haya una cuenta de referencia cerca.
- Gire una cuenta candidata y compruebe que gira libremente. Si la cuenta está atada por múltiples moléculas, exhibe un movimiento restringido.
- Gire el talón durante unas vueltas completas y descubra el radio de rotación (esta función se implementa en el software proporcionado). Preferiblemente, elija una cuenta con un radio de rotación pequeño.
NOTA: Este radio indica cuánto está descentrado el cordón del eje de la correa, que se determina aleatoriamente durante el conjunto de la correa30,31. En todos los experimentos, el descentrado mínimo de una cuenta alivia muchos artefactos asociados con la alta relación de extensión de radio de cuenta a correa que utilizamos. - Aumente la fuerza de 0 a 5 pN para identificar buenas cuentas de un solo atadura. Busque un gran cambio en el patrón de difracción de una cuenta como resultado del estiramiento de una correa de 1 kbp (o los dos mangos equivalentes de 510 pb). Si el patrón de difracción no cambia significativamente, reduzca la fuerza a cero y busque otra cuenta candidata.
NOTA: El levantamiento de ~ 300 nm de una cuenta se puede notar fácilmente a partir de las imágenes sin procesar sin iniciar realmente el proceso de seguimiento.
9. Seguimiento de cuentas para mediciones de extensión
NOTA: El seguimiento de las cuentas se realiza mediante el análisis de imágenes de cuentas en tiempo real en el software LabVIEW provisto con este artículo. El método de seguimiento y sus variantes se han utilizado en la mayoría de los sistemas convencionales de MT y se explican en la literatura previa 2,5,7,26. Al medir la posición de un cordón magnético en relación con un cordón de referencia fijo (es decir, seguimiento diferencial), las mediciones de posición se vuelven extremadamente robustas a una perturbación externa.
- Una vez que se encuentre una cuenta magnética adecuada junto con una cuenta de referencia, haga clic en el botón Calibrar para comenzar a prepararse para el seguimiento de cuentas.
- Haga clic en las cuentas de la imagen para definir las ubicaciones de las cuentas. Luego, las imágenes se recortarán a regiones de interés (ROI) (por ejemplo, 150 x 150 píxeles para una cuenta de 3 μm) alrededor de las cuentas y luego se analizarán más a fondo para extraer las coordenadas precisas de las cuentas.
- Espere a que se complete la rotación del imán. Este proceso registra las coordenadas x e y de la cuenta (calculando la correlación cruzada 2D44 o utilizando la simetría radial45 de las imágenes de la cuenta, con un rendimiento comparable) mientras gira los imanes para documentar la fijación descentrada de la cuenta31.
- Para el seguimiento en la dirección z, espere a que el software genere una tabla de búsqueda de imágenes de difracción de las cuentas a diferentes distancias del plano focal. Esto se lleva a cabo pisando la lente del objetivo con un escáner piezoeléctrico en pasos equidistantes y registrando imágenes de cuentas promediadas por fluctuación en cada posición. Luego, las coordenadas z de las cuentas en experimentos reales se determinan comparando las imágenes de cuentas en tiempo real con la tabla de búsqueda con interpolación7.
- Cuando finalice la generación de la tabla de búsqueda, habilite el seguimiento y el enfoque automático (presione Track ? y AF? Botones) y haga clic en el botón Adquirir para comenzar a registrar las posiciones de las cuentas.
NOTA: El enfoque automático es opcional, pero se recomienda corregir la deriva de la etapa en z durante la adquisición.
10. Esquemas de aplicación de fuerza
- Experimentos de rampa de fuerza: Para verificar la relación fuerza-extensión del constructo, aplique una rampa de fuerza hacia arriba y hacia abajo a una velocidad de carga constante (± 1.0 pN s−1) (Figura 4A). Por ejemplo, aplique tres rondas de un ciclo de 0-20-0 pN para verificar la longitud total de la construcción y la curva de extensión de fuerza de los mangos.
- Al especificar los parámetros de amarre en el software, superponga una curva de extensión de fuerza WLC sobre los datos medidos y determine si el cordón objetivo está atado por una construcción de muestra genuina con los mangos de ADN adecuados. Utilice la longitud de contorno conocida (por ejemplo, ~340 nm para dsDNA de 1 kbp) y la longitud de persistencia WLC (30-45 nm para dsDNA31 corto) del constructo como punto de partida. Aplique el método de corrección de extensión descrito en el paso 2.11 si es necesario.
- Si se verifica la construcción, examine la respuesta de fuerza-extensión en detalle para buscar una extensión adicional resultante de las moléculas objetivo-horquillas o complejos SNARE.
- Experimentos de fuerza constante: Varíe gradualmente la fuerza aplicada en pasos discretos para sondear la sensibilidad a la fuerza de las moléculas objetivo (Figura 4B).
NOTA: Los MT permiten experimentos de fuerza constante simples y efectivos porque la fuerza aplicada se mantiene constante cuando los imanes se mantienen quietos.- Para horquillas de ADN, aplique 4-8 pN de fuerza con pasos de 0.2-0.5 pN y mida la posición del talón para ~ 10 s en cada nivel de fuerza.
- Para complejos SNARE, aplique 14-16 pN de fuerza con pasos de 0.1-0.2 pN y mida la posición del talón para ~ 10 s en cada nivel de fuerza.
- Experimentos de salto de fuerza: Observe los eventos de transición de los complejos SNARE.
NOTA: Los experimentos de salto de fuerza, como los experimentos de fuerza constante, implican cambios en los niveles de fuerza. Sin embargo, los saltos de fuerza emplean cambios más abruptos en la fuerza aplicada, lo que permite el monitoreo de eventos desencadenados por la fuerza en las moléculas probadas, como una ruptura repentina de complejos de proteínas. Por ejemplo, dado que los complejos SNARE exhiben histéresis estructural en el ciclo de fuerza23, es informativo realizar experimentos de salto de fuerza y medir la latencia a la transición (Figura 4C).- Descomprimir: Despegamiento de una molécula VAMP2 de un complejo SNARE ternario intacto, dejando un complejo binario de sintaxina-1A y SNAP-25.
- Recomprimir: Compresión de la molécula VAMP2 descomprimida para regenerar un complejo SNARE intacto.
- Despliegue: Desmontaje completo de un complejo SNARE acompañado de disociación completa de SNAP-25. Solo VAMP2 y las moléculas de sintaxina permanecen en la construcción después del despliegue.
- Repliego: Regeneración de un complejo SNARE tras la unión de una molécula SNAP-25 libre desde el tampón.
- A 2 pN, inducir el ensamblaje de un complejo SNARE intacto esperando (~30 s) la asociación de una molécula SNAP25 libre. Se observa una disminución repentina en la extensión tras la formación de un complejo SNARE.
- Para observar eventos de descompresión, espere unos segundos a 10-12 pN, y luego muévase a 14-15 pN abruptamente con la velocidad máxima posible del motor. Dependiendo de la fuerza objetivo, el complejo SNARE exhibirá una transición reversible entre intermedios parcialmente descomprimidos (como en experimentos de fuerza constante) o un salto de ~ 25 nm a un estado más alto y descomprimido después de un tiempo de espera aleatorio (o latencia).
- Para observar los eventos de recompresión, baje la fuerza a 10-12 pN inmediatamente después de observar la descompresión. Una vez más, el complejo SNARE exhibe una transición estocástica al estado inferior con cremallera después de cierta latencia aleatoria. Si se ha producido el despliegue después de la descompresión, el complejo no se volverá a comprimir, ya que faltará una molécula SNAP-25.
- Para observar los eventos que se desarrollan, espere un período más largo después de que se observe la descompresión para detectar un aumento adicional en la extensión (~ 2 nm).
11. Análisis de datos
NOTA: Los tipos de análisis que se pueden realizar con datos de MT dependen del sistema de destino. Sin embargo, existen enfoques comunes para extraer información útil de los experimentos respectivos descritos en la Figura 4. Todos los análisis se realizan con MATLAB (R2021a) utilizando los códigos personalizados proporcionados en este artículo. Estos códigos generan gráficos utilizando los mismos datos presentados en este artículo. Tenga en cuenta que, si bien los datos sin procesar del seguimiento de 100 Hz se tomaron directamente para el análisis, los datos del seguimiento de 1,2 kHz generalmente se filtraron con la mediana (con una ventana deslizante de cinco puntos) antes del análisis para reducir el ruido (excepto para el análisis de ruido).
- Experimentos de rampa de fuerza: Analizar la relación fuerza-extensión (por ejemplo, elasticidad de polímeros) y hacer la transición de la fuerza para extraer información sobre propiedades nanomecánicas.
- Experimentos de fuerza constante: Analizar las poblaciones estatales y el tiempo de permanencia (o tasa de transición) en función de la fuerza para extraer parámetros estructurales (por ejemplo, regiones involucradas en la transición), termodinámicas (por ejemplo, diferencia de energía libre) y cinética (por ejemplo, barrera de energía) de los cambios conformacionales.
- Experimentos de salto de fuerza: Analizar la cinética de ruptura (por ejemplo, las interacciones proteína-proteína y la unión receptor-ligando) o la vida útil de los intermediarios transitorios (por ejemplo, el despliegue de biomoléculas) para extraer la estabilidad de las moléculas objetivo y sus estados.
- Como aplicaciones representativas, analice los datos de muestra para horquillas de ADN y complejos SNARE:
- Transiciones de dos estados de una horquilla de ADN: fuerza de descompresión, distancia de apertura, dependencia de la fuerza del cambio de población y asignación de estado y mediciones de tasa de transición con un modelo oculto de Markov (HMM) (códigos MATLAB proporcionados).
- Cambios conformacionales de los complejos SNARE: fuerza de descompresión, dependencia de la fuerza de los estados intermedios y latencia de descompresión, histéresis en la recompresión y comportamiento de despliegue / replegado.
NOTA: Los modelos de extensión de fuerza para mangos de ADN, horquillas de ADN y conformaciones complejas SNARE se dan en referencias anteriores14,31.
Resultados
Calibración de fuerza
Los resultados de los dos métodos de medición de fuerza (varianza de desplazamiento lateral de las perlas y análisis del espectro de potencia) difirieron en 0-2 pN (Figura 2G). De acuerdo con los resultados de la Figura 2F, podemos alcanzar de manera confiable hasta 30 pN con imanes de neodimio regulares.
Transiciones de dos estados de una horquilla de ADN de 8 pb
Prime...
Discusión
En este trabajo, introdujimos una configuración de espectroscopia de fuerza de molécula única que puede observar cambios estructurales de biomoléculas con alta precisión espaciotemporal. La cámara CMOS de alta velocidad utilizada adquiere 1.200 fotogramas s−1 a una resolución de 1.280 x 1.024, lo que permite el seguimiento de cuentas de 1,2 kHz. Sin embargo, la velocidad de las mediciones está actualmente limitada por el software de seguimiento de cuentas, por lo que el ROI generalmente se reduce a ?...
Divulgaciones
Los autores no tienen conflictos de intereses que declarar.
Agradecimientos
Este trabajo fue apoyado por la subvención de la Fundación Nacional de Investigación de Corea (NRF) financiada por el gobierno coreano (MSIT) (NRF-2022R1C1C1012176, NRF-2021R1A4A1031754 y NRF- 2021R1A6A1A10042944). S.-H.R. fue apoyado por la subvención NRF (2021R1C1C2009717).
Materiales
| Name | Company | Catalog Number | Comments |
| Materials for construct synthesis | |||
| Agarose gel electrophoresis system | Advance | Mupid-2plus | |
| DNA ladder | Bioneer | D-1037 | |
| nTaq polymerase | Enzynomics | P050A | |
| PCR purification kit | LaboPass | CMR0112 | |
| PEGylated SMCC crosslinker / SM(PEG)2 | ThermoFisher Scientific | 22102 | For SNARE–DNA coupling |
| Primer B | Bioneer | 5'-Biotin/TCGCCACCATCATTTCCA-3' | For 5-kbp force calibration construct and DNA handles |
| Primer B_hp | IDT | 5'-Biotin/TTTTTTTTTTGTTCTCTATTT TTTTAGAGAAC /AP site/ /AP site/ TCGCCACCATCATTTCCA-3' | For hairpin construct |
| Primer N | Bioneer | 5'-C6Amine/CATGTGGGTGACGCGAAA-3' | For DNA handles |
| Primer Z | Bioneer | 5'-Azide/TCGCCACCATCATTTCCA-3' | For DNA handles |
| Primer Z_5k | Bioneer | 5'-Azide/TTAGAGAGTATGGGTATATGACA TCG-3' | For 5-kbp force calibration construct |
| Primer Z_hp | Bioneer | 5'-Azide/GTGGCAGCATGACACC-3' | For hairpin construct |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| λ-DNA | Bioneer | D-2510 | Template strand for PCR |
| DNA sequences for SNARE proteins | |||
| 6×His-tagged SNAP-25b (2-206; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctat gagaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagcgg aagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCACA GCAGCGGCCTGGTGCCGCGC GGCAGCCATACTAGCGGAGAT ATCGCCGAGGACGCAGACAT GCGCAATGAGCTGGAGGAGA TGCAGAGGAGGGCTGACCAG CTGGCTGATGAGTCCCTGGA AAGCACCCGTCGCATGCTGC AGCTGGTTGAAGAGAGTAAA GATGCTGGCATCAGGACTTT GGTTATGTTGGATGAGCAAG GCGAACAACTGGAACGCATT GAGGAAGGGATGGACCAAAT CAATAAGGACATGAAAGAAG CAGAAAAGAATTTGACGGAC CTAGGAAAATTCGCCGGCCT TGCCGTGGCCCCCGCCAAC AAGCTTAAATCCAGTGATGC TTACAAAAAAGCCTGGGGC AATAATCAGGATGGAGTAGT GGCCAGCCAGCCTGCCCG TGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTG GCTTCATCCGCAGGGTAAC AAATGATGCCCGGGAAAAT GAGATGGATGAGAACCTG GAGCAGGTGAGCGGCATC ATCGGAAACCTCCGCCAC ATGGCTCTAGACATGGGCA ATGAGATTGACACCCAGA ATCGCCAGATCGACAGGA TCATGGAGAAGGCTGATT CCAACAAAACCAGAATTG ATGAAGCCAACCAACGTG CAACAAAGATGCTGGGAA GTGGTTAAggatccgaattcgag ctccgtcgacaagcttgcggccgcactc gagcaccaccaccaccaccactgagat ccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgag caataactagcataaccccttggggcct ctaaacgggtcttgaggggttttttgctga aaggaggaactatatccggat | |
| 6×His-tagged VAMP2 (2-97, L32C/I97C; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCAC AGCAGCGGCCTGGTGCCGC GCGGCAGCCATATGGCAGAT CTCTCGGCTACCGCTGCCAC CGTCCCGCCTGCCGCCCCG GCCGGCGAGGGTGGCCCCC CTGCACCTCCTCCAAATCTTA CCAGTAACAGGAGATGCCAG CAGACCCAGGCCCAGGTGG ATGAGGTGGTGGACATCATG AGGGTGAATGTGGACAAGGT CCTGGAGCGAGACCAGAAG CTATCGGAACTGGATGATCG CGCAGATGCCCTCCAGGCA GGGGCCTCCCAGTTTGAAA CAAGTGCAGCCAAGCTCAA GCGCAAATACTGGTGGAAA AACCTCAAGATGATGTGCTA Aggatccgaattcgagctccgtcg acaagcttgcggccgcactcgagcaccacca ccaccaccactgagatccggctgctaacaaa gcccgaaaggaagctgagttggctgctgcca ccgctgagcaataactagcataaccccttgg ggcctctaaacgggtcttgaggggttttttg ctgaaaggaggaactatatccggat | |
| 6×His-tagged ΔN-VAMP2 (49–96; capitalized) and Syntaxin-1A (191–267, I202C/I266C; capitalized) in pETDuet-1 | homemade | ggggaattgtgagcggataacaattcccctc tagaaataattttgtttaactttaagaagga gatataccATGGGCAGCAGCCATCA TCATCATCATCACAGCAGCGG CCTGGAAGTTCTGTTCCAGGG GCCCGGTAATGTGGACAAGGT CCTGGAGCGAGACCAGAAGCT ATCGGAACTGGATGATCGCGC AGATGCCCTCCAGGCAGGGGC CTCCCAGTTTGAAACAAGTGC AGCCAAGCTCAAGCGCAAATAC TGGTGGAAAAACCTCAAGATGAT GTAAgcggccgcataatgcttaagtcgaaca gaaagtaatcgtattgtacacggccgcataa tcgaaattaatacgactcactataggggaat tgtgagcggataacaattccccatcttagta tattagttaagtataagaaggagatatacat ATGGCCCTCAGTGAGATCGAGA CCAGGCACAGTGAGTGCATC AAGTTGGAGAACAGCATCCG GGAGCTACACGATATGTTCAT GGACATGGCCATGCTGGTGG AGAGCCAGGGGGAGATGATT GACAGGATCGAGTACAATGTG GAACACGCTGTGGACTACGTG GAGAGGGCCGTGTCTGACACC AAGAAGGCCGTCAAGTACCAG AGCAAGGCACGCAGGAAGAA GTGCATGATCTAActcgagtc tggtaaagaaaccgctgctgcgaaatttgaa cgccagcacatggactcgtctactagcgcag cttaattaacctaggctgctgccaccgctga gcaataactagcataaccccttggggcctct aaacgggtcttgaggggttttttgctgaaag gaggaactatatccggattggcgaatgggac gcgccctgtagcggcgcattaagcgcggcgg gtgtggtggttacgcgcagcgtgaccgctac acttgccagcgccctagcgcccgctcctttc gctttcttcccttcctttctcgccacgttcg ccggctttccccgtcaagctctaaatcgggg gctccctttagggttccgatttagtgcttta cggcacctcgaccccaaaaaacttgattagg gtgatggttcacgtagtgggccatcgccctg atagacggtttttcgccctttgacgttggag tccacgttctttaatagtggactcttgttcc aaactggaacaacactcaaccctatctcggt ctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctga tttaacaaaaatttaacgcgaattttaacaa aatattaacgtttacaatttctggcggcacg atggcatgagattatcaaaaaggatcttcac ctagatccttttaaattaaaaatgaagtttt aaatcaatctaaagtatatatgagtaaactt ggtctgacagttaccaatgcttaatcagtga ggcacctatctcagcgatctgtctatttcgt tcatccatagttgcctgactccccgtcgtgt agataactacgatacgggagggcttaccatc tggccccagtgctgcaatgataccgcgagac ccacgctcaccggctccagatttatcagcaa taaaccagccagccggaagggccgagcgca gaagtggtcctgcaactttatccgcctccatc cagtctattaattgttgccgggaagctagag taagtagttcgccagttaatagtttgcgcaa cgttgttgccattgctacaggcatcgtggtg tcacgctcgtcgtttggtatggcttcattca gctccggttcccaacgatcaaggcgagttac atgatcccccatgttgtgcaaaaaagcggtt agctccttcggtcctccgatcgttgtcagaa gtaagttggccgcagtgttatcactcatggt tatggcagcactgcataattctcttactgtc atgccatccgtaagatgcttttctgtgactg gtgagtactcaaccaagtcattctgagaata gtgtatgcggcgaccgagttgctcttgcccg gcgtcaatacgggataataccgcgccacata gcagaactttaaaagtgctcatcattggaaa acgttcttcggggcgaaaactctcaaggatc ttaccgctgttgagatccagttcgatgtaac ccactcgtgcacccaactgatcttcagcatc ttttactttcaccagcgtttctgggtgagcaaa aacaggaaggcaaaatgccgcaaaaaagg gaataagggcgacacggaaatgttgaatact catactcttcctttttcaatcatgattgaag catttatcagggttattgtctcatgagcgga tacatatttgaatgtatttagaaaaataaac aaataggtcatgaccaaaatcccttaacgtg agttttcgttccactgagcgtcagaccccgt agaaaagatcaaaggatcttcttgagatcct ttttttctgcgcgtaatctgctgcttgcaaa caaaaaaaccaccgctaccagcggtggtttg tttgccggatcaagagctaccaactcttttt ccgaaggtaactggcttcagcagagcgcaga taccaaatactgtccttctagtgtagccgta gttaggccaccacttcaagaactctgtagca ccgcctacatacctcgctctgctaatcctgt taccagtggctgctgccagtggcgataagtc gtgtcttaccgggttggactcaagacgatag ttaccggataaggcgcagcggtcgggctgaa cggggggttcgtgcacacagcccagcttgga gcgaacgacctacaccgaactgagataccta cagcgtgagctatgagaaagcgccacgcttccc gaagggagaaaggcggacaggtatccggta agcggcagggtcggaacaggagagcgcac gagggagcttccagggggaaacgcctggtatc tttatagtcctgtcgggtttcgccacctctg acttgagcgtcgatttttgtgatgctcgtca ggggggcggagcctatggaaaaacgccagc aacgcggcctttttacggttcctggccttttg ctggccttttgctcacatgttctttcctgcg ttatcccctgattctgtggataaccgtatta ccgcctttgagtgagctgataccgctcgccgc agccgaacgaccgagcgcagcgagtcagtg agcgaggaagcggaagagcgcctgatgcgg tattttctccttacgcatctgtgcggtatttc acaccgcatatatggtgcactctcagtacaa tctgctctgatgccgcatagttaagccagta tacactccgctatcgctacgtgactgggtca tggctgcgccccgacacccgccaacacccgc tgacgcgccctgacgggcttgtctgctcccg gcatccgcttacagacaagctgtgaccgtct ccgggagctgcatgtgtcagaggttttcacc gtcatcaccgaaacgcgcgaggcagctgcgg taaagctcatcagcgtggtcgtgaagcgatt cacagatgtctgcctgttcatccgcgtccag ctcgttgagtttctccagaagcgttaatgtc tggcttctgataaagcgggccatgttaaggg cggttttttcctgtttggtcactgatgcctc cgtgtaagggggatttctgttcatgggggta atgataccgatgaaacgagagaggatgctca cgatacgggttactgatgatgaacatgcccg gttactggaacgttgtgagggtaaacaactg gcggtatggatgcggcgggaccagagaaaaa tcactcagggtcaatgccagcgcttcgttaa tacagatgtaggtgttccacagggtagccag cagcatcctgcgatgcagatccggaacataa tggtgcagggcgctgacttccgcgtttccag actttacgaaacacggaaaccgaagaccatt catgttgttgctcaggtcgcagacgttttgc agcagcagtcgcttcacgttcgctcgcgtat cggtgattcattctgctaaccagtaaggcaa ccccgccagcctagccgggtcctcaacgaca ggagcacgatcatgctagtcatgccccgcgc ccaccggaaggagctgactgggttgaaggct ctcaagggcatcggtcgagatcccggtgcct aatgagtgagctaacttacattaattgcgtt gcgctcactgcccgctttccagtcgggaaac ctgtcgtgccagctgcattaatgaatcggcc aacgcgcggggagaggcggtttgcgtattgg gcgccagggtggtttttcttttcaccagtga gacgggcaacagctgattgcccttcaccgcc tggccctgagagagttgcagcaagcggtcca cgctggtttgccccagcaggcgaaaatcctg tttgatggtggttaacggcgggatataacat gagctgtcttcggtatcgtcgtatcccacta ccgagatgtccgcaccaacgcgcagcccgga ctcggtaatggcgcgcattgcgcccagcgcc atctgatcgttggcaaccagcatcgcagtgg gaacgatgccctcattcagcatttgcatggt ttgttgaaaaccggacatggcactccagtcg ccttcccgttccgctatcggctgaatttgat tgcgagtgagatatttatgccagccagccag acgcagacgcgccgagacagaacttaatggg cccgctaacagcgcgatttgctggtgaccca atgcgaccagatgctccacgcccagtcgcgt accgtcttcatgggagaaaataatactgttg atgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccac agcaatggcatcctggtcatccagcggatag ttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttc gacgccgcttcgttctaccatcgacaccacc acgctggcacccagttgatcggcgcgagatt taatcgccgcgacaatttgcgacggcgcgtg cagggccagactggaggtggcaacgccaatc agcaacgactgtttgcccgccagttgttgtg ccacgcggttgggaatgtaattcagctccgc catcgccgcttccactttttcccgcgttttc gcagaaacgtggctggcctggttcaccacgc gggaaacggtctgataagagacaccggcata ctctgcgacatcgtataacgttactggtttc acattcaccaccctgaattgactctcttccg ggcgctatcatgccataccgcgaaaggtttt gcgccattcgatggtgtccgggatctcgacg ctctcccttatgcgactcctgcattaggaag cagcccagtagtaggttgaggccgttgagca ccgccgccgcaaggaatggtgcatgcaagga gatggcgcccaacagtcccccggccacgggg cctgccaccatacccacgccgaaacaagcgc tcatgagcccgaagtggcgagcccgatcttc cccatcggtgatgtcggcgatataggcgcca gcaaccgcacctgtggcgccggtgatgccgg ccacgatgcgtccggcgtagaggatcgagat cgatctcgatcccgcgaaattaatacgactc actata | |
| SNAP-25b (1–206, all C to A; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcc agggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGCCGA GGACGCAGACATGCGCAATG AGCTGGAGGAGATGCAGAGG AGGGCTGACCAGCTGGCTGA TGAGTCCCTGGAAAGCACCC GTCGCATGCTGCAGCTGGTT GAAGAGAGTAAAGATGCTGG CATCAGGACTTTGGTTATGTT GGATGAGCAAGGCGAACAAC TGGAACGCATTGAGGAAGGG ATGGACCAAATCAATAAGGAC ATGAAAGAAGCAGAAAAGAAT TTGACGGACCTAGGAAAATTC GCCGGCCTTGCCGTGGCCCC CGCCAACAAGCTTAAATCCAG TGATGCTTACAAAAAAGCCTG GGGCAATAATCAGGATGGAGT AGTGGCCAGCCAGCCTGCCC GTGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTGGC TTCATCCGCAGGGTAACAAAT GATGCCCGGGAAAATGAGATG GATGAGAACCTGGAGCAGGT GAGCGGCATCATCGGAAACCT CCGCCACATGGCTCTAGACAT GGGCAATGAGATTGACACCCA GAATCGCCAGATCGACAGGAT CATGGAGAAGGCTGATTCCAA CAAAACCAGAATTGATGAAGC CAACCAACGTGCAACAAAGAT GCTGGGAAGTGGTTAA ctcgagcaccaccaccaccaccactgag atccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgagc aataactagcataaccccttggggcctc taaacgggtcttgaggggttttttgctgaa aggaggaactatatccggat | |
| Materials for protein purificaiton | |||
| 2-Mercaptoethanol | SIGMA | M3148-25ML | |
| Agar | LPS Solution | AGA500 | |
| Ampicillin, Sodium salt | PLS | AC1043-005-00 | |
| Chloramphenicol | PLS | CR1023-050-00 | |
| Competent cells (E. coli) | Novagen | 70956 | Rosetta(DE3)pLysS |
| Glycerol | SIGMA | G5516-500ML | |
| HEPES | SIGMA | H4034-100G | |
| Hydrochloric acid / HCl | SIGMA | 320331-500ML | |
| Imidazole | SIGMA | I2399-100G | |
| Isopropyl β-D-1-thiogalactopyranoside / IPTG | SIGMA | 10724815001 | |
| Kanamycin Sulfate | PLS | KC1001-005-02 | |
| Luria-Bertani (LB) Broth | LPS Solution | LB-05 | |
| Ni-NTA resin | Qiagen | 30210 | |
| PD MiniTrap G-25 (desalting column) | Cytiva | GE28-9180-07 | For instructions, see: https://www.cytivalifesciences.com/en/us/shop/chromatography/prepacked-columns/desalting-and-buffer-exchange/pd-minitrap-desalting-columns-with-sephadex-g-25-resin-p-06174 |
| Phenylmethylsulfonyl fluoride / PMSF | ThermoFisher Scientific | 36978 | |
| Plasmids for SNARE proteins | cloned in house | N/A | Available upon request |
| Protease inhibitor cocktail | genDEPOT | P3100 | |
| Sodium chloride | SIGMA | S5886-500G | |
| Sodium phosphate dibasic / Na2HPO4 | SIGMA | S7907-100G | |
| Sodium phosphate monobasic / NaH2PO4 | SIGMA | S3139-250G | |
| Tris(2-carboxyethyl)phosphine / TCEP | SIGMA | C4706-2G | |
| Trizma base | SIGMA | T1503-250G | |
| Materials for sample assembly | |||
| Biotin-PEG-SVA | LAYSAN BIO | BIO-PEG-SVA-5K-100MG & MPEG-SVA-5K-1g | For PEGylation |
| Dibenzocyclooctyne-amine / DBCO-NH2 | SIGMA | 761540-10MG | For bead coating |
| Double-sided tape | 3M | 136 | For flow cell assembly |
| Epoxy glue | DEVCON | S-208 | For flow cell assembly |
| Glass coverslip for bottom surface | VWR | 48393-251 | Rectangular, 60×24 mm, #1.5 |
| Glass coverslip for top surface | VWR | 48393-241 | Rectangular, 50×24 mm, #1.5 |
| Magnetic bead | ThermoFisher Scientific | 14301 | Dynabeads M-270 Epoxy, 2.8 μm |
| mPEG-SVA | LAYSAN BIO | mPEG-SVA 1g | For PEGylation |
| N,N-Dimethylformamide / DMF | SIGMA | D4551-250ML | For bead coating |
| N-[3-(trimethoxysilyl)propyl]ethylenediamine | SIGMA | 104884-100ML | For PEGylation |
| Neutravidin | ThermoFisher Scientific | 31000 | For sample tethering |
| Phosphate buffered saline / PBS, pH 7.2 | PLS | PR2007-100-00 | |
| Plastic syringe | Norm-ject | A5 | 5 ml, luer tip |
| Polyethylene Tubing | SCI | BB31695-PE/4 | PE-60 |
| Reference bead | SPHEROTECH | SVP-30-5 | Streptavidin-coated Polystyrene Particles; 3.0-3.4 µm |
| Syringe needle | Kovax | 21G-1 1/4'' | 21 G |
| Syringe pump | KD SCIENTIFIC | 788210 | |
| Equipment for magnetic tweezer instrument | |||
| 1-axis motorized microtranslation stage | PI | M-126.PD1 | For vertical positioning of magnets |
| 2-axis manual translation stage | ST1 | LEE400 | For alignment of magnets to the optical axis |
| Acrylic holder for magnets | DaiKwang Precision | custum order | Drawing available upon request |
| Frame grabber | Active Silicon | AS-FBD-4XCXP6-2PE8 | |
| High-speed CMOS camera | Mikrotron | EoSens 3CXP | |
| Inverted microscope | Olympus | IX73P2F-1-2 | |
| Neodymium magnets | LG magnet | ND 10x10x12t | Dimension: 10 mm × 10 mm × 12 mm; two needed |
| Objective lens | Olympus | UPLXAPO100XO | Oil-immersion, NA 1.45 |
| Objective lens nanopositioner | Mad City Labs | Nano-F100S | |
| Rotation stepper motor | AUTONICS | A3K-S545W | For rotating magnets |
| Superluminescent diode | QPHOTONICS | QSDM-680-2 | 680 nm |
| Software | |||
| LabVIEW | National Instruments | v20.0f1 | |
| MATLAB | MathWorks | v2021a |
Referencias
- Le, S., Liu, R., Lim, C. T., Yan, J. Uncovering mechanosensing mechanisms at the single protein level using magnetic tweezers. Methods. 94, 13-18 (2016).
- Choi, H. -. K., Kim, H. G., Shon, M. J., Yoon, T. -. Y. High-resolution single-molecule magnetic tweezers. Annual Review of Biochemistry. 91 (1), 33-59 (2022).
- Yang, T., Park, C., Rah, S. -. H., Shon, M. J. Nano-precision tweezers for mechanosensitive proteins and beyond. Molecules and Cells. 45 (1), 16-25 (2022).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- De Vlaminck, I., Dekker, C. Recent advances in magnetic tweezers. Annual Review of Biophysics. 41 (1), 453-472 (2012).
- Bustamante, C. J., Chemla, Y. R., Liu, S., Wang, M. D. Optical tweezers in single-molecule biophysics. Nature Reviews Methods Primers. 1, 25 (2021).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical Journal. 82 (6), 3314-3329 (2002).
- Smith, S. B., Finzi, L., Bustamante, C. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258 (5085), 1122-1126 (1992).
- Lansdorp, B. M., Tabrizi, S. J., Dittmore, A., Saleh, O. A. A high-speed magnetic tweezer beyond 10,000 frames per second. Review of Scientific Instruments. 84 (4), 044301 (2013).
- Cnossen, J. P., Dulin, D., Dekker, N. H. An optimized software framework for real-time, high-throughput tracking of spherical beads. Review of Scientific Instruments. 85 (10), 103712 (2014).
- Dulin, D., et al. High spatiotemporal-resolution magnetic tweezers: calibration and applications for DNA dynamics. Biophysical Journal. 109 (10), 2113-2125 (2015).
- Huhle, A., et al. Camera-based three-dimensional real-time particle tracking at kHz rates and Ångström accuracy. Nature Communications. 6 (1), 5885 (2015).
- Popa, I., et al. A HaloTag anchored ruler for week-long studies of protein dynamics. Journal of the American Chemical Society. 138 (33), 10546-10553 (2016).
- Shon, M. J., Kim, H., Yoon, T. -. Y. Focused clamping of a single neuronal SNARE complex by complexin under high mechanical tension. Nature Communications. 9 (1), 3639 (2018).
- Tapia-Rojo, R., Eckels, E. C., Fernández, J. M. Ephemeral states in protein folding under force captured with a magnetic tweezers design. Proceedings of the National Academy of Sciences. 116 (16), 7873-7878 (2019).
- Löf, A., et al. Multiplexed protein force spectroscopy reveals equilibrium protein folding dynamics and the low-force response of von Willebrand factor. Proceedings of the National Academy of Sciences. 116 (38), 18798-18807 (2019).
- Tapia-Rojo, R., Alonso-Caballero, A., Fernandez, J. M. Direct observation of a coil-to-helix contraction triggered by vinculin binding to talin. Science Advances. 6 (21), (2020).
- Rieu, M., et al. Parallel, linear, and subnanometric 3D tracking of microparticles with Stereo Darkfield Interferometry. Science Advances. 7 (6), (2021).
- Rieu, M., Valle-Orero, J., Ducos, B., Allemand, J. -. F., Croquette, V. Single-molecule kinetic locking allows fluorescence-free quantification of protein/nucleic-acid binding. Communications Biology. 4 (1), 1083 (2021).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences. 103 (16), 6190-6195 (2006).
- Camunas-Soler, J., Ribezzi-Crivellari, M., Ritort, F. Elastic properties of nucleic acids by single-molecule force spectroscopy. Annual Review of Biophysics. 45 (1), 65-84 (2016).
- Südhof, T. C., Rothman, J. E. Membrane fusion: grappling with SNARE and SM proteins. Science. 323 (5913), 474-477 (2009).
- Gao, Y., et al. Single reconstituted neuronal SNARE complexes zipper in three distinct stages. Science. 337 (6100), 1340-1343 (2012).
- Zorman, S., et al. Common intermediates and kinetics, but different energetics, in the assembly of SNARE proteins. eLife. 3, e03348 (2014).
- Zhang, Y., Hughson, F. M. Chaperoning SNARE folding and assembly. Annual Review of Biochemistry. 90 (1), 581-603 (2021).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H. Magnetic tweezers for single-molecule experiments. Handbook of Single-Molecule Biophysics. , 371-395 (2009).
- You, H., Le, S., Chen, H., Qin, L., Yan, J. Single-molecule manipulation of G-quadruplexes by magnetic tweezers. Journal of Visualized Experiments. (127), e56328 (2017).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical Journal. 96 (12), 5040-5049 (2009).
- Dulin, D., Barland, S., Hachair, X., Pedaci, F. Efficient illumination for microsecond tracking microscopy. PLoS One. 9 (9), e107335 (2014).
- Klaue, D., Seidel, R. Torsional stiffness of single superparamagnetic microspheres in an external magnetic field. Physical Review Letters. 102 (2), 028302 (2009).
- Shon, M. J., Rah, S. -. H., Yoon, T. -. Y. Submicrometer elasticity of double-stranded DNA revealed by precision force-extension measurements with magnetic tweezers. Science Advances. 5 (6), 1697 (2019).
- Czerwinski, F., Richardson, A. C., Oddershede, L. B. Quantifying noise in optical tweezers by Allan variance. Optics Express. 17 (15), 13255-13269 (2009).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. Review of Scientific Instruments. 83 (2), 025115 (2012).
- Ostrofet, E., Papini, F. S., Dulin, D. High spatiotemporal resolution data from a custom magnetic tweezers instrument. Data in Brief. 30, 105397 (2020).
- Yu, Z., et al. A force calibration standard for magnetic tweezers. Review of Scientific Instruments. 85 (12), 123114 (2014).
- Strick, T. R., Allemand, J. -. F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271 (5257), 1835-1837 (1996).
- Daldrop, P., Brutzer, H., Huhle, A., Kauert, D. J., Seidel, R. Extending the range for force calibration in magnetic tweezers. Biophysical Journal. 108 (10), 2550-2561 (2015).
- te Velthuis, A. J. W., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative guidelines for force calibration through spectral analysis of magnetic tweezers data. Biophysical Journal. 99 (4), 1292-1302 (2010).
- Ostrofet, E., Papini, F. S., Dulin, D. Correction-free force calibration for magnetic tweezers experiments. Scientific Reports. 8 (1), 15920 (2018).
- Seol, Y., Li, J., Nelson, P. C., Perkins, T. T., Betterton, M. D. Elasticity of short DNA molecules: theory and experiment for contour lengths of 0.6-7 µm. Biophysical Journal. 93 (12), 4360-4373 (2007).
- Burnham, D. R., Vlaminck, I. D., Henighan, T., Dekker, C. Skewed Brownian fluctuations in single-molecule magnetic tweezers. PLoS One. 9 (9), 108271 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protocols. 3 (1), 101152 (2022).
- Lee, H. -. W., et al. Profiling of protein-protein interactions via single-molecule techniques predicts the dependence of cancers on growth-factor receptors. Nature Biomedical Engineering. 2 (4), 239-253 (2018).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Parthasarathy, R. Rapid, accurate particle tracking by calculation of radial symmetry centers. Nature Methods. 9 (7), 724-726 (2012).
- Woodside, M. T., Block, S. M. Reconstructing folding energy landscapes by single-molecule force spectroscopy. Annual Review of Biophysics. 43 (1), 19-39 (2014).
- Evans, E., Ritchie, K. Dynamic strength of molecular adhesion bonds. Biophysical Journal. 72 (4), 1541-1555 (1997).
- Zhang, Y. Energetics, kinetics, and pathway of SNARE folding and assembly revealed by optical tweezers. Protein Science. 26 (7), 1252-1265 (2017).
- Chen, H., et al. Improved high-force magnetic tweezers for stretching and refolding of proteins and short DNA. Biophysical Journal. 100 (2), 517-523 (2011).
- Cho, S., et al. Tension exerted on cells by magnetic nanoparticles regulates differentiation of human mesenchymal stem cells. Biomaterials Advances. 139, 213028 (2022).
- Shon, M. J., Cohen, A. E. Nano-mechanical measurements of protein-DNA interactions with a silicon nitride pulley. Nucleic Acids Research. 44 (1), 7 (2016).
- Cheng, Y. Single-particle cryo-EM-How did it get here and where will it go. Science. 361 (6405), 876-880 (2018).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Neupane, K., et al. Direct observation of transition paths during the folding of proteins and nucleic acids. Science. 352 (6282), 239-242 (2016).
- Choi, H. -. K., et al. Watching helical membrane proteins fold reveals a common N-to-C-terminal folding pathway. Science. 366 (6469), 1150-1156 (2019).
- Kim, C., et al. Extreme parsimony in ATP consumption by 20S complexes in the global disassembly of single SNARE complexes. Nature Communications. 12 (1), 3206 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados