A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تقنية الإنتاجية العالية في الوقت الفعلي لتحديد تكوين مصائد العدلات خارج الخلية في العدلات البشرية
In This Article
Summary
نقدم طريقة آلية عالية الإنتاجية لتحديد كمية مصائد العدلات خارج الخلية (NETs) باستخدام نظام تحليل الخلايا الحية ، إلى جانب نهج الصبغة المزدوجة المعتمد على نفاذية الغشاء.
Abstract
العدلات هي خلايا سلالة نخاعية تشكل جزءا مهما من جهاز المناعة الفطري. كشف العقد الماضي عن أدوار رئيسية إضافية تلعبها العدلات في التسبب في السرطان وأمراض المناعة الذاتية ومختلف الحالات الالتهابية الحادة والمزمنة من خلال المساهمة في بدء وإدامة عدم التنظيم المناعي من خلال آليات متعددة ، بما في ذلك تشكيل مصائد العدلات خارج الخلية (NETs) ، وهي هياكل حاسمة في الدفاع عن مضادات الميكروبات. أدت القيود المفروضة على تقنيات تحديد تكوين الشبكة بطريقة غير متحيزة وقابلة للتكرار وفعالة إلى تقييد قدرتنا على فهم دور العدلات في الصحة والأمراض. نحن نصف طريقة آلية في الوقت الفعلي وعالية الإنتاجية لتحديد كمية العدلات التي تخضع لتشكيل NET باستخدام منصة تصوير الخلايا الحية إلى جانب نهج الصبغة المزدوجة المعتمد على نفاذية الغشاء باستخدام صبغتين مختلفتين من الحمض النووي لتصوير الحمض النووي داخل الخلايا وخارجها. هذه المنهجية قادرة على المساعدة في تقييم فسيولوجيا العدلات واختبار الجزيئات التي يمكن أن تستهدف تكوين الشبكة.
Introduction
مصائد العدلات خارج الخلية (NETs) هي هياكل كروماتين تشبه الويب يتم بثوقها من العدلات استجابة للمحفزات الالتهابية المختلفة. تتكون الشبكات من الحمض النووي والهستونات والعديد من البروتينات / الببتيدات المضادة للميكروبات ، والتي تحبس وتقتل مسببات الأمراض المعدية وتستدعي الاستجابات الالتهابية1.
في حين أن الشبكات مفيدة للدفاع المضيف ضد مسببات الأمراض ، فقد جذبت الانتباه كمحرك محتمل لمختلف أمراض المناعة الذاتية2 ، والتخثر3 ، وأمراض التمثيل الغذائي4 ، والنمو النقيلي للسرطانات5. على هذا النحو ، فإن تثبيط تكوين NET هو خيار علاجي محتمل لهذه الأمراض. ومع ذلك ، على الرغم من بعض الجزيئات الواعدة التي تستهدف الشبكات قيد التطوير6 ، لا يوجد حتى الآن علاج معتمد يؤثر بشكل خاص على هذه الآلية. ويعزى ذلك، جزئيا على الأقل، إلى عدم وجود طرق موضوعية وغير متحيزة وقابلة للتكرار وعالية الإنتاجية لتشكيل الشبكة.
لقد أنشأنا وأبلغنا عن طريقة جديدة باستخدام منصة تصوير الخلايا الحية ثنائية اللون 7,8. يتم تحليل صور الفاصل الزمني للعدلات الملطخة بصبغة نووية منفذة للغشاء وصبغة الحمض النووي غير المنفذة للغشاء بواسطة البرنامج ، ويتم حساب أعداد العدلات قبل وبعد تكوين NET في نقاط زمنية متعددة. نظرا لأن سلامة غشاء البلازما تفقد أثناء تكوين NET عن طريق تنظيم تفكيك Lamin B بوساطة PKCα وتفكيك Lamin A/ C بوساطة CDK4 / 6 9 ، فإن العدلات المكونة ل NET ملطخة بصبغة الحمض النووي غير المنفذة للغشاء بينما العدلات السليمة ليست كذلك. تتغلب هذه الطريقة على مشاكل التقنيات التي تم الإبلاغ عنها سابقا لتحديد تكوين الشبكة وتوفر تقديرا صافيا غير متحيز وعالي الإنتاجية وقابل للتكرار ودقيقا بطريقة آلية.
Access restricted. Please log in or start a trial to view this content.
Protocol
تم الحصول على العدلات من البشر الأصحاء بعد تقديم الموافقة المستنيرة بموجب البروتوكول المعتمد من مجلس المراجعة المؤسسية (IRB) التابع للمعاهد الوطنية للصحة (NIH). يتبع البروتوكول إرشادات لجنة أخلاقيات البحوث البشرية التابعة للمعاهد الوطنية للصحة.
1. تلطيخ العدلات وإعداد لوحة الفحص
- أخذ الدم المحيطي بموافقة خطية مستنيرة مناسبة باتباع إرشادات كل معهد وعزل العدلات باستخدام أي طريقة مرغوبة. على سبيل المثال ، طريقة Ficoll-dextran 10,11 هي طريقة شائعة الاستخدام لعزل العدلات من الدم المحيطي البشري.
ملاحظة: على الرغم من أن الطريقة التي تمت مناقشتها هنا تستخدم العدلات البشرية ، إلا أنه يمكن أيضا تحليل العدلات في الفئران باستخدام بروتوكول مماثل. - إعادة تعليق العدلات في معهد روزويل بارك التذكاري (RPMI ؛ انظر جدول المواد) 1640 متوسط عند 2.0 × 106 عدلات / مل ووضع تعليق العدلات في أنبوب طرد مركزي 1.5 مل.
ملاحظة: عادة لا نضيف مصل الأبقار الجنيني (FBS) إلى وسائط الاستزراع لأن الألبومين في FBS يمكن أن يقلل من تكوين NET12 ويمكن للنيوكلياز المستقر للحرارة في FBS أن يتحلل الشبكات13. - أضف صبغة الحمض النووي الحمراء المنفذة للغشاء (انظر جدول المواد) عند 1 ميكرولتر لكل 1.5 مل من معلق العدلات.
- احتضان في درجة حرارة الغرفة لمدة 5 دقائق في الظلام. أجهزة الطرد المركزي في 2500 × غرام لمدة 5 دقائق في درجة حرارة الغرفة وإزالة طاف.
- أعد تعليق العدلات في 1 مل من RPMI ، ثم أجهزة الطرد المركزي عند 2500 × جم لمدة 5 دقائق في درجة حرارة الغرفة وإزالة المادة الطافية. كرر 2x (اغسل إجمالي 3x).
- أعد تعليق العدلات في 1 مل من RPMI وعد باستخدام عداد خلية. تمييع تعليق العدلات إلى 1.5 × 105 العدلات / مل.
- أضف 4 ميكرولتر من صبغة الحمض النووي الخضراء غير المنفذة للغشاء المخفف مسبقا 1: 100 (انظر جدول المواد) لكل 1 مل من معلق العدلات.
- ضع 100 ميكرولتر من معلق العدلات لكل بئر في صفيحة 96 بئرا معالجة بزراعة الأنسجة الصافية (انظر جدول المواد). اضبط كل شرط في ثلاث نسخ.
ملاحظة: لقد أظهرنا سابقا7 أنه لا يوجد فرق في تكوين NET والقياس الكمي بهذه الطريقة إذا تم وضع العدلات في ألواح مع أو بدون طلاء poly-L-lysine. لذلك ، فإن استخدام لوحة معالجة زراعة الأنسجة يكفي لهذه الطريقة. - أضف 100 ميكرولتر من RPMI تحتوي على كواشف تحفيز مع أو بدون مثبط ، أو أي كاشف آخر مهم في الآبار المعنية. استخدم دائما آبار التحكم الإيجابية عن طريق إضافة 500 نانومتر فوربول 12-ميريستات 13-أسيتات (PMA) أو 2.5 ميكرومتر أيونوفور الكالسيوم (A23187) ، تم استخدام مثبط 30 ميكرومتر AKT هنا.
- ضع اللوحة في نظام تحليل الخلايا الحية (انظر جدول المواد) الموجود في حاضنة CO2 بنسبة 5٪.
ملاحظة: قد يظهر التكثيف في الجزء العلوي والسفلي من اللوحة بعد بضع دقائق من هذه الخطوة. هذا قد يمنع التصوير المناسب. امسح التكثيف وأزله جيدا قبل بدء المسح. ضع اللوحة في الجهاز ، وابدأ تشغيل البرنامج ، وأدخل بروتوكول المسح الضوئي ، ثم امسح اللوحة قبل الفحص الأول مباشرة.
2. لوحة المسح الضوئي لتصور العدلات المكونة ل NET
- ابدأ تشغيل البرنامج (انظر جدول المواد للحصول على تفاصيل البرنامج). قم بتشغيل إضافة سفينة بالضغط على + في الزاوية اليسرى العليا.
- اختر المسح الضوئي في الموعد المحدد. اختر جديد.
ملاحظة: بمجرد إنشاء هذا ، قم بتشغيل نفس بروتوكول المسح الضوئي عن طريق اختيار نسخ السابق وتحديد البروتوكول المخزن على الشاشة التالية. - اختر قياسي. اضبط إعدادات المسح الضوئي على النحو التالي: خيارات خلية تلو الأخرى: لا شيء; قنوات الصورة: المرحلة ، الأخضر (وقت الاستحواذ: 200 مللي ثانية) ، الأحمر (وقت الاستحواذ: 400 مللي ثانية) ؛ الهدف: 20x.
- اختر اللوحة المستخدمة من القائمة. حدد موقع الوعاء حيث يتم وضع اللوحة على درج نظام التصوير بالخلايا الحية.
- حدد الآبار التي توجد بها عينات وحدد عدد الصور التي يجب التقاطها لكل بئر. استنادا إلى عدد الصور لكل بئر ، قم بإنشاء مدة مسح تقديرية للوحة. عادة ، 4 صور لكل بئر كافية ؛ ومع ذلك ، قد يختلف هذا اعتمادا على ظروف وتكرار عمليات الفحص.
- أدخل المعلومات من كل بئر (على سبيل المثال ، نوع الخلية والمركب) بالنقر فوق إنشاء خريطة لوحة لتوفير اسم للدراسة.
ملاحظة: يمكن إعداد خريطة اللوحة وحفظها مسبقا باستخدام محرر خريطة اللوحة في البرنامج. يمكن استيراد بيانات خريطة اللوحة المحفوظة أثناء هذه الخطوة. إذا رغبت في ذلك ، يمكن تخطي إدخال معلومات خريطة اللوحة وتنفيذها لاحقا. يمكن أيضا الحصول على البيانات دون إدخال معلومات تخطيط اللوحة. ومع ذلك ، يوصى بإدخال معلومات اللوحة لتسهيل التحليل اللاحق. - في الشاشة التالية ، حدد Basic Analyzer كنوع التحليل ، واختر بروتوكول التحليل من القائمة المنسدلة ، والتي تعرض تعريفات التحليل المستخدمة مسبقا (وليس بروتوكولات الفحص). يمكن ترك الخلط الطيفي لكل من الأخضر والأحمر عند 0.0٪.
ملاحظة: قد يتم تخطي هذه الخطوة، إذا رغبت في ذلك. - جدولة الفحص. للتجربة هنا ، امسح كل 15-20 دقيقة لمدة 8 ساعات . اضبط وقت بدء الفحص عن طريق سحب الشريط الأبيض والرمادي أعلى الشاشة.
ملاحظة: تجنب إجراء المسح الضوئي بشكل متكرر، وإلا فقد لا يعمل الجهاز بشكل جيد بسبب ارتفاع درجة الحرارة. يجب ألا يتجاوز وقت المسح 12 ساعة لكل 24 ساعة. قد ترى تنبيها إذا كان المسح الضوئي متكررا جدا. - تحقق من إعداد المسح الضوئي على الشاشة التالية واضغط على إضافة إلى الجدول لبدء المسح. انتظر حتى يتم مسح المجموعة الأولية من الصور ضوئيا للتأكد مما إذا كان كل شيء يعمل بشكل جيد.
- في بعض الأحيان قد لا تكون الخلايا مركزة بشكل صحيح. في هذه الحالة ، تحقق مما إذا كان التكثيف موجودا (وامسحه وفقا لذلك) ، أو تم ضبط اللوحة بشكل صحيح على الدرج ، أو تم تحديد موضع اللوحة بشكل صحيح ، إلخ. إذا كانت الخلايا لا تزال عائمة ولم تستقر في قاع البئر ، فانتظر حوالي 5 دقائق قبل المسح.
3. وضع تعريف التحليل لتحديد الشبكات
- افتح الدراسة (السفينة) المراد تحليلها في علامة التبويب عرض. اضغط على تحليل التشغيل على يسار الشاشة. اختر إنشاء تعريف تحليل جديد.
- إذا تم إجراء تحليل مسبقا، فاستخدم نفس تعريف التحليل مع تعديلات طفيفة عن طريق تحديد نسخ تعريف التحليل الموجود. في هذه الحالة، انتقل إلى الخطوة 3.3. في حالة استخدام التحليل دون أي تغيير ، حدد استخدام تعريف التحليل الحالي ؛ لا ينصح بذلك لأن مستوى التألق قد يختلف بين المقايسات في أيام مختلفة ، وعادة ما تكون بعض التصحيحات الطفيفة ضرورية.
- حدد محلل أساسي. استخدم كل قنوات الصورة: المرحلة والأخضر والأحمر والتداخل.
ملاحظة: على الرغم من أننا نستخدم عادة أقنعة الأجسام الخضراء والحمراء لحساب الشبكات ، إلا أن قناع الكائن المتداخل مفيد عندما يكون الحطام مهما. في مثل هذه الحالات ، سيتم استخدام عدد الكائنات المتداخلة كبسط بدلا من عدد الكائنات الخضراء (انظر الخطوة 3.9). إذا تم استخدام عدد الكائنات المتداخلة بدلا من عدد الكائنات الخضراء، فيجب حساب جميع الإشارات الحمراء بشكل صحيح. اضبط إعداد الإشارة الحمراء لحساب جميع الأجسام الحمراء ذات الإشارة الحمراء المنخفضة في الصور الملتقطة في نقاط زمنية لاحقة ولكن ليس لحساب الحطام. - اختر 6 - 8 صور نموذجية تمثيلية للتدريب. للتجربة هنا ، قم بتضمين الصور التالية:
الصور التي تم التقاطها بين 0 إلى 20 دقيقة لتحسين إعداد الإشارة الخضراء للتعويض عن الإشارات الخضراء الدقيقة التي يمكن توليدها في العدلات غير المكونة ل NET
صور لشبكات NETs القصوى ذات التحكم الإيجابي ، تم التقاطها بين 3-6 ساعات (يختلف الوقت حسب التحفيز المستخدم) لتحسين إعداد الإشارة الخضراء لتحديد العدلات المكونة ل NET
الصور التي تم التقاطها حول النقطة الزمنية 1 h لحساب عدد الخلايا الكلية (الملطخة بصبغة حمراء) لأن الإشارة الحمراء النووية هي الأقوى حوالي 1 ساعة (عندما تستقر جميع الخلايا في قاع البئر) وتنخفض تدريجيا بسبب موت الخلية.
الصور التي تحتوي على حطام، لاستبعادها من العد. - قم بتعيين تعريف التحليل. تتوافق الأجسام الحمراء والأجسام الخضراء مع نوى جميع العدلات وتلك التي تشكل شبكات ، على التوالي. ابدأ بالمثال التالي واضغط على معاينة الحالية أو معاينة الكل، وقم بتعديل كل معلمة لتحسين النتائج:
للأخضر: تجزئة - طرح الخلفية: قبعة علوية ؛ نصف القطر: 100 ميكرومتر ؛ العتبة: 0.3 وحدة GCU ؛ تقسيم الحافة: تشغيل; حساسية الحافة: -20 ؛ تنظيف - ملء الحفرة: 100 ميكرومتر2 ؛ ضبط الحجم 0 بكسل ؛ المرشحات- المنطقة: دقيقة 20 ميكرومتر2 ، بحد أقصى 500 ميكرومتر2 ؛ الانحراف: ماكس 0.97 ؛ متوسط الكثافة: الحد الأدنى 1.00
للأحمر: تجزئة - طرح الخلفية: قبعة علوية ؛ نصف القطر: 10 ميكرومتر ؛ العتبة: 1.0 وحدة GCU ؛ انقسام الحافة: تشغيل; حساسية الحافة: -50 ؛ ملء حفرة التنظيف: 50 ميكرومتر2 ؛ ضبط الحجم 0 بكسل ؛ المرشحات- المنطقة: دقيقة 20 ميكرومتر2 ، بحد أقصى 400 ميكرومتر2 ؛ متوسط الشدة: الحد الأدنى 1.5.- إذا ظهرت إشارة خضراء مفرطة في العدلات التي لا ينبغي أن تشكل شبكات (على سبيل المثال ، العدلات غير المحفزة عند 0 دقيقة والتي تحتوي على نوى مفصصة) ، فقد يكون ذلك بسبب تجاوز الإشارة الحمراء المكتشفة في القناة الخضراء. في مثل هذه الحالة، ارجع إلى الخطوة 3.1، وافتح طبقات الصورة على يسار الشاشة، وقم بإزالة الإشارات الحمراء من القناة الخضراء باستخدام إلغاء الخلط الطيفي.
- خيار آخر هو تعيين بئر واحد للتلطيخ الفردي بصبغة حمراء نووية لتوضيح مقدار الإشارة الحمراء التي يجب إزالتها من القناة الخضراء. من ناحية أخرى ، عادة لا يؤثر نزيف الإشارة الخضراء في القناة الحمراء على التحليلات.
ملاحظة: لا يهم إذا كانت الحساسية للإشارة الحمراء منخفضة ولا يتم التقاط جميع الإشارات الحمراء في الصور التي يتم التقاطها في نقاط زمنية لاحقة (على سبيل المثال ، نقطة زمنية 6 ساعات). يعتبر الحد الأقصى لعدد الأجسام الحمراء في كل صورة إجمالي عدد العدلات في الصورة وسيتم استخدامه كمقام في الخطوة 3.9. عادة ، تبلغ الإشارات الحمراء النووية ذروتها حوالي 1 ساعة من النقطة الزمنية وتتضاءل تدريجيا بسبب موت الخلايا (الشكل 1 ب). لذلك ، اضبط إعداد الإشارة الحمراء لحساب عدد الكائنات الحمراء بشكل صحيح في الصور ذات الإشارات الحمراء القصوى.
- حدد أوقات المسح والآبار المراد تحليلها. عادة ، يجب تحليل جميع النقاط الزمنية والآبار. قم بتوفير تسمية لتعريف التحليل.
- تحقق من الملخص وابدأ التحليل. يستغرق إكمال التحليل بضع ساعات (تعتمد المدة على عدد النقاط الزمنية والآبار). بمجرد إطلاقه ، سيظهر اسم تعريف التحليل تحت اسم الدراسة في علامة التبويب عرض ، وسيظهر التاريخ الكامل عند الانتهاء.
- بعد اكتماله ، افتح الدراسة التي تم تحليلها بالنقر المزدوج فوق اسم تعريف التحليل. افتح الطبقات على يسار الشاشة وتحقق مما إذا تم تمييز كل خلية بشكل صحيح. إذا لم يتم وضع علامة بشكل صحيح ، فارجع إلى الخطوة 3.1 وكرر الخطوات اللاحقة.
- انقر على مقاييس الرسم البياني على يمين الشاشة لتصدير البيانات. حدد العدد الأخضر أو العد الأحمر أو عدد التداخل (لكل صورة)، ثم اختر النقاط الزمنية والآبار التي سيتم تصديرها. لتحديد التجميع، حدد Plate Map Replicates إذا تم تحديد جميع الآبار بشكل صحيح عند إجراء المسح.
- تصدير البيانات. احسب النسبة المئوية للخلايا المكونة ل NET لكل شرط / نقطة زمنية بالمعادلة التالية باستخدام برنامج مناسب.
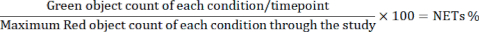
ملاحظة: السبب في أن المقام هو الحد الأقصى لعدد الأجسام الحمراء هو أن عدد الأجسام الحمراء يبلغ ذروته عند حوالي 1 ساعة من الوقت وينخفض تدريجيا بسبب موت الخلية.
Access restricted. Please log in or start a trial to view this content.
النتائج
توفر هذه الطريقة تباين الطور ، وصور الفلورسنت الأحمر (صبغة نفاذة للغشاء) وفلورسنت أخضر (صبغة غير منفذة للغشاء) تم التقاطها في كل نقطة زمنية. جنبا إلى جنب مع عملية تشكيل NET ، لوحظت التغيرات المورفولوجية في تباين الطور والصور الفلورية الحمراء ، وبمجرد اختراق الغشاء ، يمكن ملاحظة مضان أخضر (
Access restricted. Please log in or start a trial to view this content.
Discussion
الطرق الحالية لقياس الشبكات خارج الجسم الحي لها العديد من العيوب التي تحد من قدرتنا على دراسة العدلات والشبكات والأهداف العلاجية المحتملة بطريقة غير متحيزة وعالية الإنتاجية10,14. على سبيل المثال ، العد المباشر للخلايا المكونة ل NET بعد التلوين المناعي ، ...
Access restricted. Please log in or start a trial to view this content.
Disclosures
ليس للمؤلفين مصالح مالية متنافسة.
Acknowledgements
نشكر قسم التصوير بالضوء في مكتب العلوم والتكنولوجيا في المعهد الوطني لالتهاب المفاصل وأمراض الجهاز العضلي الهيكلي والجلد التابع للمعاهد الوطنية للصحة. تم دعم هذا البحث من قبل برنامج البحوث الداخلية للمعهد الوطني لالتهاب المفاصل وأمراض العضلات والعظام والجلد التابع للمعاهد الوطنية للصحة (ZIA AR041199).
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| AKT inhibitor | Calbiochem | 124028 | |
| Clear 96-well plate | Corning | 3596 | |
| Live cell analysis system | Sartorius | N/A | Incucyte Software (v2019B) |
| Membrane-impermeable DNA green dye | Thermo Fisher Scientific | S7020 | |
| Nuclear red dye | Enzo | ENZ-52406 | Neutrophil pellet becomes bluish after staining. |
| RPMI | Thermo Fisher Scientific | 11835030 | Phenol red containig RPMI can be used. |
References
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- Wigerblad, G., Kaplan, M. J. Neutrophil extracellular traps in systemic autoimmune and autoinflammatory diseases. Nat Rev Immunol. 23 (5), 274-288 (2022).
- Wagner, D. D., Heger, L. A. Thromboinflammation: From atherosclerosis to COVID-19. Arterioscler Thromb Vasc Biol. 42 (9), 1103-1112 (2022).
- Njeim, R., et al. NETosis contributes to the pathogenesis of diabetes and its complications. J Mol Endocrinol. 65 (4), R65-R76 (2020).
- De Meo, M. L., Spicer, J. D. The role of neutrophil extracellular traps in cancer progression and metastasis. Semin Immunol. 57, 101595(2021).
- Nakabo, S., Romo-Tena, J., Kaplan, M. J. Neutrophils as drivers of immune dysregulation in autoimmune diseases with skin manifestations. J Invest Dermatol. 142 (3 Pt B), 823-833 (2022).
- Gupta, S., Chan, D. W., Zaal, K. J., Kaplan, M. J. A high-throughput real-time imaging technique to quantify NETosis and distinguish mechanisms of cell death in human neutrophils. J Immunol. 200 (2), 869-879 (2018).
- Nakabo, S., Kaplan, M. J., Gupta, S. Quantification of neutrophils undergoing NET formation and distinguishing mechanisms of neutrophil cell death by use of a high-throughput method. Methods Mol Biol. 2543, 129-140 (2022).
- Singh, J., et al. Moonlighting chromatin: when DNA escapes nuclear control. Cell Death Differ. 30 (4), 861-875 (2023).
- Carmona-Rivera, C., Kaplan, M. J. Induction and quantification of NETosis. Curr Protoc Immunol. 115, 14.41.11-14.41.14 (2016).
- Hsu, A. Y., Peng, Z., Luo, H., Loison, F. Isolation of human neutrophils from whole blood and buffy coats. J Vis Exp. (175), 62837(2021).
- Neubert, E., et al. Serum and serum albumin inhibit in vitro formation of neutrophil extracellular traps (NETs). Front Immunol. 10, 12(2019).
- von Kockritz-Blickwede, M., Chow, O. A., Nizet, V. Fetal calf serum contains heat-stable nucleases that degrade neutrophil extracellular traps. Blood. 114 (25), 5245-5246 (2009).
- Zhao, W., Fogg, D. K., Kaplan, M. J. A novel image-based quantitative method for the characterization of NETosis. J Immunol Methods. 423, 104-110 (2015).
- Papayannopoulos, V. Neutrophil extracellular traps in immunity and disease. Nat Rev Immunol. 18 (2), 134-147 (2018).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved