È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Tecnica real-time ad alto rendimento per quantificare la formazione di trappole extracellulari di neutrofili nei neutrofili umani
In questo articolo
Riepilogo
Presentiamo un metodo automatizzato ad alto rendimento per quantificare le trappole extracellulari dei neutrofili (NET) utilizzando il sistema di analisi delle cellule vive, accoppiato con un approccio a doppio colorante dipendente dalla permeabilità della membrana.
Abstract
I neutrofili sono cellule di linea mieloide che costituiscono una parte cruciale del sistema immunitario innato. L'ultimo decennio ha rivelato ulteriori ruoli chiave che i neutrofili svolgono nella patogenesi del cancro, delle malattie autoimmuni e di varie condizioni infiammatorie acute e croniche, contribuendo all'inizio e alla perpetuazione della disregolazione immunitaria attraverso molteplici meccanismi, tra cui la formazione di trappole extracellulari per neutrofili (NET), che sono strutture cruciali nella difesa antimicrobica. I limiti delle tecniche per quantificare la formazione di NET in modo imparziale, riproducibile ed efficiente hanno limitato la nostra capacità di comprendere ulteriormente il ruolo dei neutrofili nella salute e nelle malattie. Descriviamo un metodo automatizzato, in tempo reale e ad alto rendimento per quantificare i neutrofili sottoposti a formazione di NET utilizzando una piattaforma di imaging di cellule vive accoppiata con un approccio a doppio colorante dipendente dalla permeabilità della membrana che utilizza due diversi coloranti di DNA per visualizzare il DNA intracellulare ed extracellulare. Questa metodologia è in grado di aiutare a valutare la fisiologia dei neutrofili e testare molecole in grado di colpire la formazione di NET.
Introduzione
Le trappole extracellulari dei neutrofili (NET) sono strutture di cromatina simili a ragnatele estruse dai neutrofili in risposta a vari stimoli infiammatori. I NET sono composti da DNA, istoni e varie proteine/peptidi antimicrobici, che intrappolano e uccidono gli agenti patogeni infettivi e invocano risposte infiammatorie1.
Sebbene i NET siano utili per la difesa dell'ospite contro gli agenti patogeni, hanno attirato l'attenzione come potenziale motore di varie malattie autoimmuni2, trombosi3, malattie metaboliche4 e crescita metastatica dei tumori5. In quanto tale, l'inibizione della formazione di NET è una potenziale opzione terapeutica per queste malattie. Tuttavia, nonostante alcune promettenti molecole mirate ai NET in fase di sviluppo6, non esiste ancora una terapia approvata che influisca specificamente su questo meccanismo. Ciò è, almeno in parte, attribuibile alla mancanza di metodi di quantificazione oggettivi, imparziali, riproducibili e ad alto rendimento per la formazione di NET.
Abbiamo stabilito e segnalato un nuovo metodo che utilizza una piattaforma di imaging a due colori su cellule vive 7,8. Le immagini time-lapse di neutrofili colorati con colorante nucleare permeabile alla membrana e colorante DNA impermeabile alla membrana vengono analizzate dal software e il numero di neutrofili pre e post-formazione di NET viene conteggiato in più punti temporali. Poiché l'integrità della membrana plasmatica viene persa durante la formazione di NET dalla regolazione deldisassemblaggio della lamina B mediata da PKCα e della lamina A/C mediata da CDK4/6 CDK4/6 9, i neutrofili che formano NET sono colorati da un colorante di DNA impermeabile alla membrana, mentre i neutrofili sani non lo sono. Questo metodo supera i problemi delle tecniche precedentemente riportate per quantificare la formazione di NET e fornisce una quantificazione imparziale, ad alto rendimento, riproducibile e accurata di NET in modo automatizzato.
Protocollo
I neutrofili da soggetti umani sani sono stati ottenuti dopo che il consenso informato è stato fornito secondo il protocollo approvato dall'Institutional Review Board (IRB) del National Institutes of Health (NIH). Il protocollo segue le linee guida del comitato etico per la ricerca umana del NIH.
1. Colorazione dei neutrofili e preparazione della piastra di analisi
- Prelevare il sangue periferico con un adeguato consenso informato scritto seguendo le linee guida di ciascun istituto e isolare i neutrofili utilizzando qualsiasi metodo desiderato. Ad esempio, il metododi Ficoll-destrano 10,11 è un metodo comunemente usato per l'isolamento dei neutrofili dal sangue periferico umano.
NOTA: Sebbene il metodo qui discusso utilizzi neutrofili umani, anche i neutrofili di topo possono essere analizzati utilizzando un protocollo simile. - Risospendere i neutrofili nel terreno 1640 del Roswell Park Memorial Institute (RPMI; vedere la tabella dei materiali) a 2,0 x 106 neutrofili/mL e posizionare la sospensione di neutrofili in una provetta da centrifuga da 1,5 mL.
NOTA: Di solito non aggiungiamo siero fetale bovino (FBS) ai terreni di coltura perché l'albumina in FBS può ridurre la formazione di NET12 e la nucleasi termostabile in FBS può degradare i NET13. - Aggiungere un colorante di DNA rosso permeabile alla membrana (vedere la tabella dei materiali) a 1 μL per 1,5 mL di sospensione di neutrofili.
- Incubare a temperatura ambiente per 5 minuti al buio. Centrifugare a 2500 x g per 5 minuti a temperatura ambiente e rimuovere il surnatante.
- Risospendere i neutrofili in 1 mL di RPMI, quindi centrifugare a 2500 x g per 5 minuti a temperatura ambiente e rimuovere il surnatante. Ripetere 2 volte (lavare il totale 3 volte).
- Risospendere i neutrofili in 1 mL di RPMI e contare utilizzando un contatore di cellule. Diluire la sospensione di neutrofili a 1,5 x 105 neutrofili/mL.
- Aggiungere 4 μL di colorante di DNA verde impermeabile alla membrana 1:100 pre-diluito (vedere Tabella dei materiali) per 1 mL di sospensione di neutrofili.
- Mettere 100 μL di sospensione di neutrofili per pozzetto in una piastra trasparente a 96 pozzetti trattata con coltura tissutale (vedere Tabella dei materiali). Impostare ogni condizione in triplice copia.
NOTA: Abbiamoprecedentemente dimostrato 7 che non vi è alcuna differenza nella formazione e quantificazione di NET con questo metodo se i neutrofili sono posti in piastre con o senza rivestimento di poli-L-lisina. Pertanto, l'uso di una piastra trattata con coltura tissutale è sufficiente per questo metodo. - Aggiungere 100 μL di RPMI contenente reagenti di stimolo con o senza inibitore o qualsiasi altro reagente di interesse nei rispettivi pozzetti. Utilizzare sempre pozzetti di controllo positivo aggiungendo 500 nM di forbolo 12-miristato 13-acetato (PMA) o 2,5 μM di ionoforo di calcio (A23187), in questo caso è stato utilizzato un inibitore AKT da 30 μM.
- Posizionare la piastra nel sistema di analisi delle cellule vive (vedere la tabella dei materiali) alloggiato in un incubatore al 5% di CO2 .
NOTA: La condensa potrebbe apparire sulla parte superiore e inferiore della piastra pochi minuti dopo questo passaggio. Ciò può inibire la corretta imaging. Pulire e rimuovere accuratamente la condensa prima di iniziare la scansione. Posizionare la lastra nella macchina, avviare il software, immettere il protocollo di scansione, quindi pulire la lastra appena prima della prima scansione.
2. Piastra di scansione per visualizzare i neutrofili che formano NET
- Avviare il software (vedere la Tabella dei materiali per i dettagli sul software). Avvia Aggiungi nave premendo + nell'angolo in alto a sinistra.
- Scegli Scansione pianificata. Scegliere New (Nuovo).
NOTA: Una volta creato, eseguire lo stesso protocollo di scansione scegliendo Copia precedente e selezionando il protocollo memorizzato nella schermata successiva. - Scegliere Standard (Standard). Impostare le impostazioni di scansione come: Opzioni cella per cella: Nessuna; Canali immagine: Fase, Verde (Tempo di acquisizione: 200 ms), Rosso (Tempo di acquisizione: 400 ms); Obiettivo: 20x.
- Scegliere la piastra utilizzata dall'elenco. Selezionare la posizione del recipiente in cui posizionare la piastra sul vassoio del sistema di imaging per cellule vive.
- Selezionare i pozzetti in cui sono presenti i campioni e decidere quante immagini acquisire per pozzetto. In base al numero di immagini per pozzetto, generare una durata di scansione stimata per la piastra. Di solito sono sufficienti 4 immagini per pozzetto; Tuttavia, questo può variare a seconda delle condizioni e della frequenza delle scansioni.
- Immettere le informazioni di ciascun pozzetto (ad esempio, tipo di cella e composto) facendo clic su Crea mappa piastra per fornire un nome allo studio.
NOTA: La mappa delle lastre può essere preparata e salvata in anticipo utilizzando l'editor delle mappe delle lastre nel software. I dati della mappa delle piastre salvati possono essere importati durante questa fase. Se lo si desidera, è possibile saltare ed eseguire l'immissione delle informazioni sulla mappa delle targhe in un secondo momento. I dati possono essere ottenuti anche senza inserire le informazioni sulla disposizione delle targhe. Tuttavia, si consiglia di inserire le informazioni sulla piastra per facilitare l'analisi successiva. - Nella schermata successiva, selezionare Basic Analyzer come tipo di analisi e scegliere Protocollo di analisi dall'elenco a discesa, che mostra le definizioni di analisi utilizzate in precedenza (non i protocolli di scansione). L'unmixing spettrale sia per il verde che per il rosso può essere lasciato allo 0,0 %.
NOTA: Se lo si desidera, questo passaggio può essere saltato. - Pianifica la scansione. Per l'esperimento qui, scansiona ogni 15-20 minuti per 8 ore . Impostare l'ora di inizio della scansione trascinando la barra bianca e grigia nella parte superiore dello schermo.
NOTA: Evitare di eseguire scansioni troppo frequenti, altrimenti la macchina potrebbe non funzionare correttamente a causa del surriscaldamento. Il tempo di scansione non deve superare le 12 ore per 24 ore. È possibile che venga visualizzato un avviso se la scansione è troppo frequente. - Controllare l'impostazione di scansione nella schermata successiva e premere Aggiungi a pianificazione per avviare la scansione. Attendere la scansione del set iniziale di immagini per confermare se tutto funziona correttamente.
- A volte le cellule potrebbero non essere messe a fuoco correttamente. In tal caso, controllare se è presente condensa (e pulire di conseguenza), se la piastra è posizionata correttamente sul vassoio o se la posizione della piastra è specificata correttamente, ecc. Se le cellule galleggiano ancora e non si sono depositate sul fondo del pozzetto, attendere circa 5 minuti prima di eseguire la scansione.
3. Impostazione della definizione di analisi per quantificare i NET
- Aprire lo studio (recipiente) da analizzare nella scheda Vista. Premere Avvia analisi a sinistra dello schermo. Selezionate Crea nuova definizione di analisi (Create New Analysis Definition).
- Se un'analisi è stata eseguita in precedenza, utilizzate la stessa definizione di analisi con modifiche minori selezionando Copia definizione analisi esistente (Copy Existing Analysis Definition). In questo caso, andare al punto 3.3. Se utilizzate l'analisi senza alcuna alterazione, selezionate Usa definizione analisi esistente (Use Existing Analysis Definition); Questo non è raccomandato perché il livello di fluorescenza può variare tra i saggi in giorni diversi e di solito sono necessarie alcune correzioni minori.
- Selezionare Analizzatore di base. Usa tutti i canali immagine: Fase, Verde, Rosso e Sovrapposizione.
NOTA: anche se in genere si usano maschere di oggetti verdi e rosse per contare le NET, la maschera di sovrapposizione degli oggetti è utile quando i detriti sono importanti. In questi casi, il numero di oggetti sovrapposti verrà utilizzato come numeratore invece del numero di oggetti verdi (vedere il passaggio 3.9). Se si utilizza il conteggio degli oggetti sovrapposti invece del conteggio degli oggetti verdi, tutti i segnali rossi devono essere conteggiati correttamente. Regolare l'impostazione del segnale rosso per contare tutti gli oggetti rossi con segnale rosso basso nelle immagini scattate in punti temporali successivi, ma non per contare i detriti. - Scegli 6-8 immagini di esempio rappresentative per l'addestramento. Per l'esperimento, includi le seguenti immagini:
Immagini scattate tra 0 e 20 minuti per ottimizzare l'impostazione del segnale verde per compensare i sottili segnali verdi che possono essere generati nei neutrofili non formanti NET
Immagini di NET massimi con controllo positivo, scattate tra 3-6 ore (il tempo varia a seconda della stimolazione utilizzata) per ottimizzare l'impostazione del segnale verde per definire i neutrofili che formano NET
Immagini scattate intorno al timepoint di 1 ora per contare il numero di cellule totali (colorate con colorante rosso) poiché il segnale rosso nucleare è il più forte intorno al timepoint di 1 ora (quando tutte le cellule si sono depositate sul fondo del pozzo) e diminuisce gradualmente a causa della morte cellulare.
Immagini contenenti detriti, per escluderli dal conteggio. - Impostate la definizione dell'analisi. Gli oggetti rossi e gli oggetti verdi corrispondono rispettivamente ai nuclei di tutti i neutrofili e a quelli che formano i NET. Iniziare con il seguente esempio di e premere Anteprima corrente o Anteprima tutto e modulare ciascun parametro per ottimizzare i risultati:
Per il verde: Segmentazione- Sottrazione di sfondo: Cappello a cilindro; Raggio: 100 μM; Soglia: 0,3 GCU; Divisione dei bordi: On; Sensibilità dei bordi: -20; Pulizia -Riempimento fori: 100 μm2; Regola la dimensione 0 pixel; Filtri- Area: min 20 μm2, max 500 μm2; Eccentricità: max 0,97; Intensità media: min 1.00
Per il rosso: Segmentazione- Sottrazione di sfondo: Cappello a cilindro; Raggio: 10 μM; Soglia: 1,0 GCU; Divisione dei bordi: ON; Sensibilità dei bordi: -50; Riempimento del foro di pulizia: 50 μm2; Regola la dimensione 0 pixel; Filtri- Area: min 20 μm2, max 400 μm2; Intensità media: min 1.5.- Se un segnale verde eccessivo appare nei neutrofili che non dovrebbero formare NET (ad esempio, neutrofili non stimolati a 0 min che hanno nuclei lobulati), può essere dovuto al trabocco del segnale rosso rilevato nel canale verde. In tal caso, tornate al punto 3.1, aprite Image Layers sulla sinistra dello schermo e rimuovete i segnali rossi dal canale verde utilizzando Spectral Unmixing.
- Un'altra opzione è quella di impostare un pozzetto per la colorazione singola con colorante rosso nucleare per chiarire quanta parte del segnale rosso deve essere rimossa dal canale verde. D'altra parte, di solito il segnale verde che sanguina nel canale rosso non influisce sulle analisi.
NOTA: Non importa se la sensibilità al segnale rosso è bassa e non tutti i segnali rossi vengono catturati nelle immagini scattate in punti temporali successivi (ad esempio, punto temporale di 6 ore). Il numero massimo di oggetti rossi in ogni immagine è considerato come il numero totale di neutrofili nell'immagine e sarà usato come denominatore al punto 3.9. Di solito, i segnali rossi nucleari raggiungono il picco intorno a 1 ora e diminuiscono gradualmente a causa della morte cellulare (Figura 1B). Pertanto, regolare l'impostazione del segnale rosso per contare correttamente gli oggetti rossi nelle immagini con i segnali rossi massimi.
- Selezionare i tempi di scansione e i pozzetti da analizzare. Di solito, tutti i punti temporali e i pozzi devono essere analizzati. Specificare un'etichetta per la definizione dell'analisi.
- Controlla il riepilogo e avvia l'analisi. Sono necessarie alcune ore per il completamento dell'analisi (la durata dipende dal numero di punti temporali e pozzetti). Una volta avviato, il nome della definizione dell'analisi verrà visualizzato sotto il nome dello studio nella scheda Vista e la data completa verrà visualizzata al termine.
- Al termine, aprire lo studio analizzato facendo doppio clic sul nome della definizione dell'analisi. Apri Livelli a sinistra dello schermo e controlla se ogni cella è contrassegnata correttamente. Se non è contrassegnato correttamente, tornare al punto 3.1 e ripetere i passaggi successivi.
- Fai clic su Metriche grafico a sinistra dello schermo per esportare i dati. Selezionare Conteggio verde, Conteggio rosso o Conteggio sovrapposizione (per immagine), quindi scegliere i punti temporali e i pozzetti da esportare. Per selezionare il raggruppamento, selezionare Repliche mappa piastre se tutti i pozzetti sono specificati correttamente al termine della scansione.
- Esportare i dati. Calcolare la percentuale di celle che formano NET per ogni condizione/punto temporale utilizzando la seguente equazione utilizzando un software appropriato.
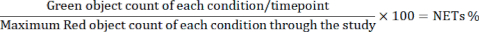
NOTA: Il motivo per cui il denominatore è il numero massimo di oggetti rossi è che il numero di oggetti rossi raggiunge il picco a circa 1 ora e diminuisce gradualmente a causa della morte cellulare.
Risultati
Questo metodo fornisce immagini a contrasto di fase, fluorescenti rosse (colorante permeabile alla membrana) e fluorescenti verdi (colorante impermeabile alla membrana) scattate in ogni punto temporale. Insieme al processo di formazione del NET, si osservano cambiamenti morfologici nelle immagini a contrasto di fase e fluorescenti rosse e, una volta violata la membrana, si può osservare la fluorescenza verde (Figura 1). In questo saggio, i neutrofili che formano NET sono generalmente rotond...
Discussione
Gli attuali metodi per quantificare i NET ex vivo presentano diversi inconvenienti che limitano la nostra capacità di studiare neutrofili, NET e potenziali bersagli terapeutici in modo imparziale e ad alto rendimento10,14. Ad esempio, il conteggio diretto delle cellule che formano NET dopo la colorazione immunofluorescente, considerato il gold standard per la quantificazione dei NET, è a basso rendimento e dipende dalla visione soggettiva dell'operator...
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti.
Riconoscimenti
Ringraziamo la Sezione di Imaging Luminoso dell'Ufficio di Scienza e Tecnologia dell'Istituto Nazionale di Artrite e Malattie Muscoloscheletriche e della Pelle del National Institutes of Health. Questa ricerca è stata supportata dal Programma di Ricerca Intramurale del National Institute of Arthritis and Musculoskeletal and Skin Diseases del National Institutes of Health (ZIA AR041199).
Materiali
| Name | Company | Catalog Number | Comments |
| AKT inhibitor | Calbiochem | 124028 | |
| Clear 96-well plate | Corning | 3596 | |
| Live cell analysis system | Sartorius | N/A | Incucyte Software (v2019B) |
| Membrane-impermeable DNA green dye | Thermo Fisher Scientific | S7020 | |
| Nuclear red dye | Enzo | ENZ-52406 | Neutrophil pellet becomes bluish after staining. |
| RPMI | Thermo Fisher Scientific | 11835030 | Phenol red containig RPMI can be used. |
Riferimenti
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- Wigerblad, G., Kaplan, M. J. Neutrophil extracellular traps in systemic autoimmune and autoinflammatory diseases. Nat Rev Immunol. 23 (5), 274-288 (2022).
- Wagner, D. D., Heger, L. A. Thromboinflammation: From atherosclerosis to COVID-19. Arterioscler Thromb Vasc Biol. 42 (9), 1103-1112 (2022).
- Njeim, R., et al. NETosis contributes to the pathogenesis of diabetes and its complications. J Mol Endocrinol. 65 (4), R65-R76 (2020).
- De Meo, M. L., Spicer, J. D. The role of neutrophil extracellular traps in cancer progression and metastasis. Semin Immunol. 57, 101595 (2021).
- Nakabo, S., Romo-Tena, J., Kaplan, M. J. Neutrophils as drivers of immune dysregulation in autoimmune diseases with skin manifestations. J Invest Dermatol. 142 (3 Pt B), 823-833 (2022).
- Gupta, S., Chan, D. W., Zaal, K. J., Kaplan, M. J. A high-throughput real-time imaging technique to quantify NETosis and distinguish mechanisms of cell death in human neutrophils. J Immunol. 200 (2), 869-879 (2018).
- Nakabo, S., Kaplan, M. J., Gupta, S. Quantification of neutrophils undergoing NET formation and distinguishing mechanisms of neutrophil cell death by use of a high-throughput method. Methods Mol Biol. 2543, 129-140 (2022).
- Singh, J., et al. Moonlighting chromatin: when DNA escapes nuclear control. Cell Death Differ. 30 (4), 861-875 (2023).
- Carmona-Rivera, C., Kaplan, M. J. Induction and quantification of NETosis. Curr Protoc Immunol. 115, 14.41.11-14.41.14 (2016).
- Hsu, A. Y., Peng, Z., Luo, H., Loison, F. Isolation of human neutrophils from whole blood and buffy coats. J Vis Exp. (175), 62837 (2021).
- Neubert, E., et al. Serum and serum albumin inhibit in vitro formation of neutrophil extracellular traps (NETs). Front Immunol. 10, 12 (2019).
- von Kockritz-Blickwede, M., Chow, O. A., Nizet, V. Fetal calf serum contains heat-stable nucleases that degrade neutrophil extracellular traps. Blood. 114 (25), 5245-5246 (2009).
- Zhao, W., Fogg, D. K., Kaplan, M. J. A novel image-based quantitative method for the characterization of NETosis. J Immunol Methods. 423, 104-110 (2015).
- Papayannopoulos, V. Neutrophil extracellular traps in immunity and disease. Nat Rev Immunol. 18 (2), 134-147 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon