このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ヒト好中球における好中球細胞外トラップ形成を定量化するリアルタイムハイスループット技術(英語)
要約
本稿では、膜透過性依存性二重色素アプローチと組み合わせた生細胞解析システムを利用して、好中球細胞外トラップ(NET)を定量する自動化されたハイスループット法を紹介します。
要約
好中球は、自然免疫系の重要な部分を形成する骨髄系細胞です。過去10年間で、好中球ががん、自己免疫疾患、およびさまざまな急性および慢性炎症状態の病因において果たす重要な役割は、抗菌防御に不可欠な構造である好中球細胞外トラップ(NET)の形成を含む複数のメカニズムを通じて免疫調節不全の開始と永続化に寄与することが明らかになりました。偏りがなく、再現性があり、効率的な方法でNET形成を定量化する技術の限界により、健康と疾患における好中球の役割をさらに理解する能力が制限されていました。本稿では、細胞内および細胞外のDNAをイメージングするために2つの異なるDNA色素を用いた膜透過性依存性二重色素アプローチと組み合わせたライブセルイメージングプラットフォームを用いて、NET形成を受ける好中球を定量する自動化されたリアルタイムのハイスループット法について説明する。この方法論は、好中球の生理機能を評価し、NET形成を標的とすることができる分子をテストするのに役立ちます。
概要
好中球細胞外トラップ(NET)は、さまざまな炎症刺激に応答して好中球から押し出されるウェブ状のクロマチン構造です。NETは、DNA、ヒストン、およびさまざまな抗菌タンパク質/ペプチドで構成されており、感染性病原体を捕捉して殺し、炎症反応を引き起こします1。
NETは病原体に対する宿主防御に有益である一方で、様々な自己免疫疾患2、血栓症3、代謝性疾患4、がんの転移増殖5の潜在的なドライバーとして注目されています。そのため、NET形成の阻害は、これらの疾患の潜在的な治療選択肢です。しかし、いくつかの有望なNETを標的とする分子が開発中であるにもかかわらず6、このメカニズムに特異的に作用する承認された治療法はまだありません。これは、少なくとも部分的には、NET形成のための客観的で、偏りがなく、再現性があり、ハイスループットの定量化方法の欠如に起因しています。
我々は、2色生細胞イメージングプラットフォームを利用した新しい手法を確立し、報告した7,8。膜透過性核色素および膜不透過性DNA色素で染色した好中球のタイムラプス画像をソフトウェアで解析し、NET形成前後の好中球の数を複数の時点でカウントします。PKCαを介したラミンBおよびCDK4/6を介したラミンA/Cの分解の制御により、NET形成中に原形質膜の完全性が失われるため9、NET形成好中球は膜不透過性のDNA色素によって染色されますが、健康な好中球は染色されません。この方法は、NET形成を定量化するために以前に報告された手法の問題を克服し、自動化された方法で偏りのない、高スループットで、再現性のある、正確なNET定量を提供します。
Access restricted. Please log in or start a trial to view this content.
プロトコル
健康なヒト被験者からの好中球は、国立衛生研究所(NIH)治験審査委員会(IRB)が承認したプロトコルに基づいてインフォームドコンセントが提供された後に取得されました。プロトコルは、NIHヒト研究倫理委員会のガイドラインに従います。
1. 好中球の染色とアッセイプレートの調製
- 各施設のガイドラインに従って、適切な書面によるインフォームドコンセントで末梢血を採取し、任意の方法で好中球を分離します。例えば、フィコール-デキストラン法10,11は、ヒト末梢血から好中球を単離するために一般的に使用される方法です。
注:ここで説明する方法はヒト好中球を使用しますが、マウス好中球も同様のプロトコルを使用して分析できます。 - 好中球をRoswell Park Memorial Institute(RPMI; 材料表を参照)1640培地で2.0 x 106 好中球/mLで再懸濁し、好中球懸濁液を1.5 mL遠心チューブに入れます。
注:FBS中のアルブミンはNET形成を減少させる可能性があり12 、FBS中の熱安定性ヌクレアーゼはNETを分解する可能性があるため、通常はウシ胎児血清(FBS)を培地に添加しません13。 - 膜透過性赤色DNA色素( 材料表を参照)を好中球懸濁液1.5 mLあたり1 μLで添加します。
- 暗所で室温で5分間インキュベートします。室温で2500 x g で5分間遠心分離し、上清を除去します。
- 好中球を1 mLのRPMIに再懸濁し、室温で2500 x g で5分間遠心分離し、上清を除去します。2回繰り返します(合計3回洗浄)。
- 好中球を 1 mL の RPMI に再懸濁し、セルカウンターを使用してカウントします。好中球懸濁液を1.5 x 105 好中球/ mLに希釈します。
- 好中球懸濁液1 mLあたり、1:100の希釈済み膜不透過性緑色DNA色素( 材料表を参照)を4 μL添加します。
- ウェルあたり100 μLの好中球懸濁液を透明な組織培養処理した96ウェルプレートに入れます( 材料表を参照)。各条件を3回に分けて設定します。
注:好中球をポリ-L-リジンコーティングの有無にかかわらずプレートに入れた場合、この分析法ではNETの形成と定量に違いがないことを以前に 示しました7。したがって、この方法には組織培養処理プレートを用いれば十分である。 - 100 μLのRPMI含有刺激試薬(阻害剤の有無にかかわらず)、または目的の他の試薬をそれぞれのウェルに加えます。ここでは、500 nM ホルボール 12-ミリスチン酸 13-酢酸(PMA)または 2.5 μM カルシウムイオノフォア(A23187)を添加して、常にポジティブコントロールウェルを使用してください。
- プレートを、5% CO2 インキュベーターに収容された生細胞解析システム(材料表を参照)に入れます。
注意: この手順の数分後に、プレートの上部と下部に結露が現れる場合があります。これにより、適切なイメージングが阻害される可能性があります。スキャンを開始する前に、結露を完全に拭いて取り除いてください。プレートをマシンに置き、ソフトウェアを起動し、スキャンプロトコルを入力してから、最初のスキャンの直前にプレートを拭きます。
2. NET形成好中球を可視化するスキャニングプレート
- ソフトウェアを起動します(ソフトウェアの詳細については 、部品表 を参照してください)。左上隅の+を押して、 Add Vessel を起動します。
- [ スケジュールに従ってスキャン] を選択します。 [新規] を選択します。
注: これを作成したら、[ Copy Previous ] を選択し、次の画面で [保存されているプロトコル] を選択して、同じスキャン プロトコルを実行します。 - [ 標準] を選択します。スキャン設定を次のように設定します:セルごとのオプション:なし。画像チャンネル:位相、緑(取得時間:200 ms)、赤(取得時間:400 ms);目的:20倍。
- リストから使用するプレートを選択します。プレートをライブセルイメージングシステムのトレイに置く容器の位置を選択します。
- サンプルが存在するウェルを選択し、ウェルごとに何枚の画像を撮影するかを決定します。ウェルあたりの画像数に基づいて、プレートの推定スキャン時間を生成します。通常、ウェルごとに 4 枚の画像で十分です。ただし、これはスキャンの条件と頻度によって異なる場合があります。
- Create Plate Map をクリックして、各ウェルからの情報(細胞タイプや化合物など)を入力し、研究の名前を入力します。
注:プレートマップは、ソフトウェアのプレートマップエディタを使用して事前に準備および保存できます。保存したプレートマップデータは、このステップでインポートできます。必要に応じて、プレートマップ情報の入力をスキップして、後で実行できます。また、プレートレイアウトの情報を入力せずにデータを取得することもできます。ただし、その後の解析を容易にするために、プレート情報を入力することをお勧めします。 - 次の画面で、分析タイプとして [Basic Analyzer ] を選択し、ドロップダウン リストから [ Analysis Protocol ] を選択すると、以前に使用した分析定義 (スキャン プロトコルではない) が表示されます。緑と赤の両方のスペクトルアンミキシングは、0.0%のままにしておくことができます。
メモ: この手順は、必要に応じてスキップできます。 - スキャンをスケジュールします。ここでの実験では、15〜20分ごとに8時間スキャンします。画面上部の白と灰色のバーをドラッグして、スキャンの開始時刻を設定します。
注意: スキャンの頻度が高すぎると、過熱によりマシンがうまく機能しなくなる可能性があります。スキャン時間は、24時間あたり12時間を超えてはなりません。スキャンの頻度が高すぎる場合は、アラートが表示されることがあります。 - 次の画面でスキャン設定を確認し、[ スケジュールに追加 ]を押してスキャンを開始します。最初の画像セットがスキャンされるまで待って、すべてが正常に機能しているかどうかを確認します。
- 細胞の焦点が合っていない場合があります。その場合は、結露がないか(それに応じて拭いてください)、プレートがトレイに正しくセットされているか、プレートの位置が正しく指定されているかなどを確認してください。細胞がまだ浮遊していて、ウェルの底に落ち着いていない場合は、約5分待ってからスキャンしてください。
3. NETを定量化するための分析定義の設定
- 分析するスタディ(容器)をViewタブで開きます。画面の左側にある [Launch Analysis ]を押します。[ 新しい分析定義の作成]を選択します。
- 以前に分析を実行したことがある場合は、[ 既存の分析定義をコピー]を選択して、同じ分析定義を少し変更して使用します。この場合は、手順 3.3 に進みます。解析を変更せずに使用する場合は 、[既存の解析定義を使用]を選択します。蛍光レベルはアッセイによって日によって異なる場合があり、通常は若干の補正が必要になるため、これは推奨されません。
- [ Basic Analyzer] を選択します。すべてのイメージ チャネル(フェーズ、緑、赤、オーバーラップ)を使用します。
注:通常、NETを数えるために緑と赤のオブジェクトマスクを使用しますが、オーバーラップオブジェクトマスクは破片が重要な場合に役立ちます。このような場合、緑色のオブジェクト数の代わりに、オーバーラップ オブジェクト数が分子として使用されます(手順 3.9 を参照)。緑のオブジェクト数の代わりにオーバーラップ オブジェクト数を使用する場合は、すべての赤色の信号を正しくカウントする必要があります。赤信号の設定を調整して、後の時点で撮影された画像で赤信号が低いすべての赤色のオブジェクトをカウントしますが、破片はカウントしません。 - トレーニング用の代表的なサンプル画像を 6 つから 8 つ選択します。ここでの実験には、次の画像を含めます。
0〜20分の間に撮影された画像は、NETを形成しない好中球で生成される可能性のある微妙な緑色の信号を補正するために緑色の信号設定を最適化します
NET形成好中球を定義するために緑色のシグナル設定を最適化するために、3〜6時間(時間は使用する刺激によって異なります)の間に撮影された、ポジティブコントロールの最大NETの画像
核赤色シグナルは1時間前後(すべての細胞がウェルの底に沈殿する時点)に最も強く、細胞死により徐々に減少するため、総細胞数(赤色色素で染色)をカウントするために1時間前後に撮影された画像。
破片を含む画像は、カウント対象から除外されます。 - 解析定義を設定します。赤い物体と緑の物体は、それぞれすべての好中球の核とNETを形成している核に対応しています。次の例から始めて、「 Preview Current 」または 「Preview All」を押し、各パラメータをモジュレートして結果を最適化します。
緑の場合:セグメンテーション-背景の減算:シルクハット;半径:100μM;しきい値:0.3 GCU;エッジ分割:オン。エッジ感度:-20;クリーンアップ -穴埋め:100μm2;サイズを 0 ピクセルに調整します。フィルター-面積:最小20μm2、最大500μm2;偏心:最大0.97;平均強度:最小1.00
赤の場合:セグメンテーション-背景減算:シルクハット;半径:10μM;しきい値:1.0 GCU;エッジ分割:オン。エッジ感度:-50;クリーンアップホールフィル:50μm2;サイズを 0 ピクセルに調整します。フィルター-面積:最小20μm2、最大400μm2;平均強度:最小1.5。- NETを形成するべきではない好中球(例:小葉核を有する0分間の刺激されていない好中球)に過剰な緑色のシグナルが現れた場合、それは緑色のチャネルで検出された赤色のシグナルのオーバーフローが原因である可能性があります。このような場合は、手順 3.1 に戻り、画面左側の イメージ レイヤー を開き、 スペクトル アンミキシングを使用して緑チャンネルから赤の信号を削除します。
- 別のオプションとして、核赤色色素による単一染色用にウェルを1つセットして、緑色チャンネルからどの程度の赤色シグナルを除去すべきかを明確にします。一方、通常、赤チャンネルに緑の信号が流れ込んでも、解析には影響しません。
注:赤信号に対する感度が低く、すべての赤信号が後の時点(たとえば、6時間の時点)で撮影された画像にキャプチャされるわけではないかどうかは関係ありません。各画像内の赤い物体数の最大数は、画像内の好中球の総数と見なされ、ステップ3.9で分母として使用されます。通常、核赤色シグナルは1時間前後でピークに達し、細胞死により徐々に減少します(図1B)。したがって、赤信号の設定を調整して、赤信号が最大の画像内の赤いオブジェクトを正しくカウントします。
- 分析するスキャン時間とウェルを選択します。通常、すべての時点と井戸を分析する必要があります。分析定義にラベルを付けます。
- 概要を確認し、分析を開始します。分析が完了するまでに数時間かかります(所要時間は時点と井戸の数によって異なります)。起動すると、分析定義の名前が[表示]タブのスタディ名の下に表示され、完了すると完全な日付が表示されます。
- 完了したら、解析定義の名前をダブルクリックして解析スタディを開きます。画面の左側にある [レイヤー ]を開き、各セルが正しくマークされているかどうかを確認します。正しくマークされていない場合は、手順 3.1 に戻り、以降の手順を繰り返します。
- 画面の左側にある [Graph Metrics ] をクリックして、データをエクスポートします。 「緑数」、「赤度数」 または「 オーバーラップ数(画像ごと)」を選択し、エクスポートするタイムポイントとウェルを選択します。グループ化の選択では、スキャンの実行時にすべてのウェルが正しく指定されている場合、 プレートマップの複製 を選択します。
- データをエクスポートします。適切なソフトウェアを使用して、各条件/時点のNET形成セルの割合を次の式で計算します。
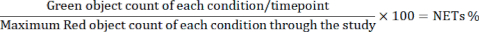
注:分母が最大赤色天体数である理由は、赤色天体の数が約1時間の時点でピークに達し、細胞死により徐々に減少するためです。
Access restricted. Please log in or start a trial to view this content.
結果
この方法では、位相差、赤色蛍光(膜透過性色素)および緑色蛍光(膜不透過性色素)の画像を各時点で取得できます。NET形成過程に伴い、位相差や赤色蛍光像に形態変化が観察され、膜が破れると緑色蛍光が観察されます(図1)。このアッセイでは、NETを形成する好中球は、ウェブ状の構造を形成するのではなく、一般的に丸いです。これは、機械の分解能が十分に高くない...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
ex vivoでNETを定量する現在の方法には、好中球、NET、および潜在的な治療標的を偏りのないハイスループットな方法で研究する能力を制限するいくつかの欠点があります10,14。例えば、NETの定量のためのゴールドスタンダードと考えられている免疫蛍光染色後のNET形成細胞の直接計数は、スループットが低く、オペレーターの主観的な見方に?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は競合する金銭的利害関係を有しません。
謝辞
米国国立衛生研究所(NIH)の国立関節炎・筋骨格・皮膚疾患研究所の科学技術局の光イメージングセクションに感謝します。この研究は、国立衛生研究所の国立関節炎および筋骨格系および皮膚疾患研究所(ZIA AR041199)の学内研究プログラムの支援を受けました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| AKT inhibitor | Calbiochem | 124028 | |
| Clear 96-well plate | Corning | 3596 | |
| Live cell analysis system | Sartorius | N/A | Incucyte Software (v2019B) |
| Membrane-impermeable DNA green dye | Thermo Fisher Scientific | S7020 | |
| Nuclear red dye | Enzo | ENZ-52406 | Neutrophil pellet becomes bluish after staining. |
| RPMI | Thermo Fisher Scientific | 11835030 | Phenol red containig RPMI can be used. |
参考文献
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- Wigerblad, G., Kaplan, M. J. Neutrophil extracellular traps in systemic autoimmune and autoinflammatory diseases. Nat Rev Immunol. 23 (5), 274-288 (2022).
- Wagner, D. D., Heger, L. A. Thromboinflammation: From atherosclerosis to COVID-19. Arterioscler Thromb Vasc Biol. 42 (9), 1103-1112 (2022).
- Njeim, R., et al. NETosis contributes to the pathogenesis of diabetes and its complications. J Mol Endocrinol. 65 (4), R65-R76 (2020).
- De Meo, M. L., Spicer, J. D. The role of neutrophil extracellular traps in cancer progression and metastasis. Semin Immunol. 57, 101595(2021).
- Nakabo, S., Romo-Tena, J., Kaplan, M. J. Neutrophils as drivers of immune dysregulation in autoimmune diseases with skin manifestations. J Invest Dermatol. 142 (3 Pt B), 823-833 (2022).
- Gupta, S., Chan, D. W., Zaal, K. J., Kaplan, M. J. A high-throughput real-time imaging technique to quantify NETosis and distinguish mechanisms of cell death in human neutrophils. J Immunol. 200 (2), 869-879 (2018).
- Nakabo, S., Kaplan, M. J., Gupta, S. Quantification of neutrophils undergoing NET formation and distinguishing mechanisms of neutrophil cell death by use of a high-throughput method. Methods Mol Biol. 2543, 129-140 (2022).
- Singh, J., et al. Moonlighting chromatin: when DNA escapes nuclear control. Cell Death Differ. 30 (4), 861-875 (2023).
- Carmona-Rivera, C., Kaplan, M. J. Induction and quantification of NETosis. Curr Protoc Immunol. 115, 14.41.11-14.41.14 (2016).
- Hsu, A. Y., Peng, Z., Luo, H., Loison, F. Isolation of human neutrophils from whole blood and buffy coats. J Vis Exp. (175), 62837(2021).
- Neubert, E., et al. Serum and serum albumin inhibit in vitro formation of neutrophil extracellular traps (NETs). Front Immunol. 10, 12(2019).
- von Kockritz-Blickwede, M., Chow, O. A., Nizet, V. Fetal calf serum contains heat-stable nucleases that degrade neutrophil extracellular traps. Blood. 114 (25), 5245-5246 (2009).
- Zhao, W., Fogg, D. K., Kaplan, M. J. A novel image-based quantitative method for the characterization of NETosis. J Immunol Methods. 423, 104-110 (2015).
- Papayannopoulos, V. Neutrophil extracellular traps in immunity and disease. Nat Rev Immunol. 18 (2), 134-147 (2018).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved