A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
عزل النوى من الأنسجة الدهنية العضلية البشرية وتسلسل الحمض النووي الريبي أحادي النوى في اتجاه مجرى النهر
In This Article
Summary
بيولوجيا الأنسجة الدهنية العضلية (IMAT) غير مستكشفة إلى حد كبير بسبب محدودية إمكانية الوصول إلى الأنسجة البشرية. هنا ، نقدم بروتوكولا مفصلا لعزل النوى وإعداد مكتبة IMAT البشري المجمد لتسلسل الحمض النووي الريبي أحادي النوى لتحديد التركيب الخلوي لهذا المستودع الدهني الفريد.
Abstract
الأنسجة الدهنية العضلية (IMAT) هي مستودع دهني غير مدروس نسبيا يقع بين ألياف العضلات. يزداد محتوى IMAT مع تقدم العمر ومؤشر كتلة الجسم ويرتبط بالأمراض الأيضية والأمراض التنكسية العضلية. ومع ذلك ، فإن فهم الخصائص البيولوجية ل IMAT وتفاعلها مع ألياف العضلات المحيطة يفتقر بشدة. في السنوات الأخيرة ، زودنا تسلسل الحمض النووي الريبي أحادي الخلية والنوى بأطالس خاصة بنوع الخلية للعديد من الأنسجة البشرية. ومع ذلك ، لا يزال التركيب الخلوي لل IMAT البشري غير مستكشف إلى حد كبير بسبب التحديات المتأصلة في إمكانية الوصول إليه من جمع الخزعة في البشر. بالإضافة إلى الكمية المحدودة من الأنسجة التي تم جمعها ، فإن معالجة IMAT البشرية معقدة بسبب قربها من الأنسجة العضلية الهيكلية واللفافة. الطبيعة المحملة بالدهون للخلايا الشحمية تجعلها غير متوافقة مع عزل الخلية الواحدة. وبالتالي ، فإن تسلسل الحمض النووي الريبي أحادي النوى هو الأمثل للحصول على نسخ عالي الأبعاد بدقة خلية واحدة ويوفر إمكانية الكشف عن بيولوجيا هذا المستودع ، بما في ذلك التركيب الخلوي الدقيق ل IMAT. هنا ، نقدم بروتوكولا مفصلا لعزل النوى وإعداد مكتبة IMAT البشري المجمد لتسلسل الحمض النووي الريبي أحادي النواة. يسمح هذا البروتوكول بتحديد سمات آلاف النوى باستخدام نهج قائم على القطيرات ، وبالتالي توفير القدرة على اكتشاف أنواع الخلايا النادرة والمنخفضة الوفرة.
Introduction
الأنسجة الدهنية العضلية (IMAT) هي مستودع دهني خارج الرحم يقيم بين ألياف العضلات وحولها1. كما هو موضح بالتفصيل في مراجعة حديثة أجراها Goodpaster et al. ، يمكن اكتشاف IMAT باستخدام التصوير المقطعي المحوسب عالي الدقة (CT) والتصوير بالرنين المغناطيسي (MRI) (الشكل 1A ، B) ويوجد حول وداخل ألياف العضلات في جميع أنحاء الجسمبأكمله 1. تختلف كمية IMAT اختلافا كبيرا بين الأفراد وتتأثر بمؤشر كتلة الجسم والعمر والجنس والعرق وقلة الحركة2،3،4. علاوة على ذلك ، يظهر ترسب IMAT بشكل شائع في الحالات المرضية المرتبطة بتنكس العضلات5 ، وقد وثقت العديد من الدراسات زيادة كتلة IMAT لدى الأفراد المصابين بالسمنة ومرض السكري من النوع 2 ومتلازمة التمثيل الغذائي ومقاومة الأنسولين6،7،8،9. ومع ذلك ، فإن الخصائص الخلوية والبيولوجية ل IMAT بدأت للتو في التفكك. وقد شكلت إمكانية الوصول المحدودة والاختلاف في مواقع IMAT ومحتواها في جميع أنحاء الجسم تحديا لجمع العينات من هذا المستودع الدهنيالفريد 2. علاوة على ذلك ، يتم "تلوث" العينات بسهولة بالعضلات الهيكلية (SM) عند جمعها ، مما يجعل الفصل بين المساهمة البيولوجية من الأنسجة المختلفة أمرا صعبا (الشكل 1C). وتحقيقا لهذه الغاية ، فإن تسلسل الحمض النووي الريبي أحادي النوى (snRNA-seq) ، الذي اكتسب اهتماما كبيرا خلال العقد الماضي ، يعمل كمنهجية مثالية للسماح بفصل أنماط التعبير الجيني المشتقة من IMAT و SM بدقة خلية واحدة. علاوة على ذلك ، فإن عزل النوى مفيد بشكل خاص للأنسجة الدهنية بسبب الخلايا الشحمية الكبيرة المحملة بالدهون ، والتي يستحيل فصلها إلى تعليق خلية واحدة دون المساس بسلامة الخلايا. أخيرا ، تمتلك هذه التقنية القدرة على اكتشاف علامات جديدة للخلايا الشحمية الخاصة ب IMAT والكشف عن تكوين ووجود مجموعات مختلفة من الخلايا السلفية ، وكذلك دراسة تباين تكوين الخلية في الظروف المرضية والطبيعية.
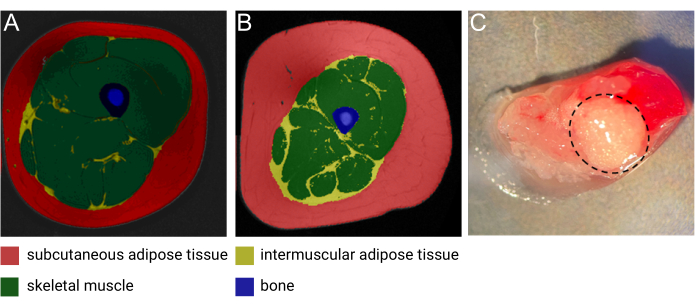
الشكل 1: صور IMAT. صورة الرنين المغناطيسي التمثيلي (MRI) ل IMAT من (أ) أنثى نحيفة في منتصف العمر و (ب) ذكر في منتصف العمر مصاب بالسمنة. الأحمر: الأنسجة الدهنية تحت الجلد ، الأصفر: الأنسجة الدهنية العضلية ، الأخضر: العضلات الهيكلية ، الأزرق: العظام. الصورة مجاملة من هيذر كورنيل ، معهد AdventHealth للبحوث الانتقالية. (C) عينة أنسجة جديدة مع IMAT (محاطة بخط أسود متقطع). الصورة مقدمة من ميغان هوبف ، معهد AdventHealth للبحوث الانتقالية وبريان بيرجمان ، جامعة كولورادو. تم تعديل هذا الرقم بإذن من Goodpaster et al.1. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
تم نشر عدد من الدراسات من صناعة الثروة الحيوانية التي تبحث في رخامي اللحوم (IMAT على وجه الخصوص) في الخنازير والدجاج والماشية باستخدام خلية واحدة (sc) و snRNA-seq10. حددت هذه الدراسات العديد من المجموعات السكانية الفرعية للخلايا الشحمية وعلامات الخلايا السلفية المحتملة ل IMAT11،12،13. ومع ذلك ، ما إذا كانت هذه التراكيب الخلوية تترجم إلى IMAT البشري غير معروف. على حد علمنا ، نظرت دراسة واحدة فقط في عدم التجانس الخلوي للعضلات البشرية مع تسلل دهني ، تم الحصول عليه من المرضى الذكور المصابين بالتهاب المفاصل العظمي الوركي ، باستخدام snRNA-seq14. أبلغ الباحثون عن عدد صغير من الخلايا الشحمية والعديد من المجموعات الفرعية السلفية الليفية (FAP) ضمن عدد كبير من النوىالعضلية 14. دراستنا هي الأولى التي تطور طريقة لاستجواب IMAT مباشرة تشريح يدويا من العضلات البشرية للتكوين الخلوي باستخدام snRNA-seq.
الأهم من ذلك ، يجب تخصيص بروتوكولات snRNA-seq للأنسجة المحددة التي تمت دراستها ، حيث أن كمية الأنسجة المتاحة والخصائص الفيزيائية للأنسجة المحددة ستحدد خطوات المعالجة المثلى. عادة ما يكون إنتاج الأنسجة ل IMAT صغيرا ، وغالبا لا يتجاوز 50 مجم ، حتى عند إجراء الخزعات الموجهة بالموجات فوق الصوتية. وبالتالي ، فإن المعالجة الصحيحة لهذا النسيج النادر أمر ضروري. نعتقد أن هذا البروتوكول سيكون بمثابة مورد قيم للباحثين الذين يدرسون IMAT البشرية.
Access restricted. Please log in or start a trial to view this content.
Protocol
كانت العينة المستخدمة في هذا البروتوكول جزءا من دراسة العضلات والتنقل والشيخوخة (SOMMA)15 ، والتي تمت الموافقة عليها من قبل مجلس المراجعة المؤسسية لمجموعة Western IRB-Copernicus Group (WCG) وتم تنفيذها وفقا لإعلان هلسنكي. قدم المشاركون موافقة خطية مستنيرة على مشاركتهم في الدراسة.
ملاحظة: تم تكييف هذا البروتوكول من بروتوكول سابق باستخدام 100 ملغ من الأنسجة الدهنية تحت الجلد في البطن البشري على منصة نانوويل16. تم تحسين البروتوكول الحالي ل 50 ملغ من IMAT البشري وإعداد المكتبة باستخدام منصة قائمة على القطيرات. قد تكون هناك حاجة إلى مزيد من تحسين هذا البروتوكول لعزل النوى من IMAT غير البشرية أو غيرها من المستودعات الدهنية.
1. تحضير المخازن المؤقتة والكواشف (الجدول 1 والجدول 2)
ملاحظة: قم بإعداد المخازن المؤقتة طازجة في يوم التجربة ولا تعيد استخدامها.
- قم بتبريد جهاز الطرد المركزي مسبقا إلى 4 درجات مئوية.
- تحضير محلول التجانس ووسط عزل النوى.
- احصل على دلاء من الثلج وأنابيب مخروطية مسبقة التبريد 2 × 15 مل.
- امزج جميع الكواشف الخاصة بمخزن التجانس (HB) في أنبوب مخروطي سعة 15 مل بالترتيب المدرج في الجدول 1. ابق على الجليد. تخلط عن طريق دوامة.
- امزج جميع كواشف وسط عزل النوى (NIM) في أنبوب مخروطي سعة 15 مل في القائمة المرتبة في الجدول 2. ابق على الجليد. تخلط عن طريق دوامة.
- قم بإعداد 10٪ Triton-X بإضافة 100 ميكرولتر من Triton X-100 إلى 900 ميكرولتر من الماء الخالي من النيوكلياز. دوامة لضمان الخلط السليم. يحفظ في درجة حرارة الغرفة (RT).
| الكاشف | الحجم (ميكرولتر) | التركيز النهائي (mM) | |
| 1x | 2x | ||
| 1 م مغل2 | 10 | 20 | 5 |
| 1 متر تريس العازلة ، درجة الحموضة 8.0 | 20 | 40 | 10 |
| 2 مليون كيلو لتر | 25 | 50 | 25 |
| 1.5 م سكروز (-4درجة مئوية) | 334 | 668 | 250 |
| 1 مللي متر DTT | 2 | 4 | 0.001 (~ 1 ميكرومتر) |
| 100x مثبطات الأنزيم البروتيني | 20 | 40 | 1x |
| سوبراسين 20 وحدة / ميكرولتر | 40 | 80 | 0.4 وحدة / ميكرولتر |
| ماء خال من النيوكلياز | 1549 | 3098 | - |
| الحجم الكلي | 2000 | 4000 | - |
الجدول 1: المخزن المؤقت للتجانس (HB). ابق على الجليد. تخلط عن طريق دوامة.
| الكاشف | الحجم (ميكرولتر) | التركيز النهائي (mM) | |
| 1x | 2x | ||
| EDTA | 0.4 | 0.8 | 0.1 |
| مثبط ريبولوك RNAse (40U / μL) | 40 | 80 | 0.8 وحدة / ميكرولتر |
| 1٪ BSA-PBS (-/-) | 1959.6 | 3919.2 | - |
| الحجم الكلي | 2000 | 4000 | - |
الجدول 2: وسط عزل النوى (NIM). ابق على الجليد. تخلط عن طريق دوامة.
2. سحق الأنسجة المجمدة (الشكل 2 أ)
- قم بإعداد محطة العمل للتجانس.
- املأ العلبة بالنيتروجين السائل (LN2).
تنبيه: عند العمل مع LN2 ، ارتد دائما نظارات واقية وقفازات تبريد. - احصل على 2x هاون ، 1x مدقة ، 1x ملعقة مغرفة صغيرة ، 1x ارتداد زجاجي ، و 1x مدقة من الفولاذ المقاوم للصدأ للحارس التلقائي.
- قم بإعداد الحارس التلقائي.
- املأ دورقا بالثلج وقم بتبريد ارتداد الزجاج مسبقا.
- املأ العلبة بالنيتروجين السائل (LN2).
- املأ مدافع الهاون (التي تحتوي على المدقة والملعقة) ب LN2 لتبريد الأدوات. دع LN2 يتبخر ويكرر.
- أثناء تبريد الأدوات ، أضف 1 مل من HB إلى ارتداد الزجاج.
- املأ كلا الهاون ب LN2 مرة أخيرة واسكب عينة IMAT 50 مجم في إحدى قذائف الهاون.
- سحق IMAT باستخدام المدقة عن طريق الضغط عليها برفق على قطعة الأنسجة لتقسيمها إلى قطع صغيرة. تأكد من سحق جميع القطع.
- سوف يتبخر LN2 ببطء أثناء سحق الأنسجة. عندما يتم سحق الأنسجة بشكل صحيح ، ولا يزال هناك 1/4 - 1/2 من هاون LN2 الأيسر ، قم بإمالة الهاون نحو شفة الهاون لجمع الأنسجة المسحوقة بواسطة الشفة. دع LN2 يتبخر تماما.
- مباشرة بعد تبخر آخر LN2 ، اغرف الأنسجة المسحوقة في الارتداد الزجاجي الذي يحتوي على 1 مل من HB.
3. تجانس الأنسجة المسحوقة
- تجانس الأنسجة المسحوقة باستخدام الحارس التلقائي. اجعل الزجاج يرتد لأعلى ولأسفل في مدقة الفولاذ المقاوم للصدأ لمدة 10 ضربات في الاتجاه الأمامي ، متبوعا ب 10 ضربات في الاتجاه العكسي.
- تأكد من أن المحلول غائم بعد التجانس ولا يحتوي على قطع مرئية من الأنسجة. غالبا ما يتوقع اللون الوردي الفاتح بسبب التلوث بالأنسجة العضلية.
- انقل التجانس إلى أنبوب منخفض الارتباط سعة 1.7 مل مبرد مسبقا على الجليد.
- استخدم 400 ميكرولتر من HB لشطف الارتداد للتأكد من نقل جميع المواد وإضافتها إلى الأنبوب.
ملاحظة: يمكن معالجة عينتين في وقت واحد. للقيام بذلك ، ضاعف كمية HB و NIM. سحق وتجانس عينة نسيج واحدة وبعد ذلك مباشرة سحق وتجانس عينة الأنسجة الثانية لتكون قادرة على أداء خطوات العزل والتنظيف بالتوازي.
4. عزل النوى وتنظيفها (الشكل 2 ب)
- أضف 14 ميكرولتر من Triton-X (10٪) إلى التجانس لتركيز 0.1٪.
- احتفظ بالأنبوب على الثلج وفي الظلام لمدة 10-15 دقيقة أثناء الدوامة كل 3 دقائق.
- قبل البلل مصفاة خلية واحدة 100 ميكرومتر ومصفاة خلية 40 ميكرومتر (لكل عينة) مع 100 ميكرولتر من RT DPBS لكل منها في أنبوب مخروطي 50 مل.
- تصفية التجانس من خلال مصفاة خلية 100 ميكرومتر.
- شطف أنبوب 1.7 مل مع 400 ميكرولتر من HB وتصفية من خلال مصفاة خلية 100 ميكرومتر.
- بعد ذلك ، قم بتصفية المحلول من خلال مصفاة الخلايا 40 ميكرومتر.
- انقل كمية متساوية من المحلول إلى أنبوبين منخفضي الربط سعة 1.7 مل مبردين مسبقا يقابلان ~ 900 ميكرولتر في كل أنبوب.
- أجهزة الطرد المركزي الأنابيب لمدة 10 دقائق عند 2700 × جم عند 4 درجات مئوية. يجب أن يكون هناك بيليه صغير مرئي بعد الطرد المركزي.
- قم بإزالة وتجاهل الطبقة الدهنية العلوية والمادة الطافية المتبقية ، تاركا محلول ~ 50 ميكرولتر من الأنبوب الأول.
- كرر للأنبوب الثاني.
- أعد تعليق الحبيبات تماما في الأنبوب الأول عن طريق سحب القطر برفق لأعلى ولأسفل 20x ونقله إلى أنبوب جديد منخفض الربط سعة 1.7 مل. تجنب خلق فقاعات.
- كرر هذا الإجراء للأنبوب الثاني وانقل المحلول المعاد تعليقه إلى نفس الأنبوب.
- أضف 500 ميكرولتر من NIM واخلطها مع الماصة.
- جهاز طرد مركزي الأنبوب بميزان عند 1000 × جم لمدة 10 دقائق عند 4 درجات مئوية.
- قم بإزالة المادة الطافية ، واترك ~ 50 ميكرولتر ، وماصة برفق لأعلى ولأسفل حتى يتم إعادة استخدام الحبيبات. اختياريا ، انقل الحبيبات المعاد تعليقها إلى أنبوب نظيف جديد إذا بقيت بعض بقايا الدهون على جانب الأنبوب.
- أضف 200 ميكرولتر من NIM واخلطها عن طريق الماصة.
5. تلطيخ النوى وعدها (الشكل 2 ج والشكل 3)
ملاحظة: لتسهيل العد ، قم بإعداد بروتوكول "عد النوى" على عداد خلية آلي ، حيث يمكن أن يؤثر ضبط المجال الساطع وقنوات DAPI على العد بشكل كبير. اضبط القنوات بحيث يتم التقاط النوى فقط وليس الحطام. تأكد من أن قناة الحقل الساطع تشير فقط إلى "الكائنات" التي تحتوي أيضا على صبغة DAPI.
- أضف 1 قطرة من محلول تلطيخ الخلايا الحية واتركه في الظلام ، على الجليد ، لمدة 15 دقيقة.
- قم بتصفية المحلول من خلال مصفاة خلية 30 ميكرومتر.
- امزج محلول النوى عن طريق السحب وأضف 10 ميكرولتر من المحلول إلى شريحة غرفة عد الخلايا.
- عد النوى باستخدام عداد الخلايا الآلي.
ملاحظة: التركيز الأمثل هو 1000 نواة / ميكرولتر المقابلة ل 1.0 × 106 / مل.- تأكد من عدم وجود كتل من النوى ، لأن هذا قد يسد الشريحة لتوليد قطرات أحادية النوى (الشكل 3).
- إذا لم يكن تركيز النوى مرتفعا بدرجة كافية ، فقم بتدوير المحلول لأسفل عند 1000 × جم لمدة 10 دقائق عند 4 درجات مئوية للحصول على حبيبات ، وإزالة المادة الطافية ، وإعادة التعليق في حجم أصغر.
- إذا كانت درجة الحطام في المحلول عالية ، فأعد تعليق محلول النوى في حجم أكبر من NIM (أي 1 مل) وقم بالتصفية من خلال مصفاة خلية 30 ميكرومتر مرة أخرى. ثم قم بالدوران لأسفل عند 1000 × جم لمدة 10 دقائق عند 4 درجات مئوية وأعد التعليق بالحجم المناسب بالنسبة لتركيز النوى.
- بعد الحصول على تركيز النوى ، انتقل مباشرة إلى الخطوة الأولى في إعداد المكتبة.
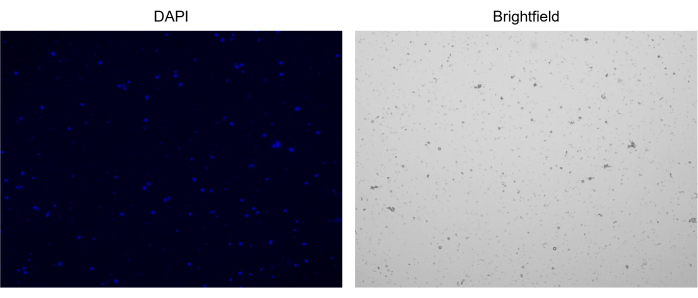
الشكل 3: تلطيخ النوى المعزولة. صورة من عداد الخلية للنوى الملطخة ب NucBlue / DAPI (الصورة اليسرى) وصورة المجال الساطع المقابلة (الصورة اليمنى). يتضح وجود كميات صغيرة من الحطام في صورة المجال الساطع. لا يحتوي عداد الخلايا الآلي المستخدم هنا على خيار لتضمين أشرطة المقياس. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
6. إعداد المكتبة ومعلمات التسلسل
- ارجع إلى بروتوكول شامل لإعداد المكتبة باستخدام نهج النوى المفردة القائم على القطيرات المتاح على صفحة الويب الخاصة بالمزود17.
- استهدف استعادة 10000 نواة مستهدفة. ومع ذلك، بالنسبة للعينات ذات المستوى العالي من الحطام أو النوى الهشة، يتوقع استرداد عدد أقل من النوى.
- قم بتخزين العينات في درجة حرارة 4 درجات مئوية لمدة تصل إلى 72 ساعة بعد الخطوة 2.3. في بروتوكول إعداد المكتبة للجمع بين معالجة المزيد من العينات بالتوازي. قم بذلك عن طريق معالجة عينتين حتى الخطوة 2.3 في يومين متتاليين ، وفي اليوم الثالث ، قم بمعالجة العينات ال 4 معا من الخطوة 3 وما بعدها في بروتوكول إعداد المكتبة.
- معلمات التسلسل: تسلسل على منصة تسلسل تهدف إلى 50000 قراءة مزدوجة النهاية لكل نواة.
ملاحظة: تم تسلسل البيانات المقدمة في هذا البروتوكول على منصة NovaSeq 6000 ، بهدف الحصول على 50000 قراءة مزدوجة النهاية لكل نواة.
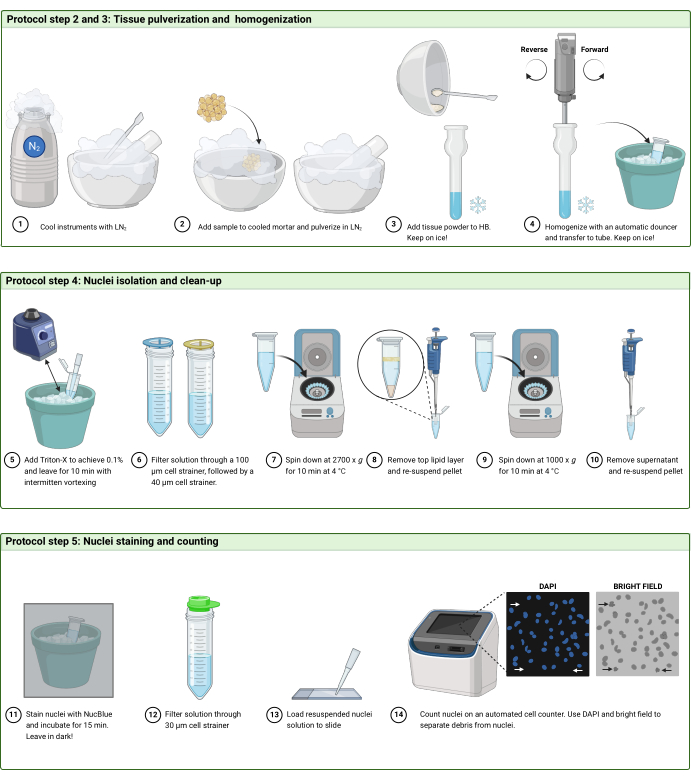
الشكل 2: سير عمل البروتوكول. رسم تخطيطي لسير العمل في (أ) الخطوتين 2 و3 و(ب) الخطوة 4 و(ج) الخطوة 5 من البروتوكول. تم إنشاء الرقم مع BioRender.com. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
7. معالجة البيانات وتحليلها
ملاحظة: في هذا البروتوكول ، يتم تقديم بعض البرامج الموصى بها وحزم R المستخدمة لمعالجة بيانات التسلسل الناتجة بإيجاز ، مع التركيز على الخطوات بعد المعالجة المسبقة الأولية (الجدول 3). توفر هذه الدراسة مقاييس عامة لمراقبة الجودة (QC) ومثالا على تقريب وإسقاط مشعب موحد (UMAP) في الشكل 4. ومع ذلك ، فإن الوصف المتعمق لتحليل المعلوماتية الحيوية خارج نطاق هذا البروتوكول. لذلك ، يمكن للقراء الرجوع إلى المراجعة الأخيرة حول أفضل الممارسات لتحليل الخلية الواحدة بواسطة Heumos et al.18.
- المعالجة المسبقة لبيانات التسلسل
- قم بتعيين النوى المفردة المقروءة إلى الجينوم المرجعي البشري GRCh38.
- تضمين قراءات intron في العد.
- قم بإجراء مراقبة الجودة وتصفية البيانات باستخدام حزمة Seurat R 19.
- احسب درجة تعقيد الخلية بقسمة عدد الجينات المكتشفة في عدد log(10) من القراءات المكتشفة.
- ارسم أهم مقاييس مراقبة الجودة باستخدام مخطط تكراري أو مخطط كمان ، بما في ذلك عدد الجينات المكتشفة لكل نواة ، ونسبة قراءة الميتوكوندريا ، ودرجة تعقيد الخلية.
- قم بتصفية النوى التي تحتوي على أقل من 200 أو أكثر من 10000 جين لكل نواة ، وأكثر من 10٪ من قراءات الميتوكوندريا ، ودرجة تعقيد أقل من 0.8.
- تطبيع البيانات وإجراء تقليل الأبعاد.
- استخدم الدالة SCTransform من Seurat لتطبيع البيانات باستخدام 2000 معلم متغير.
- بيانات الكتلة باستخدام الدالات التالية من حزمة Seurat R: RunPCA و FindNeighbors و FindClusters و RunUMAP.
- ارسم UMAP لتصور تجميع البيانات.
- تصفية الثنائيات المتوقعة باستخدام حزمة DoubletFinder R 20 وإعادة تجميع البيانات.
- قم بالتعليق على المجموعات باستخدام علامات الجينات المعروفة لأنواع الخلايا المتوقع وجودها في الأنسجة (نهج خاضع للإشراف) أو بناء على أفضل 5 جينات يتم التعبير عنها بشكل تفاضلي بين المجموعات (نهج غير خاضع للإشراف).
- استخدم decontX21لتحديد درجة تلوث الحمض النووي الريبي المحيط ولضبط مصفوفة التعبير الجيني للحمض النووي الريبي المحيط.
- قم بتضمين مصفوفة الجينات الخام كخلفية.
- احفظ كائن Seurat للاستكشاف المستقبلي للبيانات.
ملاحظة: رمز مراقبة الجودة وتحليل التجميع متاح في الملف التكميلي 1.
| حزم البرامج / R المستخدمة في سير عمل البيانات | البرامج / الحزم البديلة | خطوة المعالجة |
| سيل رينجر | ستارسولو ، كاليستو | التشذيب والمحاذاة ورسم الخرائط |
| سورات | SingleCellExperiment, Cellranger | مراقبة الجودة والتحليل واستكشاف البيانات |
| دوبليت فايندر | scds ، scdblFinder ، فرك | الكشف المزدوج |
| ديكونتكس | سوبكس، سيلبندر | تعديل الحمض النووي الريبي المحيط |
الجدول 3: برامج/أدوات لسير عمل البيانات.
Access restricted. Please log in or start a trial to view this content.
النتائج
تم تصميم سير العمل هذا لتوجيه معالجة عينات IMAT البشرية المجمدة للحصول على ملفات تعريف التعبير الجيني بدقة نواة واحدة ، مما يتيح تحديد نوع الخلية. هنا ، يتم تقديم عينة تمثيلية واحدة من IMAT من أحد المشاركين في دراسة SOMMA.
تتمثل الخطوة الأولى في أي تحليل لبيانات snRNA-seq في تقييم جودة ...
Access restricted. Please log in or start a trial to view this content.
Discussion
هناك العديد من التحديات المتأصلة للعمل مع IMAT. بالإضافة إلى إمكانية الوصول المحدودة ، غالبا ما يكون إنتاج مواد العينة نادرا جدا ، ويكاد يكون من المستحيل تجنب "تلوث" العضلات الهيكلية. للحصول على أفضل عينة ذات جودة ، يجب على المرء اختراق اللفافة العضلية عند إدخال إبرة الخزعة (للتأكد من عدم جمع...
Access restricted. Please log in or start a trial to view this content.
Disclosures
ليس لدى المؤلفين ما يكشفون عنه.
Acknowledgements
يود المؤلفون أن يشكروا بريان بيرجمان ، دكتوراه في جامعة كولورادو لتقديمه صورة خزعة IMAT في الشكل 1C من دراسة MoTrIMAT (R01AG077956). نحن ممتنون لدراسة العضلات والتنقل والشيخوخة التي توفر عينة IMAT التي تظهر منها البيانات في قسم النتائج التمثيلية. قام المعهد الوطني للشيخوخة (NIA) بتمويل دراسة العضلات والتنقل والشيخوخة (SOMMA; R01AG059416) ودراساتها المساعدة SOMMA AT (R01AG066474) و SOMMA Knee OA (R01AG070647). تم تمويل دعم البنية التحتية للدراسة جزئيا من قبل NIA Claude D. Pepper Older American Independence Centers في جامعة بيتسبرغ (P30AG024827) وجامعة ويك فورست (P30AG021332) ومعاهد العلوم السريرية والانتقالية ، بتمويل من المركز الوطني لتطوير العلوم الانتقالية ، في جامعة ويك فورست (UL1 0TR001420).
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 0.2 µm corning syringe filters | Millipore Sigma | CLS431229 | |
| 1.7 mL DNA LoBind tubes | Eppendorf | 22431021 | low-bind tubes |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 100x protease inhibitor | Thermo Fisher Scientific | 78437 | |
| 10X Magnetic Separator | 10X Genomics | 230003 | |
| 10X Vortex Adapter | 10X Genomics | 330002 | |
| 15 mL canonical tubes | Sarstedt | 6,25,54,502 | |
| 2100 Bioanalyzer | Agilent | G2939BA | |
| 50 mL conical tubes | Sarstedt | 6,25,47,254 | |
| CellRanger | Genomics | N/A | |
| Chromium iX accesory kit | 10X Genomics | PN1000323 | |
| Chromium iX Controller | 10X Genomics | PN1000326 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | PN1000127 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1 | 10X Genomics | PN1000129 | |
| Chromium Next GEM Single Cell GEM Kit v3.1 | 10X Genomics | PN1000130 | |
| Countess 3 Automated Cell Counter | Thermo Fisher Scientific | AMQAX2000 | Automated cell counter |
| Countess cell counting chamber slides | Thermo Fisher Scientific | C10228 | |
| DoubletFinder | R | N/A | |
| DPBS (no calcium, no magnesium) | Thermo Fisher Scientific | 14190144 | |
| DTT | Thermo Fisher Scientific | R0861 | |
| Dual Index Kit TT Set A, 96 rxns | 10X Genomics | PN1000215 | |
| Dynabeads MyOne SILANE | 10X Genomics | PN2000048 | |
| Falcon 100 µm Cell strainer | Corning Life Science | 352360 | |
| Falcon 40 µm Cell strainer | Corning Life Science | 352340 | |
| Glycerin (glycerol), 50% (v/v) Aqueous Solution | Ricca Chemical Company | 3290-32 | |
| KCL | Thermo Fisher Scientific | AM9640G | |
| Library Construction Kit v3.1 | 10X Genomics | PN1000196 | |
| MACS SmartStrainers (30µm) | Miltenyi Biotec | 130-098-458 | |
| Mastercycler Nexus Gradient Thermal cycler | Eppendorf | 6331000017 | |
| MgCl2 | Ambion | AM9530G | |
| Mortar and pestel | Health care logistics | 14075 | |
| NucBlue Live Ready Probes Reagent | Thermo Fisher Scientific | R37605 | |
| Nuclease Free Water (not DEPC treated) | Thermo Fisher Scientific | AM9930 | |
| Probumin Bovine Serum Albumin Fatty Acid Free, Powder | Sigma-Aldrich | 820024 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Ribolock RNAse inhibitor | Thermo Fisher Scientific | EO0382 | |
| Seurat | R | N/A | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SUPERasin 20 U/µL | Thermo Fisher Scientific | AM2695 | |
| ThermoMixer C | Eppendorf | 5382000015 | |
| Tissue homogenizer | Glass-Col | 099C K54 | |
| Tris buffer pH 8.0 | Thermo Fisher Scientific | AM9855G | |
| Triton X-100 | Thermo Fisher Scientific | AC327372500 | |
| UltraPure 0.5M EDTA pH 8.0 | Gibco | 15575020 |
References
- Goodpaster, B. H., Bergman, B. C., Brennan, A. M., Sparks, L. M. Intermuscular adipose tissue in metabolic disease. Nat Rev Endocrinol. 19 (5), 285-298 (2023).
- Sparks, L. M., Goodpaster, B. H., Bergman, B. C. The metabolic significance of intermuscular adipose tissue: Is IMAT a friend or a foe to metabolic health. Diabetes. 70 (11), 2457-2467 (2021).
- Gallagher, D., et al. Adipose tissue in muscle: A novel depot similar in size to visceral adipose tissue. Am J Clin Nutr. 81 (4), 903-910 (2005).
- Manini, T. M., et al. Reduced physical activity increases intermuscular adipose tissue in healthy young adults. Am J Clin Nutr. 85 (2), 377-384 (2007).
- Addison, O., Marcus, R. L., LaStayo, P. C., Ryan, A. S. Intermuscular fat: A review of the consequences and causes. Int J Endocrinol. 2014, 309570(2014).
- Goodpaster, B. H., et al. Obesity, regional body fat distribution, and the metabolic syndrome in older men and women. Arch Intern Med. 165 (7), 777-783 (2005).
- Goodpaster, B. H., Thaete, F. L., Kelley, D. E. Thigh adipose tissue distribution is associated with insulin resistance in obesity and in type 2 diabetes mellitus. Am J Clin Nutr. 71 (4), 885-892 (2000).
- Goodpaster, B. H., et al. Association between regional adipose tissue distribution and both type 2 diabetes and impaired glucose tolerance in elderly men. Diabetes Care. 26 (2), 372-379 (2003).
- Sachs, S., et al. Intermuscular adipose tissue directly modulates skeletal muscle insulin sensitivity in humans. Am J Physiol Endocrinol Metab. 316 (5), E866-E879 (2019).
- Ford, H., Liu, Q., Fu, X., Strieder-Barboza, C. White adipose tissue heterogeneity in the single-cell era: From mice and humans to cattle. Biology (Basel). 12 (10), 1289(2023).
- Wang, L., et al. Single-nucleus and bulk RNA sequencing reveal cellular and transcriptional mechanisms underlying lipid dynamics in high marbled pork. NPJ Sci Food. 7 (1), 23(2023).
- Li, J., et al. Identification of diverse cell populations in skeletal muscles and biomarkers for intramuscular fat of chicken by single-cell RNA sequencing. BMC Genomics. 21 (1), 752(2020).
- Lyu, P., Qi, Y., Tu, Z. J., Jiang, H. Single-cell RNA sequencing reveals heterogeneity of cultured bovine satellite cells. Front Genet. 12, 742077(2021).
- Fitzgerald, G., et al. MME+ fibro-adipogenic progenitors are the dominant adipogenic population during fatty infiltration in human skeletal muscle. Commun Biol. 6 (1), 111(2023).
- Cummings, S. R., et al. The study of muscle, mobility and aging (SOMMA): A unique cohort study about the cellular biology of aging and age-related loss of mobility. J Gerontol A Biol Sci Med Sci. 78 (11), 2083-2093 (2023).
- Whytock, K. L., et al. Isolation of nuclei from frozen human subcutaneous adipose tissue for full-length single-nuclei transcriptional profiling. STAR Protoc. 4 (1), 102054(2023).
- 10x Genomics. Chromium Single Cell 3' Reagent Kits User Guide (v3.1 Chemistry Dual Index), Document Number CG000315 RevE. , Available from: https://cdn.10xgenomics.com/image/upload/v1668017706/support-documents/CG000315_ChromiumNextGEMSingleCell3-_GeneExpression_v3.1_DualIndex__RevE.pdf (2022).
- Heumos, L., et al. Best practices for single-cell analysis across modalities. Nat Rev Genet. 24 (1), 550-572 (2023).
- Hao, Y., et al. Dictionary learning for integrative, multimodal and scalable single-cell analysis. Nat Biotechnol. 42 (2), 293-304 (2023).
- McGinnis, C. S., Murrow, L. M., Gartner, Z. J. DoubletFinder: Doublet detection in single-cell RNA sequencing data using artificial nearest neighbors. Cell Syst. 8 (4), 329-337 (2019).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (2), 57(2020).
- Common considerations for quality control filters for single cell RNA-seq data. 10X Genomics. , Available from: https://www.10xgenomics.com/analysis-guides/common-considerations-for-quality-control-filters-for-single-cell-rna-seq-data (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Mol Syst Biol. 15 (6), e8746(2019).
- Emont, M. P., et al. A single-cell atlas of human and mouse white adipose tissue. Nature. 603 (7903), 926-933 (2022).
- Hildreth, A. D., et al. Single-cell sequencing of human white adipose tissue identifies new cell states in health and obesity. Nat Immunol. 22 (5), 639-653 (2021).
- Whytock, K. L., et al. Single cell full-length transcriptome of human subcutaneous adipose tissue reveals unique and heterogeneous cell populations. iScience. 25 (8), 104772(2022).
- Probst, V., et al. Benchmarking full-length transcript single cell mRNA sequencing protocols. BMC Genomics. 23 (1), 860(2022).
- CG000148 Rev A Technical Note - Resolving cell types as a function of read depth and cell number. Technical note. 10X Genomics. , Available from: https://assets.ctfassets.net/an68im79xiti/6gDArDPBTOg4IIkYEO2Sis/803be2286bb a5ca67f353e6baf68d276/CG000148_10x_Technical _Note_Resolving_Cell_Types_as_Function_of_ Read_Depth_Cell_Number_RevA.pdf (2018).
- Gupta, A., et al. Characterization of transcript enrichment and detection bias in single-nucleus RNA-seq for mapping of distinct human adipocyte lineages. Genome Res. 32 (2), 242-257 (2022).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648(2018).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. J Am Soc Nephrol. 30 (1), 23-32 (2019).
- Kim, N., Kang, H., Jo, A., Yoo, S. -A., Lee, H. -O. Perspectives on single-nucleus RNA sequencing in different cell types and tissues. J Pathol Transl Med. 57 (1), 52-59 (2023).
- Avila Cobos, F., Alquicira-Hernandez, J., Powell, J. E., Mestdagh, P., De Preter, K. Benchmarking of cell type deconvolution pipelines for transcriptomics data. Nat Commun. 11 (1), 5650(2020).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved