Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Isolement de noyaux à partir de tissu adipeux intermusculaire humain et séquençage d’ARN à noyaux uniques en aval
Dans cet article
Résumé
La biologie du tissu adipeux intermusculaire (IMAT) est largement inexplorée en raison de l’accessibilité limitée des tissus humains. Ici, nous présentons un protocole détaillé pour l’isolement des noyaux et la préparation de banques d’IMAT humain congelé pour le séquençage de l’ARN à noyaux uniques afin d’identifier la composition cellulaire de ce dépôt adipeux unique.
Résumé
Le tissu adipeux intermusculaire (IMAT) est un dépôt adipeux relativement peu étudié situé entre les fibres musculaires. La teneur en IMAT augmente avec l’âge et l’IMC et est associée à des maladies dégénératives métaboliques et musculaires ; cependant, une compréhension des propriétés biologiques de l’IMAT et de son interaction avec les fibres musculaires environnantes fait cruellement défaut. Ces dernières années, le séquençage de l’ARN unicellulaire et nucléimétrique nous a fourni des atlas spécifiques au type cellulaire de plusieurs tissus humains. Cependant, la composition cellulaire de l’IMAT humaine reste largement inexplorée en raison des défis inhérents à son accessibilité à partir de la collecte de biopsies chez l’homme. En plus de la quantité limitée de tissus collectés, le traitement de l’IMAT humain est compliqué en raison de sa proximité avec les tissus musculaires squelettiques et les fascias. La nature chargée en lipides des adipocytes le rend incompatible avec l’isolement unicellulaire. Par conséquent, le séquençage de l’ARN à noyaux uniques est optimal pour obtenir une transcriptomique de haute dimension à une résolution cellulaire unique et offre le potentiel de découvrir la biologie de ce dépôt, y compris la composition cellulaire exacte de l’IMAT. Ici, nous présentons un protocole détaillé pour l’isolement des noyaux et la préparation de banques d’IMAT humain congelé pour le séquençage de l’ARN à noyaux uniques. Ce protocole permet le profilage de milliers de noyaux à l’aide d’une approche basée sur les gouttelettes, offrant ainsi la capacité de détecter des types de cellules rares et peu abondantes.
Introduction
Le tissu adipeux intermusculaire (IMAT) est un dépôt adipeux ectopique résidant entre et autour des fibres musculaires1. Comme décrit en détail dans une étude récente de Goodpaster et al., l’IMAT peut être détecté à l’aide de la tomodensitométrie (TDM) à haute résolution et de l’imagerie par résonance magnétique (IRM) (Figure 1A, B) et se trouve autour et dans les fibres musculaires de tout le corps1. La quantité d’IMAT varie considérablement d’un individu à l’autre et est influencée par l’IMC, l’âge, le sexe, la raceet la sédentarité2,3,4. De plus, le dépôt d’IMAT est couramment observé dans les conditions pathologiques associées à la dégénérescence musculaire5, et de nombreuses études ont documenté une augmentation de la masse IMAT chez les personnes atteintes d’obésité, de diabète de type 2, de syndrome métabolique et de résistance à l’insuline 6,7,8,9. Néanmoins, les propriétés cellulaires et biologiques de l’IMAT commencent à peine à être démêlées. L’accessibilité limitée et la variation des emplacements et du contenu de l’IMAT dans l’ensemble du corps ont rendu difficile la collecte d’échantillons de ce dépôt adipeux2 unique. De plus, les échantillons sont facilement « contaminés » par les muscles squelettiques (SM) lors de la collecte, ce qui rend difficile la séparation entre la contribution biologique des différents tissus (Figure 1C). À cette fin, le séquençage de l’ARN à noyau unique (snRNA-seq), qui a fait l’objet d’une attention considérable au cours de la dernière décennie, constitue une méthodologie idéale pour permettre la séparation des modèles d’expression génique dérivés de l’IMAT et du SM avec une résolution unicellulaire. De plus, l’isolement des noyaux est particulièrement utile pour le tissu adipeux en raison des grands adipocytes chargés de lipides, qui sont impossibles à dissocier en suspension unicellulaire sans compromettre l’intégrité des cellules. Enfin, cette technologie a le potentiel de découvrir de nouveaux marqueurs d’adipocytes spécifiques d’IMAT et de découvrir la composition et la présence de différentes populations de cellules progénitrices, ainsi que d’étudier la variation de la composition cellulaire dans des conditions pathologiques et normales.
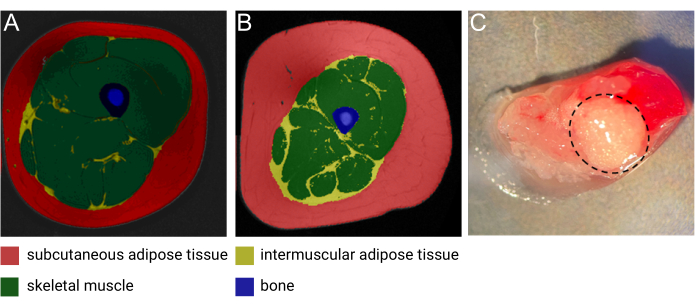
Figure 1 : Images de l’IMAT. Image représentative par résonance magnétique (IRM) de l’IMAT provenant (A) d’une femme maigre d’âge moyen et (B) d’un homme d’âge moyen obèse. Rouge : tissu adipeux sous-cutané, jaune : tissu adipeux intermusculaire, vert : muscle squelettique, bleu : os. Image reproduite avec l’aimable autorisation de Heather Cornnell, Institut de recherche translationnelle AdventHealth. (C) Échantillon de tissu frais avec IMAT (entouré d’une ligne noire pointillée). Image reproduite avec l’aimable autorisation de Meghan Hopf, AdventHealth Translational Research Institute et Bryan Bergman, Université du Colorado. Cette figure a été modifiée avec la permission de Goodpaster et al.1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Un certain nombre d’études ont été publiées dans l’industrie de l’élevage portant sur le persillage de la viande (IMAT en particulier) chez les porcs, les poulets et les bovins à l’aide de cellules uniques (sc) et de snRNA-seq10. Ces études ont permis d’identifier plusieurs sous-populations d’adipocytes et de marqueurs de cellules progénitrices potentielles d’IMAT 11,12,13 ; cependant, on ne sait pas si ces compositions cellulaires se traduisent par de l’IMAT humain. À notre connaissance, une seule étude s’est penchée sur l’hétérogénéité cellulaire du muscle humain avec infiltration graisseuse, obtenue chez des patients masculins atteints d’arthrose de la hanche, à l’aide du snRNA-seq14. Les chercheurs ont signalé une petite population d’adipocytes et plusieurs sous-populations de progéniteurs fibro-adipogéniques (PAF) au sein de la grande population de myonoyaux14. Notre étude est la première à développer une méthode pour interroger directement l’IMAT disséqué manuellement du muscle humain pour la composition cellulaire à l’aide du snRNA-seq.
Il est important de noter que les protocoles de séquençage de l’ARNsn doivent être personnalisés pour le tissu spécifique étudié, car la quantité de tissu disponible et les propriétés physiques du tissu spécifique dicteront les étapes de traitement optimales. Le rendement tissulaire pour l’IMAT est généralement faible, ne dépassant souvent pas 50 mg, même lors de la réalisation de biopsies guidées par échographie. Par conséquent, un traitement approprié de ce tissu rare est essentiel. Nous pensons que ce protocole constituera une ressource précieuse pour les chercheurs qui étudient l’IMAT humain.
Access restricted. Please log in or start a trial to view this content.
Protocole
L’échantillon utilisé pour ce protocole faisait partie de l’étude SOMMA)15, qui a été approuvée par le comité d’examen institutionnel de l’IRB-Copernicus Group (WCG) de l’Ouest et a été réalisée conformément à la Déclaration d’Helsinki. Les participants ont fourni un consentement éclairé écrit pour leur participation à l’étude.
REMARQUE : Ce protocole est adapté d’un protocole précédent utilisant 100 mg de tissu adipeux sous-cutané abdominal humain sur une plateforme basée sur des nanopuits16. Le protocole actuel est optimisé pour 50 mg d’IMAT humain et la préparation de banques à l’aide d’une plate-forme basée sur des gouttelettes. Une optimisation supplémentaire de ce protocole pour l’isolement des noyaux à partir d’IMAT non humains ou d’autres dépôts adipeux pourrait être nécessaire.
1. Préparation des tampons et des réactifs (Tableau 1 et Tableau 2)
REMARQUE : Préparez des tampons frais le jour de l’expérience et ne les réutilisez pas.
- Pré-refroidissez une centrifugeuse à 4 °C.
- Préparer le tampon d’homogénéisation et le milieu d’isolement des noyaux.
- Obtenez deux seaux de glace et pré-refroidissez 2 tubes coniques de 15 ml.
- Mélanger tous les réactifs pour le tampon d’homogénéisation (HB) dans un tube conique de 15 mL dans l’ordre indiqué au tableau 1. Restez sur la glace. Mélanger par vortex.
- Mélanger tous les réactifs pour milieu d’isolement des noyaux (MIN) dans un tube conique de 15 mL dans la liste ordonnée du tableau 2. Restez sur la glace. Mélanger par vortex.
- Préparez 10 % de Triton-X en ajoutant 100 μL de Triton X-100 à 900 μL d’eau sans nucléases. Vortex pour assurer un bon mélange. Conserver à température ambiante (RT).
| Réactif | Volume (μL) | Concentration finale (mM) | |
| 1x | 2 fois | ||
| 1 M MgCl2 | 10 | 20 | 5 |
| Tampon Tris de 1 m, pH 8,0 | 20 | 40 | 10 |
| 2 M KCl | 25 | 50 | 25 |
| 1,5 M de saccharose (-4°C) | 334 | 668 | 250 |
| 1mM TNT | 2 | 4 | 0,001 (~1 μM) |
| 100x inhibiteur de protéase | 20 | 40 | 1x |
| Superasine 20 U/μL | 40 | 80 | 0,4 U/μL |
| Eau sans nucléases | 1549 | 3098 | - |
| Total Volume | 2000 | 4000 | - |
Tableau 1 : Tampon d’homogénéisation (HB). Restez sur la glace. Mélanger par vortex.
| Réactif | Volume (μL) | Concentration finale (mM) | |
| 1x | 2 fois | ||
| EDTA | 0.4 | 0.8 | 0.1 |
| Inhibiteur de l’ARNase Ribolock (40U/μL) | 40 | 80 | 0,8 U/μL |
| 1 % BSA-PBS (-/-) | 1959.6 | 3919.2 | - |
| Total Volume | 2000 | 4000 | - |
Tableau 2 : Milieu d’isolement des noyaux (NIM). Restez sur la glace. Mélanger par vortex.
2. Pulvérisation de tissus congelés (figure 2A)
- Configurer le poste de travail pour l’homogénéisation.
- Remplissez le bidon d’azote liquide (LN2).
ATTENTION : lorsque vous travaillez avec LN2, portez toujours des lunettes et des gants cryogéniques. - Obtenez 2x mortiers, 1x pilon, 1x spatule à micro-pelle, 1x déounce en verre et 1x pilon en acier inoxydable pour le décéneur automatique.
- Configurez le dénonciateur automatique.
- Remplissez un bécher de glace et pré-refroidissez le verre.
- Remplissez le bidon d’azote liquide (LN2).
- Remplissez les 2 mortiers (contenant le pilon et la spatule) avec du LN2 pour refroidir les instruments. Laissez le LN2 s’évaporer et répétez.
- Pendant que les instruments refroidissent, ajoutez 1 mL de HB dans le verre dounce.
- Remplissez une dernière fois les deux mortiers de LN2 et versez l’échantillon IMAT de 50 mg dans l’un des mortiers.
- Pulvérisez l’IMAT à l’aide du pilon en appuyant doucement sur le morceau de tissu pour le briser en petits morceaux. Assurez-vous que tous les morceaux sont pulvérisés.
- Le LN2 va s’évaporer lentement tout en pulvérisant les tissus. Lorsque le tissu est correctement pulvérisé et qu’il reste encore 1/4 à 1/2 d’un mortier de LN2 , inclinez le mortier vers la lèvre du mortier pour recueillir le tissu pulvérisé par la lèvre. Laissez le LN2 s’évaporer complètement.
- Immédiatement après l’évaporation du dernier LN2 , versez le tissu pulvérisé dans le verre contenant 1 mL de HB.
3. Homogénéisation des tissus pulvérisés
- Homogénéiser le tissu pulvérisé à l’aide du débaucheur automatique. Amenez le verre de haut en bas sur le pilon en acier inoxydable pendant 10 coups dans le sens avant, suivis de 10 coups dans le sens inverse.
- Assurez-vous que la solution est trouble après l’homogénéisation et qu’elle ne contient aucun morceau de tissu visible. Une couleur rose clair est souvent attendue en raison de la contamination par le tissu musculaire.
- Transférez l’homogénat dans un tube pré-refroidi de 1,7 ml à faible liaison sur de la glace.
- Utilisez 400 μL de HB pour rincer le dounce pour vous assurer que tout le matériau est transféré et ajoutez-le dans le tube.
REMARQUE : Deux échantillons peuvent être traités à la fois. Pour ce faire, doublez le montant de HB et de NIM. Pulvériser et homogénéiser un échantillon de tissu et immédiatement après, pulvériser et homogénéiser le deuxième échantillon de tissu pour pouvoir effectuer les étapes d’isolement et de nettoyage en parallèle.
4. Isolement et nettoyage des noyaux (figure 2B)
- Ajouter 14 μL de Triton-X (10 %) à l’homogénat pour obtenir une concentration de 0,1 %.
- Gardez le tube sur de la glace et dans l’obscurité pendant 10 à 15 minutes tout en tourbillonnant toutes les 3 minutes.
- Prémouiller une crépine de 100 μm et une crépine de 40 μm (par échantillon) avec 100 μL de RT DPBS pour chacune dans un tube conique de 50 mL.
- Filtrer l’homogénat à travers la crépine à cellules de 100 μm.
- Rincez le tube de 1,7 mL avec 400 μL de HB et filtrez à travers la crépine à cellules de 100 μm.
- Ensuite, filtrez la solution à travers la crépine à cellules de 40 μm.
- Transférez une quantité égale de solution dans deux tubes pré-refroidis de 1,7 mL à faible liaison, correspondant à ~900 μL dans chaque tube.
- Centrifuger les tubes pendant 10 min à 2700 x g à 4 °C. Il devrait y avoir une petite pastille visible après la centrifugation.
- Retirer et jeter la couche lipidique supérieure et le surnageant restant, en laissant une solution de ~50 μL du premier tube.
- Répétez l’opération pour le deuxième tube.
- Remettez soigneusement la pastille en suspension dans le premier tube en le pipetant doucement de haut en bas 20 fois et transférez-la dans un nouveau tube à faible liaison de 1,7 ml. Évitez de créer des bulles.
- Répétez cette procédure pour le deuxième tube et transférez la solution remise en suspension dans le même tube.
- Ajouter 500 μL de NIM et mélanger avec le pipetage.
- Centrifuger le tube avec une balance à 1000 x g pendant 10 min à 4 °C.
- Retirer le surnageant, en laissant ~50 μL, et pipeter doucement de haut en bas jusqu’à ce que la pastille soit remise en suspension. Si vous le souhaitez, transférez la pastille remise en suspension dans un nouveau tube propre s’il reste des lipides sur le côté du tube.
- Ajouter 200 μL de NIM et mélanger par pipetage.
5. Coloration et comptage des noyaux (Figure 2C et Figure 3)
REMARQUE : Pour faciliter le comptage, mettez en place un protocole de « comptage des noyaux » sur un compteur de cellules automatisé, car le réglage du champ clair et des canaux DAPI peut affecter considérablement le comptage. Ajustez les canaux de manière à ce que seuls les noyaux et non les débris soient capturés. Assurez-vous que le canal de fond clair ne marque que les « objets » qui ont également une coloration DAPI.
- Ajoutez 1 goutte de la solution de coloration des cellules vivantes et laissez-la dans l’obscurité, sur de la glace, pendant 15 min.
- Filtrez la solution à travers une crépine de 30 μm.
- Mélanger la solution de noyaux par pipetage et ajouter 10 μL de la solution à une lame de chambre de comptage cellulaire.
- Comptez les noyaux à l’aide d’un compteur de cellules automatisé.
REMARQUE : La concentration optimale est de 1000 noyaux/μL correspondant à 1,0 x 106/mL.- Assurez-vous qu’il n’y a pas d’amas de noyaux, car cela pourrait obstruer la puce pour générer des gouttelettes de noyaux uniques (Figure 3).
- Si la concentration des noyaux n’est pas assez élevée, faire tourner la solution à 1000 x g pendant 10 min à 4 °C pour obtenir une pastille, retirer le surnageant et remettre en suspension dans un volume plus petit.
- Si le degré de débris dans la solution est élevé, remettre la solution de noyaux en suspension dans un plus grand volume de MNI (c.-à-d. 1 mL) et filtrer à nouveau à travers une crépine cellulaire de 30 μm. Ensuite, essorer à 1000 x g pendant 10 min à 4 °C et remettre en suspension dans un volume approprié par rapport à la concentration des noyaux.
- Après avoir obtenu la concentration des noyaux, passez directement à la première étape de la préparation de la bibliothèque.
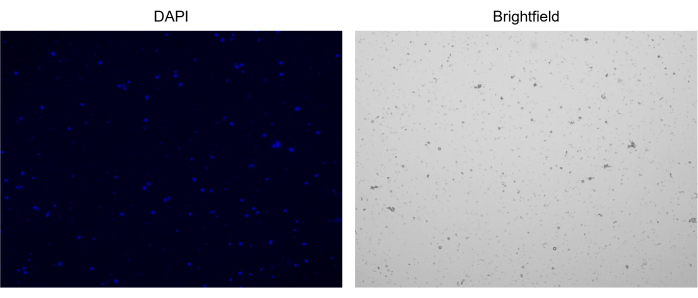
Figure 3 : Coloration de noyaux isolés. Image du compteur cellulaire de noyaux colorés avec NucBlue/DAPI (image de gauche) et l’image en fond clair correspondante (image de droite). La présence de petites quantités de débris est évidente dans l’image en champ clair. Le compteur de cellules automatisé utilisé ici n’a pas d’option pour inclure des barres d’échelle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
6. Paramètres de préparation et de séquençage de la bibliothèque
- Reportez-vous à un protocole complet pour la préparation de la banque à l’aide de l’approche des noyaux uniques basée sur des gouttelettes disponible sur la page Web du fournisseur17.
- Viser une récupération ciblée de 10 000 noyaux. Cependant, pour les échantillons présentant un niveau élevé de débris ou des noyaux fragiles, on s’attendrait à ce qu’un nombre plus faible de noyaux récupérés soit attendu.
- Conservez les échantillons à 4 °C jusqu’à 72 h après l’étape 2.3. dans le protocole de préparation de la banque pour combiner le traitement d’un plus grand nombre d’échantillons en parallèle. Pour ce faire, traitez deux échantillons jusqu’à l’étape 2.3 pendant deux jours consécutifs, et le troisième jour, traitez les 4 échantillons ensemble à partir de l’étape 3 et au-delà dans le protocole de préparation de la banque.
- Paramètres de séquençage : Séquençage sur une plateforme de séquençage visant 50 000 lectures d’extrémité appariées par noyau.
REMARQUE : Les données présentées dans ce protocole ont été séquencées sur la plateforme NovaSeq 6000, visant 50 000 lectures d’extrémités appariées par noyaux.
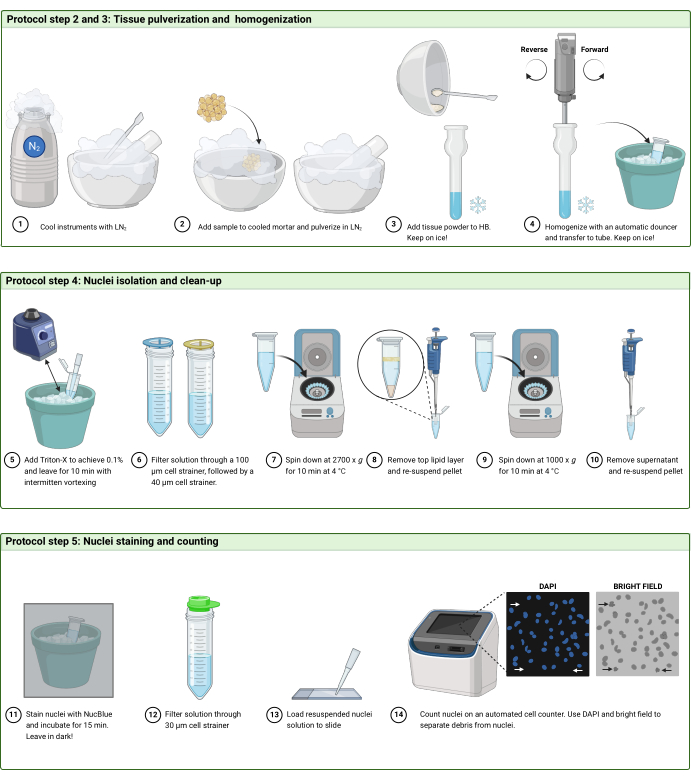
Figure 2 : flux de travail du protocole. Illustration schématique du flux de travail dans (A) les étapes 2 et 3, (B) l’étape 4 et (C) l’étape 5 du protocole. La figure a été créée avec BioRender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
7. Traitement et analyse des données
REMARQUE : Dans ce protocole, certains des logiciels et progiciels R recommandés utilisés pour traiter les données de séquençage résultantes sont brièvement présentés, en se concentrant sur les étapes après le prétraitement initial (Tableau 3). Cette étude fournit des mesures générales de contrôle de la qualité (CQ) et un exemple d’approximation et de projection de variétés uniformes (UMAP) à la figure 4. Cependant, une description approfondie de l’analyse bioinformatique n’entre pas dans le cadre de ce protocole. Par conséquent, les lecteurs peuvent se référer à la récente revue sur les meilleures pratiques pour l’analyse de cellules uniques par Heumos et al.18.
- Pré-traitement des données de séquençage
- Mappez les lectures de noyaux uniques sur le génome humain de référence GRCh38.
- Incluez les lectures d’intron dans le décompte.
- Effectuez le contrôle qualité et le filtrage des données à l’aide du package Seurat R 19.
- Calculez un score de complexité cellulaire en divisant le nombre log(10) de gènes détectés par le nombre log(10) de lectures détectées.
- Tracez les mesures de contrôle qualité les plus importantes à l’aide d’un histogramme ou d’un graphique de violon, y compris le nombre de gènes détectés par noyau, le pourcentage de lecture mitochondriale et le score de complexité cellulaire.
- Filtrez les noyaux avec moins de 200 ou plus de 10 000 gènes par noyau, plus de 10 % de lectures mitochondriales et un score de complexité inférieur à 0,8.
- Normalisez les données et effectuez une réduction de la dimensionnalité.
- Utilisez la fonction SCTransform de Seurat pour normaliser les données à l’aide de 2000 caractéristiques variables.
- Regroupez les données à l’aide des fonctions suivantes du package Seurat R : RunPCA, FindNeighbors, FindClusters et RunUMAP.
- Tracez un UMAP pour visualiser le clustering des données.
- Filtrez les doublets prévus à l’aide du package DoubletFinder R 20 et regroupez les données.
- Annoter les grappes à l’aide de marqueurs génétiques connus des types de cellules susceptibles d’être présents dans le tissu (approche supervisée) ou en fonction des 5 principaux gènes exprimés différentiellement entre les grappes (approche non supervisée).
- Utilisez decontX21pour déterminer le degré de contamination par l’ARN ambiant et pour ajuster la matrice d’expression génique pour l’ARN ambiant.
- Incluez la matrice génétique brute comme arrière-plan.
- Enregistrez l’objet Seurat pour une exploration ultérieure des données.
REMARQUE : Le code pour le contrôle de la qualité et l’analyse par grappes est disponible dans le fichier supplémentaire 1.
| Progiciels/progiciels R utilisés dans le flux de données | Logiciels/progiciels alternatifs | Étape de traitement |
| CellRanger | STARsolo, kallisto | Découpage, alignement, mappage |
| Seurat | SingleCellExperiment, Cellranger | QC, analyse et exploration de données |
| DoubletFinder | scds, scdblFinder, Scrublet | Détection de doublet |
| DecontX | SoupX, CellBender | Ajustement de l’ARN ambiant |
Tableau 3 : Logiciels/outils pour le flux de données
Access restricted. Please log in or start a trial to view this content.
Résultats
Ce flux de travail a été conçu pour guider le traitement d’échantillons IMAT humains congelés afin d’obtenir des profils d’expression génique à une résolution de noyau unique, permettant ainsi l’identification du type de cellule. Ici, un échantillon représentatif de l’IMAT provenant d’un participant à l’étude SOMMA est présenté.
La première étape de toute analyse de données de séquençage de l’ARNsn consiste à évaluer la qualité des données afin d’identi...
Access restricted. Please log in or start a trial to view this content.
Discussion
Travailler avec IMAT présente plusieurs défis inhérents. En plus de son accessibilité limitée, le rendement des échantillons est souvent très rare et la « contamination » des muscles squelettiques est presque impossible à éviter. Pour obtenir un échantillon de la meilleure qualité, il faut pénétrer dans le fascia musculaire lors de l’insertion de l’aiguille de biopsie (pour s’assurer de ne pas prélever de tissu adipeux sous-cutané) et retirer autant de tissu musculaire que possible en disséquant l...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs tiennent à remercier Bryan Bergman, PhD, à l’Université du Colorado, pour avoir fourni l’image de la biopsie IMAT dans la figure 1C de l’étude MoTrIMAT (R01AG077956). Nous sommes reconnaissants à l’Étude sur le muscle, la mobilité et le vieillissement d’avoir fourni l’échantillon IMAT à partir duquel les données sont présentées dans la section des résultats représentatifs. Le National Institute on Aging (NIA) a financé l’étude sur le muscle, la mobilité et le vieillissement (SOMMA ; R01AG059416) et ses études annexes SOMMA AT (R01AG066474) et SOMMA Knee OA (R01AG070647). Le soutien de l’infrastructure de l’étude a été financé en partie par les centres Claude D. Pepper de l’ancienne indépendance américaine de l’Université de Pittsburgh (P30AG024827) et de l’Université de Wake Forest (P30AG021332) et les instituts de sciences cliniques et translationnelles, financés par le National Center for Advancing Translational Science, de l’Université de Wake Forest (UL1 0TR001420).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 0.2 µm corning syringe filters | Millipore Sigma | CLS431229 | |
| 1.7 mL DNA LoBind tubes | Eppendorf | 22431021 | low-bind tubes |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 100x protease inhibitor | Thermo Fisher Scientific | 78437 | |
| 10X Magnetic Separator | 10X Genomics | 230003 | |
| 10X Vortex Adapter | 10X Genomics | 330002 | |
| 15 mL canonical tubes | Sarstedt | 6,25,54,502 | |
| 2100 Bioanalyzer | Agilent | G2939BA | |
| 50 mL conical tubes | Sarstedt | 6,25,47,254 | |
| CellRanger | Genomics | N/A | |
| Chromium iX accesory kit | 10X Genomics | PN1000323 | |
| Chromium iX Controller | 10X Genomics | PN1000326 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | PN1000127 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1 | 10X Genomics | PN1000129 | |
| Chromium Next GEM Single Cell GEM Kit v3.1 | 10X Genomics | PN1000130 | |
| Countess 3 Automated Cell Counter | Thermo Fisher Scientific | AMQAX2000 | Automated cell counter |
| Countess cell counting chamber slides | Thermo Fisher Scientific | C10228 | |
| DoubletFinder | R | N/A | |
| DPBS (no calcium, no magnesium) | Thermo Fisher Scientific | 14190144 | |
| DTT | Thermo Fisher Scientific | R0861 | |
| Dual Index Kit TT Set A, 96 rxns | 10X Genomics | PN1000215 | |
| Dynabeads MyOne SILANE | 10X Genomics | PN2000048 | |
| Falcon 100 µm Cell strainer | Corning Life Science | 352360 | |
| Falcon 40 µm Cell strainer | Corning Life Science | 352340 | |
| Glycerin (glycerol), 50% (v/v) Aqueous Solution | Ricca Chemical Company | 3290-32 | |
| KCL | Thermo Fisher Scientific | AM9640G | |
| Library Construction Kit v3.1 | 10X Genomics | PN1000196 | |
| MACS SmartStrainers (30µm) | Miltenyi Biotec | 130-098-458 | |
| Mastercycler Nexus Gradient Thermal cycler | Eppendorf | 6331000017 | |
| MgCl2 | Ambion | AM9530G | |
| Mortar and pestel | Health care logistics | 14075 | |
| NucBlue Live Ready Probes Reagent | Thermo Fisher Scientific | R37605 | |
| Nuclease Free Water (not DEPC treated) | Thermo Fisher Scientific | AM9930 | |
| Probumin Bovine Serum Albumin Fatty Acid Free, Powder | Sigma-Aldrich | 820024 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Ribolock RNAse inhibitor | Thermo Fisher Scientific | EO0382 | |
| Seurat | R | N/A | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SUPERasin 20 U/µL | Thermo Fisher Scientific | AM2695 | |
| ThermoMixer C | Eppendorf | 5382000015 | |
| Tissue homogenizer | Glass-Col | 099C K54 | |
| Tris buffer pH 8.0 | Thermo Fisher Scientific | AM9855G | |
| Triton X-100 | Thermo Fisher Scientific | AC327372500 | |
| UltraPure 0.5M EDTA pH 8.0 | Gibco | 15575020 |
Références
- Goodpaster, B. H., Bergman, B. C., Brennan, A. M., Sparks, L. M. Intermuscular adipose tissue in metabolic disease. Nat Rev Endocrinol. 19 (5), 285-298 (2023).
- Sparks, L. M., Goodpaster, B. H., Bergman, B. C. The metabolic significance of intermuscular adipose tissue: Is IMAT a friend or a foe to metabolic health. Diabetes. 70 (11), 2457-2467 (2021).
- Gallagher, D., et al. Adipose tissue in muscle: A novel depot similar in size to visceral adipose tissue. Am J Clin Nutr. 81 (4), 903-910 (2005).
- Manini, T. M., et al. Reduced physical activity increases intermuscular adipose tissue in healthy young adults. Am J Clin Nutr. 85 (2), 377-384 (2007).
- Addison, O., Marcus, R. L., LaStayo, P. C., Ryan, A. S. Intermuscular fat: A review of the consequences and causes. Int J Endocrinol. 2014, 309570(2014).
- Goodpaster, B. H., et al. Obesity, regional body fat distribution, and the metabolic syndrome in older men and women. Arch Intern Med. 165 (7), 777-783 (2005).
- Goodpaster, B. H., Thaete, F. L., Kelley, D. E. Thigh adipose tissue distribution is associated with insulin resistance in obesity and in type 2 diabetes mellitus. Am J Clin Nutr. 71 (4), 885-892 (2000).
- Goodpaster, B. H., et al. Association between regional adipose tissue distribution and both type 2 diabetes and impaired glucose tolerance in elderly men. Diabetes Care. 26 (2), 372-379 (2003).
- Sachs, S., et al. Intermuscular adipose tissue directly modulates skeletal muscle insulin sensitivity in humans. Am J Physiol Endocrinol Metab. 316 (5), E866-E879 (2019).
- Ford, H., Liu, Q., Fu, X., Strieder-Barboza, C. White adipose tissue heterogeneity in the single-cell era: From mice and humans to cattle. Biology (Basel). 12 (10), 1289(2023).
- Wang, L., et al. Single-nucleus and bulk RNA sequencing reveal cellular and transcriptional mechanisms underlying lipid dynamics in high marbled pork. NPJ Sci Food. 7 (1), 23(2023).
- Li, J., et al. Identification of diverse cell populations in skeletal muscles and biomarkers for intramuscular fat of chicken by single-cell RNA sequencing. BMC Genomics. 21 (1), 752(2020).
- Lyu, P., Qi, Y., Tu, Z. J., Jiang, H. Single-cell RNA sequencing reveals heterogeneity of cultured bovine satellite cells. Front Genet. 12, 742077(2021).
- Fitzgerald, G., et al. MME+ fibro-adipogenic progenitors are the dominant adipogenic population during fatty infiltration in human skeletal muscle. Commun Biol. 6 (1), 111(2023).
- Cummings, S. R., et al. The study of muscle, mobility and aging (SOMMA): A unique cohort study about the cellular biology of aging and age-related loss of mobility. J Gerontol A Biol Sci Med Sci. 78 (11), 2083-2093 (2023).
- Whytock, K. L., et al. Isolation of nuclei from frozen human subcutaneous adipose tissue for full-length single-nuclei transcriptional profiling. STAR Protoc. 4 (1), 102054(2023).
- 10x Genomics. Chromium Single Cell 3' Reagent Kits User Guide (v3.1 Chemistry Dual Index), Document Number CG000315 RevE. , Available from: https://cdn.10xgenomics.com/image/upload/v1668017706/support-documents/CG000315_ChromiumNextGEMSingleCell3-_GeneExpression_v3.1_DualIndex__RevE.pdf (2022).
- Heumos, L., et al. Best practices for single-cell analysis across modalities. Nat Rev Genet. 24 (1), 550-572 (2023).
- Hao, Y., et al. Dictionary learning for integrative, multimodal and scalable single-cell analysis. Nat Biotechnol. 42 (2), 293-304 (2023).
- McGinnis, C. S., Murrow, L. M., Gartner, Z. J. DoubletFinder: Doublet detection in single-cell RNA sequencing data using artificial nearest neighbors. Cell Syst. 8 (4), 329-337 (2019).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (2), 57(2020).
- Common considerations for quality control filters for single cell RNA-seq data. 10X Genomics. , Available from: https://www.10xgenomics.com/analysis-guides/common-considerations-for-quality-control-filters-for-single-cell-rna-seq-data (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Mol Syst Biol. 15 (6), e8746(2019).
- Emont, M. P., et al. A single-cell atlas of human and mouse white adipose tissue. Nature. 603 (7903), 926-933 (2022).
- Hildreth, A. D., et al. Single-cell sequencing of human white adipose tissue identifies new cell states in health and obesity. Nat Immunol. 22 (5), 639-653 (2021).
- Whytock, K. L., et al. Single cell full-length transcriptome of human subcutaneous adipose tissue reveals unique and heterogeneous cell populations. iScience. 25 (8), 104772(2022).
- Probst, V., et al. Benchmarking full-length transcript single cell mRNA sequencing protocols. BMC Genomics. 23 (1), 860(2022).
- CG000148 Rev A Technical Note - Resolving cell types as a function of read depth and cell number. Technical note. 10X Genomics. , Available from: https://assets.ctfassets.net/an68im79xiti/6gDArDPBTOg4IIkYEO2Sis/803be2286bb a5ca67f353e6baf68d276/CG000148_10x_Technical _Note_Resolving_Cell_Types_as_Function_of_ Read_Depth_Cell_Number_RevA.pdf (2018).
- Gupta, A., et al. Characterization of transcript enrichment and detection bias in single-nucleus RNA-seq for mapping of distinct human adipocyte lineages. Genome Res. 32 (2), 242-257 (2022).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648(2018).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. J Am Soc Nephrol. 30 (1), 23-32 (2019).
- Kim, N., Kang, H., Jo, A., Yoo, S. -A., Lee, H. -O. Perspectives on single-nucleus RNA sequencing in different cell types and tissues. J Pathol Transl Med. 57 (1), 52-59 (2023).
- Avila Cobos, F., Alquicira-Hernandez, J., Powell, J. E., Mestdagh, P., De Preter, K. Benchmarking of cell type deconvolution pipelines for transcriptomics data. Nat Commun. 11 (1), 5650(2020).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon