需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
从人肌间脂肪组织中分离细胞核和下游单核 RNA 测序
摘要
由于人体组织的可及性有限,肌间脂肪组织 (IMAT) 的生物学在很大程度上尚未得到探索。在这里,我们提出了用于单核RNA测序的冷冻人IMAT的细胞核分离和文库制备的详细方案,以鉴定这种独特脂肪库的细胞组成。
摘要
肌间脂肪组织 (IMAT) 是一种相对未被充分研究的脂肪库,位于肌肉纤维之间。IMAT含量随着年龄和BMI的增加而增加,并与代谢和肌肉退行性疾病有关;然而,对IMAT的生物学特性及其与周围肌肉纤维的相互作用的理解严重缺乏。近年来,单细胞和细胞核RNA测序为我们提供了几种人体组织的细胞类型特异性图谱。然而,由于人类 IMAT 在活检采集中具有固有的挑战,因此人类 IMAT 的细胞组成在很大程度上仍未被探索。除了收集的组织数量有限外,由于人IMAT靠近骨骼肌组织和筋膜,因此其处理也很复杂。脂肪细胞的脂质含量使其与单细胞分离不相容。因此,单核RNA测序是获得单细胞分辨率高维转录组学的最佳方法,并有可能揭示该库的生物学特性,包括IMAT的确切细胞组成。在这里,我们提出了用于单核RNA测序的冷冻人IMAT的细胞核分离和文库制备的详细方案。该协议允许使用基于液滴的方法分析数千个细胞核,从而提供检测稀有和低丰度细胞类型的能力。
引言
肌间脂肪组织 (IMAT) 是一种异位脂肪库,位于肌纤维之间和周围1。正如Goodpaster等人最近的一篇综述中详细描述的那样,IMAT可以使用高分辨率计算机断层扫描(CT)和磁共振成像(MRI)来检测(图1A,B),并且可以在整个身体的肌肉纤维周围和内部发现1。IMAT的数量因人而异,并受BMI、年龄、性别、种族和久坐的影响2,3,4。此外,IMAT 沉积常见于与肌肉退化相关的病理状况5,许多研究记录了肥胖、2 型糖尿病、代谢综合征和胰岛素抵抗患者的 IMAT 质量增加 6,7,8,9。尽管如此,IMAT的细胞和生物学特性才刚刚开始被揭示。有限的可及性以及整个身体中 IMAT 位置和内容的变化,对从这种独特的脂肪库2 收集样本提出了挑战。此外,样本在采集时很容易被骨骼肌(SM)“污染”,使得不同组织中的生物贡献之间的分离难以破译(图1C)。为此,单核RNA测序(snRNA-seq)在过去十年中获得了相当大的关注,它是一种理想的方法,可以以单细胞分辨率分离IMAT和SM衍生的基因表达模式。此外,由于脂肪细胞含有大量脂质,细胞核分离对于脂肪组织特别有用,这些脂肪细胞不可能在不损害细胞完整性的情况下解离成单细胞悬浮液。最后,该技术有可能发现IMAT特异性脂肪细胞的新标记物,揭示不同祖细胞群的组成和存在,以及研究细胞组成在病理和正常条件下的变化。
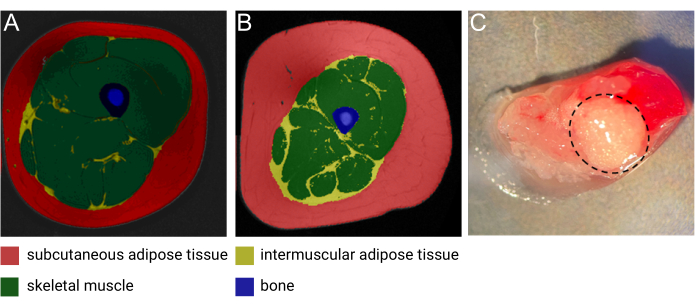
图 1:IMAT 的图像。 (A) 中年瘦女性和 (B) 肥胖中年男性的 IMAT 代表性磁共振 (MRI) 图像。红色:皮下脂肪组织,黄色:肌间脂肪组织,绿色:骨骼肌,蓝色:骨骼。图片由 AdventHealth 转化研究所的 Heather Cornnell 提供。(C) 使用IMAT的新鲜组织样本(用黑色虚线包围)。图片由AdventHealth转化研究所的Meghan Hopf和科罗拉多大学的Bryan Bergman提供。此图经 Goodpaster 等人许可修改 1. 请点击这里查看此图的较大版本.
畜牧业已经发表了许多研究,使用单细胞 (sc) 和 snRNA-seq10 调查猪、鸡和牛的肉大理石花纹(特别是 IMAT)。这些研究确定了脂肪细胞的几个亚群和 IMAT11、12、13 潜在祖细胞的标志物;然而,这些细胞组成是否转化为人类IMAT尚不清楚。据我们所知,只有一项研究使用 snRNA-seq14 研究了从患有髋关节骨关节炎的男性患者那里获得的脂肪浸润的人类肌肉的细胞异质性。研究人员报告了一个小的脂肪细胞群和大量的肌核14 群体中的几个纤维-成脂祖细胞 (FAP) 亚群。我们的研究首次开发了一种使用snRNA-seq直接从人体肌肉中手动解剖的IMAT细胞组成的方法。
重要的是,snRNA-seq的方案需要针对所研究的特定组织进行定制,因为可用组织的数量和特定组织的物理特性将决定最佳的处理步骤。IMAT的组织产量通常很小,通常不超过50mg,即使在进行超声引导活检时也是如此。因此,正确处理这种稀缺的组织至关重要。我们相信该协议将成为研究人类IMAT的研究人员的宝贵资源。
Access restricted. Please log in or start a trial to view this content.
研究方案
用于该协议的样本是肌肉、活动性和衰老研究 (SOMMA)15 的一部分,该研究由西方 IRB-哥白尼小组 (WCG) 机构审查委员会批准,并根据赫尔辛基宣言进行。参与者为参与研究提供了书面知情同意书。
注:该协议改编自先前的协议,在基于纳米孔的平台16上使用100mg人腹部皮下脂肪组织。当前方案针对 50 mg 人 IMAT 和使用基于液滴的平台进行文库制备进行了优化。可能需要进一步优化该协议,以从非人IMAT或其他脂肪库中分离细胞核。
1.缓冲液和试剂的制备(表1和表2)
注意:在实验当天准备新鲜的缓冲液,不要重复使用。
- 将离心机预冷至4°C。
- 准备均质缓冲液和细胞核分离培养基。
- 取两桶冰并预冷 2 x 15 mL 锥形管。
- 按照 表 1 中列出的顺序在 15 mL 锥形管中混合用于均质缓冲液 (HB) 的所有试剂。保持在冰上。通过涡旋混合。
- 将细胞核分离培养基 (NIM) 的所有试剂混合在 表 2 中有序列表的 15 mL 锥形管中。保持在冰上。通过涡旋混合。
- 通过向 900 μL 无核酸酶水中加入 100 μL Triton X-100 来制备 10% Triton-X。涡旋以确保正确混合。保持在室温 (RT) 下。
| 试剂 | 体积 (μL) | 终浓度 (mM) | |
| 1 倍 | 2 倍 | ||
| 1 M 氯化镁2 | 10 | 20 | 5 |
| 1 M Tris 缓冲液,pH 8.0 | 20 | 40 | 10 |
| 2 M 氯化钾 | 25 | 50 | 25 |
| 1.5M蔗糖(-4°C) | 334 | 668 | 250 |
| 1mM DTT | 2 | 4 | 0.001(~1微米) |
| 100x蛋白酶抑制剂 | 20 | 40 | 1 倍 |
| Superasin 20 U/μL | 40 | 80 | 0.4 U/μL |
| 无核酸酶水 | 1549 | 3098 | - |
| 总体积 | 2000 | 4000 | - |
表 1:均质缓冲液 (HB)。 保持在冰上。通过涡旋混合。
| 试剂 | 体积 (μL) | 终浓度 (mM) | |
| 1 倍 | 2 倍 | ||
| 乙二胺四乙酸 | 0.4 | 0.8 | 0.1 |
| Ribolock RNAse 抑制剂 (40U/μL) | 40 | 80 | 0.8 U/μL |
| 1%牛血清白蛋白-PBS(-/-) | 1959.6 | 3919.2 | - |
| 总体积 | 2000 | 4000 | - |
表 2:细胞核分离培养基 (NIM)。 保持在冰上。通过涡旋混合。
2.冷冻组织的粉碎(图2A)
- 设置用于均质化的工作站。
- 用液氮 (LN2) 填充罐子。
注意:使用 LN2 时, 请始终佩戴护目镜和冷冻手套。 - 获得 2 个研钵、1 个研杵、1 个微勺刮刀、1 个玻璃杵和 1 个不锈钢杵用于自动杵。
- 设置自动 douncer。
- 在烧杯中装满冰块,然后预冷玻璃杯。
- 用液氮 (LN2) 填充罐子。
- 用 LN2 填充 2 个研钵(包含研杵和刮刀)以冷却仪器。让 LN2 蒸发并重复。
- 当仪器冷却时,向玻璃溶液中加入 1 mL HB。
- 最后一次用 LN2 填充两个研钵,然后将 50 mg IMAT 样品倒入其中一个研钵中。
- 使用研杵将 IMAT 粉碎,轻轻地将其压在组织片上,将其分解成小块。确保所有碎片都被粉碎。
- LN2 在粉碎组织时会慢慢蒸发。当组织正确粉碎,并且还剩下 1/4 - 1/2 的 LN2 研钵时,将研钵向研钵的边缘倾斜,以收集边缘的粉碎组织。让 LN2 完全蒸发。
- 在最后一个 LN2 蒸发后,立即将粉碎的组织舀入含有 1 mL HB 的玻璃杯中。
3.粉状组织的均质化
- 使用自动落点器对粉碎的组织进行均质化。将玻璃在不锈钢杵上上下旋转 10 次,然后向相反方向 10 次。
- 确保溶液在均质化后是浑浊的,并且不含可见的组织碎片。由于被肌肉组织污染,通常会出现浅粉红色。
- 将匀浆转移到预冷的 1.7 mL 低结合管中,放在冰上。
- 使用 400 μL HB 冲洗 dounce,确保所有材料都已转移并将其添加到试管中。
注意:一次可以处理两个样品。为此,将 HB 和 NIM 的量增加一倍。粉碎和均质化一个组织样品,然后立即粉碎和均质化第二个组织样品,以便能够同时执行分离和纯化步骤。
4. 细胞核分离和净化(图2B)
- 向匀浆中加入 14 μL Triton-X (10%) 浓度为 0.1%。
- 将管子保持在冰上和黑暗中 10-15 分钟,同时每 3 分钟涡旋一次。
- 在 50 mL 锥形管中预润湿一个 100 μm 和一个 40 μm 细胞过滤器(每个样品),每个样品含 100 μL RT DPBS。
- 通过 100 μm 细胞过滤器过滤匀浆。
- 用 400 μL HB 冲洗 1.7 mL 管,并通过 100 μm 细胞过滤器过滤。
- 接下来,通过 40 μm 细胞过滤器过滤溶液。
- 将等量的溶液转移到两个预冷的 1.7 mL 低结合管中,对应于每个管中的 ~900 μL。
- 在4°C下以2700× g 离心管10分钟。 离心后应该有一个可见的小颗粒。
- 去除并丢弃顶部脂质层和剩余的上清液,从第一管中留下 ~50 μL 溶液。
- 对第二个试管重复上述步骤。
- 轻轻地上下移液 20 次,将沉淀彻底重悬于第一个管中,然后转移到新的 1.7 mL 低结合管中。避免产生气泡。
- 对第二个管重复此步骤,并将重悬溶液转移到同一管中。
- 加入 500 μL NIM 并与移液混合。
- 在4°C下用1000× g 的天平离心管10分钟。
- 取出上清液,留下 ~50 μL,轻轻地上下移液,直到重新悬浮沉淀。或者,如果管的侧面残留一些脂质,则将重悬的颗粒转移到新的干净管中。
- 加入 200 μL NIM 并通过移液混合。
5.细胞核染色和计数(图2C 和 图3)
注意:为了便于计数,请在自动细胞计数仪上设置“细胞核计数”协议,因为调整明场和DAPI通道会极大地影响计数。调整通道,以便仅捕获细胞核而不捕获碎片。确保明场通道仅标记也有 DAPI 染色的“对象”。
- 加入 1 滴活细胞染色溶液,将其置于黑暗的冰上 15 分钟。
- 通过 30 μm 细胞过滤器过滤溶液。
- 通过移液混合细胞核溶液,并将 10 μL 溶液加入细胞计数室载玻片中。
- 使用自动细胞计数仪对细胞核进行计数。
注:最佳浓度为 1000 个细胞核/μL,对应于 1.0 x 106/mL。- 确保不存在细胞核团块,因为这可能会堵塞用于生成单核液滴的芯片(图 3)。
- 如果细胞核浓度不够高,则在4°C下以1000× g 旋转溶液10分钟以获得沉淀,除去上清液,并以较小的体积重悬。
- 如果溶液中的碎片程度很高,则将细胞核溶液重悬于较大体积的 NIM(即 1 mL)中,并再次通过 30 μm 细胞过滤器过滤。然后在4°C下以1000× g 旋转10分钟,并相对于细胞核的浓度以适当的体积重悬。
- 获得细胞核浓度后,直接进行文库制备的第一步。
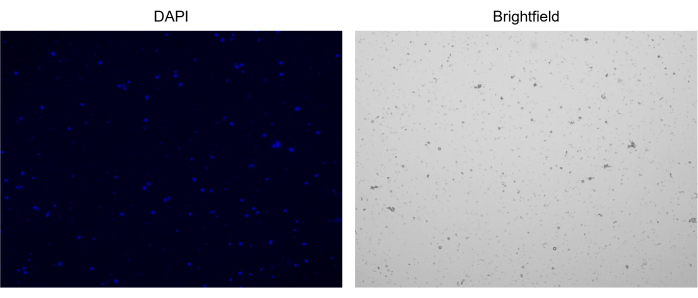
图 3:分离细胞核的染色。 用 NucBlue/DAPI 染色的细胞核细胞计数图像(左图)和相应的明场图像(右图)。在明场图像中可以明显看出少量碎片的存在。此处使用的自动细胞计数仪没有包含比例尺的选项。 请点击这里查看此图的较大版本.
6. 文库制备和测序参数
- 请参阅提供商网页17 上提供的使用基于液滴的单核方法进行文库制备的全面方案。
- 目标是实现 10,000 个目标细胞核回收率。然而,对于碎片含量高或细胞核脆弱的样品,预计回收的细胞核数量会较少。
- 步骤 2.3 后,将样品在 4°C 下储存长达 72 小时。在文库制备协议中,将更多样品的处理并行结合起来。通过连续两天处理两个样品直到步骤2.3来做到这一点,并在第三天,从步骤3开始一起处理4个样品,并在文库制备方案中继续进行。
- 测序参数:在测序平台上进行测序,目标是每个细胞核的双端读取次数达到 50,000 次。
注:该协议中提供的数据在NovaSeq 6000平台上测序,目标是每个细胞核进行50,000次双端读取。
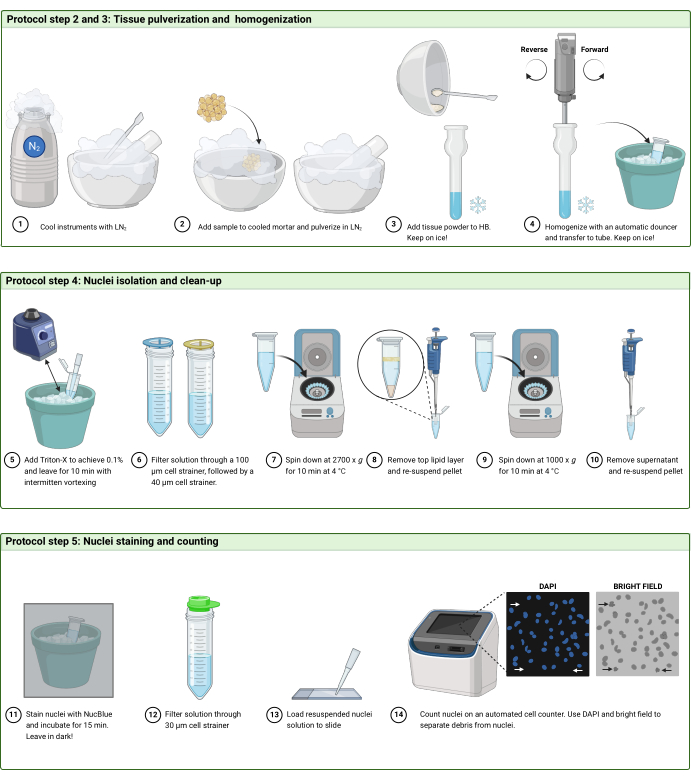
图 2:协议工作流程。 协议的 (A) 步骤 2 和 3、(B) 步骤 4 和 (C) 步骤 5 中的工作流程示意图。该图是用 BioRender.com 创作的。 请点击这里查看此图的较大版本.
7. 数据处理和分析
注:在该协议中,简要介绍了一些推荐的用于处理所得测序数据的软件和R包,重点介绍了初始预处理后的步骤(表3)。本研究提供了一般质量控制 (QC) 指标和 图 4 中的均匀流形近似和投影 (UMAP) 示例。然而,对生物信息学分析的深入描述超出了本协议的范围。因此,读者可以参考Heumos等人最近对单细胞分析最佳实践的综述[18]。
- 测序数据的预处理
- 将单个细胞核读数映射到人类参考基因组 GRCh38。
- 在计数中包括内含子读取。
- 使用 Seurat R 软件包19 执行 QC 和数据过滤。
- 通过将检测到的基因数对数(10)除以检测到的reads数(10)来计算细胞复杂性分数。
- 使用直方图或小提琴图绘制出最重要的 QC 指标,包括每个细胞核检测到的基因数量、线粒体读取百分比和细胞复杂性评分。
- 过滤掉每个细胞核少于 200 个或超过 10,000 个基因、线粒体读数大于 10% 且复杂度评分低于 0.8 的细胞核。
- 对数据进行归一化并执行降维。
- 使用 Seurat 的 SCTransform 函数,使用 2000 个变量特征对数据进行归一化。
- 使用 Seurat R 包中的以下函数对数据进行聚类: RunPCA、 FindNeighbors、 FindClusters 和 RunUMAP。
- 绘制 UMAP 以可视化数据的聚类。
- 使用 DoubletFinder R 包20 过滤掉预测的双峰,并重新聚类数据。
- 使用预期存在于组织中的细胞类型的已知基因标记(监督方法)或基于簇之间的前 5 个差异表达基因(无监督方法)注释簇。
- 使用 decontX21确定环境 RNA 污染的程度,并调整环境 RNA 的基因表达基质。
- 包括原始基因基质作为背景。
- 保存 Seurat 对象,以便将来浏览数据。
注意:QC 和聚类分析的代码可在 补充文件 1 中找到。
| 数据工作流中使用的 Software/R 包 | 替代软件/软件包 | 处理步骤 |
| CellRanger系列 | STARsolo, kallisto | 修剪、对齐、映射 |
| 修拉 | SingleCellExperiment, 细胞游侠 | 质量控制、分析和数据探索 |
| DoubletFinder(双倍取景器) | scds、scdblFinder、Scrublet | 双峰检测 |
| DecontX的 | SoupX,CellBender | 环境 RNA 调整 |
表 3:用于数据工作流的软件/工具。
Access restricted. Please log in or start a trial to view this content.
结果
该工作流程旨在指导冷冻人IMAT样品的处理,以获得单核分辨率的基因表达谱,从而实现细胞类型鉴定。在这里,展示了来自SOMMA研究参与者的一个代表性IMAT样本。
对snRNA-seq数据进行任何分析的第一步都是评估数据的质量,以识别质量较差的细胞核,这些细胞核可能会从数据集中删除。重要的是,应根据您手头的特定样本类型和数据集确定过滤步骤和阈值,因为通常评估的指?...
Access restricted. Please log in or start a trial to view this content.
讨论
与IMAT合作存在一些固有的挑战。除了可及性有限外,样品材料的产量通常非常稀缺,骨骼肌的“污染”几乎是无法避免的。为了获得最高质量的样本,在插入活检针时应穿透肌肉筋膜(以确保不收集皮下脂肪组织),并在收集后立即在显微镜下解剖样本以去除尽可能多的肌肉组织,然后进行组织学处理、快速冷冻等。这是第一种利用从骨骼肌中前瞻性收集和解剖的IMAT样本的方法。相比之下,IMAT?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的。
致谢
作者感谢科罗拉多大学的 Bryan Bergman 博士在图 1C 中提供了 MoTrIMAT 研究 (R01AG077956) 的 IMAT 活检图像。我们感谢肌肉、活动和衰老研究提供的 IMAT 样本,数据在代表性结果部分显示。美国国家老龄化研究所 (NIA) 资助了肌肉、活动性和衰老研究 (SOMMA;R01AG059416)及其辅助研究SOMMA AT(R01AG066474)和SOMMA膝关节OA (R01AG070647)。研究基础设施支持部分由匹兹堡大学 (P30AG024827) 和维克森林大学 (P30AG021332) 的 NIA Claude D. Pepper 老年美国独立中心以及临床和转化科学研究所资助,由维克森林大学国家促进转化科学中心 (UL1 0TR001420) 资助。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 0.2 µm corning syringe filters | Millipore Sigma | CLS431229 | |
| 1.7 mL DNA LoBind tubes | Eppendorf | 22431021 | low-bind tubes |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 100x protease inhibitor | Thermo Fisher Scientific | 78437 | |
| 10X Magnetic Separator | 10X Genomics | 230003 | |
| 10X Vortex Adapter | 10X Genomics | 330002 | |
| 15 mL canonical tubes | Sarstedt | 6,25,54,502 | |
| 2100 Bioanalyzer | Agilent | G2939BA | |
| 50 mL conical tubes | Sarstedt | 6,25,47,254 | |
| CellRanger | Genomics | N/A | |
| Chromium iX accesory kit | 10X Genomics | PN1000323 | |
| Chromium iX Controller | 10X Genomics | PN1000326 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | PN1000127 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1 | 10X Genomics | PN1000129 | |
| Chromium Next GEM Single Cell GEM Kit v3.1 | 10X Genomics | PN1000130 | |
| Countess 3 Automated Cell Counter | Thermo Fisher Scientific | AMQAX2000 | Automated cell counter |
| Countess cell counting chamber slides | Thermo Fisher Scientific | C10228 | |
| DoubletFinder | R | N/A | |
| DPBS (no calcium, no magnesium) | Thermo Fisher Scientific | 14190144 | |
| DTT | Thermo Fisher Scientific | R0861 | |
| Dual Index Kit TT Set A, 96 rxns | 10X Genomics | PN1000215 | |
| Dynabeads MyOne SILANE | 10X Genomics | PN2000048 | |
| Falcon 100 µm Cell strainer | Corning Life Science | 352360 | |
| Falcon 40 µm Cell strainer | Corning Life Science | 352340 | |
| Glycerin (glycerol), 50% (v/v) Aqueous Solution | Ricca Chemical Company | 3290-32 | |
| KCL | Thermo Fisher Scientific | AM9640G | |
| Library Construction Kit v3.1 | 10X Genomics | PN1000196 | |
| MACS SmartStrainers (30µm) | Miltenyi Biotec | 130-098-458 | |
| Mastercycler Nexus Gradient Thermal cycler | Eppendorf | 6331000017 | |
| MgCl2 | Ambion | AM9530G | |
| Mortar and pestel | Health care logistics | 14075 | |
| NucBlue Live Ready Probes Reagent | Thermo Fisher Scientific | R37605 | |
| Nuclease Free Water (not DEPC treated) | Thermo Fisher Scientific | AM9930 | |
| Probumin Bovine Serum Albumin Fatty Acid Free, Powder | Sigma-Aldrich | 820024 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Ribolock RNAse inhibitor | Thermo Fisher Scientific | EO0382 | |
| Seurat | R | N/A | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SUPERasin 20 U/µL | Thermo Fisher Scientific | AM2695 | |
| ThermoMixer C | Eppendorf | 5382000015 | |
| Tissue homogenizer | Glass-Col | 099C K54 | |
| Tris buffer pH 8.0 | Thermo Fisher Scientific | AM9855G | |
| Triton X-100 | Thermo Fisher Scientific | AC327372500 | |
| UltraPure 0.5M EDTA pH 8.0 | Gibco | 15575020 |
参考文献
- Goodpaster, B. H., Bergman, B. C., Brennan, A. M., Sparks, L. M. Intermuscular adipose tissue in metabolic disease. Nat Rev Endocrinol. 19 (5), 285-298 (2023).
- Sparks, L. M., Goodpaster, B. H., Bergman, B. C. The metabolic significance of intermuscular adipose tissue: Is IMAT a friend or a foe to metabolic health. Diabetes. 70 (11), 2457-2467 (2021).
- Gallagher, D., et al. Adipose tissue in muscle: A novel depot similar in size to visceral adipose tissue. Am J Clin Nutr. 81 (4), 903-910 (2005).
- Manini, T. M., et al. Reduced physical activity increases intermuscular adipose tissue in healthy young adults. Am J Clin Nutr. 85 (2), 377-384 (2007).
- Addison, O., Marcus, R. L., LaStayo, P. C., Ryan, A. S. Intermuscular fat: A review of the consequences and causes. Int J Endocrinol. 2014, 309570(2014).
- Goodpaster, B. H., et al. Obesity, regional body fat distribution, and the metabolic syndrome in older men and women. Arch Intern Med. 165 (7), 777-783 (2005).
- Goodpaster, B. H., Thaete, F. L., Kelley, D. E. Thigh adipose tissue distribution is associated with insulin resistance in obesity and in type 2 diabetes mellitus. Am J Clin Nutr. 71 (4), 885-892 (2000).
- Goodpaster, B. H., et al. Association between regional adipose tissue distribution and both type 2 diabetes and impaired glucose tolerance in elderly men. Diabetes Care. 26 (2), 372-379 (2003).
- Sachs, S., et al. Intermuscular adipose tissue directly modulates skeletal muscle insulin sensitivity in humans. Am J Physiol Endocrinol Metab. 316 (5), E866-E879 (2019).
- Ford, H., Liu, Q., Fu, X., Strieder-Barboza, C. White adipose tissue heterogeneity in the single-cell era: From mice and humans to cattle. Biology (Basel). 12 (10), 1289(2023).
- Wang, L., et al. Single-nucleus and bulk RNA sequencing reveal cellular and transcriptional mechanisms underlying lipid dynamics in high marbled pork. NPJ Sci Food. 7 (1), 23(2023).
- Li, J., et al. Identification of diverse cell populations in skeletal muscles and biomarkers for intramuscular fat of chicken by single-cell RNA sequencing. BMC Genomics. 21 (1), 752(2020).
- Lyu, P., Qi, Y., Tu, Z. J., Jiang, H. Single-cell RNA sequencing reveals heterogeneity of cultured bovine satellite cells. Front Genet. 12, 742077(2021).
- Fitzgerald, G., et al. MME+ fibro-adipogenic progenitors are the dominant adipogenic population during fatty infiltration in human skeletal muscle. Commun Biol. 6 (1), 111(2023).
- Cummings, S. R., et al. The study of muscle, mobility and aging (SOMMA): A unique cohort study about the cellular biology of aging and age-related loss of mobility. J Gerontol A Biol Sci Med Sci. 78 (11), 2083-2093 (2023).
- Whytock, K. L., et al. Isolation of nuclei from frozen human subcutaneous adipose tissue for full-length single-nuclei transcriptional profiling. STAR Protoc. 4 (1), 102054(2023).
- 10x Genomics. Chromium Single Cell 3' Reagent Kits User Guide (v3.1 Chemistry Dual Index), Document Number CG000315 RevE. , Available from: https://cdn.10xgenomics.com/image/upload/v1668017706/support-documents/CG000315_ChromiumNextGEMSingleCell3-_GeneExpression_v3.1_DualIndex__RevE.pdf (2022).
- Heumos, L., et al. Best practices for single-cell analysis across modalities. Nat Rev Genet. 24 (1), 550-572 (2023).
- Hao, Y., et al. Dictionary learning for integrative, multimodal and scalable single-cell analysis. Nat Biotechnol. 42 (2), 293-304 (2023).
- McGinnis, C. S., Murrow, L. M., Gartner, Z. J. DoubletFinder: Doublet detection in single-cell RNA sequencing data using artificial nearest neighbors. Cell Syst. 8 (4), 329-337 (2019).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (2), 57(2020).
- Common considerations for quality control filters for single cell RNA-seq data. 10X Genomics. , Available from: https://www.10xgenomics.com/analysis-guides/common-considerations-for-quality-control-filters-for-single-cell-rna-seq-data (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Mol Syst Biol. 15 (6), e8746(2019).
- Emont, M. P., et al. A single-cell atlas of human and mouse white adipose tissue. Nature. 603 (7903), 926-933 (2022).
- Hildreth, A. D., et al. Single-cell sequencing of human white adipose tissue identifies new cell states in health and obesity. Nat Immunol. 22 (5), 639-653 (2021).
- Whytock, K. L., et al. Single cell full-length transcriptome of human subcutaneous adipose tissue reveals unique and heterogeneous cell populations. iScience. 25 (8), 104772(2022).
- Probst, V., et al. Benchmarking full-length transcript single cell mRNA sequencing protocols. BMC Genomics. 23 (1), 860(2022).
- CG000148 Rev A Technical Note - Resolving cell types as a function of read depth and cell number. Technical note. 10X Genomics. , Available from: https://assets.ctfassets.net/an68im79xiti/6gDArDPBTOg4IIkYEO2Sis/803be2286bb a5ca67f353e6baf68d276/CG000148_10x_Technical _Note_Resolving_Cell_Types_as_Function_of_ Read_Depth_Cell_Number_RevA.pdf (2018).
- Gupta, A., et al. Characterization of transcript enrichment and detection bias in single-nucleus RNA-seq for mapping of distinct human adipocyte lineages. Genome Res. 32 (2), 242-257 (2022).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648(2018).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. J Am Soc Nephrol. 30 (1), 23-32 (2019).
- Kim, N., Kang, H., Jo, A., Yoo, S. -A., Lee, H. -O. Perspectives on single-nucleus RNA sequencing in different cell types and tissues. J Pathol Transl Med. 57 (1), 52-59 (2023).
- Avila Cobos, F., Alquicira-Hernandez, J., Powell, J. E., Mestdagh, P., De Preter, K. Benchmarking of cell type deconvolution pipelines for transcriptomics data. Nat Commun. 11 (1), 5650(2020).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。