È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento di nuclei dal tessuto adiposo intermuscolare umano e sequenziamento dell'RNA a singolo nucleo a valle
In questo articolo
Riepilogo
La biologia del tessuto adiposo intermuscolare (IMAT) è in gran parte inesplorata a causa della limitata accessibilità del tessuto umano. Qui, presentiamo un protocollo dettagliato per l'isolamento dei nuclei e la preparazione della libreria di IMAT umano congelato per il sequenziamento di RNA a singoli nuclei per identificare la composizione cellulare di questo deposito adiposo unico.
Abstract
Il tessuto adiposo intermuscolare (IMAT) è un deposito adiposo relativamente poco studiato situato tra le fibre muscolari. Il contenuto di IMAT aumenta con l'età e l'IMC ed è associato a malattie metaboliche e muscolo-degenerative; tuttavia, una comprensione delle proprietà biologiche dell'IMAT e della sua interazione con le fibre muscolari circostanti è gravemente carente. Negli ultimi anni, il sequenziamento dell'RNA di singole cellule e nuclei ci ha fornito atlanti specifici per tipo di cellula di diversi tessuti umani. Tuttavia, la composizione cellulare dell'IMAT umano rimane in gran parte inesplorata a causa delle sfide intrinseche della sua accessibilità dalla raccolta della biopsia nell'uomo. Oltre alla quantità limitata di tessuto raccolto, l'elaborazione dell'IMAT umano è complicata a causa della sua vicinanza al tessuto muscolare scheletrico e alla fascia. La natura lipidica degli adipociti lo rende incompatibile con l'isolamento a singola cellula. Pertanto, il sequenziamento dell'RNA a singolo nucleo è ottimale per ottenere trascrittomica ad alta dimensionalità con risoluzione di singola cellula e fornisce il potenziale per scoprire la biologia di questo deposito, inclusa l'esatta composizione cellulare di IMAT. Qui, presentiamo un protocollo dettagliato per l'isolamento dei nuclei e la preparazione della libreria di IMAT umano congelato per il sequenziamento di RNA a singoli nuclei. Questo protocollo consente la profilazione di migliaia di nuclei utilizzando un approccio basato su goccioline, fornendo così la capacità di rilevare tipi di cellule rare e poco abbondanti.
Introduzione
Il tessuto adiposo intermuscolare (IMAT) è un deposito adiposo ectopico che risiede tra e intorno alle fibre muscolari1. Come descritto in dettaglio in una recente revisione di Goodpaster et al., l'IMAT può essere rilevato utilizzando la tomografia computerizzata (TC) ad alta risoluzione e la risonanza magnetica (MRI) (Figura 1A, B) e si trova intorno e all'interno delle fibre muscolari in tutto il corpo1. La quantità di IMAT varia notevolmente da individuo a individuo ed è influenzata da BMI, età, sesso, razza e sedentarietà 2,3,4. Inoltre, la deposizione di IMAT è comunemente osservata in condizioni patologiche associate alla degenerazione muscolare5 e numerosi studi hanno documentato un aumento della massa IMAT in individui con obesità, diabete di tipo 2, sindrome metabolica e insulino-resistenza 6,7,8,9. Ciononostante, le proprietà cellulari e biologiche dell'IMAT stanno solo iniziando a essere svelate. L'accessibilità limitata e la variazione delle posizioni e dei contenuti IMAT in tutto il corpo hanno messo a dura prova la raccolta di campioni da questo deposito adiposo unico nel suo genere2. Inoltre, i campioni sono facilmente "contaminati" con il muscolo scheletrico (SM) al momento della raccolta, rendendo difficile decifrare la separazione tra il contributo biologico dei diversi tessuti (Figura 1C). A tal fine, il sequenziamento dell'RNA a singolo nucleo (snRNA-seq), che ha guadagnato notevole attenzione nell'ultimo decennio, funge da metodologia ideale per consentire la separazione di modelli di espressione genica derivati da IMAT e SM con risoluzione di una singola cellula. Inoltre, l'isolamento dei nuclei è particolarmente utile per il tessuto adiposo a causa dei grandi adipociti carichi di lipidi, che sono impossibili da dissociare in sospensione unicellulare senza compromettere l'integrità delle cellule. Infine, questa tecnologia ha il potenziale per scoprire nuovi marcatori di adipociti specifici per IMAT e scoprire la composizione e la presenza di diverse popolazioni di cellule progenitrici, nonché studiare la variazione della composizione cellulare in condizioni patologiche e normali.
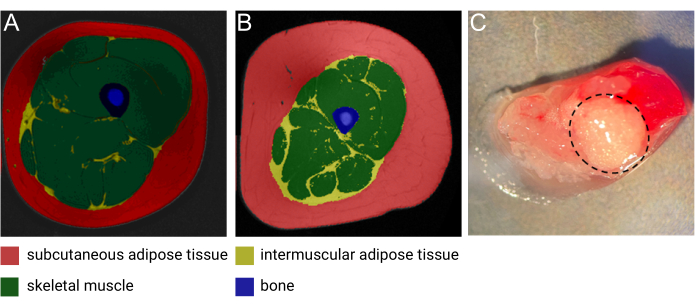
Figura 1: Immagini di IMAT. Immagine rappresentativa della risonanza magnetica (MRI) di IMAT da (A) una femmina magra di mezza età e (B) un maschio di mezza età con obesità. Rosso: tessuto adiposo sottocutaneo, giallo: tessuto adiposo intermuscolare, verde: muscolo scheletrico, blu: osso. Immagine per gentile concessione di Heather Cornnell, AdventHealth Translational Research Institute. (C) Campione di tessuto fresco con IMAT (circondato da una linea nera tratteggiata). Immagine per gentile concessione di Meghan Hopf, AdventHealth Translational Research Institute e Bryan Bergman, Università del Colorado. Questa figura è stata modificata con il permesso di Goodpaster et al.1. Clicca qui per visualizzare una versione più grande di questa figura.
Sono stati pubblicati numerosi studi dell'industria zootecnica che indagano la marezzatura della carne (IMAT in particolare) in suini, polli e bovini utilizzando cellule singole (sc) e snRNA-seq10. Questi studi hanno identificato diverse sottopopolazioni di adipociti e marcatori di potenziali cellule progenitrici di IMAT 11,12,13; tuttavia, non è noto se queste composizioni cellulari si traducano nell'IMAT umano. Per quanto ne sappiamo, solo uno studio ha esaminato l'eterogeneità cellulare del muscolo umano con infiltrazione adiposa, ottenuta da pazienti maschi con osteoartrite dell'anca, utilizzando snRNA-seq14. I ricercatori hanno riportato una piccola popolazione di adipociti e diverse sottopopolazioni di progenitori fibro-adipogenici (FAP) all'interno della grande popolazione di mionuclei14. Il nostro studio è il primo a sviluppare un metodo per interrogare direttamente l'IMAT sezionato manualmente dal muscolo umano per la composizione cellulare utilizzando snRNA-seq.
È importante sottolineare che i protocolli per l'snRNA-seq devono essere personalizzati per il tessuto specifico studiato, poiché la quantità di tessuto disponibile e le proprietà fisiche del tessuto specifico determineranno le fasi di elaborazione ottimali. La resa tissutale per l'IMAT è in genere piccola, spesso non superiore a 50 mg, anche quando si eseguono biopsie ecoguidate. Pertanto, è essenziale una corretta lavorazione di questo tessuto scarso. Riteniamo che questo protocollo servirà come risorsa preziosa per i ricercatori che studiano l'IMAT umano.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Il campione utilizzato per questo protocollo faceva parte dello Studio sui muscoli, la mobilità e l'invecchiamento (SOMMA)15, che è stato approvato dal Western IRB-Copernicus Group (WCG) Institutional Review Board ed è stato condotto in conformità con la Dichiarazione di Helsinki. I partecipanti hanno fornito il consenso informato scritto per la loro partecipazione allo studio.
NOTA : Questo protocollo è adattato da un protocollo precedente che utilizza 100 mg di tessuto adiposo sottocutaneo addominale umano su una piattaforma basata su nanopozzetti16. L'attuale protocollo è ottimizzato per 50 mg di IMAT umano e la preparazione di librerie utilizzando una piattaforma basata su goccioline. Potrebbe essere necessaria un'ulteriore ottimizzazione di questo protocollo per l'isolamento di nuclei da IMAT non umano o da altri depositi adiposi.
1. Preparazione di tamponi e reagenti (Tabella 1 e Tabella 2)
NOTA: Preparare i tamponi freschi il giorno dell'esperimento e non riutilizzarli.
- Preraffreddare una centrifuga a 4 °C.
- Preparare il tampone di omogeneizzazione e il terreno di isolamento dei nuclei.
- Procurarsi due secchi di ghiaccio e preraffreddare 2 provette coniche da 15 mL.
- Miscelare tutti i reagenti per il tampone di omogeneizzazione (HB) in una provetta conica da 15 mL secondo l'ordine indicato nella Tabella 1. Mantenere il ghiaccio. Mescolare a vortice.
- Miscelare tutti i reagenti per il mezzo di isolamento dei nuclei (NIM) in una provetta conica da 15 mL nell'elenco ordinato nella Tabella 2. Mantenere il ghiaccio. Mescolare a vortice.
- Preparare il 10% di Triton-X aggiungendo 100 μL di Triton X-100 a 900 μL di acqua priva di nucleasi. Vortice per garantire una corretta miscelazione. Conservare a temperatura ambiente (RT).
| Reagente | Volume (μL) | Concentrazione finale (mM) | |
| 1x | 2x | ||
| 1 M MgCl2 | 10 | 20 | 5 |
| 1 M Tris tampone, pH 8,0 | 20 | 40 | 10 |
| 2 M KCl | 25 | 50 | 25 |
| 1,5 M Saccarosio (-4oC) | 334 | 668 | 250 |
| 1 mM DTT | 2 | 4 | 0,001 (~1 μM) |
| 100x inibitore della proteasi | 20 | 40 | 1x |
| Superasin 20 U/μL | 40 | 80 | 0,4 U/μL |
| Acqua priva di nucleasi | 1549 | 3098 | - |
| Volume totale | 2000 | 4000 | - |
Tabella 1: Tampone di omogeneizzazione (HB). Mantenere il ghiaccio. Mescolare a vortice.
| Reagente | Volume (μL) | Concentrazione finale (mM) | |
| 1x | 2x | ||
| EDTA | 0.4 | 0.8 | 0.1 |
| Inibitore dell'RNAsi di Ribolock (40U/μL) | 40 | 80 | 0,8 U/μL |
| 1% BSA-PBS (-/-) | 1959.6 | 3919.2 | - |
| Volume totale | 2000 | 4000 | - |
Tabella 2: Mezzo di isolamento dei nuclei (NIM). Mantenere il ghiaccio. Mescolare a vortice.
2. Polverizzazione di tessuto congelato ( Figura 2A)
- Impostare la workstation per l'omogeneizzazione.
- Riempire la bombola con azoto liquido (LN2).
ATTENZIONE: quando si lavora con LN2, indossare sempre occhiali e guanti criogenici. - Ottieni 2 mortai, 1 pestello, 1 spatola a micro-paletta, 1 bocchettone di vetro e 1 pestello in acciaio inossidabile per il douncer automatico.
- Impostare il douncer automatico.
- Riempi un bicchiere di ghiaccio e pre-raffredda il bicchierino.
- Riempire la bombola con azoto liquido (LN2).
- Riempire i 2 mortai (contenenti il pestello e la spatola) con LN2 per raffreddare gli strumenti. Lasciare evaporare l'LN2 e ripetere.
- Mentre gli strumenti si raffreddano, aggiungere 1 ml di HB alla lancia di vetro.
- Riempire entrambi i mortai con LN2 un'ultima volta e versare il campione IMAT da 50 mg in uno dei mortai.
- Polverizzare l'IMAT usando il pestello premendolo delicatamente sul pezzo di tessuto per romperlo in piccoli pezzi. Assicurati che tutti i pezzi siano polverizzati.
- L'LN2 evaporerà lentamente polverizzando il tessuto. Quando il tessuto è adeguatamente polverizzato e sono rimasti ancora 1/4 - 1/2 di un mortaio di LN2 , inclinare il mortaio verso il bordo del mortaio per raccogliere il tessuto polverizzato dal labbro. Lasciare evaporare completamente l'LN2 .
- Immediatamente dopo che l'ultimo LN2 è evaporato, raccogliere il tessuto polverizzato nella bocchetta di vetro contenente 1 mL di HB.
3. Omogeneizzazione di tessuto polverizzato
- Omogeneizzare il tessuto polverizzato utilizzando il douncer automatico. Portare il bicchiere su e giù per il pestello in acciaio inossidabile per 10 colpi in avanti, seguiti da 10 colpi in senso inverso.
- Assicurarsi che la soluzione sia torbida dopo l'omogeneizzazione e non contenga pezzi di tessuto visibili. Spesso ci si aspetta un colore rosa chiaro a causa della contaminazione con il tessuto muscolare.
- Trasferire l'omogeneizzato in una provetta pre-raffreddata da 1,7 mL a basso legame su ghiaccio.
- Utilizzare 400 μl di HB per sciacquare la lancia per assicurarsi che tutto il materiale sia trasferito e aggiungerlo alla provetta.
NOTA: È possibile elaborare due campioni alla volta. Per fare ciò, raddoppia la quantità di HB e NIM. Polverizzare e omogeneizzare un campione di tessuto e subito dopo polverizzare e omogeneizzare il secondo campione di tessuto per poter eseguire le fasi di isolamento e pulizia in parallelo.
4. Isolamento e bonifica dei nuclei ( Figura 2B)
- Aggiungere 14 μl di Triton-X (10%) all'omogenato per una concentrazione dello 0,1%.
- Tenere il tubo sul ghiaccio e al buio per 10-15 minuti mentre si gira vorticosamente ogni 3 minuti.
- Pre-bagnare un filtro cellulare da 100 μm e uno da 40 μm (per campione) con 100 μL di RT DPBS per ciascuno in una provetta conica da 50 mL.
- Filtrare l'omogenato attraverso il filtro cellulare da 100 μm.
- Sciacquare la provetta da 1,7 mL con 400 μL di HB e filtrare attraverso il filtro cellulare da 100 μm.
- Quindi, filtrare la soluzione attraverso il filtro cellulare da 40 μm.
- Trasferire una quantità uguale di soluzione in due provette pre-raffreddate da 1,7 mL a basso legame corrispondenti a ~900 μL in ciascuna provetta.
- Centrifugare le provette per 10 minuti a 2700 x g a 4 °C. Dovrebbe esserci un piccolo pellet visibile dopo la centrifugazione.
- Rimuovere ed eliminare lo strato lipidico superiore e il surnatante rimanente, lasciando ~50 μL di soluzione dalla prima provetta.
- Ripetere per il secondo tubo.
- Risospendere accuratamente il pellet nella prima provetta pipettandolo delicatamente su e giù 20 volte e trasferirlo in una nuova provetta a basso legame da 1,7 mL. Evita di creare bolle.
- Ripetere questa procedura per la seconda provetta e trasferire la soluzione risoluta nella stessa provetta.
- Aggiungere 500 μl di NIM e miscelare con il pipettaggio.
- Centrifugare la provetta con una bilancia a 1000 x g per 10 minuti a 4 °C.
- Rimuovere il surnatante, lasciando ~50 μL, e pipettare delicatamente su e giù fino a quando il pellet non viene risospeso. Facoltativamente, trasferire il pellet risospeso in un nuovo tubo pulito se rimane del residuo di lipidi sul lato del tubo.
- Aggiungere 200 μl di NIM e miscelare mediante pipettaggio.
5. Colorazione e conteggio dei nuclei (Figura 2C e Figura 3)
NOTA : Per facilitare il conteggio, impostare un protocollo di "conteggio dei nuclei" su un contatore automatico di cellule, poiché la regolazione del campo luminoso e dei canali DAPI può influire notevolmente sul conteggio. Regolare i canali in modo che vengano catturati solo i nuclei e non i detriti. Assicurarsi che il canale del campo chiaro contrassegni solo gli "oggetti" che presentano anche una macchia DAPI.
- Aggiungere 1 goccia della soluzione di colorazione per cellule vive e lasciarla al buio, con ghiaccio, per 15 minuti.
- Filtrare la soluzione attraverso un filtro cellulare da 30 μm.
- Miscelare la soluzione di nuclei mediante pipettaggio e aggiungere 10 μl della soluzione a un vetrino della camera di conteggio delle cellule.
- Conta i nuclei utilizzando un contatore di cellule automatizzato.
NOTA: La concentrazione ottimale è di 1000 nuclei/μL corrispondenti a 1,0 x 106/mL.- Assicurarsi che non siano presenti grumi di nuclei, in quanto ciò potrebbe ostruire il chip per la generazione di goccioline di singoli nuclei (Figura 3).
- Se la concentrazione dei nuclei non è sufficientemente alta, centrifugare la soluzione a 1000 x g per 10 minuti a 4 °C per ottenere una pelletta, rimuovere il surnatante e risospendere in un volume più piccolo.
- Se il grado di detriti nella soluzione è elevato, risospendere la soluzione di nuclei in un volume maggiore di NIM (cioè 1 mL) e filtrare nuovamente attraverso un filtro cellulare da 30 μm. Quindi centrifugare a 1000 x g per 10 minuti a 4 °C e risospendere in volume appropriato in relazione alla concentrazione dei nuclei.
- Dopo aver ottenuto la concentrazione dei nuclei, procedere direttamente alla prima fase della preparazione della libreria.
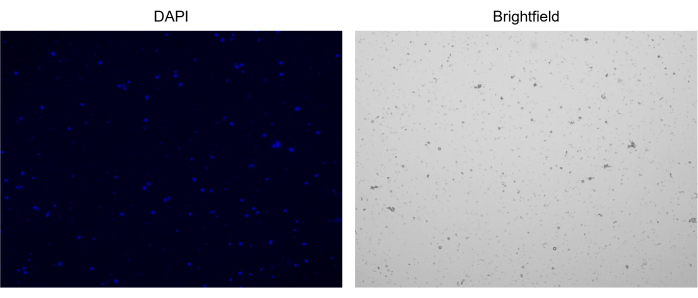
Figura 3: Colorazione di nuclei isolati. Immagine dal contatore cellulare dei nuclei colorati con NucBlue/DAPI (immagine a sinistra) e la corrispondente immagine in campo chiaro (immagine a destra). La presenza di piccole quantità di detriti è evidente nell'immagine in campo chiaro. Il contatore automatico di celle utilizzato qui non dispone di un'opzione per includere le barre della scala. Clicca qui per visualizzare una versione più grande di questa figura.
6. Parametri di preparazione e sequenziamento della libreria
- Fare riferimento a un protocollo completo per la preparazione della libreria utilizzando l'approccio a nuclei singoli basato su goccioline disponibile sulla pagina web17 del fornitore.
- Puntare a un recupero mirato di 10.000 nuclei. Tuttavia, per i campioni con un alto livello di detriti o nuclei fragili, ci si aspetterebbe un numero inferiore di nuclei recuperati.
- Conservare i campioni a 4°C per un massimo di 72 ore dopo il passaggio 2.3. nel protocollo di preparazione della libreria per combinare l'elaborazione di più campioni in parallelo. A tale scopo, elaborare due campioni fino al passaggio 2.3 per due giorni consecutivi e il terzo giorno elaborare i 4 campioni insieme dal passaggio 3 in avanti nel protocollo di preparazione della libreria.
- Parametri di sequenziamento: Sequenza su una piattaforma di sequenziamento che mira a 50.000 letture paired-end per nucleo.
NOTA: I dati presentati in questo protocollo sono stati sequenziati sulla piattaforma NovaSeq 6000, con l'obiettivo di 50.000 letture paired-end per nuclei.
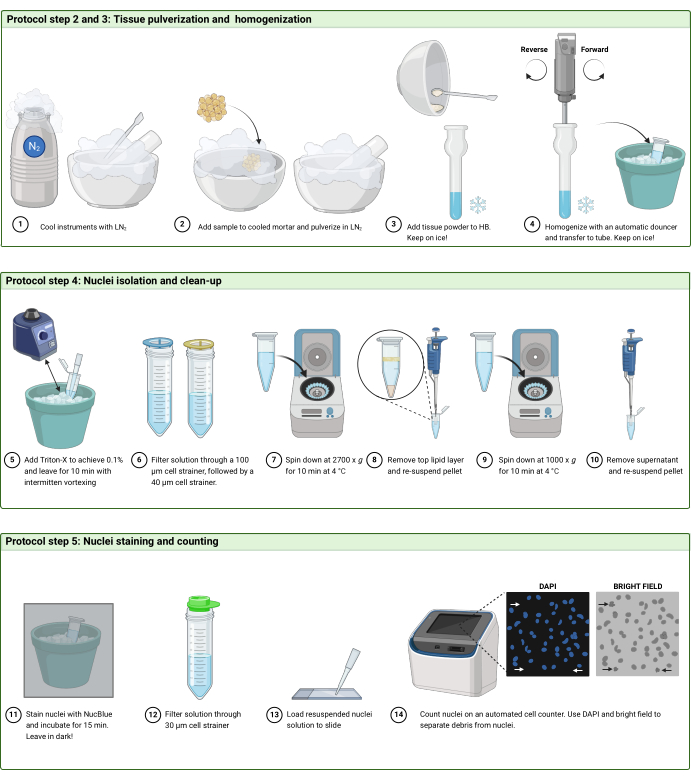
Figura 2: flusso di lavoro del protocollo. Illustrazione schematica del flusso di lavoro nei passaggi 2 e 3 (A), 4 (B) e 5 (C) del protocollo. La figura è stata creata con BioRender.com. Clicca qui per visualizzare una versione più grande di questa figura.
7. Elaborazione e analisi dei dati
NOTA : In questo protocollo, vengono brevemente introdotti alcuni dei software e dei pacchetti R consigliati utilizzati per elaborare i dati di sequenziamento risultanti, concentrandosi sui passaggi successivi alla pre-elaborazione iniziale (Tabella 3). Questo studio fornisce metriche generali di controllo della qualità (QC) e un esempio di approssimazione e proiezione uniforme del manifold (UMAP) nella Figura 4. Tuttavia, una descrizione approfondita dell'analisi bioinformatica esula dall'ambito di questo protocollo. Pertanto, i lettori possono fare riferimento alla recente revisione sulle migliori pratiche per l'analisi di singole cellule di Heumos et al.18.
- Pre-elaborazione dei dati di sequenziamento
- Mappare i singoli nuclei letti nel genoma di riferimento umano GRCh38.
- Includi le letture dell'introne nel conteggio.
- Eseguire il controllo qualità e il filtraggio dei dati utilizzando il pacchetto Seurat R 19.
- Calcola un punteggio di complessità cellulare dividendo il numero log(10) di geni rilevati per il numero log(10) di letture rilevate.
- Traccia le metriche QC più importanti utilizzando un istogramma o un grafico a violino, incluso il numero di geni rilevati per nucleo, la percentuale di lettura mitocondriale e il punteggio di complessità cellulare.
- Filtra i nuclei con meno di 200 o più di 10.000 geni per nucleo, più del 10% di letture mitocondriali e un punteggio di complessità inferiore a 0,8.
- Normalizza i dati ed esegui la riduzione della dimensionalità.
- Utilizzare la funzione SCTransform di Seurat per normalizzare i dati utilizzando 2000 funzioni variabili.
- Raggruppare i dati usando le funzioni seguenti del pacchetto Seurat R: RunPCA, FindNeighbors, FindClusters e RunUMAP.
- Tracciare un UMAP per visualizzare il clustering dei dati.
- Filtra i doppietti previsti utilizzando il pacchetto DoubletFinder R 20 e raggruppa nuovamente i dati.
- Annotare i cluster utilizzando marcatori genici noti dei tipi di cellule che si prevede siano presenti nel tessuto (approccio supervisionato) o in base ai primi 5 geni differenzialmente espressi tra i cluster (approccio non supervisionato).
- Utilizzare decontX21per determinare il grado di contaminazione dell'RNA ambientale e per regolare la matrice di espressione genica per l'RNA ambientale.
- Includi la matrice genica grezza come sfondo.
- Salvare l'oggetto Seurat per l'esplorazione futura dei dati.
NOTA: Il codice per l'analisi QC e clustering è disponibile nel file supplementare 1.
| Pacchetti software/R utilizzati nel flusso di lavoro dei dati | Software/pacchetti alternativi | Fase di lavorazione |
| CellRanger | STARsolo, kallisto | Ritaglio, allineamento, mappatura |
| Seurat | SingleCellExperiment, Cellranger | Controllo qualità, analisi ed esplorazione dei dati |
| DoubletFinder | scds, scdblFinder, Scrublet | Rilevamento doppietto |
| DecontX | ZuppaX, CellBender | Regolazione dell'RNA ambientale |
Tabella 3: Software/strumenti per il flusso di lavoro dei dati.
Access restricted. Please log in or start a trial to view this content.
Risultati
Questo flusso di lavoro è stato progettato per guidare l'elaborazione di campioni IMAT umani congelati per ottenere profili di espressione genica con risoluzione di singoli nuclei, consentendo l'identificazione del tipo di cellula. Qui viene presentato un campione IMAT rappresentativo di un partecipante allo studio SOMMA.
Il primo passo di qualsiasi analisi dei dati snRNA-seq è valutare la qualità dei dati per identificare i nuclei di scarsa qualità, che dovrebbero potenzialmente essere ri...
Access restricted. Please log in or start a trial to view this content.
Discussione
La collaborazione con IMAT presenta diverse sfide intrinseche. Oltre alla sua limitata accessibilità, la resa del materiale campione è spesso molto scarsa e la "contaminazione" del muscolo scheletrico è quasi impossibile da evitare. Per ottenere il campione della migliore qualità, è necessario penetrare la fascia muscolare quando si inserisce l'ago da biopsia (per assicurarsi di non raccogliere tessuto adiposo sottocutaneo) e rimuovere quanto più tessuto muscolare possibile sezionando il campione al microscopio sub...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori ringraziano Bryan Bergman, PhD presso l'Università del Colorado, per aver fornito l'immagine della biopsia IMAT nella Figura 1C dello studio MoTrIMAT (R01AG077956). Siamo grati per lo Studio dei Muscoli, della Mobilità e dell'Invecchiamento che fornisce il campione IMAT da cui i dati sono mostrati nella sezione dei risultati rappresentativi. Il National Institute on Aging (NIA) ha finanziato lo studio sui muscoli, la mobilità e l'invecchiamento (SOMMA; R01AG059416) e i relativi studi complementari SOMMA AT (R01AG066474) e SOMMA Knee OA (R01AG070647). Il supporto all'infrastruttura di studio è stato finanziato in parte dal NIA Claude D. Pepper Older American Independence Centers presso l'Università di Pittsburgh (P30AG024827) e la Wake Forest University (P30AG021332) e dagli Istituti di Scienze Cliniche e Traslazionali, finanziati dal National Center for Advancing Translational Science, presso la Wake Forest University (UL1 0TR001420).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.2 µm corning syringe filters | Millipore Sigma | CLS431229 | |
| 1.7 mL DNA LoBind tubes | Eppendorf | 22431021 | low-bind tubes |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 100x protease inhibitor | Thermo Fisher Scientific | 78437 | |
| 10X Magnetic Separator | 10X Genomics | 230003 | |
| 10X Vortex Adapter | 10X Genomics | 330002 | |
| 15 mL canonical tubes | Sarstedt | 6,25,54,502 | |
| 2100 Bioanalyzer | Agilent | G2939BA | |
| 50 mL conical tubes | Sarstedt | 6,25,47,254 | |
| CellRanger | Genomics | N/A | |
| Chromium iX accesory kit | 10X Genomics | PN1000323 | |
| Chromium iX Controller | 10X Genomics | PN1000326 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | PN1000127 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1 | 10X Genomics | PN1000129 | |
| Chromium Next GEM Single Cell GEM Kit v3.1 | 10X Genomics | PN1000130 | |
| Countess 3 Automated Cell Counter | Thermo Fisher Scientific | AMQAX2000 | Automated cell counter |
| Countess cell counting chamber slides | Thermo Fisher Scientific | C10228 | |
| DoubletFinder | R | N/A | |
| DPBS (no calcium, no magnesium) | Thermo Fisher Scientific | 14190144 | |
| DTT | Thermo Fisher Scientific | R0861 | |
| Dual Index Kit TT Set A, 96 rxns | 10X Genomics | PN1000215 | |
| Dynabeads MyOne SILANE | 10X Genomics | PN2000048 | |
| Falcon 100 µm Cell strainer | Corning Life Science | 352360 | |
| Falcon 40 µm Cell strainer | Corning Life Science | 352340 | |
| Glycerin (glycerol), 50% (v/v) Aqueous Solution | Ricca Chemical Company | 3290-32 | |
| KCL | Thermo Fisher Scientific | AM9640G | |
| Library Construction Kit v3.1 | 10X Genomics | PN1000196 | |
| MACS SmartStrainers (30µm) | Miltenyi Biotec | 130-098-458 | |
| Mastercycler Nexus Gradient Thermal cycler | Eppendorf | 6331000017 | |
| MgCl2 | Ambion | AM9530G | |
| Mortar and pestel | Health care logistics | 14075 | |
| NucBlue Live Ready Probes Reagent | Thermo Fisher Scientific | R37605 | |
| Nuclease Free Water (not DEPC treated) | Thermo Fisher Scientific | AM9930 | |
| Probumin Bovine Serum Albumin Fatty Acid Free, Powder | Sigma-Aldrich | 820024 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Ribolock RNAse inhibitor | Thermo Fisher Scientific | EO0382 | |
| Seurat | R | N/A | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SUPERasin 20 U/µL | Thermo Fisher Scientific | AM2695 | |
| ThermoMixer C | Eppendorf | 5382000015 | |
| Tissue homogenizer | Glass-Col | 099C K54 | |
| Tris buffer pH 8.0 | Thermo Fisher Scientific | AM9855G | |
| Triton X-100 | Thermo Fisher Scientific | AC327372500 | |
| UltraPure 0.5M EDTA pH 8.0 | Gibco | 15575020 |
Riferimenti
- Goodpaster, B. H., Bergman, B. C., Brennan, A. M., Sparks, L. M. Intermuscular adipose tissue in metabolic disease. Nat Rev Endocrinol. 19 (5), 285-298 (2023).
- Sparks, L. M., Goodpaster, B. H., Bergman, B. C. The metabolic significance of intermuscular adipose tissue: Is IMAT a friend or a foe to metabolic health. Diabetes. 70 (11), 2457-2467 (2021).
- Gallagher, D., et al. Adipose tissue in muscle: A novel depot similar in size to visceral adipose tissue. Am J Clin Nutr. 81 (4), 903-910 (2005).
- Manini, T. M., et al. Reduced physical activity increases intermuscular adipose tissue in healthy young adults. Am J Clin Nutr. 85 (2), 377-384 (2007).
- Addison, O., Marcus, R. L., LaStayo, P. C., Ryan, A. S. Intermuscular fat: A review of the consequences and causes. Int J Endocrinol. 2014, 309570(2014).
- Goodpaster, B. H., et al. Obesity, regional body fat distribution, and the metabolic syndrome in older men and women. Arch Intern Med. 165 (7), 777-783 (2005).
- Goodpaster, B. H., Thaete, F. L., Kelley, D. E. Thigh adipose tissue distribution is associated with insulin resistance in obesity and in type 2 diabetes mellitus. Am J Clin Nutr. 71 (4), 885-892 (2000).
- Goodpaster, B. H., et al. Association between regional adipose tissue distribution and both type 2 diabetes and impaired glucose tolerance in elderly men. Diabetes Care. 26 (2), 372-379 (2003).
- Sachs, S., et al. Intermuscular adipose tissue directly modulates skeletal muscle insulin sensitivity in humans. Am J Physiol Endocrinol Metab. 316 (5), E866-E879 (2019).
- Ford, H., Liu, Q., Fu, X., Strieder-Barboza, C. White adipose tissue heterogeneity in the single-cell era: From mice and humans to cattle. Biology (Basel). 12 (10), 1289(2023).
- Wang, L., et al. Single-nucleus and bulk RNA sequencing reveal cellular and transcriptional mechanisms underlying lipid dynamics in high marbled pork. NPJ Sci Food. 7 (1), 23(2023).
- Li, J., et al. Identification of diverse cell populations in skeletal muscles and biomarkers for intramuscular fat of chicken by single-cell RNA sequencing. BMC Genomics. 21 (1), 752(2020).
- Lyu, P., Qi, Y., Tu, Z. J., Jiang, H. Single-cell RNA sequencing reveals heterogeneity of cultured bovine satellite cells. Front Genet. 12, 742077(2021).
- Fitzgerald, G., et al. MME+ fibro-adipogenic progenitors are the dominant adipogenic population during fatty infiltration in human skeletal muscle. Commun Biol. 6 (1), 111(2023).
- Cummings, S. R., et al. The study of muscle, mobility and aging (SOMMA): A unique cohort study about the cellular biology of aging and age-related loss of mobility. J Gerontol A Biol Sci Med Sci. 78 (11), 2083-2093 (2023).
- Whytock, K. L., et al. Isolation of nuclei from frozen human subcutaneous adipose tissue for full-length single-nuclei transcriptional profiling. STAR Protoc. 4 (1), 102054(2023).
- 10x Genomics. Chromium Single Cell 3' Reagent Kits User Guide (v3.1 Chemistry Dual Index), Document Number CG000315 RevE. , Available from: https://cdn.10xgenomics.com/image/upload/v1668017706/support-documents/CG000315_ChromiumNextGEMSingleCell3-_GeneExpression_v3.1_DualIndex__RevE.pdf (2022).
- Heumos, L., et al. Best practices for single-cell analysis across modalities. Nat Rev Genet. 24 (1), 550-572 (2023).
- Hao, Y., et al. Dictionary learning for integrative, multimodal and scalable single-cell analysis. Nat Biotechnol. 42 (2), 293-304 (2023).
- McGinnis, C. S., Murrow, L. M., Gartner, Z. J. DoubletFinder: Doublet detection in single-cell RNA sequencing data using artificial nearest neighbors. Cell Syst. 8 (4), 329-337 (2019).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (2), 57(2020).
- Common considerations for quality control filters for single cell RNA-seq data. 10X Genomics. , Available from: https://www.10xgenomics.com/analysis-guides/common-considerations-for-quality-control-filters-for-single-cell-rna-seq-data (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Mol Syst Biol. 15 (6), e8746(2019).
- Emont, M. P., et al. A single-cell atlas of human and mouse white adipose tissue. Nature. 603 (7903), 926-933 (2022).
- Hildreth, A. D., et al. Single-cell sequencing of human white adipose tissue identifies new cell states in health and obesity. Nat Immunol. 22 (5), 639-653 (2021).
- Whytock, K. L., et al. Single cell full-length transcriptome of human subcutaneous adipose tissue reveals unique and heterogeneous cell populations. iScience. 25 (8), 104772(2022).
- Probst, V., et al. Benchmarking full-length transcript single cell mRNA sequencing protocols. BMC Genomics. 23 (1), 860(2022).
- CG000148 Rev A Technical Note - Resolving cell types as a function of read depth and cell number. Technical note. 10X Genomics. , Available from: https://assets.ctfassets.net/an68im79xiti/6gDArDPBTOg4IIkYEO2Sis/803be2286bb a5ca67f353e6baf68d276/CG000148_10x_Technical _Note_Resolving_Cell_Types_as_Function_of_ Read_Depth_Cell_Number_RevA.pdf (2018).
- Gupta, A., et al. Characterization of transcript enrichment and detection bias in single-nucleus RNA-seq for mapping of distinct human adipocyte lineages. Genome Res. 32 (2), 242-257 (2022).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648(2018).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. J Am Soc Nephrol. 30 (1), 23-32 (2019).
- Kim, N., Kang, H., Jo, A., Yoo, S. -A., Lee, H. -O. Perspectives on single-nucleus RNA sequencing in different cell types and tissues. J Pathol Transl Med. 57 (1), 52-59 (2023).
- Avila Cobos, F., Alquicira-Hernandez, J., Powell, J. E., Mestdagh, P., De Preter, K. Benchmarking of cell type deconvolution pipelines for transcriptomics data. Nat Commun. 11 (1), 5650(2020).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon