Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Выделение ядер из межмышечной жировой ткани человека и последующее секвенирование одноядерных РНК
В этой статье
Резюме
Биология межмышечной жировой ткани (IMAT) в значительной степени не изучена из-за ограниченной доступности тканей человека. Здесь мы представляем подробный протокол выделения ядер и подготовки библиотеки замороженного человеческого IMAT для секвенирования одноядерных РНК для определения клеточного состава этого уникального жирового депо.
Аннотация
Межмышечная жировая ткань (IMAT) – это относительно малоизученное жировое депо, расположенное между мышечными волокнами. Содержание IMAT увеличивается с возрастом и ИМТ и связано с метаболическими и мышечными дегенеративными заболеваниями; однако понимание биологических свойств IMAT и его взаимодействия с окружающими мышечными волокнами сильно отсутствует. В последние годы секвенирование одиночных клеток и ядер РНК позволило нам получить атласы нескольких тканей человека, специфичные для разных типов клеток. Тем не менее, клеточный состав IMAT человека остается в значительной степени неизученным из-за неотъемлемых проблем, связанных с его доступностью из биопсии у человека. В дополнение к ограниченному количеству собранной ткани, обработка IMAT человека затруднена из-за его близости к скелетной мышечной ткани и фасциям. Насыщенная липидами природа адипоцитов делает его несовместимым с выделением одиночных клеток. Следовательно, секвенирование РНК с одним ядром является оптимальным для получения многомерной транскриптомики с разрешением одной клетки и обеспечивает потенциал для раскрытия биологии этого депо, включая точный клеточный состав IMAT. Здесь мы представляем подробный протокол выделения ядер и подготовки библиотеки замороженного человеческого IMAT для секвенирования одноядерных РНК. Этот протокол позволяет профилировать тысячи ядер с использованием капельного подхода, тем самым обеспечивая возможность обнаружения редких и малораспространенных типов клеток.
Введение
Межмышечная жировая ткань (IMAT) представляет собой эктопическое жировое депо, расположенное между мышечными волокнами и вокруг них1. Как подробно описано в недавнем обзоре Goodpaster et al., IMAT может быть обнаружен с помощью компьютерной томографии (КТ) высокого разрешения и магнитно-резонансной томографии (МРТ) (рис. 1A, B) и обнаруживается вокруг и внутри мышечных волокон повсему телу. Количество IMAT сильно варьируется у разных людей и зависит от ИМТ, возраста, пола, расы и сидячего образа жизни 2,3,4. Кроме того, отложение IMAT обычно наблюдается при патологических состояниях, связанных с мышечной дегенерацией5, и многочисленные исследования документально подтвердили увеличение массы IMAT у людей с ожирением, диабетом 2 типа, метаболическим синдромом и резистентностью к инсулину 6,7,8,9. Тем не менее, клеточные и биологические свойства IMAT только начинают раскрываться. Ограниченная доступность и различия в местах расположения и содержимом IMAT по всему телу затруднили сбор образцов из этого уникального жирового депо2. Кроме того, образцы легко «загрязняются» скелетными мышцами (СМ) после сбора, что затрудняет расшифровку разделения между биологическим вкладом различных тканей (рис. 1C). С этой целью секвенирование одноядерных РНК (snRNA-seq), которое привлекло значительное внимание в течение последнего десятилетия, служит идеальной методологией, позволяющей разделить паттерны экспрессии генов, полученных от IMAT и SM, с разрешением для одной клетки. Кроме того, выделение ядер особенно полезно для жировой ткани из-за больших липидных адипоцитов, которые невозможно диссоциировать на одноклеточную суспензию без нарушения целостности клеток. Наконец, эта технология обладает потенциалом для открытия новых маркеров IMAT-специфичных адипоцитов и раскрытия состава и присутствия различных популяций клеток-предшественников, а также изучения вариаций клеточного состава в патологических и нормальных условиях.
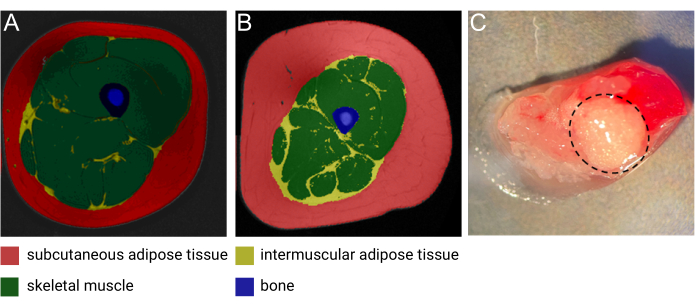
Рисунок 1: Изображения IMAT. Репрезентативное магнитно-резонансное (МРТ) изображение IMAT от (А) худощавой женщины среднего возраста и (Б) мужчины среднего возраста с ожирением. Красный: подкожная жировая клетчатка, желтый: межмышечная жировая ткань, зеленый: скелетные мышцы, синий: костные. Изображение любезно предоставлено Хизер Корннелл, Институт трансляционных исследований AdventHealth. (C) Свежий образец ткани с IMAT (обведен пунктирной черной линией). Изображение любезно предоставлено Меган Хопф из Института трансляционных исследований AdventHealth и Брайаном Бергманом из Университета Колорадо. Этот рисунок был изменен с разрешения Goodpaster et al.1. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
В области животноводства был опубликован ряд исследований, в которых изучалось мраморность мяса (в частности, IMAT) у свиней, кур и крупного рогатого скота с использованием одноклеточного (sc) и snRNA-seq10. Эти исследования выявили несколько субпопуляций адипоцитов и маркеров потенциальных клеток-предшественников IMAT 11,12,13; однако неизвестно, транслируются ли эти клеточные композиции в IMAT человека. Насколько нам известно, только в одном исследовании изучалась клеточная гетерогенность мышц человека с жировой инфильтрацией, полученная от пациентов мужского пола с остеоартрозом тазобедренного сустава, с использованием snRNA-seq14. Исследователи сообщили о небольшой популяции адипоцитов и нескольких субпопуляциях фиброадипогенных предшественников (FAP) в большой популяции миоядер14. В нашем исследовании впервые разработан метод прямого опроса IMAT, вручную препарированного из мышц человека, на предмет клеточного состава с помощью snRNA-seq.
Важно отметить, что протоколы для snRNA-seq должны быть адаптированы для конкретной изучаемой ткани, поскольку количество доступной ткани и физические свойства конкретной ткани будут диктовать оптимальные этапы обработки. Выход ткани для IMAT обычно невелик, часто не превышает 50 мг, даже при проведении биопсии под ультразвуковым контролем. Следовательно, правильная обработка этой дефицитной ткани имеет важное значение. Мы считаем, что этот протокол послужит ценным ресурсом для исследователей, изучающих IMAT человека.
Access restricted. Please log in or start a trial to view this content.
протокол
Образец, использованный для этого протокола, был частью Исследования мышц, подвижности и старения (SOMMA)15, которое было одобрено Институциональным наблюдательным советом Западной группы IRB-Copernicus (WCG) и проводилось в соответствии с Хельсинкской декларацией. Участники предоставили письменное информированное согласие на свое участие в исследовании.
Примечание: Этот протокол адаптирован по сравнению с предыдущим протоколом с использованием 100 мг подкожной жировой клетчатки брюшной полости человека на платформе на основе нанолунок16. Текущий протокол оптимизирован для 50 мг IMAT человека и подготовки библиотеки с использованием платформы на основе капель. Может потребоваться дальнейшая оптимизация этого протокола для выделения ядер из нечеловеческого IMAT или других жировых депо.
1. Приготовление буферов и реагентов (табл. 1 и табл. 2)
ПРИМЕЧАНИЕ: Приготовьте буферы свежими в день эксперимента и не используйте их повторно.
- Предварительно охладите центрифугу до 4 °C.
- Приготовьте гомогенизационный буфер и среду для выделения ядер.
- Возьмите два ведра льда и предварительно охладите 2 конические пробирки объемом 15 мл.
- Смешайте все реагенты для гомогенизационного буфера (HB) в конической пробирке объемом 15 мл в порядке, указанном в таблице 1. Держите на льду. Перемешайте путем вортексинга.
- Смешайте все реагенты для среды выделения ядер (NIM) в конической пробирке объемом 15 мл в списке, упорядоченном в таблице 2. Держите на льду. Перемешайте путем вортексинга.
- Приготовьте 10% Triton-X, добавив 100 μL Triton X-100 к 900 μL воды, не содержащей Nuclease. Вортекс для обеспечения правильного перемешивания. Хранить при комнатной температуре (RT).
| Реагент | Объем (μл) | Конечная концентрация (мМ) | |
| 1x | в 2 раза | ||
| 1 М MgCl2 | 10 | 20 | 5 |
| 1 м Трис буфер, pH 8,0 | 20 | 40 | 10 |
| 2 м KCl | 25 | 50 | 25 |
| 1,5 м сахарозы (-4оС) | 334 | 668 | 250 |
| 1 мМ DTT | 2 | 4 | 0,001 (~1 μМ) |
| 100-кратный ингибитор протеазы | 20 | 40 | 1x |
| Суперазин 20 Ед/мкл | 40 | 80 | 0,4 Ед/мкл |
| Вода без нуклеаз | 1549 | 3098 | - |
| Общий объем | 2000 | 4000 | - |
Таблица 1: Гомогенизационный буфер (HB). Держите на льду. Перемешайте путем вортексинга.
| Реагент | Объем (μл) | Конечная концентрация (мМ) | |
| 1x | в 2 раза | ||
| ЭДТА | 0.4 | 0.8 | 0.1 |
| Ингибитор риболок РНКазы (40 ЕД/мкл) | 40 | 80 | 0,8 Ед/мкл |
| 1% BSA-PBS (-/-) | 1959.6 | 3919.2 | - |
| Общий объем | 2000 | 4000 | - |
Таблица 2: Среда для выделения ядер (NIM). Держите на льду. Перемешайте путем вортексинга.
2. Измельчение замороженной ткани (Рисунок 2А)
- Настройте рабочую станцию для гомогенизации.
- Заполните канистру жидким азотом (LN2).
ВНИМАНИЕ: при работе с LN2 всегда надевайте защитные очки и криоперчатки. - Получите 2 ступки, 1 пестик, 1 шпатель для микросовка, 1 стеклянный наконечник и 1 пестик из нержавеющей стали для автоматического прыгателя.
- Настройте автоматический вышибалу.
- Наполните стакан льдом и предварительно охладите стакан.
- Заполните канистру жидким азотом (LN2).
- Наполните 2 ступки (содержащие пестик и шпатель) LN2 , чтобы охладить инструменты. Дайте LN2 испариться и повторите.
- Пока приборы остывают, добавьте 1 мл HB в стеклянную жидкость.
- Заполните обе ступы LN2 в последний раз и вылейте образец IMAT 50 мг в одну из ступок.
- Измельчите IMAT с помощью пестика, осторожно надавливая на кусок ткани, чтобы разбить его на мелкие кусочки. Убедитесь, что все детали измельчены.
- LN2 будет медленно испаряться, измельчая ткань. Когда ткань будет как следует измельчена, и останется еще 1/4 - 1/2 раствора LN2 , наклоните раствор в сторону края раствора, чтобы собрать измельченную ткань за губу. Дайте LN2 полностью испариться.
- Сразу после того, как последний LN2 испарится, зачерпните измельченную ткань в стеклянную ткань, содержащую 1 мл HB.
3. Гомогенизация измельченной ткани
- Гомогенизируйте измельченную ткань с помощью автоматического вышибалы. Поднимите и опустите пестик из нержавеющей стали вверх и вниз на 10 ударов в прямом направлении, а затем на 10 ударов в обратном направлении.
- Следите за тем, чтобы раствор после гомогенизации был мутным и не содержал видимых кусочков ткани. Светло-розовый цвет часто ожидается из-за загрязнения мышечной тканью.
- Переложите гомогенат в предварительно охлажденную пробирку объемом 1,7 мл с низким связыванием на лед.
- Используйте 400 мкл HB для промывки накидки, чтобы убедиться, что весь материал перенесен, и добавьте его в пробирку.
ПРИМЕЧАНИЕ: Одновременно можно обрабатывать два образца. Для этого увеличьте количество HB и NIM в два раза. Измельчите и гомогенизируйте один образец ткани, а затем измельчите и гомогенизируйте второй образец ткани, чтобы можно было параллельно выполнять этапы изоляции и очистки.
4. Выделение и очистка ядер (Рисунок 2B)
- Добавьте 14 мкл Triton-X (10%) в гомогенат для получения концентрации 0,1%.
- Держите трубку на льду и в темноте в течение 10-15 минут, делая завихрение каждые 3 минуты.
- Предварительно намочите один фильтр 100 мкм и один фильтр 40 мкм (на образец) 100 мкл RT DPBS для каждого в конической пробирке объемом 50 мл.
- Отфильтруйте гомогенат через сетчатый фильтр 100 μm.
- Промойте пробирку объемом 1,7 мл 400 мкл HB и отфильтруйте через клеточное сетчатое фильтр 100 мкм.
- Далее отфильтруйте раствор через сетчатый фильтр 40 мкм.
- Перелейте равное количество раствора в две предварительно охлажденные пробирки объемом 1,7 мл с низким уровнем связывания, что соответствует ~900 мкл в каждой пробирке.
- Центрифугируйте пробирки в течение 10 мин при температуре 2700 x g при 4 °C. После центрифугирования должна быть видна небольшая гранула.
- Удалите и выбросьте верхний липидный слой и оставшуюся надосадочную жидкость, оставив ~50 мкл раствора из первой пробирки.
- Повторите то же самое для второй трубки.
- Тщательно ресуспендируйте гранулу в первой пробирке, аккуратно пипетируя вверх и вниз 20 раз, и переложите в новую пробирку объемом 1,7 мл с низким уровнем связывания. Избегайте образования пузырей.
- Повторите эту процедуру для второй пробирки и перенесите ресуспендированный раствор в ту же пробирку.
- Добавьте 500 μL NIM и смешайте с пипеткой.
- Центрифугируйте пробирку с весами при давлении 1000 x g в течение 10 минут при 4 °C.
- Удалите надосадочную жидкость, оставив ~50 μл, и аккуратно пипетируйте вверх и вниз, пока гранула не будет повторно суспендирована. При желании переложите ресуспендированную гранулу в новую чистую пробирку, если на боковой стороне пробирки осталось немного липидных остатков.
- Добавьте 200 μL NIM и перемешайте с помощью пипетирования.
5. Окрашивание и подсчет ядер (Рисунок 2C и Рисунок 3)
ПРИМЕЧАНИЕ: Чтобы облегчить подсчет, настройте протокол «подсчета ядер» на автоматическом счетчике клеток, так как регулировка светлого поля и каналов DAPI может сильно повлиять на подсчет. Отрегулируйте каналы так, чтобы захватывались только ядра, а не мусор. Убедитесь, что канал светлого поля помечает только «объекты», которые также имеют пятно DAPI.
- Добавьте 1 каплю раствора для окрашивания живых клеток и оставьте в темноте, на льду, на 15 минут.
- Отфильтруйте раствор через сетчатое фильтр 30 мкм.
- Смешайте раствор ядер с помощью пипетирования и добавьте 10 мкл раствора в предметное стекло камеры для подсчета клеток.
- Подсчитайте ядра с помощью автоматического счетчика клеток.
ПРИМЕЧАНИЕ: Оптимальная концентрация составляет 1000 ядер/мкл, что соответствует 1,0 x 106/мл.- Убедитесь, что в них нет скоплений ядер, так как это может засорить чип для генерации одиночных ядер (Рисунок 3).
- Если концентрация ядер недостаточно высока, уменьшите температуру раствора при 1000 x g в течение 10 минут при 4 °C, чтобы получить гранулу, удалите надосадочную жидкость и снова суспендируйте в меньшем объеме.
- Если степень загрязнения в растворе высокая, ресуспендируйте раствор ядер в большем объеме NIM (т.е. 1 мл) и снова отфильтруйте через клеточное сетчатое фильтр с плотностью 30 мкм. Затем вращают при 1000 x g в течение 10 мин при 4 °C и повторно суспендируют в соответствующем объеме по отношению к концентрации ядер.
- После получения концентрации ядер приступайте непосредственно к первому этапу подготовки библиотеки.
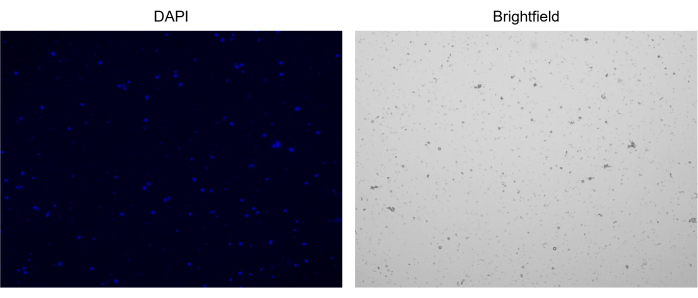
Рисунок 3: Окрашивание выделенных ядер. Изображение с клеточного счетчика ядер, окрашенных NucBlue/DAPI (левое изображение) и соответствующее изображение яркого поля (правое изображение). Присутствие незначительного количества мусора очевидно на ярком изображении поля. Используемый здесь автоматический счетчик ячеек не имеет возможности включать масштабные линейки. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
6. Подготовка библиотеки и параметры секвенирования
- Обратитесь к подробному протоколу подготовки библиотеки с использованием одноядерного подхода на основе капель, доступному на веб-страницепоставщика услуг 17.
- Стремитесь к целевому извлечению ядер на уровне 10 000. Однако для образцов с высоким содержанием мусора или хрупких ядер ожидается меньшее количество извлеченных ядер.
- Храните образцы при температуре 4°C до 72 часов после шага 2.3. в протоколе подготовки библиотеки для совмещения обработки большего количества образцов параллельно. Для этого обрабатывайте два образца до шага 2.3 в течение двух дней подряд, а на третий день обрабатывайте 4 образца вместе, начиная с шага 3 и далее в протоколе подготовки библиотеки.
- Параметры секвенирования: Секвенирование на платформе секвенирования с целью 50 000 считываний парных концов на ядро.
Данные, представленные в этом протоколе, были секвенированы на платформе NovaSeq 6000 с целью 50 000 считываний с парными концами на ядро.
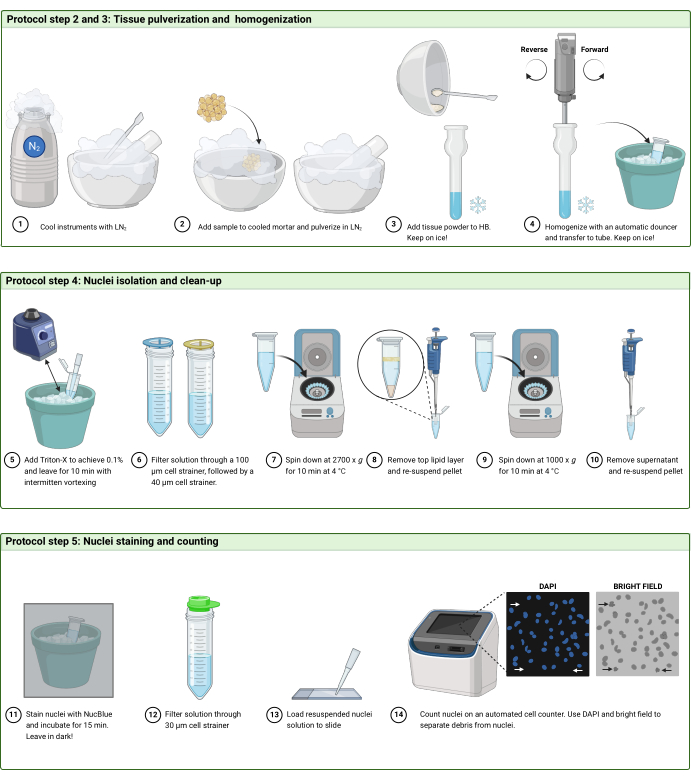
Рисунок 2: Рабочий процесс протокола. Схематическое изображение рабочего процесса на этапах (А) 2 и 3, (В) шаге 4 и (В) шаге 5 протокола. Фигура была создана с BioRender.com. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
7. Обработка и анализ данных
ПРИМЕЧАНИЕ: В этом протоколе кратко представлены некоторые из рекомендуемых программ и пакетов R, используемых для обработки полученных данных секвенирования, с акцентом на шаги после первоначальной предварительной обработки (Таблица 3). В этом исследовании представлены общие метрики контроля качества (QC) и пример равномерной аппроксимации и проекции многообразия (UMAP) на рисунке 4. Однако подробное описание биоинформатического анализа выходит за рамки данного протокола. Таким образом, читатели могут обратиться к недавнему обзору лучших практик анализа одиночных клеток, проведенному Heumos et al.18.
- Предварительная обработка данных секвенирования
- Сопоставьте чтения одиночных ядер с референсным геномом человека GRCh38.
- Включайте в подсчет чтения интронов.
- Выполнение контроля качества и фильтрации данных с помощью пакета Seurat R 19.
- Вычислите оценку сложности клетки, разделив log(10) количество обнаруженных генов на log(10) количество обнаруженных прочтений.
- Нанесите на график наиболее важные показатели контроля качества с помощью гистограммы или графика скрипки, включая количество генов, обнаруженных на ядро, процент митохондриального чтения и оценку сложности клеток.
- Отфильтруйте ядра с менее чем 200 или более 10 000 генов на ядро, более 10% митохондриальных прочтений и показателем сложности ниже 0,8.
- Нормализация данных и уменьшение размерности.
- Используйте функцию SCTransform от Seura для нормализации данных с использованием 2000 переменных признаков.
- Кластеризуйте данные с помощью следующих функций из пакета Seurat R: RunPCA, FindNeighbors, FindClusters и RunUMAP.
- Построение диаграммы UMAP для визуализации кластеризации данных.
- Отфильтруйте прогнозируемые дублеты с помощью пакета DoubletFinder R 20 и повторно кластеризуйте данные.
- Аннотируйте кластеры с использованием известных генных маркеров типов клеток, которые, как ожидается, присутствуют в ткани (контролируемый подход) или на основе 5 верхних дифференциально экспрессируемых генов между кластерами (неконтролируемый подход).
- Используйте decontX21для определения степени загрязнения окружающей РНК и корректировки матрицы экспрессии генов для окружающей РНК.
- Включите необработанную матрицу генов в качестве фона.
- Сохраните объект Seura для дальнейшего изучения данных.
ПРИМЕЧАНИЕ: Код для контроля качества и кластерного анализа доступен в дополнительном файле 1.
| Пакеты программного обеспечения/R, используемые в рабочих процессах обработки данных | Альтернативное программное обеспечение/пакеты | Этап обработки |
| Сельский рейнджер | STARsolo, kallisto | Обрезка, выравнивание, отображение |
| Сёра | SingleCellExperiment, Cellranger | Контроль качества, анализ и изучение данных |
| DoubletFinder | scds, scdblFinder, Скрублет | Обнаружение дуплетов |
| ДеконтХ | SoupX, CellBender | Настройка РНК окружающей среды |
Таблица 3: Программное обеспечение/инструменты для работы с данными.
Access restricted. Please log in or start a trial to view this content.
Результаты
Этот рабочий процесс был разработан для управления обработкой замороженных образцов IMAT человека для получения профилей экспрессии генов с разрешением одного ядра, что позволяет идентифицировать тип клеток. Здесь представлена одна репрезентативная выборка IMAT от участника исследован...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Работа с IMAT сопряжена с несколькими проблемами. Помимо ограниченной доступности, выход материала образца часто бывает очень скудным, а «загрязнения» скелетных мышц практически невозможно избежать. Для получения образца наилучшего качества следует проникнуть в мышечную фасцию при вв?...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Авторы выражают признательность Брайану Бергману, доктору философии из Университета Колорадо, за предоставление изображения биопсии IMAT на рисунке 1C из исследования MoTrIMAT (R01AG077956). Мы благодарны Study of Muscle, Mobility and Aging (Study of Muscle, Mobility and Aging) предоставила выборку IMAT, данные которой представлены в разделе репрезентативных результатов. Национальный институт по проблемам старения (NIA) финансировал исследование мышц, подвижности и старения (SOMMA; R01AG059416) и его вспомогательные исследования SOMMA AT (R01AG066474) и SOMMA Knee OA (R01AG070647). Поддержка исследовательской инфраструктуры частично финансировалась Центрами независимости пожилых американцев им. Клода Д. Пеппера в Университете Питтсбурга (P30AG024827) и Университете Уэйк Форест (P30AG021332) и Институтами клинических и трансляционных наук, финансируемыми Национальным центром развития трансляционной науки в Университете Уэйк Форест (UL1 0TR001420).
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| 0.2 µm corning syringe filters | Millipore Sigma | CLS431229 | |
| 1.7 mL DNA LoBind tubes | Eppendorf | 22431021 | low-bind tubes |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 100x protease inhibitor | Thermo Fisher Scientific | 78437 | |
| 10X Magnetic Separator | 10X Genomics | 230003 | |
| 10X Vortex Adapter | 10X Genomics | 330002 | |
| 15 mL canonical tubes | Sarstedt | 6,25,54,502 | |
| 2100 Bioanalyzer | Agilent | G2939BA | |
| 50 mL conical tubes | Sarstedt | 6,25,47,254 | |
| CellRanger | Genomics | N/A | |
| Chromium iX accesory kit | 10X Genomics | PN1000323 | |
| Chromium iX Controller | 10X Genomics | PN1000326 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | PN1000127 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1 | 10X Genomics | PN1000129 | |
| Chromium Next GEM Single Cell GEM Kit v3.1 | 10X Genomics | PN1000130 | |
| Countess 3 Automated Cell Counter | Thermo Fisher Scientific | AMQAX2000 | Automated cell counter |
| Countess cell counting chamber slides | Thermo Fisher Scientific | C10228 | |
| DoubletFinder | R | N/A | |
| DPBS (no calcium, no magnesium) | Thermo Fisher Scientific | 14190144 | |
| DTT | Thermo Fisher Scientific | R0861 | |
| Dual Index Kit TT Set A, 96 rxns | 10X Genomics | PN1000215 | |
| Dynabeads MyOne SILANE | 10X Genomics | PN2000048 | |
| Falcon 100 µm Cell strainer | Corning Life Science | 352360 | |
| Falcon 40 µm Cell strainer | Corning Life Science | 352340 | |
| Glycerin (glycerol), 50% (v/v) Aqueous Solution | Ricca Chemical Company | 3290-32 | |
| KCL | Thermo Fisher Scientific | AM9640G | |
| Library Construction Kit v3.1 | 10X Genomics | PN1000196 | |
| MACS SmartStrainers (30µm) | Miltenyi Biotec | 130-098-458 | |
| Mastercycler Nexus Gradient Thermal cycler | Eppendorf | 6331000017 | |
| MgCl2 | Ambion | AM9530G | |
| Mortar and pestel | Health care logistics | 14075 | |
| NucBlue Live Ready Probes Reagent | Thermo Fisher Scientific | R37605 | |
| Nuclease Free Water (not DEPC treated) | Thermo Fisher Scientific | AM9930 | |
| Probumin Bovine Serum Albumin Fatty Acid Free, Powder | Sigma-Aldrich | 820024 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Ribolock RNAse inhibitor | Thermo Fisher Scientific | EO0382 | |
| Seurat | R | N/A | |
| Sucrose | Sigma-Aldrich | S0389 | |
| SUPERasin 20 U/µL | Thermo Fisher Scientific | AM2695 | |
| ThermoMixer C | Eppendorf | 5382000015 | |
| Tissue homogenizer | Glass-Col | 099C K54 | |
| Tris buffer pH 8.0 | Thermo Fisher Scientific | AM9855G | |
| Triton X-100 | Thermo Fisher Scientific | AC327372500 | |
| UltraPure 0.5M EDTA pH 8.0 | Gibco | 15575020 |
Ссылки
- Goodpaster, B. H., Bergman, B. C., Brennan, A. M., Sparks, L. M. Intermuscular adipose tissue in metabolic disease. Nat Rev Endocrinol. 19 (5), 285-298 (2023).
- Sparks, L. M., Goodpaster, B. H., Bergman, B. C. The metabolic significance of intermuscular adipose tissue: Is IMAT a friend or a foe to metabolic health. Diabetes. 70 (11), 2457-2467 (2021).
- Gallagher, D., et al. Adipose tissue in muscle: A novel depot similar in size to visceral adipose tissue. Am J Clin Nutr. 81 (4), 903-910 (2005).
- Manini, T. M., et al. Reduced physical activity increases intermuscular adipose tissue in healthy young adults. Am J Clin Nutr. 85 (2), 377-384 (2007).
- Addison, O., Marcus, R. L., LaStayo, P. C., Ryan, A. S. Intermuscular fat: A review of the consequences and causes. Int J Endocrinol. 2014, 309570(2014).
- Goodpaster, B. H., et al. Obesity, regional body fat distribution, and the metabolic syndrome in older men and women. Arch Intern Med. 165 (7), 777-783 (2005).
- Goodpaster, B. H., Thaete, F. L., Kelley, D. E. Thigh adipose tissue distribution is associated with insulin resistance in obesity and in type 2 diabetes mellitus. Am J Clin Nutr. 71 (4), 885-892 (2000).
- Goodpaster, B. H., et al. Association between regional adipose tissue distribution and both type 2 diabetes and impaired glucose tolerance in elderly men. Diabetes Care. 26 (2), 372-379 (2003).
- Sachs, S., et al. Intermuscular adipose tissue directly modulates skeletal muscle insulin sensitivity in humans. Am J Physiol Endocrinol Metab. 316 (5), E866-E879 (2019).
- Ford, H., Liu, Q., Fu, X., Strieder-Barboza, C. White adipose tissue heterogeneity in the single-cell era: From mice and humans to cattle. Biology (Basel). 12 (10), 1289(2023).
- Wang, L., et al. Single-nucleus and bulk RNA sequencing reveal cellular and transcriptional mechanisms underlying lipid dynamics in high marbled pork. NPJ Sci Food. 7 (1), 23(2023).
- Li, J., et al. Identification of diverse cell populations in skeletal muscles and biomarkers for intramuscular fat of chicken by single-cell RNA sequencing. BMC Genomics. 21 (1), 752(2020).
- Lyu, P., Qi, Y., Tu, Z. J., Jiang, H. Single-cell RNA sequencing reveals heterogeneity of cultured bovine satellite cells. Front Genet. 12, 742077(2021).
- Fitzgerald, G., et al. MME+ fibro-adipogenic progenitors are the dominant adipogenic population during fatty infiltration in human skeletal muscle. Commun Biol. 6 (1), 111(2023).
- Cummings, S. R., et al. The study of muscle, mobility and aging (SOMMA): A unique cohort study about the cellular biology of aging and age-related loss of mobility. J Gerontol A Biol Sci Med Sci. 78 (11), 2083-2093 (2023).
- Whytock, K. L., et al. Isolation of nuclei from frozen human subcutaneous adipose tissue for full-length single-nuclei transcriptional profiling. STAR Protoc. 4 (1), 102054(2023).
- 10x Genomics. Chromium Single Cell 3' Reagent Kits User Guide (v3.1 Chemistry Dual Index), Document Number CG000315 RevE. , Available from: https://cdn.10xgenomics.com/image/upload/v1668017706/support-documents/CG000315_ChromiumNextGEMSingleCell3-_GeneExpression_v3.1_DualIndex__RevE.pdf (2022).
- Heumos, L., et al. Best practices for single-cell analysis across modalities. Nat Rev Genet. 24 (1), 550-572 (2023).
- Hao, Y., et al. Dictionary learning for integrative, multimodal and scalable single-cell analysis. Nat Biotechnol. 42 (2), 293-304 (2023).
- McGinnis, C. S., Murrow, L. M., Gartner, Z. J. DoubletFinder: Doublet detection in single-cell RNA sequencing data using artificial nearest neighbors. Cell Syst. 8 (4), 329-337 (2019).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (2), 57(2020).
- Common considerations for quality control filters for single cell RNA-seq data. 10X Genomics. , Available from: https://www.10xgenomics.com/analysis-guides/common-considerations-for-quality-control-filters-for-single-cell-rna-seq-data (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Mol Syst Biol. 15 (6), e8746(2019).
- Emont, M. P., et al. A single-cell atlas of human and mouse white adipose tissue. Nature. 603 (7903), 926-933 (2022).
- Hildreth, A. D., et al. Single-cell sequencing of human white adipose tissue identifies new cell states in health and obesity. Nat Immunol. 22 (5), 639-653 (2021).
- Whytock, K. L., et al. Single cell full-length transcriptome of human subcutaneous adipose tissue reveals unique and heterogeneous cell populations. iScience. 25 (8), 104772(2022).
- Probst, V., et al. Benchmarking full-length transcript single cell mRNA sequencing protocols. BMC Genomics. 23 (1), 860(2022).
- CG000148 Rev A Technical Note - Resolving cell types as a function of read depth and cell number. Technical note. 10X Genomics. , Available from: https://assets.ctfassets.net/an68im79xiti/6gDArDPBTOg4IIkYEO2Sis/803be2286bb a5ca67f353e6baf68d276/CG000148_10x_Technical _Note_Resolving_Cell_Types_as_Function_of_ Read_Depth_Cell_Number_RevA.pdf (2018).
- Gupta, A., et al. Characterization of transcript enrichment and detection bias in single-nucleus RNA-seq for mapping of distinct human adipocyte lineages. Genome Res. 32 (2), 242-257 (2022).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648(2018).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. J Am Soc Nephrol. 30 (1), 23-32 (2019).
- Kim, N., Kang, H., Jo, A., Yoo, S. -A., Lee, H. -O. Perspectives on single-nucleus RNA sequencing in different cell types and tissues. J Pathol Transl Med. 57 (1), 52-59 (2023).
- Avila Cobos, F., Alquicira-Hernandez, J., Powell, J. E., Mestdagh, P., De Preter, K. Benchmarking of cell type deconvolution pipelines for transcriptomics data. Nat Commun. 11 (1), 5650(2020).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены