Method Article
CIRCLE-seq للاستجواب في تحرير الجينات غير المستهدف
In This Article
Summary
أحد العوائق الكبيرة أمام تقنيات مثل كريسبر هو الأحداث غير المستهدفة التي يمكن أن تعطل الجينات الحيوية. "التعميم للإبلاغ في المختبر عن آثار الانقسام عن طريق التسلسل" (CIRCLE-seq) هي تقنية مصممة لتحديد مواقع الانقسام غير المقصودة. تحدد هذه الطريقة نشاط CRISPR-Cas9 على مستوى الجينوم بحساسية عالية وبدون تحيز.
Abstract
التعميم للإبلاغ في المختبر عن تأثيرات الانقسام عن طريق التسلسل (CIRCLE-seq) هو تقنية جديدة تم تطويرها للتحديد المحايد لمواقع الانقسام غير المقصودة ل CRISPR-Cas9 من خلال التسلسل المستهدف للحمض النووي المشقوق CRISPR-Cas9. يتضمن البروتوكول دائرية للحمض النووي الجيني (gDNA) ، والذي يتم معالجته لاحقا ببروتين Cas9 ودليل الحمض النووي الريبي (gRNA) ذي الأهمية. بعد العلاج ، يتم تنقية الحمض النووي المشقوق وإعداده كمكتبة لتسلسل Illumina. تنشئ عملية التسلسل قراءات مزدوجة ، مما يوفر بيانات شاملة عن كل موقع انقسام. يوفر CIRCLE-seq العديد من المزايا مقارنة بالطرق الأخرى في المختبر ، بما في ذلك الحد الأدنى من متطلبات عمق التسلسل ، والخلفية المنخفضة ، والتخصيب العالي ل gDNA المشقوق Cas9. تعزز هذه المزايا الحساسية في تحديد أحداث الانقسام المقصودة وغير المقصودة. توفر هذه الدراسة إجراء شاملا خطوة بخطوة لفحص النشاط خارج الهدف ل CRISPR-Cas9 باستخدام CIRCLE-seq. على سبيل المثال ، يتم التحقق من صحة هذا البروتوكول عن طريق رسم خرائط لمواقع الانقسام غير المقصودة على مستوى الجينوم ل CRISPR-Cas9 أثناء تعديل موضع AAVS1 . يمكن إكمال عملية CIRCLE-seq بأكملها في غضون أسبوعين ، مما يتيح وقتا كافيا لنمو الخلايا ، وتنقية الحمض النووي ، وإعداد المكتبة ، وتسلسل Illumina. يسهل إدخال بيانات التسلسل في خط أنابيب CIRCLE-seq التفسير والتحليل المبسط لمواقع الانقسام.
Introduction
شهدت هندسة الجينوم تطورات كبيرة على مدار العشرين عاما الماضية ، وكان من المعالم الرئيسية اكتشاف التكرارات المتجانسة القصيرة العنقودية المتباعدة بانتظام (CRISPR) -Cas9 في عام 20121. من خلال الاستفادة من الطبيعة القابلة للبرمجة لنوكليازات الحمض النووي البكتيرية ، تتيح تقنية CRISPR-Cas9 الاستهداف الدقيق وتعديل أي تسلسل للحمض النووي تقريبا. منذ إنشائه ، تم تحسين النظام للاعتماد فقط على نوكلياز Cas9 ودليل الحمض النووي الريبي (gRNA) لتحرير مناطق جينومية معينة. تم إثبات إمكانات CRISPR-Cas9 كعلاج علاجي في التجارب السريرية لحالات مختلفة مثل داء ليبر الخلقي ، والداء النشواني عبر الثيريتين ، وفقر الدم المنجلي ، من بينأمور أخرى 2،3،4.
يحفز CRISPR-Cas9 فواصل مزدوجة الشريطة (DSBs) ، والتي يتم حلها عادة بواسطة إحدى آليتين: الانضمام النهائي غير المتماثل المعرض للخطأ (NHEJ) أو الإصلاح الموجه بالتماثل الأكثر دقة (HDR) ، بشرط توفر قالب الحمض النووي. إن ميل CRISPR-Cas9 إلى التسبب في عمليات إدخال وحذف مرتبطة ب NHEJ (indels) ، جنبا إلى جنب مع الانقسام في المواقع الجينومية غير المقصودة ، يحد من تطبيقه في الإعدادات السريرية5،6،7،8،9،10. بالإضافة إلى ذلك ، يمكن أن تؤدي التعديلات الجينومية غير المقصودة إلى إنشاء مواقع لصق مشفرة ، أو طفرات هراء أو خاطئة ، أو تحفز داء اللونيات ، أو تمنح إمكانات الأورام للخلايا - النتائج التي لوحظت في العديد من تجارب تحرير الجينوم11،12،13،14،15. في الختام ، يعد التحديد الدقيق للنشاط غير المستهدف ل CRISPR-Cas9 أمرا بالغ الأهمية لتطبيقاته السريرية ، لا سيما في العلاجات الجينية الجهازية التي قد تغير مليارات الخلايا.
يمكن استخدام طرق مختلفة لتحديد مواقع الانقسام خارج الهدف CRISPR-Cas9 ، بما في ذلك التحديد غير المتحيز للفواصل المزدوجة الشريطة على مستوى الجينوم (GUIDE) - seq16 ، والذي يستخدم قليل قليل الأكسجين نوكليوتيدات مزدوجة الشريطة لتمييز DSBs في الخلايا الحية. ومع ذلك ، فإن انتقاد هذه الطريقة هو أن الإيجابيات الخاطئة يمكن أن تنشأ من DSBs العشوائية أو من القطع الأثرية PCR ، والتي يجب تجاهلها عن طريق استبعاد المواقع التي تم التقاطها والتي تظهر تشابها ضعيفا مع المواقع المستهدفة. الطريقة القائمة على استخدام ناقل الفيروسات العسسي المعيب (IDLV) أقل حساسية ومن المرجح أن تفوت العديد من المواقع غيرالمستهدفة 17. تتضمن الطرق الأخرى في الموقع مثل DSBCapture و BLESS و BLISS18،19،20 خلايا ثابتة وتسمية DSBs مباشرة ، ومع ذلك فهي مقيدة باعتمادها على التقاط DSB الفوري وغياب الحمض النووي الخارجي. يوفر كل من Digenome-seq21 ، وهي طريقة في المختبر ، والإثراء الانتقائي وتحديد النهايات الجينومية الموسومة عن طريق التسلسل (SITE-seq) 22 حلول التسلسل ولكن لهما قيودهما في ضوضاء الخلفية والتحليل أحادي الطرف ، على التوالي. يوفر اكتشاف Cas في الموقع خارج الأهداف والتحقق عن طريق التسلسل (Discover-Seq)23 تحديدا في الجسم الحي وفي الموقع لنشاط Cas9 عبر ربط MRE11 ، ولكنه يكتشف فقط DSBs الموجودة في وقت تحضيرالعينة 24. أخيرا ، يستخدم الاستدلال على تعديلات كريسبر (ICE) نهج المعلوماتية الحيوية لتحليل تعديلات كريسبر بقوة باستخدام بيانات سانجر25.
تصف هذه المقالة إجراء مفصلا للتعميم للإبلاغ في المختبر عن تأثيرات الانقسام عن طريق التسلسل (CIRCLE-seq): تقنية في المختبر تحدد بحساسية ونزاهة للنشاط خارج الهدف على مستوى الجينوم لنوكلياز Cas9 في مجمع مع gRNA ذي الأهمية26. يبدأ هذا النهج بزراعة الخلايا ذات الأهمية وعزل الحمض النووي ، يليه القص العشوائي من خلال الموجات فوق الصوتية المركزة ، ثم العلاج بالنوكلياز الخارجي والليغازي. تنتج هذه العملية في النهاية جزيئات الحمض النووي الدائرية مزدوجة الشريطة ، والتي يتم تنقيتها بعد ذلك من خلال معالجة DNase الآمنة للبلازميد. ثم يتعرض هذا الحمض النووي الدائري لمركب Cas9-gRNA ، الذي ينشق في كل من مواقع الانقسام المقصودة وغير المقصودة ، تاركا وراءه نهايات الحمض النووي المكشوفة التي تعمل كركائز لربط محول Illumina. تنتج هذه العملية مكتبة متنوعة من الحمض النووي الجيني (gDNA) تحتوي على طرفي كل DSB ناتجه النوكلياز ، مما يضمن أن كل قراءة تحتوي على جميع المعلومات اللازمة لكل موقع انقسام. يسمح ذلك باستخدام تسلسل Illumina مع متطلبات تغطية تسلسل أقل ، مما يميز CIRCLE-seq عن الطرق المماثلة الأخرى المذكورة أعلاه. من المهم ملاحظة أنه في حين أن CIRCLE-seq لديها حساسية أعلى خارج الهدف من البروتوكولات الأخرى كطريقة في المختبر ، فإن هذا يأتي على حساب إيجابيات كاذبة أعلى بسبب عدم وجود مشهد لاجيني موجود في طرق أخرى مثل GUIDE-seq16. بالإضافة إلى ذلك ، فإن إصلاح الحمض النووي DSB والآلات المرتبطة به غير موجودة في CIRCLE-seq ، أو إلغاء indels أو الإصلاح المناسب الذي يمكن ملاحظته لولا ذلك.
بالإضافة إلى وصف البروتوكول خطوة بخطوة لأداء CIRCLE-seq ، يتم التحقق من صحة البروتوكول من خلال تحديد مواقع الانقسام غير المقصودة على مستوى الجينوم ل CRISPR-Cas9 التي تحدث أثناء تعديل موضع AAVS1 ، على سبيل المثال. يوفر هذا البروتوكول سهل المتابعة تعليمات مفصلة ، من ثقافة الخلايا الجذعية المستحثة متعددة القدرات (iPSCs) وعزل gDNA إلى تدوير gDNA ، وانقسام Cas9-gRNA ، وإعداد المكتبة ، والتسلسل ، وتحليل خطوط الأنابيب. نظرا لمتطلبات تغطية التسلسل المنخفضة ، فإن CIRCLE-seq متاح لأي مختبر لديه إمكانية الوصول إلى تسلسل الجيل التالي.
Protocol
يتم سرد تفاصيل الكواشف والمواد الاستهلاكية والمعدات المستخدمة في هذه الدراسة في جدول المواد.
1. زراعة الخلايا (5 أيام)
- قم بتضمين عنصر تحكم سلبي في جميع أنحاء هذا البروتوكول. قم بإعداد خلايا كافية ل 25 ميكروغرام إضافية من gDNA (~ 2.0e7 خلايا لكل عينة).
- استزراع iPSCs وفقا للبروتوكول المعمولبه 27. اجمع الخلايا وأعد تعليقها في 10 مل من PBS. ماصة عينة 6 ميكرولتر من الخلايا وإعادة تعليقها بنسبة 1: 1 مع تريبان بلو. استخدم عداد الخلايا الآلي لحساب العينة.
- Aliquot 2 × 107 خلايا لكل أنبوب ، ثم تدور عند 300 × جم لمدة 3 دقائق عند 25 درجة مئوية (درجة حرارة الغرفة (RT)). هذا كاف لتكرارات متعددة. ماصة وتخلص من المادة الطافية.
2. عزل الحمض النووي الجيني (1 يوم)
- استخدم مجموعة تنقية الحمض النووي المتوفرة تجاريا لعزل الحمض النووي الجديد، باتباع تعليمات الشركة المصنعة:
- أضف 200 ميكرولتر من PBS إلى أنبوب مخروطي 15 مل يحتوي على حبيبات الخلية وإعادة التعليق. بعد ذلك، قم بإدخال 3 مل من المخزن المؤقت لتحلل الخلايا و15 ميكرولتر من البروتيناز K في الأنبوب. اقلب الأنبوب 25 مرة لخلطه جيدا. ضع الأنبوب في شاكر حمام مائي مضبوطا على 55 درجة مئوية و 150 دورة في الدقيقة لمدة 3 ساعات ، أو طوال الليل للحصول على إنتاجية مثالية للحمض النووي.
- الماصة في 15 ميكرولتر من RNase A. اقلبها 25 مرة. ضعها في حمام مائي عند 37 درجة مئوية لمدة 1 ساعة.
- برد العينة على الثلج لمدة 5 دقائق. ثم أضف 1 مل من محلول ترسيب البروتين ، والدوامة بسرعة عالية لمدة 20 ثانية ، وجهاز الطرد المركزي عند 2000 × جم لمدة 10 دقائق في RT. يجب أن تشكل البروتينات حبيبات مرئية ومضغوطة في قاع الأنبوب. إذا كانت الحبيبات غير مرئية ، فقم باحتضان العينة على الجليد لمدة 5 دقائق إضافية وجهاز الطرد المركزي مرة أخرى.
- أضف 3 مل من الأيزوبروبانول بنسبة 100٪ إلى أنبوب مخروطي جديد سعة 15 مل. قم بعمل الماصة الطافية بعناية من الخطوة 2.1.3 إلى الأنبوب. اقلب الأنبوب 50 مرة للخلط ، ثم جهاز الطرد المركزي عند 2000 × جم ودرجة حرارة الغرفة لمدة 3 دقائق. دون إزعاج حبيبات الحمض النووي ، قم بشفط المادة الطافية بعناية باستخدام ماصة باستور متصلة بمصيدة تفريغ واقلب الأنبوب على منديل نظيف وخالي من النسالة.
- ماصة 3 مل من 70٪ (حجم / حجم) من الإيثانول على حبيبات الحمض النووي واقلبها 10 مرات لغسلها. جهاز طرد مركزي عند 2,000 × جم لمدة 3 دقائق في RT. ثم اسكب بعناية المادة المطافية.
- حافظ على الأنبوب مفتوحا واترك حبيبات الحمض النووي الناتجة تجف لمدة 30 دقيقة ، مما يضمن تبخر كل الإيثانول بالكامل. ماصة في 50 ميكرولتر من محلول ترطيب الحمض النووي واخلطها جيدا عن طريق سحب العينة اللطيف.
- قم بإذابة الحمض النووي عن طريق وضع العينة في شاكر حمام مائي عند 65 درجة مئوية لمدة ساعة واحدة ، ثم اترك العينة في RT طوال الليل. قم بالطرد المركزي للعينة عند RT و 2,000 × جم لمدة دقيقة واحدة واستخدم مجموعة فحص dsDNA BR مع الأنابيب المرتبطة لتحديد كمية الحمض النووي المعزول باستخدام مقياس الفلور.
3. تحضير الحمض النووي الريبي (7 أيام)
- اطلب الحمض النووي الريبي الاصطناعي ذي الأهمية من مصدر تجاري (انظر جدول المواد). هذا البروتوكول متوافق أيضا مع crRNA / tracrRNA.
4. gRNA في اختبار الانقسام في المختبر
ملاحظة: هنا ، يتم استخدام هدف في جين AAVS1 . لاستهداف الجينات الأخرى ذات الأهمية ، صمم بادئات (الجدول 1) لتضخيم المنطقة المستهدفة واستبدال البادئات في الخطوات التالية ببادئات مخصصة.
- تحضير تفاعل تفاعل البوليميراز المتسلسل (100 ميكرومتر) ، امزج 25 ميكرولتر من Phusion Hot Start Flex 2x Master Mix (التركيز النهائي 1x) ، 0.5 ميكرولتر من AAVS1 F Primer (التركيز النهائي 0.1 ميكرومتر) ، 0.5 ميكرولتر من AAVS1 R Primer (التركيز النهائي 0.1 ميكرومتر) ، 5 ميكرولتر من gDNA (100 نانوغرام ، 20 نانوغرام / ميكرولتر ، من الخطوة 2.1.6) ، و 19 ميكرولتر من H2O الخالي من النوكلياز (الحجم الإجمالي: 50 ميكرولتر).
- استخدم معلمات الدورة الحرارية التالية: التمسخ: 98 درجة مئوية لمدة دقيقتين (دورة واحدة) ، التمسخ: 98 درجة مئوية لمدة 10 ثوان (10 دورات) ، الصلب: 72-62 درجة مئوية (-1 درجة مئوية / دورة) لمدة 15 ثانية (10 دورات) ، تمديد: 72 درجة مئوية لمدة 30 ثانية (10 دورات) ، تمسخ ، 98 درجة مئوية لمدة 10 ثوان (30 دورة). الصلب: 65 درجة مئوية لمدة 15 ثانية (30 دورة) ، التمديد: 72 درجة مئوية لمدة 30 ثانية (30 دورة) ، التمديد النهائي: 72 درجة مئوية لمدة 5 دقائق (دورة واحدة) ، الانتظار: 4 درجة مئوية إلى أجل غير مسمى.
- استخدم حبات AMPure XP لتنقية منتج تفاعل تفاعل البوليميراز المتسلسل (PCR). أولا، قم بتعبئة الماصة بأحجام 1.8x أو 90 ميكرولتر من حبات XP لمنتج PCR. الماصة عشر مرات لخلطها جيدا. اترك الخليط في RT لمدة 5 دقائق حتى يحتضن.
- باستخدام رف مغناطيسي ، افصل الخرزات عن المحلول عن طريق وضع لوحة تفاعل تفاعل تفاعل البوليميراز المتسلسل على المغناطيس لمدة 3 دقائق. ماصة من المحلول الذي تم تطهيره وتخلص منه. أضف 200 ميكرولتر من 80٪ من الإيثانول (V / v) إلى الخرزات ، واحتضن لمدة 30 ثانية ، ثم قم بإزالة الإيثانول. كرر خطوة الغسيل هذه مرتين لضمان الإزالة الكاملة للإيثانول.
- اترك العينات تجف بشكل طبيعي لمدة 3 دقائق عن طريق وضع اللوحة على المغناطيس. قم بإزالة اللوحة من المغناطيس وأضف 40 ميكرولتر من المخزن المؤقت TE ، الرقم الهيدروجيني 8.0. تخلط عن طريق سحب العينات لأعلى ولأسفل عشر مرات. اسمح للعينة بالجلوس في RT لمدة 2 دقيقة.
- ضع لوحة تفاعل تفاعل البوليميراز المتسلسل على المغناطيس لمدة دقيقة إضافية. بعد 1 دقيقة ، انقل المادة الطافية إلى طبق جديد. قم بقياس عائد تفاعل البوليميراز المتسلسل المنقى باستخدام مقياس الطيف الضوئي ، وقم بتحليله على TapeStation باستخدام شريط أنبوب بصري مع غطاء شريط أنبوب بصري ، جنبا إلى جنب مع شريط الشاشة D1000 عالي الحساسية والكواشف D1000 عالية الحساسية (السلم والمخزن المؤقت ) ، باتباع تعليمات الشركة المصنعة. قم بتخزين العينة المحضرة عند -20 درجة مئوية لمدة تصل إلى عدة أشهر.
- تمييع بروتين نوكلياز Cas9 إلى 1 ميكرومتر على النحو التالي: امزج 2 ميكرولتر من 10x Cas9 عازلة (التركيز النهائي 1x) ، 1 ميكرولتر من Cas9 Nuclease ، S. pyogenes (التركيز النهائي 1 ميكرومتر) ، و 17 ميكرولتر من H2O الخالي من النكلياز بحجم إجمالي قدره 20 ميكرولتر.
- قم بإجراء خال من RNase لمنع تدهور الحمض النووي الريبي. تمييع gRNA (من الخطوة 3.1) إلى 3 ميكرومتر في H2O إلى حجم إجمالي قدره 10 ميكرولتر.
ملاحظة: استخدم الصيغة التالية لتقدير الوزن الجزيئي ل gRNA: الوزن الجزيئي ل ssRNA (جم / مول) = (طول ssRNA (nt) × 321.47 جم / مول) + 18.02 جم / مول. كمرجع ، فإن gRNA بطول 104 nt عند 3 ميكرومتر هو حوالي 100 نانوغرام / ميكرولتر.
5. قص الحمض النووي (3 ساعات)
- قم بإعداد ME220 عن طريق توجيه ذراع التحكم أولا. ثم املأ الخزان ب H2O المنقى منقي. على الكمبيوتر المحمول لمحطة التحكم ، قم بالوصول إلى Water Works وانقر فوق تعبئة. اضبط درجة الحرارة على 4.5 درجة مئوية.
- نقل 25 ميكروغرام من gDNA إلى أنبوب دقيق (microtube-130 AFA Fiber Screw-Cap). ثم املأ الأنبوب بحجم إجمالي قدره 130 ميكرولتر ب 1x TE. استخدم الشروط التالية لقص الحمض النووي بمتوسط طول 300 نقطة أساس تقريبا: اضبط المدة على 10 ثوان ؛ ذروة الطاقة إلى 70 ؛ عامل الواجب ٪ إلى 20 ؛ دورات / انفجار إلى 50 ؛ سيؤدي كل ذلك تلقائيا إلى ضبط متوسط الطاقة على 14.0.
6. تنقية الحمض النووي الجيني المنفصم (1 ساعة)
- قسم الحمض النووي الجيني المنفصم إلى جزأين سعة كل منهما 65 ميكرولتر. قم بالتنقية باستخدام 1.8 ضعف حجم حبات XP (117 ميكرولتر) ، باتباع الإجراء الموضح في الخطوات 4.3-4.6. انقل المادة الطافية إلى لوحة PCR جديدة وقم بقياس الكمية باستخدام مقياس الطيف الضوئي.
- قم بتشغيل 1 ميكرولتر من gDNA المنفصم المبذول على TapeStation ، وفقا لتعليمات الشركة المصنعة ، لضمان قص gDNA إلى توزيع واسع يبلغ حوالي 300 نقطة أساس. إذا لزم الأمر ، قم بتخزين gDNA المنفصم عند -20 درجة مئوية لمدة تصل إلى عدة أشهر.
7. إعداد مكتبة CIRCLE-seq (3 أيام)
- محول دبوس الشعر الصلب
- قم بإعادة تعليق oSQT1288 (الجدول 1) ، محول دبوس الشعر ، إلى تركيز نهائي قدره 100 ميكرومتر في 1x TE.
- قم بإجراء تلدين المحول على النحو التالي: امزج 40 ميكرولتر من oSQT1288 (التركيز النهائي 40 ميكرومتر) ، و 10 ميكرولتر من 10x STE (التركيز النهائي 1x) ، و 50 ميكرولتر من H2O الخالي من النكلياز لحجم إجمالي يبلغ 100 ميكرولتر.
- استخدم معلمات التلدين التالية: 95 درجة مئوية لمدة 5 دقائق ، -1 درجة مئوية في الدقيقة لمدة 70 دورة ، استمر في 4 درجات مئوية إلى أجل غير مسمى.
- إجراء الإصلاح النهائي. استخدم مجموعة إعداد مكتبة HTP الخالية من تفاعل البوليميراز المتسلسل وقم بإعداد مزيج الإصلاح النهائي.
- امزج 8 ميكرولتر من H2O الخالي من Nuclease ، و 7 ميكرولتر من 10x مخزن مؤقت للإصلاح النهائي (التركيز النهائي 1x) ، و 5 ميكرولتر من مزيج إنزيم الإصلاح النهائي (الحجم النهائي 20 ميكرولتر من Total End-Repair Master Mix).
- الماصة 20 ميكرولتر من المزيج الرئيسي للإصلاح النهائي في عينة gDNA المنفصمة من الخطوات 4.3-4.6. امزج 20 ميكرولتر من End-Repair Master Mix مع 50 ميكرولتر من gDNA المنفصم للحصول على حجم نهائي يبلغ 70 ميكرولتر.
- ضع المزيج في جهاز تدوير حراري على حرارة 20 درجة مئوية لمدة 30 دقيقة ، ثم احتفظ به عند 4 درجات مئوية إلى أجل غير مسمى.
- أضف 1.7x من وحدات التخزين ، أو 120 ميكرولتر ، من حبات XP واتبع خطوات التنقية من الخطوات 4.3-4.6. Elute مع 42 ميكرولتر من TE ، درجة الحموضة 8.0. تأكد من بقاء الخرزات في الحل للخطوة التالية.
- أداء A-tailing. باستخدام مجموعة إعداد مكتبة HTP (خالية من تفاعل البوليميراز المتسلسل (PCR)، قم بإعداد A-tailing Master Mix.
- امزج 5 ميكرولتر من 10x عازلة مخلفات A (التركيز النهائي 1x) و 3 ميكرولتر من إنزيم المخلفات A (الحجم النهائي الإجمالي 8 ميكرولتر من A-tailing Master Mix).
- ماصة 8 ميكرولتر من المزيج الرئيسي A-tailing في كل عينة من الحمض النووي تحتوي على خرز من الخطوة 7.2.4 ، مع خلط 8 ميكرولتر من A-tailing Master Mix إلى 42 ميكرولتر من الحمض النووي الذي تم إصلاحه نهائيا يحتوي على حبات (الحجم النهائي الإجمالي 50 ميكرولتر). ضعها في جهاز تدوير حراري على حرارة 30 درجة مئوية لمدة 30 دقيقة. امسك عند 4 درجات مئوية إلى أجل غير مسمى.
- الماصة 1.8 مجلدات، أو 90 ميكرولتر، من محلول PEG / كلوريد الصوديوم SPRI (أحد مكونات مجموعة تحضير مكتبة HTP (خالية من تفاعل البوليميراز المتسلسل (96 تفاعلا)) إلى الحمض النووي A-tailed. تنقية الحمض النووي ذو الذيل A وفقا للخطوات 4.3-4.6. قم بتخطيب الحمض النووي ذو الذيل A في 30 ميكرولتر من TE ، درجة الحموضة 8.0. احتفظ بالخرزات في الحل للخطوة التالية.
- قم بإجراء ربط المحول. باستخدام مجموعة إعداد مكتبة HTP (خالية من PCR)، قم بإعداد Adapter Ligation Master Mix.
- امزج 10 ميكرولتر من 5x المخزن المؤقت للربط (التركيز النهائي 1x) ، و 5 ميكرولتر من DNA Ligase ، و 5 ميكرولتر من محول دبوس الشعر الملدن (40 ميكرومتر) من الخطوة 7.1.3. تأكد من التركيز النهائي 4 ميكرومتر لمزيج ربط المحول الرئيسي الكلي البالغ 20 ميكرولتر.
- ماصة 20 ميكرولتر من مزيج ربط المحول الرئيسي في كل عينة من الحمض النووي المملوثة تحتوي على حبات من الخطوة 7.3.3 (الحجم النهائي الإجمالي 50 ميكرولتر لكل عينة).
- ضعها في جهاز تدوير حراري عند 20 درجة مئوية لمدة 1 ساعة. امسك عند 4 درجات مئوية إلى أجل غير مسمى.
- انقل 1x وحدات تخزين ، أو 50 ميكرولتر ، من محلول PEG / NaCl SPRI إلى الحمض النووي المرتبط بالمحول وتنقية وفقا للخطوات 4.3-4.6. قم بصب 30 ميكرولتر من TE ، ودرجة الحموضة 8.0 ، وصب المواد الطافية في لوحة PCR جديدة شبه حواف. اجمع الحمض النووي وحدده باستخدام مقايسة dsDNA BR. إذا لزم الأمر ، قم بتخزين الحمض النووي المنقى المرتبط بالمحول لمدة تصل إلى شهر واحد عند -20 درجة مئوية.
- قم بإعداد Lambda Exonuclease / Exonuclease I (E. coli) Master Mix (يعمل في التخلص من الحمض النووي أحادي أو مزدوج الشريطة بدون محولات مرتبطة بكلا الطرفين).
- خذ 1 ميكروغرام من الحمض النووي المرتبط بالمحول من الخطوة 7.4.4 ، وقم بتخفيفه إلى 40 ميكرولتر. امزج 5 ميكرولتر من 10x exonuclease I react-buffer (التركيز النهائي 1x) ، و 4 ميكرولتر من Lambda Exonuclease (التركيز النهائي 0.4 U / μL) ، و 1 ميكرولتر من نوكلياز Exonuclease I (بكتريا قولونية) (التركيز النهائي 0.4 U / μL) للحجم الإجمالي لمزيج Lambda Exonuclease / Exonuclease I Master Mix 10 μL.
- الماصة 10 ميكرولتر من نوكلياز Lambda Exonuclease / Exonuclease I Master Mix إلى 40 ميكرولتر (1 ميكروغرام) من الحمض النووي المرتبط بالمحول (الحجم الإجمالي 50 ميكرولتر). ضعها في جهاز تدوير حراري عند 37 درجة مئوية لمدة ساعة واحدة ، ثم 75 درجة مئوية لمدة 10 دقائق. امسك عند 4 درجات مئوية إلى أجل غير مسمى.
- أحجام الماصة 1.8x ، أو 90 ميكرولتر ، من حبات XP إلى الحمض النووي المعالج ب Lambda Exonuclease / Exonuclease I. قم بتنقية وفقا للتعليمات الواردة في الخطوات 4.3-4.6. قم بتقطيع 40 ميكرولتر من TE ، درجة الحموضة 8.0. تأكد من بقاء الخرزات في محلول للخطوة الأنزيمية التالية.
- يعالج بإنزيم المستخدم وبولي نوكليوتيدات تيناز T4 (PNK). قم بإعداد USER / T4 PNK Master Mix (مطلوب لتحرير الأجزاء المتدلية 4 نقاط أساس وإعداد نهايات الحمض النووي الجاهزة للربط اللازمة لتفاعل الربط اللاحق).
- امزج 5 ميكرولتر من 10x T4 DNA Ligase Buffer (التركيز النهائي 1x) ، و 3 ميكرولتر من إنزيم المستخدم (التركيز النهائي 0.05 U / μL) ، و 2 ميكرولتر من T4 PNK (التركيز النهائي 0.4 U / μL) ، للحصول على حجم إجمالي للمزيج الرئيسي USER / PNK البالغ 10 ميكرولتر.
- ماصة 10 ميكرولتر من إنزيم المستخدم / T4 PNK Master Mix إلى 40 ميكرولتر من عينة الحمض النووي المعالجة ب lambda و exonuclease I التي تحتوي على حبات من الخطوة 7.5.3 لحجم إجمالي قدره 50 ميكرولتر. ضعها في جهاز تدوير حراري عند 37 درجة مئوية لمدة 1 ساعة. امسك عند 4 درجات مئوية إلى أجل غير مسمى.
- الماصة 1.8x الحجم، أو 90 ميكرولتر، من محلول PEG/NaCl SPRI إلى الحمض النووي المعالج ب USER/T4 PNK وتنقيتها وفقا للخطوات 4.3-4.6. Elute في 35 ميكرولتر من TE ، درجة الحموضة 8.0. صب المادة الطافية في لوحة PCR جديدة شبه حوارة. اجمع بين الحمض النووي وحدده باستخدام مقايسة dsDNA HS.
- إجراء التدوير داخل الجزيئات. تحضير مزيج التدوير الرئيسي.
- امزج 8 ميكرولتر من H2O الخالي من النوكلياز ، و 10 ميكرولتر من 10x T4 DNA ligase المؤقت (التركيز النهائي 1x) ، و 2 ميكرولتر من T4 DNA ligase (التركيز النهائي 8 U / μL) ، للحصول على حجم إجمالي لمزيج Circularization Master Mix 20 μL.
- الماصة 20 ميكرولتر من المزيج الرئيسي للتعميم إلى 500 نانوغرام من الحمض النووي المعالج بالمستخدم / PNK من الخطوة 7.6.3. قم بتخفيف 500 نانوغرام من الحمض النووي المعالج ب USER / PNK في 80 ميكرولتر ، ثم أضف 20 ميكرولتر من Circularization Master Mix (الحجم الإجمالي 100 ميكرولتر). احتضن في جهاز تدوير حراري عند 16 درجة مئوية لمدة 16 ساعة (بين عشية وضحاها).
- أضف 1x حجم ، أو 100 ميكرولتر ، من حبات XP إلى الحمض النووي الدائري وقم بتنقيتها وفقا للخطوات 4.3-4.6. قم بإزالة 38 ميكرولتر من TE ، درجة الحموضة 8.0. صب المادة الطافية في لوحة PCR جديدة شبه حوارة.
- عالج باستخدام DNase المعتمد على ATP الآمن للبلازميد. تحضير مزيج DNase الرئيسي المعتمد على ATP الآمن للبلازميد (مطلوب لتدهور الحمض النووي الخطي المتبقي).
- امزج 5 ميكرولتر من 10x عازلة تفاعل آمنة للبلازميد (التركيز النهائي 1x) ، و 2 ميكرولتر من ATP (التركيز النهائي 1 ملليمتر) ، و 5 ميكرولتر من DNase المعتمد على ATP الآمن للبلازميد (التركيز النهائي 1 وحدة / ميكرولتر) ، للحصول على حجم إجمالي للمزيج الرئيسي الآمن للبلازميد البالغ 12 ميكرولتر.
- الماصة 12 ميكرولتر من DNase Master Mix المعتمد على ATP إلى 38 ميكرولتر من الحمض النووي الدائري من الخطوة 7.7.3 (الحجم الإجمالي 50 ميكرولتر). احتضن في جهاز تدوير حراري عند 37 درجة مئوية لمدة ساعة واحدة ، ثم 70 درجة مئوية لمدة 30 دقيقة. امسك عند 4 درجات مئوية إلى أجل غير مسمى.
- الماصة 1x أحجام ، أو 50 ميكرولتر ، من حبات XP في الحمض النووي المعالج باستخدام DNase المعتمد على ATP الآمن للبلازميد وتنقية وفقا للخطوات 4.3-4.6. قم بإزالة 15 ميكرولتر من TE ، درجة الحموضة 8.0. صب المادة الطافية في لوحة PCR جديدة شبه حوارة.
- اجمع بين الحمض النووي وحدد الكمية باستخدام مقايسة dsDNA HS. إذا لزم الأمر ، قم بتخزين الحمض النووي الدائري عند -20 درجة مئوية لمدة تصل إلى عدة أشهر.
8. شق gDNA المنقى إنزيميا والدائري في المختبر (2 ساعة)
- قم بإجراء الانقسام في المختبر باستخدام مركب Cas9: gRNA. تحضير مزيج Cleavage Master في المختبر . امزج 5 ميكرولتر من 10x Cas9 Buffer (التركيز النهائي 1x) ، و 4.5 ميكرولتر من S. pyogenes Cas9 (التركيز النهائي 90 نانومتر) ، و 1.5 ميكرولتر من gRNA (التركيز النهائي 90 نانومتر) للحصول على حجم إجمالي لمزيج الانقسام الرئيسي يبلغ 11 ميكرولتر.
- احتفظ بمزيج الانقسام الرئيسي في RT لمدة 10 دقائق لتشكيل مجمعات Cas9: gRNA RNP.
- قم بتخفيف 125 نانوغرام من الحمض النووي المعالج ب DNase الآمن بالبلازميد من الخطوة 7.8.3 إلى الحجم النهائي البالغ 39 ميكرولتر. بعد ذلك ، أضف 11 ميكرولتر من Cleavage Master Mix إلى 39 ميكرولتر من الحمض النووي المعالج ب DNase الآمن للبلازميد لحجم إجمالي قدره 50 ميكرولتر.
ملاحظة: قم بتضمين عينة تحكم سلبية في هذه الخطوة ، والتي تشتمل على الحمض النووي الدائري الممزوج بالمخزن المؤقت Cas9 ، بدون مركب Cas9: gRNA. - احتضن في جهاز تدوير حراري لمدة 1 ساعة عند 37 درجة مئوية. حافظ على 4 درجات مئوية إلى أجل غير مسمى. أضف 50 ميكرولتر (حجم 1x) من حبات XP إلى الحمض النووي المشقوق في المختبر وتنقية الحمض النووي باتباع الخطوات 4.3-4.6. Elute في 42 ميكرولتر من المخزن المؤقت TE ، الرقم الهيدروجيني 8.0. تأكد من بقاء الخرزات في الحل للخطوة التالية.
9. إعداد مكتبة التسلسل من الجيل التالي (4 - 6 ساعات)
- أداء A-tailing. تحضير مزيج رئيسي الذيل.
- امزج 5 ميكرولتر من 10x A-tailing Buffer (التركيز النهائي 1x) ، و 3 ميكرولتر من إنزيم المخلفات A (الحجم الإجمالي للمزيج الرئيسي A-tailing هو 8 ميكرولتر).
- الماصة 8 ميكرولتر من المزيج الرئيسي للمخلفات A إلى 42 ميكرولتر من عينة الحمض النووي المملحة التي تحتوي على حبات من الخطوة 8.4 (الحجم الإجمالي 50 ميكرولتر). ضعها في جهاز تدوير حراري لمدة 30 دقيقة عند 30 درجة مئوية. امسك عند 4 درجات مئوية إلى أجل غير مسمى.
- الماصة 1.8x أحجام من محلول PEG / NaCl SPRI ، أو 90 ميكرولتر ، إلى الحمض النووي ذو الذيل A وتنقية الحمض النووي وفقا للخطوات 4.3-4.6. التخمير في 25 ميكرولتر من TE ، الرقم الهيدروجيني 8.0. تأكد من الاحتفاظ بالخرزات في الحل للخطوة التالية.
- قم بإجراء ربط المحول. قم بإعداد مزيج ربط المحول الرئيسي.
ملاحظة: يجب تحضير حصص الاستخدام الواحد لمحولات NEB لمنع تكوين ثنائيات المحول الناتجة عن التحلل المائي الناجم عن التجميد والذوبان ل 3 'T'.- امزج 10 ميكرولتر من 5x Ligation Buffer (التركيز النهائي 1x) ، و 5 ميكرولتر من DNA Ligase ، و 10 ميكرولتر من المحول للتسلسل (التركيز النهائي هو 3 ميكرومتر) ، ليصبح المجموع 25 ميكرولتر.
- الماصة 25 ميكرولتر من مزيج ربط المحول الرئيسي في 25 ميكرولتر من عينة الحمض النووي ذات الذيل A التي تحتوي على حبات من الخطوة 9.1.3. ضعها في جهاز تدوير حراري لمدة ساعة واحدة عند 20 درجة مئوية. امسك عند 4 درجات مئوية إلى أجل غير مسمى.
- الماصة 1x أحجام ، أو 50 ميكرولتر ، من محلول PEG / NaCl SPRI إلى الحمض النووي المرتبط بالمحول وتنقية الحمض النووي وفقا للخطوات 4.3-4.6. المذموم في 47 ميكرولتر من TE ، الرقم الهيدروجيني 8.0. تأكد من بقاء الخرزات في محلول للخطوة الأنزيمية التالية.
- إجراء العلاج باستخدام إنزيم المستخدم (يولد فجوة نوكليوتيدات واحدة في بقايا اليوراسيل).
- أضف 3 ميكرولتر من إنزيم المستخدم (المضمن في مجموعة بادئات الفهرس المزدوج) إلى عينة الحمض النووي المربوطة بالمحول والتي تحتوي على حبات من الخطوة 9.2.3. احتضان عند 37 درجة مئوية لمدة 15 دقيقة.
- أضف 35 ميكرولتر (حجم 0.7x) من محلول PEG / NaCl SPRI إلى الحمض النووي المعالج بإنزيم المستخدم وتنقية وفقا للخطوات 4.3-4.6. قم بالتخلص من 20 ميكرولتر من المخزن المؤقت TE ، الرقم الهيدروجيني 8.0. انقل المادة الطافية إلى لوحة PCR جديدة شبه حوارية وقم بقياس تركيز الحمض النووي باستخدام مقايسة dsDNA HS. يجب أن يكون التركيز المتوقع حوالي 2-5 نانوغرام / ميكرولتر.
- (اختياري) قبل الانتقال إلى الخطوة التالية ، يمكن إجراء اختيار حجم الحمض النووي باستخدام PippinHT. استخدم كاسيت PippinHT بنسبة 1.5٪ بنطاق حجم يتراوح بين 250-850 نقطة أساس. يمكن استخدام العينات الناتجة مباشرة في تفاعل البوليميراز المتسلسل في الخطوة التالية.
- إجراء PCR لإضافة الباركود
ملاحظة: تأكد من أن مجموعات تسلسل التمهيدي المختارة لكل عينة فريدة من نوعها. إذا أمكن ، يجب أن تحتوي كل عينة على رموز شريطية فريدة من نوعها i5 و i7.- قم بإعداد PCR Master Mix لإضافة الرموز الشريطية ذات الفهرس المقترن. امزج 5 ميكرولتر من H2O الخالي من النوكلياز ، و 25 ميكرولتر من 2x HotStart Ready Mix (التركيز النهائي 1x) ، و 5 ميكرولتر من i5 Primer (التركيز النهائي 1 ميكرومتر) ، و 5 ميكرولتر من i7 Primer (التركيز النهائي 1 ميكرومتر) (إجمالي حجم المزيج الرئيسي 40 ميكرولتر).
- الماصة 40 ميكرولتر من PCR Master Mix إلى 10 ميكرولتر من الحمض النووي المنقى المعالج بإنزيم المستخدم (حوالي 20 نانوغرام) من الخطوة 9.3.2 (الحجم الإجمالي 50 ميكرولتر).
- اختر ظروف التدوير الحراري PCR التالية: التمسخ: 98 درجة مئوية لمدة 45 ثانية لدورة واحدة ، التمسخ: 98 درجة مئوية لمدة 15 ثانية لمدة 20 دورة ، الصلب: 65 درجة مئوية لمدة 30 ثانية لمدة 20 دورة ، التمديد: 72 درجة مئوية لمدة 30 ثانية لمدة 20 دورة ، التمديد النهائي: 72 درجة مئوية لمدة دقيقة واحدة لدورة واحدة ، الانتظار: 4 درجة مئوية إلى أجل غير مسمى.
- أضف 0.7x من وحدات التخزين ، أو 35 ميكرولتر ، من حبات XP إلى منتج PCR وقم بتنقيتها وفقا للخطوات 4.3-4.6. Elute في 30 ميكرولتر من TE ، درجة الحموضة 8.0. صب المادة الطافية في لوحة PCR جديدة شبه حوارة. إذا لزم الأمر ، قم بتخزين الحمض النووي الدائري عند -20 درجة مئوية لمدة تصل إلى عدة أشهر.
ملاحظة: قم بتشغيل عينة من PCR على Tapestation للتحكم في جودة المكتبة وتقييم تكوين ثنائيات المحول. إذا تم اكتشاف ثنائيات المحول، فكرر الخطوة 9.5.4.
10. القياس الكمي لمكتبات CIRCLE-seq بواسطة PCR الرقمي بالقطرات (dd_PCR) (6 ساعات)
ملاحظة: يمكن أيضا إجراء القياس الكمي باستخدام qPCR أو Tapestation أو طريقة مماثلة.
- ابدأ ب 5 ميكرولتر من الحمض النووي من المكتبة (خطوة PCR 9.5.4) ممزوجة جيدا مع 45 ميكرولتر من TE الخالي من النوكلياز ، ثم قم بعمل 1:10 تخفيفات تسلسلية لكل عينة في أحجام 50 ميكرولتر ، تتراوح من 10-1 إلى 10-8 تخفيف.
- قم بإعداد حل مخزون dd_PCR Master Mix. امزج 11 ميكرولتر من مزيج 2x dd_PCR للمجسات (التركيز النهائي 1x) ، 0.055 ميكرولتر من المسبار oSQT1310 (التركيز النهائي 250 نانومتر) ، 0.055 ميكرولتر من المسبار oSQT1311 (التركيز النهائي 250 نانومتر) ، 0.099 ميكرولتر من التمهيدي oSQT1274 (التركيز النهائي 450 نانومتر) ، 0.099 ميكرولتر من التمهيدي oSQT1275 (التركيز النهائي 450 نانومتر) ، و 6.292 ميكرولتر من H2O الخالي من النوكلياز ، لإجمالي حجم dd_PCR Master Mix يبلغ 17.6 ميكرولتر. قم بإعداد مزيج رئيسي لجميع العينات للتأكد من أن الأحجام كافية لسحب العينات بدقة.
- قم بفحص أدنى ثلاثة تخفيفات (10-6،10-7 و 10-8) في نسختين (في صفيحة 96 بئرا). يجب استخدام عنصر تحكم غير قالب (NTC).
- الماصة 17.6 ميكرولتر من dd_PCR Master Mix لكل عينة على النحو التالي: امزج 17.6 ميكرولتر مع 4.4 ميكرولتر من العينة (إضافة H2O الخالي من النوكلياز إلى البئر مع NTC) ، لحجم إجمالي قدره 22 ميكرولتر. أغلق اللوحة ، ثم جهاز الطرد المركزي عند 2000 × جم لمدة 1 دقيقة في RT.
- إجراء توليد القطرات وإعادة التدوير الحراري والتحليل. باستخدام نظام Droplet Reader PCR ، انقل خرطوشة DG8 (8 آبار) إلى حامل الخرطوشة. في صف الزيت في الخرطوشة ، قم بتوزيع 70 ميكرولتر من زيت توليد القطرات للمجسات.
ملاحظة: خذ 20 ميكرولتر من العينة من الخطوة 10.1.2. وإضافته إلى صف العينة من الخرطوشة ذات 8 آبار ، وتغطية الخرطوشة بحشية مطاطية DG8 ، ووضعها في مولد القطرات ، ثم إغلاقها لبدء العملية (تلقائيا). عند الانتهاء ، قم بإزالة الخرطوشة وانقل 40 ميكرولتر من صف القطرات في الخرطوشة ذات 8 آبار إلى لوحة PCR نصف حوارية سعة 96 بئرا ، مع التأكد من الماصة ببطء.- ضع الكتلة الحرارية في مانع تسرب اللوحة PX1 PCR. سيبدأ مانع التسرب في التسخين حتى 180 درجة مئوية عند تشغيله. ضع ختم حرارة الرقائق على اللوحة ، مع التأكد من أن الخط الأحمر في الأعلى. ضع اللوحة في PX1 واضغط على Seal.
- اختر ظروف Thermocycler التالية: تنشيط الإنزيم: 95 درجة مئوية لمدة 10 دقائق لمدة دورة واحدة ، التمسخ: 94 درجة مئوية لمدة 30 ثانية لمدة 40 دورة ، التلدين / التمديد: 60 درجة مئوية لمدة دقيقة واحدة لمدة 40 دورة ، إلغاء تنشيط الإنزيم: 98 درجة مئوية لمدة 10 دقائق لمدة دورة واحدة ، الانتظار: 4 درجة مئوية إلى أجل غير مسمى.
- على قارئ القطرات ، افتح البرنامج المتوافق وحدد الآبار التي سيتم قراءتها. حدد ABS كنوع التجربة و dd_PCR Supermix للتحقيقات. اختر Ch1 غير معروف للهدف 1 و Ch2 غير معروف للهدف 2. حدد تطبيق، ثم موافق. ضع اللوحة في قارئ القطرات. بالنسبة لمجموعة الصبغة ، اختر FAM / HEX وانقر فوق تشغيل.
- تحليل نتائج dd_PCR. قم ببوابة مجموعة القطرات الموجبة المزدوجة باستخدام التحكم السلبي كمرجع. احسب متوسط القيم المكررة ، ثم اضرب في عامل التخفيف وعامل التخفيف 5 أضعاف dd_PCR.
ملاحظة: يتم حساب إجمالي النسخ لكل ميكرولتر على النحو التالي: إجمالي النسخ لكل ميكرولتر = متوسط القيمة × 5 × عامل التخفيف ، حيث تمثل "القيمة المتوسطة" متوسط قيمة القياس الكمي من Ch1 و Ch2. - اجمع جميع العينات في مكتبة واحدة بتركيزات متساوية المولار. يجب أن تحتوي المكتبة المجمعة 1x على حوالي 4.5 × 109 جزيئات وحجمها الإجمالي 5 ميكرولتر.
11. تسلسل الجيل التالي
- أرسل العينات للتسلسل إلى وكالة خارجية ، مع التأكد من ملاحظة تسلسلات المحول الصحيحة.
12. تحليل بيانات CIRCLE-seq (1-3 ساعات)
- قم بتثبيت الإصدار 2.7 من Python و Burrows-Wheeler Aligner (BWA) و SAMtools. قم بتنزيل الجينوم المرجعي (على سبيل المثال ، hg38) من http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz.
ملاحظة: إذا كان جينوم الأنواع المستهدفة غير متوفر، فيمكن تشغيل سير العمل الحسابي CIRCLE-seq في وضع مستقل عن المرجع. في هذه الحالة ، يمكن تخطي هذه الخطوة. - قم بتنزيل وتثبيت مسار CIRCLE-seq باستخدام الأوامر التالية: (1) git clone https://github.com/tsailabSJ/circleseq.git ، (2) cd circleseq ، (3) pip install -r requirements.txt.
- قم بإنشاء ملف بيان بتنسيق YAML (.yaml). يوجد أدناه نموذج بيان يمكن استخدامه مع مجموعة البيانات النموذجية المتوفرة في برنامج CIRCLE-seq لاختبار سير العمل.
ملاحظة: (1) الجينوم المرجعي: البيانات / الإدخال / CIRCLEseq_test_genome.fa ؛ (2) analysis_folder: البيانات / المخرجات؛ (3) BWA: BWA. (4) samtools: samtools. (5) read_threshold: <القيمة>؛ (6) window_size: <القيمة>؛ (7) mapq_threshold: <القيمة>؛ (8) start_threshold: <القيمة>؛ (9) gap_threshold: <القيمة>؛ (10) mismatch_threshold: <القيمة>؛ (11) merged_analysis: صحيح. (12) عينات: U2OS_EMX1 ؛ (13) الهدف: GAGTCCGAGCAGAAGAAGAANGG; (14) القراءة 1: البيانات/الإدخال/EMX1.r1.fastq.gz؛ (15) القراءة 2: البيانات/الإدخال/EMX1.r2.fastq.gz؛ (16) controlread1: البيانات/الإدخال/EMX1_control.r1.fastq.gz؛ (17) controlread2: البيانات/الإدخال/EMX1_control.r2.fastq.gz؛ (18) الوصف: U2OS. تم استخدام قيم البيان التالية: read_threshold: 4 ، window_size: 3 ، mapq_threshold: 50 ، start_threshold: 1 ، gap_threshold: 3 ، mismatch_threshold: 6 - حدد ملف FASTA للجينوم المرجعي ، ودليل الإخراج للتحليل ، والمسارات إلى أوامر BWA و SAMtools . حدد التسلسلات المستهدفة والمسارات إلى ملفات FASTQ غير المضاعفة لكل من عينات النوكلياز المشقوقة وعينات التحكم. يمكن معالجة تجارب متعددة في وقت واحد في وضع الدفعات عن طريق تضمينها جميعا في ملف بيان واحد.
- قم بتنفيذ الأمر التالي للتحليلات القياسية المستندة إلى المرجع: (1) python /path/to/circleseq.py all - manifest; (2) /path/to/manifest.yaml.
- بدلا من ذلك ، قم بتنفيذ الأمر التالي للتحليلات القياسية غير المستندة إلى المرجع: (1) Python /path / to / circleseq.py خالية من المرجع - manifest ؛ (2) /path/to/manifest.yaml.
- عند تنفيذ المسار الكامل، ابحث عن نتائج الإخراج لكل خطوة في output_folder مميزة مخصصة لتلك الخطوة المحددة.
النتائج
هنا ، يتم استخدام CIRCLE-seq للتحقيق في مواقع الانقسام التي يسببها النوكلياز ل Cas9 في مجمع مع gRNA المصمم لاستهداف موقع تكامل الفيروس المرتبط بالغدة 1 (AAVS1) باستخدام الحمض النووي المعزول من الخلايا الجذعية المستحثة متعددة القدرات (iPSCs). تم وصف هذا الحمض النووي الريبي سابقا في منشورنا27. تم عزل ما يقرب من 25 ميكروغرام من gDNA من iPSCs ، وتم قصها من خلال الموجات فوق الصوتية المركزة ، وتحديد الحجم باستخدام تنقية حبة AMPure XP لإنتاج شظايا من حوالي 300 نقطة أساس في الثانية. من هذا ال 25 ميكروغرام من الحمض النووي ، حوالي 2-5 نانوغرام من الحمض النووي تم تعميمه بنجاح لانقسام Cas9: gRNA في المختبر . تم تصوير الإجراء بأكمله في الشكل 1.
بعد إجراء وتحليل CIRCLE-seq باستخدام سير عمل الحساب الخاص بنا ، يتم عرض تصور لجميع مواقع الانقسام المكتشفة داخل وخارج الهدف في الشكل 2 أ. قدم خط أنابيب CIRCLE-seq أيضا "قراءات مدمجة" ، تم تحليلها عبر برنامج إحصائي R لإنتاج مخطط مانهاتن يوضح مواقع الانقسام التي تم اكتشافها التي يسببها النوكلياز والتي تم تعيينها على طول كل كروموسوم (الشكل 2 ب).
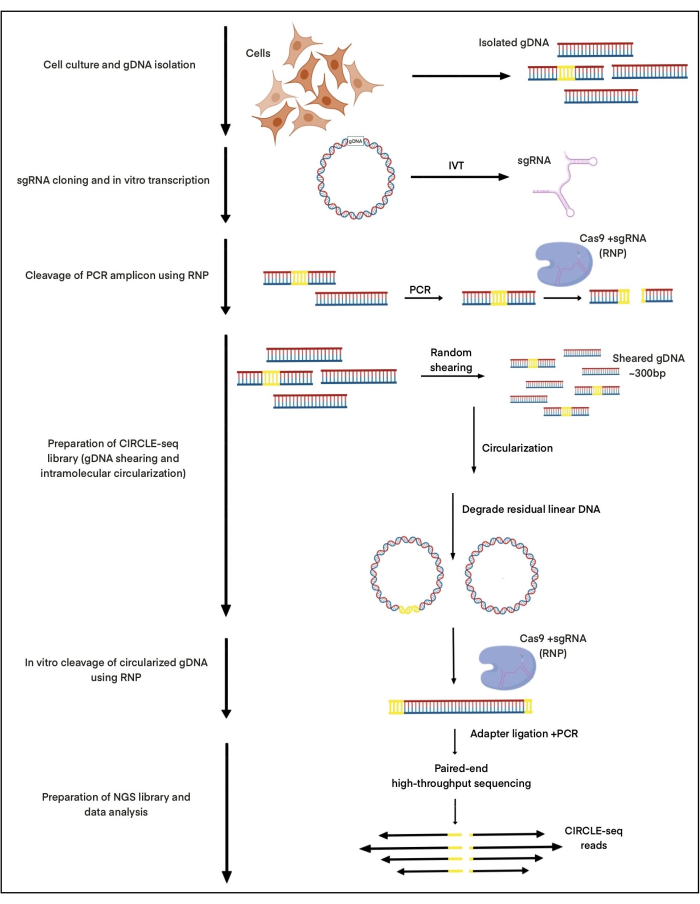
الشكل 1: مخططات سير عمل CIRCLE-seq. يشار إلى الخطوات الرئيسية للبروتوكول. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
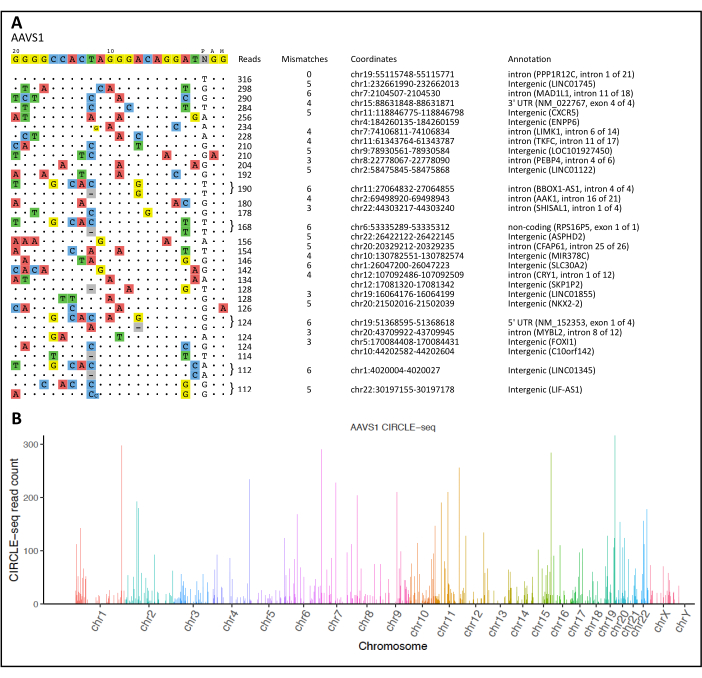
الشكل 2: تصور CIRCLE-seq ومخطط مانهاتن. (أ) محاذاة المواقع خارج الهدف مقابل الهدف المقصود لموضع AAVS1. يتم عرض التسلسل المستهدف في الجزء العلوي ، حيث يتم ترتيب الأهداف غير المستهدفة حسب عدد القراءة بترتيب تنازلي. يتم عرض الاختلافات في التسلسلات المستهدفة الأصلية بواسطة النيوكليوتيدات الملونة. يتم عرض عينة من أفضل مواقع الانقسام غير المقصودة لموضع AAVS1. (ب) مؤامرة مانهاتن توضح مواقع الانقسام غير المقصودة المكتشفة لموضع AAVS1. تمثل ارتفاعات الشريط عدد القراءة لكل موضع كروموسومي. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
| التمهيدي | التسلسل (5'-3') | تعليقات/وصف |
| AAVS1 RNA أحادي التوجيه (sgRNA) | GGGGCCACUAGGGACAGGAU | بالنسبة للبروتين الفلوري AAVS1 Locus |
| AAVS1 التمهيدي الأمامي | GCTCTGGGCGGAGGAATATG | لاختبار الانقسام في المختبر gRNA |
| AAVS1 التمهيدي العكسي | ATTCCCAGGGCCGGTTAATG | لاختبار الانقسام في المختبر gRNA |
| oSQT1288 | /5Phos / CGGTGGACCGATC / ideoxyU / ATCGGTCCACCGأتي | محول دبوس الشعر CIRCLE-seq |
| oSQT1274 | AATGATACGGCGACCACCACCGAG | TruSeq F1 |
| oSQT1275 | CAAGCAGAAGACGGCATACGAGAT | TruSeqF2 |
| oSQT1310 | /56-FAM / CCTACACGA / ZEN / CGCTCTTCCGATCT / 3IABkFQ / | مسبار TruSeq |
| oSQT1311 | /5HEX / TCGGAAGAG / ZEN / CACACGTCTGAACT / 3IABkFQ / | مسبار TruSeq |
الجدول 1: تسلسل gRNA والبادئات المستخدمة لتحليل CIRCLE-seq لموضع AAVS1 .
Discussion
هنا ، ثبت أن CIRCLE-seq هي تقنية غير متحيزة وحساسة للغاية لتحديد DSBs التي يسببها النوكلياز عبر الجينوم الناتج عن استهداف موضع AAVS1 في gDNA المشتق من iPSCs. ومن المعروف موقع AAVS1 داخل iPSCs أنه موقع الملاذ الآمن الذي غالبا ما يستخدم كموقع تكامل للجينات الخارجية باستخدام كريسبر-كاس928. درس تقريرنا الأخير إمكانات iPSCs المسمى EGFP من خلال إجراء تكامل بوساطة كريسبر لمراسل EGFP المعبر عنه بشكل أساسي في موقع AAVS1 , مما يتيح وضع العلامات وتتبع لكل من iPSCs و iPSCs المتمايزة بسبب استمرار EGFP في جميع أنحاء سلالة الخلية27. يمكن استخدام خط iPSC هذا في الجسم الحي لتقييم التوزيع العضوي للخلايا المشتقة من iPSC بعد الزرع. نظرا لأن هذا الخط الخلوي قد تم تعديله بواسطة كريسبر ويتم استخدامه أيضا لاختبار التطبيق السريري ل iPSCs ، فمن الضروري أن تكون مواقع AAVS1 المحتملة خارج الهدف معروفة واستجوابها لضمان السلامة والفعالية ، مما يجعلها مكانا مثاليا لاختبار CIRCLE-seq.
كان الاختلاف الملحوظ بين باترفيلد وآخرون ، الدراسة المنشورةسابقا 27 وهذه الدراسة هو استخدام gRNA معدل لاستهداف موضع AAVS1 . يمكن تصميم gRNA لتحسين دقة تحرير الجينوم24. تسلسل الدليل هو العامل الأكثر أهمية الذي يؤثر على الكفاءات داخل الهدف وخارجه. لذلك ، تم اختبار gRNA المختار مقابل العديد من الأدلة الأخرى ووجد أنه يتمتع بدقة فائقة. بالإضافة إلى ذلك ، أثبتت مقالة طرق حول إعادة برمجة الخلايا الليفية البشرية النتيجة القائلة بأن mRNAs ذات الغطاء الاصطناعي التي تحتوي على قواعد نووية معدلة تستفيد من التنشيط المنخفض للاستجابات المضادة للفيروسات29،30. في حين أن المناعة المنخفضة قد لا تكون ذات صلة في الفحص المختبري ، إلا أنها تصبح حاسمة عندما يكون الهدف النهائي هو تطوير علاج ذي صلة سريريا يمكن استخدامه في الخلايا الحية.
تتمتع CIRCLE-seq بالعديد من المزايا مقارنة بالطرق المماثلة. على سبيل المثال ، تسلسل Digenome-seq كلا من gDNA المشقوق بالنوكلياز وغير المشقوق ، باستخدام ~ 400 مليون قراءة21. ينتج عن هذا خلفية عالية ، مما يجعل من الصعب تصفية مواقع القطع ذات التردد المنخفض. يستخدم CIRCLE-seq فقط ~ 3-5 ملايين قراءة بسبب إثراء gDNA المشقوق بالنوكلياز ، مما يؤدي إلى خلفية منخفضة. بالإضافة إلى ذلك ، يعتمد Digenome-seq وطريقة مماثلة ، SITE-seq ، على تسلسل نهاية DNA واحدة مشقوقة بالنوكلياز. في المقابل ، تشمل قراءات CIRCLE-seq طرفي موقع القطع ، مما يتيح تحديد المواقع غير المستهدفة دون الحاجة إلى مرجع21،22،26.
تتمثل إحدى مزايا CIRCLE-seq في حساسيتها الأكبر مقارنة بالطرق التي تعتمد على زراعة الخلايا ، مثل GUIDE-seq. عندما تمت مقارنة الطريقتين ، تمكنت CIRCLE-seq من التقاط جميع المواقع غير المستهدفة التي اكتشفها GUIDE-seq واكتشفت مواقع انقسام إضافية غير مقصودة فاتتها GUIDE-seq. ومع ذلك ، فإن الاختلاف الملحوظ هو أن GUIDE-seq قد يعيقه المشهد اللاجيني ، في حين أن CIRCLE-seq يمكنه الوصول إلى الجينوم بأكمله.
كاختبار في المختبر ، يقدم CIRCLE-seq العديد من القيود ، أولها الكشف عن الإيجابيات الخاطئة. في حين أن علم التخلق سيعيق نشاط النوكلياز في مواقع معينة في الجسم الحي ، فإن الموجات فوق الصوتية تزيل هذه العقبات في المختبر ، مما يسمح بنشاط خارج الهدف في المواقع التي قد لا يمكن الوصول إليها عادة في سياق خلوي. علاوة على ذلك ، يوجد Cas9 بتركيزات عالية في هذا الاختبار في المختبر ، مما يسمح بالانقسام الذي لن يكون ممكنا في الجسم الحي. يتطلب هذا الاختبار أيضا قدرا كبيرا نسبيا من بدء gDNA ، والذي ، اعتمادا على الموارد المتاحة ، يمكن أن يلغي استخدام هذا البروتوكول. أخيرا ، من الممكن أن تكون بعض المواقع غير المستهدفة غير قابلة للكشف بسبب قيود تقنيات التسلسل الحالية من الجيل التالي.
استخدمت دراسة حديثة نهجا في السيليكو حددت خوارزميته عددا من المعلمات ذات الصلة التي يمكن من خلالها مقارنة طرق توصيف النوكلياز المختلفة ، بما في ذلك CIRCLE-seq و GUIDE-seq31. كان اثنان من المعلمات ذات الصلة هما "إثراء موقع القطع" و "النسبة المئوية للإيجابيات الكاذبة". ومن المثير للاهتمام ، أن معدل الإيجابية الكاذبة ل CIRCLE-seq تم حسابه عند 88٪ ، لكن تخصيب موقع القطع كان أعلى من الطرق الأخرى في المختبر . كشفت التحليلات المقارنة لكل طريقة أن GUIDE-seq كان الأفضل أداء ، حيث أظهر أكبر خصوصية على الهدف بمعدل إيجابي كاذب معتدلفقط 32. هذا لا يبطل CIRCLE-seq ، ولكنه يلمح إلى إمكانية استخدام CIRCLE-seq و GUIDE-seq جنبا إلى جنب ، والتحقق من صحة نتائج CIRCLE-seq مع GUIDE-seq نظرا لأن الأول يتمتع بحساسية أعلى ، في حين أن الأخير هو طريقة قائمة على الخلية مع إثراء عال لموقع القطع. تشير البيانات أيضا إلى أن تسلسل الجيل التالي القائم على الأمبليكون (NGS) يجب أن يكون الطريقة المفضلة لتحديد التعديلات الحقيقية خارج الهدف في المواقع المرشحةالمحتملة 31. تقترح هذه البيانات استراتيجية محتملة لاستخدام CIRCLE-seq ، متبوعا ب GUIDE-seq ، ثم NGS المستند إلى amplicon لفحص التأثيرات خارج الهدف.
Disclosures
المؤلفون ليس لديهم ما يكشفون عنه.
Acknowledgements
الإعراب عن عميق امتنانه للدعم التمويلي المقدم من المعاهد الوطنية للصحة (R01AR078551 و T32AR007411) ، وجمعية أبحاث انحلال البشرة الضمور (DEBRA) في النمسا ، وصندوق Gates Grubstake ، وصندوق Gates Frontiers.
Materials
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
References
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185 (2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856 (2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786 (2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058 (2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157 (2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474 (2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745 (2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved