Method Article
CIRCLE-Seq para interrogatório de edição de genes fora do alvo
Neste Artigo
Resumo
Uma barreira significativa para tecnologias como o CRISPR são os eventos fora do alvo que podem interromper genes vitais. 'Circularização para relatórios in vitro de efeitos de clivagem por sequenciamento' (CIRCLE-seq) é uma técnica projetada para identificar locais de clivagem não intencionais. Este método mapeia a atividade de todo o genoma do CRISPR-Cas9 com alta sensibilidade e sem viés.
Resumo
Circularização para Relato In Vitro de Efeitos de Clivagem por Sequenciamento (CIRCLE-seq) é uma nova técnica desenvolvida para a identificação imparcial de locais de clivagem não intencionais de CRISPR-Cas9 por meio de sequenciamento direcionado de DNA clivado de CRISPR-Cas9. O protocolo envolve a circularização do DNA genômico (gDNA), que é posteriormente tratado com a proteína Cas9 e um RNA guia (gRNA) de interesse. Após o tratamento, o DNA clivado é purificado e preparado como uma biblioteca para o sequenciamento Illumina. O processo de sequenciamento gera leituras de extremidade emparelhada, oferecendo dados abrangentes sobre cada local de clivagem. O CIRCLE-seq oferece várias vantagens sobre outros métodos in vitro , incluindo requisitos mínimos de profundidade de sequenciamento, baixo fundo e alto enriquecimento para gDNA clivado por Cas9. Essas vantagens aumentam a sensibilidade na identificação de eventos de clivagem intencionais e não intencionais. Este estudo fornece um procedimento abrangente e passo a passo para examinar a atividade fora do alvo do CRISPR-Cas9 usando o CIRCLE-seq. Por exemplo, este protocolo é validado pelo mapeamento de locais de clivagem não intencionais em todo o genoma de CRISPR-Cas9 durante a modificação do locus AAVS1 . Todo o processo CIRCLE-seq pode ser concluído em duas semanas, permitindo tempo suficiente para o crescimento celular, purificação do DNA, preparação da biblioteca e sequenciamento Illumina. A entrada de dados de sequenciamento no pipeline CIRCLE-seq facilita a interpretação e análise simplificadas dos locais de clivagem.
Introdução
A engenharia do genoma teve avanços significativos nos últimos vinte anos, com um marco importante sendo a descoberta de repetições palindrômicas curtas agrupadas regularmente interespaçadas (CRISPR)-Cas9 em 20121. Aproveitando a natureza programável das endonucleases de DNA bacteriano, a tecnologia CRISPR-Cas9 permite o direcionamento e a modificação precisos de quase todas as sequências de DNA. Desde a sua criação, o sistema foi otimizado para contar apenas com a endonuclease Cas9 e um RNA guia (gRNA) para editar regiões genômicas específicas. O potencial do CRISPR-Cas9 como terapia curativa foi demonstrado em ensaios clínicos para várias condições, como amaurose congênita de Leber, amiloidose por transtirretina e anemia falciforme, entre outras 2,3,4.
O CRISPR-Cas9 induz quebras de fita dupla (DSBs), que normalmente são resolvidas por um dos dois mecanismos: a junção de extremidade não homóloga propensa a erros (NHEJ) ou o reparo direcionado por homologia mais preciso (HDR), desde que um DNA modelo esteja disponível. A tendência do CRISPR-Cas9 de causar inserções e deleções associadas ao NHEJ (indels), juntamente com clivagem em locais genômicos não intencionais, limita sua aplicação em ambientes clínicos 5,6,7,8,9,10. Além disso, modificações genômicas não intencionais podem criar locais de emenda crípticos, mutações sem sentido ou sem sentido, induzir cromotripse ou conferir potencial oncogênico às células - resultados que foram observados em vários ensaios de edição de genoma11 , 12 , 13 , 14 , 15. Em conclusão, identificar com precisão a atividade fora do alvo do CRISPR-Cas9 é crucial para suas aplicações clínicas, particularmente em terapias genéticas sistêmicas que podem alterar bilhões de células.
Vários métodos podem ser empregados para identificar locais de clivagem fora do alvo CRISPR-Cas9, incluindo a Identificação Imparcial de Quebras de Fita Dupla em Todo o Genoma (GUIDE)-seq16, que usa oligodesoxinucleotídeos de fita dupla para marcar DSBs em células vivas. No entanto, uma crítica a esse método é que falsos positivos podem surgir de DSBs aleatórios ou de artefatos de PCR, que devem ser descartados excluindo locais capturados que apresentam baixa semelhança com os locais no alvo. O método baseado no uso de Integrase-Defective Lentiviral Vector (IDLV) é menos sensível e propenso a perder muitos locais fora do alvo17. Outros métodos in situ como DSBCapture, BLESS e BLISS 18,19,20 envolvem células fixas e rotulam DSBs diretamente, no entanto, são limitados por sua dependência da captura imediata de DSB e pela ausência de DNA exógeno. Digenome-seq21, um método in vitro, e Enriquecimento seletivo e identificação de extremidades de DNA genômico marcadas por sequenciamento (SITE-seq) 22 fornecem soluções de sequenciamento, mas têm suas limitações em ruído de fundo e análise de extremidade única, respectivamente. A descoberta de Cas in situ fora dos alvos e a verificação por sequenciamento (Discover-Seq)23 oferecem identificação in vivo e in situ da atividade de Cas9 por meio da ligação MRE11, mas detecta apenas DSBs que existem no momento da preparação da amostra24. Por fim, a Inferência de Edições CRISPR (ICE) usa uma abordagem de bioinformática para analisar de forma robusta as edições CRISPR usando dados Sanger25.
Este artigo descreve um procedimento detalhado para Circularização para Relato In Vitro de Efeitos de Clivagem por Sequenciamento (CIRCLE-seq): uma técnica in vitro que mapeia de forma sensível e imparcial a atividade fora do alvo em todo o genoma da nuclease Cas9 em um complexo com o gRNA de interesse26. Essa abordagem começa com a cultura das células de interesse e o isolamento do DNA, seguido de cisalhamento aleatório por ultrassom focalizado e, em seguida, tratamento com exonuclease e ligase. Em última análise, esse processo produz moléculas circulares de DNA de fita dupla, que são então purificadas por meio de tratamento DNase seguro para plasmídeos. Este DNA circular é então exposto ao complexo Cas9-gRNA, que cliva os locais de clivagem pretendidos e não intencionais, deixando para trás as extremidades expostas do DNA que atuam como substratos para a ligação do adaptador Illumina. Esse processo produz uma biblioteca diversificada de DNA genômico (gDNA) contendo ambas as extremidades de cada DSB induzido por nuclease, garantindo que cada leitura tenha todas as informações necessárias para cada local de clivagem. Isso permite o uso do sequenciamento Illumina com requisitos de cobertura de sequenciamento mais baixos, diferenciando o CIRCLE-seq de outros métodos semelhantes mencionados acima. É importante observar que, embora o CIRCLE-seq tenha maior sensibilidade fora do alvo do que outros protocolos como método in vitro , isso tem o custo de falsos positivos mais altos devido à ausência da paisagem epigenética presente em outros métodos, como o GUIDE-seq16. Além disso, o reparo do DNA DSB e seu maquinário associado não estão presentes no CIRCLE-seq, revogando indels ou reparos adequados que seriam observados de outra forma.
Além de descrever o protocolo passo a passo para realizar o CIRCLE-seq, o protocolo é validado pela identificação de locais de clivagem não intencionais em todo o genoma de CRISPR-Cas9 que ocorrem durante a modificação do locus AAVS1 , por exemplo. Este protocolo fácil de seguir fornece instruções detalhadas, desde a cultura de células-tronco pluripotentes induzidas (iPSCs) e isolamento de gDNA até circularização de gDNA, clivagem de Cas9-gRNA, preparação de biblioteca, sequenciamento e análise de pipeline. Dados os baixos requisitos de cobertura de sequenciamento, o CIRCLE-seq está disponível para qualquer laboratório com acesso ao sequenciamento de última geração.
Protocolo
Os detalhes dos reagentes, consumíveis e equipamentos usados para este estudo estão listados na Tabela de Materiais.
1. Cultura celular (5 dias)
- Inclua um controle negativo em todo este protocolo. Prepare células suficientes para 25 μg adicionais de gDNA (~ 2,0e7 células por amostra).
- Cultura de iPSCs de acordo com o protocolo estabelecido27. Colete as células e ressuspenda-as em 10 mL de PBS. Pipetar uma amostra de 6 μL de células e ressuspender na proporção de 1:1 com azul de tripano. Use o contador de células automatizado para contar a amostra.
- Alíquota 2 x 107 células por tubo e, em seguida, girar para baixo a 300 x g durante 3 min a 25 °C [temperatura ambiente (RT)]. Isso é suficiente para várias réplicas. Pipetar e rejeitar o sobrenadante.
2. Isolamento de DNA genômico (1 dia)
- Use o kit de purificação de DNA disponível comercialmente para isolar o gDNA, seguindo as instruções do fabricante:
- Adicione 200 μL de PBS a um tubo cônico de 15 mL contendo o pellet celular e ressuspenda. Em seguida, pipete 3 mL de tampão de lise celular e 15 μL de proteinase K no tubo. Inverta o tubo 25 vezes para misturar bem. Coloque o tubo em um agitador em banho-maria regulado para 55 °C e 150 rpm por 3 h ou durante a noite para obter o rendimento ideal do DNA.
- Pipetar em 15 μL de RNase A. Inverter 25 vezes. Colocar em banho-maria a 37 °C durante 1 h.
- Refrigere a amostra no gelo por 5 min. Em seguida, adicione 1 mL de solução de precipitação de proteínas, vórtice em alta velocidade por 20 s e centrifugue a 2000 x g por 10 min em RT. As proteínas devem formar um pellet visível e compacto no fundo do tubo. Se o pellet não estiver visível, incube a amostra no gelo por mais 5 min e centrifugue novamente.
- Adicione 3 mL de isopropanol a 100% a um novo tubo cônico de 15 mL. Pipetar cuidadosamente o sobrenadante do passo 2.1.3 para o tubo. Inverter o tubo 50 vezes para misturar e, em seguida, centrifugue a 2000 x g e temperatura ambiente por 3 min. Sem perturbar o pellet de DNA, aspire cuidadosamente o sobrenadante usando uma pipeta Pasteur conectada a uma armadilha de vácuo e inverta o tubo em um pano limpo e sem fiapos.
- Pipete 3 mL de etanol a 70% (v/v) no pellet de DNA e inverta 10 vezes para lavar. Centrifugue a 2.000 x g por 3 min em RT. Em seguida, despeje cuidadosamente o sobrenadante.
- Mantenha o tubo aberto e deixe o pellet de DNA resultante secar por 30 min, garantindo que todo o etanol tenha evaporado totalmente. Pipetar 50 μL de solução de hidratação de ADN e homogeneizar com pipetagem suave.
- Dissolver o ADN colocando a amostra num agitador em banho-maria a 65 °C durante 1 h e, em seguida, deixar a amostra em RT durante a noite. Centrifugue a amostra em RT e 2.000 x g por 1 min e use o kit de ensaio dsDNA BR com tubos associados para quantificar o DNA isolado com o fluorômetro.
3. Preparação de gRNA (7 dias)
- Encomende o gRNA sintético de interesse de uma fonte comercial (consulte a Tabela de Materiais). Este protocolo também é compatível com crRNA/tracrRNA.
4. Teste de clivagem in vitro de gRNA
NOTA: Aqui, um alvo no gene AAVS1 é usado. Para atingir outros genes de interesse, projete primers (Tabela 1) para amplificar a região alvo e substitua os primers nas etapas a seguir por primers personalizados.
- Prepare a reação de PCR: Misture 25 μL de Phusion Hot Start Flex 2x Master Mix (Concentração Final 1x), 0,5 μL de AAVS1 F Primer (Concentração final 0,1 μM), 0,5 μL de AAVS1 R Primer (Concentração final 0,1 μM), 5 μL de gDNA (100 ng, 20 ng/μL, da etapa 2.1.6) e 19 μL de H2O livre de nuclease (Volume total: 50 μL).
- Use os seguintes parâmetros do termociclador: Desnaturação: 98 °C por 2 min (1 ciclo), Desnaturação: 98 °C por 10 s (10 ciclos), Recozimento: 72-62 °C (-1 °C/ciclo) por 15 s (10 ciclos), Extensão: 72 °C por 30 s (10 ciclos), Desnaturação, 98 °C por 10 s (30 ciclos). Recozimento: 65 °C por 15 s (30 ciclos), Extensão: 72 °C por 30 s (30 ciclos), Extensão final: 72 °C por 5 min (1 ciclo), Hold: 4 °C indefinidamente.
- Use grânulos AMPure XP para purificar o produto da reação de PCR. Primeiro, pipete em volumes de 1,8x, ou 90 μL, de grânulos XP para o produto de PCR. Pipete dez vezes para misturar bem. Deixe a mistura em RT por 5 min para incubar.
- Usando um rack magnético, separe as esferas da solução colocando a placa de reação de PCR no ímã por 3 min. Pipetar a solução limpa e eliminar. Adicione 200 μL de etanol a 80% (v / v) aos grânulos, incube por 30 s e remova o etanol. Repita esta etapa de lavagem duas vezes para garantir a remoção completa do etanol.
- Deixe as amostras secarem naturalmente por 3 min, colocando a placa no ímã. Remova a placa do ímã e adicione 40 μL de tampão TE, pH 8,0. Misture pipetando para cima e para baixo dez vezes. Deixar a amostra repousar em RT durante 2 min.
- Coloque a placa de reação de PCR no ímã por mais um minuto. Após 1 min, transferir o sobrenadante para uma nova placa. Meça o rendimento de PCR purificado usando um espectrofotômetro e analise-o em um TapeStation usando uma tira de tubo óptico com uma tampa de tira de tubo óptico, juntamente com os reagentes D1000 ScreenTape de alta sensibilidade e D1000 de alta sensibilidade (escada e tampão), seguindo as instruções do fabricante. Conservar a amostra preparada a -20 °C durante vários meses.
- Dilua a proteína nuclease Cas9 a 1 μM da seguinte forma: Misture 2 μL de tampão Cas9 10x (concentração final 1x), 1 μL de nuclease Cas9, S. pyogenes (concentração final 1 μM) e 17 μL de H2O livre de nuclease em um volume total de 20 μL.
- Execute um procedimento livre de RNase para evitar a degradação do gRNA. Diluir o gRNA (da etapa 3.1) a 3 μM em H2O até um volume total de 10 μL.
NOTA: Use a seguinte fórmula para estimar o peso molecular do gRNA: Peso molecular do ssRNA (g/mol) = (comprimento do ssRNA (nt) x 321,47 g/mol) + 18,02 g/mol. Para referência, um gRNA de 104 nt de comprimento a 3 μM é de aproximadamente 100 ng/μL.
5. Cisalhamento de DNA (3 h)
- Prepare o ME220 direcionando primeiro o braço de controle. Em seguida, encha o reservatório com H2O deionizado purificado. No laptop da estação de controle, acesse Water Works e clique em Preencher. Ajuste a temperatura para 4.5 °C.
- Transfira 25 μg de gDNA para um microtubo (microtubo-130 AFA Fiber Screw-Cap). Em seguida, encha o tubo até um volume total de 130 μL com 1x TE. Use as seguintes condições para cisalhar o DNA para um comprimento médio de aprox. 300 bp: defina Duração para 10 s; Potência de pico para 70; Fator de serviço % a 20; Ciclos/Burst para 50; tudo isso definirá automaticamente a potência média para 14,0.
6. Purificação do DNA genômico cortado (1 h)
- Divida o DNA genômico cisalhado em duas porções de 65 μL cada. Purifique usando 1,8 vezes o volume de grânulos XP (117 μL), seguindo o procedimento descrito nas etapas 4.3-4.6. Transferir o sobrenadante para uma nova placa de PCR e medir a quantidade utilizando um espectrofotómetro.
- Execute 1 μL do gDNA cisalhado eluído em um TapeStation, de acordo com as instruções do fabricante, para garantir que o gDNA seja cortado para uma ampla distribuição de aprox. 300 pb. Se necessário, armazene o gDNA cisalhado a -20 °C por vários meses.
7. Preparação da biblioteca CIRCLE-seq (3 dias)
- Recozimento do adaptador Hairpin
- Ressuspenda oSQT1288 (Tabela 1), o adaptador de gancho de cabelo, a uma concentração final de 100 μM em 1x TE.
- Execute o recozimento do adaptador da seguinte forma: misture 40 μL de oSQT1288 (concentração final de 40 μM), 10 μL de 10x STE (concentração final 1x) e 50 μL de H2O livre de nuclease para um volume total de 100 μL.
- Use os seguintes parâmetros de recozimento: 95 °C por 5 min, -1 °C por min por 70 ciclos, mantenha a 4 °C indefinidamente.
- Execute o reparo final . Empregue o kit de preparação da biblioteca HTP sem PCR e prepare o Master Mix de reparo final.
- Misture 8 μL de H2O livre de nuclease, 7 μL de tampão de reparo final 10x (concentração final 1x) e 5 μL de mistura de enzimas de reparo final (volume final de 20 μL de Master Mix de reparo final total).
- Pipetar 20 μL da mistura principal de reparação final para a amostra de gDNA cortada dos passos 4.3-4.6. Misture 20 μL de End-Repair Master Mix com 50 μL de gDNA cisalhado para um volume final de 70 μL.
- Coloque a mistura em um termociclador a 20 °C por 30 min e mantenha a 4 °C indefinidamente.
- Adicione volumes de 1,7x, ou 120 μL, de grânulos XP e siga as etapas de purificação das etapas 4.3-4.6. Eluir com 42 μL de TE, pH 8,0. Certifique-se de que as contas permaneçam em solução para a próxima etapa.
- Execute A-tailing. Usando o kit de preparação de biblioteca HTP (sem PCR), prepare o A-tailing Master Mix.
- Misture 5 μL de tampão de cauda A 10x (Concentração Final 1x) e 3 μL de enzima de cauda A (volume final total de 8 μL de Master Mix de cauda A).
- Pipete 8 μL do A-tailing Master Mix em cada amostra de DNA contendo grânulos da etapa 7.2.4, misturando 8 μL de A-tailing Master Mix para 42 μL de grânulos contendo DNA reparado final (volume final total de 50 μL). Colocar num termociclador a 30 °C durante 30 min. Manter a 4 °C indefinidamente.
- Pipete 1,8 volumes, ou 90 μL, de solução de SPRI de PEG / NaCl (um componente do Kit de Preparação da Biblioteca HTP (sem PCR; 96 reações)) para o DNA de cauda A. Purifique o DNA de cauda A de acordo com as etapas 4.3-4.6. Eluir o DNA de cauda A em 30 μL de TE, pH 8,0. Mantenha as contas em solução para a próxima etapa.
- Execute a ligação do adaptador. Usando o kit de preparação de biblioteca HTP (sem PCR), prepare o Master Mix de ligação do adaptador.
- Misture 10 μL de tampão de ligação 5x (concentração final 1x), 5 μL de DNA ligase e 5 μL de adaptador de grampo de cabelo recozido (40 μM) da etapa 7.1.3. Garanta a concentração final de 4 μM para um Master Mix de ligação do adaptador total de 20 μL.
- Pipetar 20 μL de Adapter Ligation Master Mix em cada amostra de ADN eluída contendo esferas do passo 7.3.3 (volume final total de 50 μL por amostra).
- Colocar num termociclador a 20 °C durante 1 h. Manter a 4 °C indefinidamente.
- Transfira 1x volumes, ou 50 μL, de solução de SPRI de PEG / NaCl para o DNA ligado ao adaptador e purifique de acordo com as etapas 4.3-4.6. Eluir com 30 μL de TE, pH 8,0, e decantar os sobrenadantes para uma nova placa de PCR semi-contornada. Combine e quantifique o DNA usando o ensaio dsDNA BR. Se necessário, armazene o DNA purificado ligado ao adaptador por até 1 mês a -20 °C.
- Prepare o Master Mix de Exonuclease Lambda / Exonuclease I (E. coli) (funções na eliminação de DNA de fita simples ou dupla sem adaptadores ligados a ambas as extremidades).
- Pegue 1 μg de DNA ligado ao adaptador da etapa 7.4.4, diluindo-o para 40 μL. Misture 5 μL de tampão de reação 10x Exonuclease I (Concentração Final 1x), 4 μL de Exonuclease Lambda (Concentração Final 0,4 U/μL) e 1 μL de Exonuclease I (E. coli) (Concentração Final 0,4 U/μL) para um volume total de Lambda Exonuclease/Exonuclease I Master Mix de 10 μL.
- Pipetar 10 μL da Lambda Exonuclease / Exonuclease I Master Mix para 40 μL (1 μg) de DNA ligado ao adaptador (volume total de 50 μL). Coloque em um termociclador a 37 °C por 1 h e depois a 75 °C por 10 min. Manter a 4 °C indefinidamente.
- Pipete 1,8x volumes, ou 90 μL, de grânulos XP para o DNA tratado com Exonuclease Lambda / Exonuclease I. Purifique de acordo com as instruções fornecidas nas etapas 4.3-4.6. Eluir em 40 μL de TE, pH 8,0. Certifique-se de que as esferas permaneçam em solução para a próxima etapa enzimática.
- Trate com enzima USER e T4 Polynucleotide Kinase (PNK). Prepare o USER/T4 PNK Master Mix (necessário para liberar as saliências de 4 pb e preparar as extremidades de DNA prontas para ligação necessárias para a reação de ligação subsequente).
- Misture 5 μL de tampão DNA ligase T4 10x (concentração final 1x), 3 μL de enzima USER (concentração final 0,05 U/μL) e 2 μL de T4 PNK (concentração final 0,4 U/μL), para um volume total da mistura master USER/PNK de 10 μL.
- Pipete 10 μL da enzima USER / T4 PNK Master Mix para 40 μL de amostra de DNA tratada com lambda e exonuclease I contendo grânulos da etapa 7.5.3 para um volume total de 50 μL. Colocar num termociclador a 37 °C durante 1 h. Manter a 4 °C indefinidamente.
- Pipetar 1,8x volumes, ou 90 μL, de solução de SPRI de PEG/NaCl para o DNA tratado com PNK USER/T4 e purificar de acordo com as etapas 4.3-4.6. Eluir em 35 μL de TE, pH 8,0. Transvase o sobrenadante para uma nova placa de PCR semi-contornada. Combine e quantifique o DNA usando o ensaio dsDNA HS.
- Realize a circularização intramolecular. Prepare o Master Mix de Circularização.
- Misture 8 μL de H2O livre de nuclease, 10 μL de tampão 10x T4 DNA ligase (Concentração Final 1x) e 2 μL de T4 DNA ligase (Concentração Final 8 U/μL), para um volume total do Master Mix de Circularização de 20 μL.
- Pipetar 20 μL do Master Mix de Circularização para 500 ng de DNA tratado com USER/PNK da etapa 7.6.3. Diluir 500 ng de DNA tratado com USER/PNK em 80 μL e, em seguida, adicionar 20 μL do Master Mix de Circularização (volume total de 100 μL). Incubar num termociclador a 16 °C durante 16 h (durante a noite).
- Adicione 1x volumes, ou 100 μL, de grânulos XP ao DNA circularizado e purifique de acordo com as etapas 4.3-4.6. Eluir em 38 μL de TE, pH 8,0. Transvase o sobrenadante para uma nova placa de PCR semi-contornada.
- Trate com DNase dependente de ATP seguro para plasmídeo. Preparar a mistura mestra de DNase dependente de ATP segura para plasmídeos (necessária para a degradação do ADN linear residual).
- Misture 5 μL de tampão de reação seguro para plasmídeo 10x (concentração final 1x), 2 μL de ATP (concentração final 1 mM) e 5 μL de DNase dependente de ATP seguro para plasmídeo (concentração final 1 U/μL), para um volume total do Master Mix seguro para plasmídeo de 12 μL.
- Pipete 12 μL do DNase Master Mix dependente de ATP para 38 μL de DNA circularizado da etapa 7.7.3 (volume total de 50 μL). Incubar num termociclador a 37 °C durante 1 h e depois a 70 °C durante 30 min. Manter a 4 °C indefinidamente.
- Pipete 1x volumes, ou 50 μL, de grânulos XP no DNA tratado com DNase dependente de ATP seguro para plasmídeo e purifique de acordo com as etapas 4.3-4.6. Eluir em 15 μL de TE, pH 8,0. Transvase o sobrenadante para uma nova placa de PCR semi-contornada.
- Combine o DNA e quantifique usando o ensaio dsDNA HS. Se necessário, armazene o DNA circularizado a -20 °C por vários meses.
8. GDNA circularizado purificado enzimaticamente in vitro (2 h)
- Realize clivagem in vitro com o complexo Cas9:gRNA. Prepare o Master Mix de clivagem in vitro . Misture 5 μL de tampão Cas9 10x (concentração final 1x), 4,5 μL de S. Pyogenes Cas9 (concentração final 90 nM) e 1,5 μL de gRNA (concentração final 90 nM) para um volume total de clivagem Master Mix de 11 μL.
- Mantenha a mistura master de clivagem em RT por 10 min para formar complexos Cas9:gRNA RNP.
- Diluir 125 ng de DNA tratado com DNase segura para plasmídeo da etapa 7.8.3 até um volume final de 39 μL. Em seguida, adicione 11 μL do Cleavage Master Mix a 39 μL de DNA tratado com DNase segura para plasmídeo para um volume total de 50 μL.
NOTA: Inclua uma amostra de controle negativo nesta etapa, que compreende DNA circularizado misturado com tampão Cas9, sem o complexo Cas9: gRNA. - Incubar num termociclador durante 1 h a 37 °C. Manter a 4 °C indefinidamente. Adicione 50 μL (1x volume) de grânulos XP ao DNA clivado in vitro e purifique o DNA seguindo as etapas 4.3-4.6. Eluir em 42 μL de tampão TE, pH 8,0. Certifique-se de que as contas permaneçam em solução para a próxima etapa.
9. Preparação da biblioteca de sequenciamento de próxima geração (4 - 6 h)
- Execute A-tailing. Prepare o A-tailing Master Mix.
- Misture 5 μL de tampão de cauda A 10x (concentração final 1x) e 3 μL de enzima de cauda A (o volume total da mistura mestre de cauda A é de 8 μL).
- Pipetar 8 μL do Master Mix de cauda A para 42 μL de amostra de DNA eluído contendo grânulos da etapa 8.4 (volume total de 50 μL). Colocar num termociclador durante 30 min a 30 °C. Manter a 4 °C indefinidamente.
- Pipete 1,8x volumes de solução de SPRI de PEG/NaCl, ou 90 μL, para o DNA de cauda A e purifique o DNA de acordo com as etapas 4.3-4.6. Eluir em 25 μL de TE, pH 8,0. Certifique-se de manter as contas em solução para a etapa seguinte.
- Execute a ligação do adaptador. Prepare o Master Mix de ligação do adaptador.
NOTA: Alíquotas de uso único de adaptadores NEB devem ser preparadas para evitar a formação de dímeros de adaptador causados pela hidrólise induzida por congelamento e descongelamento do 3' T'.- Misture 10 μL de tampão de ligação 5x (concentração final 1x), 5 μL de DNA ligase e 10 μL de adaptador para sequenciamento (a concentração final é de 3 μM), para um total de 25 μL.
- Pipetar 25 μL do Adapter Ligation Master Mix para 25 μL de amostra de ADN com cauda em A contendo esferas do passo 9.1.3. Colocar num termociclador durante 1 h a 20 °C. Manter a 4 °C indefinidamente.
- Pipetar 1x volumes, ou 50 μL, de solução de SPRI de PEG/NaCl para o ADN ligado ao adaptador e purificar o ADN de acordo com os passos 4.3-4.6. Eluir em 47 μL de TE, pH 8,0. Certifique-se de que as esferas permaneçam em solução para a próxima etapa enzimática.
- Realize o tratamento com enzima USER (gera uma lacuna de nucleotídeo único nos resíduos de uracila).
- Adicione 3 μL de enzima USER (incluída no Kit de Primers de Índice Duplo) à amostra de DNA ligada ao adaptador contendo grânulos da etapa 9.2.3. Incubar a 37 °C durante 15 min.
- Adicione 35 μL (0,7x o volume) de solução de SPRI de PEG/NaCl ao DNA tratado com enzimas do USUÁRIO e purifique de acordo com as etapas 4.3-4.6. Eluir em 20 μL de tampão TE, pH 8,0. Transferir o sobrenadante para uma nova placa de PCR semi-contornada e medir a concentração de ADN utilizando o ensaio dsDNA HS. A concentração esperada deve ser de aprox. 2-5 ng/μL.
- (Opcional) Antes de prosseguir para a próxima etapa, a seleção do tamanho do DNA pode ser realizada usando PippinHT. Use o PippinHT de 1,5% com uma faixa de tamanho de 250-850 bp. As amostras resultantes podem ser utilizadas diretamente na PCR na próxima etapa.
- Realize PCR para adição de código de barras
NOTA: Certifique-se de que as combinações de sequência de primer escolhidas para cada amostra sejam exclusivas. Se possível, cada amostra deve ter códigos de barras i5 e i7 exclusivos.- Prepare o PCR Master Mix para adicionar códigos de barras de índice emparelhado. Misture 5 μL de H2O livre de nuclease, 25 μL de 2x HotStart Ready Mix (concentração final 1x), 5 μL de primer i5 (concentração final de 1 μM) e 5 μL de primer i7 (concentração final de 1 μM) (volume total da master mix de 40 μL).
- Pipetar 40 μL do PCR Master Mix para 10 μL de ADN purificado tratado com a enzima USER (aprox. 20 ng) do passo 9.3.2 (volume total de 50 μL).
- Escolha as seguintes condições de termociclagem de PCR: Desnaturação: 98 °C por 45 s por 1 ciclo, Desnaturação: 98 °C por 15 s por 20 ciclos, Recozimento: 65 °C por 30 s por 20 ciclos, Extensão: 72 °C por 30 s por 20 ciclos, Extensão final: 72 °C por 1 min por 1 ciclo, Hold: 4 °C indefinidamente.
- Adicione 0,7x volumes, ou 35 μL, de grânulos XP ao produto PCR e purifique de acordo com as etapas 4.3-4.6. Eluir em 30 μL de TE, pH 8,0. Transvase o sobrenadante para uma nova placa de PCR semi-contornada. Se necessário, armazene o DNA circularizado a -20 ° C por vários meses.
NOTA: Execute uma amostra do PCR no Tapestation para controlar a qualidade da biblioteca e avaliar a formação de dímeros adaptadores. Se forem detectados dímeros adaptadores, repita a etapa 9.5.4.
10. Quantificação de bibliotecas CIRCLE-seq por PCR digital de gotículas (dd_PCR) (6 h)
NOTA: A quantificação também pode ser realizada usando qPCR, Tapestation ou um método semelhante.
- Comece com 5 μL de DNA da biblioteca (etapa de PCR 9.5.4) bem misturado com 45 μL de TE livre de nuclease e, em seguida, faça diluições seriais 1:10 de cada amostra em volumes de 50 μL, variando de 10-1 a 10-8 diluição.
- Configure a solução de estoque dd_PCR Master Mix. Misture 11 μL de mistura de 2x dd_PCR para sondas (Concentração Final 1x), 0,055 μL de Sonda oSQT1310 (Concentração Final 250 nM), 0,055 μL de Sonda oSQT1311 (Concentração Final 250 nM), 0,099 μL de Primer oSQT1274 (Concentração Final 450 nM), 0,099 μL de Primer oSQT1275 (Concentração Final 450 nM) e 6,292 μL de H2O livre de nuclease, para um volume total dd_PCR Master Mix de 17,6 μL. Prepare uma mistura principal para todas as amostras para garantir que os volumes sejam suficientes para uma pipetagem precisa.
- Analise as três diluições mais baixas (10-6,10-7 e 10-8) em duplicata (em uma placa de 96 poços). Um controle não modelo (NTC) deve ser usado.
- Pipete 17,6 μL de dd_PCR Master Mix para cada amostra da seguinte forma: Misture 17,6 μL com 4,4 μL da amostra (adicionando H2O livre de nuclease ao poço com NTC), para um volume total de 22 μL. Feche a placa e centrifugue a 2000 x g por 1 min em RT.
- Realize geração, termociclagem e análise de gotículas. Usando um sistema de PCR Droplet Reader, transfira um cartucho DG8 (8 poços) para o suporte do cartucho. Na linha de óleo do cartucho, dispense 70 μL de óleo de geração de gotículas para sondas.
NOTA: Pegue 20 μL da amostra da etapa 10.1.2. e adicione-o à linha de amostra do cartucho de 8 poços, cobrindo o cartucho com a junta de borracha DG8, colocando-o no gerador de gotículas e, em seguida, fechando-o para iniciar o processo (automático). Quando terminar, remova o cartucho e mova 40 μL da fileira de gotículas do cartucho de 8 poços para uma placa de PCR de 96 poços semi-saiada, certificando-se de pipetar lentamente.- Coloque o bloco de calor no selador de placas PCR PX1. O selador começará a aquecer até 180 °C quando ligado. Coloque a vedação térmica da folha na placa, garantindo que a linha vermelha esteja no topo. Coloque a placa no PX1 e pressione Seal.
- Escolha as seguintes condições do termociclador: Ativação enzimática: 95 °C por 10 min por 1 ciclo, Desnaturação: 94 °C por 30 s por 40 ciclos, Recozimento/Extensão: 60 °C por 1 min por 40 ciclos, Desativação enzimática: 98 °C por 10 min por 1 ciclo, Hold: 4 °C indefinidamente.
- No leitor de gotículas, abra o software compatível e selecione quais poços devem ser lidos. Selecione ABS como o tipo de experimento e dd_PCR Supermix para sondas. Escolha Ch1 Desconhecido para o Alvo 1 e Ch2 Desconhecido para o Alvo 2. Selecione Aplicar e, em seguida, OK. Coloque a placa no leitor de gotículas. Para o conjunto de tinturas, escolha FAM/HEX e clique em Executar.
- Analise os resultados dd_PCR. Bloqueie a população de gotículas duplamente positivas usando o controle negativo como referência. Calcular a média dos valores duplicados e multiplicar pelo factor de diluição e pelo factor de diluição de 5 vezes do dd_PCR.
NOTA: O total de cópias por microlitro é calculado do seguinte modo: total de cópias por microlitro = valor médio × factor de diluição de 5 ×, em que o «valor médio» representa o valor médio de quantificação de Ch1 e Ch2. - Combine todas as amostras em uma única biblioteca em concentrações equimolares. A biblioteca agrupada 1x deve conter aprox. 4,5 x 109 moléculas e ter um volume total de 5 μL.
11. Sequenciamento de última geração
- Envie as amostras para sequenciamento a uma agência externa, garantindo que as sequências corretas do adaptador sejam anotadas.
12. Análise de dados CIRCLE-seq (1 - 3 h)
- Instale o Python versão 2.7, Burrows-Wheeler Aligner (BWA) e SAMtools. Baixe o genoma de referência (por exemplo, hg38) do http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz.
NOTA: Se o genoma da espécie-alvo não estiver disponível, o fluxo de trabalho computacional CIRCLE-seq pode ser executado em um modo independente de referência. Nessa situação, essa etapa pode ser ignorada. - Baixe e instale o pipeline CIRCLE-seq com os seguintes comandos: (1) git clone https://github.com/tsailabSJ/circleseq.git, (2) cd circleseq, (3) pip install -r requirements.txt.
- Crie um arquivo de manifesto no formato YAML (.yaml). Abaixo está um manifesto de exemplo que pode ser usado com o conjunto de dados de exemplo fornecido no software CIRCLE-seq para testar o fluxo de trabalho.
NOTA: (1) genoma de referência: dados / entrada / CIRCLEseq_test_genome.fa; (2) analysis_folder: dados/saída; (3) BWA: BWA; (4) Samtools: Samtools; (5) read_threshold: ; (6) window_size: ; (7) mapq_threshold: ; (8) start_threshold: ; (9) gap_threshold: ; (10) mismatch_threshold: ; (11) merged_analysis: Verdadeiro; (12) amostras: U2OS_EMX1; (13) alvo: GAGTCCGAGCAGAAGAAGAANGG; (14) read1: dados/entrada/EMX1.r1.fastq.gz; (15) read2: dados/entrada/EMX1.r2.fastq.gz; (16) controlread1: dados/entrada/EMX1_control.r1.fastq.gz; (17) controlread2: dados/entrada/EMX1_control.r2.fastq.gz; (18) descrição: U2OS. Os seguintes valores de manifesto foram usados: read_threshold: 4, window_size: 3, mapq_threshold: 50, start_threshold: 1, gap_threshold: 3, mismatch_threshold: 6 - Defina o arquivo FASTA do genoma de referência, o diretório de saída para análise e os caminhos para os comandos BWA e SAMtools . Defina as sequências de destino e os caminhos para os arquivos FASTQ demultiplexados para as amostras clivadas por nuclease e controle. Vários experimentos podem ser processados simultaneamente no modo de lote, incluindo todos eles em um único arquivo de manifesto.
- Execute o seguinte comando para análises baseadas em referência padrão: (1) python /path/to/circleseq.py all - manifesto; (2) /caminho/para/manifest.yaml.
- Como alternativa, execute o seguinte comando para análises padrão não baseadas em referência: (1) Python /path/to/circleseq.py reference-free - manifesto; (2) /caminho/para/manifest.yaml.
- Ao executar o pipeline completo, encontre os resultados de saída de cada etapa em um output_folder distinto designado para essa etapa específica.
Resultados
Aqui, o CIRCLE-seq é utilizado para investigar os locais de clivagem induzidos por nuclease de Cas9 em um complexo com o gRNA projetado para atingir o local de integração do vírus adeno-associado 1 (AAVS1) usando DNA isolado de células-tronco pluripotentes induzidas (iPSCs). Este gRNA foi descrito anteriormente em nossa publicação27. Aproximadamente 25 μg de gDNA foram isolados de iPSCs, cortados por ultrassom focalizado e o tamanho selecionado usando purificação de esferas AMPure XP para produzir fragmentos de aprox. 300 bps. A partir desses 25 μg de DNA, aprox. 2-5 ng de DNA foram circularizados com sucesso para clivagem in vitro de Cas9: gRNA. Todo o procedimento é representado na Figura 1.
Seguindo um procedimento e análise CIRCLE-seq usando nosso fluxo de trabalho de computação, uma visualização de todos os locais de clivagem dentro e fora do alvo detectados é apresentada na Figura 2A. O pipeline CIRCLE-seq também forneceu 'leituras mescladas', analisadas por meio de software estatístico R para produzir um gráfico de Manhattan mostrando os locais de clivagem induzidos por nuclease detectados mapeados ao longo de cada cromossomo (Figura 2B).
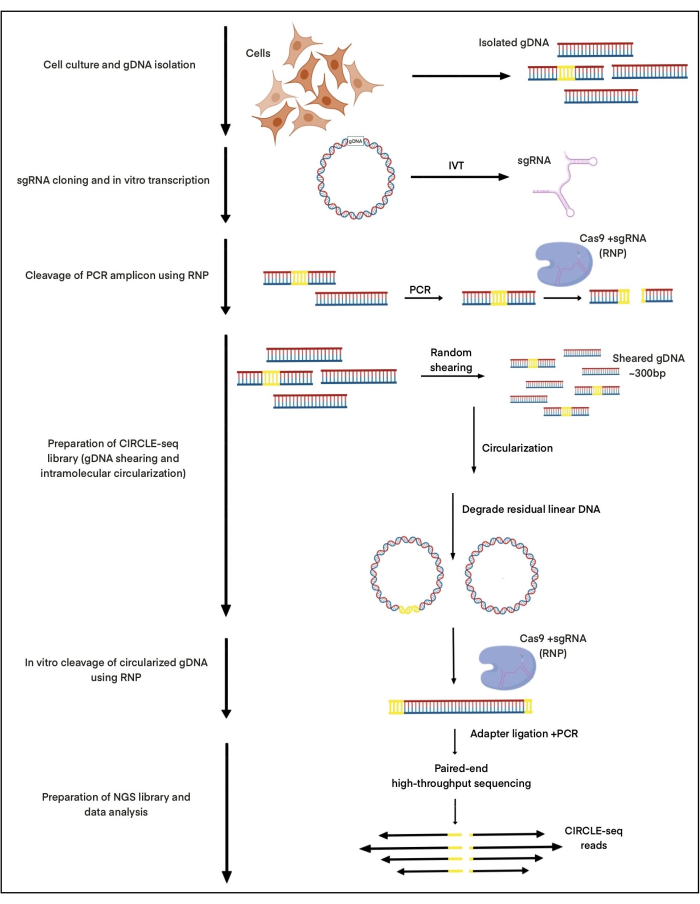
Figura 1: Esquemas de fluxo de trabalho CIRCLE-seq. As principais etapas do protocolo são indicadas. Clique aqui para ver uma versão maior desta figura.
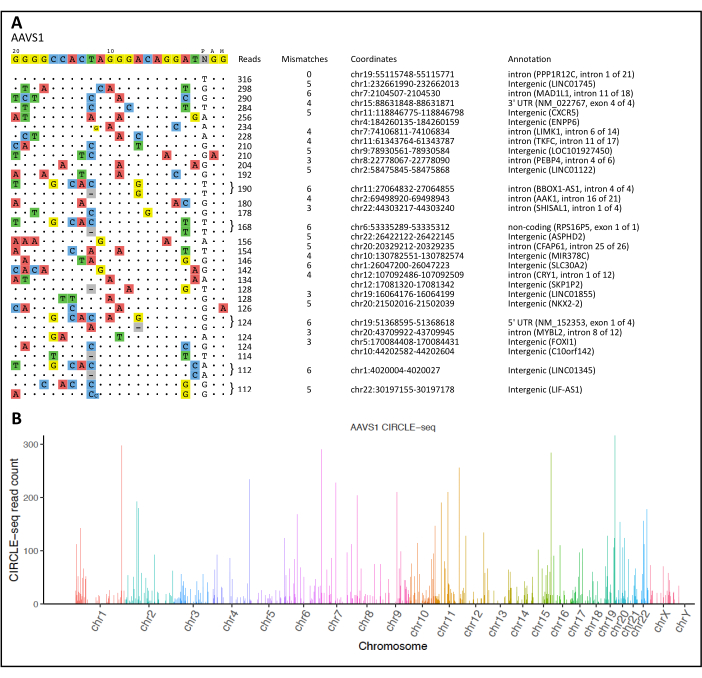
Figura 2: Visualização CIRCLE-seq e Manhattan Plot. (A) Alinhamento de locais fora do alvo contra o alvo pretendido para o locus AAVS1. A sequência de destino é exibida na parte superior, onde os alvos fora são classificados por contagem de leitura em ordem decrescente. As diferenças nas sequências alvo originais são mostradas por nucleotídeos coloridos. Uma amostra dos principais locais de clivagem não intencionais para o locus AAVS1 é mostrada. (B) Gráfico de Manhattan ilustrando os locais de clivagem não intencionais detectados para o locus AAVS1. As alturas das barras representam contagens de leitura para cada posição cromossômica. Clique aqui para ver uma versão maior desta figura.
| Cartilha | Sequência (5'-3') | Comentários/Descrição |
| AAVS1 RNA guia único (sgRNA) | GGGGCCACUAGGGACAGGAU | Para AAVS1 Locus' Fluorescent Protein Knock-In |
| AAVS1 Primer Direto | GCTCTGGGCGGAGGAATATG | Para teste de clivagem in vitro de gRNA |
| AAVS1 Primer Reverso | ATTCCCAGGGCCGGTTAATG | Para teste de clivagem in vitro de gRNA |
| oSQT1288 | /5Phos/CGGTGGACCGATGATC /ideoxyU/ATCGGTCCACCGaT | Adaptador de grampo de cabelo CIRCLE-seq |
| oSQT1274 | AATGATACGGCGACCACCGAG | TruSeq F1 |
| oSQT1275 | CAAGCAGAAGACGGCATACGAGAT | TruSeqF2 |
| oSQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | Sonda TruSeq |
| oSQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | Sonda TruSeq |
Tabela 1: Sequências de gRNA e primers usados para análise CIRCLE-seq do locus AAVS1 .
Discussão
Aqui, o CIRCLE-seq demonstrou ser uma técnica imparcial e altamente sensível para identificar DSBs induzidos por nuclease em todo o genoma resultante do direcionamento do locus AAVS1 no gDNA derivado de iPSCs. O local AAVS1 dentro de iPSCs é conhecido como um locus de porto seguro que é frequentemente usado como um local de integração de genes exógenos usando CRISPR-Cas928. Nosso relatório recente estudou o potencial de iPSCs marcadas com EGFP realizando a integração mediada por CRISPR de um repórter EGFP expresso constitutivamente no local AAVS1 , o que permite a rotulagem e rastreamento de iPSCs e iPSCs diferenciados devido à persistência de EGFP em toda a linhagemda célula 27. Esta linha iPSC pode ser usada in vivo para avaliar a distribuição do organismo de células derivadas de iPSC após o transplante. Como esta linha celular foi modificada por CRISPR e também está sendo usada para testar a aplicação clínica de iPSCs, é imperativo que os potenciais locais AAVS1 fora do alvo sejam conhecidos e interrogados para garantir segurança e eficácia, tornando-o um locus ideal para testar CIRCLE-seq.
Uma diferença notável entre Butterfield et al., estudo publicado anteriormente27 e este foi o uso de um gRNA modificado para atingir o locus AAVS1. Um gRNA pode ser projetado para melhorar a precisão da edição do genoma24. A sequência guia é o fator mais crítico que influencia as eficiências dentro e fora do alvo. Portanto, o gRNA escolhido foi testado em vários outros guias e apresentou fidelidade superior. Além disso, um artigo de métodos sobre a reprogramação de fibroblastos humanos comprovou a descoberta de que mRNAs sintéticos contendo nucleobases modificadas se beneficiam da baixa ativação de respostas antivirais29,30. Embora a baixa imunogenicidade possa não ser relevante em um ensaio in vitro, ela se torna crucial quando o objetivo final é desenvolver uma terapia clinicamente relevante que possa ser usada em células vivas.
O CIRCLE-seq tem muitas vantagens sobre métodos semelhantes. Por exemplo, as sequências Digenome-seq clivado e não clivado gDNA, usando ~ 400 milhões de leituras21. Isso resulta em um fundo alto, dificultando a filtragem de locais de corte de boa-fé de baixa frequência. O CIRCLE-seq usa apenas ~ 3-5 milhões de leituras devido ao enriquecimento do gDNA clivado por nuclease, resultando em baixo fundo. Além disso, o Digenome-seq e um método semelhante, o SITE-seq, dependem do sequenciamento de uma única extremidade de DNA clivada por nuclease. Em contraste, as leituras CIRCLE-seq incluem ambas as extremidades do local de corte, permitindo a identificação de locais fora do alvo sem a necessidade de uma referência 21,22,26.
Uma vantagem do CIRCLE-seq é sua maior sensibilidade em comparação com os métodos que dependem de cultura de células, como o GUIDE-seq. Quando os dois métodos foram comparados, o CIRCLE-seq foi capaz de capturar todos os locais fora do alvo detectados pelo GUIDE-seq e descobriu locais de clivagem não intencionais adicionais que o GUIDE-seq havia perdido. No entanto, uma diferença notável é que o GUIDE-seq pode ser prejudicado pelo cenário epigenético, enquanto o CIRCLE-seq pode acessar todo o genoma.
Como ensaio in vitro , o CIRCLE-seq apresenta várias limitações, a primeira das quais é a detecção de falsos positivos. Enquanto a epigenética impedirá a atividade da nuclease em certos locais in vivo, a ultrassonografia remove esses obstáculos in vitro, permitindo a atividade fora do alvo em locais que normalmente não são acessíveis em um contexto celular. Além disso, Cas9 está presente em altas concentrações neste ensaio in vitro , permitindo clivagem que de outra forma não seria possível in vivo. Este ensaio também requer uma quantidade relativamente grande de gDNA inicial, o que, dependendo dos recursos disponíveis, pode anular o uso deste protocolo. Por fim, é possível que alguns locais fora do alvo sejam indetectáveis devido às limitações das atuais tecnologias de sequenciamento de última geração.
Um estudo recente usou uma abordagem in silico cujo algoritmo identificou uma série de parâmetros relevantes para comparar diferentes métodos de caracterização de nucleases, incluindo CIRCLE-seq e GUIDE-seq31. Dois dos parâmetros relevantes foram 'enriquecimento no local de corte' e '% de falsos positivos'. Curiosamente, a taxa de falso-positivos do CIRCLE-seq foi calculada em 88%, mas seu enriquecimento no local de corte foi maior do que os outros métodos in vitro . Análises comparativas de todos os métodos revelaram que o GUIDE-seq foi o melhor desempenho, pois demonstrou a maior especificidade no alvo com apenas uma taxa moderada de falso-positivos32. Isso não invalida o CIRCLE-seq, mas sugere a possibilidade de utilizar o CIRCLE-seq e o GUIDE-seq em conjunto, validando as descobertas do CIRCLE-seq com o GUIDE-seq, uma vez que o primeiro tem maior sensibilidade, enquanto o último é um método baseado em células com alto enriquecimento no local de corte. Os dados também indicam que o sequenciamento de próxima geração (NGS) baseado em amplicon deve ser o método preferido para identificar modificações genuínas fora do alvo em locais candidatos a potencial31. Esses dados sugerem uma estratégia potencial de usar CIRCLE-seq, seguido por GUIDE-seq e, em seguida, NGS baseado em amplicon para examinar efeitos fora do alvo.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Estendendo a mais profunda gratidão pelo apoio financeiro fornecido pelos Institutos Nacionais de Saúde (R01AR078551 e T32AR007411), pela Associação de Pesquisa de Epidermólise Bolhosa Distrófica (DEBRA) Áustria, pelo Fundo Gates Grubstake e pelo Fundo Gates Frontiers.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
Referências
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185 (2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856 (2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786 (2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058 (2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157 (2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474 (2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745 (2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados