Method Article
CIRCLE-Seq zur Abfrage von Off-Target-Gen-Editing
In diesem Artikel
Zusammenfassung
Ein wesentliches Hindernis für Technologien wie CRISPR sind die Off-Target-Ereignisse, die lebenswichtige Gene stören können. "Circularization for In Vitro Reporting of Cleavage Effects by Sequencing" (CIRCLE-seq) ist eine Technik zur Identifizierung unbeabsichtigter Spaltstellen. Diese Methode bildet die genomweite Aktivität von CRISPR-Cas9 mit hoher Sensitivität und ohne Verzerrung ab.
Zusammenfassung
Circularization for In Vitro Reporting of Cleavage Effects by Sequencing (CIRCLE-seq) ist eine neuartige Technik, die für die unparteiische Identifizierung unbeabsichtigter Spaltstellen von CRISPR-Cas9 durch gezielte Sequenzierung von CRISPR-Cas9-gespaltener DNA entwickelt wurde. Das Protokoll beinhaltet die Zirkularisierung genomischer DNA (gDNA), die anschließend mit dem Cas9-Protein und einer Guide-RNA (gRNA) von Interesse behandelt wird. Nach der Behandlung wird die gespaltene DNA gereinigt und als Bibliothek für die Illumina-Sequenzierung vorbereitet. Der Sequenzierungsprozess generiert Paired-End-Reads, die umfassende Daten zu jeder Spaltstelle liefern. CIRCLE-seq bietet mehrere Vorteile gegenüber anderen In-vitro-Methoden , darunter minimale Anforderungen an die Sequenzierungstiefe, einen geringen Hintergrund und eine hohe Anreicherung für Cas9-gespaltene gDNA. Diese Vorteile erhöhen die Sensitivität bei der Identifizierung sowohl beabsichtigter als auch unbeabsichtigter Spaltungsereignisse. Diese Studie bietet ein umfassendes Schritt-für-Schritt-Verfahren zur Untersuchung der Off-Target-Aktivität von CRISPR-Cas9 mit Hilfe von CIRCLE-seq. Als Beispiel wird dieses Protokoll validiert, indem genomweite unbeabsichtigte Spaltstellen von CRISPR-Cas9 während der Modifikation des AAVS1-Locus kartiert werden. Der gesamte CIRCLE-seq-Prozess kann in zwei Wochen abgeschlossen werden, so dass genügend Zeit für das Zellwachstum, die DNA-Aufreinigung, die Vorbereitung der Bibliothek und die Illumina-Sequenzierung bleibt. Die Eingabe von Sequenzierungsdaten in die CIRCLE-seq-Pipeline ermöglicht eine optimierte Interpretation und Analyse von Spaltstellen.
Einleitung
In den letzten zwanzig Jahren hat das Genom-Engineering bedeutende Fortschritte gemacht, wobei ein wichtiger Meilenstein die Entdeckung von CRISPR-Cas9 (Clustered Regularly Interspaced Short Palindromic Repeats) im Jahr 2012war 1. Die CRISPR-Cas9-Technologie nutzt die programmierbare Natur bakterieller DNA-Endonukleasen und ermöglicht die präzise Ausrichtung und Modifikation nahezu jeder DNA-Sequenz. Seit seiner Einführung wurde das System so optimiert, dass es sich nur auf die Cas9-Endonuklease und eine Guide-RNA (gRNA) verlässt, um bestimmte genomische Regionen zu bearbeiten. Das Potenzial von CRISPR-Cas9 als kurative Therapie wurde in klinischen Studien unter anderem für verschiedene Erkrankungen wie Leber-Amaurose, Transthyretin-Amyloidose und Sichelzellenanämie nachgewiesen 2,3,4.
CRISPR-Cas9 induziert Doppelstrangbrüche (DSBs), die typischerweise durch einen von zwei Mechanismen aufgelöst werden: die fehleranfällige nicht-homologe Endverbindung (NHEJ) oder die präzisere homologiegerichtete Reparatur (HDR), sofern eine Template-DNA verfügbar ist. Die Tendenz von CRISPR-Cas9, NHEJ-assoziierte Insertionen und Deletionen (Indels) zu verursachen, zusammen mit Spaltungen an unbeabsichtigten genomischen Stellen, schränkt seine Anwendung im klinischen Umfeld ein 5,6,7,8,9,10. Darüber hinaus können unbeabsichtigte genomische Modifikationen kryptische Spleißstellen, Nonsense- oder Missense-Mutationen erzeugen, Chromothripsis induzieren oder onkogenes Potenzial für Zellen verleihen – Ergebnisse, die in mehreren Genom-Editing-Studien beobachtet wurden 11,12,13,14,15. Zusammenfassend lässt sich sagen, dass die genaue Identifizierung der Off-Target-Aktivität von CRISPR-Cas9 für seine klinischen Anwendungen von entscheidender Bedeutung ist, insbesondere in systemischen Gentherapien, die Milliarden von Zellen verändern können.
Verschiedene Methoden können eingesetzt werden, um CRISPR-Cas9-Off-Target-Spaltstellen zu identifizieren, einschließlich der genomweiten unverzerrten Identifizierung von Doppelstrangbrüchen (GUIDE)-seq 16, bei der doppelsträngige Oligodeoxynukleotide verwendet werden, um DSBs in lebenden Zellen zu markieren. Ein Kritikpunkt an dieser Methode ist jedoch, dass falsch positive Ergebnisse durch zufällige DSBs oder durch PCR-Artefakte entstehen können, die verworfen werden müssen, indem erfasste Stellen ausgeschlossen werden, die eine geringe Ähnlichkeit mit den On-Target-Stellen aufweisen. Die Methode, die auf der Verwendung von Integrase-defekten lentiviralen Vektoren (IDLV) basiert, ist weniger empfindlich und übersieht wahrscheinlich viele Off-Target-Stellen17. Andere in situ-Methoden wie DSBCapture, BLESS und BLISS 18,19,20 beziehen fixierte Zellen ein und markieren DSBs direkt, sind jedoch durch ihre Abhängigkeit von sofortigem DSB-Capture und das Fehlen exogener DNA eingeschränkt. Digenome-seq21, eine In-vitro-Methode, und Selective enrichment and Identification of Tagged genomic DNA Ends by sequencing (SITE-seq)22 bieten beide Sequenzierungslösungen, haben jedoch ihre Grenzen in Bezug auf Hintergrundrauschen bzw. Single-End-Analyse. Die Entdeckung von in situ Cas-Off-Targets und die Verifizierung durch Sequenzierung (Discover-Seq)23 bietet in vivo und in situ die Identifizierung der Cas9-Aktivität über MRE11-Bindung, weist jedoch nur DSBs nach, die zum Zeitpunkt der Probenvorbereitung vorhandensind 24. Schließlich verwendet Inference of CRISPR Edits (ICE) einen bioinformatischen Ansatz, um CRISPR-Bearbeitungen anhand von Sanger-Daten robust zu analysieren25.
Dieser Artikel beschreibt ein detailliertes Verfahren für die Zirkularisierung für die In-vitro-Berichterstattung über Spaltungseffekte durch Sequenzierung (CIRCLE-seq): eine in vitro-Technik, die die genomweite Off-Target-Aktivität der Cas9-Nuklease in einem Komplex mit der gRNA von Interesse sensitiv und unparteiisch abbildet26. Dieser Ansatz beginnt mit der Kultivierung der interessierenden Zellen und der Isolierung der DNA, gefolgt von der zufälligen Scherung durch gezielte Ultraschalluntersuchung und der anschließenden Behandlung mit Exonuklease und Ligase. Dieser Prozess erzeugt schließlich zirkuläre doppelsträngige DNA-Moleküle, die dann durch eine plasmidsichere DNase-Behandlung gereinigt werden. Diese zirkuläre DNA wird dann dem Cas9-gRNA-Komplex ausgesetzt, der sich sowohl an beabsichtigten als auch an unbeabsichtigten Spaltungsstellen spaltet und freiliegende DNA-Enden hinterlässt, die als Substrate für die Illumina-Adapterligation dienen. Dieser Prozess erzeugt eine vielfältige Bibliothek genomischer DNA (gDNA), die beide Enden jedes Nuklease-induzierten DSB enthält, wodurch sichergestellt wird, dass jeder Read alle für jede Spaltstelle erforderlichen Informationen enthält. Dies ermöglicht die Verwendung der Illumina-Sequenzierung mit geringeren Anforderungen an die Sequenzierungsabdeckung, wodurch sich CIRCLE-seq von anderen ähnlichen Methoden abhebt, die oben erwähnt wurden. Es ist wichtig zu beachten, dass CIRCLE-seq zwar eine höhere Off-Target-Sensitivität als andere Protokolle als In-vitro-Methode aufweist, dies jedoch auf Kosten höherer falsch-positiver Ergebnisse geht, da die epigenetische Landschaft, die bei anderen Methoden wie GUIDE-seq16 vorhanden ist, fehlt. Darüber hinaus sind die DSB-DNA-Reparatur und die damit verbundene Maschinerie in CIRCLE-seq nicht vorhanden, wodurch Indels oder eine ordnungsgemäße Reparatur, die sonst beobachtet worden wären, aufgehoben werden.
Neben der Beschreibung des Schritt-für-Schritt-Protokolls zur Durchführung von CIRCLE-seq wird das Protokoll durch die Identifizierung genomweiter unbeabsichtigter Spaltstellen von CRISPR-Cas9 validiert, die beispielsweise während der Modifikation des AAVS1-Locus auftreten. Dieses leicht verständliche Protokoll bietet detaillierte Anweisungen, von der Kultivierung induzierter pluripotenter Stammzellen (iPSCs) und der gDNA-Isolierung bis hin zur gDNA-Zirkularisierung, Cas9-gRNA-Spaltung, Bibliotheksvorbereitung, Sequenzierung und Pipeline-Analyse. Angesichts der geringen Anforderungen an die Sequenzierungsabdeckung steht CIRCLE-seq jedem Labor zur Verfügung, das Zugang zu Next-Generation-Sequencing hat.
Protokoll
Die Einzelheiten zu den Reagenzien, Verbrauchsmaterialien und Geräten, die für diese Studie verwendet wurden, sind in der Materialtabelle aufgeführt.
1. Zellkultur (5 Tage)
- Nehmen Sie eine Negativkontrolle in dieses Protokoll auf. Bereiten Sie genügend Zellen für weitere 25 μg gDNA (~2,0e, 7 Zellen pro Probe) vor.
- Kultivierung von iPSCs gemäß dem festgelegten Protokoll27. Sammeln Sie die Zellen und resuspendieren Sie sie in 10 ml PBS. Eine 6-μl-Zellprobe pipettieren und im Verhältnis 1:1 mit Trypanblau resuspendieren. Verwenden Sie einen automatisierten Zellzähler, um die Probe zu zählen.
- Aliquot 2 x 107 Zellen pro Röhrchen, dann 3 min lang bei 25 °C (Raumtemperatur (RT)) bei 300 x g herunterschleudern. Dies ist ausreichend für mehrere Replikate. Pipettieren Sie den Überstand ab und entsorgen Sie ihn.
2. Isolierung genomischer DNA (1 Tag)
- Verwenden Sie das im Handel erhältliche DNA-Aufreinigungskit, um die gDNA gemäß den Anweisungen des Herstellers zu isolieren:
- Geben Sie 200 μl PBS in ein konisches 15-ml-Röhrchen, das das Zellpellet enthält, und resuspendieren Sie es. Pipettieren Sie dann 3 ml Zelllysepuffer und 15 μl Proteinase K in das Röhrchen. Drehen Sie das Rohr 25 Mal um, um es gründlich zu mischen. Legen Sie das Röhrchen 3 Stunden lang oder über Nacht in einen Wasserbadschüttler, der auf 55 °C und 150 U/min eingestellt ist, um eine optimale DNA-Ausbeute zu erzielen.
- In 15 μl RNase A pipettieren. 25 Mal invertieren. 1 h in ein Wasserbad bei 37 °C stellen.
- Die Probe 5 Minuten lang auf Eis kalt stellen. Geben Sie dann 1 ml Proteinfällungslösung hinzu, wirbeln Sie 20 s lang bei hoher Geschwindigkeit vor und zentrifugieren Sie bei 2000 x g für 10 min bei RT. Die Proteine sollten ein sichtbares, kompaktes Pellet am Boden des Röhrchens bilden. Wenn das Pellet nicht sichtbar ist, inkubieren Sie die Probe weitere 5 Minuten auf Eis und zentrifugieren Sie erneut.
- Geben Sie 3 ml 100 % Isopropanol in ein neues konisches 15-ml-Röhrchen. Den Überstand aus Schritt 2.1.3 vorsichtig in das Röhrchen pipettieren. Drehen Sie das Röhrchen 50 Mal um, um es zu mischen, und zentrifugieren Sie es dann bei 2000 x g und Raumtemperatur für 3 min. Ohne das DNA-Kügelchen zu stören, saugen Sie den Überstand vorsichtig mit einer Pasteur-Pipette ab, die mit einer Vakuumfalle verbunden ist, und drehen Sie das Röhrchen mit einem sauberen, fusselfreien Tuch um.
- Pipettieren Sie 3 ml 70 % (v/v) Ethanol auf das DNA-Pellet und invertieren Sie es 10 Mal, um es zu waschen. Zentrifugieren Sie bei 2.000 x g für 3 min bei RT. Gießen Sie dann den Überstand vorsichtig ab.
- Halten Sie das Röhrchen offen und lassen Sie das resultierende DNA-Pellet 30 Minuten lang trocknen, um sicherzustellen, dass das gesamte Ethanol vollständig verdampft ist. 50 μl DNA-Hydratationslösung pipettieren und durch vorsichtiges Pipettieren gründlich mischen.
- Die DNA wird aufgelöst, indem die Probe 1 h lang bei 65 °C in einen Wasserbadschüttler gegeben wird, und lassen Sie die Probe dann über Nacht bei RT stehen. Zentrifugieren Sie die Probe bei RT und 2.000 x g für 1 min und verwenden Sie das dsDNA BR Assay Kit mit den zugehörigen Röhrchen, um die isolierte DNA mit dem Fluorometer zu quantifizieren.
3. Vorbereitung der gRNA (7 Tage)
- Bestellen Sie die gewünschte synthetische gRNA bei einer kommerziellen Quelle (siehe Materialtabelle). Dieses Protokoll ist auch mit crRNA/tracrRNA kompatibel.
4. gRNA-In-vitro-Spaltungstest
HINWEIS: Hier wird ein Ziel im AAVS1-Gen verwendet. Um auf andere interessante Gene abzuzielen, entwerfen Sie Primer (Tabelle 1), um die Zielregion zu amplifizieren, und ersetzen Sie die Primer in den folgenden Schritten durch benutzerdefinierte Primer.
- Bereiten Sie die PCR-Reaktion vor: Mischen Sie 25 μl Phusion Hot Start Flex 2x Master Mix (Endkonzentration 1x), 0,5 μl AAVS1 F Primer (Endkonzentration 0,1 μM), 0,5 μl AAVS1 R Primer (Endkonzentration 0,1 μM), 5 μl gDNA (100 ng, 20 ng/μl, aus Schritt 2.1.6) und 19 μl nukleasefreies H2O (Gesamtvolumen: 50 μL).
- Verwenden Sie die folgenden Thermocycler-Parameter: Denaturierung: 98 °C für 2 min (1 Zyklus), Denaturierung: 98 °C für 10 s (10 Zyklen), Glühen: 72-62 °C (-1 °C/Zyklus) für 15 s (10 Zyklen), Verlängerung: 72 °C für 30 s (10 Zyklen), Denaturierung, 98 °C für 10 s (30 Zyklen). Glühen: 65 °C für 15 s (30 Zyklen), Verlängerung: 72 °C für 30 s (30 Zyklen), Enddehnung: 72 °C für 5 min (1 Zyklus), Halten: 4 °C auf unbestimmte Zeit.
- Verwenden Sie AMPure XP-Kügelchen, um das Produkt der PCR-Reaktion aufzureinigen. Pipettieren Sie zunächst 1,8-fache Volumina oder 90 μl XP-Kügelchen in das PCR-Produkt. Zehnmal pipettieren, um es gründlich zu mischen. Lassen Sie die Mischung 5 Minuten lang bei RT inkubieren.
- Trennen Sie die Kügelchen mit einem Magnetgestell von der Lösung, indem Sie die PCR-Reaktionsplatte für 3 Minuten auf den Magneten legen. Die geklärte Lösung abpipettieren und verwerfen. Geben Sie 200 μl 80%iges Ethanol (v/v) zu den Kügelchen, inkubieren Sie 30 s lang und entfernen Sie dann das Ethanol. Wiederholen Sie diesen Waschschritt zweimal, um eine vollständige Entfernung des Ethanols zu gewährleisten.
- Lassen Sie die Proben 3 Minuten lang auf natürliche Weise trocknen, indem Sie die Platte auf den Magneten legen. Entfernen Sie die Platte vom Magneten und fügen Sie 40 μl TE-Puffer mit einem pH-Wert von 8,0 hinzu. Mischen Sie, indem Sie zehnmal auf und ab pipettieren. Lassen Sie die Probe 2 Minuten lang bei RT ruhen.
- Legen Sie die PCR-Reaktionsplatte für eine weitere Minute auf den Magneten. Nach 1 Minute den Überstand auf eine neue Platte umfüllen. Messen Sie die gereinigte PCR-Ausbeute mit einem Spektralphotometer und analysieren Sie sie auf einer TapeStation unter Verwendung eines optischen Tubenstreifenstreifens mit einer Kappe des optischen Tubenstreifens zusammen mit den Reagenzien High Sensitivity D1000 ScreenTape und High Sensitivity D1000 (Leiter und Puffer) gemäß den Anweisungen des Herstellers. Lagern Sie die vorbereitete Probe bis zu mehreren Monaten bei -20 °C.
- Cas9-Nukleaseprotein wie folgt auf 1 μM verdünnen: Mischen Sie 2 μl 10x Cas9-Puffer (Endkonzentration 1x), 1 μl Cas9-Nuklease, S. pyogenes (Endkonzentration 1 μM) und 17 μl nukleasefreies H2O in einem Gesamtvolumen von 20 μl.
- Führen Sie ein RNase-freies Verfahren durch, um den gRNA-Abbau zu verhindern. Die gRNA (aus Schritt 3.1) wird in H2O auf 3 μM auf ein Gesamtvolumen von 10 μl verdünnt.
HINWEIS: Verwenden Sie die folgende Formel, um das Molekulargewicht der gRNA zu schätzen: Molekulargewicht der ssRNA (g/mol) = (Länge der ssRNA (nt) x 321,47 g/mol) + 18,02 g/mol. Zum Vergleich: Eine 104 nt lange gRNA bei 3 μM entspricht etwa 100 ng/μL.
5. DNA-Schere (3 h)
- Bereiten Sie den ME220 vor, indem Sie zuerst den Querlenker referenzieren. Füllen Sie dann das Reservoir mit gereinigtem deionisiertem H2O. Greifen Sie auf dem Laptop der Kontrollstation auf Water Works zu und klicken Sie auf Füllen. Stellen Sie die Temperatur auf 4,5 °C ein.
- Übertragen Sie 25 μg gDNA in ein Mikroröhrchen (Mikroröhrchen-130 AFA Fiber Screw-Cap). Füllen Sie dann das Röhrchen bis zu einem Gesamtvolumen von 130 μL mit 1x TE. Verwenden Sie die folgenden Bedingungen, um die DNA auf eine durchschnittliche Länge von ca. 300 bp zu scheren: Setzen Sie die Dauer auf 10 s; Spitzenleistung auf 70; Einschaltfaktor % bis 20; Zyklen/Burst bis 50; All dies setzt die durchschnittliche Leistung automatisch auf 14,0.
6. Reinigung von gescherter genomischer DNA (1 h)
- Teilen Sie die gescherte genomische DNA in zwei Portionen zu je 65 μl. Reinigen Sie mit dem 1,8-fachen Volumen der XP-Kügelchen (117 μl) nach dem in den Schritten 4.3 bis 4.6 beschriebenen Verfahren. Übertragen Sie den Überstand auf eine neue PCR-Platte und messen Sie die Menge mit einem Spektralphotometer.
- Lassen Sie 1 μL der eluierten gescherten gDNA auf einer TapeStation laufen, gemäß den Anweisungen des Herstellers, um sicherzustellen, dass die gDNA auf eine breite Verteilung von ca. 300 bp geschert wird. Lagern Sie die gescherte gDNA bei Bedarf bis zu mehreren Monaten bei -20 °C.
7. Vorbereitung der CIRCLE-seq-Bibliothek (3 Tage)
- Glühen des Hairpin-Adapters
- Resuspendieren Sie oSQT1288 (Tabelle 1), den Hairpin-Adapter, auf eine Endkonzentration von 100 μM in 1x TE.
- Führen Sie das Adapterglühen wie folgt durch: Mischen Sie 40 μl oSQT1288 (Endkonzentration 40 μM), 10 μl 10x STE (Endkonzentration 1x) und 50 μl nukleasefreies H2O für ein Gesamtvolumen von 100 μl.
- Verwenden Sie die folgenden Glühparameter: 95 °C für 5 Minuten, -1 °C pro Minute für 70 Zyklen, auf unbestimmte Zeit bei 4 °C halten.
- Endreparatur durchführen . Verwenden Sie das PCR-freie HTP-Bibliotheksvorbereitungskit und bereiten Sie den End-Repair-Mastermix vor.
- Mischen Sie 8 μl Nuklease-freies H2O, 7 μl 10x Endreparaturpuffer (Endkonzentration 1x) und 5 μl Endreparatur-Enzymmischung (Endvolumen 20 μl Gesamtendreparatur-Mastermix).
- Pipettieren Sie 20 μl des Endreparatur-Mastermixes in die gescherte gDNA-Probe aus den Schritten 4.3 bis 4.6. Mischen Sie 20 μl End-Repair Master Mix mit 50 μl gescherter gDNA für ein Endvolumen von 70 μl.
- Die Mischung 30 Minuten lang bei 20 °C in einen Thermocycler geben und dann unbegrenzt bei 4 °C halten.
- Fügen Sie 1,7-fache Volumina oder 120 μl XP-Kügelchen hinzu und befolgen Sie die Aufreinigungsschritte in den Schritten 4.3 bis 4.6. Eluieren mit 42 μL TE, pH 8,0. Stellen Sie sicher, dass die Kügelchen für den nächsten Schritt in Lösung bleiben.
- Führen Sie ein A-tailing durch. Bereiten Sie mit dem HTP Library Preparation Kit (PCR-frei) den A-tailing Master Mix vor.
- Mischen Sie 5 μl 10x A-Tailing-Puffer (Endkonzentration 1x) und 3 μL A-Tailing-Enzym (Gesamtendvolumen von 8 μl A-Tailing-Mastermix).
- Pipettieren Sie 8 μl des A-Tailing-Mastermixes in jede DNA-Probe, die Kügelchen aus Schritt 7.2.4 enthält, und mischen Sie 8 μl A-Tailing-Mastermix mit 42 μl endreparierter DNA, die Kügelchen enthalten (Gesamtendvolumen von 50 μl). In einem Thermocycler bei 30 °C 30 min erhitzen. Auf unbestimmte Zeit bei 4 °C halten.
- Pipettieren Sie 1,8 Volumen oder 90 μl PEG/NaCl SPRI-Lösung (eine Komponente des HTP Library Preparation Kit (PCR-frei; 96 Reaktionen)) in die A-seitige DNA. Reinigen Sie die A-seitige DNA gemäß den Schritten 4.3-4.6. Eluieren Sie die A-seitige DNA in 30 μl TE, pH 8,0. Bewahren Sie die Kügelchen für den nächsten Schritt in Lösung auf.
- Führen Sie die Adapterligation durch. Bereiten Sie mit dem HTP Library Preparation (PCR-free) Kit den Adapter Ligation Master Mix vor.
- Mischen Sie 10 μl 5x Ligationspuffer (Endkonzentration 1x), 5 μl DNA-Ligase und 5 μl geglühten Hairpin-Adapter (40 μM) aus Schritt 7.1.3. Stellen Sie die Endkonzentration von 4 μM für einen Gesamtadapter-Ligatur-Mastermix von 20 μl sicher.
- Pipettieren Sie 20 μl Adapter Ligation Master Mix in jede eluierte DNA-Probe, die Beads aus Schritt 7.3.3 enthält (Gesamtendvolumen von 50 μl pro Probe).
- In einen Thermocycler bei 20 °C 1 h stellen. Auf unbestimmte Zeit bei 4 °C halten.
- 1x Volumen oder 50 μl PEG/NaCl SPRI-Lösung auf die adapterligierte DNA übertragen und gemäß den Schritten 4.3-4.6 aufreinigen. Eluieren Sie mit 30 μl TE, pH 8,0, und dekantieren Sie die Überstände in eine neue PCR-Platte mit halbem Rand. Kombinieren und quantifizieren Sie die DNA mit dem dsDNA BR-Assay. Lagern Sie bei Bedarf aufgereinigte adapterligierte DNA bis zu 1 Monat bei -20 °C.
- Bereiten Sie den Lambda Exonuclease/Exonuclease I (E. coli) Master Mix vor (funktioniert bei der Eliminierung von einzel- oder doppelsträngiger DNA ohne Adapter, die an beiden Enden ligiert sind).
- Nehmen Sie 1 μg Adapter Ligated DNA aus Schritt 7.4.4 und verdünnen Sie es auf 40 μl. Mischen Sie 5 μl 10x Exonuklease I Reaktionspuffer (Endkonzentration 1x), 4 μl Lambda Exonuklease (Endkonzentration 0,4 U/μl) und 1 μl Exonuklease I (E. coli) (Endkonzentration 0,4 U/μl) für ein Gesamtvolumen des Lambda Exonuklease/Exonuclease I Master Mix von 10 μl.
- Pipettieren Sie 10 μl des Lambda Exonuklease/Exonuclease I Master Mix auf 40 μl (1 μg) adapterligierte DNA (Gesamtvolumen von 50 μl). In einen Thermocycler 1 h bei 37 °C und dann 10 min bei 75 °C stellen. Auf unbestimmte Zeit bei 4 °C halten.
- Pipettieren Sie das 1,8-fache Volumen oder 90 μl XP-Kügelchen auf die mit Lambda Exonuklease/Exonuklease I behandelte DNA. Reinigen Sie gemäß den Anweisungen in den Schritten 4.3-4.6. Eluieren in 40 μl TE, pH 8,0. Stellen Sie sicher, dass die Kügelchen für den nächsten enzymatischen Schritt in Lösung bleiben.
- Behandeln Sie mit USER-Enzym und T4-Polynukleotidkinase (PNK). Bereiten Sie den USER/T4 PNK Master Mix vor (erforderlich, um die 4-bp-Überhänge zu lösen und die ligationsbereiten DNA-Enden vorzubereiten, die für die nachfolgende Ligationsreaktion erforderlich sind).
- Mischen Sie 5 μl 10x T4 DNA-Ligase-Puffer (Endkonzentration 1x), 3 μl USER-Enzym (Endkonzentration 0,05 U/μl) und 2 μl T4 PNK (Endkonzentration 0,4 U/μl), um ein Gesamtvolumen des USER/PNK-Mastermixes von 10 μl zu erhalten.
- Pipettieren Sie 10 μl des USER Enzyme/T4 PNK Master Mix auf 40 μl Lambda- und Exonuklease I-behandelte DNA-Probe, die Kügelchen aus Schritt 7.5.3 enthält, für ein Gesamtvolumen von 50 μl. In einen Thermocycler bei 37 °C 1 h stellen. Auf unbestimmte Zeit bei 4 °C halten.
- Pipettieren Sie ein 1,8-faches Volumen oder 90 μl PEG/NaCl SPRI-Lösung in die mit USER/T4 PNK behandelte DNA und reinigen Sie sie gemäß den Schritten 4.3-4.6. Eluieren in 35 μl TE, pH 8,0. Dekantieren Sie den Überstand in eine neue PCR-Platte mit halbem Rand. Kombinieren und quantifizieren Sie die DNA mit dem dsDNA HS-Assay.
- Führen Sie eine intramolekulare Zirkularisierung durch. Bereiten Sie den Zirkularisierungs-Mastermix vor.
- Mischen Sie 8 μl nukleasefreies H2O, 10 μl 10x T4-DNA-Ligase-Puffer (Endkonzentration 1x) und 2 μl T4-DNA-Ligase (Endkonzentration 8 U/μl), um ein Gesamtvolumen des Zirkularisierungs-Mastermixes von 20 μl zu erhalten.
- Pipettieren Sie 20 μl des Zirkularisierungs-Mastermixes auf 500 ng DNA, die mit USER/PNK aus Schritt 7.6.3 behandelt wurde. Verdünnen Sie 500 ng DNA, die mit USER/PNK behandelt wurde, in 80 μl und fügen Sie dann 20 μl des Zirkularisierungs-Mastermixes (Gesamtvolumen von 100 μl) hinzu. In einem Thermocycler bei 16 °C 16 h (über Nacht) inkubieren.
- Fügen Sie der zirkulierten DNA 1x Volumen oder 100 μl XP-Kügelchen hinzu und reinigen Sie sie gemäß den Schritten 4.3-4.6. Eluieren in 38 μl TE, pH 8,0. Dekantieren Sie den Überstand in eine neue PCR-Platte mit halbem Rand.
- Behandlung mit plasmidsicherer ATP-abhängiger DNase. Bereiten Sie den plasmidsicheren ATP-abhängigen DNase-Mastermix vor (erforderlich für den Abbau von verbleibender linearer DNA).
- Mischen Sie 5 μl 10x plasmidsicheren Reaktionspuffer (Endkonzentration 1x), 2 μl ATP (Endkonzentration 1 mM) und 5 μl plasmidsichere ATP-abhängige DNase (Endkonzentration 1 U/μl), um ein Gesamtvolumen des plasmidsicheren Mastermixes von 12 μl zu erhalten.
- Pipettieren Sie 12 μl des ATP-abhängigen DNase-Mastermixes auf 38 μl zirkularisierte DNA aus Schritt 7.7.3 (Gesamtvolumen von 50 μl). In einem Thermocycler 1 h bei 37 °C und dann 30 min lang bei 70 °C inkubieren. Auf unbestimmte Zeit bei 4 °C halten.
- Pipettieren Sie 1x Volumen oder 50 μl XP-Kügelchen in die DNA, die mit Plasmid-Safe, ATP-abhängiger DNase behandelt wurde, und reinigen Sie sie gemäß den Schritten 4.3-4.6. Eluieren in 15 μl TE, pH 8,0. Dekantieren Sie den Überstand in eine neue PCR-Platte mit halbem Rand.
- Kombinieren Sie die DNA und quantifizieren Sie sie mit dem dsDNA HS-Assay. Lagern Sie die zirkularisierte DNA bei Bedarf bis zu mehreren Monaten bei -20 °C.
8. Spaltung enzymatisch gereinigter, zirkularisierter gDNA in vitro (2 h)
- Führen Sie eine In-vitro-Spaltung mit dem Cas9:gRNA-Komplex durch. Bereiten Sie den in vitro Cleavage Master Mix vor. Mischen Sie 5 μl 10x Cas9-Puffer (Endkonzentration 1x), 4,5 μl S. pyogenes Cas9 (Endkonzentration 90 nM) und 1,5 μl gRNA (Endkonzentration 90 nM) für ein Gesamtvolumen des Spaltungs-Master-Mixes von 11 μl.
- Halten Sie den Spaltungs-Mastermix 10 Minuten lang bei RT, um Cas9:gRNA-RNP-Komplexe zu bilden.
- 125 ng plasmidsicherer DNase-behandelter DNA aus Schritt 7.8.3 auf ein Endvolumen von 39 μl verdünnen. Fügen Sie dann 11 μl des Spaltungs-Mastermixes zu 39 μl plasmidsicherer DNase-behandelter DNA hinzu, um ein Gesamtvolumen von 50 μl zu erhalten.
HINWEIS: Nehmen Sie in diesen Schritt eine Negativkontrollprobe auf, die aus zirkularisierter DNA besteht, die mit Cas9-Puffer gemischt ist, ohne den Cas9:gRNA-Komplex. - In einem Thermocycler 1 h bei 37 °C inkubieren. Auf unbestimmte Zeit bei 4 °C halten. Geben Sie 50 μl (1x Volumen) XP-Kügelchen zu der in vitro gespaltenen DNA und reinigen Sie die DNA gemäß den Schritten 4.3-4.6. Eluieren Sie in 42 μl TE-Puffer, pH 8,0. Stellen Sie sicher, dass die Kügelchen für den nächsten Schritt in Lösung bleiben.
9. Vorbereitung der Next-Generation-Sequencing-Bibliothek (4 - 6 h)
- Führen Sie ein A-tailing durch. Bereiten Sie den A-tailing Master Mix vor.
- Mischen Sie 5 μl 10x A-Tailing-Puffer (Endkonzentration 1x) und 3 μl A-Tailing-Enzym (Gesamtvolumen des A-Tailing-Mastermixes beträgt 8 μl).
- Pipettieren Sie 8 μl des A-Tailing Master Mix auf 42 μl eluierte DNA-Probe, die Kügelchen aus Schritt 8.4 enthält (Gesamtvolumen von 50 μl). 30 min bei 30 °C in einen Thermocycler geben. Auf unbestimmte Zeit bei 4 °C halten.
- Pipettieren Sie 1,8-fache Volumina der PEG/NaCl SPRI-Lösung oder 90 μl in die A-seitige DNA und reinigen Sie die DNA gemäß den Schritten 4.3-4.6. Eluieren in 25 μl TE, pH 8,0. Achten Sie darauf, dass die Kügelchen für den folgenden Schritt in Lösung bleiben.
- Führen Sie die Adapterligation durch. Bereiten Sie den Adapter Ligation Master Mix vor.
HINWEIS: Einmal-Aliquots von NEB-Adaptern müssen vorbereitet werden, um die Bildung von Adapterdimeren zu verhindern, die durch die Frost-Tau-induzierte Hydrolyse des 3' T' verursacht werden.- Mischen Sie 10 μl 5x Ligationspuffer (Endkonzentration 1x), 5 μl DNA-Ligase und 10 μl Adapter für die Sequenzierung (Endkonzentration beträgt 3 μM), für insgesamt 25 μl.
- Pipettieren Sie 25 μl des Adapter-Ligations-Mastermixes in eine 25 μl A-seitige DNA-Probe, die Kügelchen aus Schritt 9.1.3 enthält. 1 h bei 20 °C in einen Thermocycler stellen. Auf unbestimmte Zeit bei 4 °C halten.
- Pipettieren Sie 1x Volumen oder 50 μl PEG/NaCl SPRI-Lösung auf die adapterligierte DNA und reinigen Sie die DNA gemäß den Schritten 4.3-4.6. Eluieren in 47 μl TE, pH 8,0. Stellen Sie sicher, dass die Kügelchen für den nächsten enzymatischen Schritt in Lösung bleiben.
- Führen Sie die Behandlung mit dem USER-Enzym durch (erzeugt eine Einzelnukleotidlücke an Uracilresten).
- Geben Sie 3 μl USER-Enzym (im Dual Index Primers Kit enthalten) in die adapterligierte DNA-Probe, die die Kügelchen aus Schritt 9.2.3 enthält. Bei 37 °C 15 min inkubieren.
- Geben Sie 35 μl (0,7x Volumen) PEG/NaCl SPRI-Lösung in die mit dem USER-Enzym behandelte DNA und reinigen Sie sie gemäß den Schritten 4.3-4.6. Eluieren Sie in 20 μl TE-Puffer, pH 8,0. Übertragen Sie den Überstand auf eine neue PCR-Platte mit halbem Rand und messen Sie die DNA-Konzentration mit dem dsDNA HS-Assay. Die erwartete Konzentration sollte ca. 2-5 ng/μL betragen.
- (Fakultativ) Bevor mit dem nächsten Schritt fortgefahren wird, kann die Auswahl der DNA-Größe mit PippinHT durchgeführt werden. Verwenden Sie die 1,5 % PippinHT-Kassette mit einem Größenbereich von 250-850 bp. Die anfallenden Proben können im nächsten Schritt direkt in der PCR verwertet werden.
- PCR für das Hinzufügen von Barcodes durchführen
HINWEIS: Stellen Sie sicher, dass die für jede Probe gewählten Primersequenzkombinationen eindeutig sind. Wenn möglich, sollte jede Probe mit eindeutigen i5- und i7-Barcodes versehen sein.- Bereiten Sie den PCR-Master-Mix vor, um Barcodes mit gepaartem Index hinzuzufügen. Mischen Sie 5 μl nukleasefreies H2O, 25 μl 2x HotStart Ready Mix (Endkonzentration 1x), 5 μl i5 Primer (Endkonzentration 1 μM) und 5 μl i7 Primer (Endkonzentration 1 μM) (Gesamtvolumen des Mastermixes von 40 μl).
- Pipettieren Sie 40 μl des PCR-Mastermixes auf 10 μl gereinigte DNA, die mit dem USER-Enzym (ca. 20 ng) aus Schritt 9.3.2 behandelt wurde (Gesamtvolumen von 50 μl).
- Wählen Sie die folgenden PCR-Thermocycling-Bedingungen: Denaturierung: 98 °C für 45 s für 1 Zyklus, Denaturierung: 98 °C für 15 s für 20 Zyklen, Glühen: 65 °C für 30 s für 20 Zyklen, Verlängerung: 72 °C für 30 s für 20 Zyklen, Endverlängerung: 72 °C für 1 min für 1 Zyklus, Halten: 4 °C auf unbestimmte Zeit.
- Geben Sie 0,7-fache Volumina oder 35 μl XP-Beads zum PCR-Produkt und reinigen Sie es gemäß den Schritten 4.3-4.6. Eluieren Sie in 30 μl TE, pH 8,0. Dekantieren Sie den Überstand in eine neue PCR-Platte mit halbem Rand. Lagern Sie die zirkuläre DNA bei Bedarf bis zu mehreren Monaten bei -20 °C.
HINWEIS: Führen Sie eine PCR-Probe auf der Tapestation durch, um die Qualität der Bibliothek zu kontrollieren und die Bildung von Adapterdimeren zu beurteilen. Wenn Adapter-Dimere erkannt werden, wiederholen Sie Schritt 9.5.4.
10. Quantifizierung von CIRCLE-seq-Bibliotheken mittels digitaler Tröpfchen-PCR (dd_PCR) (6 h)
HINWEIS: Die Quantifizierung kann auch mit qPCR, Tapestation oder einer ähnlichen Methode durchgeführt werden.
- Beginnen Sie mit 5 μl DNA aus der Bibliothek (PCR-Schritt 9.5.4), die gut mit 45 μl nukleasefreier TE gemischt wurden, und führen Sie dann 1:10 serielle Verdünnungen jeder Probe in 50 μL-Volumina durch, die von 10-1 bis 10-8 Verdünnung reichen.
- Richten Sie die dd_PCR Master Mix-Lagerlösung ein. Mischen Sie 11 μl 2x dd_PCR Mischung für Sonden (Endkonzentration 1x), 0,055 μl Sonde oSQT1310 (Endkonzentration 250 nM), 0,055 μl Sonde oSQT1311 (Endkonzentration 250 nM), 0,099 μl Primer oSQT1274 (Endkonzentration 450 nM), 0,099 μl Primer oSQT1275 (Endkonzentration 450 nM) und 6,292 μl nukleasefreies H2O, für insgesamt dd_PCR Master-Mix-Volumen von 17,6 μl. Bereiten Sie für alle Proben einen Mastermix vor, um sicherzustellen, dass die Volumina für ein genaues Pipettieren ausreichen.
- Untersuchen Sie die drei niedrigsten Verdünnungen (10-6, 10-7 und 10-8) in doppelter Ausführung (in einer 96-Well-Platte). Es muss ein Non-Template-Control (NTC) verwendet werden.
- Pipettieren Sie 17,6 μl dd_PCR Master Mix wie folgt in jede Probe: Mischen Sie 17,6 μl mit 4,4 μl der Probe (fügen Sie nukleasefreies H2O in die Vertiefung mit NTC hinzu), um ein Gesamtvolumen von 22 μl zu erhalten. Verschließen Sie die Platte, zentrifugieren Sie dann bei 2000 x g für 1 min bei RT.
- Führen Sie die Tröpfchengenerierung, das Thermocycling und die Analyse durch. Übertragen Sie mit einem Droplet Reader PCR-System eine DG8-Kartusche (8-Well) in den Kartuschenhalter. Geben Sie in der Ölreihe der Kartusche 70 μl Öl zur Tröpfchenerzeugung für Sonden ab.
HINWEIS: Entnehmen Sie 20 μl der Probe aus Schritt 10.1.2. und fügen Sie es der Probenreihe der 8-Well-Kartusche hinzu, bedecken Sie die Kartusche mit der DG8-Gummidichtung, setzen Sie sie in den Tröpfchengenerator ein und schließen Sie ihn dann, um den Vorgang zu starten (automatisch). Wenn Sie fertig sind, entfernen Sie die Kartusche und geben Sie 40 μl aus der Tröpfchenreihe der 8-Well-Kartusche in eine 96-Well-PCR-Platte mit halbem Rand, wobei Sie darauf achten, dass das Pipettieren langsam erfolgt.- Legen Sie den Heizblock in den PCR-Plattenversiegeler PX1. Das Versiegelungsgerät beginnt sich beim Einschalten auf bis zu 180 °C aufzuheizen. Legen Sie die Folienversiegelung auf die Platte und stellen Sie sicher, dass die rote Linie oben liegt. Setzen Sie die Platte in den PX1 ein und drücken Sie auf Siegel.
- Wählen Sie die folgenden Thermocycler-Bedingungen: Enzymaktivierung: 95 °C für 10 min für 1 Zyklus, Denaturierung: 94 °C für 30 s für 40 Zyklen, Glühen/Verlängern: 60 °C für 1 min für 40 Zyklen, Enzymdeaktivierung: 98 °C für 10 min für 1 Zyklus, Halten: 4 °C auf unbestimmte Zeit.
- Öffnen Sie auf dem Droplet-Reader die kompatible Software und wählen Sie aus, welche Wells ausgelesen werden sollen. Wählen Sie ABS als Versuchstyp und dd_PCR Supermix für Sonden aus. Wählen Sie Ch1 Unknown für Target 1 und Ch2 Unknown für Target 2 aus. Wählen Sie Anwenden und dann OK aus. Legen Sie die Platte in den Tröpfchenleser. Wählen Sie für den Farbstoffsatz FAM/HEX und klicken Sie auf Ausführen.
- Analysieren Sie die dd_PCR Ergebnisse. Gate der doppelt positiven Tröpfchenpopulation mit der negativen Kontrolle als Referenz. Berechnen Sie den Durchschnitt der doppelten Werte und multiplizieren Sie ihn mit dem Verdünnungsfaktor und dem 5-fachen Verdünnungsfaktor der dd_PCR.
HINWEIS: Die Gesamtzahl der Kopien pro Mikroliter wird wie folgt berechnet: Gesamtzahl der Kopien pro Mikroliter = Mittelwert × 5 × Verdünnungsfaktor, wobei der "Mittelwert" den durchschnittlichen Bestimmungswert von Ch1 und Ch2 darstellt. - Kombinieren Sie alle Proben in einer einzigen Bibliothek bei äquimolaren Konzentrationen. Die 1x gepoolte Bibliothek sollte ca. 4,5 x 109 Moleküle enthalten und ein Gesamtvolumen von 5 μL haben.
11. Sequenzierung der nächsten Generation
- Senden Sie die Proben zur Sequenzierung an eine externe Agentur und stellen Sie sicher, dass die richtigen Adaptersequenzen notiert sind.
12. CIRCLE-seq Datenanalyse (1 - 3 h)
- Installieren Sie Python Version 2.7, Burrows-Wheeler Aligner (BWA) und SAMtools. Laden Sie das Referenzgenom (z. B. hg38) von http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz herunter.
HINWEIS: Wenn das Genom der Zielspezies nicht verfügbar ist, kann der CIRCLE-seq-Berechnungsworkflow in einem referenzunabhängigen Modus ausgeführt werden. In diesem Fall kann dieser Schritt übersprungen werden. - Laden Sie die CIRCLE-seq-Pipeline mit den folgenden Befehlen herunter, und installieren Sie sie: (1) git clone https://github.com/tsailabSJ/circleseq.git, (2) cd circleseq, (3) pip install -r requirements.txt.
- Erstellen Sie eine Manifestdatei im YAML-Format (.yaml). Nachfolgend finden Sie ein Beispielmanifest, das mit dem in der CIRCLE-seq-Software bereitgestellten Beispieldatensatz verwendet werden kann, um den Workflow zu testen.
HINWEIS: (1) Referenzgenom: data/input/CIRCLEseq_test_genome.fa; (2) analysis_folder: Daten/Ausgabe; (3) bwa: bwa; (4) samtools: samtools; (5) read_threshold: ; (6) window_size: ; (7) mapq_threshold: ; (8) start_threshold: ; (9) gap_threshold: ; (10) mismatch_threshold: ; (11) merged_analysis: Wahr; (12) Proben: U2OS_EMX1; (13) Ziel: GAGTCCGAGCAGAAGAAGAANGG; (14) Lesen1: Daten/Eingabe/EMX1.r1.fastq.gz; (15) read2: Daten/Eingabe/EMX1.r2.fastq.gz; (16) controlread1: Daten/Eingabe/EMX1_control.r1.fastq.gz; (17) controlread2: Daten/Eingabe/EMX1_control.r2.fastq.gz; (18) Beschreibung: U2OS. Die folgenden Manifestwerte wurden verwendet: read_threshold: 4, window_size: 3, mapq_threshold: 50, start_threshold: 1, gap_threshold: 3, mismatch_threshold: 6 - Definieren Sie die FASTA-Datei des Referenzgenoms, das Ausgabeverzeichnis für die Analyse und die Pfade zu den Befehlen BWA und SAMtools . Definieren Sie die Zielsequenzen und die Pfade zu den demultiplexierten FASTQ-Dateien sowohl für die Nuklease-gespaltenen als auch für die Kontrollproben. Mehrere Experimente können gleichzeitig im Batchmodus verarbeitet werden, indem alle in einer einzigen Manifestdatei eingeschlossen werden.
- Führen Sie den folgenden Befehl für standardmäßige referenzbasierte Analysen aus: (1) python /Pfad/zum/circleseq.py alles - Manifest; (2) /Pfad/zum/Manifest.yaml.
- Alternativ können Sie den folgenden Befehl für standardmäßige, nicht referenzbasierte Analysen ausführen: (1) Python /Pfad/zum/circleseq.py referenzfrei - Manifest; (2) /Pfad/zum/manifest.yaml.
- Wenn Sie die vollständige Pipeline ausführen, suchen Sie die Ausgabeergebnisse jedes Schritts in einem eindeutigen output_folder , der für diesen bestimmten Schritt vorgesehen ist.
Ergebnisse
Hier wird CIRCLE-seq verwendet, um die Nuklease-induzierten Spaltstellen von Cas9 in einem Komplex mit der gRNA zu untersuchen, der auf die Adeno-assoziierte Virusintegrationsstelle 1 (AAVS1) abzielt, wobei DNA verwendet wird, die aus induzierten pluripotenten Stammzellen (iPSCs) isoliert wurde. Diese gRNA wurde bereits in unserer Publikationbeschrieben 27. Etwa 25 μg gDNA wurden aus iPS-Zellen isoliert, durch fokussierte Ultraschalluntersuchung geschert und die Größe mittels AMPure XP Bead-Aufreinigung ausgewählt, um Fragmente von ca. 300 bps zu erhalten. Von diesen 25 μg DNA wurden ca. 2-5 ng DNA für die in vitro Cas9:gRNA-Spaltung erfolgreich zirkuliert. Die gesamte Vorgehensweise ist in Abbildung 1 dargestellt.
Nach einem CIRCLE-seq-Verfahren und einer Analyse unter Verwendung unseres Berechnungsworkflows ist in Abbildung 2A eine Visualisierung aller detektierten On- und Off-Target-Spaltstellen dargestellt. Die CIRCLE-seq-Pipeline lieferte auch "Merged Reads", die mit der statistischen Software R analysiert wurden, um ein Manhattan-Diagramm zu erhalten, das die detektierten Nuklease-induzierten Spaltstellen entlang jedes Chromosoms zeigt (Abbildung 2B).
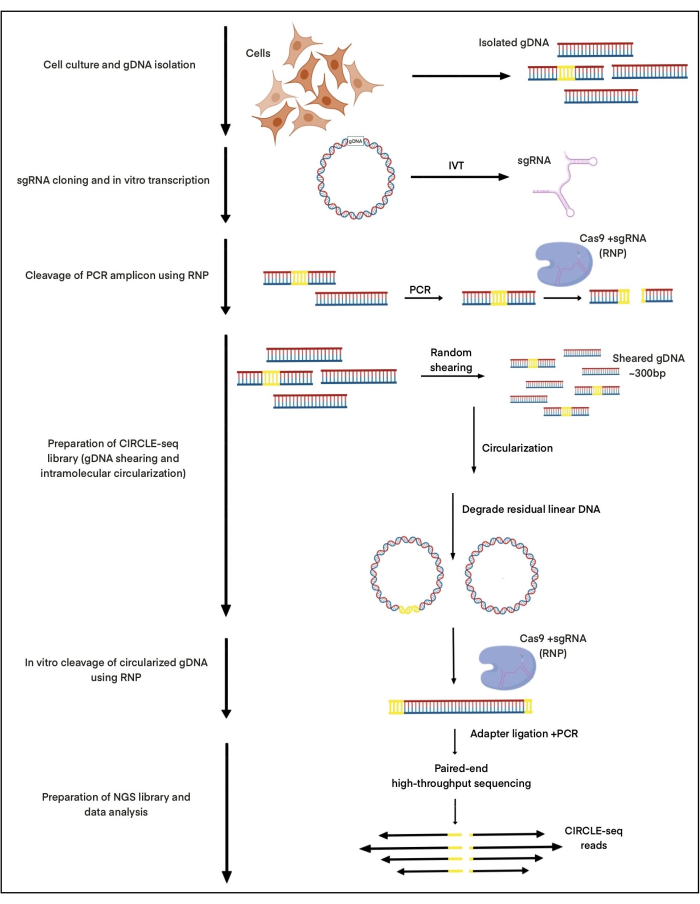
Abbildung 1: Schaltpläne des CIRCLE-seq-Workflows. Die wichtigsten Schritte des Protokolls sind angegeben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
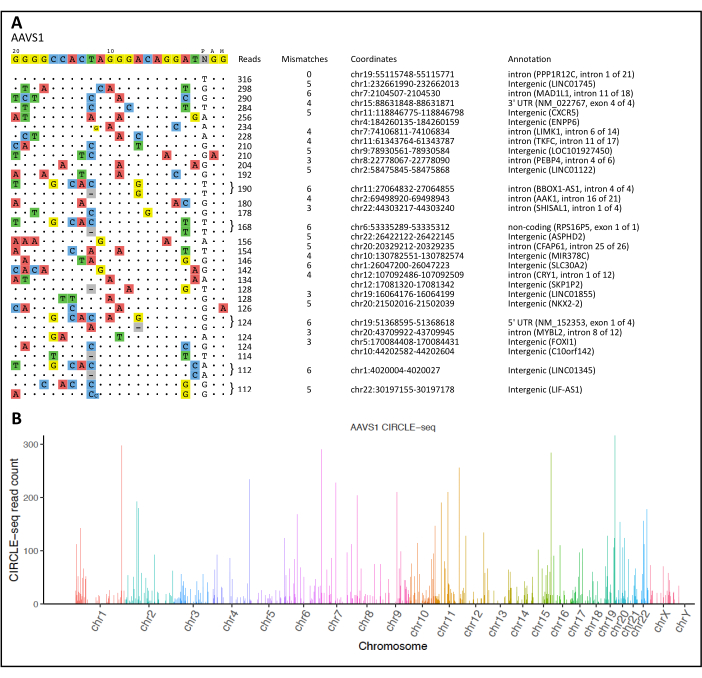
Abbildung 2: CIRCLE-seq-Visualisierung und Manhattan-Plot. (A) Ausrichtung von Off-Target-Stellen gegen das beabsichtigte Ziel für den AAVS1-Locus. Die Zielsequenz wird oben angezeigt, wobei die Off-Targets nach der Anzahl der Lesevorgänge in absteigender Reihenfolge geordnet sind. Unterschiede in den ursprünglichen Zielsequenzen werden durch farbige Nukleotide dargestellt. Eine Stichprobe der wichtigsten unbeabsichtigten Spaltstellen für den AAVS1-Locus ist dargestellt. (B) Manhattan-Diagramm, das die entdeckten unbeabsichtigten Spaltstellen für den AAVS1-Locus veranschaulicht. Die Balkenhöhen stellen die Leseanzahl für jede Chromosomenposition dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Fibel | Sequenz (5'-3') | Kommentare/Beschreibung |
| AAVS1 Einzelne Guide-RNA (sgRNA) | GGGGCCACUAGGGACAGGAU | Für den fluoreszierenden Protein-Knock-in des AAVS1-Locus |
| AAVS1 Forward Primer | GCTCTGGGCGGAGGAATAT | Für den gRNA-In-vitro-Spaltungstest |
| AAVS1 Reverse Primer | ATTCCCAGGGCCGGTTAATG | Für den gRNA-In-vitro-Spaltungstest |
| oSQT1288 | /5Phos/CGGTGGACCGATGATC /ideoxyU/ATCGGTCCACCGaT | CIRCLE-seq Haarnadel-Adapter |
| oSQT1274 | AATGATACGGCGACCACCGAG | TruSeq F1 |
| oSQT1275 | CAAGCAGAAGACGGCATACGAGAT | TruSeqF2 |
| oSQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | TruSeq-Sonde |
| oSQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | TruSeq-Sonde |
Tabelle 1: Sequenzen von gRNA und Primern, die für die CIRCLE-seq-Analyse des AAVS1-Locus verwendet wurden.
Diskussion
Hier wird gezeigt, dass CIRCLE-seq eine unverzerrte und hochempfindliche Technik zur Identifizierung von Nuklease-induzierten DSBs im gesamten Genom ist, die sich aus der Ausrichtung auf den AAVS1-Locus in gDNA ergeben, die aus iPSCs gewonnen wird. Die AAVS1-Stelle in iPSCs ist als Safe-Harbor-Locus bekannt, der häufig als Integrationsstelle für exogene Gene unter Verwendung von CRISPR-Cas9 verwendet wird28. Unser aktueller Bericht untersuchte das Potenzial von EGFP-markierten iPSCs durch die CRISPR-vermittelte Integration eines konstitutiv exprimierten EGFP-Reporters in die AAVS1-Stelle , was die Markierung und Verfolgung sowohl von iPSCs als auch von differenzierten iPSCs aufgrund der Persistenz von EGFP in der gesamten Zelllinie ermöglicht27. Diese iPSC-Linie kann in vivo verwendet werden, um die organismische Verteilung von iPSC-abgeleiteten Zellen nach der Transplantation zu bewerten. Da diese Zelllinie CRISPR-modifiziert wurde und auch zur Erprobung der klinischen Anwendung von iPSCs verwendet wird, ist es zwingend erforderlich, dass die potenziellen AAVS1-Off-Target-Stellen bekannt sind und abgefragt werden, um Sicherheit und Wirksamkeit zu gewährleisten, was sie zu einem idealen Ort für den Test von CIRCLE-seq macht.
Ein bemerkenswerter Unterschied zwischen Butterfield et al., einer zuvor veröffentlichten Studie27 und dieser, war die Verwendung einer modifizierten gRNA, die auf den AAVS1-Locus abzielte. Eine gRNA kann so gestaltet werden, dass sie die Genauigkeit der Genomeditierung verbessert24. Die Leitsequenz ist der kritischste Faktor, der die On- und Off-Target-Effizienz beeinflusst. Daher wurde die ausgewählte gRNA gegen mehrere andere Leitfäden getestet und für überlegen befunden. Darüber hinaus untermauerte ein Methodenartikel über die Reprogrammierung humaner Fibroblasten die Erkenntnis, dass synthetische gekapselte mRNAs, die modifizierte Nukleobasen enthalten, von einer geringen Aktivierung antiviraler Antworten profitieren29,30. Während eine geringe Immunogenität in einem In-vitro-Assay möglicherweise nicht relevant ist, wird sie entscheidend, wenn das ultimative Ziel darin besteht, eine klinisch relevante Therapie zu entwickeln, die in lebenden Zellen eingesetzt werden kann.
CIRCLE-seq hat viele Vorteile gegenüber ähnlichen Methoden. Zum Beispiel sequenziert Digenome-seq sowohl nuklease-gespaltene als auch ungespaltene gDNA unter Verwendung von ~400 Millionen Reads21. Dies führt zu einem hohen Hintergrund, der es schwierig macht, niederfrequente Bona-Fide-Schnittstellen herauszufiltern. CIRCLE-seq verwendet aufgrund der Anreicherung von Nuklease-gespaltener gDNA nur ~3-5 Millionen Reads, was zu einem niedrigen Hintergrund führt. Darüber hinaus beruhen Digenome-seq und eine ähnliche Methode, SITE-seq, auf der Sequenzierung eines einzelnen Nuklease-gespaltenen DNA-Endes. Im Gegensatz dazu schließen CIRCLE-seq-Lesevorgänge beide Enden der Schnittstelle ein, was die Identifizierung von Off-Target-Stellen ermöglicht, ohne dass eine Referenz 21,22,26 erforderlich ist.
Ein Vorteil von CIRCLE-seq ist die höhere Sensitivität im Vergleich zu Methoden, die auf Zellkulturen basieren, wie z. B. GUIDE-seq. Beim Vergleich der beiden Methoden war CIRCLE-seq in der Lage, alle von GUIDE-seq erkannten Off-Target-Stellen zu erfassen und zusätzliche unbeabsichtigte Spaltstellen aufzudecken, die GUIDE-seq übersehen hatte. Ein bemerkenswerter Unterschied besteht jedoch darin, dass GUIDE-seq durch die epigenetische Landschaft behindert werden kann, während CIRCLE-seq auf das gesamte Genom zugreifen kann.
Als In-vitro-Assay weist CIRCLE-seq mehrere Einschränkungen auf, von denen die erste der Nachweis von falsch positiven Ergebnissen ist. Während die Epigenetik die Nukleaseaktivität an bestimmten Stellen in vivo behindert, beseitigt Ultraschall diese Hindernisse in vitro und ermöglicht eine Off-Target-Aktivität an Stellen, die normalerweise in einem zellulären Kontext nicht zugänglich sind. Darüber hinaus ist Cas9 in diesem in vitro Assay in hohen Konzentrationen vorhanden, was eine Spaltung ermöglicht, die sonst in vivo nicht möglich wäre. Dieser Assay erfordert auch eine relativ große Menge an Start-gDNA, was je nach verfügbaren Ressourcen die Verwendung dieses Protokolls zunichte machen kann. Schließlich ist es möglich, dass einige Off-Target-Stellen aufgrund der Einschränkungen der aktuellen Next-Gen-Sequencing-Technologien nicht nachweisbar sind.
In einer kürzlich durchgeführten Studie wurde ein In-silico-Ansatz verwendet, dessen Algorithmus eine Reihe relevanter Parameter identifizierte, mit denen verschiedene Nuklease-Charakterisierungsmethoden, einschließlich CIRCLE-seq und GUIDE-seq31, verglichen werden konnten. Zwei der relevanten Parameter waren "Anreicherung an der Schnittstelle" und "% falsch positive Ergebnisse". Interessanterweise wurde die Falsch-Positiv-Rate von CIRCLE-seq mit 88 % berechnet, aber die Anreicherung an der Schnittstelle war um ein Vielfaches höher als bei den anderen In-vitro-Methoden . Vergleichende Analysen aller Methoden zeigten, dass GUIDE-seq die beste Leistung erbrachte, da es mit nur einer moderaten Falsch-Positiv-Rate die größte On-Target-Spezifität aufwies32. Dies macht CIRCLE-seq nicht ungültig, sondern deutet vielmehr auf die Möglichkeit hin, CIRCLE-seq und GUIDE-seq zusammen zu verwenden, was die Ergebnisse von CIRCLE-seq mit GUIDE-seq validiert, da ersteres eine höhere Sensitivität aufweist, während letzteres eine zellbasierte Methode mit hoher Anreicherung an der Schnittstelle ist. Die Daten deuten auch darauf hin, dass die Amplicon-basierte Next-Generation-Sequencing (NGS) die bevorzugte Methode zur Identifizierung echter Off-Target-Modifikationen an potenziellen Kandidatenstellen sein sollte31. Diese Daten deuten auf eine mögliche Strategie hin, bei der CIRCLE-seq, gefolgt von GUIDE-seq und dann amplikonbasiertem NGS verwendet wird, um Off-Target-Effekte zu untersuchen.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Wir danken den National Institutes of Health (R01AR078551 und T32AR007411), der Dystrophic Epidermolysis Bullosa Research Association (DEBRA) Österreich, dem Gates Grubstake Fund und dem Gates Frontiers Fund für die finanzielle Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
Referenzen
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185(2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856(2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786(2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058(2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157(2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474(2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745(2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten