Method Article
Hedef Dışı Gen Düzenlemenin Sorgulanması için CIRCLE-Seq
Bu Makalede
Özet
CRISPR gibi teknolojilerin önündeki önemli bir engel, hayati genleri bozabilecek hedef dışı olaylardır. 'Dizileme ile Bölünme Etkilerinin İn Vitro Raporlaması için Döngüselleştirme' (CIRCLE-seq), istenmeyen bölünme bölgelerini belirlemek için tasarlanmış bir tekniktir. Bu yöntem, CRISPR-Cas9'un genom çapındaki aktivitesini yüksek hassasiyetle ve önyargısız olarak haritalandırır.
Özet
Dizileme ile Bölünme Etkilerinin İn Vitro Raporlaması için Döngüselleştirme (CIRCLE-seq), CRISPR-Cas9 parçalanmış DNA'nın hedeflenen dizilimi yoluyla CRISPR-Cas9'un istenmeyen bölünme bölgelerinin tarafsız bir şekilde tanımlanması için geliştirilmiş yeni bir tekniktir. Protokol, daha sonra Cas9 proteini ve ilgilenilen bir kılavuz RNA (gRNA) ile muamele edilen genomik DNA'nın (gDNA) dairesel hale getirilmesini içerir. Tedaviyi takiben, parçalanmış DNA saflaştırılır ve Illumina dizilimi için bir kütüphane olarak hazırlanır. Sıralama işlemi, her bölünme bölgesi hakkında kapsamlı veriler sunan eşleştirilmiş uç okumalar oluşturur. CIRCLE-seq, minimum dizileme derinliği gereksinimleri, düşük arka plan ve Cas9 parçalı gDNA için yüksek zenginleştirme dahil olmak üzere diğer in vitro yöntemlere göre çeşitli avantajlar sağlar. Bu avantajlar, hem amaçlanan hem de istenmeyen bölünme olaylarını belirlemede hassasiyeti artırır. Bu çalışma, CIRCLE-seq kullanarak CRISPR-Cas9'un hedef dışı aktivitesini incelemek için kapsamlı, adım adım bir prosedür sağlar. Örnek olarak, bu protokol, AAVS1 lokusunun modifikasyonu sırasında CRISPR-Cas9'un genom çapında istenmeyen bölünme bölgelerinin haritalanmasıyla doğrulanır. Tüm CIRCLE-seq süreci iki hafta içinde tamamlanabilir, bu da hücre büyümesi, DNA saflaştırması, kütüphane hazırlığı ve Illumina dizilimi için yeterli zaman sağlar. Sıralama verilerinin CIRCLE-seq boru hattına girişi, bölünme bölgelerinin kolaylaştırılmış yorumlanmasını ve analizini kolaylaştırır.
Giriş
Genom mühendisliği, son yirmi yılda önemli ilerlemeler kaydetti ve önemli bir dönüm noktası, 2012'de kümelenmiş düzenli aralıklı kısa palindromik tekrarların (CRISPR)-Cas9'un keşfi oldu1. Bakteriyel DNA endonükleazlarının programlanabilir doğasından yararlanan CRISPR-Cas9 teknolojisi, hemen hemen her DNA dizisinin hassas bir şekilde hedeflenmesini ve değiştirilmesini sağlar. Başlangıcından bu yana, sistem belirli genomik bölgeleri düzenlemek için yalnızca Cas9 endonükleazına ve bir kılavuz RNA'ya (gRNA) dayanacak şekilde optimize edilmiştir. CRISPR-Cas9'un iyileştirici bir tedavi olarak potansiyeli, diğerlerinin yanı sıra Leber'in konjenital amaurozu, transtiretin amiloidozu ve orak hücreli anemi gibi çeşitli durumlar için klinik çalışmalarda gösterilmiştir 2,3,4.
CRISPR-Cas9, tipik olarak iki mekanizmadan biri ile çözülen çift sarmallı kırılmaları (DSB'ler) indükler: hataya eğilimli homolog olmayan uç birleştirme (NHEJ) veya daha kesin homolojiye yönelik onarım (HDR), bir şablon DNA'nın mevcut olması koşuluyla. CRISPR-Cas9'un istenmeyen genomik bölgelerde bölünme ile birlikte NHEJ ile ilişkili eklemelere ve delesyonlara (indels) neden olma eğilimi, klinik ortamlardauygulanmasını sınırlar 5,6,7,8,9,10. Ek olarak, istenmeyen genomik modifikasyonlar, kriptik ekleme bölgeleri, anlamsız veya yanlış anlamlı mutasyonlar oluşturabilir, kromotripsis indükleyebilir veya çeşitli genom düzenleme denemelerinde gözlemlenen hücrelere onkojenik potansiyel verebilir 11,12,13,14,15. Sonuç olarak, CRISPR-Cas9'un hedef dışı aktivitesini doğru bir şekilde tanımlamak, özellikle milyarlarca hücreyi değiştirebilen sistemik gen terapilerinde klinik uygulamaları için çok önemlidir.
Canlı hücrelerde DSB'leri etiketlemek için çift sarmallı oligodeoksinükleotidleri kullanan Çift sarmallı kırılmaların Genom Çapında Tarafsız Tanımlanması (GUIDE)-seq16 dahil olmak üzere CRISPR-Cas9 hedef dışı bölünme bölgelerini tanımlamak için çeşitli yöntemler kullanılabilir. Bununla birlikte, bu yönteme yönelik bir eleştiri, yanlış pozitiflerin rastgele DSB'lerden veya PCR artefaktlarından kaynaklanabileceğidir ve bu, hedeflenen sitelere zayıf benzerlik gösteren yakalanan siteler hariç tutularak atılması gerekir. İntegraz Kusurlu Lentiviral Vektör (IDLV) kullanımına dayanan yöntem daha az hassastır ve birçok hedef dışı bölgeyi gözden kaçırma olasılığı yüksektir17. DSBCapture, BLESS ve BLISS 18,19,20 gibi diğer yerinde yöntemler, sabit hücreleri içerir ve DSB'leri doğrudan etiketler, ancak bunlar, anında DSB yakalamaya bağımlılıkları ve eksojen DNA'nın yokluğu ile sınırlıdır. Bir in vitro yöntem olan Digenome-seq21 ve Dizileme ile Etiketli genomik DNA Uçlarının Seçici zenginleştirilmesi ve Tanımlanması (SITE-seq)22'nin her ikisi de dizileme çözümleri sağlar, ancak sırasıyla arka plan gürültüsü ve tek uçlu analizde sınırlamaları vardır. Yerinde Cas hedef dışı keşfi ve sıralama ile doğrulama (Discover-Seq)23, MRE11 bağlanması yoluyla Cas9 aktivitesinin in vivo ve yerinde tanımlanmasını sağlar, ancak yalnızca numune hazırlama sırasında mevcut olan DSB'leri tespit eder24. Son olarak, CRISPR Düzenlemelerinin Çıkarımı (ICE), Sanger verilerini25 kullanarak CRISPR düzenlemelerini sağlam bir şekilde analiz etmek için biyoinformatik bir yaklaşım kullanır.
Bu makale, Dizileme ile Bölünme Etkilerinin İn Vitro Raporlaması için Daireselleştirme için ayrıntılı bir prosedürü açıklamaktadır (CIRCLE-seq): Cas9 nükleazının genom çapında hedef dışı aktivitesini ilgilenilen gRNA ile bir kompleks içinde hassas ve tarafsız bir şekilde haritalayan bir in vitro teknik26. Bu yaklaşım, ilgilenilen hücrelerin kültürlenmesi ve DNA'nın izole edilmesiyle başlar, ardından odaklanmış ultrasonikasyon yoluyla rastgele kesme, ardından eksonükleaz ve ligaz tedavisi ile başlar. Bu işlem nihayetinde dairesel çift sarmallı DNA molekülleri üretir ve bunlar daha sonra plazmid güvenli DNaz tedavisi ile saflaştırılır. Bu dairesel DNA daha sonra Cas9-gRNA kompleksine maruz bırakılır, bu da hem amaçlanan hem de istenmeyen bölünme bölgelerinde parçalanır ve Illumina adaptör ligasyonu için substrat görevi gören açıkta kalan DNA uçlarını geride bırakır. Bu işlem, nükleazın neden olduğu her bir DSB'nin her iki ucunu içeren çeşitli bir genomik DNA (gDNA) kütüphanesi üretir ve her okumanın her bir bölünme bölgesi için gerekli tüm bilgilere sahip olmasını sağlar. Bu, CIRCLE-seq'i yukarıda belirtilen diğer benzer yöntemlerden ayırarak, daha düşük dizileme kapsamı gereksinimleriyle Illumina dizilemesinin kullanılmasına izin verir. CIRCLE-seq'in in vitro bir yöntem olarak diğer protokollerden daha yüksek hedef dışı duyarlılığa sahip olmasına rağmen, bunun GUIDE-seq16 gibi diğer yöntemlerde bulunan epigenetik manzaranın olmaması nedeniyle daha yüksek yanlış pozitifler pahasına geldiğini belirtmek önemlidir. Ek olarak, DSB DNA onarımı ve ilgili mekanizması, CIRCLE-seq'te, aksi takdirde gözlemlenecek olan indelleri veya uygun onarımı iptal etmede mevcut değildir.
CIRCLE-seq'i gerçekleştirmek için adım adım protokolün açıklanmasına ek olarak, protokol, örnek olarak AAVS1 lokusunun modifikasyonu sırasında meydana gelen CRISPR-Cas9'un genom çapında istenmeyen bölünme bölgelerini tanımlayarak doğrulanır. Bu takip etmesi kolay protokol, indüklenmiş pluripotent kök hücrelerin (iPSC'ler) kültürü ve gDNA izolasyonundan gDNA daireselleştirmesine, Cas9-gRNA bölünmesine, kütüphane hazırlamaya, dizilemeye ve boru hattı analizine kadar ayrıntılı talimatlar sağlar. Düşük dizileme kapsamı gereksinimleri göz önüne alındığında, CIRCLE-seq, yeni nesil dizilemeye erişimi olan herhangi bir laboratuvar tarafından kullanılabilir.
Protokol
Bu çalışma için kullanılan reaktiflerin, sarf malzemelerinin ve ekipmanın ayrıntıları Malzeme Tablosunda listelenmiştir.
1. Hücre kültürü (5 gün)
- Bu protokol boyunca negatif bir kontrol ekleyin. Ek 25 μg gDNA için yeterli hücre hazırlayın (örnek başına ~ 2.0e7 hücre).
- Kurulan protokole göre kültür iPSC'leri27. Hücreleri toplayın ve 10 mL PBS'de yeniden süspanse edin. 6 μL'lik bir hücre örneğini pipetleyin ve tripan mavisi ile 1: 1 oranında yeniden süspanse edin. Örneği saymak için otomatik hücre sayacını kullanın.
- Tüp başına 2 x 107 hücre ekleyin, ardından 25 ° C'de 3 dakika boyunca 300 x g'da döndürün (Oda sıcaklığı (RT)). Bu, birden çok çoğaltma için yeterlidir. Süpernatanı pipetleyin ve atın.
2. Genomik DNA izolasyonu (1 gün)
- Üreticinin talimatlarını izleyerek gDNA'yı izole etmek için piyasada bulunan DNA saflaştırma kitini kullanın:
- Hücre peletini içeren 15 mL'lik konik bir tüpe 200 μL PBS ekleyin ve yeniden süspanse edin. Daha sonra, tüpe 3 mL hücre lizis tamponu ve 15 μL proteinaz K pipetleyin. İyice karıştırmak için tüpü 25 kez ters çevirin. Tüpü, optimum DNA verimi için 3 saat veya gece boyunca 55 ° C ve 150 rpm'ye ayarlanmış bir su banyosu çalkalayıcısına yerleştirin.
- 15 μL RNase A'da pipetleyin ve 25 kez ters çevirin. 37 °C'de 1 saat su banyosuna koyun.
- Numuneyi buz üzerinde 5 dakika soğutun. Daha sonra, 1 mL protein çökeltme çözeltisi ekleyin, 20 saniye boyunca yüksek hızda girdap yapın ve RT'de 10 dakika boyunca 2000 x g'da santrifüjleyin. Proteinler, tüpün dibinde görünür, kompakt bir pelet oluşturmalıdır. Pelet görünmüyorsa, numuneyi 5 dakika daha buz üzerinde inkübe edin ve tekrar santrifüjleyin.
- Yeni bir 15 mL konik tüpe 3 mL %100 izopropanol ekleyin. Adım 2.1.3'teki süpernatanı dikkatlice tüpe pipetleyin. Karıştırmak için tüpü 50 kez ters çevirin, ardından 2000 x g'da ve oda sıcaklığında 3 dakika santrifüjleyin. DNA peletini bozmadan, bir vakum tuzağına bağlı bir Pasteur pipeti kullanarak süpernatanı dikkatlice aspire edin ve tüpü temiz, tüy bırakmayan bir mendil üzerinde ters çevirin.
- DNA peletinin üzerine 3 mL %70 (h/h) etanol pipetleyin ve yıkamak için 10 kez ters çevirin. RT'de 3 dakika boyunca 2.000 x g'da santrifüjleyin. Ardından, süpernatanı dikkatlice dökün.
- Tüpü açık tutun ve elde edilen DNA peletinin 30 dakika kurumasını bekleyin, böylece tüm etanolün tamamen buharlaştığından emin olun. 50 μL DNA hidrasyon solüsyonunda pipetleyin ve nazikçe pipetleyerek iyice karıştırın.
- Numuneyi 1 saat boyunca 65 °C'de bir su banyosu çalkalayıcıya yerleştirerek DNA'yı çözün, ardından numuneyi gece boyunca RT'de bırakın. Numuneyi 1 dakika boyunca RT ve 2.000 x g'da santrifüjleyin ve izole edilen DNA'yı florometre ile ölçmek için ilgili tüplerle birlikte dsDNA BR Test Kitini kullanın.
3. gRNA'nın hazırlanması (7 gün)
- İlgilendiğiniz sentetik gRNA'yı ticari bir kaynaktan sipariş edin (bkz. Malzeme Tablosu). Bu protokol aynı zamanda crRNA/tracrRNA ile de uyumludur.
4. gRNA in vitro bölünme testi
NOT: Burada, AAVS1 genindeki bir hedef kullanılır. İlgilenilen diğer genleri hedeflemek için, hedef bölgeyi amplifiye etmek için primerler (Tablo 1) tasarlayın ve aşağıdaki adımlarda primerleri özel primerlerle değiştirin.
- PCR reaksiyonunu hazırlayın: 25 μL Phusion Hot Start Flex 2x Master Mix (Son Konsantrasyon 1x), 0.5 μL AAVS1 F Primer (Son konsantrasyon 0.1 μM), 0.5 μL AAVS1 R Primer (Nihai konsantrasyon 0.1 μM), 5 μL gDNA (100 ng, 20 ng/μL, adım 2.1.6'dan itibaren) ve 19 μL Nukelaz içermeyen H2O(Toplam hacim: 50 μL).
- Aşağıdaki termodöngüleyici parametrelerini kullanın: Denatürasyon: 2 dk (1 döngü) için 98 °C, Deatürasyon: 10 sn (10 döngü) için 98 °C, Tavlama: 15 sn (10 döngü) için 72-62 °C (-1 °C/döngü), Uzatma: 30 sn (10 döngü) için 72 °C, Deatürasyon, 10 sn (30 döngü) için 98 °C. Tavlama: 15 sn (30 döngü) için 65 °C, Uzatma: 30 sn (30 döngü) için 72 °C, Son Uzatma: 5 dakika (1 döngü) için 72 °C, Tutma: 4 °C süresiz olarak.
- PCR reaksiyonunun ürününü saflaştırmak için AMPure XP boncuklarını kullanın. İlk olarak, PCR ürününe 1,8x hacim veya 90 μL XP boncukları pipetleyin. İyice karıştırmak için on kez pipetleyin. Karışımı inkübe etmek için 5 dakika RT'de bırakın.
- Manyetik bir raf kullanarak, PCR reaksiyon plakasını 3 dakika boyunca mıknatısın üzerine yerleştirerek boncukları çözeltiden ayırın. Temizlenmiş çözeltiyi pipetleyin ve atın. Boncuklara 200 μL %80 etanol (h/h) ekleyin, 30 saniye inkübe edin ve ardından etanolü çıkarın. Etanolün tamamen çıkarılmasını sağlamak için bu yıkama adımını iki kez tekrarlayın.
- Plakayı mıknatısın üzerine yerleştirerek numunelerin 3 dakika doğal olarak kurumasını bekleyin. Plakayı mıknatıstan çıkarın ve 40 μL TE tamponu, pH 8.0 ekleyin. On kez yukarı ve aşağı pipetleyerek karıştırın. Numunenin 2 dakika RT'de oturmasına izin verin.
- PCR reaksiyon plakasını bir dakika daha mıknatısın üzerine yerleştirin. 1 dakika sonra süpernatanı yeni bir plakaya aktarın. Saflaştırılmış PCR verimini bir spektrofotometre kullanarak ölçün ve üreticinin talimatlarını izleyerek Yüksek Hassasiyetli D1000 ScreenTape ve Yüksek Hassasiyetli D1000 reaktifleri (merdiven ve tampon) ile birlikte optik tüp şerit kapaklı bir optik tüp şeridi kullanarak bir TapeStation üzerinde analiz edin. Hazırlanan numuneyi -20 °C'de birkaç aya kadar saklayın.
- Cas9 nükleaz proteinini aşağıdaki gibi 1 μM'ye seyreltin: 2 μL 10x Cas9 tamponu (Son konsantrasyon 1x), 1 μL Cas9 Nükleaz, S. pyogenes (Son Konsantrasyon 1 μM) ve 17 μL Nukelaz içermeyen H2O'yu toplam 20 μL hacimde karıştırın.
- gRNA bozulmasını önlemek için RNaz içermeyen bir prosedür gerçekleştirin. gRNA'yı (adım 3.1'den itibaren) H2O'da 3 μM'ye toplam 10 μL hacme seyreltin.
NOT: gRNA'nın moleküler ağırlığını tahmin etmek için aşağıdaki formülü kullanın: ssRNA'nın moleküler ağırlığı (g/mol) = (ssRNA uzunluğu (nt) x 321.47 g/mol) + 18.02 g/mol. Referans olarak, 3 μM'de 104 nt uzunluğunda bir gRNA yaklaşık 100 ng/μL'dir.
5. DNA kesme (3 saat)
- Önce kontrol kolunu hedef alarak ME220'yi hazırlayın. Ardından, rezervuarı saflaştırılmış deiyonize H2O ile doldurun. Kontrol istasyonu dizüstü bilgisayarında Water Works'e erişin ve Doldur'a tıklayın. Sıcaklığı 4,5 °C'ye ayarlayın.
- 25 μg gDNA'yı bir mikrotüpe aktarın (mikrotüp-130 AFA Fiber Vidalı Kapak). Ardından, tüpü 1x TE ile toplam 130 μL hacme kadar doldurun. DNA'yı ortalama yaklaşık 300 bp uzunluğa kadar kesmek için aşağıdaki koşulları kullanın: Süreyi 10 s'ye ayarlayın; 70'e kadar tepe gücü; Görev Faktörü % 20'ye kadar; 50'ye kadar döngü/seri çekim; bunların tümü Ort Gücünü otomatik olarak 14.0'a ayarlayacaktır.
6. Kesilmiş genomik DNA'nın saflaştırılması (1 saat)
- Kesilmiş genomik DNA'yı her biri 65 μL'lik iki parçaya bölün. Adım 4.3-4.6'da belirtilen prosedürü izleyerek XP boncuklarının hacminin 1,8 katı (117 μL) kullanarak saflaştırın. Süpernatanı yeni bir PCR plakasına aktarın ve miktarı bir spektrofotometre kullanarak ölçün.
- gDNA'nın yaklaşık 300 bp'lik geniş bir dağılıma göre kesildiğinden emin olmak için üreticinin talimatlarına göre ayrıştırılmış kesilmiş gDNA'dan 1 μL'yi bir TapeStation üzerinde çalıştırın. Gerekirse, kesilmiş gDNA'yı -20 °C'de birkaç aya kadar saklayın.
7. CIRCLE-seq kütüphanesinin hazırlanması (3 gün)
- Saç tokası adaptörü tavlama
- Saç tokası adaptörü olan oSQT1288'i (Tablo 1), 1x TE'de 100 μM'lik bir nihai konsantrasyona yeniden askıya alın.
- Adaptör tavlamasını şu şekilde gerçekleştirin: toplam 100 μL hacim için 40 μL oSQT1288 (Son Konsantrasyon 40 μM), 10 μL 10x STE (Son Konsantrasyon 1x) ve 50 μL Nükleaz İçermeyen H2O'yu karıştırın.
- Aşağıdaki tavlama parametrelerini kullanın: 5 dakika boyunca 95 ° C, 70 döngü için dakikada -1 ° C, süresiz olarak 4 ° C'de tutun.
- Bitiş onarımı gerçekleştirin. PCR İçermeyen HTP Kitaplığı Hazırlama Kitini kullanın ve Son Onarım Ana Karışımını hazırlayın.
- 8 μL Nükleaz İçermeyen H2O, 7 μL 10x son onarım tamponu (Son Konsantrasyon 1x) ve 5 μL son onarım enzim karışımını (son hacim 20 μL Toplam Son Onarım Ana Karışımı) karıştırın.
- 20 μL son onarım ana karışımını 4.3-4.6 adımlarından kesilmiş gDNA örneğine pipetleyin. 70 μL'lik bir son hacim için 20 μL End-Repair Master Mix'i 50 μL kesilmiş gDNA ile karıştırın.
- Karışımı 30 dakika boyunca 20 °C'de bir termo döngüleyiciye koyun, ardından 4 °C'de süresiz olarak tutun.
- 1,7x hacim veya 120 μL XP boncukları ekleyin ve 4.3-4.6 arasındaki saflaştırma adımlarını izleyin. 42 μL TE ile Elute, pH 8.0. Bir sonraki adım için boncukların çözelti içinde kaldığından emin olun.
- A-tailing gerçekleştirin. HTP Kütüphane Hazırlama (PCR içermeyen) Kitini kullanarak A-tailing Master Mix'i hazırlayın.
- 5 μL 10x A-kuyruk tamponu (Nihai Konsantrasyon 1x) ve 3 μL A-kuyruk enzimi (toplam nihai hacim 8 μL A-kuyruk Ana Karışımı) karıştırın.
- Adım 7.2.4'ten itibaren boncuklar içeren her bir DNA örneğine 8 μL A-tailing Master Mix'i pipetleyin, 8 μL A-tailing Master Mix'i boncuklar içeren 42 μL Uçtan Onarılmış DNA ile karıştırın (toplam nihai hacim 50 μL). 30 °C'de 30 dakika boyunca bir termocycler'a yerleştirin. Süresiz olarak 4 °C'de tutun.
- 1.8 hacim veya 90 μL PEG / NaCl SPRI solüsyonu (HTP Kütüphane Hazırlama Kitinin bir bileşeni (PCR içermez; 96 reaksiyon)) A kuyruklu DNA'ya pipetleyin. A kuyruklu DNA'yı adım 4.3-4.6'ya göre saflaştırın. A kuyruklu DNA'yı 30 μL TE, pH 8.0'da elute edin. Bir sonraki adım için boncukları çözelti içinde tutun.
- Adaptör ligasyonu gerçekleştirin. HTP Kitaplık Hazırlama (PCR içermeyen) Kitini kullanarak Adaptör Ligation Master Mix'i hazırlayın.
- Adım 7.1.3'ten 10 μL 5x ligasyon tamponu (Son Konsantrasyon 1x), 5 μL DNA Ligaz ve 5 μL tavlanmış firkete adaptörünü (40 μM) karıştırın. Toplam 20 μL'lik bir Adaptör Ligation Master Karışımı için Son Konsantrasyonun 4 μM olduğundan emin olun.
- Adım 7.3.3'ten (numune başına toplam 50 μL toplam nihai hacim) boncuklar içeren her ayrıştırılmış DNA örneğine 20 μL Adaptör Ligation Master Karışımı pipetleyin.
- 20 °C'de 1 saat boyunca bir termo döngüleyiciye yerleştirin. Süresiz olarak 4 °C'de tutun.
- 1x hacim veya 50 μL PEG/NaCl SPRI solüsyonunu adaptörle bağlanmış DNA'ya aktarın ve adım 4.3-4.6'ya göre saflaştırın. 30 μL TE, pH 8.0 ile elute edin ve süpernatanları yeni bir yarı etekli PCR plakasına boşaltın. dsDNA BR testini kullanarak DNA'yı birleştirin ve nicelleştirin. Gerekirse, saflaştırılmış adaptörle bağlanmış DNA'yı -20 ° C'de 1 aya kadar saklayın.
- Lambda Eksonükleaz/Eksonükleaz I (E. coli) Master Mix'i hazırlayın (her iki uca bağlı adaptörler olmadan tek veya çift sarmallı DNA'yı ortadan kaldırmada işlev görür).
- Adım 7.4.4'ten 1 μg Adaptör Bağlanmış DNA alın ve 40 μL'ye seyreltin. 5 μL 10x Eksonükleaz I reaksiyon tamponu (Son Konsantrasyon 1x), 4 μL Lambda Eksonükleaz (Son Konsantrasyon 0.4 U/μL) ve 1 μL Eksonükleaz I (E. coli) (Son Konsantrasyon 0.4 U/μL) 10 μL'lik Lambda Eksonükleaz/Eksonükleaz I Master Karışımının toplam hacmi için.
- 10 μL Lambda Eksonükleaz/Eksonükleaz I Master Karışımını 40 μL (1 μg) adaptörle bağlanmış DNA'ya (toplam hacim 50 μL) pipetleyin. 37 °C'de 1 saat, ardından 75 °C'de 10 dakika boyunca bir termo döngüleyiciye yerleştirin. Süresiz olarak 4 °C'de tutun.
- Lambda Eksonükleaz/Eksonükleaz I ile muamele edilmiş DNA'ya 1,8x hacim veya 90 μL XP boncukları pipetleyin. Adım 4.3-4.6'da verilen talimatlara göre arındırın. 40 μL TE, pH 8.0 içinde elüte. Bir sonraki enzimatik adım için boncukların çözelti içinde kaldığından emin olun.
- KULLANICI Enzimi ve T4 Polinükleotid Kinaz (PNK) ile tedavi edin. USER/T4 PNK Master Mix'i hazırlayın (4 bp'lik çıkıntıları serbest bırakmak ve sonraki ligasyon reaksiyonu için gerekli olan ligasyona hazır DNA uçlarını hazırlamak için gereklidir).
- 5 μL 10x T4 DNA Ligaz Tamponu (Son Konsantrasyon 1x), 3 μL USER Enzimi (Son Konsantrasyon 0.05 U/μL) ve 2 μL T4 PNK (Son Konsantrasyon 0.4 U/μL) karıştırın, toplam 10 μL USER/PNK ana karışımı hacmi için.
- Toplam 50 μL'lik bir hacim için 7.5.3. adımdan boncuklar içeren 40 μL lambda ve eksonükleaz I ile muamele edilmiş DNA örneğine 10 μL USER Enzim/T4 PNK Master Karışımı pipetleyin. 37 °C'de 1 saat süreyle bir termo döngüleyiciye yerleştirin. Süresiz olarak 4 °C'de tutun.
- USER/T4 PNK ile muamele edilen DNA'ya 1,8x hacim veya 90 μL PEG/NaCl SPRI solüsyonu pipetleyin ve adım 4.3-4.6'ya göre saflaştırın. 35 μL TE, pH 8.0 içinde elüte. Süpernatanı yeni bir yarı etekli PCR plakasına boşaltın. dsDNA HS testini kullanarak DNA'yı birleştirin ve nicelleştirin.
- Molekül içi daireselleştirme gerçekleştirin. Daireselleştirme ana karışımını hazırlayın.
- 8 μL Nükleaz içermeyen H2O, 10 μL 10x T4 DNA ligaz tamponu (Nihai Konsantrasyon 1x) ve 2 μL T4 DNA ligaz (Nihai Konsantrasyon 8 U/μL) karıştırın, toplam 20 μL'lik Daireselleştirme Ana Karışımı hacmi için.
- Adım 7.6.3'ten itibaren USER/PNK ile muamele edilmiş 500 ng DNA'ya 20 μL Daireselleştirme Ana Karışımını pipetleyin. USER/PNK ile muamele edilmiş 500 ng DNA'yı 80 μL'de seyreltin, ardından 20 μL Daireselleştirme Ana Karışımı (toplam hacim 100 μL) ekleyin. 16 ° C'de 16 saat (gece boyunca) bir termodöngüleyicide inkübe edin.
- Dairesel DNA'ya 1x hacim veya 100 μL XP boncukları ekleyin ve adım 4.3-4.6'ya göre saflaştırın. 38 μL TE, pH 8.0'da elüte. Süpernatanı yeni bir yarı etekli PCR plakasına boşaltın.
- Plazmid Güvenli ATP'ye Bağımlı DNaz ile tedavi edin. Plazmid Güvenli ATP'ye Bağımlı DNaz Master Karışımını hazırlayın (Artık lineer DNA'nın bozunması için gereklidir).
- 5 μL 10x Plazmid Güvenli Reaksiyon Tamponu (Son Konsantrasyon 1x), 2 μL ATP (Son Konsantrasyon 1 mM) ve 5 μL Plazmid Güvenli ATP'ye Bağımlı DNaz (Son Konsantrasyon 1 U/μL) karıştırın, toplam 12 μL'lik Plazmid-Güvenli Master Karışım hacmi için.
- ATP'ye bağımlı DNase Master Mix'in 12 μL'sini adım 7.7.3'ten (toplam 50 μL hacim) 38 μL dairesel DNA'ya pipetleyin. 37 °C'de 1 saat, ardından 70 °C'de 30 dakika inkübe edin. Süresiz olarak 4 °C'de tutun.
- Plazmid-Güvenli ATP'ye Bağımlı DNaz ile muamele edilmiş DNA'ya 1x hacim veya 50 μL XP boncukları pipetleyin ve adım 4.3-4.6'ya göre saflaştırın. 15 μL TE, pH 8.0 içinde elüte. Süpernatanı yeni bir yarı etekli PCR plakasına boşaltın.
- DNA'yı birleştirin ve dsDNA HS testini kullanarak nicelleştirin. Gerekirse, dairesel DNA'yı birkaç aya kadar -20 °C'de saklayın.
8. Enzimatik olarak saflaştırılmış, dairesel gDNA'nın in vitro olarak parçalanması (2 saat)
- Cas9:gRNA kompleksi ile in vitro bölünme gerçekleştirin. In vitro Cleavage Master Mix'i hazırlayın. 5 μL 10x Cas9 Tamponu (Son Konsantrasyon 1x), 4.5 μL S. Pyogenes Cas9 (Nihai Konsantrasyon 90 nM) ve 1.5 μL gRNA'yı (Son Konsantrasyon 90 nM) toplam 11 μL'lik Cleavage Master Mix hacmi için karıştırın.
- Cas9:gRNA RNP kompleksleri oluşturmak için bölünme ana karışımını RT'de 10 dakika tutun.
- 125 ng Plazmid-Güvenli DNaz ile muamele edilmiş DNA'yı adım 7.8.3'ten 39 μL'lik bir nihai hacme seyreltin. Ardından, toplam 50 μL'lik bir hacim için 39 μL Plazmid-Güvenli DNaz ile muamele edilmiş DNA'ya 11 μL Cleavage Master Mix ekleyin.
NOT: Bu adıma, Cas9:gRNA kompleksi olmadan, Cas9 tamponu ile karıştırılmış dairesel DNA'yı içeren bir negatif kontrol numunesi ekleyin. - 37 °C'de 1 saat boyunca bir termodöngüleyicide inkübe edin. Süresiz olarak 4 ° C'de koruyun. İn vitro parçalanmış DNA'ya 50 μL (1x hacim) XP boncukları ekleyin ve 4.3-4.6 adımlarını izleyerek DNA'yı saflaştırın. 42 μL TE tamponunda Elute, pH 8.0. Bir sonraki adım için boncukların çözelti içinde kaldığından emin olun.
9. Yeni nesil dizileme kütüphanesinin hazırlanması (4 - 6 saat)
- A-tailing gerçekleştirin. A-tailing Master Mix'i hazırlayın.
- 5 μL 10x A-kuyruk Tamponu (Nihai Konsantrasyon 1x) ve 3 μL A-kuyruk Enzimini karıştırın (A-kuyruk ana karışımının toplam hacmi 8 μL'dir).
- 8 μL'lik A-tailing Master Mix'i, adım 8.4'ten (toplam 50 μL hacim) boncuklar içeren 42 μL ayrıştırılmış DNA örneğine pipetleyin. 30 ° C'de 30 dakika boyunca bir termo döngüleyiciye koyun. Süresiz olarak 4 °C'de tutun.
- A kuyruklu DNA'ya 1.8x hacim PEG / NaCl SPRI solüsyonu veya 90 μL pipetleyin ve DNA'yı 4.3-4.6 adımlarına göre saflaştırın. 25 μL TE, pH 8.0 içinde elüte. Bir sonraki adım için boncukları çözelti içinde tuttuğunuzdan emin olun.
- Adaptör ligasyonu gerçekleştirin. Adaptör ligation ana karışımını hazırlayın.
NOT: 3' T'nin donma-çözülme kaynaklı hidrolizinin neden olduğu adaptör dimerlerinin oluşumunu önlemek için NEB adaptörlerinin tek kullanımlık alikotları hazırlanmalıdır.- Dizileme için 10 μL 5x Ligasyon Tamponu (Son Konsantrasyon 1x), 5 μL DNA Ligaz ve 10 μL Adaptörü (Nihai konsantrasyon 3 μM'dir) toplam 25 μL karıştırın.
- 25μL Adaptör Ligation Master Karışımını, adım 9.1.3'ten itibaren boncuklar içeren 25 μL A kuyruklu DNA örneğine pipetleyin. 20 °C'de 1 saat boyunca bir termo döngüleyiciye koyun. Süresiz olarak 4 °C'de tutun.
- Adaptörle bağlanmış DNA'ya 1x hacim veya 50 μL PEG/NaCl SPRI solüsyonu pipetleyin ve DNA'yı 4.3-4.6 adımlarına göre saflaştırın. 47 μL TE, pH 8.0 içinde elüte. Bir sonraki enzimatik adım için boncukların çözelti içinde kaldığından emin olun.
- KULLANICI Enzimi ile tedavi yapın (urasil kalıntılarında tek bir nükleotid boşluğu oluşturur).
- Adım 9.2.3'ten itibaren boncuklar içeren adaptörle bağlanmış DNA örneğine 3 μL USER enzimi (Çift İndeksli Primer Kitinde bulunur) ekleyin. 37 °C'de 15 dakika inkübe edin.
- KULLANICI enzimi ile muamele edilen DNA'ya 35 μL (0.7x hacim) PEG / NaCl SPRI çözeltisi ekleyin ve 4.3-4.6 adımlarına göre saflaştırın. 20 μL TE tamponunda elute, pH 8.0. Süpernatanı yeni bir yarı etekli PCR plakasına aktarın ve dsDNA HS testini kullanarak DNA konsantrasyonunu ölçün. Beklenen konsantrasyon yaklaşık 2-5 ng/μL olmalıdır.
- (İsteğe bağlı) Bir sonraki adıma geçmeden önce PippinHT kullanılarak DNA boyutu seçimi gerçekleştirilebilir. 250-850 bp boyut aralığına sahip %1,5 PippinHT kaseti kullanın. Elde edilen numuneler, bir sonraki adımda doğrudan PCR'de kullanılabilir.
- Barkod ekleme için PCR gerçekleştirin
NOT: Her numune için seçilen astar dizisi kombinasyonlarının benzersiz olduğundan emin olun. Mümkünse, her numunenin benzersiz i5 ve i7 barkodları olmalıdır.- Eşleştirilmiş indeksli barkodlar eklemek için PCR Master Mix'i hazırlayın. 5 μL Nükleaz içermeyen H2O, 25 μL 2x HotStart Hazır Karışım (Son Konsantrasyon 1x), 5 μL i5 Astar (Son Konsantrasyon 1 μM) ve 5 μL i7 Astar (Son Konsantrasyon 1 μM) (toplam ana karışım hacmi 40 μL) karıştırın.
- 40 μL PCR Master Mix'i, adım 9.3.2'den (toplam 50 μL hacim) USER enzimi (yaklaşık 20 ng) ile muamele edilmiş 10 μL saflaştırılmış DNA'ya pipetleyin.
- Aşağıdaki PCR termodöngü koşullarını seçin: Denatürasyon: 1 döngü için 45 saniye için 98 °C, Denatürasyon: 20 döngü için 15 saniye için 98 °C, Tavlama: 20 döngü için 30 saniye için 65 °C, Uzatma: 20 döngü için 30 saniye için 72 °C, Son Uzatma: 1 döngü için 1 dakika boyunca 72 °C, Tutun: 4 °C süresiz olarak.
- PCR ürününe 0,7x hacim veya 35 μL XP boncukları ekleyin ve 4.3-4.6 adımlarına göre saflaştırın. 30 μL TE, pH 8.0 içinde elüte. Süpernatanı yeni bir yarı etekli PCR plakasına boşaltın. Gerekirse, dairesel DNA'yı birkaç aya kadar -20 ° C'de saklayın.
NOT: Kitaplığın kalitesini kontrol etmek ve adaptör dimerlerinin oluşumunu değerlendirmek için Tapestation'da bir PCR örneği çalıştırın. Adaptör karartıcıları algılanırsa, adım 9.5.4'ü tekrarlayın.
10. CIRCLE-seq kütüphanelerinin damlacık dijital PCR ile miktar tayini (dd_PCR) (6 saat)
NOT: Kantifikasyon, qPCR, Tapestation veya benzer bir yöntem kullanılarak da gerçekleştirilebilir.
- Kütüphaneden 5 μL DNA ile başlayın (PCR aşaması 9.5.4) 45 μL nükleaz içermeyen TE ile iyice karıştırın, daha sonra her numunenin 50 μL hacimlerde 10-10 ila 10-8 seyreltme arasında değişen 1:10 seri seyreltmesini yapın.
- dd_PCR Master Mix stok çözümünü ayarlayın. Problar için 11 μL 2x dd_PCR karışımı karıştırın (Son Konsantrasyon 1x), 0.055 μL Prob oSQT1310 (Son Konsantrasyon 250 nM), 0.055 μL Prob oSQT1311 (Son Konsantrasyon 250 nM), 0.099 μL Primer oSQT1274 (Nihai Konsantrasyon 450 nM), 0.099 μL Primer oSQT1275 (Nihai Konsantrasyon 450 nM) ve 6.292 μL Nükleaz içermeyen H2O, 17,6 μL'lik toplam dd_PCR Master Mix hacmi için. Hacimlerin doğru pipetleme için yeterli olduğundan emin olmak için tüm numuneler için bir ana karışım hazırlayın.
- En düşük üç seyreltmeyi (10-6,10-7 ve 10-8) iki kez (96 oyuklu bir plakada) test edin. Şablon olmayan bir denetim (NTC) kullanılmalıdır.
- Her numuneye 17,6 μL dd_PCR Master Mix pipetleyin: Toplam 22 μL'lik bir hacim için 17,6 μL'yi 4,4 μL numune ile karıştırın (NTC ile kuyucuğa nükleaz içermeyen H2O ekleyerek). Plakayı kapatın, ardından RT'de 1 dakika boyunca 2000 x g'da santrifüjleyin.
- Damlacık üretimi, termodöngü ve analiz gerçekleştirin. Bir Damlacık Okuyucu PCR sistemi kullanarak, bir DG8 kartuşunu (8 kuyulu) kartuş tutucuya aktarın. Kartuşun yağ sırasında, problar için 70 μL damlacık üretim yağı dağıtın.
NOT: 20 μL alınamp10.1.2 adımından itibaren. ve 8 kuyulu kartuşun numune sırasına ekleyin, kartuşu DG8 lastik conta ile kaplayın, damlacık üretecine yerleştirin ve ardından işleme başlamak için kapatın (otomatik). Tamamlandığında, kartuşu çıkarın ve 40 oyuklu kartuşun damlacık sırasından 8 μL'yi yarı etekli 96 oyuklu bir PCR plakasına taşıyın ve yavaşça pipetlediğinizden emin olun.- Isı bloğunu PX1 PCR plaka kapatıcıya yerleştirin. Sızdırmazlık maddesi açıldığında 180 °C'ye kadar ısınmaya başlayacaktır. Folyo ısı contasını plakaya yerleştirin ve kırmızı çizginin üstte olduğundan emin olun. Plakayı PX1'e yerleştirin ve Mühür'e basın.
- Aşağıdaki Termodöngüleyici koşullarını seçin: Enzim Aktivasyonu: 1 döngü için 10 dakika boyunca 95 °C, Denatürasyon: 40 döngü için 30 saniye boyunca 94 °C, Tavlama/Uzatma: 40 döngü için 1 dakika boyunca 60 °C, Enzim Devre Dışı Bırakma: 1 döngü için 10 dakika boyunca 98 °C, Tutma: 4 °C süresiz olarak.
- Damlacık okuyucuda uyumlu yazılımı açın ve hangi kuyucukların okunacağını seçin. Deney türü olarak ABS'yi seçin ve problar için Supermix'i dd_PCR . Hedef 1 için Ch1 Bilinmiyor'u ve Hedef 2 için Ch2 Bilinmiyor'u seçin. Uygula'yı ve ardından Tamam'ı seçin. Plakayı damlacık okuyucuya yerleştirin. Boya seti için FAM/HEX'i seçin ve Çalıştır'a tıklayın.
- En dd_PCR sonuçları analiz edin. Negatif kontrolü referans olarak kullanarak çift pozitif damlacık popülasyonunu geçitleyin. Yinelenen değerlerin ortalamasını hesaplayın, ardından seyreltme faktörü ve dd_PCR 5 kat seyreltme faktörü ile çarpın.
NOT: Mikrolitre başına toplam kopya sayısı şu şekilde hesaplanır: mikrolitre başına toplam kopya = seyreltme faktörü × 5 × ortalama değer, burada 'ortalama değer' Ch1 ve Ch2'den ortalama niceleme değerini temsil eder. - Tüm örnekleri eşmolar konsantrasyonlarda tek bir kitaplıkta birleştirin. 1x havuzlanmış kütüphane yaklaşık 4.5 x 109 molekül içermeli ve toplam hacmi 5 μL'dir.
11. Yeni nesil dizileme
- Numuneleri sıralama için harici bir kuruma gönderin ve doğru adaptör dizilerinin not edildiğinden emin olun.
12. CIRCLE-seq veri analizi (1 - 3 saat)
- Python sürüm 2.7, Burrows-Wheeler Aligner (BWA) ve SAMtools'u yükleyin. Referans genomu (örneğin, hg38) http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz'dan indirin.
NOT: Hedef türün genomu mevcut değilse, CIRCLE-seq hesaplama iş akışı referanstan bağımsız bir modda çalıştırılabilir. Bu durumda, bu adım atlanabilir. - Aşağıdaki komutlarla CIRCLE-seq ardışık düzenini indirin ve kurun: (1) git clone https://github.com/tsailabSJ/circleseq.git, (2) cd circleseq, (3) pip install -r requirements.txt.
- YAML biçiminde (.yaml) bir bildirim dosyası oluşturun. Aşağıda, iş akışını test etmek için CIRCLE-seq yazılımında sağlanan örnek veri kümesiyle kullanılabilecek örnek bir bildirim verilmiştir.
NOT: (1) referans genomu: data/input/CIRCLEseq_test_genome.fa; (2) analysis_folder: veri / çıktı; (3) bwa: bwa; (4) Samtools: Samtools; (5) read_threshold: ; (6) window_size: ; (7) mapq_threshold: ; (8) start_threshold: ; (9) gap_threshold: ; (10) mismatch_threshold: ; (11) merged_analysis: Doğru; (12) Örnekler: U2OS_EMX1; (13) hedef: GAGTCCGAGCAGAAGAAGAANGG; (14) okuma1: veri/giriş/EMX1.r1.fastq.gz; (15) Oku2: veri/giriş/EMX1.r2.fastq.gz; (16) ControlRead1: veri/giriş/EMX1_control.r1.fastq.gz; (17) ControlRead2: veri/giriş/EMX1_control.r2.fastq.gz; (18) açıklama: U2OS. Aşağıdaki bildirim değerleri kullanılmıştır: read_threshold: 4, window_size: 3, mapq_threshold: 50, start_threshold: 1, gap_threshold: 3, mismatch_threshold: 6 - Referans genom FASTA dosyasını, analiz için çıktı dizinini ve BWA ve SAMtools komutlarının yollarını tanımlayın. Hem nükleaz parçalanmış hem de kontrol örnekleri için hedef dizileri ve çoğullamayı kaldırılmış FASTQ dosyalarının yollarını tanımlayın. Birden çok deneme, tümü tek bir bildirim dosyasına dahil edilerek toplu modda aynı anda işlenebilir.
- Standart başvuru tabanlı analizler için aşağıdaki komutu yürütün: (1) python /path/to/circleseq.py all - manifest; (2) /path/to/manifest.yaml.
- Alternatif olarak, referans tabanlı olmayan standart analizler için aşağıdaki komutu yürütün: (1) Python /path/to/circleseq.py referanssız - manifest; (2) /path/to/manifest.yaml.
- Tam işlem hattını yürütürken, her adımın çıkış sonuçlarını, söz konusu adım için belirlenmiş ayrı bir output_folder bulun.
Sonuçlar
Burada, CIRCLE-seq, indüklenmiş pluripotent kök hücrelerden (iPSC'ler) izole edilen DNA'yı kullanarak adeno-ilişkili virüs entegrasyon bölgesi 1'i (AAVS1) hedeflemek üzere tasarlanmış gRNA ile bir kompleks içinde Cas9'un nükleaz kaynaklı bölünme bölgelerini araştırmak için kullanılır. Bu gRNA daha önce27 numaralı yayınımızda tanımlanmıştı. iPSC'lerden yaklaşık 25 μg gDNA izole edildi, odaklanmış ultrasonikasyon yoluyla kesildi ve yaklaşık 300 bps'lik parçalar elde etmek için AMPure XP boncuk saflaştırması kullanılarak boyut seçildi. Bu 25 μg DNA'dan, yaklaşık 2-5 ng DNA, in vitro Cas9:gRNA bölünmesi için başarılı bir şekilde dairesel hale getirildi. Prosedürün tamamı Şekil 1'de gösterilmiştir.
Hesaplama iş akışımızı kullanan bir CIRCLE-seq prosedürü ve analizinin ardından, tespit edilen tüm hedef içi ve dışı bölünme bölgelerinin bir görselleştirmesi Şekil 2A'da sunulmaktadır. CIRCLE-seq boru hattı ayrıca, her bir kromozom boyunca haritalanan tespit edilen nükleaz kaynaklı bölünme bölgelerini gösteren bir Manhattan grafiği elde etmek için R istatistiksel yazılımı aracılığıyla analiz edilen 'birleştirilmiş okumalar' sağladı (Şekil 2B).
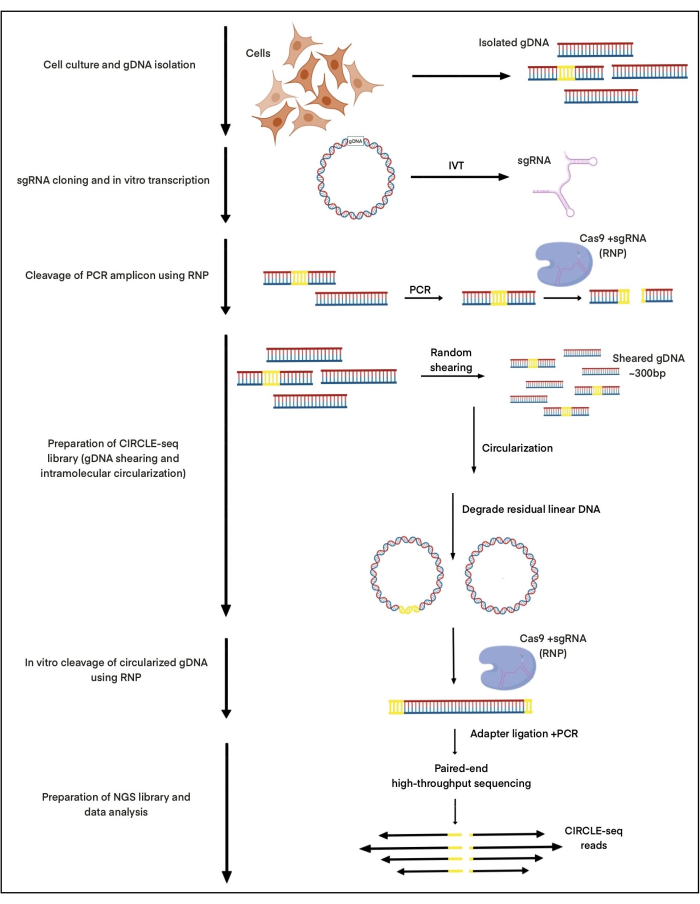
Şekil 1: CIRCLE-seq iş akışı şemaları. Protokolün ana adımları belirtilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
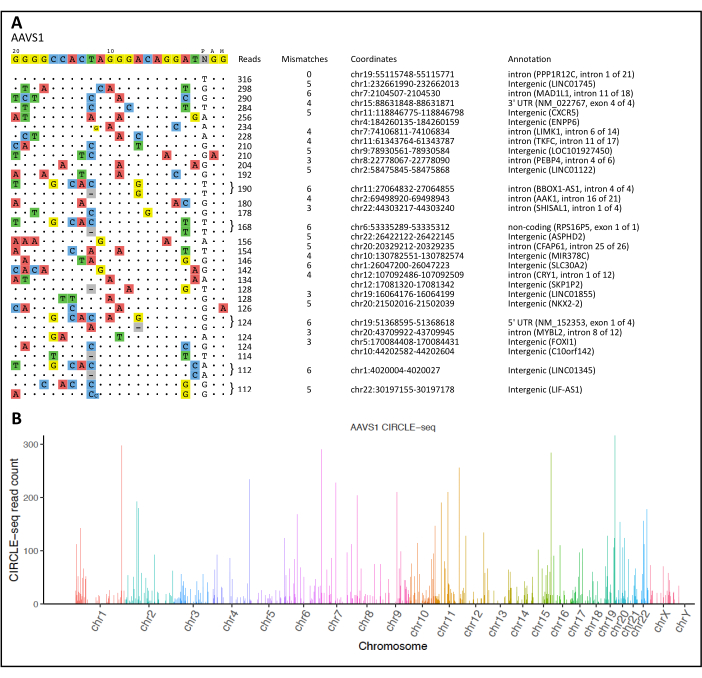
Şekil 2: CIRCLE-seq görselleştirmesi ve Manhattan Plot. (A) Hedef dışı bölgelerin AAVS1 lokusu için amaçlanan hedefe göre hizalanması. Hedef dizi en üstte görüntülenir ve burada hedef dışı hedefler okuma sayısına göre azalan sırada sıralanır. Orijinal hedef dizilerdeki farklılıklar renkli nükleotidlerle gösterilir. AAVS1 lokusu için en üst istenmeyen bölünme bölgelerinin bir örneği gösterilmektedir. (B) AAVS1 lokusu için tespit edilen istenmeyen bölünme bölgelerini gösteren Manhattan grafiği. Çubuk yükseklikleri, her kromozomal pozisyon için okuma sayılarını temsil eder. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Astar | Sıra (5'-3') | Yorumlar/Açıklama |
| AAVS1 Tek kılavuz RNA (sgRNA) | GGGGCCACUAGGGACAGGAU | AAVS1 Locus'un Floresan Protein Knock-In için |
| AAVS1 İleri Astar | GCTCTGGGCGGAGGAATG | gRNA in vitro bölünme testi için |
| AAVS1 Ters Astar | ATTCCCAGGGCCGGTTAATG | gRNA in vitro bölünme testi için |
| oSQT1288 | /5Phos/CGGTGGACCGATGATC /ideoxyU/ATCGGTCCACCGaT | CIRCLE-seq saç tokası adaptörü |
| oSQT1274 | AATGATACGGCGACCACCGAG | TruSeq F1 |
| oSQT1275 | CAAGCAGAAGACGGCATACGAGAT | TruSeqF2 |
| oSQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | TruSeq probu |
| oSQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | TruSeq probu |
Tablo 1: AAVS1 lokusunun CIRCLE-seq analizi için kullanılan gRNA ve primer dizileri.
Tartışmalar
Burada, CIRCLE-seq'in, iPSC'lerden türetilen gDNA'daki AAVS1 lokusunu hedeflemekten kaynaklanan genom boyunca nükleaz kaynaklı DSB'leri tanımlamak için tarafsız ve oldukça hassas bir teknik olduğu gösterilmiştir. iPSC'ler içindeki AAVS1 bölgesi, genellikle CRISPR-Cas928 kullanılarak eksojen genlerin bir entegrasyon bölgesi olarak kullanılan güvenli bir liman lokusu olarak bilinir. Son raporumuz, EGFP'nin hücrenin soyu boyunca kalıcılığı nedeniyle hem iPSC'lerin hem de farklılaşmış iPSC'lerin etiketlenmesini ve izlenmesini sağlayan, yapısal olarak ifade edilen bir EGFP raportörünün AAVS1 bölgesine CRISPR aracılı entegrasyonunu gerçekleştirerek EGFP etiketli iPSC'lerin potansiyelini inceledi27. Bu iPSC hattı, transplantasyondan sonra iPSC'den türetilmiş hücrelerin organizma dağılımını değerlendirmek için in vivo olarak kullanılabilir. Bu hücre hattı CRISPR ile modifiye edildiğinden ve aynı zamanda iPSC'lerin klinik uygulamasını test etmek için kullanıldığından, güvenlik ve etkinliği sağlamak için potansiyel AAVS1 hedef dışı bölgelerin bilinmesi ve sorgulanması zorunludur, bu da onu CIRCLE-seq'i test etmek için ideal bir yer haline getirir.
Butterfield ve ark., daha önce yayınlanmış çalışma27 ile bu çalışma arasındaki dikkate değer bir fark, AAVS1 lokusunu hedeflemek için modifiye edilmiş bir gRNA'nın kullanılmasıydı. Genom düzenleme doğruluğunu artırmak için bir gRNA tasarlanabilir24. Kılavuz sırası, hedef içi ve dışı verimlilikleri etkileyen en kritik faktördür. Bu nedenle, seçilen gRNA diğer birkaç kılavuza karşı test edildi ve üstün aslına uygun olduğu bulundu. Ek olarak, insan fibroblastlarının yeniden programlanmasına ilişkin bir yöntem makalesi, modifiye edilmiş nükleobazlar içeren sentetik kapaklı mRNA'ların antiviral yanıtların düşük aktivasyonundan yararlandığı bulgusunu doğrulamıştır29,30. Düşük immünojenisite bir in vitro testte ilgili olmasa da, nihai hedef canlı hücrelerde kullanılabilecek klinik olarak ilgili bir tedavi geliştirmek olduğunda çok önemli hale gelir.
CIRCLE-seq, benzer yöntemlere göre birçok avantaja sahiptir. Örneğin, Digenome-seq, ~ 400 milyon okuma21 kullanarak hem nükleaz ile bölünmüş hem de ayrılmamış gDNA'yı diziliyor. Bu, yüksek bir arka planla sonuçlanır ve düşük frekanslı iyi niyetli kesim alanlarını filtrelemeyi zorlaştırır. CIRCLE-seq, nükleaz parçalanmış gDNA'nın zenginleşmesi nedeniyle yalnızca ~ 3-5 milyon okuma kullanır ve bu da düşük arka plan ile sonuçlanır. Ek olarak, Digenome-seq ve benzer bir yöntem olan SITE-seq, tek bir nükleaz parçalanmış DNA ucunun dizilenmesine dayanır. Buna karşılık, CIRCLE-seq okumaları, kesilen bölgenin her iki ucunu da içerir ve referans 21,22,26'ya ihtiyaç duymadan hedef dışı sitelerin tanımlanmasını sağlar.
CIRCLE-seq'in bir avantajı, GUIDE-seq gibi hücre kültürüne dayanan yöntemlere kıyasla daha fazla hassasiyetidir. İki yöntem karşılaştırıldığında, CIRCLE-seq, GUIDE-seq tarafından tespit edilen tüm hedef dışı bölgeleri yakalayabildi ve GUIDE-seq'in gözden kaçırdığı ek istenmeyen bölünme bölgelerini ortaya çıkardı. Bununla birlikte, dikkate değer bir fark, GUIDE-seq'in epigenetik manzara tarafından engellenebilmesi, CIRCLE-seq'in ise tüm genoma erişebilmesidir.
Bir in vitro test olarak CIRCLE-seq, ilki yanlış pozitiflerin tespiti olan çeşitli sınırlamalar sunar. Epigenetik, in vivo olarak belirli bölgelerde nükleaz aktivitesini engelleyecek olsa da, ultrasonikasyon bu engelleri in vitro olarak ortadan kaldırır ve normalde hücresel bağlamda erişilemeyen yerlerde hedef dışı aktiviteye izin verir. Ayrıca, Cas9 bu in vitro testte yüksek konsantrasyonlarda bulunur ve aksi takdirde in vivo olarak mümkün olmayacak bölünmeye izin verir. Bu tahlil aynı zamanda, mevcut kaynaklara bağlı olarak, bu protokolün kullanımını reddedebilecek nispeten büyük miktarda başlangıç gDNA'sı gerektirir. Son olarak, mevcut yeni nesil dizileme teknolojilerinin sınırlamaları nedeniyle bazı hedef dışı sitelerin tespit edilemez olması mümkündür.
Yakın tarihli bir çalışma, algoritması CIRCLE-seq ve GUIDE-seq31 dahil olmak üzere farklı nükleaz karakterizasyon yöntemlerini karşılaştırmak için bir dizi ilgili parametreyi tanımlayan bir in silico yaklaşım kullandı. İlgili parametrelerden ikisi 'kesim yeri zenginleştirme' ve '% yanlış pozitifler' idi. İlginç bir şekilde, CIRCLE-seq'in yanlış pozitiflik oranı %88 olarak hesaplandı, ancak kesim yeri zenginleştirmesi diğer in vitro yöntemlere göre kat kat daha yüksekti. Her yöntemin karşılaştırmalı analizleri, GUIDE-seq'in en iyi performans gösteren olduğunu ortaya koydu, çünkü yalnızca orta derecede bir yanlış pozitif oranı32 ile en yüksek hedef özgüllüğünü gösterdi. Bu, CIRCLE-seq'i geçersiz kılmaz, daha ziyade CIRCLE-seq ve GUIDE-seq'i birlikte kullanma olasılığına işaret eder ve CIRCLE-seq'in bulgularını GUIDE-seq ile doğrular, çünkü birincisi daha yüksek hassasiyete sahipken, ikincisi yüksek kesim bölgesi zenginleştirmesine sahip hücre bazlı bir yöntemdir. Veriler ayrıca, amplikon tabanlı yeni nesil dizilemenin (NGS), potansiyel aday bölgelerdeki gerçek hedef dışı değişiklikleri tanımlamak için tercih edilen yöntem olması gerektiğini göstermektedir31. Bu veriler, hedef dışı etkileri incelemek için CIRCLE-seq, ardından GUIDE-seq ve ardından amplikon tabanlı NGS kullanma stratejisine yönelik potansiyel bir strateji önermektedir.
Açıklamalar
Yazarların ifşa edecek hiçbir şeyi yok.
Teşekkürler
Ulusal Sağlık Enstitüleri (R01AR078551 ve T32AR007411), Distrofik Epidermolizis Bullosa Araştırma Derneği (DEBRA) Avusturya, Gates Grubstake Fonu ve Gates Frontiers Fonu tarafından sağlanan finansman desteği için en derin şükranlarımızı sunarız.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
Referanslar
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185 (2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856 (2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786 (2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058 (2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157 (2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474 (2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745 (2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır