Method Article
CIRCLE-Seq לחקירת עריכת גנים מחוץ למטרה
In This Article
Summary
מחסום משמעותי לטכנולוגיות כמו CRISPR הוא אירועים מחוץ למטרה שיכולים לשבש גנים חיוניים. 'סירקולריזציה לדיווח במבחנה על השפעות מחשוף על ידי ריצוף' (CIRCLE-seq) היא טכניקה שנועדה לזהות אתרי מחשוף לא מכוונים. שיטה זו ממפה את הפעילות הכלל-גנומית של CRISPR-Cas9 ברגישות גבוהה וללא משוא פנים.
Abstract
סירקולריזציה לדיווח חוץ גופי על השפעות מחשוף על ידי ריצוף (CIRCLE-seq) היא טכניקה חדשה שפותחה לזיהוי חסר פניות של אתרי מחשוף לא מכוונים של CRISPR-Cas9 באמצעות ריצוף ממוקד של DNA שסוע CRISPR-Cas9. הפרוטוקול כולל מעגל DNA גנומי (gDNA), אשר מטופל לאחר מכן בחלבון Cas9 וב-RNA מנחה (gRNA) מעניין. לאחר הטיפול, ה-DNA השסוע מטוהר ומוכן כספרייה לריצוף אילומינה. תהליך הריצוף מייצר קריאות קצה זוגיות, המציע נתונים מקיפים על כל אתר מחשוף. CIRCLE-seq מספק מספר יתרונות על פני שיטות אחרות במבחנה , כולל דרישות עומק ריצוף מינימליות, רקע נמוך והעשרה גבוהה עבור gDNA שסוע Cas9. יתרונות אלה מגבירים את הרגישות בזיהוי אירועי מחשוף מכוונים ולא מכוונים. מחקר זה מספק נוהל מקיף, שלב אחר שלב, לבחינת הפעילות מחוץ למטרה של CRISPR-Cas9 באמצעות CIRCLE-seq. כדוגמה, פרוטוקול זה מאומת על ידי מיפוי אתרי מחשוף לא מכוונים ברחבי הגנום של CRISPR-Cas9 במהלך שינוי לוקוס AAVS1 . ניתן להשלים את כל תהליך ה-CIRCLE-seq תוך שבועיים, מה שמאפשר מספיק זמן לצמיחת תאים, טיהור DNA, הכנת ספרייה וריצוף Illumina. הקלט של נתוני ריצוף לצינור CIRCLE-seq מאפשר פרשנות וניתוח יעילים של אתרי מחשוף.
Introduction
הנדסת הגנום ראתה התקדמות משמעותית בעשרים השנים האחרונות, כאשר אבן דרך מרכזית הייתה הגילוי של חזרות פלינדרומיות קצרות במרווחים קבועים (CRISPR)-Cas9 בשנת 20121. טכנולוגיית CRISPR-Cas9 ממנפת את האופי הניתן לתכנות של אנדונוקלאזות DNA חיידקיות, ומאפשרת מיקוד ושינוי מדויקים של כמעט כל רצף DNA. מאז הקמתה, המערכת עברה אופטימיזציה להסתמך רק על אנדונוקלאז Cas9 ו-RNA מנחה (gRNA) לעריכת אזורים גנומיים ספציפיים. הפוטנציאל של CRISPR-Cas9 כטיפול מרפא הוכח בניסויים קליניים למצבים שונים כגון אמורוזיס מולדת של לבר, עמילואידוזיס טרנסתירטין ואנמיה חרמשית, בין היתר 2,3,4.
CRISPR-Cas9 גורם להפסקות דו-גדיליות (DSBs), אשר נפתרות בדרך כלל על ידי אחד משני מנגנונים: חיבור קצה לא הומולוגי הנוטה לשגיאות (NHEJ) או תיקון מכוון הומולוגיה מדויק יותר (HDR), בתנאי שתבנית DNA זמינה. הנטייה של CRISPR-Cas9 לגרום להוספות ומחיקות הקשורות ל-NHEJ (indels), יחד עם מחשוף באתרים גנומיים לא מכוונים, מגבילה את יישומו במסגרות קליניות 5,6,7,8,9,10. בנוסף, שינויים גנומיים לא מכוונים יכולים ליצור אתרי חיבור קריפטיים, מוטציות שטויות או מוטציות שגויות, לגרום לכרומותריפסיס או להעניק פוטנציאל אונקוגני לתוצאות תאים שנצפו במספר ניסויים בעריכת גנום 11,12,13,14,15. לסיכום, זיהוי מדויק של הפעילות מחוץ למטרה של CRISPR-Cas9 הוא חיוני ליישומים הקליניים שלו, במיוחד בטיפולים גנטיים מערכתיים שעשויים לשנות מיליארדי תאים.
ניתן להשתמש בשיטות שונות לזיהוי אתרי מחשוף מחוץ למטרה של CRISPR-Cas9, כולל זיהוי בלתי מוטה של שברים דו-גדיליים (GUIDE)-seq16, המשתמש באוליגודאוקסינוקלאוטידים דו-גדיליים כדי לתייג DSBs בתאים חיים. עם זאת, ביקורת על שיטה זו היא שתוצאות חיוביות כוזבות יכולות לנבוע מ-DSB אקראיים או מחפצי PCR, אותם יש להשליך על ידי אי הכללת אתרים שנלכדו המראים דמיון גרוע לאתרים על היעד. השיטה המבוססת על שימוש ב-Integrase-Defective Lentiviral Vector (IDLV) פחות רגישה וסביר להניח שתפספס אתרים רבים מחוץ למטרה17. שיטות אחרות באתרן כמו DSBCapture, BLESS ו-BLISS 18,19,20 מערבות תאים קבועים ומסמנות DSBs ישירות, אולם הן מוגבלות על ידי התלות שלהן בלכידת DSB מיידית והיעדר DNA אקסוגני. Digenome-seq21, שיטת מבחנה, והעשרה סלקטיבית וזיהוי של קצוות DNA גנומיים מתויגים על ידי ריצוף (SITE-seq)22 שניהם מספקים פתרונות ריצוף אך יש להם מגבלות ברעשי רקע וניתוח חד-קצה, בהתאמה. גילוי מחוץ ליעדי Cas באתרם ואימות על ידי ריצוף (Discover-Seq)23 מציע זיהוי in vivo ובאתרו של פעילות Cas9 באמצעות קשירת MRE11, אך מזהה רק DSBs הקיימים בזמן הכנת הדגימה24. לבסוף, הסקת עריכות CRISPR (ICE) משתמשת בגישה ביואינפורמטית כדי לנתח בצורה חזקה עריכות CRISPR באמצעות נתוני Sanger25.
מאמר זה מתאר נוהל מפורט למעגל לדיווח חוץ גופי על השפעות מחשוף על ידי ריצוף (CIRCLE-seq): טכניקה במבחנה הממפה ברגישות וללא משוא פנים את הפעילות מחוץ למטרה של נוקלאז Cas9 במתחם עם ה-gRNA המעניין26. גישה זו מתחילה בתרבית התאים המעניינים ובידוד ה-DNA, ולאחר מכן גזירה אקראית באמצעות אולטרסאונד ממוקד, ולאחר מכן טיפול באקסונוקלאז וליגאז. תהליך זה מייצר בסופו של דבר מולקולות DNA דו-גדיליות מעגליות, אשר מטוהרות לאחר מכן באמצעות טיפול ב-DNase בטוח לפלסמיד. DNA מעגלי זה נחשף לאחר מכן לקומפלקס Cas9-gRNA, אשר מתבקע באתרי מחשוף מיועדים ולא מכוונים, ומשאיר אחריו קצוות DNA חשופים המשמשים כמצעים לקשירת מתאם Illumina. תהליך זה מייצר ספרייה מגוונת של DNA גנומי (gDNA) המכילה את שני הקצוות של כל DSB המושרה על ידי נוקלאז, ומבטיחה שלכל קריאה יש את כל המידע הדרוש לכל אתר מחשוף. זה מאפשר שימוש ברצף Illumina עם דרישות כיסוי ריצוף נמוכות יותר, מה שמבדיל את CIRCLE-seq משיטות דומות אחרות שהוזכרו לעיל. חשוב לציין שבעוד של-CIRCLE-seq יש רגישות גבוהה יותר מחוץ למטרה מאשר לפרוטוקולים אחרים כשיטת מבחנה , זה בא על חשבון חיובים כוזבים גבוהים יותר בשל היעדר הנוף האפיגנטי הקיים בשיטות אחרות כגון GUIDE-seq16. בנוסף, תיקון DNA DSB והמנגנון הקשור אליו אינם קיימים ב-CIRCLE-seq, מבטלים אינדלים או תיקון תקין שאחרת היו נצפים.
בנוסף לתיאור הפרוטוקול שלב אחר שלב לביצוע CIRCLE-seq, הפרוטוקול מאומת על ידי זיהוי אתרי מחשוף לא מכוונים ברחבי הגנום של CRISPR-Cas9 המתרחשים במהלך השינוי של לוקוס AAVS1 , כדוגמה. פרוטוקול קל לביצוע זה מספק הוראות מפורטות, החל מתרבית של תאי גזע פלוריפוטנטיים מושרים (iPSCs) ובידוד gDNA ועד למחזור gDNA, מחשוף Cas9-gRNA, הכנת ספרייה, ריצוף וניתוח צנרת. בהתחשב בדרישות כיסוי הריצוף הנמוכות, CIRCLE-seq זמין לכל מעבדה עם גישה לריצוף מהדור הבא.
Protocol
פרטי הריאגנטים, החומרים המתכלים והציוד המשמשים למחקר זה מפורטים בטבלת החומרים.
1. תרבית תאים (5 ימים)
- כלול בקרה שלילית לאורך פרוטוקול זה. הכן מספיק תאים ל-25 מיקרוגרם נוספים של gDNA (~2.0e7 תאים לדגימה).
- תרבית iPSCs על פי הפרוטוקולשנקבע 27. אסוף את התאים והשהה אותם מחדש ב -10 מ"ל של PBS. פיפטה דגימה של 6 מיקרוליטר של תאים והשעיה ביחס של 1:1 עם טריפן כחול. השתמש במונה תאים אוטומטי כדי לספור את הדגימה.
- בחר 2 x 107 תאים לכל צינור, ואז סובב מטה ב -300 x גרם למשך 3 דקות ב -25 מעלות צלזיוס (טמפרטורת החדר (RT)). זה מספיק עבור שכפולים מרובים. פיפטה והשליכו את הסופרנטנט.
2. בידוד DNA גנומי (יום אחד)
- השתמש בערכת טיהור ה-DNA הזמינה מסחרית כדי לבודד את ה-gDNA, בהתאם להוראות היצרן:
- הוסף 200 מיקרוליטר של PBS לצינור חרוטי של 15 מ"ל המכיל את גלולת התא והשהה מחדש. לאחר מכן, פיפטה 3 מ"ל של מאגר ליזה של תאים ו-15 מיקרוליטר של פרוטאינאז K לתוך הצינור. הפוך את הצינור 25 פעמים כדי לערבב היטב. יש להניח את השפופרת בשייקר אמבט מים המכוון ל-55 מעלות צלזיוס ו-150 סל"ד למשך 3 שעות, או למשך הלילה לקבלת תפוקת DNA אופטימלית.
- פיפטה ב-15 מיקרוליטר של RNase A. הפוך 25 פעמים. מניחים באמבט מים בטמפרטורה של 37 מעלות צלזיוס למשך שעה.
- מצננים את הדגימה על קרח למשך 5 דקות. לאחר מכן, הוסף 1 מ"ל של תמיסת משקעים חלבון, מערבולת במהירות גבוהה למשך 20 שניות, וצנטריפוגה ב-2000 x גרם למשך 10 דקות ב-RT. חלבונים צריכים ליצור כדור גלוי וקומפקטי בתחתית הצינור. אם הגלולה אינה נראית לעין, דגרו את הדגימה על קרח למשך 5 דקות נוספות וצנטריפוגה שוב.
- הוסף 3 מ"ל של 100% איזופרופנול לצינור חרוטי חדש של 15 מ"ל. פיפטה בזהירות את הסופרנטנט משלב 2.1.3 לתוך הצינור. הפוך את הצינור 50 פעמים לערבוב, ואז צנטריפוגה בחום של 2000 x גרם וטמפרטורת החדר למשך 3 דקות. מבלי להפריע לכדור ה-DNA, שאפו בזהירות את הסופרנטנט באמצעות פיפטת פסטר המחוברת למלכודת ואקום והפכו את הצינור על מגבון נקי ונטול מוך.
- פיפטה 3 מ"ל של 70% אתנול (v/v) על כדור ה-DNA והפוך 10 פעמים לכביסה. צנטריפוגה ב-2,000 x גרם למשך 3 דקות ב-RT. לאחר מכן, שפכו בזהירות את הסופרנטנט.
- שמור את הצינור פתוח ואפשר לגלולת ה-DNA המתקבלת להתייבש למשך 30 דקות, וודא שכל האתנול התאדה במלואו. פיפטה ב-50 מיקרוליטר של תמיסת הידרציה של DNA ומערבבים היטב על ידי פיפטינג עדין.
- ממיסים את ה-DNA על ידי הנחת הדגימה בשייקר אמבט מים בטמפרטורה של 65 מעלות צלזיוס למשך שעה, ולאחר מכן השאירו את הדגימה בחוץ ב-RT למשך הלילה. צנטריפוגה את הדגימה ב-RT ו-2,000 x g למשך דקה אחת והשתמש בערכת הבדיקה dsDNA BR עם צינורות קשורים כדי לכמת את ה-DNA המבודד עם הפלואורומטר.
3. הכנת gRNA (7 ימים)
- להזמין את ה-gRNA הסינתטי המעניין ממקור מסחרי (ראה טבלת חומרים). פרוטוקול זה תואם גם ל-crRNA/tracrRNA.
4. בדיקת מחשוף gRNA במבחנה
הערה: כאן, נעשה שימוש במטרה בגן AAVS1 . כדי למקד גנים אחרים מעניינים, תכנן פריימרים (טבלה 1) כדי להגביר את אזור היעד ולהחליף את הפריימרים בשלבים הבאים בפריימרים מותאמים אישית.
- הכן את תגובת ה-PCR: מערבבים 25 מיקרוליטר של Phusion Hot Start Flex 2x Master Mix (ריכוז סופי 1x), 0.5 מיקרוליטר של פריימר AAVS1 F (ריכוז סופי 0.1 מיקרומטר), 0.5 מיקרוליטר של פריימר AAVS1 R (ריכוז סופי 0.1 מיקרומטר), 5 מיקרוליטר של gDNA (100 ננוגרם, 20 ננוגרם/מיקרוליטר, משלב 2.1.6), ו-19 מיקרוליטר של H2O ללא נוקלאז (נפח כולל: 50 מיקרוליטר).
- השתמש בפרמטרים התרמו-ציקלרים הבאים: דנטורציה: 98 מעלות צלזיוס למשך 2 דקות (מחזור אחד), דנטורציה: 98 מעלות צלזיוס למשך 10 שניות (10 מחזורים), חישול: 72-62 מעלות צלזיוס (-1 מעלות צלזיוס למחזור) למשך 15 שניות (10 מחזורים), הארכה: 72 מעלות צלזיוס למשך 30 שניות (10 מחזורים), דנטורציה, 98 מעלות צלזיוס למשך 10 שניות (30 מחזורים). חישול: 65 מעלות צלזיוס למשך 15 שניות (30 מחזורים), הארכה: 72 מעלות צלזיוס למשך 30 שניות (30 מחזורים), הארכה סופית: 72 מעלות צלזיוס למשך 5 דקות (מחזור אחד), החזקה: 4 מעלות צלזיוס ללא הגבלת זמן.
- השתמש בחרוזי AMPure XP כדי לטהר את התוצר של תגובת ה-PCR. ראשית, פיפטה בנפח של פי 1.8, או 90 מיקרוליטר, של חרוזי XP לתוצר ה-PCR. פיפטה עשר פעמים לערבב היטב. השאירו את התערובת ב-RT למשך 5 דקות לדגירה.
- בעזרת מתלה מגנטי, הפרד את החרוזים מהתמיסה על ידי הנחת לוחית תגובת ה-PCR על המגנט למשך 3 דקות. פיפטה את התמיסה שנוקה והשליך. הוסף 200 מיקרוליטר של 80% אתנול (v/v) לחרוזים, דגר למשך 30 שניות ולאחר מכן הסר את האתנול. חזור על שלב כביסה זה פעמיים כדי להבטיח הסרה מלאה של האתנול.
- הניחו לדגימות להתייבש באופן טבעי במשך 3 דקות על ידי הנחת הצלחת על המגנט. הסר את הצלחת מהמגנט והוסף 40 מיקרוליטר של מאגר TE, pH 8.0. מערבבים על ידי פיפטינג למעלה ולמטה עשר פעמים. אפשר לדגימה לשבת ב-RT למשך 2 דקות.
- הנח את לוחית תגובת ה-PCR על המגנט למשך דקה נוספת. לאחר דקה אחת, העבירו את הסופרנטנט לצלחת חדשה. מדוד את תפוקת ה-PCR המטוהר באמצעות ספקטרופוטומטר, ונתח אותו ב-TapeStation באמצעות פס צינור אופטי עם מכסה פס צינור אופטי, יחד עם ריאגנטים D1000 ScreenTape ברגישות גבוהה ורגישות גבוהה D1000 (סולם ומאגר), בהתאם להוראות היצרן. אחסן את הדגימה המוכנה בטמפרטורה של -20 מעלות צלזיוס עד מספר חודשים.
- יש לדלל חלבון נוקלאז Cas9 ל-1 מיקרומטר באופן הבא: מערבבים 2 מיקרוליטר של 10x Cas9 buffer (ריכוז סופי 1x), 1 מיקרוליטר של Cas9 Nuclease, S. pyogenes (ריכוז סופי 1 מיקרומטר), ו-17 מיקרוליטר של H2O נטול נוקלאז בנפח כולל של 20 מיקרוליטר.
- בצע הליך נטול RNase כדי למנוע פירוק gRNA. דילול gRNA (משלב 3.1) ל-3 מיקרומטר ב-H2O לנפח כולל של 10 מיקרוליטר.
הערה: השתמש בנוסחה הבאה כדי להעריך את המשקל המולקולרי של gRNA: משקל מולקולרי של ssRNA (g/mol) = (אורך ssRNA (nt) x 321.47 g/mol) + 18.02 g/mol. לעיון, gRNA באורך 104 nt ב-3 μM הוא כ-100 ננוגרם/מיקרוליטר.
5. גזירת DNA (3 שעות)
- הכן את ה-ME220 על ידי ביות זרוע הבקרה. לאחר מכן, מלאו את המאגר ב- H2O מטוהר דה-יוניזציה. במחשב הנייד של תחנת הבקרה, גש ל-Water Works ולחץ על מילוי. התאם את הטמפרטורה ל-4.5 מעלות צלזיוס.
- העבר 25 מיקרוגרם של gDNA לתוך מיקרו-צינור (מיקרו-צינור-130 AFA סיבים בורג-פקק). לאחר מכן, מלאו את הצינור לנפח כולל של 130 מיקרוליטר עם 1x TE. השתמש בתנאים הבאים כדי לגזור את ה-DNA לאורך ממוצע של כ-300 bp: הגדר את משך הזמן ל-10 שניות; שיא הספק ל-70; מקדם מכס % עד 20; מחזורים/פרץ ל-50; כל אלה יגדירו אוטומטית את ה-Avg Power ל-14.0.
6. טיהור DNA גנומי גזוז (1 שעות)
- חלקו את ה- DNA הגנומי הגזוז לשני חלקים של 65 מיקרוליטר כל אחד. טהר באמצעות פי 1.8 מנפח חרוזי XP (117 מיקרוליטר), בהתאם להליך המתואר בשלבים 4.3-4.6. העבירו את הסופרנטנט ללוחית PCR חדשה ומדדו את הכמות באמצעות ספקטרופוטומטר.
- הפעל 1 מיקרוליטר של ה-gDNA הגזוז הגזוז על TapeStation, על פי הוראות היצרן, כדי להבטיח שה-gDNA נגזז להפצה רחבה של כ. 300 bp. במידת הצורך, אחסן את ה-gDNA הגזוז בטמפרטורה של -20 מעלות צלזיוס עד מספר חודשים.
7. הכנת ספריית CIRCLE-seq (3 ימים)
- חישול מתאם סיכת ראש
- השעו מחדש את oSQT1288 (טבלה 1), מתאם סיכת הראש, לריכוז סופי של 100 מיקרומטר ב-1x TE.
- בצע את חישול המתאם באופן הבא: מערבבים 40 מיקרוליטר של oSQT1288 (ריכוז סופי 40 מיקרומטר), 10 מיקרוליטר של 10x STE (ריכוז סופי 1x), ו-50 מיקרוליטר של H2O ללא נוקלאז לנפח כולל של 100 מיקרוליטר.
- השתמש בפרמטרי החישול הבאים: 95 מעלות צלזיוס למשך 5 דקות, -1 מעלות צלזיוס לדקה למשך 70 מחזורים, החזק ב-4 מעלות צלזיוס ללא הגבלת זמן.
- בצע תיקון סיום . השתמשו בערכת ההכנה לספריית HTP ללא PCR והכינו את ה-Master Mix לתיקון הסופי.
- יש לערבב 8 מיקרוליטר של H2O נטול נוקלאז, 7 מיקרוליטר של מאגר תיקון קצה 10x (ריכוז סופי 1x) ו-5 מיקרוליטר של תערובת אנזימים לתיקון קצה (נפח סופי 20 מיקרוליטר של Total End-Repair Master Mix).
- פיפטה 20 מיקרוליטר של מאסטר תיקון הקצה מתערבבים בדגימת ה-gDNA הגזוז משלבים 4.3-4.6. מערבבים 20 מיקרוליטר של End-Repair Master Mix עם 50 מיקרוליטר של gDNA גזוז לנפח סופי של 70 מיקרוליטר.
- מניחים את התערובת בתרמוסייקלר בטמפרטורה של 20 מעלות צלזיוס למשך 30 דקות, ואז מחזיקים בטמפרטורה של 4 מעלות צלזיוס ללא הגבלת זמן.
- הוסף פי 1.7 נפחים, או 120 מיקרוליטר של חרוזי XP ובצע את שלבי הטיהור משלבים 4.3-4.6. עם 42 מיקרוליטר TE, pH 8.0. ודא שהחרוזים נשארים בתמיסה לשלב הבא.
- בצע זנב A. באמצעות ערכת ההכנה לספריית HTP (ללא PCR), הכינו את תערובת המאסטר לזנב A.
- מערבבים 5 מיקרוליטר של מאגר זנב A 10x (ריכוז סופי 1x) ו-3 מיקרוליטר של אנזים זנב A (נפח סופי כולל של 8 מיקרוליטר של תערובת מאסטר זנב A).
- פיפטה 8 מיקרוליטר של תערובת מאסטר זנב A לכל דגימת DNA המכילה חרוזים משלב 7.2.4, ערבוב של 8 מיקרוליטר של תערובת מאסטר זנב A ל-42 מיקרוליטר של DNA מתוקן קצה המכיל חרוזים (נפח סופי כולל של 50 מיקרוליטר). מניחים בתרמוסייקלר בטמפרטורה של 30 מעלות צלזיוס למשך 30 דקות. החזק ב-4 מעלות צלזיוס ללא הגבלת זמן.
- פיפטה 1.8 נפחים, או 90 מיקרוליטר, של תמיסת PEG/NaCl SPRI (רכיב של ערכת ההכנה של ספריית HTP (ללא PCR; 96 תגובות)) ל-DNA זנב A. טהר את ה-DNA עם זנב A לפי שלבים 4.3-4.6. יש לסלק את ה-DNA עם זנב A ב-30 מיקרוליטר של TE, pH 8.0. שמור את החרוזים בתמיסה לשלב הבא.
- בצע קשירת מתאם. באמצעות ערכת HTP Library Preparation (ללא PCR), הכינו את Adapter Ligation Master Mix.
- מערבבים 10 מיקרוליטר של מאגר קשירה פי 5 (ריכוז סופי 1x), 5 מיקרוליטר של DNA ליגאז ו-5 מיקרוליטר של מתאם סיכת ראש מחוסל (40 מיקרומטר) משלב 7.1.3. ודא את הריכוז הסופי של 4 מיקרומטר עבור תערובת מאסטר כוללת של קשירת מתאם של 20 מיקרוליטר.
- פיפטה 20 מיקרוליטר של תערובת מאסטר קשירת מתאם לכל דגימת DNA מסולסלת המכילה חרוזים משלב 7.3.3 (נפח סופי כולל של 50 מיקרוליטר לדגימה).
- מניחים בתרמו-סייקלר בטמפרטורה של 20 מעלות צלזיוס למשך שעה. החזק ב-4 מעלות צלזיוס ללא הגבלת זמן.
- העבירו 1x נפחים, או 50 מיקרוליטר, של תמיסת PEG/NaCl SPRI ל-DNA הקשור במתאם וטהרו לפי שלבים 4.3-4.6. יש להוציא 30 מיקרוליטר TE, pH 8.0, ולשפוך את הסופרנטנטים לתוך צלחת PCR חדשה עם חצאית למחצה. שלב וכימת את ה-DNA באמצעות בדיקת dsDNA BR. במידת הצורך, יש לאחסן DNA מטוהר הקשור במתאם למשך עד חודש בטמפרטורה של -20 מעלות צלזיוס.
- הכן את תערובת המאסטר של Lambda Exonuclease/Exonuclease I (E. coli) Master Mix (מתפקד בסילוק DNA חד-גדילי או דו-גדילי ללא מתאמים הקשורים לשני הקצוות).
- קח 1 מיקרוגרם של DNA קשור מתאם משלב 7.4.4, ודלל אותו ל-40 מיקרוליטר. מערבבים 5 מיקרוליטר של 10x Exonuclease I מאגר תגובה (ריכוז סופי 1x), 4 מיקרוליטר של Lambda Exonuclease (ריכוז סופי 0.4 U/μL), ו-1 מיקרוליטר של Exonuclease I (E. coli) (ריכוז סופי 0.4 U/μL) לנפח כולל של Lambda Exonuclease/Exonuclease I Master Mix של 10 מיקרוליטר.
- פיפטה 10 מיקרוליטר של Lambda Exonuclease/Exonuclease I Master Mix ל-40 מיקרוליטר (1 מיקרוגרם) של DNA קשור במתאם (נפח כולל של 50 מיקרוליטר). מניחים בתרמו-סייקלר בטמפרטורה של 37 מעלות צלזיוס למשך שעה, ולאחר מכן 75 מעלות צלזיוס למשך 10 דקות. החזק ב-4 מעלות צלזיוס ללא הגבלת זמן.
- פיפטה פי 1.8 נפחים, או 90 מיקרוליטר, של חרוזי XP ל-DNA שטופל ב-Lambda Exonuclease/Exonuclease I. טהר לפי ההוראות שניתנו בשלבים 4.3-4.6. נשטף ב-40 מיקרוליטר של TE, pH 8.0. ודא שהחרוזים נשארים בתמיסה לשלב האנזימטי הבא.
- יש לטפל באנזים USER וב-T4 Polynucleotide Kinase (PNK). הכן את USER/T4 PNK Master Mix (נדרש לשחרור ה-4-bp ולהכנת קצוות ה-DNA המוכנים לקשירה הדרושים לתגובת הקשירה הבאה).
- מערבבים 5 מיקרוליטר של 10x T4 DNA Ligase Buffer (ריכוז סופי 1x), 3 מיקרוליטר של אנזים USER (ריכוז סופי 0.05 U/μL), ו-2 מיקרוליטר של T4 PNK (ריכוז סופי 0.4 U/μL), לנפח כולל של תערובת המאסטר USER/PNK של 10 מיקרוליטר.
- פיפטה 10 מיקרוליטר של USER Enzyme/T4 PNK Master Mix ל-40 מיקרוליטר של דגימת DNA שטופלה בלמבדה ואקסונוקלאז I המכילה חרוזים משלב 7.5.3 לנפח כולל של 50 מיקרוליטר. מניחים בתרמו-סייקלר בטמפרטורה של 37 מעלות צלזיוס למשך שעה. החזק ב-4 מעלות צלזיוס ללא הגבלת זמן.
- פיפטה פי 1.8 נפחים, או 90 מיקרוליטר, של תמיסת PEG/NaCl SPRI ל-DNA שטופל ב-USER/T4 PNK ומטהרת לפי שלבים 4.3-4.6. הוצאה ב-35 מיקרוליטר של TE, pH 8.0. שפכו את הסופרנטנט לתוך צלחת PCR חדשה עם חצאית למחצה. שלב וכימת את ה-DNA באמצעות מבחן dsDNA HS.
- לבצע מעגל תוך מולקולרי. הכן את תערובת מאסטר המחזור.
- מערבבים 8 מיקרוליטר של H2O ללא נוקלאז, 10 מיקרוליטר של 10x T4 DNA ligase buffer (ריכוז סופי 1x), ו-2 מיקרוליטר של T4 DNA ligase (ריכוז סופי 8 U/μL), לנפח כולל של Circularization Master Mix של 20 מיקרוליטר.
- פיפטה 20 מיקרוליטר של תערובת מאסטר סירקולריזציה ל-500 ננוגרם של DNA שטופל ב-USER/PNK משלב 7.6.3. יש לדלל 500 ננוגרם של DNA שטופל ב-USER/PNK ב-80 מיקרוליטר, ולאחר מכן להוסיף 20 מיקרוליטר של Circularization Master Mix (נפח כולל של 100 מיקרוליטר). דגירה בתרמו-סייקלר בטמפרטורה של 16 מעלות צלזיוס למשך 16 שעות (לילה).
- הוסף נפח 1x, או 100 מיקרוליטר, של חרוזי XP ל-DNA המעגלי וטהר לפי שלבים 4.3-4.6. נשטף ב-38 מיקרוליטר של TE, pH 8.0. שפכו את הסופרנטנט לתוך צלחת PCR חדשה עם חצאית למחצה.
- טפל ב-DNA תלוי ATP בטוח לפלסמיד. הכן את תערובת המאסטר של DNase תלויה ב-ATP בטוחה לפלסמיד (נדרשת לפירוק ה-DNA הליניארי השיורי).
- מערבבים 5 מיקרוליטר של מאגר תגובה בטוח לפלסמיד פי 10 (ריכוז סופי 1x), 2 מיקרוליטר של ATP (ריכוז סופי 1 מ"מ), ו-5 מיקרוליטר של DNase תלוי ATP בטוח לפלסמיד (ריכוז סופי 1 U/μL), לנפח כולל של תערובת המאסטר הבטוחה לפלסמיד של 12 מיקרוליטר.
- פיפטה 12 מיקרוליטר של תערובת מאסטר DNase תלויה ב-ATP ל-38 מיקרוליטר של DNA מחזורי משלב 7.7.3 (נפח כולל של 50 מיקרוליטר). דגירה בתרמו-סייקלר בטמפרטורה של 37 מעלות צלזיוס למשך שעה, ולאחר מכן 70 מעלות צלזיוס למשך 30 דקות. החזק ב-4 מעלות צלזיוס ללא הגבלת זמן.
- פיפטה 1x נפחים, או 50 מיקרוליטר, של חרוזי XP לתוך ה-DNA שטופל ב-DNase תלוי ATP בטוח לפלסמיד ומטהרים לפי שלבים 4.3-4.6. הוצאה ב-15 מיקרוליטר של TE, pH 8.0. שפכו את הסופרנטנט לתוך צלחת PCR חדשה עם חצאית למחצה.
- שלב את ה-DNA וכימת באמצעות מבחן dsDNA HS. במידת הצורך, אחסן את ה-DNA המעגלי ב-20 מעלות צלזיוס עד מספר חודשים.
8. חיתוך gDNA מטוהר אנזימטית, מחזורי במבחנה (2 שעות)
- בצע מחשוף במבחנה עם קומפלקס Cas9:gRNA. מכינים את תערובת המאסטר של מחשוף במבחנה . מערבבים 5 מיקרוליטר של 10x Cas9 Buffer (ריכוז סופי 1x), 4.5 מיקרוליטר של S. Pyogenes Cas9 (ריכוז סופי 90 ננומטר) ו-1.5 מיקרוליטר של gRNA (ריכוז סופי 90 ננומטר) לקבלת נפח Cleavage Master Mix כולל של 11 מיקרוליטר.
- שמור את תערובת מאסטר המחשוף ב-RT למשך 10 דקות ליצירת קומפלקסים Cas9:gRNA RNP.
- לדלל 125 ננוגרם של DNA שטופל ב-DNA בטוח לפלסמיד משלב 7.8.3 לנפח סופי של 39 מיקרוליטר. לאחר מכן, הוסף 11 מיקרוליטר של Cleavage Master Mix ל-DNA שטופל ב-DNA בטוח לפלסמיד של 39 מיקרוליטר לנפח כולל של 50 מיקרוליטר.
הערה: כלול דגימת בקרה שלילית בשלב זה, הכוללת DNA מעגלי מעורבב עם מאגר Cas9, ללא קומפלקס Cas9:gRNA. - דגירה בתרמו-סייקלר למשך שעה בטמפרטורה של 37 מעלות צלזיוס. יש לשמור על טמפרטורה של 4 מעלות צלזיוס ללא הגבלת זמן. הוסף 50 מיקרוליטר (נפח 1x) של חרוזי XP ל-DNA השסוע במבחנה וטהר את ה-DNA לפי שלבים 4.3-4.6. נשטף ב-42 מיקרוליטר של מאגר TE, pH 8.0. ודא שהחרוזים נשארים בתמיסה לשלב הבא.
9. הכנת ספריית הרצף של הדור הבא (4 - 6 שעות)
- בצע זנב A. הכן את תערובת המאסטר A-tailing.
- מערבבים 5 מיקרוליטר של מאגר זנב A 10x (ריכוז סופי פי 1), ו-3 מיקרוליטר של אנזים זנב A (הנפח הכולל של תערובת המאסטר של זנב A הוא 8 מיקרוליטר).
- פיפטה 8 מיקרוליטר של תערובת מאסטר זנב A עד 42 מיקרוליטר של דגימת DNA מסולסלת המכילה חרוזים משלב 8.4 (נפח כולל של 50 מיקרוליטר). מניחים בתרמוסייקלר למשך 30 דקות בחום של 30 מעלות צלזיוס. החזק ב-4 מעלות צלזיוס ללא הגבלת זמן.
- פיפטה פי 1.8 נפחים של תמיסת PEG/NaCl SPRI, או 90 מיקרוליטר, ל-DNA בעל זנב A ומטהרים את ה-DNA לפי שלבים 4.3-4.6. נשטף ב-25 מיקרוליטר של TE, pH 8.0. הקפד לשמור את החרוזים בתמיסה לשלב הבא.
- בצע קשירת מתאם. הכן את Adapter Ligation Master Mix.
הערה: יש להכין מינויים חד פעמיים של מתאמי NEB על מנת למנוע היווצרות של דימרים של מתאם הנגרמים על ידי הידרוליזה הנגרמת על ידי הקפאה-הפשרה של ה-3' T'.- מערבבים 10 מיקרוליטר של מאגר קשירה פי 5 (ריכוז סופי 1x), 5 מיקרוליטר של DNA ליגאז ו-10 מיקרוליטר של מתאם לריצוף (הריכוז הסופי הוא 3 מיקרומטר), בסך הכל 25 מיקרוליטר.
- פיפטה 25μL של תערובת המאסטר של קשירת המתאם לתוך 25 מיקרוליטר של דגימת DNA עם זנב A המכילה חרוזים משלב 9.1.3. מניחים בתרמוסייקלר למשך שעה בחום של 20 מעלות צלזיוס. החזק ב-4 מעלות צלזיוס ללא הגבלת זמן.
- פיפטה 1x נפחים, או 50 מיקרוליטר, של תמיסת PEG/NaCl SPRI ל-DNA הקשור במתאם ומטהרים את ה-DNA לפי שלבים 4.3-4.6. נפלט ב-47 מיקרוליטר של TE, pH 8.0. ודא שהחרוזים נשארים בתמיסה לשלב האנזימטי הבא.
- בצע טיפול באנזים USER (יוצר פער נוקלאוטידים בודד בשאריות אורציל).
- הוסף 3 מיקרוליטר של אנזים USER (כלול בערכת פריימרים של אינדקס כפול) לדגימת ה-DNA הקשורה במתאם המכילה חרוזים משלב 9.2.3. דגירה בטמפרטורה של 37 מעלות צלזיוס למשך 15 דקות.
- הוסף 35 מיקרוליטר (נפח פי 0.7) של תמיסת PEG/NaCl SPRI ל-DNA שטופל באנזים USER וטהר לפי שלבים 4.3-4.6. נשטף ב-20 מיקרוליטר של מאגר TE, pH 8.0. העבירו את הסופרנטנט לצלחת PCR חדשה עם חצאית למחצה ומדדו את ריכוז ה-DNA באמצעות בדיקת dsDNA HS. הריכוז הצפוי צריך להיות כ. 2-5 ננוגרם/מיקרוליטר.
- (אופציונלי) לפני שתמשיך לשלב הבא, ניתן לבצע בחירת גודל DNA באמצעות PippinHT. השתמש בקלטת 1.5% PippinHT בטווח מידות של 250-850 bp. ניתן להשתמש ישירות בדגימות המתקבלות ב-PCR בשלב הבא.
- בצע PCR להוספת ברקוד
הערה: ודא ששילובי רצף הפריימר שנבחרו עבור כל דגימה הם ייחודיים. במידת האפשר, כל דגימה צריכה לכלול ברקודים ייחודיים של i5 ו-i7.- הכן את PCR Master Mix כדי להוסיף ברקודים בעלי אינדקס מזווג. מערבבים 5 מיקרוליטר של H2O ללא נוקלאז, 25 מיקרוליטר של 2x HotStart Ready Mix (ריכוז סופי 1x), 5 מיקרוליטר של פריימר i5 (ריכוז סופי 1 מיקרומטר), ו-5 מיקרוליטר של פריימר i7 (ריכוז סופי 1 מיקרומטר) (נפח תערובת מאסטר כולל של 40 מיקרוליטר).
- פיפטה 40 מיקרוליטר של PCR Master Mix ל-10 מיקרוליטר של DNA מטוהר שטופל באנזים USER (כ-20 ננוגרם) משלב 9.3.2 (נפח כולל של 50 מיקרוליטר).
- בחר את תנאי ה-PCR הבאים: דנטורציה: 98 מעלות צלזיוס למשך 45 שניות למשך מחזור אחד, דנטורציה: 98 מעלות צלזיוס למשך 15 שניות למשך 20 מחזורים, חישול: 65 מעלות צלזיוס למשך 30 שניות למשך 20 מחזורים, הארכה: 72 מעלות צלזיוס למשך 30 שניות למשך 20 מחזורים, הארכה סופית: 72 מעלות צלזיוס למשך דקה אחת למשך מחזור אחד, החזקה: 4 מעלות צלזיוס ללא הגבלת זמן.
- הוסף 0.7x נפחים, או 35 מיקרוליטר, של חרוזי XP למוצר ה-PCR וטהר לפי שלבים 4.3-4.6. יש לנטרל 30 מיקרוליטר של TE, pH 8.0. שפכו את הסופרנטנט לתוך צלחת PCR חדשה עם חצאית למחצה. במידת הצורך, אחסן את ה-DNA המעגלי ב-20 מעלות צלזיוס עד מספר חודשים.
הערה: הפעל דגימה של ה-PCR ב-Tapestation כדי לשלוט באיכות הספרייה ולהעריך את היווצרותם של דימרים של מתאמים. אם זוהו דימרים של מתאם, חזור על שלב 9.5.4.
10. כימות ספריות CIRCLE-seq על ידי PCR דיגיטלי טיפתי (dd_PCR) (6 שעות)
הערה: ניתן לבצע כימות גם באמצעות qPCR, Tapestation או שיטה דומה.
- התחל עם 5 מיקרוליטר של DNA מהספרייה (שלב PCR 9.5.4) מעורבב היטב עם 45 מיקרוליטר של TE נטול נוקלאז, ולאחר מכן בצע דילול סדרתי של 1:10 של כל דגימה בנפחים של 50 מיקרוליטר, הנעים בין 10-1 ל-10-8 דילול.
- הגדר את פתרון המלאי של dd_PCR Master Mix. מערבבים 11 מיקרוליטר של תערובת 2x dd_PCR לבדיקות (ריכוז סופי 1x), 0.055 מיקרוליטר של בדיקה oSQT1310 (ריכוז סופי 250 ננומטר), 0.055 מיקרוליטר של בדיקה oSQT1311 (ריכוז סופי 250 ננומטר), 0.099 מיקרוליטר של פריימר oSQT1274 (ריכוז סופי 450 ננומטר), 0.099 מיקרוליטר של פריימר oSQT1275 (ריכוז סופי 450 ננומטר), ו-6.292 מיקרוליטר של H2O ללא נוקלאז, לנפח מאסטר מיקס dd_PCR כולל של 17.6 מיקרוליטר. הכן תערובת מאסטר לכל הדגימות כדי להבטיח שהנפחים מספיקים לפיפטינג מדויק.
- בדוק את שלושת הדילולים הנמוכים ביותר (10-6,10-7 ו-10-8) בכפול (בצלחת של 96 בארות). יש להשתמש בפקד שאינו תבנית (NTC).
- פיפטה 17.6 מיקרוליטר של תערובת מאסטר dd_PCR לכל דגימה באופן הבא: מערבבים 17.6 מיקרוליטר עם 4.4 מיקרוליטר מהדגימה (הוספת H2O נטול נוקלאז לבאר עם NTC), לנפח כולל של 22 מיקרוליטר. אוטמים את הצלחת, ואז צנטריפוגה ב 2000 x גרם למשך דקה אחת ב- RT.
- בצע יצירת טיפות, מחזור תרמי וניתוח. באמצעות מערכת Droplet Reader PCR, העבר מחסנית DG8 (8 בארות) לתוך מחזיק המחסנית. בשורת השמן של המחסנית, הוצא 70 מיקרוליטר של שמן לייצור טיפות לבדיקות.
הערה: קח 20 מיקרוליטר מהדגימה משלב 10.1.2. והוסף אותו לשורת המדגם של מחסנית 8 הבארות, מכסה את המחסנית באטם הגומי DG8, מכניס אותה לגנרטור הטיפות ואז סוגר אותה כדי להתחיל בתהליך (אוטומטי). בסיום, הסר את המחסנית והעבר 40 מיקרוליטר משורת הטיפות של מחסנית 8 הבארות לתוך צלחת PCR חצי חצאית של 96 בארות, הקפד לבצע פיפטה לאט.- הנח את בלוק החום לתוך אוטם צלחת PX1 PCR. הסילר יתחיל להתחמם עד 180 מעלות צלזיוס כאשר הוא מופעל. הנח את אטם חום נייר הכסף על הצלחת, וודא שהקו האדום נמצא למעלה. הנח את הצלחת לתוך ה-PX1 ולחץ על החותם.
- בחר את תנאי התרמו-ציקלר הבאים: הפעלת אנזימים: 95 מעלות צלזיוס למשך 10 דקות למשך מחזור אחד, דנטורציה: 94 מעלות צלזיוס למשך 30 שניות למשך 40 מחזורים, חישול/הארכה: 60 מעלות צלזיוס למשך דקה למשך 40 מחזורים, השבתת אנזימים: 98 מעלות צלזיוס למשך 10 דקות למשך מחזור אחד, החזקה: 4 מעלות צלזיוס ללא הגבלת זמן.
- בקורא הטיפות, פתח את התוכנה התואמת ובחר אילו בארות יש לקרוא. בחר ABS כסוג הניסוי ו-dd_PCR Supermix עבור בדיקות. בחר Ch1 לא ידוע עבור יעד 1 ו-Ch2 לא ידוע עבור יעד 2. בחר החל ולאחר מכן אישור. הכניסו את הצלחת לקורא הטיפות. עבור ערכת הצבעים, בחר FAM/HEX ולחץ על הפעל.
- נתח את התוצאות dd_PCR. שער את אוכלוסיית הטיפות החיוביות הכפולות באמצעות הבקרה השלילית כהפניה. חשב את ממוצע הערכים הכפולים, ולאחר מכן הכפל במקדם הדילול ובגורם הדילול פי 5 של dd_PCR.
הערה: סך העותקים למיקרוליטר מחושב באופן הבא: סך העותקים למיקרוליטר = ערך ממוצע × 5 × גורם דילול, כאשר 'ערך ממוצע' מייצג את ערך הכימות הממוצע מ-Ch1 ו-Ch2. - שלב את כל הדגימות לספרייה אחת בריכוזים שווי ערך. הספרייה המאגרת 1x צריכה להכיל כ. 4.5 x 109 מולקולות ובעלת נפח כולל של 5 מיקרוליטר.
11. ריצוף מהדור הבא
- שלח את הדגימות לריצוף לסוכנות חיצונית, וודא שרצפי המתאם הנכונים מצוינים .
12. ניתוח נתוני CIRCLE-seq (1 - 3 שעות)
- התקן את Python גרסה 2.7, Burrows-Wheeler Aligner (BWA) ו-SAMtools. הורד את גנום הייחוס (למשל, hg38) מ-http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz.
הערה: אם הגנום של מין היעד אינו זמין, ניתן להפעיל את זרימת העבודה החישובית CIRCLE-seq במצב שאינו תלוי בהפניה. במצב זה, ניתן לדלג על שלב זה. - הורד והתקן את צינור CIRCLE-seq עם הפקודות הבאות: (1) git clone https://github.com/tsailabSJ/circleseq.git, (2) cd circleseq, (3) pip install -r requirements.txt.
- צור קובץ מניפסט בתבנית YAML ( .yaml). להלן מניפסט לדוגמה שניתן להשתמש בו עם מערך הנתונים לדוגמה המסופק בתוכנת CIRCLE-seq כדי לבדוק את זרימת העבודה.
הערה: (1) גנום ייחוס: נתונים/קלט/CIRCLEseq_test_genome.fa; (2) analysis_folder: נתונים/פלט; (3) BWA: BWA; (4) סמול: סמול; (5) read_threshold: <ערך>; (6) window_size: <ערך>; (7) mapq_threshold: <ערך>; (8) start_threshold: <ערך>; (9) gap_threshold: <ערך>; (10) mismatch_threshold: <ערך>; (11) merged_analysis: נכון; (12) דוגמאות: U2OS_EMX1; (13) יעד: GAGTCCGAGCAGAAGAAGAANGG; (14) read1: נתונים/קלט/EMX1.r1.fastq.gz; (15) read2: נתונים/קלט/EMX1.r2.fastq.gz; (16) controlread1: נתונים/קלט/EMX1_control.r1.fastq.gz; (17) controlread2: נתונים/קלט/EMX1_control.r2.fastq.gz; (18) תיאור: U2OS. נעשה שימוש בערכי המניפסט הבאים: read_threshold: 4, window_size: 3, mapq_threshold: 50, start_threshold: 1, gap_threshold: 3, mismatch_threshold: 6 - הגדר את קובץ ה-FASTA של גנום הייחוס, את ספריית הפלט לניתוח ואת הנתיבים לפקודות BWA ו-SAMtools . הגדר את רצפי היעד ואת הנתיבים לקבצי ה-FASTQ המפורקים הן עבור דגימות הנוקלאז והן עבור דגימות הבקרה. ניתן לעבד מספר ניסויים בו זמנית במצב אצווה על ידי הכללת כולם בקובץ מניפסט יחיד.
- בצע את הפקודה הבאה לניתוחים סטנדרטיים מבוססי התייחסות: (1) python /path/to/circleseq.py all - manifest; (2) /path/to/manifest.yaml.
- לחלופין, בצע את הפקודה הבאה עבור ניתוחים סטנדרטיים שאינם מבוססי הפניה: (1) Python /path/to/circleseq.py ללא הפניות - מניפסט; (2) /path/to/manifest.yaml.
- בעת ביצוע הצינור המלא, מצא את תוצאות הפלט של כל שלב output_folder נפרדת המיועדת לשלב ספציפי זה.
תוצאות
כאן, CIRCLE-seq משמש לחקירת אתרי המחשוף המושרים על ידי נוקלאז של Cas9 בקומפלקס עם ה-gRNA שנועד למקד לאתר אינטגרציה של הנגיף הקשור לאדנו 1 (AAVS1) באמצעות DNA מבודד מתאי גזע פלוריפוטנטיים מושרים (iPSCs). gRNA זה תואר בעבר בפרסום שלנו27. כ-25 מיקרוגרם של gDNA בודדו מ-iPSCs, נחתכו באמצעות אולטרסאונד ממוקד, והגודל נבחר באמצעות טיהור חרוזי AMPure XP כדי להניב שברים של כ-300 bps. מ-25 מיקרוגרם זה של DNA, כ-2-5 ננוגרם של DNA עבר בהצלחה מחשוף Cas9:gRNA במבחנה . ההליך כולו מתואר באיור 1.
לאחר הליך CIRCLE-seq וניתוח באמצעות זרימת העבודה החישובית שלנו, הדמיה של כל אתרי המחשוף שזוהו על המטרה ומחוצה לה מוצגת באיור 2A. צינור CIRCLE-seq סיפק גם 'קריאות ממוזגות', שנותחו באמצעות תוכנה סטטיסטית R כדי להניב תרשים מנהטן המציג את אתרי המחשוף המושרים על ידי נוקלאז שזוהו וממופים לאורך כל כרומוזום (איור 2B).
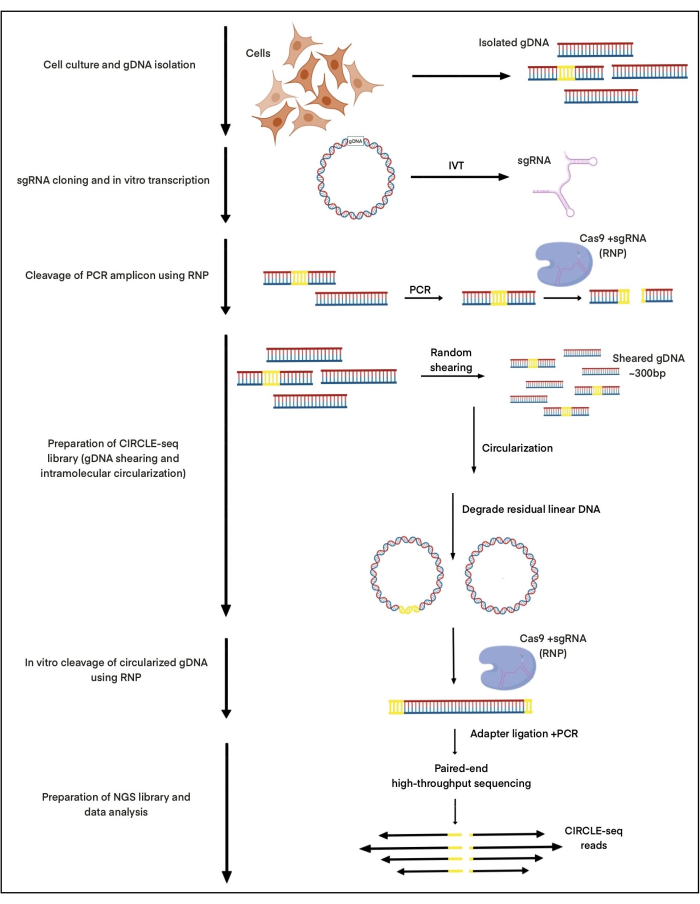
איור 1: סכמות זרימת עבודה של CIRCLE-seq. השלבים העיקריים של הפרוטוקול מצוינים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
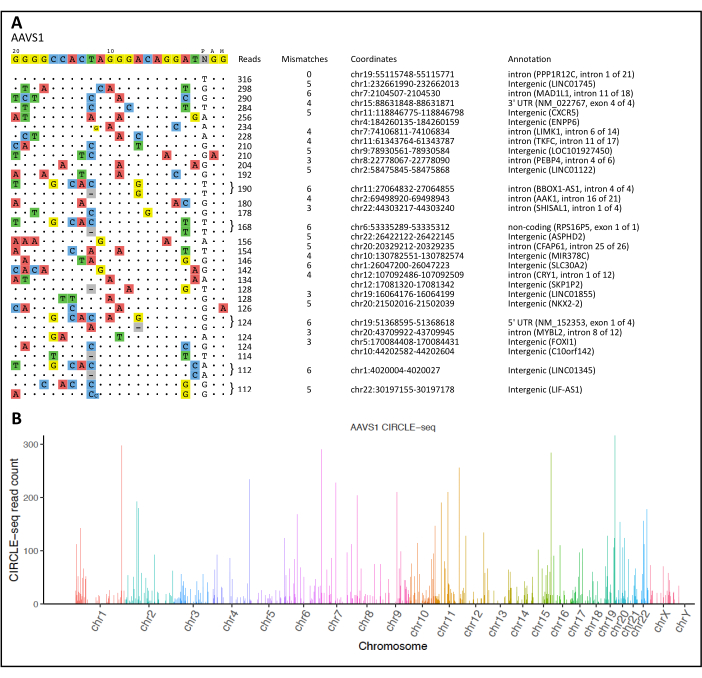
איור 2: ויזואליזציה של CIRCLE-seq ותרשים מנהטן. (A) יישור של אתרים מחוץ למטרה כנגד המטרה המיועדת עבור לוקוס AAVS1. רצף היעד מוצג בחלק העליון, כאשר מחוץ ליעדים מדורגים לפי ספירת קריאה בסדר יורד. הבדלים ברצפי המטרה המקוריים מוצגים על ידי נוקלאוטידים צבעוניים. מוצגת דוגמה של אתרי המחשוף הלא מכוונים המובילים עבור לוקוס AAVS1. (B) תרשים מנהטן הממחיש את אתרי המחשוף הלא מכוונים שזוהו עבור לוקוס AAVS1. גבהי העמודות מייצגים ספירת קריאה עבור כל מיקום כרומוזומלי. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| תחל | רצף (5'-3') | הערות/תיאור |
| AAVS1 RNA מוביל יחיד (sgRNA) | GGGGCCACUAGGGACAGGAU | עבור חלבון פלואורסצנטי של AAVS1 Locus |
| AAVS1 פריימר קדימה | GCTCTGGGCGGAGGAATATG | לבדיקת מחשוף gRNA במבחנה |
| AAVS1 פריימר הפוך | ATTCCCAGGGCCGGTTAATG | לבדיקת מחשוף gRNA במבחנה |
| oSQT1288 | /5Phos/CGGTGGACCGATC /ideoxyU/ATCGGTCCACCGaT | מתאם סיכת ראש CIRCLE-seq |
| oSQT1274 | AATGATACGGCGACCACCGAG | TruSeq F1 |
| oSQT1275 | CAAGCAGAAGACGGCATACGAGAT | TruSeqF2 |
| oSQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | בדיקת TruSeq |
| oSQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | בדיקת TruSeq |
טבלה 1: רצפים של gRNA ופריימרים המשמשים לניתוח CIRCLE-seq של לוקוס AAVS1 .
Discussion
כאן, CIRCLE-seq הוכח כטכניקה בלתי משוחדת ורגישה ביותר לזיהוי DSBs המושרים על ידי נוקלאז על פני הגנום הנובע מהתמקדות בלוקוס AAVS1 ב-gDNA שמקורו ב-iPSCs. אתר AAVS1 בתוך iPSCs ידוע כאתר נמל מבטחים המשמש לעתים קרובות כאתר אינטגרציה של גנים אקסוגניים באמצעות CRISPR-Cas928. הדו"ח האחרון שלנו בחן את הפוטנציאל של iPSCs המסומנים על ידי EGFP על ידי ביצוע אינטגרציה בתיווך CRISPR של מדווח EGFP המתבטא באופן מכונן באתר AAVS1 , המאפשר תיוג ומעקב הן של iPSCs והן של iPSCs מובחנים עקב ההתמדה של EGFP לאורך שושלת התא27. ניתן להשתמש בקו iPSC זה in vivo כדי להעריך את התפלגות האורגניזם של תאים שמקורם ב-iPSC לאחר ההשתלה. מכיוון שקו תאים זה עבר שינוי CRISPR ומשמש גם לבדיקת היישום הקליני של iPSCs, הכרחי שאתרי AAVS1 הפוטנציאליים מחוץ למטרה יהיו ידועים ונחקרים כדי להבטיח בטיחות ויעילות, מה שהופך אותו למיקום אידיאלי לבדיקת CIRCLE-seq.
הבדל בולט בין באטרפילד ואחרים, מחקר27 שפורסם בעבר לבין מחקר זה היה השימוש ב-gRNA שונה כדי להתמקד בלוקוס AAVS1. ניתן לעצב gRNA כדי לשפר את דיוק עריכת הגנום24. רצף המנחה הוא הגורם הקריטי ביותר המשפיע על היעילות על המטרה ומחוצה לה. לכן, ה-gRNA שנבחר נבדק מול מספר מדריכים אחרים ונמצא בעל נאמנות מעולה. בנוסף, מאמר שיטות על תכנות מחדש של פיברובלסטים אנושיים אישש את הממצא כי mRNAs סינתטיים עם מכסה המכילים נוקלאובסיסים מותאמים נהנים מהפעלה נמוכה של תגובות אנטי-ויראליות29,30. בעוד שאימונוגניות נמוכה עשויה שלא להיות רלוונטית בבדיקה חוץ גופית, היא הופכת להיות קריטית כאשר המטרה הסופית היא לפתח טיפול רלוונטי מבחינה קלינית שניתן להשתמש בו בתאים חיים.
ל-CIRCLE-seq יתרונות רבים על פני שיטות דומות. לדוגמה, Digenome-seq מרצף גם gDNA שסוע נוקלאז וגם לא מקוטע, תוך שימוש ב~400 מיליון קריאות21. התוצאה היא רקע גבוה, מה שמקשה על סינון אתרי חיתוך בתדירות נמוכה. CIRCLE-seq משתמש רק ב~3-5 מיליון קריאות עקב העשרת gDNA שסוע בנוקלאז, וכתוצאה מכך רקע נמוך. בנוסף, Digenome-seq ושיטה דומה, SITE-seq, מסתמכים על ריצוף קצה DNA יחיד שסוע בנוקלאז. לעומת זאת, קריאות CIRCLE-seq כוללות את שני הקצוות של אתר החיתוך, מה שמאפשר זיהוי אתרים מחוץ למטרה ללא צורך בהתייחסות 21,22,26.
היתרון של CIRCLE-seq הוא הרגישות הגדולה יותר שלו בהשוואה לשיטות המסתמכות על תרבית תאים, כגון GUIDE-seq. כאשר הושוו שתי השיטות, CIRCLE-seq הצליח ללכוד את כל האתרים מחוץ למטרה שזוהו על ידי GUIDE-seq וחשף אתרי מחשוף לא מכוונים נוספים ש-GUIDE-seq החמיץ. עם זאת, הבדל בולט הוא ש-GUIDE-seq עשוי להיות מעוכב על ידי הנוף האפיגנטי, בעוד ש-CIRCLE-seq יכול לגשת לכל הגנום.
כמבחן במבחנה , CIRCLE-seq מציג מספר מגבלות, הראשונה שבהן היא זיהוי תוצאות חיוביות כוזבות. בעוד שאפיגנטיקה תעכב את פעילות הנוקלאז באתרים מסוימים in vivo, אולטרה-סאונד מסיר את המכשולים הללו במבחנה, ומאפשר פעילות מחוץ למטרה במקומות שבדרך כלל אינם נגישים בהקשר תאי. יתר על כן, Cas9 קיים בריכוזים גבוהים בבדיקה חוץ גופית זו, מה שמאפשר מחשוף שלא היה אפשרי אחרת in vivo. בדיקה זו דורשת גם כמות גדולה יחסית של gDNA התחלתי, אשר, בהתאם למשאבים הזמינים, יכול לשלול את השימוש בפרוטוקול זה. לבסוף, ייתכן שחלק מהאתרים מחוץ ליעד יהיו בלתי ניתנים לגילוי בשל המגבלות של טכנולוגיות הריצוף הנוכחיות של הדור הבא.
מחקר שנערך לאחרונה השתמש בגישת in silico שהאלגוריתם שלה זיהה מספר פרמטרים רלוונטיים איתם ניתן להשוות שיטות אפיון נוקלאז שונות, כולל CIRCLE-seq ו-GUIDE-seq31. שניים מהפרמטרים הרלוונטיים היו 'העשרה באתר' ו'% תוצאות חיוביות כוזבות'. מעניין ששיעור החיוביים השגויים של CIRCLE-seq חושב ב-88%, אך ההעשרה באתר החיתוך שלו הייתה גבוהה פי שניים משיטות המבחנה האחרות. ניתוחים השוואתיים של כל שיטה גילו ש-GUIDE-seq היה בעל הביצועים הטובים ביותר, מכיוון שהוא הפגין את הספציפיות הגדולה ביותר על המטרה עם שיעור חיובי כוזב מתון בלבד32. זה לא פוסל את CIRCLE-seq, אלא רומז על האפשרות להשתמש ב-CIRCLE-seq ו-GUIDE-seq במקביל, ולאמת את הממצאים של CIRCLE-seq עם GUIDE-seq מכיוון שלראשון יש רגישות גבוהה יותר, בעוד שהאחרון הוא שיטה מבוססת תאים עם העשרה גבוהה של אתר חיתוך. הנתונים מצביעים גם על כך שריצוף הדור הבא (NGS) מבוסס אמפליקון צריך להיות השיטה המועדפת לזיהוי שינויים אמיתיים מחוץ למטרה באתרים מועמדים פוטנציאליים31. נתונים אלה מרמזים על אסטרטגיה פוטנציאלית של שימוש ב-CIRCLE-seq, ואחריו GUIDE-seq, ולאחר מכן NGS מבוסס אמפליקון כדי לבחון השפעות מחוץ למטרה.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
אנו מביעים את הכרת התודה העמוקה ביותר על תמיכת המימון הניתנת על ידי המכונים הלאומיים לבריאות (R01AR078551 ו-T32AR007411), האגודה לחקר אפידרמוליזה דיסטרופית בולוזה (DEBRA) אוסטריה, קרן גייטס גרובסטיק וקרן גבולות גייטס.
Materials
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
References
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185(2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856(2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786(2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058(2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157(2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474(2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745(2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved