Method Article
CIRCLE-Seq pour l’interrogation de l’édition de gènes hors cible
Dans cet article
Résumé
Un obstacle important aux technologies comme CRISPR est les événements hors cible qui peuvent perturber les gènes vitaux. La « circularisation pour la notification in vitro des effets de clivage par séquençage » (CIRCLE-seq) est une technique conçue pour identifier les sites de clivage non intentionnels. Cette méthode cartographie l’activité à l’échelle du génome de CRISPR-Cas9 avec une sensibilité élevée et sans biais.
Résumé
La circularisation pour la déclaration in vitro des effets de clivage par séquençage (CIRCLE-seq) est une nouvelle technique développée pour l’identification impartiale des sites de clivage non intentionnels de CRISPR-Cas9 par séquençage ciblé de l’ADN clivé de CRISPR-Cas9. Le protocole implique la circularisation de l’ADN génomique (ADNg), qui est ensuite traité avec la protéine Cas9 et un ARN guide (ARNg) d’intérêt. Après le traitement, l’ADN clivé est purifié et préparé comme une banque pour le séquençage Illumina. Le processus de séquençage génère des lectures d’extrémités appariées, offrant des données complètes sur chaque site de clivage. CIRCLE-seq offre plusieurs avantages par rapport aux autres méthodes in vitro , notamment des exigences minimales de profondeur de séquençage, un faible bruit de fond et un enrichissement élevé pour l’ADNg clivé en Cas9. Ces avantages améliorent la sensibilité dans l’identification des événements de clivage intentionnels et non intentionnels. Cette étude fournit une procédure complète, étape par étape, pour examiner l’activité hors cible de CRISPR-Cas9 à l’aide de CIRCLE-seq. À titre d’exemple, ce protocole est validé en cartographiant les sites de clivage non intentionnel de CRISPR-Cas9 à l’échelle du génome lors de la modification du locus AAVS1 . L’ensemble du processus CIRCLE-seq peut être achevé en deux semaines, ce qui laisse suffisamment de temps pour la croissance cellulaire, la purification de l’ADN, la préparation de la banque et le séquençage Illumina. L’entrée des données de séquençage dans le pipeline CIRCLE-seq facilite l’interprétation et l’analyse simplifiées des sites de clivage.
Introduction
L’ingénierie génomique a connu des progrès significatifs au cours des vingt dernières années, avec une étape majeure étant la découverte de répétitions palindromiques courtes groupées et régulièrement espacées (CRISPR)-Cas9 en 20121. Tirant parti de la nature programmable des endonucléases d’ADN bactérien, la technologie CRISPR-Cas9 permet un ciblage et une modification précis de presque toutes les séquences d’ADN. Depuis sa création, le système a été optimisé pour s’appuyer uniquement sur l’endonucléase Cas9 et un ARN guide (ARNg) pour modifier des régions génomiques spécifiques. Le potentiel de CRISPR-Cas9 en tant que thérapie curative a été démontré dans des essais cliniques pour diverses affections telles que l’amaurose congénitale de Leber, l’amylose à transthyrétine et l’anémie falciforme, entre autres 2,3,4.
CRISPR-Cas9 induit des cassures double brin (DSB), qui sont généralement résolues par l’un des deux mécanismes suivants : la jonction d’extrémités non homologues sujette aux erreurs (NHEJ) ou la réparation dirigée par homologie (HDR), plus précise, à condition qu’un ADN matrice soit disponible. La tendance de CRISPR-Cas9 à provoquer des insertions et des délétions (indels) associées à la NHEJ, ainsi qu’un clivage sur des sites génomiques non intentionnels, limite son application en milieu clinique 5,6,7,8,9,10. De plus, des modifications génomiques involontaires peuvent créer des sites d’épissage cryptiques, des mutations non-sens ou faux-sens, induire une chromothripsie ou conférer un potentiel oncogénique aux cellules - résultats qui ont été observés dans plusieurs essais d’édition du génome 11,12,13,14,15 . En conclusion, l’identification précise de l’activité hors cible de CRISPR-Cas9 est cruciale pour ses applications cliniques, en particulier dans les thérapies géniques systémiques qui peuvent altérer des milliards de cellules.
Diverses méthodes peuvent être utilisées pour identifier les sites de clivage hors cible de CRISPR-Cas9, notamment GUIDE-seq16, qui utilise des oligodésoxynucléotides double brin pour marquer les CDB dans les cellules vivantes. Néanmoins, une critique de cette méthode est que les faux positifs peuvent provenir de DSB aléatoires ou d’artefacts PCR, qui doivent être éliminés en excluant les sites capturés qui présentent une faible similitude avec les sites ciblés. La méthode basée sur l’utilisation du vecteur lentiviral défectueux en intégrase (IDLV) est moins sensible et risque de manquer de nombreux sites hors cible17. D’autres méthodes in situ telles que DSBCapture, BLESS et BLISS 18,19,20 impliquent des cellules fixes et marquent directement les DSB, mais elles sont limitées par leur dépendance à la capture immédiate des DSB et l’absence d’ADN exogène. Digenome-seq21, une méthode in vitro, et SITE-seq (SITE-seq)22 fournissent tous deux des solutions de séquençage, mais ont leurs limites en termes de bruit de fond et d’analyse à une extrémité, respectivement. La découverte de Cas hors cibles in situ et la vérification par séquençage (Discover-Seq)23 offrent une identification in vivo et in situ de l’activité de Cas9 via la liaison MRE11, mais ne détectent que les DSB qui existent au moment de la préparation de l’échantillon24. Enfin, l’inférence de modifications CRISPR (ICE) utilise une approche bioinformatique pour analyser de manière robuste les modifications CRISPR à l’aide des données de Sanger25.
Cet article décrit une procédure détaillée de circularisation pour le rapport in vitro des effets de clivage par séquençage (CIRCLE-seq) : une technique in vitro qui cartographie de manière sensible et impartiale l’activité hors cible à l’échelle du génome de la nucléase Cas9 dans un complexe avec l’ARNg d’intérêt26. Cette approche commence par la culture des cellules d’intérêt et l’isolement de l’ADN, suivie d’un cisaillement aléatoire par ultrasons focalisés, puis d’un traitement à l’exonucléase et à la ligase. Ce processus produit finalement des molécules d’ADN double brin circulaires, qui sont ensuite purifiées par un traitement par DNase sans danger pour les plasmides. Cet ADN circulaire est ensuite exposé au complexe Cas9-gRNA, qui se fissure aux sites de clivage intentionnels et non intentionnels, laissant derrière lui des extrémités d’ADN exposées qui agissent comme des substrats pour la ligature de l’adaptateur Illumina. Ce processus produit une bibliothèque diversifiée d’ADN génomique (ADNg) contenant les deux extrémités de chaque DSB induite par une nucléase, garantissant que chaque lecture dispose de toutes les informations nécessaires pour chaque site de clivage. Cela permet l’utilisation du séquençage Illumina avec des exigences de couverture de séquençage plus faibles, ce qui distingue CIRCLE-seq des autres méthodes similaires mentionnées ci-dessus. Il est important de noter que si CIRCLE-seq a une sensibilité hors cible plus élevée que d’autres protocoles en tant que méthode in vitro , cela se fait au prix d’un nombre plus élevé de faux positifs en raison de l’absence du paysage épigénétique présent dans d’autres méthodes telles que GUIDE-seq16. De plus, la réparation de l’ADN DSB et sa machinerie associée ne sont pas présentes dans CIRCLE-seq, abrogeant les indels ou la réparation appropriée qui serait autrement observée.
En plus de décrire le protocole étape par étape pour effectuer CIRCLE-seq, le protocole est validé en identifiant les sites de clivage non intentionnels de CRISPR-Cas9 à l’échelle du génome qui se produisent lors de la modification du locus AAVS1 , à titre d’exemple. Ce protocole facile à suivre fournit des instructions détaillées, de la culture de cellules souches pluripotentes induites (iPSC) et de l’isolement de l’ADNg à la circularisation de l’ADNg, au clivage de l’ARNg Cas9, à la préparation de la banque, au séquençage et à l’analyse du pipeline. Compte tenu des faibles exigences de couverture de séquençage, CIRCLE-seq est disponible pour tout laboratoire ayant accès au séquençage de nouvelle génération.
Protocole
Les détails des réactifs, des consommables et de l’équipement utilisés pour cette étude sont répertoriés dans la table des matériaux.
1. Culture cellulaire (5 jours)
- Incluez un contrôle négatif tout au long de ce protocole. Préparez suffisamment de cellules pour 25 μg supplémentaires d’ADNg (~2,0e7 cellules par échantillon).
- Culture des iPSC selon le protocole établi27. Prélever les cellules et les remettre en suspension dans 10 mL de PBS. Pipeter un échantillon de cellules de 6 μL et remettre en suspension dans un rapport de 1:1 avec du bleu de trypan. Utilisez un compteur de cellules automatisé pour compter l’échantillon.
- Aliquote 2 x 107 cellules par tube, puis essorage à 300 x g pendant 3 min à 25 °C (température ambiante (RT)). Ceci est suffisant pour plusieurs répétitions. Retirez la pipette et jetez le surnageant.
2. Isolement de l’ADN génomique (1 jour)
- Utilisez le kit de purification d’ADN disponible dans le commerce pour isoler l’ADNg, en suivant les instructions du fabricant :
- Ajouter 200 μL de PBS dans un tube conique de 15 mL contenant la pastille cellulaire et remettre en suspension. Ensuite, pipetez 3 ml de tampon de lyse cellulaire et 15 μL de protéinase K dans le tube. Retournez le tube 25 fois pour bien mélanger. Placez le tube dans un agitateur à bain-marie réglé à 55 °C et 150 tr/min pendant 3 h, ou toute la nuit pour un rendement optimal en ADN.
- Pipette dans 15 μL de RNase A. Inverser 25 fois. Placer au bain-marie à 37 °C pendant 1 h.
- Réfrigérer l’échantillon sur de la glace pendant 5 min. Ensuite, ajoutez 1 mL de solution de précipitation des protéines, faites un vortex à grande vitesse pendant 20 s et centrifugez à 2000 x g pendant 10 min à l’heure inverse. Les protéines doivent former une pastille visible et compacte au fond du tube. Si la pastille n’est pas visible, incubez l’échantillon sur de la glace pendant 5 minutes supplémentaires et centrifugez-le à nouveau.
- Ajouter 3 ml d’isopropanol à 100 % dans un nouveau tube conique de 15 ml. Pipeter soigneusement le surnageant de l’étape 2.1.3 dans le tube. Retournez le tube 50 fois pour mélanger, puis centrifugez à 2000 x g et à température ambiante pendant 3 min. Sans perturber la pastille d’ADN, aspirez soigneusement le surnageant à l’aide d’une pipette Pasteur reliée à un piège à vide et retournez le tube sur une lingette propre et non pelucheuse.
- Pipetez 3 ml d’éthanol à 70 % (v/v) sur la pastille d’ADN et retournez-la 10 fois pour la laver. Centrifugeuse à 2 000 x g pendant 3 min à RT. Ensuite, versez soigneusement le surnageant.
- Gardez le tube ouvert et laissez sécher la pastille d’ADN résultante pendant 30 min, en vous assurant que tout l’éthanol s’est complètement évaporé. Pipeter dans 50 μL de solution d’hydratation d’ADN et bien mélanger par pipetage doux.
- Dissoudre l’ADN en plaçant l’échantillon dans un agitateur à 65 °C pendant 1 h, puis laisser l’échantillon à l’extérieur pendant la nuit. Centrifugez l’échantillon à RT et 2 000 x g pendant 1 min et utilisez le kit de dosage dsDNA BR avec les tubes associés pour quantifier l’ADN isolé avec le fluorimètre.
3. Préparation de l’ARNg (7 jours)
- Commandez l’ARNg synthétique d’intérêt auprès d’une source commerciale (voir le tableau des matériaux). Ce protocole est également compatible avec l’ARNcr/ARNtracr.
4. Test de clivage in vitro de l’ARNg
REMARQUE : Ici, une cible du gène AAVS1 est utilisée. Pour cibler d’autres gènes d’intérêt, concevez des amorces (tableau 1) pour amplifier la région cible et remplacez les amorces dans les étapes suivantes par des amorces personnalisées.
- Préparez la réaction PCR : Mélangez 25 μL de Phusion Hot Start Flex 2x Master Mix (concentration finale 1x), 0,5 μL d’amorce AAVS1 F (concentration finale 0,1 μM), 0,5 μL d’amorce AAVS1 R (concentration finale 0,1 μM), 5 μL d’ADNg (100 ng, 20 ng/μL, à partir de l’étape 2.1.6) et 19 μL de H2O sans nucléase (Volume total : 50 μL).
- Utilisez les paramètres suivants du thermocycleur : Dénaturation : 98 °C pendant 2 min (1 cycle), Dénaturation : 98 °C pendant 10 s (10 cycles), Recuit : 72-62 °C (-1 °C/cycle) pendant 15 s (10 cycles), Extension : 72 °C pendant 30 s (10 cycles), Dénaturation, 98 °C pendant 10 s (30 cycles). Recuit : 65 °C pendant 15 s (30 cycles), Extension : 72 °C pendant 30 s (30 cycles), Extension finale : 72 °C pendant 5 min (1 cycle), Maintien : 4 °C indéfiniment.
- Utilisez des billes AMPure XP pour purifier le produit de la réaction PCR. Tout d’abord, pipetez des volumes de 1,8 fois, soit 90 μL, de billes XP sur le produit PCR. Pipetez dix fois pour bien mélanger. Laissez le mélange à RT pendant 5 min pour incuber.
- À l’aide d’un support magnétique, séparez les billes de la solution en plaçant la plaque de réaction PCR sur l’aimant pendant 3 min. Pipetez la solution éliminée et jetez-la. Ajoutez 200 μL d’éthanol à 80 % (v/v) dans les billes, incubez pendant 30 s, puis retirez l’éthanol. Répétez cette étape de lavage deux fois pour assurer l’élimination complète de l’éthanol.
- Laissez sécher les échantillons naturellement pendant 3 min en posant la plaque sur l’aimant. Retirez la plaque de l’aimant et ajoutez 40 μL de tampon TE, pH 8,0. Mélanger en pipetant de haut en bas dix fois. Laissez l’échantillon reposer à RT pendant 2 min.
- Placez la plaque de réaction PCR sur l’aimant pendant une minute supplémentaire. Après 1 min, transférez le surnageant dans une nouvelle assiette. Mesurez le rendement de la PCR purifiée à l’aide d’un spectrophotomètre et analysez-le sur une TapeStation à l’aide d’une bande de tube optique avec un capuchon de bande de tube optique, ainsi que des réactifs High Sensitivity D1000 ScreenTape et High Sensitivity D1000 (échelle et tampon), en suivant les instructions du fabricant. Conservez l’échantillon préparé à -20 °C pendant plusieurs mois au maximum.
- Diluer la protéine nucléase Cas9 à 1 μM comme suit : Mélanger 2 μL de 10 tampons Cas9 (concentration finale 1x), 1 μL de nucléase Cas9, S. pyogenes (concentration finale 1 μM) et 17 μL de H2O sans nucléase dans un volume total de 20 μL.
- Effectuez une procédure sans RNase pour prévenir la dégradation de l’ARNg. Diluer l’ARNg (à partir de l’étape 3.1) à 3 μM dansH2O jusqu’à un volume total de 10 μL.
REMARQUE : Utilisez la formule suivante pour estimer la masse moléculaire de l’ARNg : Masse moléculaire de l’ARNsb (g/mol) = (longueur de l’ARNsb (nt) x 321,47 g/mol) + 18,02 g/mol. À titre de référence, un ARNg de 104 nt de long à 3 μM correspond à environ 100 ng/μL.
5. Cisaillement de l’ADN (3 h)
- Préparez le ME220 en renvoyant d’abord le bras de commande. Ensuite, remplissez le réservoir avec du H2O désionisé purifié. Sur l’ordinateur portable du poste de contrôle, accédez à Water Works et cliquez sur Remplir. Réglez la température à 4,5 °C.
- Transférez 25 μg d’ADNg dans un microtube (microtube-130 AFA Fiber Screw-Cap). Ensuite, remplissez le tube jusqu’à un volume total de 130 μL avec 1x TE. Utilisez les conditions suivantes pour cisailler l’ADN à une longueur moyenne d’environ 300 pb : réglez la durée sur 10 s ; puissance de crête à 70 ; Facteur de droit % à 20 ; cycles/rafale jusqu’à 50 ; tous ces paramètres régleront automatiquement la puissance moyenne sur 14,0.
6. Purification de l’ADN génomique cisaillé (1 h)
- Divisez l’ADN génomique cisaillé en deux portions de 65 μL chacune. Purifiez en utilisant 1,8 fois le volume de billes XP (117 μL), en suivant la procédure décrite aux étapes 4.3-4.6. Transférez le surnageant sur une nouvelle plaque de PCR et mesurez la quantité à l’aide d’un spectrophotomètre.
- Exécutez 1 μL de l’ADNg cisaillé élué sur une TapeStation, conformément aux instructions du fabricant, pour s’assurer que l’ADNg est cisaillé à une large distribution d’environ 300 pb. Si nécessaire, conservez l’ADNg cisaillé à -20 °C pendant plusieurs mois au maximum.
7. Préparation de la bibliothèque CIRCLE-seq (3 jours)
- Recuit de l’adaptateur d’épingle à cheveux
- Réintroduire l’oSQT1288 (Tableau 1), l’adaptateur en épingle à cheveux, à une concentration finale de 100 μM dans 1x TE.
- Effectuez le recuit de l’adaptateur comme suit : mélangez 40 μL d’oSQT1288 (concentration finale 40 μM), 10 μL de 10x STE (concentration finale 1x) et 50 μL de H2O sans nucléase pour un volume total de 100 μL.
- Utilisez les paramètres de recuit suivants : 95 °C pendant 5 min, -1 °C par minute pendant 70 cycles, maintenir à 4 °C indéfiniment.
- Effectuer la réparation de fin . Utilisez le kit de préparation de la bibliothèque HTP sans PCR et préparez le Master Mix de réparation finale.
- Mélangez 8 μL de H2O sans nucléases, 7 μL de 10 tampons de réparation en bout (concentration finale 1x) et 5 μL de mélange d’enzymes de réparation en bout (volume final 20 μL de Total End-Repair Master Mix).
- Pipeter 20 μL du mélange maître de réparation finale dans l’échantillon d’ADNg cisaillé des étapes 4.3 à 4.6. Mélangez 20 μL de Master Mix End-Repair avec 50 μL d’ADNg cisaillé pour un volume final de 70 μL.
- Placez le mélange dans un thermocycleur à 20 °C pendant 30 min, puis maintenez-le à 4 °C indéfiniment.
- Ajoutez 1,7 fois les volumes, soit 120 μL, de billes XP et suivez les étapes de purification des étapes 4.3 à 4.6. Éluée avec 42 μL d’ET, pH 8,0. Assurez-vous que les billes restent en solution pour l’étape suivante.
- Effectuez le A-tailing. À l’aide du kit de préparation de la bibliothèque HTP (sans PCR), préparez le Master Mix A-tailing.
- Mélangez 5 μL de 10 tampons de résidus A (concentration finale 1x) et 3 μL d’enzyme de résidus A (volume final total de 8 μL de Master Mix de résidus A).
- Pipeter 8 μL du A-tailing Master Mix dans chaque échantillon d’ADN contenant des billes à partir de l’étape 7.2.4, en mélangeant 8 μL de A-tailing Master Mix à 42 μL d’ADN réparé contenant des billes (volume final total de 50 μL). Placer dans un thermocycleur à 30 °C pendant 30 min. Maintenir à 4 °C indéfiniment.
- Pipeter 1,8 volume, soit 90 μL, de la solution PEG/NaCl SPRI (un composant du kit de préparation de la bibliothèque HTP (sans PCR ; 96 réactions)) à l’ADN à queue A. Purifiez l’ADN A selon les étapes 4.3-4.6. Éluer l’ADN de la queue A dans 30 μL d’ET, pH 8,0. Gardez les billes en solution pour l’étape suivante.
- Effectuez la ligature de l’adaptateur. À l’aide du kit de préparation de la bibliothèque HTP (sans PCR), préparez l’Adapter Ligation Master Mix.
- Mélangez 10 μL de tampon de ligature 5x (concentration finale 1x), 5 μL d’ADN ligase et 5 μL d’adaptateur d’épingle à cheveux recuit (40 μM) de l’étape 7.1.3. Assurez-vous que la concentration finale est de 4 μM pour un Master Mix total de 20 μL.
- Pipeter 20 μL d’Adapter Ligation Master Mix dans chaque échantillon d’ADN élué contenant des billes à partir de l’étape 7.3.3 (volume final total de 50 μL par échantillon).
- Placer dans un thermocycleur à 20 °C pendant 1 h. Maintenir à 4 °C indéfiniment.
- Transférez 1 volume (50 μL) de solution de PEG/NaCl SPRI dans l’ADN ligaturé par l’adaptateur et purifiez selon les étapes 4.3 à 4.6. Éluer avec 30 μL d’ET, pH 8,0, et décanter les surnageants dans une nouvelle plaque PCR semi-jupe. Combinez et quantifiez l’ADN à l’aide du test dsDNA BR. Si nécessaire, conservez l’ADN purifié ligaturé par un adaptateur jusqu’à 1 mois à -20 °C.
- Préparez le Master Mix Lambda Exonucléase/Exonucléase I (E. coli) (fonctions d’élimination de l’ADN simple ou double brin sans adaptateurs ligaturés aux deux extrémités).
- Prélever 1 μg d’ADN ligaturé adaptateur de l’étape 7.4.4 et le diluer à 40 μL. Mélangez 5 μL de tampon réactionnel 10x Exonucléase I (concentration finale 1x), 4 μL d’exonucléase Lambda (concentration finale 0,4 U/μL) et 1 μL d’exonucléase I (E. coli) (concentration finale 0,4 U/μL) pour un volume total de Master Mix Lambda Exonucléase/Exonucléase I de 10 μL.
- Pipetez 10 μL du Master Mix Lambda Exonucléase/Exonucléase I à 40 μL (1 μg) d’ADN ligaturé par adaptateur (volume total de 50 μL). Placer dans un thermocycleur à 37 °C pendant 1 h, puis à 75 °C pendant 10 min. Maintenir à 4 °C indéfiniment.
- Pipetez des volumes de 1,8 fois, soit 90 μL, de billes XP sur l’ADN traité à l’exonucléase/exonucléase I Lambda. Purifiez selon les instructions données aux étapes 4.3 à 4.6. Éluée dans 40 μL d’ET, pH 8,0. Assurez-vous que les billes restent en solution pour l’étape enzymatique suivante.
- Traiter avec l’enzyme USER et la polynucléotide kinase T4 (PNK). Préparez le Master Mix PNK USER/T4 (nécessaire pour libérer les surplombs de 4 pb et pour préparer les extrémités de l’ADN prêtes à être ligaturées nécessaires à la réaction de ligature ultérieure).
- Mélangez 5 μL de tampon d’ADN ligase 10x T4 (concentration finale 1x), 3 μL d’enzyme USER (concentration finale 0,05 U/μL) et 2 μL de T4 PNK (concentration finale 0,4 U/μL), pour un volume total du mélange maître USER/PNK de 10 μL.
- Pipeter 10 μL du Master Mix USER Enzyme/T4 PNK à 40 μL d’échantillon d’ADN traité lambda et exonucléase I contenant des billes de l’étape 7.5.3 pour un volume total de 50 μL. Placer dans un thermocycleur à 37 °C pendant 1 h. Maintenir à 4 °C indéfiniment.
- Pipeter des volumes 1,8x, soit 90 μL, de la solution de PEG/NaCl SPRI sur l’ADN traité avec USER/T4 PNK et purifier selon les étapes 4.3-4.6. Élution dans 35 μL d’ET, pH 8,0. Décantez le surnageant dans une nouvelle plaque PCR semi-jupe. Combinez et quantifiez l’ADN à l’aide du test dsDNA HS.
- Effectuer une circularisation intramoléculaire. Préparez le Master Mix de circularisation.
- Mélangez 8 μL de H2O sans nucléases, 10 μL de tampon d’ADN ligase 10x T4 (concentration finale 1x) et 2 μL d’ADN ligase T4 (concentration finale 8 U/μL), pour un volume total de 20 μL de Master Mix de circularisation.
- Pipette de 20 μL du Master Mix de circularisation à 500 ng d’ADN traité avec USER/PNK à partir de l’étape 7.6.3. Diluez 500 ng d’ADN traité avec USER/PNK dans 80 μL, puis ajoutez 20 μL du Master Mix de circularisation (volume total de 100 μL). Incuber dans un thermocycleur à 16 °C pendant 16 h (toute la nuit).
- Ajoutez 1 volume de billes de XP à l’ADN circularisé et purifiez-le selon les étapes 4.3 à 4.6. Éluer dans 38 μL d’ET, pH 8,0. Décantez le surnageant dans une nouvelle plaque PCR semi-jupe.
- Traiter avec de la DNase dépendante de l’ATP sans danger pour les plasmides. Préparez le mélange maître de DNase ATP-dépendante sans danger pour les plasmides (nécessaire à la dégradation de l’ADN linéaire résiduel).
- Mélangez 5 μL de tampon de réaction 10x sans danger pour les plasmides (concentration finale 1x), 2 μL d’ATP (concentration finale 1 mM) et 5 μL de DNase dépendante de l’ATP sans danger pour les plasmides (concentration finale 1 U/μL), pour un volume total de 12 μL de Master Mix sans danger pour les plasmides.
- Pipeter 12 μL du DNase Master Mix dépendant de l’ATP à 38 μL d’ADN circularisé à partir de l’étape 7.7.3 (volume total de 50 μL). Incuber dans un thermocycleur à 37 °C pendant 1 h, puis à 70 °C pendant 30 min. Maintenir à 4 °C indéfiniment.
- Pipeter 1 volume x, soit 50 μL, de billes XP dans l’ADN traité avec la DNase ATP-dépendante sans danger pour les plasmides et purifier selon les étapes 4.3 à 4.6. Éluée dans 15 μL d’ET, pH 8,0. Décantez le surnageant dans une nouvelle plaque PCR semi-jupe.
- Combinez l’ADN et quantifiez à l’aide du test dsDNA HS. Si nécessaire, conservez l’ADN circulaire à -20 °C pendant plusieurs mois.
8. Civrage de l’ADNg circulaire et purifié enzymatiquement in vitro (2 h)
- Réaliser un clivage in vitro avec le complexe Cas9 :gRNA. Préparez le Master Mix pour clivage in vitro . Mélangez 5 μL de tampon 10x Cas9 (concentration finale 1x), 4,5 μL de S. Pyogenes Cas9 (concentration finale 90 nM) et 1,5 μL d’ARNg (concentration finale 90 nM) pour un volume total de mélange principal de clivage de 11 μL.
- Maintenir le mélange maître de clivage à RT pendant 10 min pour former des complexes RNP Cas9 :gRNA.
- Diluer 125 ng d’ADN traité par DNase sans danger pour les plasmides à partir de l’étape 7.8.3 jusqu’à un volume final de 39 μL. Ensuite, ajoutez 11 μL du Cleavage Master Mix à 39 μL d’ADN traité par DNase sans danger pour les plasmides, pour un volume total de 50 μL.
REMARQUE : Incluez dans cette étape un échantillon de contrôle négatif, qui comprend de l’ADN circularisé mélangé à un tampon Cas9, sans le complexe Cas9 :ARNg. - Incuber dans un thermocycleur pendant 1 h à 37 °C. Maintenir à 4 °C indéfiniment. Ajoutez 50 μL (1x volume) de billes XP à l’ADN clivé in vitro et purifiez l’ADN en suivant les étapes 4.3-4.6. Éluer dans 42 μL de tampon TE, pH 8,0. Assurez-vous que les billes restent en solution pour l’étape suivante.
9. Préparation de la bibliothèque de séquençage de nouvelle génération (4 - 6 h)
- Effectuez le A-tailing. Préparez le A-tailing Master Mix.
- Mélangez 5 μL de tampon de queue A 10x (concentration finale 1x) et 3 μL d’enzyme de queue A (le volume total du mélange maître de queue A est de 8 μL).
- Pipeter 8 μL du A-tailing Master Mix à 42 μL d’échantillon d’ADN élué contenant des billes à partir de l’étape 8.4 (volume total de 50 μL). Placer dans un thermocycleur pendant 30 min à 30 °C. Maintenir à 4 °C indéfiniment.
- Pipeter 1,8 fois des volumes de la solution PEG/NaCl SPRI, soit 90 μL, sur l’ADN à queue A et purifier l’ADN selon les étapes 4.3 à 4.6. Éluée dans 25 μL d’ET, pH 8,0. Assurez-vous de garder les billes en solution pour l’étape suivante.
- Effectuez la ligature de l’adaptateur. Préparez l’adaptateur Ligation Master Mix.
REMARQUE : Des aliquotes à usage unique des adaptateurs NEB doivent être préparées afin d’éviter la formation de dimères d’adaptateur causée par l’hydrolyse du 3' T’induite par le gel-dégel.- Mélangez 10 μL de tampon de ligature 5x (concentration finale 1x), 5 μL d’ADN ligase et 10 μL d’adaptateur pour le séquençage (la concentration finale est de 3 μM), pour un total de 25 μL.
- Pipeter 25 μL de l’adaptateur Ligation Master Mix dans 25 μL d’échantillon d’ADN à queue A contenant des billes de l’étape 9.1.3. Placer dans un thermocycleur pendant 1 h à 20 °C. Maintenir à 4 °C indéfiniment.
- Pipeter 1 fois des volumes, soit 50 μL, de la solution de PEG/NaCl SPRI sur l’ADN ligaturé par adaptateur et purifier l’ADN selon les étapes 4.3-4.6. Éluée dans 47 μL d’ET, pH 8,0. Assurez-vous que les billes restent en solution pour l’étape enzymatique suivante.
- Effectuer le traitement avec l’enzyme USER (génère un seul espace nucléotidique au niveau des résidus d’uracile).
- Ajoutez 3 μL d’enzyme USER (inclus dans le kit d’amorces à double index) à l’échantillon d’ADN ligaturé par adaptateur contenant des billes à partir de l’étape 9.2.3. Incuber à 37 °C pendant 15 min.
- Ajouter 35 μL (volume 0,7x) de solution de PEG/NaCl SPRI à l’ADN traité par enzyme USER et purifier selon les étapes 4.3-4.6. Eluté dans 20 μL de tampon TE, pH 8,0. Transférez le surnageant sur une nouvelle plaque PCR semi-jupe et mesurez la concentration d’ADN à l’aide du test dsDNA HS. La concentration attendue doit être d’environ 2-5 ng/μL.
- (Facultatif) Avant de passer à l’étape suivante, la sélection de la taille de l’ADN peut être effectuée à l’aide de PippinHT. Utilisez la cassette PippinHT à 1,5 % avec une gamme de tailles de 250 à 850 pb. Les échantillons résultants peuvent être directement utilisés dans la PCR à l’étape suivante.
- Effectuer une PCR pour l’ajout de codes-barres
REMARQUE : Assurez-vous que les combinaisons de séquences d’amorces choisies pour chaque échantillon sont uniques. Si possible, chaque échantillon doit avoir des codes-barres i5 et i7 uniques.- Préparez le PCR Master Mix pour ajouter des codes-barres à index appariés. Mélangez 5 μL de H2O sans nucléases, 25 μL de 2x HotStart Ready Mix (concentration finale 1x), 5 μL d’apprêt i5 (concentration finale 1 μM) et 5 μL d’apprêt i7 (concentration finale 1 μM) (volume total de mélange principal de 40 μL).
- Pipeter 40 μL du PCR Master Mix à 10 μL d’ADN purifié traité avec l’enzyme USER (environ 20 ng) à partir de l’étape 9.3.2 (volume total de 50 μL).
- Choisissez les conditions de thermocyclage PCR suivantes : Dénaturation : 98 °C pendant 45 s pour 1 cycle, Dénaturation : 98 °C pendant 15 s pour 20 cycles, Recuit : 65 °C pendant 30 s pendant 20 cycles, Extension : 72 °C pendant 30 s pendant 20 cycles, Extension finale : 72 °C pendant 1 min pour 1 cycle, Maintien : 4 °C indéfiniment.
- Ajoutez 0,7 fois des volumes, soit 35 μL, de billes XP au produit PCR et purifiez selon les étapes 4.3 à 4.6. Éluer dans 30 μL d’ET, pH 8,0. Décantez le surnageant dans une nouvelle plaque PCR semi-jupe. Si nécessaire, conservez l’ADN circularisé à -20°C pendant plusieurs mois maximum.
REMARQUE : Exécutez un échantillon de la PCR sur Tapestation pour contrôler la qualité de la bibliothèque et évaluer la formation des dimères de l’adaptateur. Si des dimères de l’adaptateur sont détectés, répétez l’étape 9.5.4.
10. Quantification des banques CIRCLE-seq par PCR numérique en gouttelettes (dd_PCR) (6 h)
REMARQUE : La quantification peut également être effectuée à l’aide de la qPCR, de la Tapestation ou d’une méthode similaire.
- Commencer avec 5 μL d’ADN de la banque (étape 9.5.4 de la PCR) bien mélangés avec 45 μL d’ET sans nucléases, puis effectuer des dilutions en série de 1:10 de chaque échantillon dans des volumes de 50 μL, allant de 10-1 à 10-8 dilutions.
- Configurez la solution mère dd_PCR Master Mix. Mélanger 11 μL de 2x dd_PCR mélange pour sondes (concentration finale 1x), 0,055 μL de sonde oSQT1310 (concentration finale 250 nM), 0,055 μL de sonde oSQT1311 (concentration finale 250 nM), 0,099 μL d’amorce oSQT1274 (concentration finale 450 nM), 0,099 μL d’amorce oSQT1275 (concentration finale 450 nM) et 6,292 μL de H2O sans nucléase, pour un volume total de dd_PCR Master Mix de 17,6 μL. Préparez un mélange maître pour tous les échantillons afin de vous assurer que les volumes sont suffisants pour un pipetage précis.
- Analysez les trois dilutions les plus faibles (10-6, 10-7 et 10-8) en double (dans une plaque de 96 puits). Un contrôle sans modèle (NTC) doit être utilisé.
- Pipeter 17,6 μL de dd_PCR Master Mix sur chaque échantillon comme suit : Mélanger 17,6 μL avec 4,4 μL de l’échantillon (en ajoutant du H2O sans nucléases dans le puits avec du NTC), pour un volume total de 22 μL. Sceller la plaque, puis centrifuger à 2000 x g pendant 1 min à RT.
- Effectuez la génération de gouttelettes, le thermocyclage et l’analyse. À l’aide d’un système PCR Droplet Reader, transférez une cartouche DG8 (8 puits) dans le support de cartouche. Dans la rangée d’huile de la cartouche, distribuer 70 μL d’huile de génération de gouttelettes pour les sondes.
REMARQUE : Prélever 20 μL de l’échantillon de l’étape 10.1.2. et ajoutez-le à la rangée d’échantillons de la cartouche à 8 puits, en recouvrant la cartouche avec le joint en caoutchouc DG8, en le plaçant dans le générateur de gouttelettes, puis en le fermant pour commencer le processus (automatique). Une fois l’opération terminée, retirez la cartouche et déplacez 40 μL de la rangée de gouttelettes de la cartouche à 8 puits dans une plaque PCR semi-jupe à 96 puits, en veillant à pipeter lentement.- Placez le bloc de chaleur dans la scelleuse de plaques PX1 PCR. La soudeuse commencera à chauffer jusqu’à 180 °C lorsqu’elle est allumée. Placez le thermoscellage en aluminium sur la plaque, en vous assurant que la ligne rouge est sur le dessus. Placez la plaque dans le PX1 et appuyez sur Seal.
- Choisissez les conditions suivantes du thermocycleur : Activation enzymatique : 95 °C pendant 10 min pour 1 cycle, Dénaturation : 94 °C pendant 30 s pendant 40 cycles, Recuit/Extension : 60 °C pendant 1 min pendant 40 cycles, Désactivation enzymatique : 98 °C pendant 10 min pendant 1 cycle, Maintien : 4 °C indéfiniment.
- Sur le lecteur de gouttelettes, ouvrez le logiciel compatible et sélectionnez les puits à lire. Sélectionnez ABS comme type d’expérience et dd_PCR Supermix pour les sondes. Choisissez Ch1 inconnu pour la cible 1 et Ch2 inconnu pour la cible 2. Sélectionnez Appliquer, puis OK. Insérez la plaque dans le lecteur de gouttelettes. Pour le jeu de colorants, choisissez FAM/HEX et cliquez sur Exécuter.
- Analysez les résultats dd_PCR. Gate la population de gouttelettes double positive en utilisant le témoin négatif comme référence. Calculez la moyenne des valeurs en double, puis multipliez par le facteur de dilution et le facteur de dilution 5 du dd_PCR.
REMARQUE : Le nombre total de copies par microlitre est calculé comme suit : nombre total de copies par microlitre = valeur moyenne × 5 × facteur de dilution, où la « valeur moyenne » représente la valeur moyenne de quantification des canaux 1 et 2. - Combinez tous les échantillons en une seule banque à des concentrations équimolaires. La bibliothèque 1x regroupée doit contenir environ 4,5 x 109 molécules et avoir un volume total de 5 μL.
11. Séquençage de nouvelle génération
- Soumettez les échantillons pour le séquençage à un organisme externe, en veillant à ce que les séquences d’adaptateur correctes soient notées.
12. Analyse des données CIRCLE-seq (1 - 3 h)
- Installez Python version 2.7, Burrows-Wheeler Aligner (BWA) et SAMtools. Téléchargez le génome de référence (par exemple, hg38) à partir de http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz.
REMARQUE : Si le génome de l’espèce cible n’est pas disponible, le flux de travail de calcul CIRCLE-seq peut être exécuté en mode indépendant de la référence. Dans cette situation, cette étape peut être ignorée. - Téléchargez et installez le pipeline CIRCLE-seq à l’aide des commandes suivantes : (1) git clone https://github.com/tsailabSJ/circleseq.git, (2) cd circleseq, (3) pip install -r requirements.txt.
- Créez un fichier manifeste au format YAML (.yaml). Vous trouverez ci-dessous un exemple de manifeste qui peut être utilisé avec l’exemple de jeu de données fourni dans le logiciel CIRCLE-seq pour tester le flux de travail.
REMARQUE : (1) génome de référence : data/input/CIRCLEseq_test_genome.fa ; (2) analysis_folder : données/sorties ; (3) BWA : BWA ; (4) Samtools : Samtools ; (5) read_threshold : < Valeur > ; (6) window_size : < Valeur > ; (7) mapq_threshold : < Valeur > ; (8) start_threshold : < Valeur > ; (9) gap_threshold : < Valeur > ; (10) mismatch_threshold : < Valeur > ; (11) merged_analysis : Vrai ; (12) échantillons : U2OS_EMX1 ; (13) cible : GAGTCCGAGCAGAAGAAGAANGG ; (14) read1 : données/entrées/EMX1.r1.fastq.gz ; (15) read2 : données/entrées/EMX1.r2.fastq.gz ; (16) controlread1 : données/entrées/EMX1_control.r1.fastq.gz ; (17) controlread2 : données/entrées/EMX1_control.r2.fastq.gz ; (18) description : U2OS. Les valeurs manifestes suivantes ont été utilisées : read_threshold : 4, window_size : 3, mapq_threshold : 50, start_threshold : 1, gap_threshold : 3, mismatch_threshold : 6 - Définissez le fichier FASTA du génome de référence, le répertoire de sortie pour l’analyse et les chemins d’accès aux commandes BWA et SAMtools . Définissez les séquences cibles et les chemins d’accès aux fichiers FASTQ démultiplexés pour les échantillons clivés par nucléase et les échantillons de contrôle. Plusieurs expériences peuvent être traitées simultanément en mode batch en les incluant toutes dans un seul fichier manifeste.
- Exécutez la commande suivante pour les analyses standard basées sur des références : (1) python /path/to/circleseq.py all - manifest ; (2) /path/to/manifest.yaml.
- Vous pouvez également exécuter la commande suivante pour les analyses standard non basées sur des références : (1) Python /path/to/circleseq.py reference-free - manifest ; (2) /chemin/vers/manifest.yaml.
- Lors de l’exécution du pipeline complet, recherchez les résultats de sortie de chaque étape dans un output_folder distinct désigné pour cette étape spécifique.
Résultats
Ici, CIRCLE-seq est utilisé pour étudier les sites de clivage induits par les nucléases de Cas9 dans un complexe avec l’ARNg conçu pour cibler le site d’intégration du virus adéno-associé 1 (AAVS1) en utilisant de l’ADN isolé de cellules souches pluripotentes induites (iPSC). Cet ARNg a déjà été décrit dans notre publication27. Environ 25 μg d’ADNg ont été isolés à partir d’iPSC, cisaillés par ultrasons focalisés, et la taille a été sélectionnée à l’aide de la purification de billes AMPure XP pour produire des fragments d’environ 300 pb. À partir de ces 25 μg d’ADN, environ 2 à 5 ng d’ADN ont été circularisés avec succès pour le clivage in vitro de Cas9 :gRNA. L’ensemble de la procédure est illustré à la figure 1.
À la suite d’une procédure CIRCLE-seq et d’une analyse à l’aide de notre flux de travail de calcul, une visualisation de tous les sites de clivage détectés sur et hors cible est présentée dans la figure 2A. Le pipeline CIRCLE-seq a également fourni des « lectures fusionnées », analysées via le logiciel statistique R pour produire un graphique de Manhattan montrant les sites de clivage induits par les nucléases détectés cartographiés le long de chaque chromosome (Figure 2B).
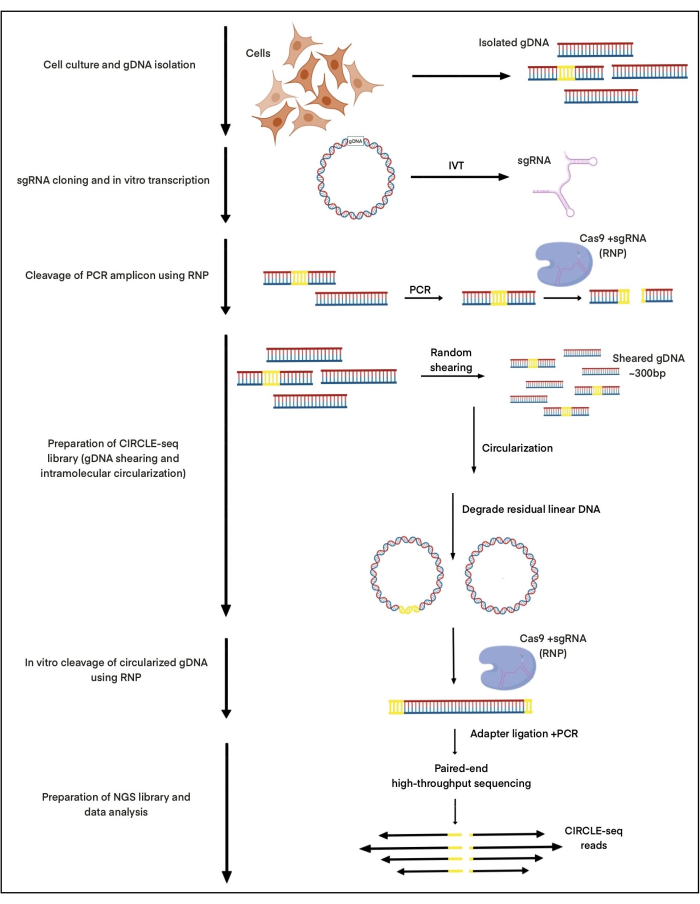
Figure 1 : Schémas du flux de travail CIRCLE-seq. Les principales étapes du protocole sont indiquées. Veuillez cliquer ici pour voir une version agrandie de cette figure.
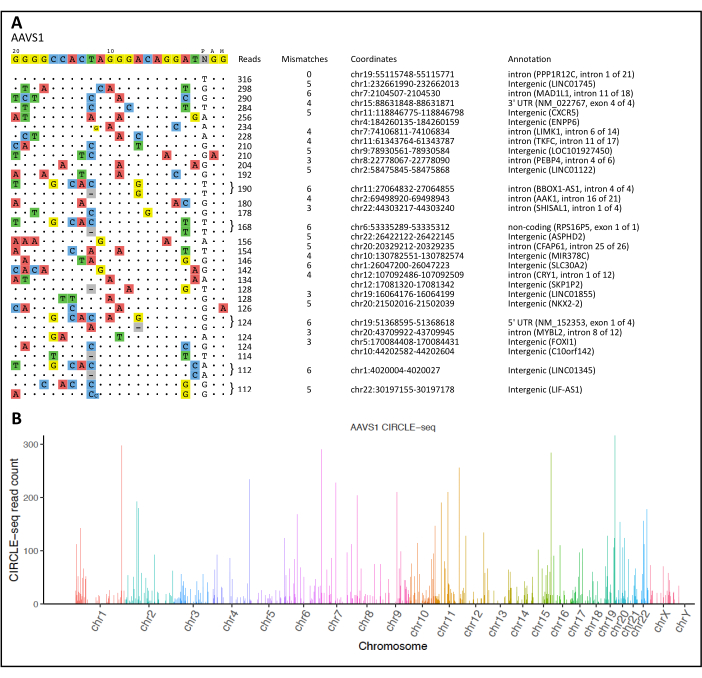
Figure 2 : Visualisation de CIRCLE-seq et du graphique de Manhattan. (A) Alignement des sites hors cible par rapport à la cible prévue pour le locus AAVS1. La séquence cible est affichée en haut, où les hors cibles sont classées par nombre de lectures dans l’ordre décroissant. Les différences dans les séquences cibles d’origine sont mises en évidence par des nucléotides colorés. Un échantillon des principaux sites de clivage non intentionnel du locus AAVS1 est présenté. (B) Graphique de Manhattan illustrant les sites de clivage non intentionnels détectés pour le locus AAVS1. Les hauteurs de barre représentent le nombre de lectures pour chaque position chromosomique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Amorce | Séquence (5'-3') | Commentaires/Description |
| AAVS1 ARN guide unique (ARNsg) | GGGGCCACUAGGGACAGGAU | Pour l’inhibition de la protéine fluorescente du locus AAVS1 |
| AAVS1 Amorce avant | GCTCTGGGCGGAGGAATATG | Pour le test de clivage in vitro de l’ARNg |
| AAVS1 Amorce inversée | ATTCCCAGGGCCGGTTAATG | Pour le test de clivage in vitro de l’ARNg |
| oSQT1288 | /5Phos/CGGTGGACCGATGATC /ideoxyU/ATCGGTCCACCGaT | Adaptateur d’épingle à cheveux CIRCLE-seq |
| oSQT1274 | AATGATACGGCGACCACCGAG | TruSeq F1 |
| oSQT1275 | CAAGCAGAAGACGGCATACGAGAT | TruSeqF2 |
| oSQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | Sonde TruSeq |
| oSQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | Sonde TruSeq |
Tableau 1 : Séquences d’ARNg et d’amorces utilisées pour l’analyse CIRCLE-seq du locus AAVS1 .
Discussion
Ici, CIRCLE-seq s’avère être une technique non biaisée et très sensible pour identifier les DSB induites par les nucléases dans le génome résultant du ciblage du locus AAVS1 dans l’ADNg dérivé des iPSC. Le site AAVS1 au sein des iPSC est bien connu comme un locus de sphère de sécurité qui est souvent utilisé comme site d’intégration de gènes exogènes à l’aide de CRISPR-Cas928. Notre récent rapport a étudié le potentiel des iPSC marquées à l’EGFP en effectuant l’intégration médiée par CRISPR d’un rapporteur EGFP exprimé constitutivement dans le site AAVS1 , ce qui permet le marquage et le suivi des iPSC et des iPSC différenciées en raison de la persistance de l’EGFP tout au long de la lignée de la cellule27. Cette lignée d’iPSC peut être utilisée in vivo pour évaluer la distribution de l’organisme des cellules dérivées d’iPSC après transplantation. Comme cette lignée cellulaire a été modifiée par CRISPR et qu’elle est également utilisée pour tester l’application clinique des iPSC, il est impératif que les sites hors cible potentiels d’AAVS1 soient connus et interrogés pour garantir l’innocuité et l’efficacité, ce qui en fait un lieu idéal pour tester CIRCLE-seq.
Une différence notable entre l’étude27 de Butterfield et al., précédemment publiée, et celle-ci était l’utilisation d’un ARNg modifié pour cibler le locus AAVS1. Un ARNg peut être conçu pour améliorer la précision de l’édition du génome24. La séquence de guidage est le facteur le plus critique qui influence l’efficacité sur et hors cible. Par conséquent, l’ARNg choisi a été testé par rapport à plusieurs autres guides et s’est avéré d’une fidélité supérieure. De plus, un article sur les méthodes de reprogrammation des fibroblastes humains a corroboré la découverte que les ARNm synthétiques coiffés contenant des nucléobases modifiées bénéficient d’une faible activation des réponses antivirales29,30. Bien qu’une faible immunogénicité puisse ne pas être pertinente dans un essai in vitro, elle devient cruciale lorsque l’objectif ultime est de développer une thérapie cliniquement pertinente pouvant être utilisée dans des cellules vivantes.
CIRCLE-seq présente de nombreux avantages par rapport aux méthodes similaires. Par exemple, Digenome-seq séquence à la fois l’ADNg clivé par nucléase et l’ADNg non clivé, en utilisant ~400 millions de lectures21. Il en résulte un bruit de fond élevé, ce qui rend difficile le filtrage des sites de coupe de bonne foi à basse fréquence. CIRCLE-seq n’utilise que ~3 à 5 millions de lectures en raison de l’enrichissement de l’ADNg clivé par nucléase, ce qui entraîne un faible bruit de fond. De plus, Digenome-seq et une méthode similaire, SITE-seq, reposent sur le séquençage d’une seule extrémité d’ADN clivée par nucléase. En revanche, les lectures CIRCLE-seq incluent les deux extrémités du site de coupe, ce qui permet d’identifier les sites hors cible sans avoir besoin d’une référence 21,22,26.
L’un des avantages de CIRCLE-seq est sa plus grande sensibilité par rapport aux méthodes qui reposent sur la culture cellulaire, telles que GUIDE-seq. Lorsque les deux méthodes ont été comparées, CIRCLE-seq a été en mesure de capturer tous les sites hors cible détectés par GUIDE-seq et de découvrir d’autres sites de clivage non intentionnels que GUIDE-seq avait manqués. Cependant, une différence notable est que GUIDE-seq peut être entravé par le paysage épigénétique, tandis que CIRCLE-seq peut accéder à l’ensemble du génome.
En tant que test in vitro , CIRCLE-seq présente plusieurs limites, dont la première est la détection des faux positifs. Alors que l’épigénétique entrave l’activité des nucléases à certains sites in vivo, les ultrasons éliminent ces obstacles in vitro, permettant une activité hors cible à des endroits qui ne sont normalement pas accessibles dans un contexte cellulaire. De plus, Cas9 est présent à des concentrations élevées dans cet essai in vitro , ce qui permet un clivage qui ne serait pas possible autrement in vivo. Ce test nécessite également une quantité relativement importante d’ADNg de départ, ce qui, en fonction des ressources disponibles, peut annuler l’utilisation de ce protocole. Enfin, il est possible que certains sites hors cible soient indétectables en raison des limites des technologies actuelles de séquençage de nouvelle génération.
Une étude récente a utilisé une approche in silico dont l’algorithme a identifié un certain nombre de paramètres pertinents avec lesquels comparer différentes méthodes de caractérisation des nucléases, notamment CIRCLE-seq et GUIDE-seq31. Deux des paramètres pertinents étaient l'«enrichissement en site coupé » et le « % de faux positifs ». Il est intéressant de noter que le taux de faux positifs de CIRCLE-seq a été calculé à 88 %, mais que son enrichissement en site coupé était beaucoup plus élevé que celui des autres méthodes in vitro . L’analyse comparative de chaque méthode a révélé que GUIDE-seq était le plus performant, car il a démontré la plus grande spécificité sur la cible avec seulement un taux modéré de faux positifs32. Cela n’invalide pas CIRCLE-seq, mais fait plutôt allusion à la possibilité d’utiliser CIRCLE-seq et GUIDE-seq en tandem, validant les résultats de CIRCLE-seq avec GUIDE-seq puisque le premier a une sensibilité plus élevée, tandis que le second est une méthode basée sur les cellules avec un enrichissement élevé du site de coupe. Les données indiquent également que le séquençage de nouvelle génération (NGS) basé sur les amplicons devrait être la méthode privilégiée pour identifier les modifications hors cible authentiques sur les sites candidats potentiels31. Ces données suggèrent une stratégie potentielle d’utilisation de CIRCLE-seq, suivi de GUIDE-seq, puis du NGS basé sur l’amplicon pour examiner les effets hors cible.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous exprimons notre profonde gratitude pour le soutien financier fourni par les National Institutes of Health (R01AR078551 et T32AR007411), l’Association de recherche sur l’épidermolyse bulleuse dystrophique (DEBRA) Autriche, le Gates Grubstake Fund et le Gates Frontiers Fund.
matériels
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
Références
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185(2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856(2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786(2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058(2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157(2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474(2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745(2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon