Method Article
CIRCLE-Seq для исследования нецелевого редактирования генов
В этой статье
Резюме
Существенным препятствием для таких технологий, как CRISPR, являются нецелевые события, которые могут нарушить работу жизненно важных генов. «Циркуляризация для сообщения in vitro об эффектах расщепления методом секвенирования» (CIRCLE-seq) — это метод, предназначенный для выявления непреднамеренных участков расщепления. Этот метод картирует полногеномную активность CRISPR-Cas9 с высокой чувствительностью и без смещения.
Аннотация
Циркуляризация для сообщения in vitro об эффектах расщепления методом секвенирования (CIRCLE-seq) — это новый метод, разработанный для беспристрастной идентификации непреднамеренных сайтов расщепления CRISPR-Cas9 путем целенаправленного секвенирования расщепленной ДНК CRISPR-Cas9. Протокол включает в себя циркуляризацию геномной ДНК (гДНК), которая впоследствии обрабатывается белком Cas9 и направляющей РНК (гРНК), представляющей интерес. После обработки расщепленная ДНК очищается и подготавливается в виде библиотеки для секвенирования Illumina. В процессе секвенирования генерируются парные чтения, которые предоставляют исчерпывающие данные о каждом участке расщепления. CIRCLE-seq имеет ряд преимуществ по сравнению с другими методами in vitro , включая минимальные требования к глубине секвенирования, низкий фон и высокую обогащенность гДНК, расщепленной Cas9. Эти преимущества повышают чувствительность при выявлении как преднамеренных, так и непреднамеренных событий расщепления. В этом исследовании представлена всесторонняя, пошаговая процедура изучения нецелевой активности CRISPR-Cas9 с помощью CIRCLE-seq. Например, этот протокол валидируется путем картирования непреднамеренных сайтов расщепления CRISPR-Cas9 по всему геному во время модификации локуса AAVS1 . Весь процесс CIRCLE-seq может быть завершен за две недели, что дает достаточно времени для роста клеток, очистки ДНК, подготовки библиотеки и секвенирования Illumina. Ввод данных секвенирования в конвейер CIRCLE-seq упрощает интерпретацию и анализ сайтов расщепления.
Введение
За последние двадцать лет в геномной инженерии был достигнут значительный прогресс, главным из которых стало открытие в 2012 году кластеризованных регулярно чередующихся коротких палиндромных повторов (CRISPR)-Cas91. Используя программируемую природу бактериальных ДНК-эндонуклеаз, технология CRISPR-Cas9 позволяет точно нацеливаться и модифицировать практически любую последовательность ДНК. С момента своего создания система была оптимизирована для использования только эндонуклеазы Cas9 и направляющей РНК (гРНК) для редактирования конкретных областей генома. Потенциал CRISPR-Cas9 в качестве лечебной терапии был продемонстрирован в клинических испытаниях для лечения различных заболеваний, таких как врожденный амавроз Лебера, транстиретиновый амилоидоз и серповидноклеточная анемия, среди прочих 2,3,4.
CRISPR-Cas9 индуцирует двухцепочечные разрывы (DSB), которые обычно разрешаются одним из двух механизмов: подверженным ошибкам негомологичном соединении концов (NHEJ) или более точной гомологически направленной репарации (HDR), при условии наличия матричной ДНК. Тенденция CRISPR-Cas9 вызывать NHEJ-ассоциированные вставки и делеции (инделы), наряду с расщеплением в непреднамеренных геномных участках, ограничивает его применение в клинических условиях 5,6,7,8,9,10. Кроме того, непреднамеренные геномные модификации могут создавать загадочные сайты сплайсинга, бессмысленные или миссенс-мутации, вызывать хромоттрипсис или придавать онкогенный потенциал клеткам — результаты, которые наблюдались в нескольких исследованиях редактирования генома 11,12,13,14,15 . В заключение следует отметить, что точная идентификация нецелевой активности CRISPR-Cas9 имеет решающее значение для его клинического применения, особенно в системной генной терапии, которая может изменить миллиарды клеток.
Для идентификации сайтов-расщепления CRISPR-Cas9 вне мишени могут быть использованы различные методы, в том числе полногеномная непредвзятая идентификация двухцепочечных разрывов (GUIDE)-seq16, в которой используются двухцепочечные олигодоксинуклеотиды для маркировки DSB в живых клетках. Тем не менее, критика этого метода заключается в том, что ложные срабатывания могут возникать из-за случайных DSB или артефактов ПЦР, которые должны быть отброшены, исключив захваченные сайты, которые демонстрируют плохое сходство с целевыми сайтами. Метод, основанный на использовании интегразного дефективного лентивирусного вектора (IDLV), менее чувствителен и с вероятностью пропускает многие нецелевые участки17. Другие методы in situ, такие как DSBCapture, BLESS и BLISS 18,19,20, задействуют фиксированные клетки и маркируют DSB напрямую, однако они ограничены их зависимостью от немедленного захвата DSB и отсутствием экзогенной ДНК. Digenome-seq21, метод in vitro, и селективное обогащение и идентификация меченых геномных концов ДНК методом секвенирования (SITE-seq)22 предоставляют решения для секвенирования, но имеют свои ограничения в отношении фонового шума и анализа одного конца, соответственно. Обнаружение in situ немишеней Cas и верификация путем секвенирования (Discover-Seq)23 обеспечивает идентификацию активности Cas9 in vivo и in situ посредством связывания MRE11, но обнаруживает только те DSB, которые существуют на момент подготовки образца24. Наконец, Inference of CRISPR Edits (ICE) использует биоинформатический подход для надежного анализа правок CRISPR с использованием данных Сэнгера25.
В этой статье подробно описана процедура циркуляризации для In vitro Reporting of Cleavage Effects by Sequencing (CIRCLE-seq): метод in vitro, который чувствительно и беспристрастно отображает полногеномную нецелевую активность нуклеазы Cas9 в комплексе с представляющей интерес гРНК26. Этот подход начинается с культивирования интересующих клеток и выделения ДНК, за которым следует случайный сдвиг с помощью сфокусированного ультразвука, а затем лечение экзонуклеазой и лигазой. В конечном итоге в результате этого процесса образуются кольцевые двухцепочечные молекулы ДНК, которые затем очищаются с помощью плазмидной ДНКазной обработки. Затем эта кольцевая ДНК подвергается воздействию комплекса Cas9-gRNA, который расщепляется как в преднамеренных, так и в непреднамеренных местах расщепления, оставляя после себя обнаженные концы ДНК, которые действуют как субстраты для лигирования адаптера Illumina. В результате этого процесса создается разнообразная библиотека геномной ДНК (гДНК), содержащая оба конца каждого нуклеазно-индуцированного DSB, гарантируя, что каждое чтение содержит всю информацию, необходимую для каждого сайта расщепления. Это позволяет использовать секвенирование Illumina с более низкими требованиями к покрытию секвенирования, что отличает CIRCLE-seq от других подобных методов, упомянутых выше. Важно отметить, что, хотя CIRCLE-seq действительно имеет более высокую нецелевую чувствительность, чем другие протоколы в качестве метода in vitro , это происходит за счет более высоких ложноположительных результатов из-за отсутствия эпигенетического ландшафта, который присутствует в других методах, таких как GUIDE-seq16. Кроме того, репарация ДНК DSB и связанные с ней механизмы отсутствуют в CIRCLE-seq, что отменяет инделы или надлежащую репарацию, которые в противном случае наблюдались бы.
В дополнение к описанию пошагового протокола для выполнения CIRCLE-seq, протокол валидируется путем идентификации непреднамеренных сайтов расщепления CRISPR-Cas9 по всему геному, которые происходят во время модификации локуса AAVS1 , в качестве примера. Этот простой в использовании протокол содержит подробные инструкции, от культивирования индуцированных плюрипотентных стволовых клеток (ИПСК) и выделения гДНК до циркуляризации гДНК, расщепления Cas9-гРНК, подготовки библиотеки, секвенирования и анализа трубопроводов. Учитывая низкие требования к покрытию секвенирования, CIRCLE-seq доступен для любой лаборатории, имеющей доступ к секвенированию нового поколения.
протокол
Подробная информация о реагентах, расходных материалах и оборудовании, использованных для данного исследования, приведена в Таблице материалов.
1. Культивирование клеток (5 дней)
- Включите негативный контроль во весь этот протокол. Подготовьте достаточное количество клеток для дополнительных 25 мкг гДНК (~2,0e7 клеток на образец).
- Культивирование ИПСК в соответствии с установленным протоколом27. Соберите клетки и повторно суспендируйте их в 10 мл PBS. Пипеткой наберите образец клеток объемом 6 мкл и ресуспендируйте в соотношении 1:1 трипановым синим. Используйте автоматический счетчик клеток для подсчета образца.
- Аликвотьте 2 x 10по 7 ячеек в пробирке, затем вращайте при 300 x g в течение 3 минут при 25 °C (Комнатная температура (RT)). Этого достаточно для нескольких репликаций. Отсоедините пипетку и выбросьте надосадочную жидкость.
2. Выделение геномной ДНК (1 день)
- Используйте имеющийся в продаже набор для очистки ДНК для выделения гДНК, следуя инструкциям производителя:
- Добавьте 200 мкл PBS в коническую пробирку объемом 15 мл, содержащую клеточную гранулу, и восстановите. Затем пипеткой в пробирку ввели 3 мл буфера для лизиса клеток и 15 мкл протеиназы К. Переверните трубку 25 раз, чтобы тщательно перемешать. Поместите пробирку в шейкер на водяной бане, установленный на 55 °C и 150 об/мин, на 3 часа или на ночь для оптимального выхода ДНК.
- Пипетку в 15 мкл РНКазы А. Инвертировать 25 раз. Поставьте на водяную баню при температуре 37 °C на 1 час.
- Охладите образец на льду в течение 5 минут. Затем добавьте 1 мл раствора для осаждения белка, вонзите на высокой скорости в течение 20 с и центрифугируйте при 2000 x g в течение 10 минут при RT. Белки должны образовать видимую компактную гранулу на дне пробирки. Если гранула не видна, инкубируйте образец на льду еще 5 минут и снова центрифугируйте.
- Добавьте 3 мл 100% изопропанола в новую коническую пробирку объемом 15 мл. Осторожно введите надосадочную жидкость с шага 2.1.3 в пробирку. Переверните пробирку 50 раз, чтобы перемешать, затем центрифугируйте при температуре 2000 x g и комнатной температуре в течение 3 минут. Не повреждая гранулу ДНК, осторожно аспирируйте надосадочную жидкость с помощью пастеровской пипетки, подключенной к вакуумной ловушке, и переверните пробирку на чистую безворсовую салфетку.
- Нанесите пипеткой 3 мл 70% (v/v) этанола на гранулу ДНК и переверните 10 раз для промывки. Центрифугируйте при 2 000 x g в течение 3 мин при RT. Затем аккуратно слейте надосадочную жидкость.
- Держите пробирку открытой и дайте полученной грануле ДНК высохнуть в течение 30 минут, убедившись, что весь этанол полностью испарился. Пипетку в 50 мкл раствора для гидратации ДНК и тщательно перемешайте с помощью щадящего пипетирования.
- Растворите ДНК, поместив образец в шейкер с водяной баней при температуре 65 °C на 1 час, затем оставьте образец при температуре RT на ночь. Центрифугируйте образец при RT и 2000 x g в течение 1 минуты и используйте набор для анализа dsDNA BR с соответствующими пробирками для количественного определения выделенной ДНК с помощью флуориметра.
3. Подготовка гРНК (7 дней)
- Закажите интересующую вас синтетическую гРНК из коммерческого источника (см. Таблицу материалов). Этот протокол также совместим с crRNA/tracrRNA.
4. Тест на расщепление гРНК in vitro
ПРИМЕЧАНИЕ: Здесь используется мишень в гене AAVS1 . Чтобы нацелиться на другие гены, представляющие интерес, разработайте праймеры (табл. 1) для амплификации целевой области и замените праймеры на следующих этапах специальными праймерами.
- Приготовьте ПЦР-реакцию: смешайте 25 мкл праймера AAVS1 R Праймера (конечная концентрация 0,1 мкМ), 5 мкл гДНК (100 нг, 20 нг/л, начиная с этапа 2.1.6) и 19 мкл нуклеазы H 2 O (общий объем: 50 μл).
- Используйте следующие параметры амплификатора: денатурация: 98 °C в течение 2 мин (1 цикл), денатурация: 98 °C в течение 10 с (10 циклов), отжиг: 72-62 °C (-1 °C/цикл) в течение 15 с (10 циклов), расширение: 72 °C в течение 30 с (10 циклов), денатурация, 98 °C в течение 10 с (30 циклов). Отжиг: 65 °C в течение 15 с (30 циклов), расширение: 72 °C в течение 30 с (30 циклов), окончательное расширение: 72 °C в течение 5 мин (1 цикл), удержание: 4 °C на неопределенный срок.
- Используйте шарики AMPure XP для очистки продукта ПЦР-реакции. Во-первых, пипетируйте в 1,8-кратном объеме или 90 мкл шариков XP в продукт для ПЦР. Пипеткой десять раз тщательно перемешать. Оставьте смесь на RT на 5 минут для инкубации.
- С помощью магнитного штатива отделите шарики от раствора, поместив реакционную пластину ПЦР на магнит на 3 минуты. Отпижите очищенный раствор пипеткой и выбросьте. Добавьте в гранулы 200 μл 80% этанола (v/v) в гранулы, инкубируйте в течение 30 с, а затем удалите этанол. Повторите этот этап промывки дважды, чтобы обеспечить полное удаление этанола.
- Дайте образцам высохнуть естественным путем в течение 3 минут, поместив пластину на магнит. Снимите пластину с магнита и добавьте 40 μL TE буфера, pH 8,0. Перемешайте, пипетируя вверх и вниз десять раз. Оставьте образец на RT в течение 2 минут.
- Поместите реакционную пластину ПЦР на магнит еще на минуту. Через 1 минуту переложите надосадочную жидкость в новую тарелку. Измерьте выход очищенной ПЦР с помощью спектрофотометра и проанализируйте его на TapeStation с помощью полоски для оптической трубки с колпачком из оптической трубки, а также высокочувствительной ленты D1000 ScreenTape и высокочувствительных реагентов D1000 (лестница и буфер), следуя инструкциям производителя. Приготовленный образец хранят при температуре -20 °C до нескольких месяцев.
- Разбавьте белок нуклеазы Cas9 до 1 мкМ следующим образом: смешайте 2 мкл 10x буфера Cas9 (конечная концентрация 1x), 1 мкл Nuclease Cas9, S. pyogenes (конечная концентрация 1 мкМ) и 17 мкл безнуклеазного H2O в общем объеме 20 мкл.
- Выполните процедуру, не содержащую РНКазы, чтобы предотвратить деградацию гРНК. Разбавляют гРНК (начиная со стадии 3.1) до 3 мкМ вН2О до общего объема 10 мкл.
ПРИМЕЧАНИЕ: Используйте следующую формулу для оценки молекулярной массы гРНК: Молекулярная масса ссРНК (г/моль) = (длина ссРНК (nt) x 321,47 г/моль) + 18,02 г/моль. Для справки, длинная гРНК длиной 104 нт с концентрацией 3 мкМ составляет примерно 100 нг/мкл.
5. Сдвиг ДНК (3 часа)
- Подготовьте ME220, сначала наведя на себя рычаг управления. Затем заполните резервуар очищенным деионизированным H2O. На ноутбуке станции управления откройте Water Works и нажмите « Заполнить». Отрегулируйте температуру до 4,5 °C.
- Перенесите 25 г гДНК в микропробирку (microtube-130 AFA Fiber Screw-Cap). Затем наполните пробирку до общего объема 130 мкл 1x TE. Используйте следующие условия для сдвига ДНК до средней длины около 300.н.: установите Продолжительность на 10 с; Пиковая мощность до 70; Коэффициент пошлины % до 20; Циклы/Burst до 50; все это автоматически установит среднюю мощность на 14,0.
6. Очищение срезанной геномной ДНК (1 ч)
- Разделите срезанную геномную ДНК на две части по 65 мкл каждая. Очистите, используя в 1,8 раза больше объема шариков XP (117 μл), следуя процедуре, описанной в шагах 4.3-4.6. Перенесите надосадочную жидкость на новую ПЦР-планшет и измерьте количество с помощью спектрофотометра.
- Пропустите 1 мкл элюированной срезанной гДНК на ленточную станцию в соответствии с инструкциями производителя, чтобы убедиться, что гДНК срезана до широкого распределения около 300.о. При необходимости храните срезанную гДНК при температуре -20 °C до нескольких месяцев.
7. Подготовка библиотеки CIRCLE-seq (3 дня)
- Отжиг адаптера шпильки
- Ресуспендируйте oSQT1288 (Таблица 1), адаптер для шпилек, до конечной концентрации 100 мкМ в 1x TE.
- Выполните отжиг адаптером следующим образом: смешайте 40 мкл oSQT1288 (конечная концентрация 40 мкМ), 10 мкл 10x STE (конечная концентрация 1x) и 50 мкл Nuclease-Free H2O в общем объеме 100 μл.
- Используйте следующие параметры отжига: 95 °C в течение 5 минут, -1 °C в минуту в течение 70 циклов, держать при 4 °C неограниченное время.
- Выполнить Завершить ремонт . Используйте набор для подготовки библиотеки HTP без ПЦР и приготовьте мастер-смесь для конечного ремонта.
- Смешайте 8 мкл Nuclease-Free H2O, 7 мкл 10-кратного буфера для эндокоррекции (конечная концентрация 1x) и 5 мкл смеси ферментов для конечного восстановления (окончательный объем 20 мкл общей мастер-смеси для конечного восстановления).
- Пипетка 20 мкл мастер-смеси для конечного восстановления в образец срезанной гДНК на этапах 4.3-4.6. Смешайте 20 μL End-Repair Master Mix с 50 μL срезанной гДНК до конечного объема 70 μL.
- Поместите смесь в амплификатор при температуре 20 °C на 30 минут, затем подержите при температуре 4 °C неопределенно долго.
- Добавьте в 1,7 раза, или 120 μл, гранул XP и следуйте шагам очистки из шагов 4.3-4.6. Элюировать 42 мкл TE, pH 8,0. Убедитесь, что шарики остаются в растворе для следующего шага.
- Выполните A-tailing. Используя набор для подготовки библиотеки HTP (без ПЦР), приготовьте мастер-смесь для хвостохранилища.
- Смешайте 5 мкл 10x A-tailing буфера (конечная концентрация 1x) и 3 мкл фермента A-tailing (общий конечный объем 8 мкл A-tailing Master Mix).
- Пипеткой введите 8 мкл A-tailing Master Mix в каждый образец ДНК, содержащий гранулы, начиная с этапа 7.2.4, смешав 8 мкл A-tailing Master Mix с 42 μL гранул, содержащих End-Repaired DNA (общий конечный объем 50 мкл). Поместите в амплификатор при температуре 30 °C на 30 минут. Держите при температуре 4 °C неограниченное время.
- Пипетка 1,8 объема, или 90 μл, раствора PEG/NaCl SPRI (компонент набора для подготовки библиотеки HTP (без ПЦР; 96 реакций)) в ДНК с А-образным хвостом. Очистите ДНК с А-образным хвостом в соответствии с шагами 4.3-4.6. Элюируйте ДНК с А-образным хвостом в 30 мкл TE, pH 8,0. Оставьте бусины в растворе для следующего шага.
- Выполните перевязку адаптера. Используя набор для подготовки библиотеки HTP (без ПЦР), приготовьте мастер-смесь для лигирования адаптера.
- Смешайте 10 мкл 5-кратного буфера для лигирования (конечная концентрация 1x), 5 мкл ДНК-лигазы и 5 мкл отожженного адаптера для шпилек (40 мкМ) на шаге 7.1.3. Обеспечьте конечную концентрацию 4 мкМ для общей мастер-смеси Adapter Ligation Master Mix 20 μл.
- Пипеткой 20 мкл мастер-смеси для лигирования адаптера в каждый элюированный образец ДНК, содержащий гранулы, начиная с шага 7.3.3 (общий конечный объем 50 мкл на образец).
- Поместите в амплификатор при температуре 20 °C на 1 час. Держите при температуре 4 °C неограниченное время.
- Перенесите 1x объем, или 50 μл, раствора PEG/NaCl SPRI в ДНК, лигированную адаптером, и очистите в соответствии с этапами 4.3-4.6. Разбавьте 30 мкл TE, pH 8,0, и сцедите надосадочную жидкость в новую полузакрытую ПЦР-планшет. Объедините и количественно оцените ДНК с помощью анализа dsDNA BR. При необходимости храните очищенную ДНК, лигированную адаптером, до 1 месяца при температуре -20 °C.
- Приготовьте мастер-смесь для лямбда-экзонуклеазы/экзонуклеазы I (E. coli) (функционирует в устранении одно- или двухцепочечной ДНК без лигированных с обоих концов адаптеров).
- Возьмите 1 г адаптерной лигированной ДНК с шага 7.4.4, разбавив ее до 40 μл. Смешайте 5 мкл 10x реакционного буфера экзонуклеазы I (конечная концентрация 1x), 4 мкл экзонуклеазы лямбда (конечная концентрация 0,4 ед/мкл) и 1 мкл экзонуклеазы I (E. coli) (конечная концентрация 0,4 ед/мкл) с общим объемом мастер-смеси экзонуклеазы лямбда/экзонуклеазы I 10 мкл.
- Пипетка 10 мкл смеси Lambda Exonuclease/Exonuclease I Master Mix в 40 мкл (1 мкг) адаптерно-лигированной ДНК (общий объем 50 мкл). Поместите в амплификатор при температуре 37 °C на 1 час, затем при 75 °C на 10 минут. Держите при температуре 4 °C неограниченное время.
- Пипетка в 1,8 объема, или 90 μл, гранул XP в ДНК, обработанную экзонуклеазой/экзонуклеазой I. Очищайте в соответствии с инструкциями, приведенными в шагах 4.3-4.6. Элюировать в 40 мкл TE, pH 8,0. Убедитесь, что шарики остаются в растворе для следующего ферментативного этапа.
- Лечите ферментом USER и полинуклеотидкиназой Т4 (PNK). Приготовьте мастер-смесь USER/T4 PNK (требуется для снятия выступов 4.о. и подготовки готовых к лигированию концов ДНК, необходимых для последующей реакции лигирования).
- Смешайте 5 мкл 10x T4 буфера ДНК-лигазы (конечная концентрация 1x), 3 мкл фермента USER (конечная концентрация 0,05 ед/мкл) и 2 мкл T4 PNK (конечная концентрация 0,4 ед/мкл), чтобы общий объем мастер-смеси USER/PNK составил 10 мкл.
- Пипетка 10 мкл USER Enzyme/T4 PNK Master Mix в 40 мкл обработанного лямбда и экзонуклеазы I образца ДНК, содержащего гранулы из этапа 7.5.3 общего объема 50 мкл. Поместите в амплификатор при температуре 37 °C на 1 час. Держите при температуре 4 °C неограниченное время.
- Пипетку в объеме 1,8x или 90 мкл раствора PEG/NaCl SPRI в ДНК, обработанную USER/T4 PNK, и очистите в соответствии с этапами 4.3-4.6. Разбавьте в 35 мкл TE, pH 8,0. Сцедите надосадочную жидкость в новую ПЦР-планшет с полуюбкой. Объедините и количественно оцените ДНК с помощью анализа dsDNA HS.
- Выполнение внутримолекулярной циркуляризации. Приготовьте мастер-смесь для циркуляризации.
- Смешайте 8 мкл не содержащего нуклеазы H2O, 10 мкл 10-кратного буфера Т4-ДНК-лигазы (конечная концентрация 1х) и 2 мкл ДНК-лигазы Т4 (конечная концентрация 8 ЕД/мкл), чтобы общий объем циркуляризационной мастер-смеси составил 20 мкл.
- Пипетка 20 мкл Circularization Master Mix до 500 нг ДНК, обработанной USER/PNK, начиная с шага 7.6.3. Разведите 500 нг ДНК, обработанной USER/PNK, в 80 μл, затем добавьте 20 μL Circularization Master Mix (общий объем 100 μл). Инкубировать в амплификаторе при температуре 16 °C в течение 16 ч (на ночь).
- Добавьте 1 объем, или 100 μл, гранул XP в циркуляризованную ДНК и очистите в соответствии с шагами 4.3-4.6. Разбавить в 38 мкл TE, pH 8,0. Сцедите надосадочную жидкость в новую ПЦР-планшет с полуюбкой.
- Лечение плазмидно-безопасной АТФ-зависимой ДНКазой. Приготовьте безопасную для плазмид АТФ-зависимую ДНКазную мастер-смесь (требуется для деградации остаточной линейной ДНК).
- Смешайте 5 мкл 10-кратного плазмидного безопасного реакционного буфера (конечная концентрация 1x), 2 мкл АТФ (конечная концентрация 1 мМ) и 5 мкл плазмидно-безопасной АТФ-зависимой ДНКазы (конечная концентрация 1 ЕД/мкл), чтобы получить общий объем плазмидно-безопасной мастер-смеси 12 мкл.
- Пипетка 12 мкл АТФ-зависимой ДНКазы до 38 мкл циркуляризованной ДНК с шага 7.7.3 (общий объем 50 мкл). Выдерживать в амплификаторе при температуре 37 °C в течение 1 часа, затем при 70 °C в течение 30 минут. Держите при температуре 4 °C неограниченное время.
- Пипетку в 1 объеме или 50 мкл шариков XP в ДНК, обработанную плазмидно-безопасной АТФ-зависимой ДНКазой, и очистите в соответствии с шагами 4.3-4.6. Разбавьте в 15 мкл TE, pH 8,0. Сцедите надосадочную жидкость в новую ПЦР-планшет с полуюбкой.
- Объедините ДНК и количественно оцените с помощью анализа dsDNA HS. При необходимости храните циркуляризованную ДНК при температуре -20 °C до нескольких месяцев.
8. Расщепление ферментативно очищенной, циркуляризированной гДНК in vitro (2 ч)
- Выполнение расщепления in vitro с помощью комплекса Cas9:gRNA. Приготовьте мастер-смесь для декольте in vitro . Смешайте 5 мкл 10x буфера Cas9 (конечная концентрация 1x), 4,5 мкл S. pyogenes Cas9 (конечная концентрация 90 нМ) и 1,5 мкл гРНК (конечная концентрация 90 нМ) так, чтобы общий объем мастер-смеси Craavage составил 11 мкл.
- Выдержите мастер-смесь для расщепления при температуре RT в течение 10 минут для образования комплексов Cas9:gRNA RNP.
- Разбавьте 125 нг ДНК, обработанной плазмидной ДНКазой, начиная с этапа 7.8.3 до конечного объема 39 мкл. Затем добавьте 11 μL Craavage Master Mix к 39 μL Plasmid-Safe DNase-обработанной ДНКазы общим объемом 50 μL.
ПРИМЕЧАНИЕ: На этом этапе включите отрицательный контрольный образец, который содержит циркуляризованную ДНК, смешанную с буфером Cas9, без комплекса Cas9:gРНК. - Выдерживать в амплификаторе в течение 1 ч при температуре 37 °C. Поддерживать при температуре 4 °C неограниченное время. Добавьте 50 мкл (1x объем) гранул XP к ДНК, расщепленной in vitro, и очистите ДНК, следуя шагам 4.3-4.6. Разбавьте 42 мкл TE буфера, pH 8,0. Убедитесь, что шарики остаются в растворе для следующего шага.
9. Подготовка библиотеки секвенирования нового поколения (4 - 6 ч)
- Выполните A-tailing. Приготовьте смесь A-tailing Master Mix.
- Смешайте 5 мкл 10x A-tailing Buffer (конечная концентрация 1x) и 3 мкл A-tailing Enzyme (общий объем мастер-смеси A-tailing 8 μL).
- Дозируйте 8 μL A-tailing Master Mix на 42 μL элюированного образца ДНК, содержащего гранулы, начиная с шага 8.4 (общий объем 50 μL). Поместите в термоамплификатор на 30 минут при температуре 30 °C. Держите при температуре 4 °C неограниченное время.
- Пипетируйте в 1,8 раза больше раствора PEG/NaCl SPRI, или 90 μл, в ДНК с А-образным хвостом и очистите ДНК в соответствии с шагами 4.3-4.6. Разбавить в 25 мкл TE, pH 8,0. Убедитесь, что шарики находятся в растворе для следующего шага.
- Выполните перевязку адаптера. Приготовьте мастер-смесь для лигирования адаптера.
ПРИМЕЧАНИЕ: Одноразовые аликвоты адаптеров NEB должны быть подготовлены таким образом, чтобы предотвратить образование димеров адаптера, вызванных гидролизом 3'T', вызванным замерзанием-оттаиванием.- Смешайте 10 μL 5x Ligation Buffer (конечная концентрация 1x), 5 μL DNA Ligase и 10 μL Adapter для секвенирования (конечная концентрация 3 μM), всего 25 μL.
- Пипетка 25 мкл мастер-смеси для лигирования адаптера в 25 мкл образца ДНК с А-образным хвостом, содержащего шарики, из этапа 9.1.3. Поместите в амплификатор на 1 час при температуре 20 °C. Держите при температуре 4 °C неограниченное время.
- Пипетируйте 1x объем, или 50 μл, раствора PEG/NaCl SPRI в ДНК, лигированную адаптером, и очистите ДНК в соответствии с этапами 4.3-4.6. Элюировать в 47 мкл TE, pH 8,0. Убедитесь, что шарики остаются в растворе для следующего ферментативного этапа.
- Проведите обработку ферментом USER Enzyme (образует однонуклеотидный разрыв в остатках урацила).
- Добавьте 3 мкл фермента USER (входит в комплект Dual Index Primers Kit) в образец ДНК, лигированный адаптером, содержащий гранулы из этапа 9.2.3. Выдерживать при температуре 37 °C в течение 15 минут.
- Добавьте 35 мкл (0,7x объема) раствора PEG/NaCl SPRI в ДНК, обработанную ферментом USER, и очистите в соответствии с шагами 4.3-4.6. Разбавьте в 20 мкл TE буфера, pH 8,0. Перенесите надосадочную жидкость на новую ПЦР-планшет с полуюбкой и измерьте концентрацию ДНК с помощью анализа dsDNA HS. Ожидаемая концентрация должна составлять около 2-5 нг/мкл.
- (Дополнительный) Прежде чем перейти к следующему этапу, можно провести подбор размера ДНК с помощью PippinHT. Используйте кассету с 1,5% PippinHT с диапазоном размеров 250-850.о. Полученные образцы могут быть непосредственно использованы в ПЦР на следующем этапе.
- Проведение ПЦР для добавления штрих-кода
ПРИМЕЧАНИЕ: Убедитесь, что выбранные комбинации последовательностей праймеров для каждого образца уникальны. По возможности каждый образец должен иметь уникальные штрих-коды i5 и i7.- Подготовьте PCR Master Mix для добавления штрихкодов с парным индексом. Смешайте 5 μл безнуклеазного H2O, 25 μл 2x HotStart Ready Mix (конечная концентрация 1x), 5 μл i5 Primer (конечная концентрация 1 μM) и 5 μL i7 Primer (конечная концентрация 1 μM) (общий объем мастер-смеси 40 μL).
- Пипетка 40 мкл PCR Master Mix на 10 мкл очищенной ДНК, обработанной ферментом USER (около 20 нг) с шага 9.3.2 (общий объем 50 мкл).
- Выберите следующие условия термоциклирования ПЦР: денатурация: 98 °C в течение 45 с для 1 цикла, денатурация: 98 °C в течение 15 с для 20 циклов, отжиг: 65 °C в течение 30 с для 20 циклов, расширение: 72 °C в течение 30 с для 20 циклов, окончательное расширение: 72 °C в течение 1 мин для 1 цикла, удержание: 4 °C на неопределенный срок.
- Добавьте 0,7x объема, или 35 μл, гранул XP в продукт для ПЦР и очистите в соответствии с шагами 4.3-4.6. Элюировать в 30 мкл TE, pH 8,0. Сцедите надосадочную жидкость в новую ПЦР-планшет с полуюбкой. При необходимости храните циркуляризованную ДНК при температуре -20°C до нескольких месяцев.
ПРИМЕЧАНИЕ: Запустите образец ПЦР на Tapestation, чтобы проверить качество библиотеки и оценить образование димеров адаптера. Если димеры адаптера обнаружены, повторите шаг 9.5.4.
10. Количественная оценка библиотек CIRCLE-seq методом капельной цифровой ПЦР (dd_PCR) (6 ч)
ПРИМЕЧАНИЕ: Количественная оценка также может быть выполнена с использованием количественной ПЦР, Tapestation или аналогичного метода.
- Начните с 5 μL ДНК из библиотеки (шаг ПЦР 9.5.4), хорошо смешанных с 45 μL безнуклеазного TE, затем сделайте последовательные разведения 1:10 каждого образца в объемах 50 μL, в диапазоне от 10-1 до 10-8 разведения.
- Настройте складское решение dd_PCR Master Mix. Смешайте 11 мкл 2x dd_PCR смеси для зондов (конечная концентрация 1x), 0,055 мкл зонда oSQT1310 (конечная концентрация 250 нМ), 0,055 мкл зонда oSQT1311 (конечная концентрация 250 нМ), 0,099 мкл праймера oSQT1274 (конечная концентрация 450 нМ), 0,099 мкл праймера oSQT1275 (конечная концентрация 450 нМ) и 6,292 мкл безнуклеазного H2O, общий объем dd_PCR Master Mix составляет 17,6 μл. Подготовьте мастер-смесь для всех образцов, чтобы убедиться, что объемы достаточны для точного пипетирования.
- Проводят анализ трех самых низких разведений (10-6,10-7 и 10-8) в двух экземплярах (в 96-луночном планшете). Необходимо использовать элемент управления, не являющийся шаблоном (NTC).
- Пипетка 17,6 мкл мастер-смеси dd_PCR каждому образцу следующим образом: Смешайте 17,6 мкл с 4,4 мкл образца (добавив в лунку с NTC безнуклеазный H2O), в общем объеме 22 мкл. Запечатайте планшет, затем центрифугируйте при 2000 x g в течение 1 минуты при RT.
- Выполнение генерации капель, термоциклирования и анализа. С помощью системы ПЦР Droplet Reader перенесите картридж DG8 (8-луночный) в держатель картриджа. В масляный ряд картриджа налейте 70 μл масла для генерации капель для зондов.
ПРИМЕЧАНИЕ: Возьмите 20 мкл образца с шага 10.1.2. и добавить его в ряд образцов 8-луночного картриджа, накрыв картридж резиновой прокладкой DG8, поместив его в генератор капель, а затем закрыв его, чтобы начать процесс (автоматический). Когда закончите, извлеките картридж и переместите 40 μL из капельного ряда 8-луночного картриджа в 96-луночный ПЦР-планшет с полуюбкой, следя за тем, чтобы пипетировать медленно.- Поместите нагревательный блок в запайщик пластин PX1 PCR. При включении герметик начнет нагреваться до 180 °C. Наденьте термосвариватель фольги на пластину, убедившись, что красная линия находится сверху. Поместите пластину в PX1 и нажмите Seal.
- Выберите следующие условия работы амплификатора: Активация фермента: 95 °C в течение 10 мин в течение 1 цикла, Денатурация: 94 °C в течение 30 с в течение 40 циклов, Отжиг/удлинение: 60 °C в течение 1 мин в течение 40 циклов, Деактивация фермента: 98 °C в течение 10 мин в течение 1 цикла, Удержание: 4 °C в течение неопределенного времени.
- На считывателе капель откройте совместимое программное обеспечение и выберите, какие лунки необходимо считывать. Выберите ABS в качестве типа эксперимента и dd_PCR Supermix для зондов. Выберите Ch1 Unknown для цели 1 и Ch2 Unknown для цели 2. Выберите «Применить», затем «ОК». Вставьте пластину в считыватель капель. Для набора красителей выберите FAM/HEX и нажмите « Запустить».
- Проанализируйте dd_PCR результаты. Забей двойную положительную популяцию капель, используя отрицательный контроль в качестве эталона. Рассчитайте среднее арифметическое повторяющихся значений, затем умножьте на коэффициент разбавления и 5-кратный коэффициент разбавления dd_PCR.
ПРИМЕЧАНИЕ: Общее количество копий на микролитр рассчитывается следующим образом: общее количество копий на микролитр = среднее значение × 5 × коэффициента разбавления, где «среднее значение» представляет собой среднее количественное значение из Ch1 и Ch2. - Объедините все образцы в единую библиотеку при эквимолярных концентрациях. Объединенная библиотека 1x должна содержать около 4,5 x 109 молекул и иметь общий объем 5 μL.
11. Секвенирование нового поколения
- Отправьте образцы для секвенирования во внешнее агентство, убедившись, что отмечены правильные последовательности адаптеров.
12. Анализ данных CIRCLE-seq (1 - 3 ч)
- Установите Python версии 2.7, Burrows-Wheeler Aligner (BWA) и SAMtools. Загрузите референсный геном (например, hg38) из http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz.
Примечание: Если геном целевого вида недоступен, вычислительный рабочий процесс CIRCLE-seq может быть запущен в режиме, не зависящем от ссылки. В этой ситуации этот шаг можно пропустить. - Скачайте и установите конвейер CIRCLE-seq с помощью следующих команд: (1) git clone https://github.com/tsailabSJ/circleseq.git, (2) cd circleseq, (3) pip install -r requirements.txt.
- Создайте файл манифеста в формате YAML (.yaml). Ниже приведен пример манифеста, который можно использовать с примером набора данных, предоставленным в программном обеспечении CIRCLE-seq, для тестирования рабочего процесса.
ПРИМЕЧАНИЕ: (1) референсный геном: data/input/CIRCLEseq_test_genome.fa; (2) analysis_folder: данные/вывод; (3) BWA: BWA; (4) Samtools: Samtools; (5) read_threshold: <ценность>; (6) window_size: <ценность>; (7) mapq_threshold: <ценность>; (8) start_threshold: <ценность>; (9) gap_threshold: <ценность>; (10) mismatch_threshold: <ценность>; (11) merged_analysis: Верно; (12) образцы: U2OS_EMX1; (13) цель: GAGTCCGAGCAGAAGAANGG; (14) Чтение1: данные/ввод/EMX1.r1.fastq.gz; (15) Чтение2: данные/ввод/EMX1.r2.fastq.gz; (16) controlread1: данные/ввод/EMX1_control.r1.fastq.gz; (17) controlread2: данные/ввод/EMX1_control.r2.fastq.gz; (18) описание: U2OS. Использовались следующие значения манифеста: read_threshold: 4, window_size: 3, mapq_threshold: 50, start_threshold: 1, gap_threshold: 3, mismatch_threshold: 6 - Определите файл FASTA референсного генома, выходной каталог для анализа и пути к командам BWA и SAMtools . Определите целевые последовательности и пути к демультиплексированным файлам FASTQ как для нуклеазно-расщепленных, так и для контрольных образцов. Несколько экспериментов могут быть обработаны одновременно в пакетном режиме, если включить их все в один файл манифеста.
- Выполните следующую команду для стандартного анализа, основанного на ссылках: (1) python /path/to/circleseq.py all - manifest; (2) /path/to/manifest.yaml.
- В качестве альтернативы выполните следующую команду для стандартного анализа, не основанного на ссылках: (1) Python /path/to/circleseq.py reference-free - manifest; (2) /путь/к/manifest.yaml.
- При выполнении полного конвейера найдите выходные результаты каждого шага в отдельной output_folder , предназначенной для этого конкретного шага.
Результаты
В данном случае CIRCLE-seq используется для исследования индуцированных нуклеазами сайтов расщепления Cas9 в комплексе с гРНК, предназначенной для нацеливания на аденоассоциированный сайт интеграции вируса 1 (AAVS1) с использованием ДНК, выделенной из индуцированных плюрипотентных стволовых клеток (iPSCs). Эта гРНК ранее была описана в нашей публикации27. Около 25 мкг гДНК было выделено из ИПСК с помощью сфокусированного ультразвука и выбран размер с помощью гранулированной очистки AMPure XP для получения фрагментов размером около 300 бит/с. Из этих 25 мкг ДНК около 2-5 нг ДНК были успешно циркуляризованы для расщепления in vitro Cas9:gРНК. Вся процедура показана на рисунке 1.
После процедуры CIRCLE-seq и анализа с использованием нашего вычислительного процесса, визуализация всех обнаруженных сайтов расщепления на цели и вне ее представлена на рисунке 2A. Конвейер CIRCLE-seq также предоставлял «объединенные чтения», проанализированные с помощью статистического программного обеспечения R, чтобы получить Манхэттенский график, показывающий обнаруженные участки расщепления, индуцированные нуклеазами, картированные вдоль каждой хромосомы (рис. 2B).
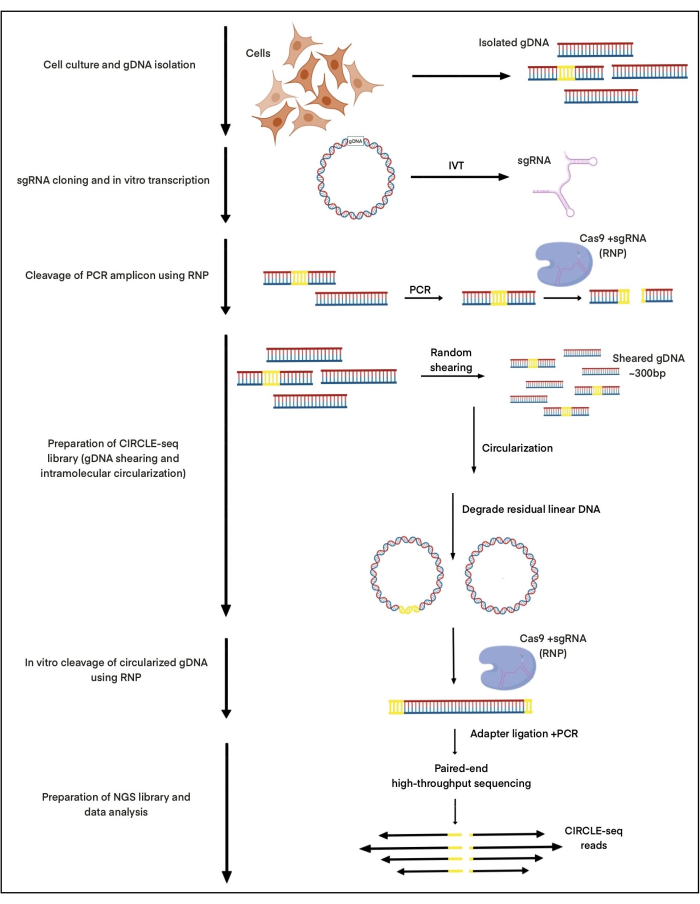
Рисунок 1: Схемы рабочего процесса CIRCLE-seq. Обозначены основные этапы работы протокола. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
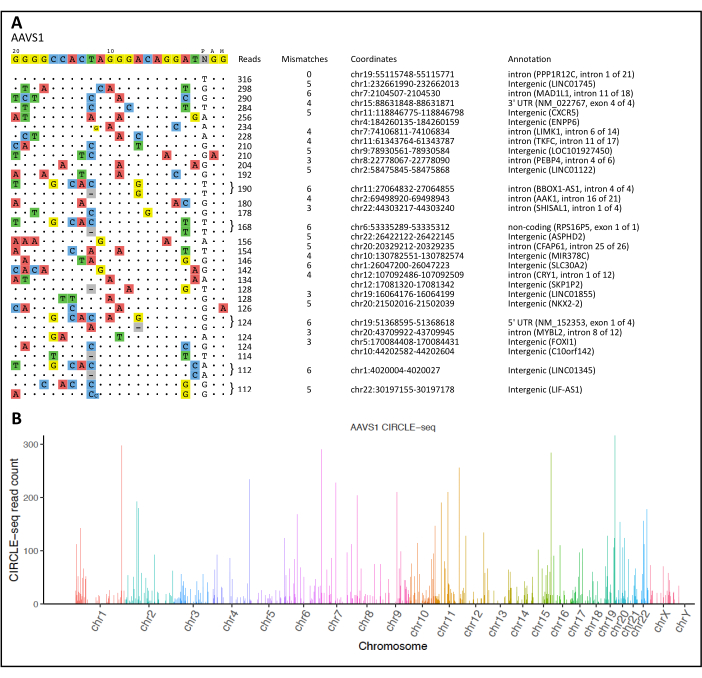
Рисунок 2: Визуализация CIRCLE-seq и Манхэттенский график . (A) Выравнивание нецелевых сайтов относительно намеченной цели для локуса AAVS1. Целевая последовательность отображается вверху, где нецелевые объекты ранжируются по количеству прочтений в порядке убывания. Различия в исходных последовательностях-мишенях показаны цветными нуклеотидами. Показана выборка верхних непреднамеренных сайтов расщепления для локуса AAVS1. (B) Манхэттенский график, иллюстрирующий обнаруженные непреднамеренные участки расщепления для локуса AAVS1. Высота столбцов представляет собой количество прочтений для каждой хромосомной позиции. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
| Букварь | Последовательность (5'-3') | Комментарии/Описание |
| AAVS1 Одиночная направляющая РНК (sgRNA) | ГГГГКККАКУАГГГАКАГГАУ | Для удара флуоресцентного белка локуса AAVS1 |
| AAVS1 Форвардный праймер | GCTCTGGGCGGAGGAATATG | Для теста на расщепление гРНК in vitro |
| AAVS1 Обратная грунтовка | ATTCCCAGGGGGTTAATG | Для теста на расщепление гРНК in vitro |
| oSQT1288 | /5Phos/CGGTGGACCGATC /ideoxyU/ATCGGTCCACCGaT | Адаптер для шпильки CIRCLE-seq |
| oSQT1274 | AATGATACGGCGACCACCGAG | ТруSeq F1 |
| oSQT1275 | CAAGCAGAAGAGGGCATACGAGAT | TruSeqF2 |
| oSQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | Щуп TruSeq |
| oSQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | Щуп TruSeq |
Таблица 1: Последовательности гРНК и праймеров, использованных для анализа CIRCLE-seq локуса AAVS1 .
Обсуждение
В данной работе показано, что CIRCLE-seq является непредвзятым и высокочувствительным методом идентификации индуцированных нуклеазами DSB по всему геному, возникающих в результате нацеливания на локус AAVS1 в гДНК, полученной из iPSCs. Сайт AAVS1 в ИПСК хорошо известен как локус «безопасной гавани», который часто используется в качестве сайта интеграции экзогенных генов с использованием CRISPR-Cas928. В нашем недавнем отчете изучался потенциал меченых EGFP иПСК путем выполнения CRISPR-опосредованной интеграции конститутивно экспрессированного репортера EGFP в сайт AAVS1 , что позволяет маркировать и отслеживать как ИПСК, так и дифференцированные ИПСК благодаря персистенции EGFP по всей клеточнойлинии27. Эта линия iPSC может быть использована in vivo для оценки распределения клеток, полученных из iPSC, в организме после трансплантации. Поскольку эта клеточная линия была модифицирована CRISPR и также используется для тестирования клинического применения ИПСК, крайне важно, чтобы потенциальные участки AAVS1 были известны и исследованы для обеспечения безопасности и эффективности, что делает ее идеальным местом для тестирования CIRCLE-seq.
Заметное различие между ранее опубликованным исследованием27 и этим исследованием заключалось в использовании модифицированной гРНК для нацеливания на локус AAVS1. ГРНК может быть сконструирована для повышения точности редактированиягенома 24. Последовательность направляющих является наиболее важным фактором, влияющим на целевую и нецелевую эффективность. Таким образом, выбранная гРНК была протестирована в сравнении с несколькими другими проводниками и показала превосходную точность. Кроме того, в статье о методах перепрограммирования фибробластов человека был подтвержден вывод о том, что синтетические кэпированные мРНК, содержащие модифицированные нуклеиновые основания, выигрывают от низкой активации противовирусных реакций29,30. В то время как низкая иммуногенность может быть нерелевантной в анализе in vitro, она приобретает решающее значение, когда конечной целью является разработка клинически значимой терапии, которая может быть использована в живых клетках.
CIRCLE-seq имеет много преимуществ по сравнению с аналогичными методами. Например, Digenome-seq секвенирует как нуклеазно-расщепленную, так и разрозненную гДНК, используя ~400 миллионов прочтений21. Это приводит к высокому фону, что затрудняет фильтрацию низкочастотных сайтов добросовестной резки. CIRCLE-seq использует только ~3-5 миллионов прочтений из-за обогащения гДНК, расщепленной нуклеазой, что приводит к низкому фону. Кроме того, Digenome-seq и аналогичный метод, SITE-seq, основаны на секвенировании одного конца ДНК, расщепленного нуклеазой. В отличие от этого, операции чтения CIRCLE-seq включают оба конца участка разреза, что позволяет идентифицировать участки, не являющиеся мишенью, без необходимости использования ссылки 21,22,26.
Преимуществом CIRCLE-seq является его большая чувствительность по сравнению с методами, основанными на клеточных культурах, такими как GUIDE-seq. Когда эти два метода были сравнены, CIRCLE-seq смог захватить все нецелевые сайты, обнаруженные GUIDE-seq, и обнаружил дополнительные непреднамеренные участки расщепления, которые GUIDE-seq пропустил. Тем не менее, заметное различие заключается в том, что GUIDE-seq может быть затруднен эпигенетическим ландшафтом, в то время как CIRCLE-seq может получить доступ ко всему геному.
В качестве анализа in vitro , CIRCLE-seq имеет несколько ограничений, первым из которых является обнаружение ложноположительных результатов. В то время как эпигенетика будет препятствовать активности нуклеазы в определенных участках in vivo, ультразвуковое исследование устраняет эти препятствия in vitro, позволяя нецелевой активности в местах, которые обычно могут быть недоступны в клеточном контексте. Кроме того, Cas9 присутствует в высоких концентрациях в этом анализе in vitro , что позволяет проводить расщепление, которое в противном случае было бы невозможно in vivo. Этот анализ также требует относительно большого количества исходной гДНК, что, в зависимости от имеющихся ресурсов, может свести на нет использование этого протокола. Наконец, некоторые нецелевые сайты могут быть необнаруживаемыми из-за ограничений современных технологий секвенирования нового поколения.
В недавнем исследовании использовался подход in silico , алгоритм которого определил ряд релевантных параметров для сравнения различных методов характеризации нуклеаз, включая CIRCLE-seq и GUIDE-seq31. Двумя важными параметрами были «обогащение сайта на вырезанном сайте» и «% ложных срабатываний». Интересно, что частота ложноположительных результатов CIRCLE-seq составила 88%, но его обогащение в месте разреза было в несколько раз выше, чем при использовании других методов in vitro . Сравнительный анализ каждого метода показал, что GUIDE-seq показал наилучшие результаты, поскольку он продемонстрировал наибольшую специфичность в отношении мишени при умеренном проценте ложноположительныхрезультатов 32. Это не опровергает CIRCLE-seq, а скорее намекает на возможность использования CIRCLE-seq и GUIDE-seq в тандеме, подтверждая результаты CIRCLE-seq с помощью GUIDE-seq, поскольку первый имеет более высокую чувствительность, в то время как второй является клеточным методом с высоким обогащением сайта разреза. Данные также указывают на то, что секвенирование нового поколения (NGS) на основе ампликона должно быть предпочтительным методом для идентификации подлинных нецелевых модификаций в потенциальных сайтах-кандидатах31. Эти данные предполагают потенциальную стратегию использования CIRCLE-seq, за которым следует GUIDE-seq, а затем NGS на основе ампликона для изучения побочных эффектов.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
выражая глубочайшую благодарность за финансовую поддержку, предоставленную Национальными институтами здравоохранения (R01AR078551 и T32AR007411), Ассоциацией по исследованию дистрофического буллезного эпидермолиза (DEBRA) Австрии, Фондом Гейтса и Фондом Гейтса Frontiers.
Материалы
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
Ссылки
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185 (2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856 (2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786 (2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058 (2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157 (2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474 (2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745 (2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены