Method Article
CIRCLE-Seq per l'interrogazione dell'editing genetico off-target
In questo articolo
Riepilogo
Un ostacolo significativo a tecnologie come CRISPR sono gli eventi fuori bersaglio che possono interrompere i geni vitali. CIRCLE-seq ("Circularization for In Vitro Reporting of Cleavage Effects by Sequencing") è una tecnica progettata per identificare i siti di scissione non intenzionali. Questo metodo mappa l'attività dell'intero genoma di CRISPR-Cas9 con alta sensibilità e senza bias.
Abstract
La circolarizzazione per la segnalazione in vitro degli effetti di clivaggio mediante sequenziamento (CIRCLE-seq) è una nuova tecnica sviluppata per l'identificazione imparziale di siti di clivaggio non intenzionali di CRISPR-Cas9 attraverso il sequenziamento mirato del DNA cliveggiato di CRISPR-Cas9. Il protocollo prevede la circolarizzazione del DNA genomico (gDNA), che viene successivamente trattato con la proteina Cas9 e un RNA guida (gRNA) di interesse. Dopo il trattamento, il DNA scisso viene purificato e preparato come libreria per il sequenziamento Illumina. Il processo di sequenziamento genera letture paired-end, offrendo dati completi su ciascun sito di scissione. CIRCLE-seq offre diversi vantaggi rispetto ad altri metodi in vitro , tra cui requisiti minimi di profondità di sequenziamento, basso background e alto arricchimento per il gDNA clivato Cas9. Questi vantaggi aumentano la sensibilità nell'identificare gli eventi di scissione intenzionali e non intenzionali. Questo studio fornisce una procedura completa e passo-passo per esaminare l'attività fuori bersaglio di CRISPR-Cas9 utilizzando CIRCLE-seq. Ad esempio, questo protocollo è convalidato mappando i siti di scissione non intenzionali dell'intero genoma di CRISPR-Cas9 durante la modifica del locus AAVS1 . L'intero processo CIRCLE-seq può essere completato in due settimane, lasciando tempo sufficiente per la crescita cellulare, la purificazione del DNA, la preparazione della libreria e il sequenziamento Illumina. L'inserimento dei dati di sequenziamento nella pipeline CIRCLE-seq facilita l'interpretazione e l'analisi semplificate dei siti di scissione.
Introduzione
L'ingegneria del genoma ha visto progressi significativi negli ultimi vent'anni, con una pietra miliare importante che è stata la scoperta di brevi ripetizioni palindromiche raggruppate regolarmente interspaziate (CRISPR)-Cas9 nel 20121. Sfruttando la natura programmabile delle endonucleasi del DNA batterico, la tecnologia CRISPR-Cas9 consente di indirizzare e modificare con precisione quasi tutte le sequenze di DNA. Fin dal suo inizio, il sistema è stato ottimizzato per fare affidamento solo sull'endonucleasi Cas9 e su un RNA guida (gRNA) per modificare specifiche regioni genomiche. Il potenziale di CRISPR-Cas9 come terapia curativa è stato dimostrato in studi clinici per varie condizioni come l'amaurosi congenita di Leber, l'amiloidosi da transtiretina e l'anemia falciforme, tra le altre 2,3,4.
CRISPR-Cas9 induce rotture a doppio filamento (DSB), che sono tipicamente risolte da uno dei due meccanismi: l'unione terminale non omologa soggetta a errori (NHEJ) o la più precisa riparazione diretta dall'omologia (HDR), a condizione che sia disponibile un DNA stampo. La tendenza di CRISPR-Cas9 a causare inserzioni e delezioni associate a NHEJ (indels), insieme alla scissione in siti genomici non intenzionali, ne limita l'applicazione in contesti clinici 5,6,7,8,9,10. Inoltre, le modifiche genomiche non intenzionali possono creare siti di splicing criptici, mutazioni nonsenso o missenso, indurre cromotripsi o conferire potenziale oncogenico alle cellule, esiti che sono stati osservati in diversi studi di editing genomico 11,12,13,14,15. In conclusione, identificare con precisione l'attività off-target di CRISPR-Cas9 è fondamentale per le sue applicazioni cliniche, in particolare nelle terapie geniche sistemiche che possono alterare miliardi di cellule.
Vari metodi possono essere impiegati per identificare i siti di scissione fuori bersaglio di CRISPR-Cas9, tra cui l'identificazione imparziale a livello di genoma delle rotture a doppio filamento (GUIDE)-seq16, che utilizza oligodesossinucleotidi a doppio filamento per marcare i DSB nelle cellule viventi. Tuttavia, una critica a questo metodo è che i falsi positivi possono derivare da DSB casuali o da artefatti PCR, che devono essere scartati escludendo i siti catturati che mostrano scarsa somiglianza con i siti target. Il metodo basato sull'uso del vettore lentivirale difettoso dell'integrasi (IDLV) è meno sensibile e probabilmente non rileva molti siti fuori bersaglio17. Altri metodi in situ come DSBCapture, BLESS e BLISS 18,19,20 coinvolgono cellule fisse e marcano direttamente i DSB, tuttavia sono limitati dalla loro dipendenza dalla cattura immediata dei DSB e dall'assenza di DNA esogeno. Il digenome-seq21, un metodo in vitro, e l'arricchimento selettivo e l'identificazione di estremità genomiche marcate mediante sequenziamento (SITE-seq)22 forniscono entrambi soluzioni di sequenziamento, ma hanno i loro limiti rispettivamente nel rumore di fondo e nell'analisi single-end. La scoperta di Cas off-target in situ e la verifica mediante sequenziamento (Discover-Seq)23 offre l'identificazione in vivo e in situ dell'attività di Cas9 tramite il legame MRE11, ma rileva solo i DSB esistenti al momento della preparazione del campione24. Infine, l'Inference of CRISPR Edits (ICE) utilizza un approccio bioinformatico per analizzare in modo robusto le modifiche CRISPR utilizzando i dati Sanger25.
Questo articolo descrive una procedura dettagliata per la circolarizzazione per la segnalazione in vitro degli effetti di scissione mediante sequenziamento (CIRCLE-seq): una tecnica in vitro che mappa in modo sensibile e imparziale l'attività off-target dell'intero genoma della nucleasi Cas9 in un complesso con il gRNA di interesse26. Questo approccio inizia con la coltura delle cellule di interesse e l'isolamento del DNA, seguito dal taglio casuale attraverso ultrasuoni focalizzati, quindi dal trattamento con esonucleasi e ligasi. Questo processo produce infine molecole di DNA circolari a doppio filamento, che vengono poi purificate attraverso il trattamento con DNasi sicuro per i plasmidi. Questo DNA circolare viene quindi esposto al complesso Cas9-gRNA, che si sfalda sia nei siti di scissione previsti che in quelli non intenzionali, lasciando dietro di sé le estremità del DNA esposte che fungono da substrati per la legatura dell'adattatore Illumina. Questo processo produce una libreria diversificata di DNA genomico (gDNA) contenente entrambe le estremità di ciascun DSB indotto dalla nucleasi, assicurando che ogni lettura abbia tutte le informazioni necessarie per ogni sito di scissione. Ciò consente l'uso del sequenziamento Illumina con requisiti di copertura del sequenziamento inferiori, distinguendo CIRCLE-seq da altri metodi simili sopra menzionati. È importante notare che, sebbene CIRCLE-seq abbia una sensibilità fuori bersaglio più elevata rispetto ad altri protocolli come metodo in vitro , ciò comporta un costo di falsi positivi più elevati a causa dell'assenza del panorama epigenetico presente in altri metodi come GUIDE-seq16. Inoltre, la riparazione del DNA DSB e i suoi macchinari associati non sono presenti in CIRCLE-seq, abrogando gli indel o la corretta riparazione che altrimenti si osserverebbero.
Oltre a descrivere il protocollo passo-passo per eseguire CIRCLE-seq, il protocollo è convalidato identificando ad esempio i siti di scissione non intenzionali di CRISPR-Cas9 a livello di genoma che si verificano durante la modifica del locus AAVS1 . Questo protocollo di facile consultazione fornisce istruzioni dettagliate, dalla coltura di cellule staminali pluripotenti indotte (iPSC) e l'isolamento del gDNA alla circolarizzazione del gDNA, alla scissione del gRNA Cas9-gRNA, alla preparazione della libreria, al sequenziamento e all'analisi della pipeline. Dati i bassi requisiti di copertura del sequenziamento, CIRCLE-seq è disponibile per qualsiasi laboratorio con accesso al sequenziamento di nuova generazione.
Protocollo
I dettagli dei reagenti, dei materiali di consumo e delle attrezzature utilizzate per questo studio sono elencati nella Tabella dei materiali.
1. Coltura cellulare (5 giorni)
- Includi un controllo negativo in tutto questo protocollo. Preparare un numero sufficiente di cellule per ulteriori 25 μg di gDNA (~2.0e7 cellule per campione).
- Colture iPSC secondo il protocollostabilito 27. Raccogliere le cellule e risospenderle in 10 mL di PBS. Pipettare un campione di cellule da 6 μl e risospendere in rapporto 1:1 con il blu di tripano. Utilizzare il contatore automatico di celle per contare il campione.
- Aliquotare 2 x 107 celle per provetta, quindi centrifugare a 300 x g per 3 minuti a 25 °C (temperatura ambiente (RT)). Questo è sufficiente per più repliche. Pipettare ed eliminare il surnatante.
2. Isolamento del DNA genomico (1 giorno)
- Utilizzare il kit di purificazione del DNA disponibile in commercio per isolare il gDNA, seguendo le istruzioni del produttore:
- Aggiungere 200 μl di PBS a una provetta conica da 15 mL contenente il pellet cellulare e ririspendere. Quindi, pipettare 3 mL di tampone di lisi cellulare e 15 μL di proteinasi K nella provetta. Capovolgere il tubo 25 volte per mescolare accuratamente. Posizionare la provetta in un agitatore a bagnomaria impostato a 55 °C e 150 giri/min per 3 ore, o durante la notte per una resa ottimale del DNA.
- Pipettare in 15 μl di RNasi A. Capovolgere 25 volte. Mettere a bagnomaria a 37 °C per 1 ora.
- Raffreddare il campione con ghiaccio per 5 minuti. Quindi, aggiungere 1 mL di soluzione di precipitazione proteica, agitare ad alta velocità per 20 s e centrifugare a 2000 x g per 10 minuti a RT. Le proteine dovrebbero formare un pellet visibile e compatto sul fondo della provetta. Se il pellet non è visibile, incubare il campione su ghiaccio per altri 5 minuti e centrifugare nuovamente.
- Aggiungere 3 mL di isopropanolo al 100% a una nuova provetta conica da 15 mL. Pipettare con cautela il surnatante del passaggio 2.1.3 nella provetta. Capovolgere la provetta 50 volte per mescolare, quindi centrifugare a 2000 x g e temperatura ambiente per 3 minuti. Senza disturbare il pellet di DNA, aspirare con cura il surnatante utilizzando una pipetta Pasteur collegata a una trappola a vuoto e capovolgere il tubo su un panno pulito e privo di lanugine.
- Pipettare 3 mL di etanolo al 70% (v/v) sul pellet di DNA e capovolgere 10 volte per lavare. Centrifugare a 2.000 x g per 3 minuti a RT. Quindi, versare con cura il surnatante.
- Tenere la provetta aperta e lasciare asciugare il pellet di DNA risultante per 30 minuti, assicurandosi che tutto l'etanolo sia completamente evaporato. Pipettare in 50 μl di soluzione di idratazione del DNA e miscelare accuratamente mediante pipettaggio delicato.
- Sciogliere il DNA ponendo il campione in un agitatore a bagnomaria a 65 °C per 1 ora, quindi lasciare il campione in RT per una notte. Centrifugare il campione a RT e 2.000 x g per 1 minuto e utilizzare il kit di saggio dsDNA BR con le provette associate per quantificare il DNA isolato con il fluorimetro.
3. Preparazione del gRNA (7 giorni)
- Ordina il gRNA sintetico di interesse da una fonte commerciale (vedi Tabella dei materiali). Questo protocollo è compatibile anche con crRNA/tracrRNA.
4. Test di scissione in vitro del gRNA
NOTA: In questo caso, viene utilizzato un bersaglio nel gene AAVS1 . Per indirizzare altri geni di interesse, progettare primer (Tabella 1) per amplificare la regione target e sostituire i primer nei passaggi successivi con primer personalizzati.
- Preparare la reazione PCR: Miscelare 25 μL di Phusion Hot Start Flex 2x Master Mix (Concentrazione finale 1x), 0,5 μL di AAVS1 F Primer (Concentrazione finale 0,1 μM), 0,5 μL di AAVS1 R Primer (Concentrazione finale 0,1 μM), 5 μL di gDNA (100 ng, 20 ng/μL, dal passaggio 2.1.6) e 19 μL di H2O privo di nucleasi (Volume totale: 50 μl).
- Utilizzare i seguenti parametri del termociclatore: Denaturazione: 98 °C per 2 min (1 ciclo), Denaturazione: 98 °C per 10 s (10 cicli), Ricottura: 72-62 °C (-1 °C/ciclo) per 15 s (10 cicli), Estensione: 72 °C per 30 s (10 cicli), Denaturazione, 98 °C per 10 s (30 cicli). Ricottura: 65 °C per 15 s (30 cicli), Estensione: 72 °C per 30 s (30 cicli), Estensione finale: 72 °C per 5 min (1 ciclo), Mantenimento: 4 °C a tempo indeterminato.
- Utilizzare le perle AMPure XP per purificare il prodotto della reazione PCR. Innanzitutto, pipettare in volumi 1,8x, o 90 μl, di perline XP nel prodotto PCR. Pipettare dieci volte per mescolare accuratamente. Lasciare incubare la miscela in RT per 5 minuti.
- Utilizzando una rastrelliera magnetica, separare le perle dalla soluzione posizionando la piastra di reazione PCR sul magnete per 3 minuti. Pipettare la soluzione chiarificata e gettarla. Aggiungere 200 μl di etanolo all'80% (v/v) alle perle, incubare per 30 s e quindi rimuovere l'etanolo. Ripetere questa fase di lavaggio due volte per garantire la completa rimozione dell'etanolo.
- Lasciare asciugare i campioni naturalmente per 3 minuti posizionando la piastra sul magnete. Rimuovere la piastra dal magnete e aggiungere 40 μl di tampone TE, pH 8,0. Miscelare pipettando su e giù dieci volte. Lasciare riposare il campione a RT per 2 minuti.
- Posizionare la piastra di reazione PCR sul magnete per un altro minuto. Dopo 1 minuto, trasferire il surnatante in una nuova piastra. Misurare la resa della PCR purificata utilizzando uno spettrofotometro e analizzarla su una TapeStation utilizzando una striscia di provette ottiche con un tappo per provette ottiche, insieme a ScreenTape D1000 ad alta sensibilità e reagenti D1000 ad alta sensibilità (ladder e tampone), seguendo le istruzioni del produttore. Conservare il campione preparato a -20 °C per un massimo di diversi mesi.
- Diluire la proteina nucleasi Cas9 a 1 μM come segue: Miscelare 2 μL di tampone Cas9 10x (concentrazione finale 1x), 1 μL di nucleasi Cas9, S. pyogenes (concentrazione finale 1 μM) e 17 μL di H2O privo di nucleasi in un volume totale di 20 μL.
- Eseguire una procedura priva di RNasi per prevenire la degradazione del gRNA. Diluire il gRNA (dal passaggio 3.1) a 3 μM in H2O fino a un volume totale di 10 μL.
NOTA: Utilizzare la seguente formula per stimare il peso molecolare del gRNA: Peso molecolare dell'ssRNA (g/mol) = (lunghezza dell'ssRNA (nt) x 321,47 g/mol) + 18,02 g/mol. Per riferimento, un gRNA lungo 104 nt a 3 μM è circa 100 ng/μL.
5. Tranciatura del DNA (3 ore)
- Preparare l'ME220 puntando prima il braccio di controllo. Quindi, riempire il serbatoio con H2O deionizzato purificato. Sul laptop della stazione di controllo, accedi a Water Works e fai clic su Riempi. Regolare la temperatura a 4,5 °C.
- Trasferire 25 μg di gDNA in una microprovetta (microtubo-130 AFA Fiber Screw-Cap). Quindi, riempire la provetta fino a un volume totale di 130 μl con 1x TE. Utilizzare le seguenti condizioni per tranciare il DNA a una lunghezza media di circa 300 bp: impostare Durata su 10 s; Potenza di picco a 70; Fattore di dazio % a 20; Cicli/raffica a 50; tutto ciò imposterà automaticamente la potenza media su 14.0.
6. Purificazione del DNA genomico tranciato (1 h)
- Dividi il DNA genomico tranciato in due porzioni da 65 μl ciascuna. Purificare utilizzando 1,8 volte il volume delle perle XP (117 μL), seguendo la procedura descritta nei passaggi 4.3-4.6. Trasferire il surnatante in una nuova piastra PCR e misurare la quantità utilizzando uno spettrofotometro.
- Eseguire 1 μL di gDNA tranciato eluito su una TapeStation, secondo le istruzioni del produttore, per garantire che il gDNA venga tranciato a un'ampia distribuzione di circa 300 bp. Se necessario, conservare il gDNA tranciato a -20 °C per un massimo di diversi mesi.
7. Preparazione della libreria CIRCLE-seq (3 giorni)
- Ricottura dell'adattatore a forcina
- Risospendere oSQT1288 (Tabella 1), l'adattatore a forcina, a una concentrazione finale di 100 μM in 1x TE.
- Eseguire la ricottura dell'adattatore come segue: miscelare 40 μL di oSQT1288 (Concentrazione finale 40 μM), 10 μL di 10x STE (Concentrazione finale 1x) e 50 μL di H2O privo di nucleasi per un volume totale di 100 μL.
- Utilizzare i seguenti parametri di ricottura: 95 °C per 5 minuti, -1 °C al minuto per 70 cicli, mantenere a 4 °C a tempo indeterminato.
- Eseguire Termina riparazione . Utilizzare il kit di preparazione della libreria HTP senza PCR e preparare la miscela master di riparazione finale.
- Miscelare 8 μL di H2O privo di nucleasi, 7 μL di tampone di riparazione terminale 10x (concentrazione finale 1x) e 5 μL di miscela enzimatica di riparazione finale (volume finale 20 μL di miscela master Total End-Repair).
- Pipettare 20 μl della miscela master per la riparazione finale nel campione di gDNA tranciato dai passaggi 4.3-4.6. Miscelare 20 μl di End-Repair Master Mix con 50 μl di gDNA tranciato per un volume finale di 70 μl.
- Mettere la miscela in un termociclatore a 20 °C per 30 minuti, quindi mantenere a 4 °C a tempo indeterminato.
- Aggiungere 1,7 volte i volumi, o 120 μL, di perle XP e seguire i passaggi di purificazione dai passaggi 4.3-4.6. Eluire con 42 μl di TE, pH 8,0. Assicurati che le perle rimangano in soluzione per il passaggio successivo.
- Eseguire l'A-tailing. Utilizzando il kit di preparazione della libreria HTP (senza PCR), preparare la miscela master A-tailing.
- Miscelare 5 μl di tampone A-tailing 10x (concentrazione finale 1x) e 3 μl di enzima A-tailing (volume finale totale di 8 μl di A-tailing Master Mix).
- Pipettare 8 μl di A-tailing Master Mix in ciascun campione di DNA contenente le microsfere del passaggio 7.2.4, mescolando 8 μl di A-tailing Master Mix con 42 μl di DNA contenente microsfere di end-repaired (volume finale totale di 50 μl). Mettere in termociclatore a 30 °C per 30 min. Mantenere a 4 °C a tempo indeterminato.
- Pipettare 1,8 volumi, o 90 μL, di soluzione PEG/NaCl SPRI (un componente del kit di preparazione della libreria HTP (senza PCR; 96 reazioni)) al DNA con coda A. Purificare il DNA della coda A secondo i passaggi 4.3-4.6. Eluire il DNA con coda A in 30 μL di TE, pH 8,0. Conservare le perline in soluzione per il passaggio successivo.
- Eseguire la legatura dell'adattatore. Utilizzando il kit di preparazione della libreria HTP (senza PCR), preparare la miscela master di legatura dell'adattatore.
- Miscelare 10 μl di tampone di legatura 5x (concentrazione finale 1x), 5 μl di DNA ligasi e 5 μl di adattatore a forcina ricotto (40 μM) dal passaggio 7.1.3. Garantire la concentrazione finale di 4 μM per una miscela master di legatura dell'adattatore totale di 20 μl.
- Pipettare 20 μl di Adapter Ligation Master Mix in ciascun campione di DNA eluito contenente le microsfere del passaggio 7.3.3 (volume finale totale di 50 μl per campione).
- Porre in termociclatore a 20 °C per 1 ora. Mantenere a 4 °C a tempo indeterminato.
- Trasferire 1x volumi, o 50 μL, di soluzione PEG/NaCl SPRI al DNA legato con adattatore e purificare secondo i passaggi 4.3-4.6. Eluire con 30 μl di TE, pH 8,0 e decantare i surnatanti in una nuova piastra PCR semi-bordata. Combinare e quantificare il DNA utilizzando il test dsDNA BR. Se necessario, conservare il DNA purificato legato con adattatore per un massimo di 1 mese a -20 °C.
- Preparare il Master Mix Lambda Esonucleasi/Esonucleasi I (E. coli) (funzioni nell'eliminazione del DNA a singolo o doppio filamento senza adattatori legati a entrambe le estremità).
- Prelevare 1 μg di DNA legato con adattatore dal passaggio 7.4.4, diluindolo a 40 μl. Miscelare 5 μL di 10 tampone di reazione Exonuclease I (Concentrazione finale 1x), 4 μL di Lambda Exonuclease (Concentrazione finale 0,4 U/μL) e 1 μL di Esonucleasi I (E. coli) (Concentrazione finale 0,4 U/μL) per un volume totale di Lambda Exonuclease/Exonuclease I Master Mix di 10 μL.
- Pipettare 10 μl della miscela master Lambda Exonucleasi/Exonuclease I in 40 μl (1 μg) di DNA legato con adattatore (volume totale di 50 μl). Porre in termociclatore a 37 °C per 1 ora, quindi a 75 °C per 10 minuti. Mantenere a 4 °C a tempo indeterminato.
- Pipettare 1,8 volte i volumi, o 90 μL, di perle XP nel DNA trattato con Lambda Exonucleasi/Esonucleasi I. Purificare secondo le istruzioni fornite nei passaggi 4.3-4.6. Eluire in 40 μL di TE, pH 8,0. Assicurarsi che le perle rimangano in soluzione per il passaggio enzimatico successivo.
- Trattare con l'enzima USER e la polinucleotide chinasi T4 (PNK). Preparare la USER/T4 PNK Master Mix (necessaria per rilasciare le sporgenze di 4 bp e per preparare le estremità del DNA pronte per la legatura necessarie per la successiva reazione di legatura).
- Miscelare 5 μl di tampone DNA ligasi T4 10x (concentrazione finale 1x), 3 μl di enzima USER (concentrazione finale 0,05 U/μL) e 2 μl di PNK T4 (concentrazione finale 0,4 U/μl), per un volume totale della miscela master USER/PNK di 10 μl.
- Pipettare 10 μl di USER Enzyme/T4 PNK Master Mix in 40 μl di campione di DNA trattato con lambda ed esonucleasi I contenente le perle del passaggio 7.5.3 per un volume totale di 50 μl. Porre in termociclatore a 37 °C per 1 ora. Mantenere a 4 °C a tempo indeterminato.
- Pipettare 1,8 volte i volumi, o 90 μl, della soluzione di PEG/NaCl SPRI nel DNA trattato con USER/T4 PNK e purificare secondo i passaggi 4.3-4.6. Eluire in 35 μl di TE, pH 8,0. Decantare il surnatante in una nuova piastra PCR semi-gonnata. Combinare e quantificare il DNA utilizzando il test dsDNA HS.
- Eseguire la circolarizzazione intramolecolare. Preparare il Master Mix di circolarizzazione.
- Miscelare 8 μL di H2O privo di nucleasi, 10 μL di tampone DNA ligasi T4 10x (concentrazione finale 1x) e 2 μl di DNA ligasi T4 (concentrazione finale 8 U/μL), per un volume totale della Master Mix di circolarizzazione di 20 μL.
- Pipettare 20 μl della Circularization Master Mix in 500 ng di DNA trattato con USER/PNK dal passaggio 7.6.3. Diluire 500 ng di DNA trattato con USER/PNK in 80 μL, quindi aggiungere 20 μL di Circularization Master Mix (volume totale di 100 μL). Incubare in termociclatore a 16 °C per 16 ore (tutta la notte).
- Aggiungere 1x volume, o 100 μL, di perle XP al DNA circolarizzato e purificare secondo i passaggi 4.3-4.6. Eluire in 38 μL di TE, pH 8,0. Decantare il surnatante in una nuova piastra PCR semi-gonnata.
- Trattare con DNasi ATP-dipendente sicura per i plasmidi. Preparare la miscela master di DNasi ATP-dipendente sicura per il plasmide (necessaria per la degradazione del DNA lineare residuo).
- Miscelare 5 μL di 10 μl di tampone di reazione sicuro per i plasmidi (concentrazione finale 1x), 2 μl di ATP (concentrazione finale 1 mM) e 5 μl di DNasi ATP-dipendente sicura per i plasmidi (concentrazione finale 1 U/μL), per un volume totale della miscela master sicura per i plasmidi di 12 μl.
- Pipettare 12 μl della DNase Master Mix ATP-dipendente in 38 μl di DNA circolarizzato dal passaggio 7.7.3 (volume totale di 50 μl). Incubare in termociclatore a 37 °C per 1 ora, quindi a 70 °C per 30 minuti. Mantenere a 4 °C a tempo indeterminato.
- Pipettare 1 volume, o 50 μL, di perle XP nel DNA trattato con DNasi ATP-dipendente sicura per il plasmide e purificare secondo i passaggi 4.3-4.6. Eluire in 15 μL di TE, pH 8,0. Decantare il surnatante in una nuova piastra PCR semi-gonnata.
- Combinare il DNA e quantificare utilizzando il test dsDNA HS. Se necessario, conservare il DNA circolarizzato a -20 °C per un massimo di diversi mesi.
8. Scissione di gDNA circolarizzato enzimaticamente purificato in vitro (2 ore)
- Eseguire la scissione in vitro con il complesso Cas9:gRNA. Preparare il Master Mix di Scissione in vitro . Miscelare 5 μl di tampone Cas9 10x (concentrazione finale 1x), 4,5 μl di S. pyogenes Cas9 (concentrazione finale 90 nM) e 1,5 μl di gRNA (concentrazione finale 90 nM) per un volume totale di Cleavage Master Mix di 11 μl.
- Mantenere la miscela master di scissione a RT per 10 minuti per formare complessi Cas9:gRNA RNP.
- Diluire 125 ng di DNA trattato con DNasi sicuro per il plasmide dal passaggio 7.8.3 fino a un volume finale di 39 μl. Quindi, aggiungere 11 μl di Cleavage Master Mix a 39 μl di DNA trattato con DNasi sicura per il plasmide per un volume totale di 50 μl.
NOTA: Includere un campione di controllo negativo in questa fase, che comprende DNA circolarizzato mescolato con tampone Cas9, senza il complesso Cas9:gRNA. - Incubare in termociclatore per 1 ora a 37 °C. Mantenere a 4 °C a tempo indeterminato. Aggiungere 50 μl (1x volume) di perle XP al DNA scisso in vitro e purificare il DNA seguendo i passaggi 4.3-4.6. Eluire in 42 μl di tampone TE, pH 8,0. Assicurarsi che le perle rimangano in soluzione per il passaggio successivo.
9. Preparazione della libreria di sequenziamento di nuova generazione (4 - 6 ore)
- Eseguire l'A-tailing. Preparare l'A-tailing Master Mix.
- Miscelare 5 μl di tampone A-tailing 10x (concentrazione finale 1x) e 3 μl di enzima A-tailing (il volume totale della miscela master A-tailing è 8 μl).
- Pipettare 8 μl di A-tailing Master Mix in 42 μl di campione di DNA eluito contenente le microsfere del passaggio 8.4 (volume totale di 50 μl). Mettere in termociclatore per 30 minuti a 30 °C. Mantenere a 4 °C a tempo indeterminato.
- Pipettare 1,8 volte i volumi della soluzione PEG/NaCl SPRI, o 90 μl, nel DNA con coda A e purificare il DNA secondo i passaggi 4.3-4.6. Eluire in 25 μL di TE, pH 8,0. Assicurati di conservare le perline in soluzione per il passaggio successivo.
- Eseguire la legatura dell'adattatore. Preparare l'adattatore Ligation Master Mix.
NOTA: Le aliquote monouso degli adattatori NEB devono essere preparate per evitare la formazione di dimeri dell'adattatore causati dall'idrolisi indotta dal gelo-disgelo del 3' T'.- Miscelare 10 μl di 5 tamponi di legatura (concentrazione finale 1x), 5 μl di DNA ligasi e 10 μl di adattatore per il sequenziamento (la concentrazione finale è 3 μM), per un totale di 25 μl.
- Pipettare 25 μl di Adapter Ligation Master Mix in 25 μl di campione di DNA con coda A contenente le microsfere del passaggio 9.1.3. Porre in termociclatore per 1 ora a 20 °C. Mantenere a 4 °C a tempo indeterminato.
- Pipettare 1x volumi, o 50 μL, di soluzione PEG/NaCl SPRI nel DNA legato con adattatore e purificare il DNA secondo i passaggi 4.3-4.6. Eluire in 47 μL di TE, pH 8,0. Assicurarsi che le perle rimangano in soluzione per il passaggio enzimatico successivo.
- Eseguire il trattamento con l'enzima USER (genera un singolo gap nucleotidico sui residui di uracile).
- Aggiungere 3 μL di enzima USER (incluso nel kit Dual Index Primers) al campione di DNA legato con adattatore contenente le perle del passaggio 9.2.3. Incubare a 37 °C per 15 min.
- Aggiungere 35 μL (0,7 volte il volume) di soluzione PEG/NaCl SPRI al DNA trattato con l'enzima USER e purificare secondo i passaggi 4.3-4.6. Eluire in 20 μl di tampone TE, pH 8,0. Trasferire il surnatante in una nuova piastra PCR semi-gonna e misurare la concentrazione di DNA utilizzando il test dsDNA HS. La concentrazione prevista dovrebbe essere di circa 2-5 ng/μL.
- (Facoltativo) Prima di procedere alla fase successiva, è possibile effettuare la selezione delle dimensioni del DNA utilizzando PippinHT. Utilizzare la cassetta PippinHT all'1,5% con una gamma di dimensioni compresa tra 250 e 850 bp. I campioni risultanti possono essere utilizzati direttamente nella PCR nella fase successiva.
- Eseguire la PCR per l'aggiunta di codici a barre
NOTA: Assicurarsi che le combinazioni di sequenze di primer scelte per ciascun campione siano univoche. Se possibile, ogni campione dovrebbe avere codici a barre i5 e i7 univoci.- Preparare la PCR Master Mix per aggiungere codici a barre con indici accoppiati. Miscelare 5 μL di H2O privo di nucleasi, 25 μL di 2x HotStart Ready Mix (concentrazione finale 1x), 5 μl di primer i5 (concentrazione finale 1 μM) e 5 μl di primer i7 (concentrazione finale 1 μM) (volume totale della master mix di 40 μl).
- Pipettare 40 μl della PCR Master Mix in 10 μl di DNA purificato trattato con l'enzima USER (circa 20 ng) dalla fase 9.3.2 (volume totale di 50 μl).
- Scegliere le seguenti condizioni di termociclo PCR: Denaturazione: 98 °C per 45 s per 1 ciclo, Denaturazione: 98 °C per 15 s per 20 cicli, Ricottura: 65 °C per 30 s per 20 cicli, Estensione: 72 °C per 30 s per 20 cicli, Estensione finale: 72 °C per 1 min per 1 ciclo, Mantenimento: 4 °C a tempo indeterminato.
- Aggiungere 0,7 volte i volumi, o 35 μl, di perle di XP al prodotto PCR e purificare secondo i passaggi 4.3-4.6. Eluire in 30 μL di TE, pH 8,0. Decantare il surnatante in una nuova piastra PCR semi-gonnata. Se necessario, conservare il DNA circolarizzato a -20°C per un massimo di diversi mesi.
NOTA: Eseguire un campione della PCR su Tapestation per controllare la qualità della libreria e valutare la formazione di dimeri adattatori. Se vengono rilevati dimeri dell'adattatore, ripetere il passaggio 9.5.4.
10. Quantificazione di librerie CIRCLE-seq mediante PCR digitale a goccia (dd_PCR) (6 ore)
NOTA: La quantificazione può essere eseguita anche utilizzando qPCR, Tapestation o un metodo simile.
- Iniziare con 5 μL di DNA dalla libreria (fase PCR 9.5.4) ben miscelati con 45 μL di TE privo di nucleasi, quindi effettuare diluizioni seriali 1:10 di ciascun campione in volumi da 50 μL, con diluizioni da 10-1 a 10-8 .
- Impostare la soluzione stock dd_PCR Master Mix. Miscela 11 μL di miscela 2x dd_PCR per sonde (Concentrazione finale 1x), 0,055 μL di sonda oSQT1310 (Concentrazione finale 250 nM), 0,055 μL di sonda oSQT1311 (Concentrazione finale 250 nM), 0,099 μL di Primer oSQT1274 (Concentrazione finale 450 nM), 0,099 μL di Primer oSQT1275 (Concentrazione finale 450 nM) e 6,292 μL di H2O privo di nucleasi, per un volume totale dd_PCR Master Mix di 17,6 μL. Preparare una miscela master per tutti i campioni per garantire che i volumi siano sufficienti per un pipettaggio accurato.
- Dosare le tre diluizioni più basse (10-6,10-7 e 10-8) in duplicato (in una piastra a 96 pozzetti). È necessario utilizzare un controllo non modello (NTC).
- Pipettare 17,6 μl di dd_PCR Master Mix per ciascun campione come segue: Miscelare 17,6 μl con 4,4 μl di campione (aggiungendo H2O privo di nucleasi al pozzetto con NTC), per un volume totale di 22 μl. Sigillare la piastra, quindi centrifugare a 2000 x g per 1 min a RT.
- Esegui la generazione di goccioline, il ciclo termico e l'analisi. Utilizzando un sistema PCR Droplet Reader, trasferire una cartuccia DG8 (8 pozzetti) nel supporto della cartuccia. Nella fila dell'olio della cartuccia, erogare 70 μL di olio per la generazione di goccioline per le sonde.
NOTA: Prelevare 20 μl del campione dal passaggio 10.1.2. e aggiungerlo alla fila di campioni della cartuccia a 8 pozzetti, coprendo la cartuccia con la guarnizione in gomma DG8, inserendola nel generatore di goccioline e quindi chiudendola per iniziare il processo (automatico). Al termine, rimuovere la cartuccia e spostare 40 μl dalla fila di goccioline della cartuccia a 8 pozzetti in una piastra PCR a 96 pozzetti semi-bordata, assicurandosi di pipettare lentamente.- Posizionare il blocco termico nella sigillatrice per piastre PCR PX1. Il sigillante inizierà a riscaldarsi fino a 180 °C quando viene acceso. Metti la termosaldatura in alluminio sulla piastra, assicurandoti che la linea rossa sia in alto. Posizionare la piastra nel PX1 e premere Seal.
- Scegliere le seguenti condizioni del termociclatore: Attivazione enzimatica: 95 °C per 10 minuti per 1 ciclo, Denaturazione: 94 °C per 30 s per 40 cicli, Ricottura/Estensione: 60 °C per 1 min per 40 cicli, Disattivazione enzimatica: 98 °C per 10 min per 1 ciclo, Mantenimento: 4 °C a tempo indeterminato.
- Sul lettore di droplet, aprire il software compatibile e selezionare i pozzetti da leggere. Selezionare ABS come tipo di esperimento e dd_PCR Supermix per le sonde. Scegliere Ch1 Sconosciuto per il target 1 e Ch2 Unknown per il target 2. Selezionare Applica, quindi OK. Inserire la piastra nel lettore di gocce. Per il set di coloranti, scegli FAM/HEX e fai clic su Esegui.
- Analizza i risultati dd_PCR. Gate la popolazione di goccioline doppie positive utilizzando il controllo negativo come riferimento. Calcola la media dei valori duplicati, quindi moltiplica per il fattore di diluizione e il fattore di diluizione di 5 volte del dd_PCR.
NOTA: Le copie totali per microlitro sono calcolate come segue: copie totali per microlitro = valore medio × fattore di diluizione di 5 ×, dove il "valore medio" rappresenta il valore medio di quantificazione da Ch1 e Ch2. - Combina tutti i campioni in un'unica libreria a concentrazioni equimolari. La libreria aggregata 1x dovrebbe contenere circa 4,5 x 109 molecole e avere un volume totale di 5 μL.
11. Sequenziamento di nuova generazione
- Inviare i campioni per il sequenziamento a un'agenzia esterna, assicurandosi che vengano annotate le sequenze di adattatori corrette.
12. Analisi dei dati CIRCLE-seq (1 - 3 ore)
- Installare Python versione 2.7, Burrows-Wheeler Aligner (BWA) e SAMtools. Scarica il genoma di riferimento (ad es. hg38) da http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz.
NOTA: Se il genoma della specie target non è disponibile, il flusso di lavoro computazionale CIRCLE-seq può essere eseguito in modalità indipendente dal riferimento. In questo caso, questo passaggio può essere saltato. - Scarica e installa la pipeline CIRCLE-seq con i seguenti comandi: (1) git clone https://github.com/tsailabSJ/circleseq.git, (2) cd circleseq, (3) pip install -r requirements.txt.
- Creare un file manifesto in formato YAML (.yaml). Di seguito è riportato un manifesto di esempio che può essere utilizzato con il set di dati di esempio fornito nel software CIRCLE-seq per testare il flusso di lavoro.
NOTA: (1) genoma di riferimento: dati/input/CIRCLEseq_test_genome.fa; (2) analysis_folder: dati/output; (3) BWA: BWA; (4) samtools: samtools; (5) read_threshold: ; (6) window_size: ; (7) mapq_threshold: ; (8) start_threshold: ; (9) gap_threshold: ; (10) mismatch_threshold: ; (11) merged_analysis: Vero; (12) campioni: U2OS_EMX1; (13) obiettivo: GAGTCCGAGCAGAAGAAGAANGG; (14) Read1: dati/input/EMX1.r1.fastq.gz; (15) Read2: dati/input/EMX1.r2.fastq.gz; (16) controlread1: dati/input/EMX1_control.r1.fastq.gz; (17) controlread2: dati/input/EMX1_control.r2.fastq.gz; (18) descrizione: U2OS. Sono stati utilizzati i seguenti valori manifesto: read_threshold: 4, window_size: 3, mapq_threshold: 50, start_threshold: 1, gap_threshold: 3, mismatch_threshold: 6 - Definire il file FASTA del genoma di riferimento, la directory di output per l'analisi e i percorsi dei comandi BWA e SAMtools . Definire le sequenze target e i percorsi dei file FASTQ demultiplexati sia per i campioni di clivaggio nucleasi che per quelli di controllo. È possibile elaborare più esperimenti contemporaneamente in modalità batch includendoli tutti in un unico file manifesto.
- Eseguire il seguente comando per le analisi standard basate su riferimenti: (1) python /percorso/di/circleseq.py all - manifest; (2) /percorso/di/manifest.yaml.
- In alternativa, eseguire il seguente comando per analisi standard non basate su riferimenti: (1) Python /percorso/a/circleseq.py senza riferimenti - manifesto; (2) /percorso/del/manifest.yaml.
- Quando si esegue la pipeline completa, trovare i risultati di output di ogni passaggio in un output_folder distinto designato per quel passaggio specifico.
Risultati
Qui, CIRCLE-seq viene utilizzato per studiare i siti di scissione indotti dalla nucleasi di Cas9 in un complesso con il gRNA progettato per colpire il sito di integrazione del virus adeno-associato 1 (AAVS1) utilizzando DNA isolato da cellule staminali pluripotenti indotte (iPSC). Questo gRNA è stato precedentemente descritto nella nostra pubblicazione27. Circa 25 μg di gDNA sono stati isolati da iPSC, tranciati mediante ultrasuoni focalizzati e la dimensione selezionata utilizzando la purificazione a microsfere AMPure XP per produrre frammenti di circa 300 bps. Da questi 25 μg di DNA, circa 2-5 ng di DNA sono stati circolarizzati con successo per la scissione in vitro di Cas9:gRNA. L'intera procedura è illustrata nella Figura 1.
A seguito di una procedura CIRCLE-seq e di un'analisi utilizzando il nostro flusso di lavoro di calcolo, nella Figura 2A viene presentata una visualizzazione di tutti i siti di scissione rilevati sul bersaglio e fuori bersaglio. La pipeline CIRCLE-seq ha anche fornito "letture unite", analizzate tramite il software statistico R per produrre un grafico di Manhattan che mostra i siti di scissione indotti dalla nucleasi rilevati mappati lungo ciascun cromosoma (Figura 2B).
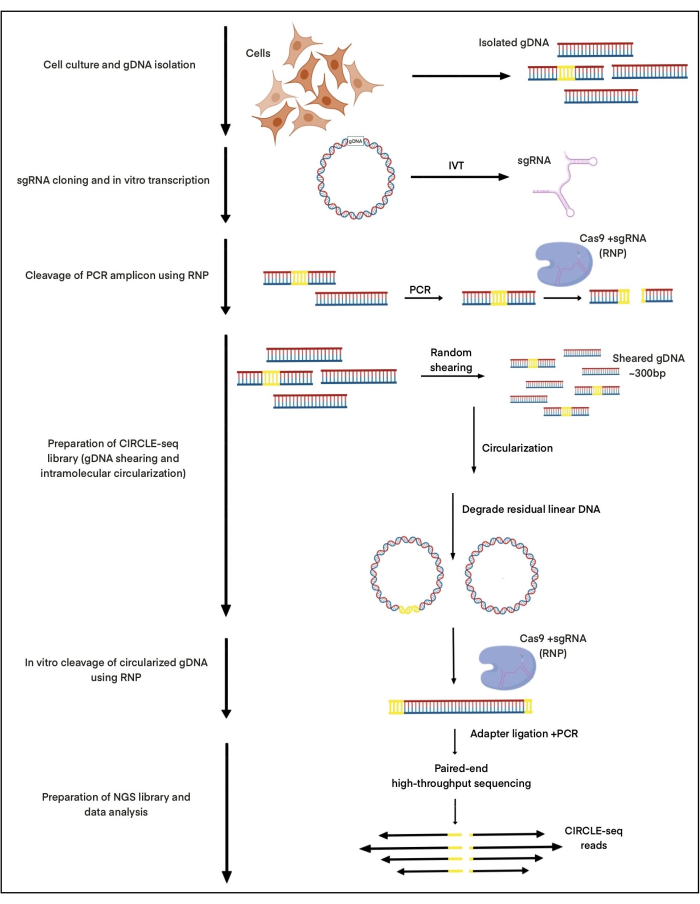
Figura 1: Schemi del flusso di lavoro CIRCLE-seq. Sono indicati i passaggi principali del protocollo. Clicca qui per visualizzare una versione più grande di questa figura.
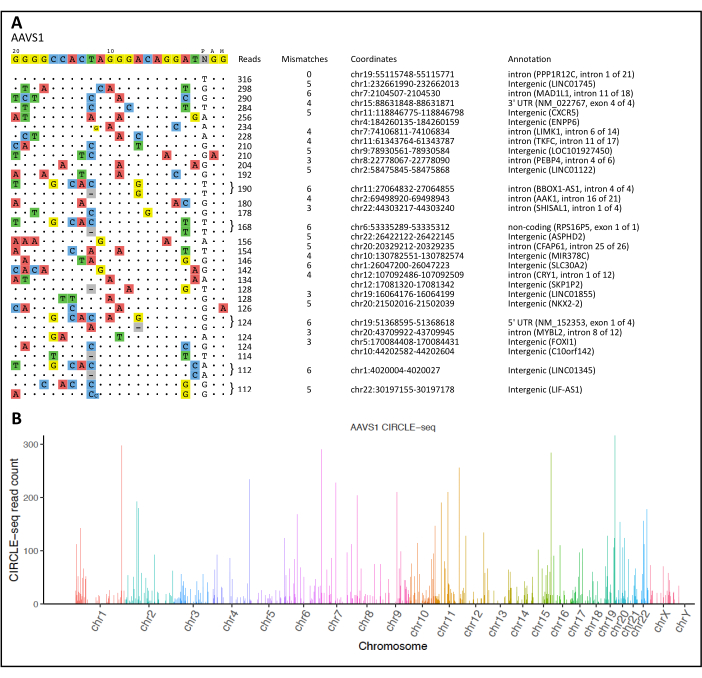
Figura 2: Visualizzazione CIRCLE-seq e Manhattan Plot. (A) Allineamento dei siti fuori bersaglio rispetto al bersaglio previsto per il locus AAVS1. La sequenza di destinazione viene visualizzata nella parte superiore, dove gli obiettivi non target vengono classificati in base al numero di letture in ordine decrescente. Le differenze nelle sequenze target originali sono mostrate da nucleotidi colorati. Viene mostrato un esempio dei principali siti di scissione non intenzionali per il locus AAVS1. (B) Grafico di Manhattan che illustra i siti di scissione non intenzionali rilevati per il locus AAVS1. Le altezze delle barre rappresentano il conteggio delle letture per ogni posizione cromosomica. Clicca qui per visualizzare una versione più grande di questa figura.
| Abbecedario | Sequenza (5'-3') | Commenti/Descrizione |
| AAVS1 Singolo RNA guida (sgRNA) | GGGGCCACUAGGGACAGGAU | per il knock-in della proteina fluorescente di AAVS1 Locus |
| AAVS1 Primer in avanti | GCTCTGGGCGGAGGAATATG | Per il test di scissione in vitro del gRNA |
| AAVS1 Primer inverso | ATTCCCAGGGCCGGTTAATG | Per il test di scissione in vitro del gRNA |
| oSQT1288 | /5Phos/CGGTGGACCGATGATC /ideoxyU/ATCGGTCCACCGaT | Adattatore per forcina CIRCLE-seq |
| oSQT1274 | AATGATACGGCGACCACCGAG | TruSeq F1 |
| oSQT1275 | CAAGCAGAAGACGGCATACGAGAT | TruSeqF2 |
| oSQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | Sonda TruSeq |
| oSQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | Sonda TruSeq |
Tabella 1: Sequenze di gRNA e primer utilizzati per l'analisi CIRCLE-seq del locus AAVS1 .
Discussione
In questo caso, CIRCLE-seq ha dimostrato di essere una tecnica imparziale e altamente sensibile per identificare le DSB indotte dalla nucleasi in tutto il genoma risultanti dal targeting del locus AAVS1 nel gDNA derivato dalle iPSC. Il sito AAVS1 all'interno delle iPSC è ben noto come locus di approdo sicuro che viene spesso utilizzato come sito di integrazione di geni esogeni utilizzando CRISPR-Cas928. Il nostro recente rapporto ha studiato il potenziale delle iPSC marcate con EGFP eseguendo l'integrazione mediata da CRISPR di un reporter EGFP costitutivamente espresso nel sito AAVS1 , che consente la marcatura e il tracciamento sia delle iPSC che delle iPSC differenziate a causa della persistenza di EGFP in tutto il lignaggio della cellula27. Questa linea di iPSC può essere utilizzata in vivo per valutare la distribuzione nell'organismo delle cellule derivate da iPSC dopo il trapianto. Poiché questa linea cellulare è stata modificata con CRISPR e viene utilizzata anche per testare l'applicazione clinica delle iPSC, è imperativo che i potenziali siti off-target di AAVS1 siano noti e interrogati per garantire la sicurezza e l'efficacia, rendendola un locus ideale per testare CIRCLE-seq.
Una notevole differenza tra lo studio27 precedentemente pubblicato da Butterfield et al. e questo è stato l'uso di un gRNA modificato per colpire il locus AAVS1. Un gRNA può essere progettato per migliorare l'accuratezza dell'editing del genoma24. La sequenza di guida è il fattore più critico che influenza l'efficienza sul bersaglio e fuori bersaglio. Pertanto, il gRNA scelto è stato testato con diverse altre guide e si è scoperto che aveva una fedeltà superiore. Inoltre, un articolo sui metodi sulla riprogrammazione dei fibroblasti umani ha confermato la scoperta che gli mRNA sintetici con cappuccio contenenti basi nucleotiche modificate beneficiano di una bassa attivazione delle risposte antivirali29,30. Sebbene una bassa immunogenicità possa non essere rilevante in un test in vitro, diventa cruciale quando l'obiettivo finale è sviluppare una terapia clinicamente rilevante che possa essere utilizzata nelle cellule viventi.
CIRCLE-seq offre molti vantaggi rispetto a metodi simili. Ad esempio, il digenoma-seq sequenzia sia il gDNA scisso che quello non scisso della nucleasi, utilizzando ~ 400 milioni di letture21. Ciò si traduce in uno sfondo elevato, che rende difficile filtrare i siti di taglio in buona fede a bassa frequenza. CIRCLE-seq utilizza solo ~3-5 milioni di letture a causa dell'arricchimento del gDNA tagliato con nucleasi, con conseguente basso background. Inoltre, il digenome-seq e un metodo simile, SITE-seq, si basano sul sequenziamento di una singola estremità del DNA scissa dalla nucleasi. Al contrario, le letture CIRCLE-seq includono entrambe le estremità del sito tagliato, consentendo l'identificazione di siti fuori bersaglio senza la necessità di un riferimento 21,22,26.
Un vantaggio di CIRCLE-seq è la sua maggiore sensibilità rispetto ai metodi che si basano sulla coltura cellulare, come GUIDE-seq. Quando i due metodi sono stati confrontati, CIRCLE-seq è stato in grado di catturare tutti i siti fuori bersaglio rilevati da GUIDE-seq e ha scoperto ulteriori siti di scissione non intenzionali che GUIDE-seq aveva perso. Tuttavia, una differenza notevole è che GUIDE-seq può essere ostacolato dal panorama epigenetico, mentre CIRCLE-seq può accedere all'intero genoma.
Come test in vitro , CIRCLE-seq presenta diverse limitazioni, la prima delle quali è il rilevamento di falsi positivi. Mentre l'epigenetica ostacolerà l'attività della nucleasi in determinati siti in vivo, l'ultrasonicazione rimuove questi ostacoli in vitro, consentendo attività fuori bersaglio in luoghi che potrebbero normalmente non essere accessibili in un contesto cellulare. Inoltre, Cas9 è presente ad alte concentrazioni in questo test in vitro , consentendo una scissione che non sarebbe altrimenti possibile in vivo. Questo test richiede anche una quantità relativamente grande di gDNA di partenza che, a seconda delle risorse disponibili, può negare l'uso di questo protocollo. Infine, è possibile che alcuni siti fuori bersaglio non siano rilevabili a causa delle limitazioni delle attuali tecnologie di sequenziamento di nuova generazione.
Un recente studio ha utilizzato un approccio in silico il cui algoritmo ha identificato una serie di parametri rilevanti con cui confrontare diversi metodi di caratterizzazione delle nucleasi, tra cui CIRCLE-seq e GUIDE-seq31. Due dei parametri pertinenti erano l'«arricchimento del sito di taglio» e la «% di falsi positivi». È interessante notare che il tasso di falsi positivi di CIRCLE-seq è stato calcolato all'88%, ma il suo arricchimento nel sito di taglio è stato di gran lunga superiore rispetto agli altri metodi in vitro . Le analisi comparative di ogni metodo hanno rivelato che GUIDE-seq era il migliore, in quanto dimostrava la massima specificità sul bersaglio con solo un moderato tasso di falsi positivi32. Ciò non invalida CIRCLE-seq, ma piuttosto suggerisce la possibilità di utilizzare CIRCLE-seq e GUIDE-seq in tandem, convalidando i risultati di CIRCLE-seq con GUIDE-seq poiché il primo ha una sensibilità più elevata, mentre il secondo è un metodo basato su cellule con un elevato arricchimento del sito di taglio. I dati indicano anche che il sequenziamento di nuova generazione (NGS) basato su ampliconi dovrebbe essere il metodo preferito per identificare autentiche modifiche fuori bersaglio in potenziali siti candidati31. Questi dati suggeriscono una potenziale strategia di utilizzo di CIRCLE-seq, seguito da GUIDE-seq e quindi NGS basato su ampliconi per esaminare gli effetti off-target.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Esprimo la più profonda gratitudine per il sostegno finanziario fornito dal National Institutes of Health (R01AR078551 e T32AR007411), dalla Dystrophic Epidermolysis Bullosa Research Association (DEBRA) Austria, dal Gates Grubstake Fund e dal Gates Frontiers Fund.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
Riferimenti
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185(2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856(2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786(2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058(2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157(2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474(2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745(2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon