Method Article
CIRCLE-SEQ para la interrogación de la edición de genes fuera del objetivo
En este artículo
Resumen
Un obstáculo importante para tecnologías como CRISPR son los eventos fuera del objetivo que pueden alterar genes vitales. La «circularización para la notificación in vitro de los efectos de la escisión mediante secuenciación» (CIRCLE-seq) es una técnica diseñada para identificar los sitios de escisión no intencionados. Este método mapea la actividad de CRISPR-Cas9 en todo el genoma con alta sensibilidad y sin sesgos.
Resumen
La circularización para la notificación in vitro de los efectos de la escisión por secuenciación (CIRCLE-seq) es una técnica novedosa desarrollada para la identificación imparcial de sitios de escisión no intencionados de CRISPR-Cas9 mediante la secuenciación dirigida del ADN escindido de CRISPR-Cas9. El protocolo consiste en circularizar el ADN genómico (ADNg), que posteriormente se trata con la proteína Cas9 y un ARN guía (ARNg) de interés. Después del tratamiento, el ADN escindido se purifica y se prepara como una biblioteca para la secuenciación de Illumina. El proceso de secuenciación genera lecturas de extremos emparejados, ofreciendo datos completos sobre cada sitio de escisión. CIRCLE-seq proporciona varias ventajas sobre otros métodos in vitro , incluidos los requisitos mínimos de profundidad de secuenciación, el bajo fondo y el alto enriquecimiento para el ADNg escindido en Cas9. Estas ventajas mejoran la sensibilidad en la identificación de eventos de escisión intencionados y no intencionados. Este estudio proporciona un procedimiento completo, paso a paso, para examinar la actividad fuera del objetivo de CRISPR-Cas9 utilizando CIRCLE-seq. A modo de ejemplo, este protocolo se valida mediante el mapeo de sitios de escisión no intencionada de CRISPR-Cas9 en todo el genoma durante la modificación del locus AAVS1 . Todo el proceso CIRCLE-seq se puede completar en dos semanas, lo que permite tiempo suficiente para el crecimiento celular, la purificación del ADN, la preparación de la biblioteca y la secuenciación de Illumina. La entrada de datos de secuenciación en la canalización CIRCLE-seq facilita la interpretación y el análisis simplificados de los sitios de escisión.
Introducción
La ingeniería genómica ha experimentado avances significativos en los últimos veinte años, con un hito importante siendo el descubrimiento de las repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR)-Cas9 en 20121. Aprovechando la naturaleza programable de las endonucleasas de ADN bacteriano, la tecnología CRISPR-Cas9 permite la selección y modificación precisas de casi cualquier secuencia de ADN. Desde sus inicios, el sistema se ha optimizado para depender solo de la endonucleasa Cas9 y un ARN guía (ARNg) para editar regiones genómicas específicas. El potencial de CRISPR-Cas9 como terapia curativa ha sido demostrado en ensayos clínicos para diversas afecciones como la amaurosis congénita de Leber, la amiloidosis por transtiretina y la anemia falciforme, entre otras 2,3,4.
CRISPR-Cas9 induce roturas de doble cadena (DSB), que normalmente se resuelven mediante uno de dos mecanismos: la unión de extremos no homólogos (NHEJ) propensa a errores o la reparación dirigida por homología (HDR) más precisa, siempre que se disponga de un ADN molde. La tendencia de CRISPR-Cas9 a causar inserciones y deleciones asociadas a NHEJ (indels), junto con escisiones en sitios genómicos no deseados, limita su aplicación en entornos clínicos 5,6,7,8,9,10. Además, las modificaciones genómicas no intencionadas pueden crear sitios de empalme crípticos, mutaciones sin sentido o sin sentido, inducir cromotripsis o conferir potencial oncogénico a las células, resultados que se han observado en varios ensayos de edición del genoma 11,12,13,14,15. En conclusión, la identificación precisa de la actividad fuera del objetivo de CRISPR-Cas9 es crucial para sus aplicaciones clínicas, particularmente en terapias génicas sistémicas que pueden alterar miles de millones de células.
Se pueden emplear varios métodos para identificar los sitios de escisión fuera del objetivo de CRISPR-Cas9, incluida la identificación insesgada de roturas de doble cadena en todo el genoma (GUIDE)-seq16, que utiliza oligodesoxinucleótidos de doble cadena para marcar DSB en células vivas. Sin embargo, una crítica a este método es que los falsos positivos pueden surgir de DSB aleatorios o de artefactos de PCR, que deben descartarse excluyendo los sitios capturados que muestran poca similitud con los sitios en el objetivo. El método basado en el uso del vector lentiviral defectuoso por integrasa (IDLV) es menos sensible y es probable que pase por alto muchos sitios fuera del objetivo17. Otros métodos in situ como DSBCapture, BLESS y BLISS 18,19,20 involucran células fijas y etiquetan DSB directamente, sin embargo, están limitados por su dependencia de la captura inmediata de DSB y la ausencia de ADN exógeno. Digenome-seq21, un método in vitro, y Selective enrichment and Identification of Tagged genomic DNA Ends by sequencing (SITE-seq)22 proporcionan soluciones de secuenciación, pero tienen sus limitaciones en el ruido de fondo y el análisis de un solo extremo, respectivamente. El descubrimiento in situ de Cas fuera de los objetivos y la verificación por secuenciación (Discover-Seq)23 ofrece la identificación in vivo e in situ de la actividad de Cas9 a través de la unión a MRE11, pero solo detecta los DSB que existen en el momento de la preparación de la muestra24. Por último, Inference of CRISPR Edits (ICE) utiliza un enfoque bioinformático para analizar de forma robusta las ediciones CRISPR utilizando datos de Sanger25.
Este artículo describe un procedimiento detallado para la circularización para el informe in vitro de los efectos de la escisión por secuenciación (CIRCLE-seq): una técnica in vitro que mapea de manera sensible e imparcial la actividad fuera del objetivo de todo el genoma de la nucleasa Cas9 en un complejo con el ARNg de interés26. Este enfoque comienza con el cultivo de las células de interés y el aislamiento del ADN, seguido de un cizallamiento aleatorio mediante ultrasonidos focalizados, y luego un tratamiento con exonucleasa y ligasa. En última instancia, este proceso produce moléculas circulares de ADN de doble cadena, que luego se purifican mediante un tratamiento de DNasa seguro para plásmidos. A continuación, este ADN circular se expone al complejo Cas9-gRNA, que se escinde tanto en los sitios de escisión intencionados como en los no intencionados, dejando atrás los extremos del ADN expuestos que actúan como sustratos para la ligadura del adaptador Illumina. Este proceso produce una biblioteca diversa de ADN genómico (ADNg) que contiene ambos extremos de cada DSB inducida por nucleasa, lo que garantiza que cada lectura tenga toda la información necesaria para cada sitio de escisión. Esto permite el uso de la secuenciación de Illumina con requisitos de cobertura de secuenciación más bajos, lo que diferencia a CIRCLE-seq de otros métodos similares mencionados anteriormente. Es importante tener en cuenta que, si bien CIRCLE-seq tiene una mayor sensibilidad fuera del objetivo que otros protocolos como método in vitro , esto se produce a costa de un mayor número de falsos positivos debido a la ausencia del panorama epigenético que está presente en otros métodos como GUIDE-seq16. Además, la reparación del ADN DSB y su maquinaria asociada no están presentes en CIRCLE-seq, lo que anula los indels o la reparación adecuada que de otro modo se observaría.
Además de describir el protocolo paso a paso para realizar CIRCLE-seq, el protocolo se valida identificando los sitios de escisión no intencionada de CRISPR-Cas9 en todo el genoma que se producen durante la modificación del locus AAVS1 , por ejemplo. Este protocolo fácil de seguir proporciona instrucciones detalladas, desde el cultivo de células madre pluripotentes inducidas (iPSC) y el aislamiento de ADNg hasta la circularización del ADNg, la escisión de Cas9-gRNA, la preparación de bibliotecas, la secuenciación y el análisis de tuberías. Dados los bajos requisitos de cobertura de secuenciación, CIRCLE-seq está disponible para cualquier laboratorio con acceso a la secuenciación de próxima generación.
Protocolo
Los detalles de los reactivos, consumibles y equipos utilizados para este estudio se enumeran en la Tabla de Materiales.
1. Cultivo celular (5 días)
- Incluya un control negativo en todo este protocolo. Prepare suficientes células para 25 μg adicionales de ADNg (~2.0e7 células por muestra).
- Cultivo de iPSCs según el protocolo establecido27. Recoja las células y vuelva a suspenderlas en 10 mL de PBS. Pipetear una muestra de células de 6 μL y volver a suspender en una proporción de 1:1 con azul de tripano. Utilice el contador de células automatizado para contar la muestra.
- Alícuota 2 x 107 celdas por tubo, luego girar a 300 x g durante 3 min a 25 °C (temperatura ambiente (RT)). Esto es suficiente para varias réplicas. Pipetear y desechar el sobrenadante.
2. Aislamiento de ADN genómico (1 día)
- Utilice el kit de purificación de ADN disponible en el mercado para aislar el ADNg, siguiendo las instrucciones del fabricante:
- Agregue 200 μL de PBS a un tubo cónico de 15 mL que contenga el pellet de celda y vuelva a suspender. A continuación, pipetee 3 mL de tampón de lisis celular y 15 μL de proteinasa K en el tubo. Invierta el tubo 25 veces para mezclar bien. Coloque el tubo en un agitador de baño de agua ajustado a 55 °C y 150 rpm durante 3 h, o toda la noche para obtener un rendimiento óptimo de ADN.
- Pipetear en 15 μL de RNasa A. Invertir 25 veces. Colocar al baño maría a 37 °C durante 1 h.
- Enfríe la muestra en hielo durante 5 min. A continuación, añadir 1 mL de solución de precipitación de proteínas, vórtice a alta velocidad durante 20 s y centrifugar a 2000 x g durante 10 min a RT. Las proteínas deben formar una pastilla visible y compacta en el fondo del tubo. Si el gránulo no es visible, incube la muestra en hielo durante 5 minutos más y vuelva a centrifugar.
- Agregue 3 mL de isopropanol al 100% a un nuevo tubo cónico de 15 mL. Pipetear con cuidado el sobrenadante del paso 2.1.3 en el tubo. Invierta el tubo 50 veces para mezclar, luego centrifugar a 2000 x g y temperatura ambiente durante 3 min. Sin alterar la bolita de ADN, aspire cuidadosamente el sobrenadante con una pipeta Pasteur conectada a una trampa de vacío e invierta el tubo en una toallita limpia y sin pelusa.
- Pipetear 3 mL de etanol al 70% (v/v) en la bolita de ADN e invertir 10 veces para lavar. Centrífuga a 2.000 x g durante 3 min a RT. Luego, vierta con cuidado el sobrenadante.
- Mantenga el tubo abierto y deje que la bolita de ADN resultante se seque durante 30 minutos, asegurándose de que todo el etanol se haya evaporado por completo. Pipetear 50 μL de solución de hidratación de ADN y mezclar bien mediante un pipeteo suave.
- Disuelva el ADN colocando la muestra en un agitador al baño de agua a 65 °C durante 1 h, luego deje la muestra en RT durante la noche. Centrifugar la muestra a RT y 2.000 x g durante 1 minuto y utilizar el kit de ensayo dsDNA BR con tubos asociados para cuantificar el ADN aislado con el fluorómetro.
3. Preparación del ARNg (7 días)
- Solicite el ARNg sintético de interés de una fuente comercial (consulte la Tabla de materiales). Este protocolo también es compatible con crRNA/tracrRNA.
4. Prueba de escisión in vitro de ARNg
NOTA: En este caso, se utiliza una diana del gen AAVS1 . Para dirigirse a otros genes de interés, diseñe cebadores (Tabla 1) para amplificar la región objetivo y reemplace los cebadores en los siguientes pasos con cebadores personalizados.
- Preparación de la reacción de PCR: Mezclar 25 μL de Phusion Hot Start Flex 2x Master Mix (concentración final 1x), 0,5 μL de cebador AAVS1 F (concentración final 0,1 μM), 0,5 μL de cebador AAVS1 R (concentración final 0,1 μM), 5 μL de ADNg (100 ng, 20 ng/μL, del paso 2.1.6) y 19 μL de H2O libre de nucleasas (volumen total: 50 μL).
- Utilice los siguientes parámetros del termociclador: Desnaturalización: 98 °C durante 2 min (1 ciclo), Desnaturalización: 98 °C durante 10 s (10 ciclos), Recocido: 72-62 °C (-1 °C/ciclo) durante 15 s (10 ciclos), Extensión: 72 °C durante 30 s (10 ciclos), Desnaturalización, 98 °C durante 10 s (30 ciclos). Recocido: 65 °C durante 15 s (30 ciclos), Extensión: 72 °C durante 30 s (30 ciclos), Extensión final: 72 °C durante 5 min (1 ciclo), Mantenimiento: 4 °C indefinidamente.
- Utilice las perlas AMPure XP para purificar el producto de la reacción PCR. En primer lugar, pipetee en volúmenes 1,8x, o 90 μL, de perlas XP en el producto de PCR. Pipetear diez veces para mezclar bien. Deje la mezcla en RT durante 5 min para incubar.
- Con una rejilla magnética, separe las perlas de la solución colocando la placa de reacción PCR en el imán durante 3 minutos. Pipetear la solución aclarada y desecharla. Agregue 200 μL de etanol al 80% (v/v) a las perlas, incube durante 30 s y luego retire el etanol. Repita este paso de lavado dos veces para asegurar la eliminación completa del etanol.
- Deje que las muestras se sequen naturalmente durante 3 minutos colocando la placa en el imán. Retire la placa del imán y añada 40 μL de tampón TE, pH 8,0. Mezclar pipeteando hacia arriba y hacia abajo diez veces. Deje que la muestra se asiente en RT durante 2 minutos.
- Coloque la placa de reacción PCR en el imán durante un minuto más. Después de 1 minuto, transfiera el sobrenadante a un plato nuevo. Mida el rendimiento de la PCR purificada con un espectrofotómetro y analícelo en una TapeStation utilizando una tira de tubo óptico con un tapón de tira de tubo óptico, junto con reactivos D1000 ScreenTape de alta sensibilidad y D1000 de alta sensibilidad (escalera y tampón), siguiendo las instrucciones del fabricante. Almacene la muestra preparada a -20 °C durante varios meses.
- Diluya la proteína nucleasa Cas9 a 1 μM de la siguiente manera: Mezcle 2 μL de tampón Cas9 10x (concentración final 1x), 1 μL de nucleasa Cas9, S. pyogenes (concentración final 1 μM) y 17 μL de H2O libre de nucleasa en un volumen total de 20 μL.
- Realice un procedimiento sin ARNasa para evitar la degradación del ARNg. Diluir el ARNg (a partir del paso 3.1) a 3 μM en H2O hasta un volumen total de 10 μL.
NOTA: Utilice la siguiente fórmula para estimar el peso molecular del ARNg: Peso molecular del ARNs (g/mol) = (longitud del ARNs (nt) x 321,47 g/mol) + 18,02 g/mol. Como referencia, un ARNg de 104 nt de largo a 3 μM es de aproximadamente 100 ng/μL.
5. Cizallamiento de ADN (3 h)
- Prepare el ME220 colocando primero el brazo de control. A continuación, llene el depósito con H2O desionizado purificado. En la computadora portátil de la estación de control, acceda a Water Works y haga clic en Llenar. Ajuste la temperatura a 4,5 °C.
- Transfiera 25 μg de ADNg a un microtubo (microtube-130 AFA Fiber Screw-Cap). A continuación, llene el tubo hasta un volumen total de 130 μL con 1x TE. Utilice las siguientes condiciones para cortar el ADN a una longitud media de aprox. 300 pb: establezca Duración en 10 s; Potencia máxima a 70; Factor de derecho % a 20; ciclos/ráfaga a 50; todo lo cual establecerá automáticamente la potencia promedio en 14.0.
6. Purificación del ADN genómico cortado (1 h)
- Divida el ADN genómico cortado en dos porciones de 65 μL cada una. Purifique utilizando 1,8 veces el volumen de las perlas XP (117 μL), siguiendo el procedimiento descrito en los pasos 4.3-4.6. Transfiera el sobrenadante a una nueva placa de PCR y mida la cantidad con un espectrofotómetro.
- Ejecute 1 μL del ADNg esquilado eluido en una TapeStation, de acuerdo con las instrucciones del fabricante, para garantizar que el ADNg se corte a una distribución amplia de aproximadamente 300 pb. Si es necesario, almacene el ADNg cortado a -20 °C durante varios meses.
7. Preparación de la biblioteca CIRCLE-seq (3 días)
- Recocido de adaptador de horquilla
- Vuelva a suspender oSQT1288 (Tabla 1), el adaptador de horquilla, hasta una concentración final de 100 μM en 1x TE.
- Realice el recocido del adaptador de la siguiente manera: mezcle 40 μL de oSQT1288 (concentración final de 40 μM), 10 μL de 10x STE (concentración final 1x) y 50 μL de H2O libre de nucleasas para un volumen total de 100 μL.
- Utilice los siguientes parámetros de recocido: 95 °C durante 5 min, -1 °C por min durante 70 ciclos, mantener a 4 °C indefinidamente.
- Realizar la reparación final. Emplee el kit de preparación de bibliotecas HTP sin PCR y prepare la mezcla maestra de reparación final.
- Mezcle 8 μL de H2O sin nucleasas, 7 μL de tampón de reparación final 10x (concentración final 1x) y 5 μL de mezcla de enzimas de reparación final (volumen final 20 μL de mezcla maestra de reparación final total).
- Pipetear 20 μL de la mezcla maestra de reparación final en la muestra de ADNg cortada de los pasos 4.3-4.6. Mezcle 20 μL de End-Repair Master Mix con 50 μL de ADNg cortado para obtener un volumen final de 70 μL.
- Coloque la mezcla en un termociclador a 20 °C durante 30 minutos, luego manténgala a 4 °C indefinidamente.
- Agregue 1,7 volúmenes, o 120 μL, de perlas XP y siga los pasos de purificación de los pasos 4.3-4.6. Eluir con 42 μL de TE, pH 8,0. Asegúrese de que las cuentas permanezcan en solución para el siguiente paso.
- Realizar A-tailing. Con el kit de preparación de bibliotecas HTP (sin PCR), prepare la mezcla maestra de cola A.
- Mezcle 5 μL de tampón de relaves A 10x (concentración final 1x) y 3 μL de enzima de relaves A (volumen final total de 8 μL de mezcla maestra de relaves A).
- Pipetear 8 μL de la mezcla maestra de cola A en cada muestra de ADN que contenga perlas del paso 7.2.4, mezclando 8 μL de mezcla maestra de cola A con 42 μL de perlas que contengan ADN reparado al final (volumen final total de 50 μL). Colocar en un termociclador a 30 °C durante 30 min. Mantener a 4 °C indefinidamente.
- Pipetee 1,8 volúmenes, o 90 μL, de solución SPRI de PEG/NaCl (un componente del kit de preparación de la biblioteca HTP (sin PCR; 96 reacciones)) al ADN de cola A. Purifique el ADN de la cola A de acuerdo con los pasos 4.3-4.6. Eluir el ADN de la cola A en 30 μL de TE, pH 8.0. Mantenga las cuentas en solución para el siguiente paso.
- Realizar la ligadura del adaptador. Con el kit de preparación de la biblioteca HTP (sin PCR), prepare la mezcla maestra de ligadura del adaptador.
- Mezcle 10 μL de tampón de ligadura 5x (concentración final 1x), 5 μL de ADN ligasa y 5 μL de adaptador de horquilla recocido (40 μM) del paso 7.1.3. Asegure la concentración final de 4 μM para una mezcla maestra de ligadura de adaptador total de 20 μL.
- Pipetear 20 μL de Adapter Ligation Master Mix en cada muestra de ADN eluida que contenga perlas del paso 7.3.3 (volumen final total de 50 μL por muestra).
- Colocar en un termociclador a 20 °C durante 1 h. Mantener a 4 °C indefinidamente.
- Transfiera 1x volúmenes, o 50 μL, de la solución SPRI de PEG/NaCl al ADN ligado al adaptador y purifique de acuerdo con los pasos 4.3-4.6. Eluir con 30 μL de TE, pH 8,0 y decantar los sobrenadantes en una nueva placa de PCR con semifaldón. Combine y cuantifique el ADN mediante el ensayo dsDNA BR. Si es necesario, almacene el ADN purificado ligado al adaptador durante un máximo de 1 mes a -20 °C.
- Prepare la mezcla maestra de exonucleasa lambda/exonucleasa I (E. coli) (funciones en la eliminación de ADN monocatenario o bicatenario sin adaptadores ligados a ambos extremos).
- Tome 1 μg de ADN ligado adaptador del paso 7.4.4, diluyéndolo a 40 μL. Mezcle 5 μL de tampón de reacción de exonucleasa I 10x (concentración final 1x), 4 μL de exonucleasa lambda (concentración final de 0,4 U/μL) y 1 μL de exonucleasa I (E. coli) (concentración final de 0,4 U/μL) para obtener un volumen total de la mezcla maestra de exonucleasa lambda/exonucleasa I de 10 μL.
- Pipetee 10 μL de la mezcla maestra de exonucleasa/exonucleasa I de Lambda a 40 μL (1 μg) de ADN ligado a un adaptador (volumen total de 50 μL). Coloque en un termociclador a 37 °C durante 1 h, luego a 75 °C durante 10 min. Mantener a 4 °C indefinidamente.
- Pipetee volúmenes 1,8x, o 90 μL, de perlas XP en el ADN tratado con exonucleasa/exonucleasa I. Purifique de acuerdo con las instrucciones dadas en los pasos 4.3-4.6. Eluido en 40 μL de TE, pH 8.0. Asegúrese de que las perlas permanezcan en solución para el siguiente paso enzimático.
- Tratar con la enzima USER y la polinucleótido quinasa T4 (PNK). Prepare la mezcla maestra PNK USER/T4 (necesaria para liberar los voladizos de 4 pb y para preparar los extremos de ADN listos para la ligadura necesarios para la reacción de ligadura posterior).
- Mezcle 5 μL de tampón de ADN ligasa T4 10x (concentración final 1x), 3 μL de enzima USER (concentración final 0,05 U/μL) y 2 μL de T4 PNK (concentración final 0,4 U/μL), para un volumen total de la mezcla maestra USER/PNK de 10 μL.
- Pipetear 10 μL de USER Enzyme/T4 PNK Master Mix en 40 μL de muestra de ADN tratada con lambda y exonucleasa I que contiene perlas del paso 7.5.3 para un volumen total de 50 μL. Colocar en un termociclador a 37 °C durante 1 h. Mantener a 4 °C indefinidamente.
- Pipetear 1,8 volúmenes, o 90 μL, de solución PEG/NaCl SPRI en el ADN tratado con USER/T4 PNK y purificar de acuerdo con los pasos 4.3-4.6. Eluido en 35 μL de TE, pH 8,0. Decantar el sobrenadante en una nueva placa PCR semifaldón. Combine y cuantifique el ADN utilizando el ensayo dsDNA HS.
- Realizar circularización intramolecular. Prepara la mezcla maestra de circularización.
- Mezcle 8 μL de H2O libre de nucleasas, 10 μL de tampón de ADN ligasa T4 10x (Concentración final 1x) y 2 μL de ADN ligasa T4 (Concentración final 8 U/μL), para obtener un volumen total de la mezcla maestra de circularización de 20 μL.
- Pipetear 20 μL de la mezcla maestra de circularización en 500 ng de ADN tratado con USER/PNK a partir del paso 7.6.3. Diluir 500 ng de ADN tratado con USER/PNK en 80 μL, luego agregar 20 μL de la Mezcla Maestra de Circularización (volumen total de 100 μL). Incubar en un termociclador a 16 °C durante 16 h (toda la noche).
- Agregue 1x volúmenes, o 100 μL, de perlas XP al ADN circularizado y purifique de acuerdo con los pasos 4.3-4.6. Eluido en 38 μL de TE, pH 8.0. Decantar el sobrenadante en una nueva placa PCR semifaldón.
- Trate con DNasa dependiente de ATP segura para plásmidos. Prepare la mezcla maestra de DNasa dependiente de ATP segura para plásmidos (necesaria para la degradación del ADN lineal residual).
- Mezcle 5 μL de tampón de reacción seguro para plásmidos 10x (concentración final 1x), 2 μL de ATP (concentración final 1 mM) y 5 μL de DNasa dependiente de ATP segura para plásmidos (concentración final 1 U/μL), para obtener un volumen total de la mezcla maestra segura para plásmidos de 12 μL.
- Pipetear 12 μL de la mezcla maestra de DNasa dependiente de ATP a 38 μL de ADN circularizado del paso 7.7.3 (volumen total de 50 μL). Incubar en un termociclador a 37 °C durante 1 h, luego a 70 °C durante 30 min. Mantener a 4 °C indefinidamente.
- Pipetear 1x volúmenes, o 50 μL, de perlas XP en el ADN tratado con DNasa dependiente de ATP segura para plásmidos y purificar de acuerdo con los pasos 4.3-4.6. Eluido en 15 μL de TE, pH 8,0. Decantar el sobrenadante en una nueva placa PCR semifaldón.
- Combine el ADN y cuantifique utilizando el ensayo dsDNA HS. Si es necesario, almacene el ADN circularizado a -20 °C durante varios meses.
8. Escisión enzimáticamente purificada, ADNg circularizada in vitro (2 h)
- Realizar escisión in vitro con el complejo Cas9:gRNA. Prepara la mezcla maestra de escisión in vitro . Mezcle 5 μL de tampón Cas9 10x (concentración final 1x), 4,5 μL de S. Pyogenes Cas9 (concentración final de 90 nM) y 1,5 μL de ARNg (concentración final de 90 nM) para obtener un volumen total de mezcla maestra de escisión de 11 μL.
- Mantenga la mezcla maestra de escisión en RT durante 10 minutos para formar complejos Cas9:gRNA RNP.
- Diluir 125 ng de ADN tratado con ADNasa seguro para plásmidos del paso 7.8.3 hasta un volumen final de 39 μL. A continuación, añada 11 μL de la mezcla maestra de escisión a 39 μL de ADN tratado con DNasa seguro para plásmidos hasta obtener un volumen total de 50 μL.
NOTA: Incluya una muestra de control negativo en este paso, que comprenda ADN circularizado mezclado con tampón Cas9, sin el complejo Cas9:gRNA. - Incubar en un termociclador durante 1 h a 37 °C. Mantener a 4 °C indefinidamente. Añada 50 μL (1x volumen) de perlas XP al ADN escindido in vitro y purifique el ADN siguiendo los pasos 4.3-4.6. Eluido en 42 μL de tampón TE, pH 8,0. Asegúrese de que las cuentas permanezcan en la solución para el siguiente paso.
9. Preparación de la biblioteca de secuenciación de próxima generación (4 - 6 h)
- Realizar A-tailing. Prepara la mezcla maestra de cola A.
- Mezcle 5 μL de tampón de relaves A 10x (concentración final 1x) y 3 μL de enzima de relaves A (el volumen total de la mezcla maestra de relaves A es de 8 μL).
- Pipetear 8 μL de la mezcla maestra de cola A hasta 42 μL de muestra de ADN eluida que contenga perlas del paso 8.4 (volumen total de 50 μL). Coloque en un termociclador durante 30 min a 30 °C. Mantener a 4 °C indefinidamente.
- Pipetear 1,8 veces volúmenes de solución PEG/NaCl SPRI, o 90 μL, en el ADN de la cola A y purificar el ADN de acuerdo con los pasos 4.3-4.6. Eluido en 25 μL de TE, pH 8.0. Asegúrese de mantener las cuentas en solución para el siguiente paso.
- Realizar la ligadura del adaptador. Prepare la mezcla maestra de ligadura del adaptador.
NOTA: Se deben preparar alícuotas de un solo uso de adaptadores NEB para evitar la formación de dímeros adaptadores causados por la hidrólisis inducida por congelación y descongelación de la 3' T'.- Mezcle 10 μL de tampón de ligadura 5x (concentración final 1x), 5 μL de ADN ligasa y 10 μL de adaptador para secuenciación (la concentración final es de 3 μM), para un total de 25 μL.
- Pipetear 25 μL de la mezcla maestra de ligadura adaptadora en 25 μL de muestra de ADN de cola A que contiene perlas del paso 9.1.3. Colocar en un termociclador durante 1 h a 20 °C. Mantener a 4 °C indefinidamente.
- Pipetear 1x volúmenes, o 50 μL, de solución SPRI de PEG/NaCl en el ADN ligado al adaptador y purificar el ADN de acuerdo con los pasos 4.3-4.6. Eluido en 47 μL de TE, pH 8.0. Asegúrese de que las perlas permanezcan en solución para el siguiente paso enzimático.
- Realizar el tratamiento con la enzima USER (genera un espacio de un solo nucleótido en los residuos de uracilo).
- Añada 3 μL de enzima USER (incluida en el kit de cebadores de doble índice) a la muestra de ADN ligada al adaptador que contiene perlas del paso 9.2.3. Incubar a 37 °C durante 15 min.
- Añada 35 μL (0,7x volumen) de solución SPRI de PEG/NaCl al ADN tratado con enzimas del USUARIO y purifique de acuerdo con los pasos 4.3-4.6. Eluido en 20 μL de tampón TE, pH 8,0. Transfiera el sobrenadante a una nueva placa de PCR con semifaldón y mida la concentración de ADN utilizando el ensayo dsDNA HS. La concentración esperada debe ser de aprox. 2-5 ng/μL.
- (Opcional) Antes de continuar con el siguiente paso, la selección del tamaño del ADN se puede llevar a cabo utilizando PippinHT. Utilice el casete PippinHT al 1,5% con un rango de tamaño de 250-850 pb. Las muestras resultantes se pueden utilizar directamente en la PCR en el siguiente paso.
- Realizar PCR para la adición de códigos de barras
NOTA: Asegúrese de que las combinaciones de secuencias de cebadores elegidas para cada muestra sean únicas. Si es posible, cada muestra debe tener códigos de barras i5 e i7 únicos.- Prepare la mezcla maestra de PCR para agregar códigos de barras de índice emparejado. Mezcle 5 μL de H2O sin nucleasas, 25 μL de 2x HotStart Ready Mix (concentración final 1x), 5 μL de cebador i5 (concentración final 1 μM) y 5 μL de cebador i7 (concentración final 1 μM) (volumen total de mezcla maestra de 40 μL).
- Pipetear 40 μL de la mezcla maestra de PCR por 10 μL de ADN purificado tratado con la enzima USER (aprox. 20 ng) del paso 9.3.2 (volumen total de 50 μL).
- Elija las siguientes condiciones de termociclado PCR: Desnaturalización: 98 °C durante 45 s durante 1 ciclo, Desnaturalización: 98 °C durante 15 s durante 20 ciclos, Recocido: 65 °C durante 30 s durante 20 ciclos, Extensión: 72 °C durante 30 s durante 20 ciclos, Extensión final: 72 °C durante 1 min durante 1 ciclo, Mantenimiento: 4 °C indefinidamente.
- Añada 0,7 volúmenes, o 35 μL, de perlas XP al producto de PCR y purifique de acuerdo con los pasos 4.3-4.6. Eluido en 30 μL de TE, pH 8.0. Decantar el sobrenadante en una nueva placa PCR semifaldón. Si es necesario, almacene el ADN circularizado a -20 °C durante varios meses.
NOTA: Ejecute una muestra de la PCR en Tapestation para controlar la calidad de la biblioteca y evaluar la formación de dímeros adaptadores. Si se detectan dímeros adaptadores, repita el paso 9.5.4.
10. Cuantificación de librerías CIRCLE-seq por PCR digital en gotas (dd_PCR) (6 h)
NOTA: La cuantificación también se puede realizar mediante qPCR, Tapestation o un método similar.
- Comience con 5 μL de ADN de la biblioteca (paso de PCR 9.5.4) bien mezclado con 45 μL de TE libre de nucleasas, luego haga diluciones seriadas 1:10 de cada muestra en volúmenes de 50 μL, que van desde 10-1 hasta 10-8 diluciones.
- Configure la solución madre dd_PCR Master Mix. Mezcla 11 μL de 2x dd_PCR mezcla para sondas (Concentración final 1x), 0,055 μL de Sonda oSQT1310 (Concentración final 250 nM), 0,055 μL de Sonda oSQT1311 (Concentración final 250 nM), 0,099 μL de Primer oSQT1274 (Concentración final 450 nM), 0,099 μL de Primer oSQT1275 (Concentración final 450 nM) y 6,292 μL de H2O, libre de nucleasasa, para un volumen total de mezcla maestra de dd_PCR de 17,6 μL. Prepare una mezcla maestra para todas las muestras para asegurarse de que los volúmenes sean suficientes para un pipeteo preciso.
- Ensaye las tres diluciones más bajas (10-6, 10-7 y 10-8) por duplicado (en una placa de 96 pocillos). Se debe utilizar un control sin plantilla (NTC).
- Pipetear 17,6 μL de dd_PCR Master Mix en cada muestra de la siguiente manera: Mezclar 17,6 μL con 4,4 μL de la muestra (añadiendo H2O sin nucleasa al pocillo con NTC), para un volumen total de 22 μL. Selle la placa, luego centrifugue a 2000 x g durante 1 min en RT.
- Realice la generación de gotas, el termociclado y el análisis. Con un sistema de PCR con lector de gotas, transfiera un cartucho DG8 (8 pocillos) al soporte del cartucho. En la fila de aceite del cartucho, dispense 70 μL de aceite de generación de gotas para sondas.
NOTA: Tome 20 μL de la muestra del paso 10.1.2. y agréguelo a la fila de muestra del cartucho de 8 pocillos, cubriendo el cartucho con la junta de goma DG8, colocándolo en el generador de gotas y luego cerrándolo para comenzar el proceso (automático). Cuando haya terminado, retire el cartucho y mueva 40 μL de la fila de gotas del cartucho de 8 pocillos a una placa de PCR de 96 pocillos con faldón semi-excavado, asegurándose de pipetear lentamente.- Coloque el bloque de calor en el sellador de placas PX1 PCR. El sellador comenzará a calentarse hasta 180 °C cuando se encienda. Coloque el sello térmico de papel de aluminio en la placa, asegurándose de que la línea roja esté en la parte superior. Coloque la placa en el PX1 y presione Seal.
- Elija las siguientes condiciones del termociclador: Activación de enzimas: 95 °C durante 10 min durante 1 ciclo, Desnaturalización: 94 °C durante 30 s durante 40 ciclos, Recocido/Extensión: 60 °C durante 1 min durante 40 ciclos, Desactivación de enzimas: 98 °C durante 10 min durante 1 ciclo, Mantener: 4 °C indefinidamente.
- En el lector de gotas, abra el software compatible y seleccione los pocillos que desea leer. Seleccione ABS como tipo de experimento y dd_PCR Supermix para las sondas. Elija Ch1 Desconocido para el objetivo 1 y Ch2 Desconocido para el Objetivo 2. Seleccione Aplicar y, a continuación, Aceptar. Coloque la placa en el lector de gotas. Para el conjunto de tintes, elija FAM/HEX y haga clic en Ejecutar.
- Analice los resultados dd_PCR. Cierre la población de gotas positivas dobles utilizando el control negativo como referencia. Calcule el promedio de los valores duplicados, luego multiplíquelo por el factor de dilución y el factor de dilución de 5 veces del dd_PCR.
NOTA: El total de copias por microlitro se calcula de la siguiente manera: total de copias por microlitro = valor medio × 5 × factor de dilución, donde el "valor medio" representa el valor medio de cuantificación de Ch1 y Ch2. - Combine todas las muestras en una sola biblioteca a concentraciones equimolares. La biblioteca agrupada 1x debe contener aproximadamente 4,5 x 109 moléculas y tener un volumen total de 5 μL.
11. Secuenciación de nueva generación
- Envíe las muestras para su secuenciación a una agencia externa, asegurándose de que se anoten las secuencias adaptadoras correctas.
12. Análisis de datos CIRCLE-seq (1 - 3 h)
- Instale Python versión 2.7, Burrows-Wheeler Aligner (BWA) y SAMtools. Descargue el genoma de referencia (por ejemplo, hg38) de http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz.
NOTA: Si el genoma de la especie objetivo no está disponible, el flujo de trabajo computacional CIRCLE-seq se puede ejecutar en un modo independiente de la referencia. En esta situación, se puede omitir este paso. - Descargue e instale la canalización CIRCLE-seq con los siguientes comandos: (1) git clone https://github.com/tsailabSJ/circleseq.git, (2) cd circleseq, (3) pip install -r requirements.txt.
- Cree un archivo de manifiesto en formato YAML (.yaml). A continuación se muestra un manifiesto de muestra que se puede utilizar con el conjunto de datos de ejemplo proporcionado en el software CIRCLE-seq para probar el flujo de trabajo.
NOTA: (1) genoma de referencia: data/input/CIRCLEseq_test_genome.fa; (2) analysis_folder: datos/salida; (3) BWA: BWA; (4) Samtools: Samtools; (5) read_threshold: ; (6) window_size: ; (7) mapq_threshold: ; (8) start_threshold: ; (9) gap_threshold: ; (10) mismatch_threshold: ; (11) merged_analysis: Cierto; (12) Muestras: U2OS_EMX1; (13) objetivo: GAGTCCGAGCAGAAGAAGAANGG; (14) Read1: datos/entrada/EMX1.r1.fastq.gz; (15) Read2: datos/entrada/EMX1.r2.fastq.gz; (16) ControlRead1: datos/entrada/EMX1_control.r1.fastq.gz; (17) ControlRead2: datos/entrada/EMX1_control.r2.fastq.gz; (18) descripción: U2OS. Se utilizaron los siguientes valores de manifiesto: read_threshold: 4, window_size: 3, mapq_threshold: 50, start_threshold: 1, gap_threshold: 3, mismatch_threshold: 6 - Defina el archivo FASTA del genoma de referencia, el directorio de salida para el análisis y las rutas de acceso a los comandos BWA y SAMtools . Defina las secuencias de destino y las rutas a los archivos FASTQ demultiplexados para las muestras escindidas con nucleasa y de control. Se pueden procesar varios experimentos simultáneamente en modo por lotes incluyéndolos todos en un único archivo de manifiesto.
- Ejecute el siguiente comando para análisis estándar basados en referencias: (1) python /ruta/a/circleseq.py todo - manifiesto; (2) /ruta/a/manifest.yaml.
- Alternativamente, ejecute el siguiente comando para análisis estándar no basados en referencias: (1) Python /path/to/circleseq.py reference-free - manifest; (2) /ruta/a/manifiesto.yaml.
- Al ejecutar la canalización completa, busque los resultados de salida de cada paso en un output_folder distinto designado para ese paso específico.
Resultados
Aquí, CIRCLE-seq se utiliza para investigar los sitios de escisión inducidos por nucleasas de Cas9 en un complejo con el ARNg diseñado para dirigirse al sitio de integración de virus adenoasociado 1 (AAVS1) utilizando ADN aislado de células madre pluripotentes inducidas (iPSC). Este ARNg fue descrito previamente en nuestra publicación27. Se aislaron aproximadamente 25 μg de ADNg de las iPSC, se cortaron mediante ultrasonidos focalizados y se seleccionó el tamaño mediante la purificación de perlas AMPure XP para obtener fragmentos de aproximadamente 300 bps. A partir de estos 25 μg de ADN, se circularizaron con éxito aprox. 2-5 ng de ADN para la escisión in vitro Cas9:gRNA. Todo el procedimiento se muestra en la Figura 1.
Siguiendo un procedimiento y análisis de CIRCLE-seq utilizando nuestro flujo de trabajo de cálculo, en la Figura 2A se presenta una visualización de todos los sitios de escisión detectados dentro y fuera del objetivo. La canalización CIRCLE-seq también proporcionó "lecturas combinadas", analizadas a través del software estadístico R para producir un gráfico de Manhattan que muestra los sitios de escisión inducidos por la nucleasa detectados mapeados a lo largo de cada cromosoma (Figura 2B).
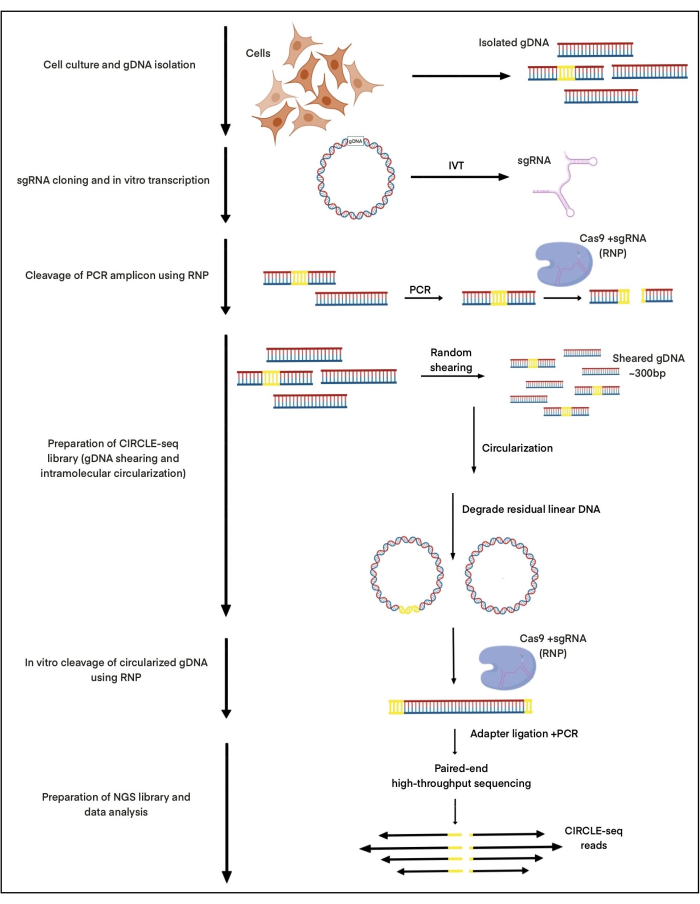
Figura 1: Esquemas del flujo de trabajo CIRCLE-seq. Se indican los pasos principales del protocolo. Haga clic aquí para ver una versión más grande de esta figura.
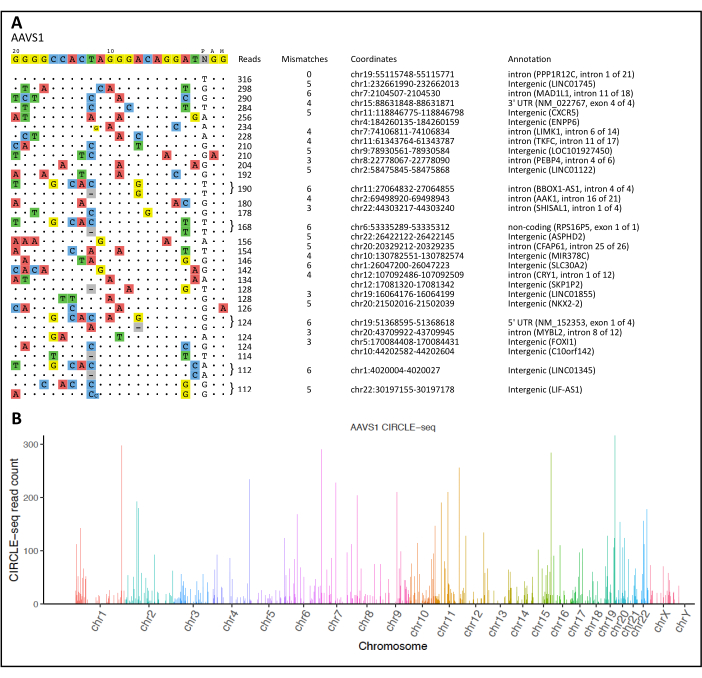
Figura 2: Visualización de CIRCLE-seq y Diagrama de Manhattan. (A) Alineación de sitios fuera del objetivo con el objetivo previsto para el locus AAVS1. La secuencia de objetivos se muestra en la parte superior, donde los objetivos fuera de lugar se clasifican por recuento de lecturas en orden descendente. Las diferencias en las secuencias diana originales se muestran mediante nucleótidos coloreados. Se muestra una muestra de los principales sitios de escisión no intencional para el locus AAVS1. (B) Gráfico de Manhattan que ilustra los sitios de escisión no intencional detectados para el locus AAVS1. Las alturas de las barras representan los recuentos de lecturas para cada posición cromosómica. Haga clic aquí para ver una versión más grande de esta figura.
| Cebador | Secuencia (5'-3') | Comentarios/Descripción |
| AAVS1 ARN guía simple (sgRNA) | GGGGCCACUAGGGACAGGAU | Para el knock-in de proteínas fluorescentes del locus AAVS1 |
| Imprimación AAVS1 Forward | GCTCTGGGCGGAGGAATATG | Para la prueba de escisión in vitro de ARNg |
| Imprimación inversa AAVS1 | ATTCCCAGGGCCGGTTAATG | Para la prueba de escisión in vitro de ARNg |
| oSQT1288 | /5Phos/CGGTGGACCGATGATC /ideoxyU/ATCGGTCCACCGaT | Adaptador de horquilla CIRCLE-seq |
| oSQT1274 | AATGATACGGCGACCACCGAG | TruSeq F1 |
| oSQT1275 | CAAGCAGAAGACGGCATACGAGAT | TruSeqF2 |
| oSQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | Sonda TruSeq |
| oSQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | Sonda TruSeq |
Tabla 1: Secuencias de ARNg y cebadores utilizados para el análisis CIRCLE-seq del locus AAVS1 .
Discusión
Aquí, se demuestra que CIRCLE-seq es una técnica imparcial y altamente sensible para identificar DSB inducidos por nucleasas en todo el genoma como resultado de dirigirse al locus AAVS1 en el ADNg derivado de iPSC. El sitio AAVS1 dentro de las iPSC es bien conocido como un locus de puerto seguro que a menudo se usa como un sitio de integración de genes exógenos usando CRISPR-Cas928. Nuestro reciente informe estudió el potencial de las iPSCs marcadas con EGFP mediante la integración mediada por CRISPR de un reportero de EGFP expresado constitutivamente en el sitio AAVS1 , lo que permite el etiquetado y el seguimiento de tanto iPSCs como iPSCs diferenciadas debido a la persistencia de EGFP en todo el linaje de la célula27. Esta línea de iPSC se puede utilizar in vivo para evaluar la distribución del organismo de las células derivadas de iPSC después del trasplante. Dado que esta línea celular ha sido modificada con CRISPR y también se está utilizando para probar la aplicación clínica de iPSC, es imperativo que se conozcan e interroguen los posibles sitios fuera del objetivo de AAVS1 para garantizar la seguridad y la eficacia, lo que la convierte en un lugar ideal para probar CIRCLE-seq.
Una diferencia notable entre el estudio27 de Butterfield et al., publicado anteriormente y este, fue el uso de un ARNg modificado para dirigirse al locus AAVS1. Un ARNg puede diseñarse para mejorar la precisión de la edición del genoma24. La secuencia guía es el factor más crítico que influye en la eficiencia dentro y fuera del objetivo. Por lo tanto, el ARNg elegido se probó con varias otras guías y se encontró que tenía una fidelidad superior. Además, un artículo sobre métodos sobre la reprogramación de fibroblastos humanos corroboró el hallazgo de que los ARNm sintéticos con tapa que contienen nucleobases modificadas se benefician de una baja activación de las respuestas antivirales29,30. Si bien la inmunogenicidad baja puede no ser relevante en un ensayo in vitro, se vuelve crucial cuando el objetivo final es desarrollar una terapia clínicamente relevante que pueda usarse en células vivas.
CIRCLE-seq tiene muchas ventajas sobre métodos similares. Por ejemplo, Digenome-seq secuencia tanto el ADNg escindido por nucleasa como el desatado, utilizando ~400 millones de lecturas21. Esto da como resultado un fondo alto, lo que dificulta el filtrado de sitios de corte de buena fe de baja frecuencia. CIRCLE-seq solo usa ~ 3-5 millones de lecturas debido al enriquecimiento del ADNg escindido por nucleasa, lo que resulta en un fondo bajo. Además, Digenome-seq y un método similar, SITE-seq, se basan en la secuenciación de un solo extremo de ADN escindido por nucleasa. Por el contrario, las lecturas CIRCLE-seq incluyen ambos extremos del sitio de corte, lo que permite la identificación de sitios fuera del objetivo sin la necesidad de una referencia 21,22,26.
Una ventaja de CIRCLE-seq es su mayor sensibilidad en comparación con los métodos que se basan en el cultivo celular, como GUIDE-seq. Cuando se compararon los dos métodos, CIRCLE-seq fue capaz de capturar todos los sitios fuera del objetivo detectados por GUIDE-seq y descubrió sitios de escisión no deseados adicionales que GUIDE-seq había pasado por alto. Sin embargo, una diferencia notable es que GUIDE-seq puede verse obstaculizado por el panorama epigenético, mientras que CIRCLE-seq puede acceder a todo el genoma.
Como ensayo in vitro , CIRCLE-seq presenta varias limitaciones, la primera de las cuales es la detección de falsos positivos. Mientras que la epigenética impedirá la actividad de la nucleasa en determinados sitios in vivo, la ultrasonicación elimina estos obstáculos in vitro, permitiendo la actividad fuera del objetivo en lugares que normalmente no serían accesibles en un contexto celular. Además, Cas9 está presente en altas concentraciones en este ensayo in vitro , lo que permite una escisión que de otro modo no sería posible in vivo. Este ensayo también requiere una cantidad relativamente grande de ADNg inicial, que, dependiendo de los recursos disponibles, puede negar el uso de este protocolo. Por último, es posible que algunos sitios fuera del objetivo sean indetectables debido a las limitaciones de las tecnologías actuales de secuenciación de próxima generación.
Un estudio reciente utilizó un enfoque in silico cuyo algoritmo identificó una serie de parámetros relevantes con los que comparar diferentes métodos de caracterización de nucleasas, incluidos CIRCLE-seq y GUIDE-seq31. Dos de los parámetros relevantes fueron el «enriquecimiento en el lugar de corte» y el «% de falsos positivos». Curiosamente, la tasa de falsos positivos de CIRCLE-seq se calculó en un 88%, pero su enriquecimiento en el sitio de corte fue mucho mayor que el de los otros métodos in vitro . Los análisis comparativos de cada método revelaron que GUIDE-seq fue el de mejor rendimiento, ya que demostró la mayor especificidad en el objetivo con una tasa de falsos positivos moderada32. Esto no invalida CIRCLE-seq, sino que insinúa la posibilidad de utilizar CIRCLE-seq y GUIDE-seq en tándem, validando los hallazgos de CIRCLE-seq con GUIDE-seq, ya que el primero tiene una mayor sensibilidad, mientras que el segundo es un método basado en células con un alto enriquecimiento del sitio de corte. Los datos también indican que la secuenciación de próxima generación (NGS) basada en amplicones debería ser el método preferido para identificar modificaciones genuinas fuera del objetivo en posibles sitios candidatos31. Estos datos sugieren una estrategia potencial de uso de CIRCLE-seq, seguido de GUIDE-seq, y luego NGS basado en amplicones para examinar los efectos fuera del objetivo.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Expresamos nuestro más profundo agradecimiento por el apoyo financiero proporcionado por los Institutos Nacionales de Salud (R01AR078551 y T32AR007411), la Asociación de Investigación de la Epidermólisis Bullosa Distrófica (DEBRA) de Austria, el Fondo Gates Grubstake y el Fondo Gates Frontiers.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
Referencias
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185 (2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856 (2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786 (2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058 (2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157 (2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474 (2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745 (2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados