A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تحديد وقياس المستقلبات المشوشة في المرضى المصابين بأمراض خطيرة باستخدام التمثيل الغذائي القائم على الرنين المغناطيسي النووي
In This Article
Summary
يستخدم التحليل الطيفي بالرنين المغناطيسي النووي (NMR) لتحديد خلل التنظيم في المستقلبات لدى المرضى الذين يعانون من أمراض مختلفة. تسمح هذه التقنية بالقياس الكمي للمستقلبات المشوشة ، وكشف الرؤى الفيزيولوجية المرضية. هنا ، نصف الإجراء خطوة بخطوة للنهج القائم على الرنين المغناطيسي النووي لتوصيف التمثيل الغذائي للمرضى.
Abstract
تبرز الأيض كنهج مهم يعكس استجابة الفرد للظروف الفيزيولوجية المرضية. تطور التحليل الطيفي بالرنين المغناطيسي النووي (NMR) كأداة لتحديد اختلالات التمثيل الغذائي في المرضى المصابين بأمراض خطيرة المصابين بحالات مثل متلازمة الضائقة التنفسية الحادة (ARDS) والتهاب البنكرياس الحاد الوخيم (SAP) وإصابة الكلى الحادة (AKI) والإنتان. يتم تسجيل البيانات الطيفية من عينة المصل لمجموعة الدراسة والمجموعة الضابطة باستخدام مطياف الرنين المغناطيسي النووي 800 ميجاهرتز ومعالجتها باستخدام أدوات معالجة وتحليل الرنين المغناطيسي النووي. علاوة على ذلك ، يتم إجراء تحليل إحصائي صارم ، مثل الاختبارات أحادية المتغير ومتعددة المتغيرات ، لتحديد مستقلبات مهمة ، والتي يتم تحديدها بعد ذلك بدقة وتحديدها كميا باستخدام برنامج القياس الكمي لمستقلبات الرنين المغناطيسي النووي. بالإضافة إلى ذلك ، يسلط تحليل المسار الضوء على الدورات الكيميائية الحيوية المشوشة التي تؤدي إلى شدة المرض. من خلال هذا النهج الشامل ، يهدف الباحثون إلى اكتساب رؤى أعمق حول التغيرات الأيضية المرتبطة بهذه الأمراض الخطيرة ، مما قد يمهد الطريق لفهم أفضل للمرض وتحسين استراتيجيات التشخيص والعلاج.
Introduction
على الرغم من الجهود المستمرة لتوفير تشخيص فعال للأمراض في جميع أنحاء العالم ، إلا أن العلاج الموجه لم يحقق بعد إمكاناته الحقيقية. أدت الأساليب المختلفة ، مثل النسخ ، والبروتينات ، وما إلى ذلك ، إلى تحديد العديد من المؤشرات الحيوية ، لكنها لم تحمل فائدة سريرية كافية بسبب نقص الحساسية والخصوصية1،2. يعد العلاج الموجه تحديا كبيرا في بعض الأمراض متعددة العوامل ، مما يؤدي في النهاية إلى ارتفاع معدل الوفيات. هناك حاجة إلى فهم أفضل للآلية الكامنة والفيزيولوجيا المرضية للأمراض المعقدة الموجودة مع عدم تجانس واسع. لذلك ، في هذا الصدد ، أحدث تطور التمثيل الغذائي ثورة في التطور العلاجي ، والذي قد يساعد في النهاية في تصميم أنظمة العلاج لمختلف الأمراض الخطيرة مثل متلازمة الضائقة التنفسية الحادة (ARDS) ، والإنتان ، والتهاب البنكرياس الحاد الوخيم (SAP).
التمثيل الغذائي هو نهج شامل يهدف إلى تحديد وقياس جزيئات الوزن الجزيئي الصغيرة (المستقلبات مثل الأحماض الأمينية والدهون والببتيدات والأحماض العضوية والفيتامينات) عبر مختلف السوائل الحيوية أو الخلايا أو مستخلصات الأنسجة. تلعب هذه المستقلبات ، التي تزن عادة أقل من 1500 دالتون ، أدوارا نشطة في العمليات الكيميائية الحيوية ، مما يعكس مخططا تدريجيا للحالة البيولوجية للكائن الحي. وهي تشمل ركائز للعمليات الأنزيمية الرئيسية ، والوسيطة في المسارات البيولوجية ، والمنتجات الثانوية لعملية التمثيل الغذائي الخلوي. وبالتالي ، تلتقط التمثيل الغذائي بصمة مفصلة للتأثيرات الغذائية والتفاعلات الدوائية وحالات المرض. تعد تغيرات المستقلب مؤشرات حساسة للغاية لعملية التمثيل الغذائي والمسارات البيولوجية ، مما يسمح بالارتباطات مع التعبيرات المظهرية والتشوهات الفيزيولوجية المرضيةالناتجة 3،4. يمكن أن تكون الاختلافات الأولية في المستقلبات بمثابة مؤشرات مبكرة لشدة المرض ، بينما قد تساعد التغييرات الزمنية في مراقبة فعالية العلاج وتطور المرض والنتائج السريرية5،6،7. وبالتالي ، تعزز الأيض المقايسات السريرية والعديد من مناهج omics الأخرى من خلال إعادة تعريف الأمراض من خلال نقاط النهاية السريرية والفسيولوجية والكيميائيةالحيوية 8،9،10،11،12،13. يتم استخدام القدرات التحليلية لعمليات التمثيل الغذائي لمراقبة وتحديد قابلية الإصابة بالمرض من خلال تركيزات مستقلبة متغيرة14،15.
في هذا السياق ، ظهر كل من قياس الطيف الكتلي (MS) والتحليل الطيفي بالرنين المغناطيسي النووي (NMR) كمنصات تحليلية أساسية لتنميط المستقلب في العينات البيولوجية. تستخدم هذه الطرق لتحديد وقياس المستقلبات المستهدفة وغير المستهدفة16،17،18. كل منصة لها مزاياها وقيودها ، لكن الطبيعة غير المدمرة للرنين المغناطيسي النووي تجعلها مفضلة في العديد من الدراسات في الجسم الحي ولتوصيف بنية المركبات غير المعروفة ، لا سيما في المراحل الأولى من أبحاث التمثيل الغذائي. تجزئة العينة والاشتقاق والتأين المطلوبة قبل أن يؤدي MS إلى حدوث تحيزات وغالبا ما يؤدي إلى فقدان العينة ، مما يؤثر على الميزات الديناميكية التي يمكن أن يلتقطها التحليل الطيفي بالرنين المغناطيسي النووي بأقل قدر ممكن من تحضير العينة أو بدونها. القيد الأساسي للرنين المغناطيسي النووي هو حساسيته المنخفضة مقارنة بمرض التصلب العصبي المتعدد ، والذي يوفر حدا أدنى للكشف ، مما يجعل من الصعب اكتشاف مستقلبات أقل وفرة19. ومع ذلك ، فإن التطورات مثل المغناطيس فائق التوصيل عالي الدقة ، ومجسات الرنين المغناطيسي النووي المبردة بالتبريد ، والتقنيات التي تعزز الحساسية قد خففت من هذا القيد20،21،22. كنهج تكميلي لعلم الجينوم والبروتينات ، يكتسب التنميط الأيضي باستخدام التحليل الطيفي بالرنين المغناطيسي النووي زخما كتقنية مفضلة23،24،25. إن الحد الأدنى من إعداد العينة في الرنين المغناطيسي النووي وقابلية التكرار والتكرار يجعله أداة قيمة لالتقاط السمات الديناميكية المتأصلة في المستقلبات على الرغم من تحديات الحساسية26.
أجرت العديد من المجموعات البحثية عمليات التمثيل الغذائي بنجاح ، وحددت الملف الأيضي غير المنظم للمرضى الذين يعانون من أمراض مختلفة27 مثل متلازمة الضائقة التنفسية الحادة28،29،30،31،32،33 ، والالتهاب الرئوي34 ، والإنتان7 ، وحصوات المرارة35 ، والتهاب البنكرياس36. كانت دراسات التمثيل الغذائي القائمة على الرنين المغناطيسي النووي للمرضى المصابين بأمراض خطيرة مفيدة في تتبع التقدم من متلازمة الاستجابة الالتهابية الجهازية (SIRS) إلى متلازمة الخلل الوظيفي للأعضاء المتعددة (MODS) ، والتي تعد سببا رئيسيا لوفيات ومراضة وحدة العناية المركزة (ICU) 37. في دراسة أجراها سترينجر وآخرون ، تم استخدام عينات البلازما لفحص التغيرات الأيضية في المرضى الذين يعانون من إصابة الرئة الحادة الناجمة عن تعفن الدم (ALI) مقارنة بمرضى التحكم38. عكست المستقلبات الرئيسية التي تم العثور عليها مرتفعة في هذه الدراسة التجريبية مسارات التمثيل الغذائي المعنية وارتباطها بالدرجات السريرية. تم توسيع هذا البحث ليشمل التمثيل الغذائي في المصل للتمييز بين الإنتان والمراحل المبكرة من آليات إصابة الرئة في متلازمة الضائقة التنفسيةالحادة 32. بالإضافة إلى ذلك ، حددت دراسة أخرى في متلازمة الضائقة التنفسية الحادة مؤشرات حيوية قوية للمصل تميز بوضوح بين إصابة الرئة الحادة / متلازمة الضائقة التنفسية الحادة والضوابط الصحية ، مما يقدم رؤى حول التغيرات الأيضية الجهازية المقابلة للبداية الحادة لإصابة الرئة39،40.
التحليل الطيفي بالرنين المغناطيسي النووي هو تقنية تحليلية عالية الإنتاجية وآلية توفر معلومات قوية وغير متحيزة حول بصمة التمثيل الغذائي التي تكشف عن الفيزيولوجيا المرضية الأساسية38. يتوقف التطبيق السريري والتفسير البيولوجي لبيانات الرنين المغناطيسي النووي على الحصول على أطياف عالية الجودة تحتوي على معلومات غنية. لذلك ، يعد ضمان جمع البيانات ومعالجتها وتحليلها بدقة ووحدة وصياغة جيدة أمرا بالغ الأهمية. لذلك ، فإن الهدف من هذه الدراسة هو الاستفادة من الخطوات الأساسية لعمليات التمثيل الغذائي القائمة على الرنين المغناطيسي النووي لتحديد وقياس المستقلبات. تسلط هذه الدراسة الضوء على الخطوات الرئيسية للبروتوكول المطلوب لدراسة التمثيل الغذائي السريري (الشكل 1) ، مثل اختيار العينات المناسبة ، وجمعها وتخزينها ، ومعالجة العينات وإعدادها ، والحصول على البيانات وتحليلها ، وتحديد وقياس المستقلب محل الاهتمام ، وفي النهاية ، تفسير النتائج في سياق سريري لاشتقاق الرؤى ذات الصلة. كل خطوة من هذه الخطوات ضرورية للاستفادة من التحليل الطيفي بالرنين المغناطيسي النووي في التمثيل الغذائي للكشف عن رؤى بيولوجية وسريرية مهمة.
Protocol
تم الحصول على الموافقة الأخلاقية (رمز IEC: 2022-71-PhD-126) من IEC في معهد سانجاي غاندي للدراسات العليا للعلوم الطبية (SGPGIMS) ، لكناو. تم أخذ موافقات خطية ومستنيرة من المرضى أو أقاربهم لإجراء الدراسة ونشر البيانات لغرض البحث. علاوة على ذلك ، تم إجراء البحث وفقا للإرشادات المؤسسية.
1. تصميم الدراسة والتصريح الأخلاقي
- حدد حجم العينة لكل مجموعة. مراجعة ووضع معايير الاختيار للمشاركين بعناية. بالإضافة إلى ذلك ، إذا كان تصميم الدراسة يتطلب إدراج عينات بشرية أو حيوانية ، فاحصل على تصريح أخلاقي من اللجنة الأخلاقية المؤسسية المناسبة (IEC) قبل البدء في جمع العينات.
- حدد مرضى متلازمة الضائقة التنفسية الحادة المصنفين على أنهم خفيفون ومتوسطون وشديدون ، بناء على معايير التشخيص لتعريف برلين 2012 بناء على الضغط الجزئي للأكسجين في الدم الشرياني (PaO2) إلى نسبة الأكسجين المستوحم (FiO2) (P / F). يعتبر مرضى متلازمة الضائقة التنفسية الحادة الذين يعانون من P / F من 300 إلى 200 معتدلين ، و 200-100 معتدلين ، وأقل من 100 على أنهمشديدون 38. هنا ، ركزنا على مجموعتين: مرضى متلازمة الضائقة التنفسية الحادة الخفيف والمتوسط (انظر الجدول 1).
ملاحظة: تتضمن الدراسة استخدام المصل الذي تم الحصول عليه من عينات الدم لإجراء التمثيل الغذائي القائم على الرنين المغناطيسي النووي. في هذه الدراسة ، كان متوسط عمر المشاركين المدرجين كمرجع 42 عاما (± 10.9 سنوات) ، مع توزيع بين الجنسين من الذكور و 6 إناث.
| باب | نسبة P / F |
| متوسط | 300-200 |
| المعتدل | 200-100 |
| شديد | 100-0 |
الجدول 1: تصنيف مرضى متلازمة الضائقة التنفسية الحادة.
2. اختيار العينة وجمعها ومعالجتها
- قم بعزل المصل باتباع الخطوات الموضحة أدناه.
- للتجربة ، اجمع 2 مل من عينات الدم من الشرايين باستخدام إبرة معقمة في قنينة عادية (بدون إضافات) وقارورة معقمة. اترك العينة لتتخثر لمدة 30 دقيقة في درجة حرارة الغرفة. لهذه الدراسة ، تم اختيار 4 مرضى تم تشخيص إصابتهم بمتلازمة الضائقة التنفسية الحادة الخفيفة ، و 4 مرضى يعانون من متلازمة الضائقة التنفسية الحادة المعتدلة.
- قم بالطرد المركزي للعينة عند 3100 × جم لمدة 15 دقيقة لفصل المصل25. انقل المصل إلى أنابيب طرد مركزي دقيقة معقمة ، مما يجعل حصصا ذات حجم أصغر (أي 400-500 ميكرولتر) ، وقم بتسميته بشكل مناسب (اسم المريض ، رقم CR ، نقطة اليوم ، إلخ) ، وتخزينها عند -80 درجة مئوية للتحليل المستقبلي.
ملاحظة: في هذه المرحلة ، يمكن إيقاف التجربة مؤقتا واستئنافها في وقت مناسب.
- قم بمعالجة عينة المصل قبل تجارب الرنين المغناطيسي النووي باستخدام الخطوات التالية.
- قم بإذابة العينة قبل إجراء تجارب الرنين المغناطيسي النووي. امزج 250 ميكرولتر من كمية المصل مع 250 ميكرولتر من محلول عازلة الفوسفات الملحي (يحتوي على 100٪ D2O ، 0.9٪ كلوريد الصوديوم ، 50 ملي مولار عازلة فوسفات الصوديوم ، درجة الحموضة 7.4) لتقليل تباين الأس الهيدروجيني.
- دوامة الخليط لبضع ثوان (~ 15 ثانية) للتأكد من خلطه بشكل متجانس. أخيرا ، انقل العينة المحضرة إلى أنبوب الرنين المغناطيسي النووي النظيف مع إدراج متحد المحور يحتوي على بروبيونات ثلاثي ميثيل سيليل (TSP) بتركيز 0.05 ملي مولار ، والذي يعمل كمعيار خارجي للمعايرة والقياسالكمي 41.
ملاحظة: كمرجع ، يتم استخدام مركب خامل وغير تفاعلي لا يتفاعل مع التحليلات. تشمل المركبات المرجعية الأخرى شائعة الاستخدام Tetramethylsilane (TMS) وثلاثي ميثيل سيليل بروبانسلفونات الصوديوم (DSS).
3. تجارب الرنين المغناطيسي النووي
ملاحظة: ينصب التركيز على تحديد الجزيئات الصغيرة في عينات المصل ، لذلك استخدمنا تسلسل نبضات Carr-Purcell-Meiboom-Gill (CPMG) ، والذي يثبط إشارات الجزيئات الكبيرة. تم تسجيل جميع عينات المصل في هذه الدراسة باستخدام مطياف الرنين المغناطيسي النووي 800 ميجاهرتز المجهز برأس مسبار عكسي عريض النطاق TCI 5 مم مبرد بالتبريد وتدرج z محمي.
- ضع العينة في المغناطيس. اكتب الأمر wrpa (اذكر رقم التجربة) واضغط على Enter لإعداد تجربة بروتون باستخدام برنامج نبض cpmg. اذكر اسم المريض والتفاصيل الأخرى اللازمة من خلال النقر على خيار العنوان.
- قم بقفل المجال المغناطيسي عن طريق كتابة الأمر Lock ، واضغط على Enter ، وحدد الخيار 90٪ H2O و 10٪ D2O أيضا. قم بضبط المسبار ومطابقته يدويا بواسطة ATMM لتحسين كفاءة نبضات التردد اللاسلكي وزيادة الحساسية. قم بتنفيذ حشوة التدرج 1D بالنقر فوق topshim.
- اضبط معلمات الاكتساب التالية، بما في ذلك تأخير الاسترخاء بمقدار 5 ثوان، وعرض اكتساح طيفي يبلغ 12 جزءا في المليون (جزء في المليون)، ووقت صدى قدره 300 ميكرو ثانية. يمكن تعديل كل هذه المعلمات بسهولة عن طريق تحديد خيار معلمات الاكتساب في اللوحة العلوية.
- قم بتطبيق توسيع الخط بمقدار 0.3 هرتز باستخدام وظيفة النافذة الأسية ، وجمع البيانات إلى 64,000 نقطة عبر 128 عملية فحص. يمكن تعديل هذه المعلمات بالمثل عن طريق تحديد الخيار معلمات الاكتساب في اللوحة العلوية. بالنقر فوق معلمات الاكتساب ، ستظهر قائمة بالمواصفات ، ويمكن تعديل السمات المذكورة ، جنبا إلى جنب مع المعلمات الأخرى ، أثناء التحسين.
- الحصول على أطياف الرنين المغناطيسي النووي باستخدام أدوات معالجة وتحليل الرنين المغناطيسي النووي. بمجرد الحصول على الأطياف النهائية ، اكتب الأمر apk و absn واضغط على Enter لإجراء تصحيح الطور وتصحيح خط الأساس ، على التوالي. انقر فوق خيار معايرة المحور الموجود على اللوحة العلوية ثم قم بمعايرة ذروة TSP إلى 0 جزء في المليون تلقائيا أو يدويا.
- نقل البيانات التي تم الحصول عليها من النظام إلى محطة العمل ، حيث يتم إجراء مزيد من المعالجة والتحليل.
ملاحظة: اكتملت المرحلة التجريبية الآن ويمكن إيقافها مؤقتا. يمكن إجراء المعالجة المسبقة للبيانات وتحليلها وتفسيرها بسهولة.
4. المعالجة المسبقة للبيانات
- قم بإجراء المعالجة المسبقة للبيانات باستخدام أدوات معالجة وتحليل الرنين المغناطيسي النووي وبرنامج قياس كمية مستقلب الرنين المغناطيسي النووي.
- في بعض الأحيان ، لا يكفي التصحيح التلقائي فقط ، وفي هذه الحالة ، يكون التصحيح اليدوي ضروريا أيضا. لإجراء تصحيح الطور يدويا ، انقر فوق الخيار عملية في شريط القائمة العلوي لأدوات معالجة وتحليل الرنين المغناطيسي النووي. ثم انقر فوق ضبط المرحلة ، واسحب الماوس ، وراقب حتى يتم تصحيح الأطياف بشكل أساسي. بمجرد الانتهاء من ذلك ، انقر فوق خيار الحفظ والإرجاع (موجود في شكل رمز) المتاح في نفس شريط القائمة.
- لإجراء تصحيح خط الأساس اليدوي (طريقة ويتاكر) ، افتح الملف الطيفي في وحدة المعالج الخاصة ببرنامج قياس كمية مستقلب الرنين المغناطيسي النووي. حدد الخيار تصحيح خط الأساس. ثم حدد الخيار طريقة ويتاكر. الآن ضع النقاط في قاعدة القمم في الأطياف بأكملها (0.0 إلى 10 جزء في المليون عادة في حالة المصل أو حسب توفر المستقلب في الأطياف) لضبط القاعدة. بعد تصحيح جميع الملفات الطيفية ، احفظها في مجلد واحد. يمكن إجراء مزيد من المعالجة ، إما باستخدام المعالج أو وحدة ملف التعريف الخاصة ببرنامج القياس الكمي لمستقلب الرنين المغناطيسي النووي ، باستخدام نفس الملف المحفوظ بالتنسيق المحدد.
- قم بإجراء binning باستخدام وحدة ملفات التعريف الخاصة بنفس البرنامج. حدد الخيار أدوات، ثم عملية الدفعات، ثم التجميع الطيفي.
- لإنشاء ورقة التجميع النهائية لاستخدامها في التحليل الإحصائي، حدد المجلد الذي يحتوي على جميع الملفات الطيفية المصححة للخط الأساسي بعد النقر فوق التجميع الطيفي.
- قسم الأطياف إلى عدد محدد من الصناديق المتساوية الحجم بعرض طيفي محدد ، يتراوح من 0.01 إلى 0.04 جزء في المليون (يمكن تحسينها وفقا للدراسة). تعمل هذه العملية على تبسيط وتنظيم البيانات الطيفية ، مما يسهل تحليلا أكثر اتساقا. حدد حجم الجرافة جنبا إلى جنب مع قيم البداية والنهاية جزء في المليون وقم بتعيين المجلد حيث سيتم حفظ أوراق تجميع الإخراج.
- استبعاد مناطق التحول الكيميائي المقابلة للماء والمذيب TSP (4,8-5,2 و0,0-0,7 جزء في المليون) لمنع التداخل الطيفي 42,43,44.
- يحتوي جدول البيانات هذا على أسماء العينات في صف واحد وقيم الحاوية المختلفة جنبا إلى جنب مع جزء في المليون في الصفوف الأخرى. قبل استخدام هذه الورقة للتحليل الإحصائي، قم بتعديلها عن طريق تنظيم العينات في مجموعات لتحليلها وحفظها بتنسيق محدد بفواصل (CSV) فقط.
ملاحظة: على سبيل المثال ، عند مقارنة المجموعة الخفيفة مع المجموعة المتوسطة من مرضى متلازمة الضائقة التنفسية الحادة ، قمنا بترتيب البيانات بملصقات خفيفة ومعتدلة ، وأضفنا رقما تسلسليا ، وحفظنا الملف بتنسيق CSV.
5. التحليل الإحصائي
ملاحظة: تعمل ورقة التجميع التي تم الحصول عليها من الخطوة السابقة كملف إدخال للتحليل الإحصائي. في هذه الدراسة ، تم استخدام وحدة التحليل الإحصائي (عامل واحد) لبرنامج التحليل الإحصائي لمواد التمثيل الغذائي.
- قبل التحليل الإحصائي ، قم بإجراء خطوة التطبيع باستخدام المجموع والسجل والباريتو ولكن يمكن أن تختلف عبر دراسات مختلفة45.
- إجراء تحليل أحادي المتغير ومتعدد المتغيرات للتمييز بين المجموعات المختلفة وتحديد دقة المستقلبات المحددة.
- إجراء تحليل المكون الرئيسي (PCA) ، والتحليل التمييزي الجزئي للمربع الأدنى (PLS-DA) ، والتحليل التمييزي الجزئي المتعامد للمربع الأدنى (OPLS-DA) لمراقبة التمييز بين المجموعات التي تؤدي إلى درجات الأهمية المتغيرة للإسقاط (VIP) لكل مستقلب مسؤول عن الاختلاف في ملف التمثيل الغذائي.
- قم بإجراء طرق أحادية المتغير ، مثل اختبار الطالب t و ANOVA ، لتحديد المستقلبات المهمة بناء على معايير وجود قيمة p أقل من 0.05. ينتج عن كلا الاختبارين مخططات شعيرات صندوقية توضح الفرق في التركيز النسبي للمستقلبات.
- قم بإجراء تحليل المؤشرات الحيوية ، والذي ينتج عنه مساحة أسفل الرسم البياني لمنحنى خصائص تشغيل جهاز الاستقبال (AUROC). تعتبر قيمة AUC (المنطقة تحت المنحنى) > 0.8 مهمة46.
- حدد القائمة النهائية للمستقلبات المهمة باستخدام معايير درجة VIP>1 ، والقيمة الاحتمالية المصححة ب Bonferroni<0.0544 ، و AUC > 0.846.
- تحديد المستقلبات كما هو موضح أدناه.
- يوفر Binning مستقلبات مهمة في شكل قيم جزء في المليون بدلا من الأسماء. حدد المستقلبات المقابلة لقيم جزء في المليون المحددة باستخدام قواعد بيانات مثل بنك الرنين المغناطيسي البيولوجي (BMRB) 47وقاعدة بيانات التمثيل الغذائي البشري (HMDB).
- بخلاف ذلك ، استخدم الأدبيات المنشورةسابقا 42،44 لتحديد الهوية والتحقق من صحتها.
- استخدم وحدة ملف التعريف الخاصة ببرنامج قياس كمية مستقلبات الرنين المغناطيسي النووي لتأكيد المستقلبات المحددة بقيم جزء في المليون.
6. القياس الكمي للمستقلبات باستخدام برنامج قياس كمي مستقلب الرنين المغناطيسي النووي
ملاحظة: يتم استخدام وحدة ملف تعريف البرنامج على نطاق واسع لتحديد المستقلبات المحددة.
- قبل البدء في القياس الكمي ، قم بمعايرة المركب المرجعي المحدد للدراسة (ملعقة صغيرة مستخدمة في هذه الدراسة) باستخدام وحدة المعالج. انقر فوق الخيار معايرة CSI (مؤشر الشكل الكيميائي). اذكر تركيز المركب المرجعي (0.05 ملي مولار لهذه الدراسة). بعد ذلك ، اسحب ذروة البرنامج إلى الذروة المرجعية الطيفية واضبط ارتفاعها وعرضها بشكل صحيح. بمجرد أن تتناسب القمم المرجعية الطيفية مع ذروة البرنامج ، انقر فوق قبول.
- افتح الأطياف المطلوبة في وحدة ملف التعريف وحدد المكتبة المركبة المناسبة للدراسة.
- في الجزء السفلي من الشاشة ، يتم عرض قائمة بالمستقلبات. حدد المستقلب محل الاهتمام من هذه القائمة. بمجرد تحديده ، سيظهر جزء في المليون المحتمل من المستقلب المختار في الزاوية العلوية من الأطياف.
- حدد قيمة جزء في المليون في الزاوية العلوية وقم بتكبير الأطياف إلى مقياس جزء في المليون المحدد للمستقلب.
- في هذه المرحلة ، ستظهر قمتان على الشاشة: واحدة تمثل البيانات المسجلة والأخرى ، معروضة كخط منقط ، تمثل ذروة البرنامج. محاذاة كلا القمتين. اسحب ذروة البرنامج واضبط ارتفاعه وموضعه ليتناسب مع الذروة الموجودة في العينة.
- بمجرد محاذاة المستقلب بشكل صحيح ، يمكن الحصول على تركيز المستقلب من القيمة المعروضة تحت تركيز العنوان بالمللي مولار لهذا المستقلب (الشكل 1).
- اتبع نفس الإجراء لجميع المستقلبات والعينات وتصدير القيم الكمية في جدول بيانات. لإنشاء ملف الإخراج ، انقر فوق خيار العمليات المجمعة ضمن قائمة الأدوات . حدد المجلد المطلوب لحفظ ملف الإخراج.
7. تحليل المسار
ملاحظة: يتم استخدام المستقلبات المهمة التي تم تحديدها بعد التحليل لتحديد المسارات الرئيسية التي تؤثر بشكل مباشر على النتائج في مجموعات المرض. يتم استخدام وحدة تحليل المسار لبرنامج التحليل الإحصائي لمواد الأيض وقاعدة بيانات KEGG بشكل عام لهذا الغرض.
- أدخل القائمة التي تم الحصول عليها من المستقلبات المهمة في الوحدة. يتم إنشاء قائمة تحتوي على أسماء المسارات.
- حدد المسارات النهائية بناء على معيار وجود عامل تأثير أكبر من 0.1. تشير قيم التأثير الهامة إلى المسارات المتعلقة بالمرض30.
- قم بتحليل المسارات الناتجة بدقة لتوضيح عدم التنظيم المرتبط شدة المرض. أحد عيوب هذه الطريقة هو عدم كفاءتها في تحديد المسارات غير المنظمة عندما تكون المستقلبات المهمة أقل من 3.
- للتعامل مع نفس الشيء ، يمكن أيضا تحديد المستقلب المحدد وارتباطه بخطورة المرض باستخدام الأدبيات المنشورة سابقا. قم بإجراء مسح شامل للأدبيات ولاحظ ما إذا كان المستقلب المحدد يتبع أيضا نفس الاتجاه حسب اهتمامك.
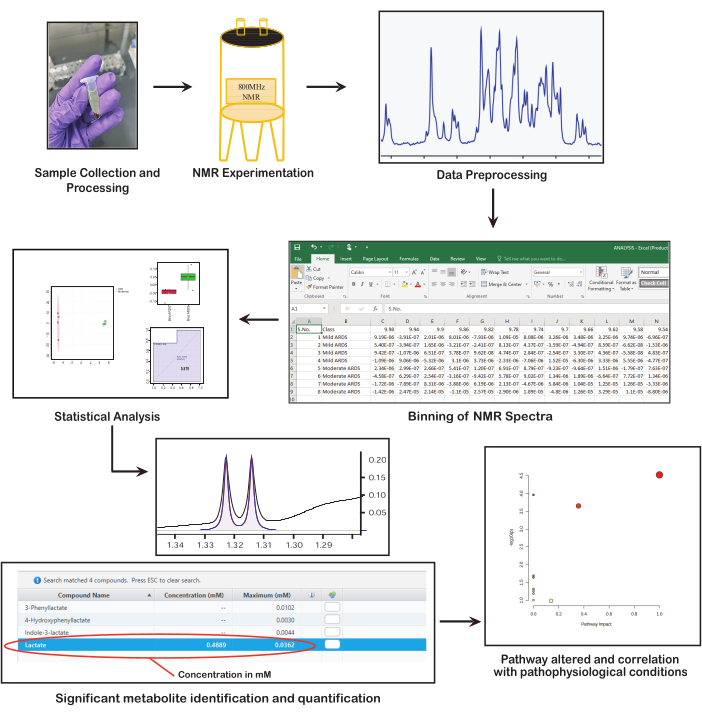
الشكل 1: الخطوات الأساسية في التمثيل الغذائي القائم على الرنين المغناطيسي النووي. يعرض الشكل الخطوات الرئيسية في التمثيل الغذائي القائم على الرنين المغناطيسي النووي: جمع العينات وإعدادها ، وإجراء التحليل الطيفي بالرنين المغناطيسي النووي ، والمعالجة المسبقة للبيانات ، وإجراء التحليل الإحصائي ، وتحديد المستقلبات وقياسها كميا ، وتفسير الأهمية البيولوجية. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
النتائج
لإجراء دراسة التمثيل الغذائي ، من الضروري تحديد حجم العينة والمجموعات المحددة التي سيتم تحليلها. يعد اختيار حجم عينة مناسب أمرا ضروريا للحصول على نتائج مهمة ترتبط بدقة مع شدةالمرض 48. ومع ذلك ، في هذا العمل بالذات ، استخدمنا حجم عينة صغير لتوضيح الخطوات المت?...
Discussion
تحدد التمثيل الغذائي المستقلبات وتقيدها بكفاءة ، مستهدفة دورات التمثيل الغذائي التي تصبح مختلة أثناء المرض. تعتمد جودة النتائج على التنفيذ الدقيق لكل خطوة في نهج التمثيل الغذائي. كل مرحلة ، من اختيار العينات وجمعها إلى تحديد المسار ، حاسمة في تحديد العوامل الأساسية الت...
Disclosures
ويعلن أصحاب البلاغ عدم وجود مصالح مالية متنافسة.
Acknowledgements
تعترف AS بأكاديمية البحث العلمي والابتكاري (AcSIR) للتسجيل (رقم التسجيل 10BB22A71002). كما تعترف AS بمنظمة البحث والتطوير الدفاعي (DRDO) للزمالة. نحن نقدر مركز البحوث الطبية الحيوية (CBMR) لتوفير مرفق مطياف الرنين المغناطيسي النووي 800 ميجاهرتز والتمويل من خلال المشروع الداخلي (CBMR / IMR / 0008/2021). كما نعرب عن تقديرنا لقسم طب الرعاية الحرجة (CCM) ، SGPGIMS ، للدعم المستمر. نحن نقدر مساعدة العديد من الممرضات ، والأهم من ذلك ، المرضى المسجلين في هذه الدراسة. تم تمويل هذه الدراسة من قبل المشروع الداخلي (CBMR / IMR / 0008/2021) التابع لمركز البحوث الطبية الحيوية (CBMR) والمشروع الخارجي (No. LSRB / 01/15001 / LSRB-404 / PEE & BS / 2023) من منظمة البحث والتطوير الدفاعي (DRDO).
Materials
| Name | Company | Catalog Number | Comments |
| Centirfuge | Sigma aldrich | 3-18KS | |
| Chenomx NMR suite | NMR Suite, v9, Chenomx Inc., Edmonton, Canada | NMR metabolite quantification software | |
| Co-axial insert | Sigma aldrich | Z278513 | |
| Deuterim oxide | Sigma aldrich | 151882 | |
| Eppendorf tubes | Tarsons | 500020 | |
| Metaboanalyst | Wishart Research Group | Metabolomics statistical analysis software | |
| NMR tube | Wilmad | Z412007 | 5mm diameter |
| Pipette | Eppendorf research plus | 3123000039 | 0-100 μl |
| Sample collection vials | Tarsons cryo chill vials | 523194 | |
| Sodium azide | Sigma aldrich | S2002 | |
| Sodium chloride crystal | Sigma aldrich | S9625 | |
| Sodium phosphate dibasic | Sigma aldrich | 567550 | |
| Sodium phosphate monobasic | Sigma aldrich | S0751 | |
| Topspin 3.6.4 | Bruker | NMR processing and analysis tool | |
| Tsp salt | Sigma aldrich | 269913 |
References
- Rubenfeld, G. D. Confronting the frustrations of negative clinical trials in acute respiratory distress syndrome. Ann. Am. Thorac. Soc. 12 (Supplement 1), S58-S63 (2015).
- Ware, L. B., et al. Prognostic and pathogenetic value of combining clinical and biochemical indices in patients with acute lung injury. Chest. 137 (2), 288-296 (2010).
- Johnson, C. H., Julijana, I., Gary, S. Metabolomics: beyond biomarkers and towards mechanisms. Nat Rev Mol Cell Biol. 17 (7), 451-459 (2016).
- Patti, G. J., Oscar, Y., Gary, S. Metabolomics: the apogee of the omics trilogy. Nat Rev Mol Cell Biol. 13 (4), 263-269 (2012).
- Nicholson, J. K., John, C. L. Metabonomics. Nature. 455 (7216), 1054-1056 (2008).
- Schnackenberg, L. K., Beger, R. D. Monitoring the health to disease continuum with global metabolic profiling and systems biology. Pharmacogenomics. 7 (7), 1077-1086 (2006).
- Pandey, S., Siddiqui, M. A., Azim, A., Sinha, N. Metabolic fingerprint of patients showing responsiveness to treatment of septic shock in intensive care unit. Magn Reson Mater Phys Biol Med. 36 (4), 659-669 (2023).
- Mamas, M., Dunn, W. B., Neyses, L., Goodacre, R. The role of metabolites and metabolomics in clinically applicable biomarkers of disease. Arch. Toxicol. 85, 5-17 (2011).
- Dunn, W. B., Broadhurst, D. I., Atherton, H. J., Goodacre, R., Griffin, J. L. Systems level studies of mammalian metabolomes: the roles of mass spectrometry and nuclear magnetic resonance spectroscopy. Chem Soc Rev. 40 (1), 387-426 (2011).
- Dettmer, K., Bruce, D. H. Metabolomics--a new exciting field within the" omics" sciences. Environ Health Perspect. 112 (7), A396-A397 (2004).
- Goodacre, R., Vaidyanathan, S., Dunn, W. B., Harrigan, G. G., Kell, D. B. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 22 (5), 245-252 (2004).
- Lindon, J. C., Nicholson, J. K., Holmes, E., Everett, J. R. Metabonomics: metabolic processes studied by NMR spectroscopy of biofluids. Concepts Magn Reson Educ J. 12 (5), 289-320 (2000).
- Bernini, P., et al. Individual human phenotypes in metabolic space and time. J Proteome Res. 8 (9), 4264-4271 (2009).
- Nicholson, J. K., et al. Metabolic phenotyping in clinical and surgical environments. Nature. 491 (7424), 384-392 (2012).
- Vinayavekhin, N., Homan, E. A., Saghatelian, A. Exploring disease through metabolomics. ACS Chem Biol. 5 (1), 91-103 (2010).
- Dunn, W. B., Ellis, D. I. Metabolomics: current analytical platforms and methodologies. TrAC Trends Anal Chem. 24 (4), 285-294 (2005).
- Dunn, W. B., Bailey, N. J. C., Johnson, H. E. Measuring the metabolome: current analytical technologies. Analyst. 130 (5), 606-625 (2005).
- Bingol, K. Recent advances in targeted and untargeted metabolomics by NMR and MS/NMR methods. High-throughput. 7 (2), 9 (2018).
- Emwas, A. H. M. The strengths and weaknesses of NMR spectroscopy and mass spectrometry with particular focus on metabolomics research. Methods Mol Biol. 1277, 161-193 (2015).
- Markley, J. L., et al. The future of NMR-based metabolomics. Curr Opin Biotechnol. 43, 34-40 (2017).
- Marchand, J., Martineau, E., Guitton, Y., Dervilly-Pinel, G., Giraudeau, P. Multidimensional NMR approaches towards highly resolved, sensitive and high-throughput quantitative metabolomics. Curr Opin Biotechnol. 43, 49-55 (2017).
- Eghbalnia, H. R., et al. Increasing rigor in NMR-based metabolomics through validated and open source tools. Curr Opin Biotechnol. 43, 56-61 (2017).
- Vignoli, A., et al. High-throughput metabolomics by 1D NMR. Angew Chem Int Ed. 58 (4), 968-994 (2019).
- Sundekilde, U. K., Eggers, N., Bertram, H. C. NMR-based metabolomics of food. Methods Mol Biol. 2037, 335-344 (2019).
- Garcia-Perez, I., et al. Identifying unknown metabolites using NMR-based metabolic profiling techniques. Nat Protoc. , 1-30 (2020).
- Dona, A. C., et al. A guide to the identification of metabolites in NMR-based metabonomics/metabolomics experiments. Comput Struct Biotechnol J. 14, 135-153 (2016).
- Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N., Siddiqui, M. H. Metabolomics: an emerging potential approach to decipher critical illnesses. Biophys Chem. 267, 106462 (2020).
- McNicholas, B. A., et al. Impact of early acute kidney injury on management and outcome in patients with acute respiratory distress syndrome: a secondary analysis of a multicenter observational study. Crit. Care Med. 47 (9), 1216-1225 (2019).
- Ambruso, S. L., et al. Inter-organ communication in homeostasis and disease: Lung metabolomics after ischemic acute kidney injury reveals increased oxidative stress, altered energy production, and ATP depletion. Am J Physiol Lung Cell Mol Physiol. 321 (1), L50 (2021).
- Viswan, A., et al. Metabolomics based predictive biomarker model of ARDS: A systemic measure of clinical hypoxemia. PloS One. 12 (11), e0187545 (2017).
- Metwaly, S. M., Brent, W. W. Systems biology ARDS research with a focus on metabolomics. Metabolites. 10 (5), 207 (2020).
- Stringer, K. A., et al. Metabolic consequences of sepsis-induced acute lung injury revealed by plasma 1H-nuclear magnetic resonance quantitative metabolomics and computational analysis. Am J Physiol Lung Cell Mol Physiol. 300 (1), L4-L11 (2011).
- Singh, A., Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N. Unveiling Pathophysiological Insights: Serum Metabolic Dysregulation in Acute Respiratory Distress Syndrome Patients with Acute Kidney Injury. J Proteome Res. 23 (10), 4216-4228 (2024).
- Slupsky, C. M. NMR-based analysis of metabolites in urine provides rapid diagnosis and etiology of pneumonia. Biomark Med. 4 (2), 195-197 (2010).
- Siddiqui, M. A., et al. NMR spectroscopy-based analysis of gallstones of cancerous and benign gallbladders from different geographical regions of the Indian subcontinent. Plos One. 18 (6), e0286979 (2023).
- Li, J., et al. 1HNMR-based metabolomic profile of rats with experimental acute pancreatitis. BMC Gastroenterol. 14, 1-7 (2014).
- Mao, H., et al. Systemic metabolic changes of traumatic critically ill patients revealed by an NMR-based metabonomic approach. J Proteome Res. 8 (12), 5423-5430 (2009).
- Stringer, K. A., Jones, A. E., Puskatich, M. A., Karnovsky, A., Serkova, N. J. 1H-nuclear magnetic resonance (NMR)-detected lipids associated with apoptosis differentiate early acute respiratory distress syndrome (ARDS) from sepsis. C63. LUNG INJURY AND REPAIR: TWO TO TANGO. Am J Resp Crit Care Med. 189, A5000 (2014).
- Viswan, A., Singh, C., Kayastha, A. M., Azim, A., Sinha, N. An NMR based panorama of the heterogeneous biology of acute respiratory distress syndrome (ARDS) from the standpoint of metabolic biomarkers. NMR Biomed. 33 (2), e4192 (2020).
- Singh, C., et al. Metabolic profiling of human lung injury by 1 H high-resolution nuclear magnetic resonance spectroscopy of blood serum. Metabolomics. 11, 166-174 (2015).
- Pandey, S., Siddiqui, M. A., Trigun, S. K., Azim, A., Sinha, N. Gender-specific association of oxidative stress and immune response in septic shock mortality using NMR-based metabolomics. Mol Omics. 18 (2), 143-153 (2022).
- Nagana, G. A., Gowda, Y. N., Raftery, D. Expanding the limits of human blood metabolite quantitation using NMR spectroscopy. Anal Chem. 87 (1), 706-715 (2015).
- Gowda, G. A. N., Raftery, D. Quantitating metabolites in protein precipitated serum using NMR spectroscopy. Anal Chem. 86 (11), 5433-5440 (2014).
- Gowda, G. A. N., Raftery, D. NMR-based metabolomics. Adv Exp Med Biol. 1280, 19-37 (2021).
- Xia, J., Psychogios, N., Young, N., Wishart, D. S. MetaboAnalyst: a web server for metabolomic data analysis and interpretation. Nucleic Acids Res. 37 (suppl_2), W652-W660 (2009).
- orbacıoğlu, &. #. 3. 5. 0. ;. K., Aksel, G. Receiver operating characteristic curve analysis in diagnostic accuracy studies: A guide to interpreting the area under the curve value. Turk J Emerg Med. 23 (4), 195 (2023).
- Ulrich, E. L., et al. BioMagResBank. Nucleic Acids Res. 36 (suppl_1), D402-D408 (2007).
- Nyamundanda, G., Gormley, I. C., Fan, Y., Gallagher, W. M., Brennan, L. MetSizeR: selecting the optimal sample size for metabolomic studies using an analysis based approach. BMC bioinformatics. 14, 1-8 (2013).
- Beckonert, O., et al. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts. Nat Protoc. 2 (11), 2692 (2007).
- Dona, A. C., et al. Precision high-throughput proton NMR spectroscopy of human urine, serum, and plasma for large-scale metabolic phenotyping. Anal Chem. 86 (19), 9887-9894 (2014).
- Ala-Korpela, M. Potential role of body fluid 1H NMR metabonomics as a prognostic and diagnostic tool. Expert Rev Mol Diagn. 7 (6), 761-773 (2007).
- Takeda, I., et al. Understanding the human salivary metabolome. NMR Biomed. 22 (6), 577-584 (2009).
- Callejon-Leblic, B. e. m., García-Barrera, T., Pereira-Vega, A., Gómez-Ariza, J. L. Metabolomic study of serum, urine and bronchoalveolar lavage fluid based on gas chromatography mass spectrometry to delve into the pathology of lung cancer. J Pharm Biomed Anal. 163, 122-129 (2019).
- Van Oort, P. M. P., et al. Exhaled breath metabolomics for the diagnosis of pneumonia in intubated and mechanically-ventilated intensive care unit (ICU)-patients. Int J Mol Sci. 18 (2), 449 (2017).
- Serkova, N. J., Niemann, C. U. Pattern recognition and biomarker validation using quantitative 1H-NMR-based metabolomics. Expert Rev Mol Diagn. 6 (5), 717-731 (2006).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation times. Rev Sci Instrum. 29 (8), 688-691 (1958).
- Xi, Y., Rocke, D. M. Baseline correction for NMR spectroscopic metabolomics data analysis. BMC bioinformatics. 9, 1-10 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved