Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Identifizierung und Quantifizierung gestörter Metaboliten bei kritisch kranken Patienten mittels NMR-basierter Metabolomik
In diesem Artikel
Zusammenfassung
Die Kernspinresonanzspektroskopie (NMR) wird eingesetzt, um eine Fehlregulation der Metaboliten bei Patienten mit verschiedenen Erkrankungen zu identifizieren. Diese Technik ermöglicht die Quantifizierung der gestörten Metaboliten und entwirrt die pathophysiologischen Erkenntnisse. Hier beschreiben wir Schritt für Schritt das Vorgehen des NMR-basierten Ansatzes zur metabolischen Charakterisierung der Patienten.
Zusammenfassung
Die Metabolomik entwickelt sich zu einem wichtigen Ansatz, um die Reaktion des Individuums auf pathophysiologische Bedingungen widerzuspiegeln. Die Kernspinresonanzspektroskopie (NMR) hat sich zu einem Instrument entwickelt, um metabolische Fehlregulationen bei kritisch kranken Patienten zu identifizieren, die an Erkrankungen wie dem akuten Atemnotsyndrom (ARDS), der schweren akuten Pankreatitis (SAP), dem akuten Nierenversagen (AKI) und der Sepsis leiden. Die spektralen Daten der Serumprobe der Studien- und Kontrollgruppe werden mit einem 800 MHz NMR-Spektrometer aufgezeichnet und mit NMR-Verarbeitungs- und Analysewerkzeugen verarbeitet. Darüber hinaus wird eine strenge statistische Analyse, wie z. B. univariate und multivariate Tests, durchgeführt, um signifikante Metaboliten zu lokalisieren, die dann mit Hilfe von NMR-Metaboliten-Quantifizierungssoftware genau identifiziert und quantifiziert werden. Darüber hinaus hebt die Signalweganalyse die gestörten biochemischen Zyklen hervor, die zur Schwere der Erkrankung führen. Durch diesen umfassenden Ansatz wollen die Forscher tiefere Einblicke in die metabolischen Veränderungen gewinnen, die mit diesen kritischen Krankheiten verbunden sind, und so möglicherweise den Weg für ein besseres Verständnis der Krankheit und verbesserte Diagnose- und Behandlungsstrategien ebnen.
Einleitung
Trotz kontinuierlicher Bemühungen, weltweit eine effiziente Krankheitsdiagnose zu ermöglichen, hat die zielgerichtete Therapie ihr wahres Potenzial noch nicht ausgeschöpft. Verschiedene Ansätze, wie z.B. Transkriptomik, Proteomik usw., haben zur Identifizierung mehrerer Biomarker geführt, die jedoch aufgrund der mangelnden Sensitivität und Spezifität keinen ausreichenden klinischen Nutzen hatten 1,2. Die zielgerichtete Therapie ist bei einigen multifaktoriellen Erkrankungen eine große Herausforderung, die schließlich zu einer höheren Sterblichkeit führt. Es besteht ein Bedarf an einem besseren Verständnis des zugrundeliegenden Mechanismus und der Pathophysiologie komplexer Krankheiten, die mit einer großen Heterogenität bestehen. In dieser Hinsicht hat die Entwicklung der Metabolomik die therapeutische Entwicklung revolutioniert, was schließlich dazu beitragen kann, die Behandlungsschemata für verschiedene kritische Krankheiten wie das akute Atemnotsyndrom (ARDS), die Sepsis und die schwere akute Pankreatitis (SAP) anzupassen.
Die Metabolomik ist ein umfassender Ansatz, der darauf abzielt, Moleküle mit kleinem Molekulargewicht (Metaboliten wie Aminosäuren, Lipide, Peptide, organische Säuren und Vitamine) in verschiedenen Bioflüssigkeiten, Zellen oder Gewebeextrakten zu identifizieren und zu quantifizieren. Diese Metaboliten, die typischerweise weniger als 1500 Da wiegen, spielen eine aktive Rolle in biochemischen Prozessen und spiegeln einen progressiven Abriss des biologischen Zustands des Organismus wider. Dazu gehören Substrate für wichtige enzymatische Prozesse, Zwischenprodukte in biologischen Stoffwechselwegen und Nebenprodukte des Zellstoffwechsels. Folglich erfasst die Metabolomik einen detaillierten Fingerabdruck von Ernährungseinflüssen, Arzneimittelwechselwirkungen und Krankheitszuständen. Metabolitenveränderungen sind hochempfindliche Indikatoren für den Stoffwechsel und die biologischen Signalwege, die Korrelationen mit phänotypischen Expressionen und daraus resultierenden pathophysiologischen Anomalien ermöglichen 3,4. Anfängliche Schwankungen der Metaboliten können als Frühindikatoren für den Schweregrad der Erkrankung dienen, während zeitliche Veränderungen bei der Überwachung der Behandlungswirksamkeit, des Krankheitsverlaufs und der klinischen Ergebnisse hilfreich sein können 5,6,7. Die Metabolomik verbessert somit klinische Assays und verschiedene andere Omics-Ansätze, indem sie Krankheiten durch klinische, physiologische und biochemische Endpunkte neu definiert 8,9,10,11,12,13. Die analytischen Fähigkeiten der Metabolomik werden genutzt, um die Krankheitsanfälligkeit durch veränderte Metabolitenkonzentrationen zu überwachen und zu bestimmen14,15.
In diesem Zusammenhang haben sich sowohl die Massenspektrometrie (MS) als auch die Kernspinresonanzspektroskopie (NMR) als primäre analytische Plattformen für die Erstellung von Metabolitenprofilen in biologischen Proben herausgestellt. Diese Methoden werden sowohl zur gezielten als auch zur ungezielten Identifizierung und Quantifizierung von Metaboliten verwendet 16,17,18. Jede Plattform hat ihre Vor- und Nachteile, aber die zerstörungsfreie Natur der NMR macht sie in verschiedenen In-vivo-Studien und für die Charakterisierung der Struktur unbekannter Verbindungen, insbesondere in der Anfangsphase der Metabolomik-Forschung, vorzuziehen. Die Probenfraktionierung, Derivatisierung und Ionisation, die vor der MS erforderlich sind, kann zu Verzerrungen führen und häufig zu Probenverlusten führen, was sich auf die dynamischen Merkmale auswirkt, die die NMR-Spektroskopie mit minimaler oder keiner Probenvorbereitung erfassen kann. Die Haupteinschränkung der NMR ist ihre geringere Empfindlichkeit im Vergleich zu MS, die eine niedrigere Nachweisgrenze bietet, was es schwierig macht, weniger häufig vorkommende Metaboliten nachzuweisen19. Fortschritte wie hochauflösende supraleitende Magnete, kryogenisch gekühlte NMR-Sonden und Techniken, die die Empfindlichkeit erhöhen, haben diese Einschränkung jedoch gemildert 20,21,22. Als komplementärer Ansatz zur Genomik und Proteomik gewinnt die Erstellung von Stoffwechselprofilen mittels NMR-Spektroskopie als bevorzugte Technik an Bedeutung 23,24,25. Die minimale Probenvorbereitung, Reproduzierbarkeit und Wiederholbarkeit der NMR machen sie trotz ihrer Empfindlichkeitsprobleme zu einem wertvollen Werkzeug für die Erfassung der inhärenten dynamischen Merkmale von Metaboliten26.
Mehrere Forschungsgruppen haben erfolgreich Metabolomik durchgeführt und das dysregulierte Stoffwechselprofil von Patienten für verschiedene Krankheiten27 wie ARDS 28,29,30,31,32,33, Lungenentzündung 34, Sepsis7, Gallensteine35 und Pankreatitis36 bestimmt. NMR-basierte Metabolomik-Studien an kritisch kranken Patienten haben maßgeblich dazu beigetragen, das Fortschreiten vom systemischen Entzündungsreaktionssyndrom (SIRS) zum Multiorgandysfunktionssyndrom (MODS) zu verfolgen, das eine der Hauptursachen für Mortalität und Morbidität auf Intensivstationen ist37. In einer Studie von Stringer et al. wurden Plasmaproben verwendet, um metabolische Veränderungen bei Patienten mit Sepsis-induzierter akuter Lungenschädigung (ALI) im Vergleich zu Kontrollpatienten zu untersuchen38. Die in dieser Pilotstudie als erhöht eingestuften Schlüsselmetaboliten spiegelten die beteiligten Stoffwechselwege und ihre Assoziation mit den klinischen Ergebnissen wider. Diese Forschung wurde auf die Serummetabolomik ausgeweitet, um die Sepsis von den frühen Stadien der Lungenverletzungsmechanismen bei ARDSzu unterscheiden 32. Darüber hinaus identifizierte eine weitere Studie in ARDS potente Serum-Biomarker, die eindeutig zwischen akutem Lungenversagen/ARDS und gesunden Kontrollen unterscheiden und Einblicke in systemische metabolische Veränderungen bieten, die dem akuten Auftreten von Lungenschäden entsprechen39,40.
Die NMR-Spektroskopie ist eine automatisierte Hochdurchsatz-Analysetechnik, die robuste und unvoreingenommene Informationen über den metabolischen Fingerabdruck liefert, die die zugrunde liegende Pathophysiologie aufdecken38. Die klinische Anwendung und biologische Interpretation von NMR-Daten hängt von der Gewinnung hochwertiger Spektren ab, die reichhaltige Informationen enthalten. Daher ist es von entscheidender Bedeutung, eine genaue, einheitliche und gut formulierte Datenerfassung, -verarbeitung und -analyse zu gewährleisten. Daher ist es das Ziel dieser Arbeit, die wesentlichen Schritte der NMR-basierten Metabolomik für die Identifizierung und Quantifizierung von Metaboliten zu nutzen. Diese Studie beleuchtet die wichtigsten Schritte des Protokolls, die für klinische Metabolomik-Studien erforderlich sind (Abbildung 1), wie z. B. die Auswahl geeigneter Proben, die Entnahme und Lagerung, die Probenverarbeitung und -vorbereitung, die Datenerfassung und -analyse, die Identifizierung und Quantifizierung des interessierenden Metaboliten und schließlich die Interpretation der Ergebnisse in einem klinischen Kontext, um relevante Erkenntnisse abzuleiten. Jeder dieser Schritte ist unerlässlich, um die NMR-Spektroskopie in der Metabolomik zu nutzen, um wichtige biologische und klinische Erkenntnisse zu gewinnen.
Protokoll
Die ethische Zulassung (IEC-Code: 2022-71-PhD-126) wurde von der IEC des Sanjay Gandhi Postgraduate Institute of Medical Sciences (SGPGIMS), Lucknow, erhalten. Es wurden schriftliche und informierte Einwilligungen von den Patienten oder ihren Angehörigen eingeholt, um die Studie durchzuführen und die Daten zu Forschungszwecken zu veröffentlichen. Darüber hinaus wurde die Forschung nach den institutionellen Richtlinien durchgeführt.
1. Studiendesign und ethische Freigabe
- Bestimmen Sie den Stichprobenumfang für jede Gruppe. Überprüfen Sie die Auswahlkriterien für die Teilnehmer sorgfältig und legen Sie sie fest. Wenn das Studiendesign die Einbeziehung von menschlichen oder tierischen Proben erfordert, holen Sie außerdem die ethische Freigabe der zuständigen institutionellen Ethikkommission (IEC) ein, bevor Sie mit der Probenentnahme beginnen.
- Ausgewählte ARDS-Patienten, die als leicht, mittelschwer und schwer eingestuft werden, basierend auf den diagnostischen Kriterien der Definition von Berlin 2012, die auf dem Partialdruck des Sauerstoffs im arteriellen Blut (PaO2) zum Anteil des eingeatmeten Sauerstoffs (FiO2) (P/F) basiert. Die ARDS-Patienten mit einem P/F-Bereich von 300-200 gelten als leicht, 200-100 als mittelschwer und unter 100 als schwer38. Hier haben wir uns auf zwei Gruppen konzentriert: leichte und mittelschwere ARDS-Patienten (siehe Tabelle 1).
HINWEIS: Die Studie umfasst die Verwendung von Serum, das aus Blutproben gewonnen wird, um NMR-basierte Metabolomik durchzuführen. In dieser Studie hatten die als Referenz eingeschlossenen Teilnehmer ein Durchschnittsalter von 42 Jahren (± 10,9 Jahre) mit einer Geschlechterverteilung von 2 Männern und 6 Frauen.
| Kategorie | P/F-Verhältnis |
| Leicht | 300-200 |
| Mäßig | 200-100 |
| Schwer | 100-0 |
Tabelle 1: Kategorisierung von ARDS-Patienten.
2. Probenauswahl, -entnahme und -verarbeitung
- Führen Sie die Serumisolierung durch, indem Sie die unten beschriebenen Schritte ausführen.
- Entnehmen Sie zu Experimenten 2 ml Blutproben aus den Arterien mit einer sterilen Nadel in einem glatten (ohne Zusatzstoffe) und sterilen Fläschchen. Lassen Sie die Probe 30 Minuten lang bei Raumtemperatur gerinnen. Für diese Studie wurden 4 Patienten mit leichtem ARDS und 4 Patienten mit mittelschwerem ARDS ausgewählt.
- Zentrifugieren Sie die Probe 15 Minuten lang bei 3100 x g , um das Serum25 zu trennen. Das Serum wird in sterile Mikrozentrifugenröhrchen überführt, wobei Aliquots mit kleinerem Volumen (d. h. 400-500 μl) erstellt werden, sie entsprechend gekennzeichnet werden (Name des Patienten, CR-Nr., Tagespunkt usw.) und für zukünftige Analysen bei -80 °C gelagert werden.
HINWEIS: Zu diesem Zeitpunkt kann das Experiment zu einem geeigneten Zeitpunkt angehalten und fortgesetzt werden.
- Verarbeiten Sie die Serumprobe vor NMR-Experimenten mit den folgenden Schritten.
- Tauen Sie die Probe auf, bevor Sie die NMR-Experimente durchführen. Mischen Sie 250 μl Aliquot des Serums mit 250 μl Kochsalzphosphat-Pufferlösung (enthält 100 % D2O, 0,9 % NaCl, 50 mM Natriumphosphatpuffer, pH 7,4), um pH-Schwankungen zu minimieren.
- Wirbeln Sie die Mischung einige Sekunden lang (~15 s) ein, um sicherzustellen, dass sie homogen vermischt wird. Schließlich wird die vorbereitete Probe in ein sauberes NMR-Röhrchen mit einem koaxialen Einsatz überführt, der Trimethylsilylpropionat (TSP) in einer Konzentration von 0,05 mM enthält, das als externer Standard für die Kalibrierung und Quantifizierung dient41.
HINWEIS: Als Referenz wird eine inerte und nicht reaktive Verbindung verwendet, die nicht mit den Analyten interagiert. Weitere häufig verwendete Referenzverbindungen sind Tetramethylsilan (TMS) und Natriumtrimethylsilylpropansulfonat (DSS).
3. NMR-Experimente
HINWEIS: Der Schwerpunkt liegt auf der Identifizierung kleiner Moleküle in den Serumproben, daher haben wir die Carr-Purcell-Meiboom-Gill (CPMG)-Pulssequenz verwendet, die Makromolekülsignale unterdrückt. Alle Serumproben in dieser Studie wurden mit einem 800 MHz NMR-Spektrometer aufgezeichnet, das mit einem kryogenisch gekühlten Dreifachresonanz-TCI-Sondenkopf mit 5 mm Breitband und einem abgeschirmten Z-Gradienten ausgestattet war.
- Legen Sie die Probe in den Magneten. Schreiben Sie den Befehl wrpa (Experimentnummer angeben) und drücken Sie die Eingabetaste, um ein Protonenexperiment mit dem cpmg-Pulsprogramm einzurichten. Nennen Sie den Namen des Patienten und andere notwendige Details, indem Sie auf die Option Titel klicken.
- Sperren Sie das Magnetfeld, indem Sie den Befehl Lock schreiben , drücken Sie die Eingabetaste und wählen Sie die Option 90% H2O und 10% D2O weiter. Stimmen Sie die Sonde manuell mit ATMM ab, um die Effizienz von Hochfrequenzimpulsen zu optimieren und die Empfindlichkeit zu maximieren. Führen Sie das 1D-Gradienten-Shimming durch, indem Sie auf topshim klicken.
- Stellen Sie die folgenden Erfassungsparameter ein, darunter eine Relaxationsverzögerung von 5 s, eine spektrale Sweep-Breite von 12 parts per million (ppm) und eine Echozeit von 300 μs. Alle diese Parameter können einfach geändert werden, indem Sie die Option Erfassungsparameter im oberen Bereich auswählen.
- Wenden Sie eine Linienverbreiterung von 0,3 Hz mit einer Exponentialfensterfunktion an und sammeln Sie Daten in 64.000 Punkten über 128 Scans. Diese Parameter können auf ähnliche Weise geändert werden, indem Sie die Option Erfassungsparameter im oberen Bereich auswählen. Wenn Sie auf Akquisitionsparameter klicken, erscheint eine Liste der Spezifikationen, und die genannten Attribute können zusammen mit anderen Parametern während der Optimierung geändert werden.
- Erfassen Sie die NMR-Spektren mit NMR-Verarbeitungs- und Analysewerkzeugen. Sobald die endgültigen Spektren erhalten sind, schreiben Sie den Befehl apk und absn und drücken Sie die Eingabetaste, um die Phasenkorrektur bzw. die Basislinienkorrektur durchzuführen. Klicken Sie auf die Option Achse kalibrieren im oberen Bereich und kalibrieren Sie den TSP-Peak entweder automatisch oder manuell auf 0 ppm.
- Übertragen Sie die erfassten Daten vom System auf die Workstation, wo die weitere Verarbeitung und Analyse erfolgt.
HINWEIS: Die Versuchsphase ist nun abgeschlossen und kann pausiert werden. Die Vorverarbeitung, Analyse und Interpretation von Daten kann bequem durchgeführt werden.
4. Vorverarbeitung von Daten
- Führen Sie die Datenvorverarbeitung mit NMR-Verarbeitungs- und Analysewerkzeugen und NMR-Metaboliten-Quantifizierungssoftware durch.
- Manchmal reicht eine automatische Korrektur nicht aus, und in diesem Fall ist auch eine manuelle Korrektur erforderlich. Um die Phasenkorrektur manuell durchzuführen, klicken Sie auf die Option Verarbeiten in der oberen Menüleiste der NMR-Verarbeitungs- und Analysewerkzeuge. Klicken Sie dann auf Phase anpassen, ziehen Sie die Maus und beobachten Sie, bis die Spektren phasenkorrigiert sind. Wenn Sie fertig sind, klicken Sie auf die Option Speichern und Zurückkehren (in Form eines Symbols), die in derselben Menüleiste verfügbar ist.
- Um eine manuelle Baseline-Korrektur (Whittaker-Methode) durchzuführen, öffnen Sie die Spektraldatei im Prozessormodul der NMR-Metaboliten-Quantifizierungssoftware. Wählen Sie die Option Baseline-Korrektur aus. Wählen Sie dann die Option Whittaker-Methode. Setzen Sie nun die Punkte an die Basis der Peaks in den gesamten Spektren (0,0 bis 10 ppm, normalerweise im Falle von Serum oder je nach Verfügbarkeit des Metaboliten in den Spektren), um die Basis anzupassen. Nachdem alle Spektraldateien grundlegend korrigiert wurden, speichern Sie sie in einem einzigen Ordner. Die weitere Verarbeitung, entweder unter Verwendung des Prozessors oder des Profiler-Moduls der NMR-Metaboliten-Quantifizierungssoftware, kann mit derselben Datei durchgeführt werden, die in dem spezifischen Format gespeichert ist.
- Führen Sie das Binning mit dem Profiler-Modul derselben Software durch. Wählen Sie die Option Extras, dann Stapelverarbeitung und dann Spektrales Binning aus.
- Um das endgültige Binning-Sheet für die statistische Analyse zu generieren, wählen Sie den Ordner aus, der alle baseline-korrigierten Spektraldateien enthält, nachdem Sie auf Spektral-Binning geklickt haben.
- Teilen Sie die Spektren in eine festgelegte Anzahl gleich großer Bins mit einer definierten spektralen Breite auf, die von 0,01 bis 0,04 ppm reicht (kann gemäß der Studie optimiert werden). Dieser Prozess vereinfacht und organisiert die Spektraldaten und ermöglicht so eine konsistentere Analyse. Geben Sie die Bucket-Größe zusammen mit den ppm-Werten für Start und Ende an und legen Sie den Ordner fest, in dem die Ausgabe-Binning-Blätter gespeichert werden.
- Die chemischen Verschiebungsbereiche, die Wasser und dem Lösungsmittel TSP entsprechen (4,8-5,2 und 0,0-0,7 ppm), sind auszuschließen, um spektrale Interferenzenzu vermeiden 42,43,44.
- Diese Tabelle enthält die Stichprobennamen in einer Zeile und die verschiedenen Klassenwerte zusammen mit ppm in den anderen Zeilen. Bevor Sie dieses Blatt für die statistische Analyse verwenden, ändern Sie es, indem Sie die Stichproben für die Analyse in Gruppen organisieren und nur im CSV-Format (Comma-Limited) speichern.
HINWEIS: Beim Vergleich der milden Gruppe mit der moderaten Gruppe von ARDS-Patienten haben wir beispielsweise die Daten mit leichten und mittelschweren Beschriftungen versehen, eine Seriennummer hinzugefügt und die Datei im CSV-Format gespeichert.
5. Statistische Analyse
HINWEIS: Das Binning-Sheet, das Sie im vorherigen Schritt erhalten haben, dient als Eingabedatei für die statistische Analyse. In dieser Studie wurde das Modul für die statistische Analyse (ein Faktor) der statistischen Analysesoftware für die Metabolomik verwendet.
- Führen Sie vor der statistischen Analyse den Normalisierungsschritt mit Summe, Logarithmus und Pareto durch, der jedoch in verschiedenen Studien variierenkann 45.
- Führen Sie univariate und multivariate Analysen durch, um die verschiedenen Gruppen zu unterscheiden und die Genauigkeit der identifizierten Metaboliten zu bestimmen.
- Führen Sie eine Hauptkomponentenanalyse (PCA), eine partielle Diskriminanzanalyse der kleinsten Quadrate (PLS-DA) und eine orthogonale partielle Diskriminanzanalyse der kleinsten Quadrate (OPLS-DA) durch, um die Unterscheidung zwischen den Gruppen zu beobachten, die für jeden Metaboliten, der für den Unterschied im Stoffwechselprofil verantwortlich ist, zu VIP-Werten (Variable Importance of Projection) führt.
- Führen Sie univariate Methoden durch, wie z. B. den studentischen t-Test und die ANOVA, um die signifikanten Metaboliten auf der Grundlage des Kriteriums eines p-Werts von weniger als 0,05 zu identifizieren. Beide Tests liefern Box-Whisker-Diagramme, die den Unterschied in der relativen Konzentration der Metaboliten zeigen.
- Führen Sie eine Biomarker-Analyse durch, die einen Bereich unter dem AUROC-Diagramm (Receiver Operating Characteristic Curve) ergibt. Der AUC-Wert (Fläche unter der Kurve) > 0,8 gilt als signifikant46.
- Wählen Sie die endgültige Liste der signifikanten Metaboliten anhand der Kriterien VIP-Score>1, Bonferroni-korrigierter p-Wert<0,0544 und AUC > 0,846 aus.
- Identifizieren Sie die Metaboliten wie unten beschrieben.
- Binning liefert signifikante Metaboliten in Form von ppm-Werten anstelle von Namen. Identifizieren Sie die Metaboliten, die bestimmten ppm-Werten entsprechen, mithilfe von Datenbanken wie der Biological Magnetic Resonance Bank (BMRB)47und der Human Metabolome Database (HMDB).
- Verwenden Sie außerdem bereits veröffentlichte Literatur42,44 zur Identifizierung und Validierung.
- Verwenden Sie das Profiler-Modul der NMR-Metaboliten-Quantifizierungssoftware, um die identifizierten Metaboliten mit ihren ppm-Werten zu bestätigen.
6. Quantifizierung von Metaboliten mit Hilfe von NMR-Software zur Quantifizierung von Metaboliten
HINWEIS: Das Profiler-Modul der Software wird häufig verwendet, um die identifizierten Metaboliten zu quantifizieren.
- Bevor Sie mit der Quantifizierung beginnen, kalibrieren Sie die für die Studie ausgewählte Referenzsubstanz (in dieser Studie verwendeter TSP) mit dem Prozessormodul. Klicken Sie auf die Option CSI kalibrieren (chemischer Formindikator). Nennen Sie die Konzentration der Referenzverbindung (0,05 mM für diese Studie). Ziehen Sie dann den Software-Peak auf den spektralen Referenzpeak und passen Sie dessen Höhe und Breite richtig an. Sobald die spektralen Referenzpeaks mit dem Peak der Software übereinstimmen, klicken Sie auf Akzeptieren.
- Öffnen Sie die gewünschten Spektren im Profiler-Modul und wählen Sie die entsprechende Verbindungsbibliothek für die Studie aus.
- Am unteren Rand des Bildschirms wird eine Liste der Metaboliten angezeigt. Wählen Sie den gewünschten Metaboliten aus dieser Liste aus. Nach der Auswahl erscheinen die möglichen ppm des gewählten Metaboliten in der oberen Ecke der Spektren.
- Wählen Sie den ppm-Wert in der oberen Ecke aus und zoomen Sie in die Spektren auf die spezifische ppm-Skala des Metaboliten.
- An diesem Punkt erscheinen zwei Peaks auf dem Bildschirm: Einer stellt die aufgezeichneten Daten dar und der andere, der als gepunktete Linie dargestellt wird und den Software-Peak darstellt. Richten Sie beide Spitzen aus. Ziehen Sie den Software-Peak und passen Sie seine Höhe und Position an den im Sample vorhandenen Peak an.
- Nach der richtigen Ausrichtung kann die Konzentration des Metaboliten aus dem Wert ermittelt werden, der unter der Überschrift Konzentration in mM für diesen Metaboliten angezeigt wird (Abbildung 1).
- Befolgen Sie das gleiche Verfahren für alle Metaboliten und Proben und exportieren Sie die quantifizierten Werte in eine Tabelle. Um die Ausgabedatei zu generieren, klicken Sie im Menü Extras auf die Option Batch-Operationen. Geben Sie den gewünschten Ordner zum Speichern der Ausgabedatei an.
7. Analyse des Signalwegs
HINWEIS: Die nach der Analyse identifizierten signifikanten Metaboliten werden verwendet, um die Hauptsignalwege zu bestimmen, die das Ergebnis in Krankheitsgruppen direkt beeinflussen. Hierfür werden in der Regel das Modul Signalweganalyse der statistischen Analysesoftware Metabolomics und die Datenbank KEGG eingesetzt.
- Geben Sie die erhaltene Liste der signifikanten Metaboliten in das Modul ein. Es wird eine Liste mit den Namen der Pfade generiert.
- Wählen Sie die endgültigen Pfade basierend auf dem Kriterium eines Impact-Faktors größer als 0,1 aus. Signifikante Wirkungswerte deuten auf krankheitsbezogene Signalwegehin 30.
- Analysieren Sie die resultierenden Signalwege gründlich, um die mit der Schwere der Erkrankung verbundene Dysregulation aufzuklären. Einer der Nachteile dieser Methode ist ihre Ineffizienz bei der Identifizierung der fehlregulierten Signalwege, wenn die signifikanten Metaboliten weniger als 3 betragen.
- Um dies zu bewältigen, kann der identifizierte Metabolit und seine Korrelation mit dem Schweregrad der Erkrankung auch anhand der zuvor veröffentlichten Literatur bestimmt werden. Führen Sie eine gründliche Literaturrecherche durch und beobachten Sie, ob der identifizierte Metabolit auch dem gleichen Trend folgt, der Ihren Interessen entspricht.
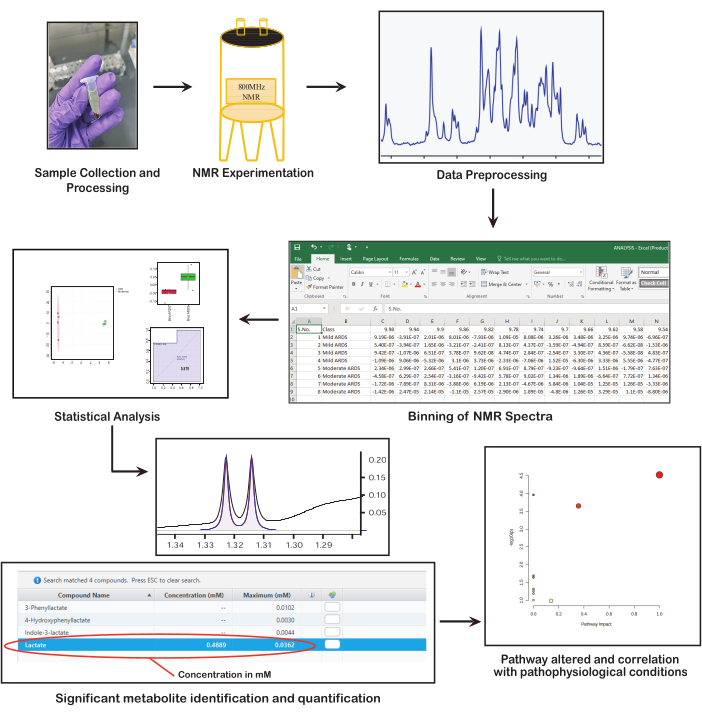
Abbildung 1: Grundlegende Schritte in der NMR-basierten Metabolomik. Die Abbildung zeigt die wichtigsten Schritte der NMR-basierten Metabolomik: Entnahme und Aufbereitung von Proben, Durchführung der NMR-Spektroskopie, Vorverarbeitung von Daten, Durchführung statistischer Analysen, Identifizierung und Quantifizierung von Metaboliten und Interpretation der biologischen Signifikanz. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Um eine Metabolomik-Studie durchzuführen, ist es wichtig, die Stichprobengröße und die spezifischen Gruppen zu bestimmen, die analysiert werden sollen. Die Auswahl einer angemessenen Stichprobengröße ist unerlässlich, um signifikante Ergebnisse zu erhalten, die genau mit dem Schweregrad der Erkrankung korrelieren48. In dieser speziellen Arbeit haben wir jedoch eine kleine Stichprobengröße verwendet, um die Schritte bei der Identifizierung und Quantifizieru...
Diskussion
Die Metabolomik identifiziert und quantifiziert effizient Metaboliten und zielt auf die Stoffwechselzyklen ab, die während der Krankheit gestört werden. Die Qualität der Ergebnisse hängt von der sorgfältigen Ausführung jedes Schritts im Metabolomik-Ansatz ab. Jede Phase, von der Probenauswahl und -entnahme bis zur Identifizierung des Signalwegs, ist entscheidend für die genaue Identifizierung der Hauptfaktoren, die zur Krankheit beitragen. Vor der Durchführung von Metabolomics is...
Offenlegungen
Die Autoren erklären, dass keine konkurrierenden finanziellen Interessen bestehen.
Danksagungen
Die AS dankt der Akademie für wissenschaftliche und innovative Forschung (AcSIR) für die Registrierung (Registrierungsnummer 10BB22A71002). AS dankt auch der Defence Research and Development Organization (DRDO) für das Stipendium. Wir danken dem Centre of Biomedical Research (CBMR) für die Bereitstellung der 800-MHz-NMR-Spektrometer-Einrichtung und die Finanzierung im Rahmen des intramuralen Projekts (CBMR/IMR/0008/2021). Wir danken auch der Abteilung für Intensivmedizin (CCM), SGPGIMS, für ihre ständige Unterstützung. Wir danken vielen Pflegekräften und vor allem den Patienten, die an dieser Studie teilgenommen haben. Diese Studie wurde durch das intramurale Projekt (CBMR/IMR/0008/2021) des Centre of Biomedical Research (CBMR) und durch das extramurale Projekt (Nr. LSRB/01/15001/LSRB-404/PEE&BS/2023) der Defence Research and Development Organization (DRDO).
Materialien
| Name | Company | Catalog Number | Comments |
| Centirfuge | Sigma aldrich | 3-18KS | |
| Chenomx NMR suite | NMR Suite, v9, Chenomx Inc., Edmonton, Canada | NMR metabolite quantification software | |
| Co-axial insert | Sigma aldrich | Z278513 | |
| Deuterim oxide | Sigma aldrich | 151882 | |
| Eppendorf tubes | Tarsons | 500020 | |
| Metaboanalyst | Wishart Research Group | Metabolomics statistical analysis software | |
| NMR tube | Wilmad | Z412007 | 5mm diameter |
| Pipette | Eppendorf research plus | 3123000039 | 0-100 μl |
| Sample collection vials | Tarsons cryo chill vials | 523194 | |
| Sodium azide | Sigma aldrich | S2002 | |
| Sodium chloride crystal | Sigma aldrich | S9625 | |
| Sodium phosphate dibasic | Sigma aldrich | 567550 | |
| Sodium phosphate monobasic | Sigma aldrich | S0751 | |
| Topspin 3.6.4 | Bruker | NMR processing and analysis tool | |
| Tsp salt | Sigma aldrich | 269913 |
Referenzen
- Rubenfeld, G. D. Confronting the frustrations of negative clinical trials in acute respiratory distress syndrome. Ann. Am. Thorac. Soc. 12 (Supplement 1), S58-S63 (2015).
- Ware, L. B., et al. Prognostic and pathogenetic value of combining clinical and biochemical indices in patients with acute lung injury. Chest. 137 (2), 288-296 (2010).
- Johnson, C. H., Julijana, I., Gary, S. Metabolomics: beyond biomarkers and towards mechanisms. Nat Rev Mol Cell Biol. 17 (7), 451-459 (2016).
- Patti, G. J., Oscar, Y., Gary, S. Metabolomics: the apogee of the omics trilogy. Nat Rev Mol Cell Biol. 13 (4), 263-269 (2012).
- Nicholson, J. K., John, C. L. Metabonomics. Nature. 455 (7216), 1054-1056 (2008).
- Schnackenberg, L. K., Beger, R. D. Monitoring the health to disease continuum with global metabolic profiling and systems biology. Pharmacogenomics. 7 (7), 1077-1086 (2006).
- Pandey, S., Siddiqui, M. A., Azim, A., Sinha, N. Metabolic fingerprint of patients showing responsiveness to treatment of septic shock in intensive care unit. Magn Reson Mater Phys Biol Med. 36 (4), 659-669 (2023).
- Mamas, M., Dunn, W. B., Neyses, L., Goodacre, R. The role of metabolites and metabolomics in clinically applicable biomarkers of disease. Arch. Toxicol. 85, 5-17 (2011).
- Dunn, W. B., Broadhurst, D. I., Atherton, H. J., Goodacre, R., Griffin, J. L. Systems level studies of mammalian metabolomes: the roles of mass spectrometry and nuclear magnetic resonance spectroscopy. Chem Soc Rev. 40 (1), 387-426 (2011).
- Dettmer, K., Bruce, D. H. Metabolomics--a new exciting field within the" omics" sciences. Environ Health Perspect. 112 (7), A396-A397 (2004).
- Goodacre, R., Vaidyanathan, S., Dunn, W. B., Harrigan, G. G., Kell, D. B. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 22 (5), 245-252 (2004).
- Lindon, J. C., Nicholson, J. K., Holmes, E., Everett, J. R. Metabonomics: metabolic processes studied by NMR spectroscopy of biofluids. Concepts Magn Reson Educ J. 12 (5), 289-320 (2000).
- Bernini, P., et al. Individual human phenotypes in metabolic space and time. J Proteome Res. 8 (9), 4264-4271 (2009).
- Nicholson, J. K., et al. Metabolic phenotyping in clinical and surgical environments. Nature. 491 (7424), 384-392 (2012).
- Vinayavekhin, N., Homan, E. A., Saghatelian, A. Exploring disease through metabolomics. ACS Chem Biol. 5 (1), 91-103 (2010).
- Dunn, W. B., Ellis, D. I. Metabolomics: current analytical platforms and methodologies. TrAC Trends Anal Chem. 24 (4), 285-294 (2005).
- Dunn, W. B., Bailey, N. J. C., Johnson, H. E. Measuring the metabolome: current analytical technologies. Analyst. 130 (5), 606-625 (2005).
- Bingol, K. Recent advances in targeted and untargeted metabolomics by NMR and MS/NMR methods. High-throughput. 7 (2), 9 (2018).
- Emwas, A. H. M. The strengths and weaknesses of NMR spectroscopy and mass spectrometry with particular focus on metabolomics research. Methods Mol Biol. 1277, 161-193 (2015).
- Markley, J. L., et al. The future of NMR-based metabolomics. Curr Opin Biotechnol. 43, 34-40 (2017).
- Marchand, J., Martineau, E., Guitton, Y., Dervilly-Pinel, G., Giraudeau, P. Multidimensional NMR approaches towards highly resolved, sensitive and high-throughput quantitative metabolomics. Curr Opin Biotechnol. 43, 49-55 (2017).
- Eghbalnia, H. R., et al. Increasing rigor in NMR-based metabolomics through validated and open source tools. Curr Opin Biotechnol. 43, 56-61 (2017).
- Vignoli, A., et al. High-throughput metabolomics by 1D NMR. Angew Chem Int Ed. 58 (4), 968-994 (2019).
- Sundekilde, U. K., Eggers, N., Bertram, H. C. NMR-based metabolomics of food. Methods Mol Biol. 2037, 335-344 (2019).
- Garcia-Perez, I., et al. Identifying unknown metabolites using NMR-based metabolic profiling techniques. Nat Protoc. , 1-30 (2020).
- Dona, A. C., et al. A guide to the identification of metabolites in NMR-based metabonomics/metabolomics experiments. Comput Struct Biotechnol J. 14, 135-153 (2016).
- Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N., Siddiqui, M. H. Metabolomics: an emerging potential approach to decipher critical illnesses. Biophys Chem. 267, 106462 (2020).
- McNicholas, B. A., et al. Impact of early acute kidney injury on management and outcome in patients with acute respiratory distress syndrome: a secondary analysis of a multicenter observational study. Crit. Care Med. 47 (9), 1216-1225 (2019).
- Ambruso, S. L., et al. Inter-organ communication in homeostasis and disease: Lung metabolomics after ischemic acute kidney injury reveals increased oxidative stress, altered energy production, and ATP depletion. Am J Physiol Lung Cell Mol Physiol. 321 (1), L50 (2021).
- Viswan, A., et al. Metabolomics based predictive biomarker model of ARDS: A systemic measure of clinical hypoxemia. PloS One. 12 (11), e0187545 (2017).
- Metwaly, S. M., Brent, W. W. Systems biology ARDS research with a focus on metabolomics. Metabolites. 10 (5), 207 (2020).
- Stringer, K. A., et al. Metabolic consequences of sepsis-induced acute lung injury revealed by plasma 1H-nuclear magnetic resonance quantitative metabolomics and computational analysis. Am J Physiol Lung Cell Mol Physiol. 300 (1), L4-L11 (2011).
- Singh, A., Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N. Unveiling Pathophysiological Insights: Serum Metabolic Dysregulation in Acute Respiratory Distress Syndrome Patients with Acute Kidney Injury. J Proteome Res. 23 (10), 4216-4228 (2024).
- Slupsky, C. M. NMR-based analysis of metabolites in urine provides rapid diagnosis and etiology of pneumonia. Biomark Med. 4 (2), 195-197 (2010).
- Siddiqui, M. A., et al. NMR spectroscopy-based analysis of gallstones of cancerous and benign gallbladders from different geographical regions of the Indian subcontinent. Plos One. 18 (6), e0286979 (2023).
- Li, J., et al. 1HNMR-based metabolomic profile of rats with experimental acute pancreatitis. BMC Gastroenterol. 14, 1-7 (2014).
- Mao, H., et al. Systemic metabolic changes of traumatic critically ill patients revealed by an NMR-based metabonomic approach. J Proteome Res. 8 (12), 5423-5430 (2009).
- Stringer, K. A., Jones, A. E., Puskatich, M. A., Karnovsky, A., Serkova, N. J. 1H-nuclear magnetic resonance (NMR)-detected lipids associated with apoptosis differentiate early acute respiratory distress syndrome (ARDS) from sepsis. C63. LUNG INJURY AND REPAIR: TWO TO TANGO. Am J Resp Crit Care Med. 189, A5000 (2014).
- Viswan, A., Singh, C., Kayastha, A. M., Azim, A., Sinha, N. An NMR based panorama of the heterogeneous biology of acute respiratory distress syndrome (ARDS) from the standpoint of metabolic biomarkers. NMR Biomed. 33 (2), e4192 (2020).
- Singh, C., et al. Metabolic profiling of human lung injury by 1 H high-resolution nuclear magnetic resonance spectroscopy of blood serum. Metabolomics. 11, 166-174 (2015).
- Pandey, S., Siddiqui, M. A., Trigun, S. K., Azim, A., Sinha, N. Gender-specific association of oxidative stress and immune response in septic shock mortality using NMR-based metabolomics. Mol Omics. 18 (2), 143-153 (2022).
- Nagana, G. A., Gowda, Y. N., Raftery, D. Expanding the limits of human blood metabolite quantitation using NMR spectroscopy. Anal Chem. 87 (1), 706-715 (2015).
- Gowda, G. A. N., Raftery, D. Quantitating metabolites in protein precipitated serum using NMR spectroscopy. Anal Chem. 86 (11), 5433-5440 (2014).
- Gowda, G. A. N., Raftery, D. NMR-based metabolomics. Adv Exp Med Biol. 1280, 19-37 (2021).
- Xia, J., Psychogios, N., Young, N., Wishart, D. S. MetaboAnalyst: a web server for metabolomic data analysis and interpretation. Nucleic Acids Res. 37 (suppl_2), W652-W660 (2009).
- orbacıoğlu, &. #. 3. 5. 0. ;. K., Aksel, G. Receiver operating characteristic curve analysis in diagnostic accuracy studies: A guide to interpreting the area under the curve value. Turk J Emerg Med. 23 (4), 195 (2023).
- Ulrich, E. L., et al. BioMagResBank. Nucleic Acids Res. 36 (suppl_1), D402-D408 (2007).
- Nyamundanda, G., Gormley, I. C., Fan, Y., Gallagher, W. M., Brennan, L. MetSizeR: selecting the optimal sample size for metabolomic studies using an analysis based approach. BMC bioinformatics. 14, 1-8 (2013).
- Beckonert, O., et al. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts. Nat Protoc. 2 (11), 2692 (2007).
- Dona, A. C., et al. Precision high-throughput proton NMR spectroscopy of human urine, serum, and plasma for large-scale metabolic phenotyping. Anal Chem. 86 (19), 9887-9894 (2014).
- Ala-Korpela, M. Potential role of body fluid 1H NMR metabonomics as a prognostic and diagnostic tool. Expert Rev Mol Diagn. 7 (6), 761-773 (2007).
- Takeda, I., et al. Understanding the human salivary metabolome. NMR Biomed. 22 (6), 577-584 (2009).
- Callejon-Leblic, B. e. m., García-Barrera, T., Pereira-Vega, A., Gómez-Ariza, J. L. Metabolomic study of serum, urine and bronchoalveolar lavage fluid based on gas chromatography mass spectrometry to delve into the pathology of lung cancer. J Pharm Biomed Anal. 163, 122-129 (2019).
- Van Oort, P. M. P., et al. Exhaled breath metabolomics for the diagnosis of pneumonia in intubated and mechanically-ventilated intensive care unit (ICU)-patients. Int J Mol Sci. 18 (2), 449 (2017).
- Serkova, N. J., Niemann, C. U. Pattern recognition and biomarker validation using quantitative 1H-NMR-based metabolomics. Expert Rev Mol Diagn. 6 (5), 717-731 (2006).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation times. Rev Sci Instrum. 29 (8), 688-691 (1958).
- Xi, Y., Rocke, D. M. Baseline correction for NMR spectroscopic metabolomics data analysis. BMC bioinformatics. 9, 1-10 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten