A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
זיהוי וכימות של מטבוליטים מטורפים בחולים קריטיים באמצעות מטבולומיקה מבוססת NMR
In This Article
Summary
ספקטרוסקופיית תהודה מגנטית גרעינית (NMR) משמשת לזיהוי חוסר ויסות במטבוליטים בחולים עם מחלות שונות. טכניקה זו מאפשרת לכמת את המטבוליטים המופרעים, ולחשוף את התובנות הפתופיזיולוגיות. כאן, אנו מתארים את ההליך שלב אחר שלב של הגישה מבוססת NMR לאפיון מטבולי של החולים.
Abstract
מטבולומיקה מסתמנת כגישה משמעותית המשקפת את תגובת הפרט לתנאים פתופיזיולוגיים. ספקטרוסקופיית תהודה מגנטית גרעינית (NMR) התפתחה ככלי לזיהוי הפרעות מטבוליות בחולים קריטיים הסובלים ממצבים כמו תסמונת מצוקה נשימתית חריפה (ARDS), דלקת לבלב חריפה חמורה (SAP), פגיעה חריפה בכליות (AKI) ואלח דם. הנתונים הספקטרליים מדגימת הסרום של קבוצת המחקר והביקורת נרשמים באמצעות ספקטרומטר NMR של 800 MHz ומעובדים באמצעות כלי עיבוד וניתוח NMR. יתר על כן, ניתוח סטטיסטי קפדני, כגון בדיקות חד-משתניות ורב-משתניות, מבוצע כדי לאתר מטבוליטים משמעותיים, אשר לאחר מכן מזוהים ומכומתים במדויק באמצעות תוכנת כימות מטבוליטים NMR. בנוסף, ניתוח מסלולים מדגיש את המחזורים הביוכימיים המופרעים שגורמים לחומרת המחלה. באמצעות גישה מקיפה זו, החוקרים שואפים להשיג תובנות עמוקות יותר לגבי השינויים המטבוליים הקשורים למחלות קריטיות אלה, מה שעשוי לסלול את הדרך להבנה טובה יותר של המחלה ולשפר את אסטרטגיות האבחון והטיפול.
Introduction
למרות מאמצים מתמשכים לספק אבחון מחלות יעיל ברחבי העולם, טיפול ממוקד עדיין לא השיג את הפוטנציאל האמיתי שלו. גישות שונות, כגון תעתוק, פרוטאומיקה וכו ', הביאו לזיהוי של מספר סמנים ביולוגיים, אך אלה לא החזיקו מספיק תועלת קלינית בגלל חוסר רגישות וספציפיות 1,2. טיפול ממוקד מטרה הוא אתגר גדול בכמה מחלות מולטיפקטוריאליות, ובסופו של דבר מוביל לתמותה גבוהה יותר. יש צורך בהבנה טובה יותר של המנגנון הבסיסי והפתופיזיולוגיה של מחלות מורכבות הקיימות עם הטרוגניות רחבה. לכן, בהקשר זה, התפתחות המטבולומיקה חוללה מהפכה בפיתוח הטיפולי, אשר בסופו של דבר עשוי לסייע בהתאמת משטרי הטיפול במחלות קריטיות שונות כגון תסמונת מצוקה נשימתית חריפה (ARDS), אלח דם ודלקת לבלב חריפה חמורה (SAP).
מטבולומיקה היא גישה מקיפה שמטרתה לזהות ולכמת מולקולות קטנות בעלות משקל מולקולרי (מטבוליטים כגון חומצות אמינו, שומנים, פפטידים, חומצות אורגניות וויטמינים) על פני נוזלים ביולוגיים, תאים או תמציות רקמות שונות. מטבוליטים אלה, שבדרך כלל שוקלים פחות מ -1500 Da, ממלאים תפקידים פעילים בתהליכים ביוכימיים, המשקפים מתאר מתקדם של המצב הביולוגי של האורגניזם. הם כוללים מצעים לתהליכים אנזימטיים מרכזיים, מתווכים במסלולים ביולוגיים ותוצרי לוואי של חילוף החומרים בתאים. כתוצאה מכך, מטבולומיקה לוכדת טביעת אצבע מפורטת של השפעות תזונתיות, אינטראקציות בין תרופתיות ומצבי מחלה. שינויים מטבוליטים הם אינדיקטורים רגישים מאוד של מטבוליזם ומסלולים ביולוגיים, המאפשרים קורלציות עם ביטויים פנוטיפיים והפרעות פתופיזיולוגיות הנובעות מכך 3,4. שינויים ראשוניים במטבוליטים יכולים לשמש אינדיקטורים מוקדמים לחומרת המחלה, בעוד ששינויים זמניים עשויים לסייע במעקב אחר יעילות הטיפול, התקדמות המחלה ותוצאות קליניות 5,6,7. מטבולומיקה משפרת אפוא את הבדיקות הקליניות וגישות אומיקס שונות אחרות על ידי הגדרה מחדש של מחלות באמצעות נקודות קצה קליניות, פיזיולוגיות וביוכימיות 8,9,10,11,12,13. היכולות האנליטיות של מטבולומיקה משמשות לניטור וקביעת רגישות המחלה באמצעות שינויים בריכוזי מטבוליטים14,15.
בהקשר זה, הן ספקטרומטריית מסה (MS) והן ספקטרוסקופיית תהודה מגנטית גרעינית (NMR) התפתחו כפלטפורמות האנליטיות העיקריות ליצירת פרופיל מטבוליטים בדגימות ביולוגיות. שיטות אלה משמשות לזיהוי וכימות ממוקדים ולא ממוקדים של מטבוליטים 16,17,18. לכל פלטפורמה יתרונות ומגבלות, אך האופי הלא הרסני של תמ"ג הופך אותה לעדיפה במחקרי in vivo שונים ובאפיון מבנה של תרכובות לא ידועות, במיוחד בשלבים הראשונים של מחקר המטבולומיקה. שבר הדגימה, הדה-ריווטציה והיינון הנדרשים לפני טרשת נפוצה יכולים להציג הטיות ולעתים קרובות לגרום לאובדן דגימה, המשפיעים על התכונות הדינמיות שספקטרוסקופיית NMR יכולה ללכוד עם הכנת דגימה מינימלית או ללא הכנת דגימה. המגבלה העיקרית של NMR היא רגישותו הנמוכה יותר בהשוואה לטרשת נפוצה, המציעה גבול זיהוי נמוך יותר, מה שהופך אותו למאתגר לזהות מטבוליטים פחות נפוצים19. עם זאת, פיתוחים כגון מגנטים מוליכי-על ברזולוציה גבוהה, בדיקות NMR מקוררות קריוגנית וטכניקות המשפרות את הרגישות מיתנו מגבלה זו 20,21,22. כגישה משלימה לגנומיקה ופרוטאומיקה, פרופיל מטבולי באמצעות ספקטרוסקופיית NMR צובר תאוצה כטכניקה מועדפת 23,24,25. הכנת הדגימות, יכולת השחזור והחזרתיות המינימליות של NMR הופכות אותו לכלי רב ערך ללכידת התכונות הדינמיות הטבועות במטבוליטים למרות אתגרי הרגישות שלו26.
מספר קבוצות מחקר ביצעו מטבולומיקה בהצלחה, ואיתרו את הפרופיל המטבולי הלא מווסת של חולים במחלות שונות27 כגון ARDS 28,29,30,31,32,33, דלקת ריאות34, אלח דם7, אבני מרה35 ודלקת הלבלב 36. מחקרי מטבולומיקה מבוססי NMR על חולים במצב קריטי סייעו במעקב אחר ההתקדמות מתסמונת תגובה דלקתית מערכתית (SIRS) לתסמונת תפקוד לקוי של איברים מרובים (MODS), שהיא גורם מוביל לתמותה ותחלואה ביחידות טיפול נמרץ37. במחקר שנערך על ידי Stringer et al., דגימות פלזמה שימשו לבחינת שינויים מטבוליים בחולים עם פגיעה ריאתית חריפה הנגרמת מאלח דם (ALI) בהשוואה לחולי ביקורת38. מטבוליטים מרכזיים שנמצאו גבוהים במחקר פיילוט זה שיקפו את המסלולים המטבוליים המעורבים ואת הקשר שלהם לציונים קליניים. מחקר זה הורחב למטבוליזם בסרום כדי להבדיל בין אלח דם לבין השלבים המוקדמים של מנגנוני פגיעה ריאתית ב- ARDS32. בנוסף, מחקר אחר ב- ARDS זיהה סמנים ביולוגיים חזקים בסרום המבדילים באופן מובהק בין פגיעה ריאתית חריפה / ARDS לבין בקרות בריאות, ומציע תובנות לגבי שינויים מטבוליים מערכתיים המתאימים להתפרצות החריפה של פגיעה ריאתית39,40.
ספקטרוסקופיית NMR היא טכניקה אנליטית אוטומטית בעלת תפוקה גבוהה המספקת מידע חזק ובלתי משוחד על טביעת האצבע המטבולית החושפת את הפתופיזיולוגיה הבסיסית38. היישום הקליני והפרשנות הביולוגית של נתוני NMR תלויים בקבלת ספקטרום איכותי המכיל מידע עשיר. לכן, הבטחת איסוף, עיבוד וניתוח נתונים מדויקים, אחידים ומנוסחים היטב היא חיונית. לכן, מטרת מחקר זה היא למנף את השלבים החיוניים של מטבולומיקה מבוססת NMR לזיהוי וכימות של מטבוליטים. מחקר זה מדגיש את השלבים העיקריים של הפרוטוקול הנדרש למחקר מטבולומיקה קלינית (איור 1), כגון בחירת דגימות מתאימות, איסוף ואחסון, עיבוד והכנת דגימות, רכישה וניתוח נתונים, זיהוי וכימות המטבוליט המעניין, ובסופו של דבר, פרשנות התוצאות בהקשר קליני כדי להפיק תובנות רלוונטיות. כל אחד מהשלבים הללו חיוני למינוף ספקטרוסקופיית NMR במטבולומיקה כדי לחשוף תובנות ביולוגיות וקליניות משמעותיות.
Protocol
אישור אתי (קוד חברת החשמל: 2022-71-PhD-126) התקבל מחברת החשמל של המכון למדעי הרפואה סנג'אי גנדי (SGPGIMS), לאקנאו. הסכמות בכתב ודעת נלקחו מהמטופלים או מקרוביהם לבצע את המחקר ולפרסם את הנתונים לצורכי מחקר. יתר על כן, המחקר נערך בהתאם להנחיות המוסדיות.
1. עיצוב המחקר ואישור אתי
- קבע את גודל המדגם עבור כל קבוצה. סקור בקפידה וקבע את קריטריוני הבחירה למשתתפים. בנוסף, אם תכנון המחקר דורש הכללת דגימות אדם או בעלי חיים, יש לקבל אישור אתי מוועדת האתיקה המוסדית המתאימה (IEC) לפני תחילת איסוף הדגימות.
- חולי ARDS נבחרים מסווגים כקלים, בינוניים וחמורים, בהתבסס על קריטריוני האבחון של הגדרת ברלין 2012 בהתבסס על הלחץ החלקי של חמצן בדם עורקי (PaO2) לשבר החמצן בהשראת (FiO2) (P / F). חולי ARDS עם טווח P/F של 300-200 נחשבים קלים, 200-100 בינוניים, ומתחת ל-100 כחמורים38. כאן התמקדנו בשתי קבוצות: חולי ARDS קלים ובינוניים (ראו טבלה 1).
הערה: המחקר כולל שימוש בסרום המתקבל מדגימות דם לביצוע מטבולומיקה מבוססת NMR. במחקר זה, המשתתפים שנכללו להתייחסות היו בגיל ממוצע של 42 שנים (± 10.9 שנים), עם התפלגות מגדרית של 2 גברים ו -6 נקבות.
| קטגוריה | יחס P/F |
| מתון | 300-200 |
| מתון | 200-100 |
| חמור | 100-0 |
טבלה 1: סיווג חולי ARDS.
2. בחירה, איסוף ועיבוד של דוגמאות
- בצעו בידוד בסרום על ידי ביצוע השלבים המתוארים להלן.
- לצורך ניסויים, לאסוף 2 מ"ל של דגימות דם מן העורקים באמצעות מחט סטרילית רגיל (ללא תוספים) בקבוקון סטרילי. השאירו את הדגימה להיקרש במשך 30 דקות בטמפרטורת החדר. למחקר זה נבחרו 4 חולים שאובחנו עם ARDS, ו -4 חולים עם ARDS בינוני.
- צנטריפוגה את הדגימה ב 3100 x גרם במשך 15 דקות כדי להפריד את הסרום25. להעביר את הסרום לתוך צינורות microcentrifuge סטריליים, ביצוע aliquots של נפח קטן יותר (כלומר, 400-500 μL), לתייג אותם כראוי (שם המטופל, CR מספר, נקודת יום, וכו '), ולאחסן אותם ב -80 ° C לניתוח עתידי.
הערה: בשלב זה, ניתן להשהות את הניסוי ולחדשו בזמן נוח.
- עבד את דגימת הסרום לפני ניסויי NMR באמצעות השלבים הבאים.
- הפשירו את הדגימה לפני ביצוע ניסויי NMR. יש לערבב 250 μL של aliquot של הסרום עם 250 μL של תמיסת חיץ פוספט מלוחים (המכילה 100% D2O, 0.9% NaCl, 50 mM נתרן פוספט חיץ pH, pH 7.4) כדי למזער את השונות pH.
- מערבבים את התערובת למשך מספר שניות (~ 15 שניות) כדי להבטיח שהיא מעורבבת בצורה הומוגנית. לבסוף, להעביר את הדגימה המוכנה לצינור NMR נקי עם תוספת קואקסיאלית המכילה trimethylsilylyl propionate (TSP) בריכוז של 0.05 mM, המשמש תקן חיצוני לכיול וכימות41.
הערה: לשם השוואה, נעשה שימוש בתרכובת אינרטית ולא תגובתית שאינה מקיימת אינטראקציה עם האנליטים. תרכובות ייחוס נפוצות אחרות כוללות טטרמתילסילן (TMS) ונתרן טרימתילסילפרופילפרופאנסולפונט (DSS).
3. ניסויי תמ"ג
הערה: ההתמקדות היא בזיהוי מולקולות קטנות בדגימות הסרום, ולכן השתמשנו ברצף הדופק Carr-Purcell-Meiboom-Gill (CPMG), המדכא אותות מקרומולקולה. כל דגימות הסרום במחקר זה תועדו באמצעות ספקטרומטר NMR של 800 MHz המצויד בראש בדיקה הפוך TCI בעל תהודה משולשת מקוררת קריוגנית, בפס רחב 5 מ"מ ובשיפוע z מסוכך.
- הכניסו את הדגימה למגנט. כתוב את הפקודה wrpa (ציין מספר ניסוי) והקש Enter כדי להגדיר ניסוי פרוטון עם תוכנית הדופק cpmg. ציין את שם המטופל ופרטים אחרים הדרושים על ידי לחיצה על האפשרות כותרת.
- נעל את השדה המגנטי על-ידי כתיבת הפקודה נעל, הקש Enter ובחר באפשרות 90% H2O ו- 10% D2O נוספים . כוונן והתאם את הגשושית באופן ידני באמצעות ATMM כדי למטב את היעילות של פולסי תדר רדיו ולמקסם את הרגישות. בצע שיימינג שיפוע 1D על ידי לחיצה על topshim.
- הגדר את פרמטרי הרכישה הבאים, כולל עיכוב הרפיה של 5 שניות, רוחב טאטוא ספקטרלי של 12 חלקים למיליון (ppm) וזמן הד של 300 μs. כל הפרמטרים הללו ניתן לשנות בקלות על ידי בחירת האפשרות פרמטרים רכישה בחלונית העליונה.
- החל הרחבת קו של 0.3 הרץ באמצעות פונקציית חלון מעריכי, איסוף נתונים ל- 64,000 נקודות על פני 128 סריקות. ניתן לשנות פרמטרים אלה באופן דומה על ידי בחירת האפשרות פרמטרים רכישה בחלונית העליונה. על ידי לחיצה על פרמטרים רכישה, תופיע רשימה של מפרטים, ואת התכונות שהוזכרו, יחד עם פרמטרים אחרים, ניתן לשנות במהלך אופטימיזציה.
- רכוש את ספקטרום ה- NMR באמצעות כלי עיבוד וניתוח NMR. לאחר קבלת הספקטרום הסופי, כתוב את הפקודה apk ו - absn ולחץ על Enter כדי לבצע תיקון פאזה ותיקון קו בסיס, בהתאמה. לחץ על האפשרות כיול ציר הנמצאת בחלונית העליונה ולאחר מכן כייל את שיא TSP ל- 0 עמודים לדקה באופן אוטומטי או ידני.
- העבר את הנתונים שנרכשו מהמערכת לתחנת העבודה, שם נעשה עיבוד וניתוח נוספים.
הערה: שלב הניסוי הושלם כעת וניתן להשהותו. עיבוד מקדים, ניתוח ופרשנות של נתונים יכולים להתבצע בנוחות.
4. עיבוד מקדים של נתונים
- בצע את עיבוד הנתונים מראש באמצעות כלי עיבוד וניתוח NMR ותוכנה לכימות מטבוליטים NMR.
- לפעמים, רק תיקון אוטומטי אינו מספיק, ובמקרה זה, תיקון ידני הוא גם הכרחי. כדי לבצע תיקון פאזה באופן ידני, לחץ על האפשרות תהליך בשורת התפריטים העליונה של כלי עיבוד וניתוח NMR. לאחר מכן לחץ על התאמת פאזה, גרור את העכבר והתבונן עד לתיקון הפאזה של הספקטרום. לאחר שתסיים, לחץ על שמור והחזיר אפשרות (קיימת בצורה של סמל) זמין באותה שורת תפריטים.
- כדי לבצע תיקון בסיסי ידני (שיטת Whittaker), פתח את הקובץ הספקטרלי במודול המעבד של תוכנת כימות המטבוליטים NMR. בחרו באפשרות 'תיקון קו בסיס'. לאחר מכן בחר באפשרות Whittaker Method. כעת שים את הנקודות בבסיס הפסגות בספקטרום כולו (0.0 עד 10 ppm, בדרך כלל במקרה של סרום או לפי זמינות המטבוליט בספקטרה) כדי להתאים את הבסיס. לאחר שכל הקבצים הספקטרליים תוקנו בקו הבסיס, שמור אותם בתיקיה אחת. עיבוד נוסף, באמצעות המעבד או מודול profiler של תוכנת כימות מטבוליטים NMR, יכול להתבצע באמצעות אותו קובץ שנשמר בפורמט הספציפי.
- בצע binning באמצעות מודול profiler של אותה תוכנה. בחר באפשרות כלים, לאחר מכן תהליך אצווה ולאחר מכן בינינג ספקטרלי.
- ליצירת גיליון האיגוד הסופי שישמש לניתוח סטטיסטי, בחר בתיקייה המכילה את כל הקבצים הספקטרליים שתוקנו על-ידי קווי הבסיס לאחר לחיצה על Spectral binning.
- חלק את הספקטרום למספר מוגדר של פחים בגודל שווה עם רוחב ספקטרלי מוגדר, הנע בין 0.01 ל- 0.04 עמודים לדקה (ניתן למטב בהתאם למחקר). תהליך זה מפשט ומארגן את הנתונים הספקטרליים, ומאפשר ניתוח עקבי יותר. ציין את גודל הדלי יחד עם ערכי עמודים לדקה בהתחלה ובסוף וציין את התיקיה שבה יישמרו גיליונות איחוד הפלט.
- לא לכלול את אזורי השינוי הכימי המתאימים למים ואת TSP הממס (4.8-5.2 ו- 0.0-0.7 ppm) כדי למנוע הפרעות ספקטרליות 42,43,44.
- גיליון אלקטרוני זה מכיל את השמות לדוגמה בשורה אחת ואת ערכי הסלים השונים יחד עם ppm בשורות האחרות. לפני השימוש בגיליון זה לניתוח סטטיסטי, שנה אותו על-ידי ארגון הדגימות לקבוצות לצורך ניתוח ושמירתו בתבנית המופרדת באמצעות פסיקים (CSV) בלבד.
הערה: לדוגמה, כאשר השווינו את הקבוצה הקלה לקבוצה המתונה של חולי ARDS, סידרנו את הנתונים עם תוויות קלות ובינוניות, הוספנו מספר סידורי ושמרנו את הקובץ בפורמט CSV.
5. ניתוח סטטיסטי
הערה: גיליון האיגוד שהתקבל מהשלב הקודם משמש כקובץ הקלט לניתוח סטטיסטי. במחקר זה נעשה שימוש במודול הניתוח הסטטיסטי (גורם אחד) של תוכנת ניתוח סטטיסטי מטבולומי.
- לפני ניתוח סטטיסטי, בצע את שלב הנורמליזציה באמצעות סכום, יומן ופארטו, אך הוא יכול להשתנות בין מחקרים שונים45.
- בצע ניתוח חד משתני ורב-משתני כדי להבחין בין הקבוצות השונות ולקבוע את דיוק המטבוליטים שזוהו.
- בצע ניתוח רכיבים עיקריים (PCA), ניתוח דיסקרימיננטי חלקי הכי פחות ריבועי (PLS-DA) וניתוח דיסקרימיננטי חלקי אורתוגונלי (OPLS-DA) כדי לבחון אפליה בין הקבוצות המניבות ציוני חשיבות משתנה של היטל (VIP) עבור כל מטבוליט האחראי להבדל בפרופיל המטבולי.
- בצע שיטות חד משתניות, כגון מבחן t סטודנט ו- ANOVA, כדי לזהות את המטבוליטים המשמעותיים בהתבסס על הקריטריונים של בעל ערך p קטן מ -0.05. שתי הבדיקות מניבות חלקות שפם קופסה המראות את ההבדל בריכוז היחסי של המטבוליטים.
- בצע ניתוח סמנים ביולוגיים, אשר מניב אזור מתחת לגרף עקומת ההפעלה האופיינית של המקלט (AUROC). ערך AUC (שטח מתחת לעקומה) > 0.8 נחשב משמעותי46.
- בחר את הרשימה הסופית של מטבוליטים משמעותיים באמצעות הקריטריונים ציון VIP>1, ערך p מתוקן Bonferroni<0.0544, ו- AUC > 0.846.
- זהה מטבוליטים כמתואר להלן.
- Binning מספק מטבוליטים משמעותיים בצורה של ערכי ppm ולא שמות. זהה את המטבוליטים המתאימים לערכי ppm ספציפיים באמצעות מסדי נתונים כגון בנק התהודה המגנטית הביולוגית (BMRB)47ומסד הנתונים של חילוף החומרים האנושי (HMDB).
- מלבד אלה, השתמש בספרות42,44 שפורסמה בעבר לצורך זיהוי ואימות.
- השתמש במודול profiler של תוכנת כימות מטבוליטים NMR כדי לאשר את המטבוליטים המזוהים עם ערכי ppm שלהם.
6. כימות מטבוליטים באמצעות תוכנת כימות מטבוליטים NMR
הערה: מודול הפרופיילר של התוכנה נמצא בשימוש נרחב כדי לכמת את המטבוליטים שזוהו.
- לפני תחילת הכימות, כייל את תרכובת הייחוס שנבחרה למחקר (כפית המשמשת במחקר זה) באמצעות מודול המעבד. לחץ על האפשרות כיול CSI (מחוון צורה כימית). ציין את ריכוז תרכובת הייחוס (0.05 mM עבור מחקר זה). לאחר מכן, גרור את שיא התוכנה אל שיא הייחוס הספקטרלי והתאם את גובהו ורוחבו כראוי. ברגע ששיאי הייחוס הספקטרליים מתאימים לשיא של התוכנה, לחץ על קבל.
- פתח את הספקטרום הרצוי במודול הפרופיילר ובחר את ספריית התרכובות המתאימה למחקר.
- בתחתית המסך מוצגת רשימה של מטבוליטים. בחר את המטבוליט של עניין מרשימה זו. לאחר שנבחר, ppm האפשרי של המטבוליט שנבחר יופיע בפינה העליונה של הספקטרה.
- בחר ערך Ppm בפינה העליונה והתקרב לספקטרום לקנה המידה הספציפי ppm של המטבוליט.
- בשלב זה יופיעו על המסך שני שיאים: האחד מייצג את הנתונים המוקלטים והשני, המוצג כקו מקווקו, המייצג את שיא התוכנה. יישר את שתי הפסגות. גרור את שיא התוכנה וכוונן את גובהו ומיקומו כך שיתאימו לשיא הקיים בדגימה.
- לאחר יישור נכון, ניתן לקבל את ריכוז המטבוליט מהערך המוצג תחת הכותרת ריכוז ב-mM עבור אותו מטבוליט (איור 1).
- בצע את אותו הליך עבור כל המטבוליטים והדגימות וייצא את הערכים המכומתים בגיליון אלקטרוני. כדי ליצור את קובץ הפלט, לחץ על פעולות אצווה אפשרות תחת כלים תפריט. ציין את התיקייה הרצויה לשמירת קובץ הפלט.
7. ניתוח מסלול
הערה: המטבוליטים המשמעותיים שזוהו לאחר הניתוח משמשים לקביעת המסלולים העיקריים המשפיעים ישירות על התוצאה בקבוצות המחלה. מודול ניתוח המסלול של תוכנת ניתוח סטטיסטי מטבולומיקה ומסד הנתונים KEGG משמשים בדרך כלל למטרה זו.
- הזן את הרשימה המתקבלת של מטבוליטים משמעותיים למודול. נוצרת רשימה המכילה את שמות הנתיבים.
- בחר את המסלולים הסופיים בהתבסס על קריטריון של גורם השפעה גדול מ- 0.1. ערכי השפעה משמעותיים מצביעים על מסלולים הקשורים למחלה30.
- לנתח את המסלולים המתקבלים ביסודיות כדי להבהיר את dysregulation הקשורים לחומרת המחלה. אחד היתרונות של שיטה זו הוא חוסר היעילות שלה בזיהוי מסלולים dysregulated כאשר מטבוליטים משמעותיים הם פחות מ 3.
- כדי להתמודד עם אותו דבר, מטבוליט מזוהה ואת הקשר שלה עם חומרת המחלה ניתן לקבוע גם באמצעות הספרות שפורסמה בעבר. בצע סקר ספרות יסודי ובדוק אם המטבוליט המזוהה עוקב גם הוא אחר אותה מגמה לפי העניין שלך.
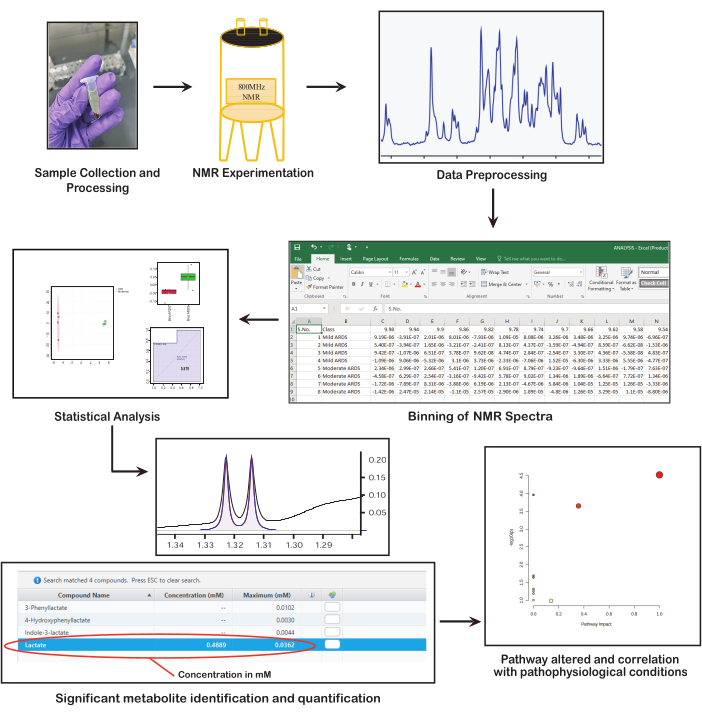
איור 1: שלבים בסיסיים במטבולומיקה מבוססת NMR. האיור מציג שלבים מרכזיים במטבולומיקה מבוססת NMR: איסוף והכנת דגימות, ביצוע ספקטרוסקופיית NMR, עיבוד מקדים של נתונים, ביצוע ניתוח סטטיסטי, זיהוי וכימות מטבוליטים ופענוח מובהקות ביולוגית. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
תוצאות
כדי לבצע מחקר מטבולומי, חיוני לקבוע את גודל המדגם ואת הקבוצות הספציפיות שינותחו. בחירת גודל מדגם מתאים חיונית להשגת תוצאות משמעותיות המתאימות במדויק לחומרת המחלה48. עם זאת, בעבודה ספציפית זו, השתמשנו בגודל מדגם קטן כדי להדגים את השלבים הכרוכים בזיהוי וכימות ...
Discussion
מטבולומיקה מזהה ומכמתת ביעילות מטבוליטים, ומכוונת למחזורים המטבוליים שמשתבשים במהלך המחלה. איכות התוצאות תלויה בביצוע קפדני של כל שלב בגישה המטבולומית. כל שלב, החל מבחירת הדגימה ואיסוף ועד לזיהוי המסלולים, הוא קריטי בזיהוי מדויק של הגורמים העיקריים התורמים למחלה. לפני ...
Disclosures
המחברים מצהירים כי אין אינטרס כלכלי מתחרה.
Acknowledgements
AS מודה לאקדמיה למחקר מדעי וחדשני (AcSIR) לרישום (מספר רישום 10BB22A71002). AS גם מודה לארגון המחקר והפיתוח הביטחוני (DRDO) על המלגה. אנו מודים למרכז למחקר ביו-רפואי (CBMR) על אספקת מתקן ספקטרומטר NMR של 800 מגה-הרץ ומימון באמצעות פרויקט האינטרמורל (CBMR/IMR/0008/2021). אנו מודים גם למחלקה לרפואת טיפול נמרץ (CCM), SGPGIMS, על תמיכה מתמדת. אנו מכירים בעזרתן של אחיות רבות, כמו גם, והכי חשוב, של החולים שנרשמו למחקר זה. מחקר זה מומן על ידי פרויקט האינטרמורל (CBMR/IMR/0008/2021) של המרכז למחקר ביו-רפואי (CBMR) ועל ידי הפרויקט החוץ-ציורי (No. LSRB/01/15001/LSRB-404/PEE&BS/2023) של ארגון המחקר והפיתוח הביטחוני (DRDO).
Materials
| Name | Company | Catalog Number | Comments |
| Centirfuge | Sigma aldrich | 3-18KS | |
| Chenomx NMR suite | NMR Suite, v9, Chenomx Inc., Edmonton, Canada | NMR metabolite quantification software | |
| Co-axial insert | Sigma aldrich | Z278513 | |
| Deuterim oxide | Sigma aldrich | 151882 | |
| Eppendorf tubes | Tarsons | 500020 | |
| Metaboanalyst | Wishart Research Group | Metabolomics statistical analysis software | |
| NMR tube | Wilmad | Z412007 | 5mm diameter |
| Pipette | Eppendorf research plus | 3123000039 | 0-100 μl |
| Sample collection vials | Tarsons cryo chill vials | 523194 | |
| Sodium azide | Sigma aldrich | S2002 | |
| Sodium chloride crystal | Sigma aldrich | S9625 | |
| Sodium phosphate dibasic | Sigma aldrich | 567550 | |
| Sodium phosphate monobasic | Sigma aldrich | S0751 | |
| Topspin 3.6.4 | Bruker | NMR processing and analysis tool | |
| Tsp salt | Sigma aldrich | 269913 |
References
- Rubenfeld, G. D. Confronting the frustrations of negative clinical trials in acute respiratory distress syndrome. Ann. Am. Thorac. Soc. 12 (Supplement 1), S58-S63 (2015).
- Ware, L. B., et al. Prognostic and pathogenetic value of combining clinical and biochemical indices in patients with acute lung injury. Chest. 137 (2), 288-296 (2010).
- Johnson, C. H., Julijana, I., Gary, S. Metabolomics: beyond biomarkers and towards mechanisms. Nat Rev Mol Cell Biol. 17 (7), 451-459 (2016).
- Patti, G. J., Oscar, Y., Gary, S. Metabolomics: the apogee of the omics trilogy. Nat Rev Mol Cell Biol. 13 (4), 263-269 (2012).
- Nicholson, J. K., John, C. L. Metabonomics. Nature. 455 (7216), 1054-1056 (2008).
- Schnackenberg, L. K., Beger, R. D. Monitoring the health to disease continuum with global metabolic profiling and systems biology. Pharmacogenomics. 7 (7), 1077-1086 (2006).
- Pandey, S., Siddiqui, M. A., Azim, A., Sinha, N. Metabolic fingerprint of patients showing responsiveness to treatment of septic shock in intensive care unit. Magn Reson Mater Phys Biol Med. 36 (4), 659-669 (2023).
- Mamas, M., Dunn, W. B., Neyses, L., Goodacre, R. The role of metabolites and metabolomics in clinically applicable biomarkers of disease. Arch. Toxicol. 85, 5-17 (2011).
- Dunn, W. B., Broadhurst, D. I., Atherton, H. J., Goodacre, R., Griffin, J. L. Systems level studies of mammalian metabolomes: the roles of mass spectrometry and nuclear magnetic resonance spectroscopy. Chem Soc Rev. 40 (1), 387-426 (2011).
- Dettmer, K., Bruce, D. H. Metabolomics--a new exciting field within the" omics" sciences. Environ Health Perspect. 112 (7), A396-A397 (2004).
- Goodacre, R., Vaidyanathan, S., Dunn, W. B., Harrigan, G. G., Kell, D. B. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 22 (5), 245-252 (2004).
- Lindon, J. C., Nicholson, J. K., Holmes, E., Everett, J. R. Metabonomics: metabolic processes studied by NMR spectroscopy of biofluids. Concepts Magn Reson Educ J. 12 (5), 289-320 (2000).
- Bernini, P., et al. Individual human phenotypes in metabolic space and time. J Proteome Res. 8 (9), 4264-4271 (2009).
- Nicholson, J. K., et al. Metabolic phenotyping in clinical and surgical environments. Nature. 491 (7424), 384-392 (2012).
- Vinayavekhin, N., Homan, E. A., Saghatelian, A. Exploring disease through metabolomics. ACS Chem Biol. 5 (1), 91-103 (2010).
- Dunn, W. B., Ellis, D. I. Metabolomics: current analytical platforms and methodologies. TrAC Trends Anal Chem. 24 (4), 285-294 (2005).
- Dunn, W. B., Bailey, N. J. C., Johnson, H. E. Measuring the metabolome: current analytical technologies. Analyst. 130 (5), 606-625 (2005).
- Bingol, K. Recent advances in targeted and untargeted metabolomics by NMR and MS/NMR methods. High-throughput. 7 (2), 9 (2018).
- Emwas, A. H. M. The strengths and weaknesses of NMR spectroscopy and mass spectrometry with particular focus on metabolomics research. Methods Mol Biol. 1277, 161-193 (2015).
- Markley, J. L., et al. The future of NMR-based metabolomics. Curr Opin Biotechnol. 43, 34-40 (2017).
- Marchand, J., Martineau, E., Guitton, Y., Dervilly-Pinel, G., Giraudeau, P. Multidimensional NMR approaches towards highly resolved, sensitive and high-throughput quantitative metabolomics. Curr Opin Biotechnol. 43, 49-55 (2017).
- Eghbalnia, H. R., et al. Increasing rigor in NMR-based metabolomics through validated and open source tools. Curr Opin Biotechnol. 43, 56-61 (2017).
- Vignoli, A., et al. High-throughput metabolomics by 1D NMR. Angew Chem Int Ed. 58 (4), 968-994 (2019).
- Sundekilde, U. K., Eggers, N., Bertram, H. C. NMR-based metabolomics of food. Methods Mol Biol. 2037, 335-344 (2019).
- Garcia-Perez, I., et al. Identifying unknown metabolites using NMR-based metabolic profiling techniques. Nat Protoc. , 1-30 (2020).
- Dona, A. C., et al. A guide to the identification of metabolites in NMR-based metabonomics/metabolomics experiments. Comput Struct Biotechnol J. 14, 135-153 (2016).
- Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N., Siddiqui, M. H. Metabolomics: an emerging potential approach to decipher critical illnesses. Biophys Chem. 267, 106462 (2020).
- McNicholas, B. A., et al. Impact of early acute kidney injury on management and outcome in patients with acute respiratory distress syndrome: a secondary analysis of a multicenter observational study. Crit. Care Med. 47 (9), 1216-1225 (2019).
- Ambruso, S. L., et al. Inter-organ communication in homeostasis and disease: Lung metabolomics after ischemic acute kidney injury reveals increased oxidative stress, altered energy production, and ATP depletion. Am J Physiol Lung Cell Mol Physiol. 321 (1), L50 (2021).
- Viswan, A., et al. Metabolomics based predictive biomarker model of ARDS: A systemic measure of clinical hypoxemia. PloS One. 12 (11), e0187545 (2017).
- Metwaly, S. M., Brent, W. W. Systems biology ARDS research with a focus on metabolomics. Metabolites. 10 (5), 207 (2020).
- Stringer, K. A., et al. Metabolic consequences of sepsis-induced acute lung injury revealed by plasma 1H-nuclear magnetic resonance quantitative metabolomics and computational analysis. Am J Physiol Lung Cell Mol Physiol. 300 (1), L4-L11 (2011).
- Singh, A., Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N. Unveiling Pathophysiological Insights: Serum Metabolic Dysregulation in Acute Respiratory Distress Syndrome Patients with Acute Kidney Injury. J Proteome Res. 23 (10), 4216-4228 (2024).
- Slupsky, C. M. NMR-based analysis of metabolites in urine provides rapid diagnosis and etiology of pneumonia. Biomark Med. 4 (2), 195-197 (2010).
- Siddiqui, M. A., et al. NMR spectroscopy-based analysis of gallstones of cancerous and benign gallbladders from different geographical regions of the Indian subcontinent. Plos One. 18 (6), e0286979 (2023).
- Li, J., et al. 1HNMR-based metabolomic profile of rats with experimental acute pancreatitis. BMC Gastroenterol. 14, 1-7 (2014).
- Mao, H., et al. Systemic metabolic changes of traumatic critically ill patients revealed by an NMR-based metabonomic approach. J Proteome Res. 8 (12), 5423-5430 (2009).
- Stringer, K. A., Jones, A. E., Puskatich, M. A., Karnovsky, A., Serkova, N. J. 1H-nuclear magnetic resonance (NMR)-detected lipids associated with apoptosis differentiate early acute respiratory distress syndrome (ARDS) from sepsis. C63. LUNG INJURY AND REPAIR: TWO TO TANGO. Am J Resp Crit Care Med. 189, A5000 (2014).
- Viswan, A., Singh, C., Kayastha, A. M., Azim, A., Sinha, N. An NMR based panorama of the heterogeneous biology of acute respiratory distress syndrome (ARDS) from the standpoint of metabolic biomarkers. NMR Biomed. 33 (2), e4192 (2020).
- Singh, C., et al. Metabolic profiling of human lung injury by 1 H high-resolution nuclear magnetic resonance spectroscopy of blood serum. Metabolomics. 11, 166-174 (2015).
- Pandey, S., Siddiqui, M. A., Trigun, S. K., Azim, A., Sinha, N. Gender-specific association of oxidative stress and immune response in septic shock mortality using NMR-based metabolomics. Mol Omics. 18 (2), 143-153 (2022).
- Nagana, G. A., Gowda, Y. N., Raftery, D. Expanding the limits of human blood metabolite quantitation using NMR spectroscopy. Anal Chem. 87 (1), 706-715 (2015).
- Gowda, G. A. N., Raftery, D. Quantitating metabolites in protein precipitated serum using NMR spectroscopy. Anal Chem. 86 (11), 5433-5440 (2014).
- Gowda, G. A. N., Raftery, D. NMR-based metabolomics. Adv Exp Med Biol. 1280, 19-37 (2021).
- Xia, J., Psychogios, N., Young, N., Wishart, D. S. MetaboAnalyst: a web server for metabolomic data analysis and interpretation. Nucleic Acids Res. 37 (suppl_2), W652-W660 (2009).
- orbacıoğlu, &. #. 3. 5. 0. ;. K., Aksel, G. Receiver operating characteristic curve analysis in diagnostic accuracy studies: A guide to interpreting the area under the curve value. Turk J Emerg Med. 23 (4), 195 (2023).
- Ulrich, E. L., et al. BioMagResBank. Nucleic Acids Res. 36 (suppl_1), D402-D408 (2007).
- Nyamundanda, G., Gormley, I. C., Fan, Y., Gallagher, W. M., Brennan, L. MetSizeR: selecting the optimal sample size for metabolomic studies using an analysis based approach. BMC bioinformatics. 14, 1-8 (2013).
- Beckonert, O., et al. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts. Nat Protoc. 2 (11), 2692 (2007).
- Dona, A. C., et al. Precision high-throughput proton NMR spectroscopy of human urine, serum, and plasma for large-scale metabolic phenotyping. Anal Chem. 86 (19), 9887-9894 (2014).
- Ala-Korpela, M. Potential role of body fluid 1H NMR metabonomics as a prognostic and diagnostic tool. Expert Rev Mol Diagn. 7 (6), 761-773 (2007).
- Takeda, I., et al. Understanding the human salivary metabolome. NMR Biomed. 22 (6), 577-584 (2009).
- Callejon-Leblic, B. e. m., García-Barrera, T., Pereira-Vega, A., Gómez-Ariza, J. L. Metabolomic study of serum, urine and bronchoalveolar lavage fluid based on gas chromatography mass spectrometry to delve into the pathology of lung cancer. J Pharm Biomed Anal. 163, 122-129 (2019).
- Van Oort, P. M. P., et al. Exhaled breath metabolomics for the diagnosis of pneumonia in intubated and mechanically-ventilated intensive care unit (ICU)-patients. Int J Mol Sci. 18 (2), 449 (2017).
- Serkova, N. J., Niemann, C. U. Pattern recognition and biomarker validation using quantitative 1H-NMR-based metabolomics. Expert Rev Mol Diagn. 6 (5), 717-731 (2006).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation times. Rev Sci Instrum. 29 (8), 688-691 (1958).
- Xi, Y., Rocke, D. M. Baseline correction for NMR spectroscopic metabolomics data analysis. BMC bioinformatics. 9, 1-10 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved