このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
NMRベースのメタボロミクスを用いた重症患者における代謝異常物の同定と定量
要約
核磁気共鳴(NMR)分光法は、さまざまな疾患を持つ患者の代謝産物の調節不全を特定するために使用されます。この技術により、乱れた代謝物の定量化が可能になり、病態生理学的な洞察が解き明かされます。ここでは、患者の代謝特性評価のためのNMRベースのアプローチの段階的な手順について説明します。
要約
メタボロミクスは、病態生理学的状態に対する個人の反応を反映する重要なアプローチとして浮上しています。核磁気共鳴(NMR)分光法は、急性呼吸窮迫症候群(ARDS)、重症急性膵炎(SAP)、急性腎障害(AKI)、敗血症などの疾患に苦しむ重症患者の代謝調節不全を特定するツールとして進化してきました。研究群と対照群の血清サンプルからのスペクトルデータは、800 MHz NMR分光計を使用して記録され、NMR処理および分析ツールを使用して処理されます。さらに、単変量検定や多変量検定などの厳密な統計解析を行い、有意な代謝物を特定し、NMR代謝物定量ソフトウェアを用いて正確に同定・定量します。さらに、パスウェイ解析では、病気の重症化をもたらす乱れた生化学的サイクルが強調されています。この包括的なアプローチを通じて、研究者はこれらの重篤な疾患に関連する代謝変化についてより深い洞察を得ることを目指しており、疾患に対する理解を深め、診断および治療戦略を改善するための道を開く可能性があります。
概要
世界中で効率的な疾患診断を提供するための継続的な努力にもかかわらず、標的療法はまだその真の可能性を達成していません。トランスクリプトミクス、プロテオミクスなどのさまざまなアプローチにより、いくつかのバイオマーカーが同定されましたが、感度と特異性が欠如していたため、これらは十分な臨床的有用性を保持していませんでした1,2。標的療法は、一部の多因子疾患では大きな課題であり、最終的には死亡率の上昇につながります。幅広い不均一性を持つ複雑な疾患の根底にあるメカニズムと病態生理学について、より深く理解する必要があります。このように、メタボロミクスの進化は治療法の開発に革命をもたらし、最終的には急性呼吸窮迫症候群(ARDS)、敗血症、重症急性膵炎(SAP)などのさまざまな重篤な疾患の治療計画を調整するのに役立つ可能性があります。
メタボロミクスは、さまざまな生体液、細胞、または組織抽出物にわたる低分子量分子(アミノ酸、脂質、ペプチド、有機酸、ビタミンなどの代謝物)を同定および定量することを目的とした包括的なアプローチです。これらの代謝産物は、通常1500 Da未満の重量で、生物の生物学的状態の進行的な輪郭を反映して、生化学的プロセスで積極的な役割を果たします。それらには、主要な酵素プロセスの基質、生物学的経路の中間体、および細胞代謝の副産物が含まれます。その結果、メタボロミクスは、食事の影響、薬物相互作用、および病状の詳細なフィンガープリントを捉えます。代謝物の変化は、代謝と生物学的経路の非常に感度の高い指標であり、表現型の発現と結果として生じる病態生理学的異常との相関を可能にします3,4。代謝物の初期変動は、疾患の重症度の初期指標として役立つ可能性がありますが、時間的な変化は、治療の有効性、疾患の進行、および臨床転帰のモニタリングに役立つ可能性があります5,6,7。このように、メタボロミクスは、臨床、生理学、生化学のエンドポイント8,9,10,11,12,13を通じて疾患を再定義することにより、臨床アッセイやその他のさまざまなオミクスアプローチを強化します。メタボロミクスの分析能力は、代謝物濃度の変化による疾患感受性をモニターおよび決定するために採用されています14,15。
このような状況において、質量分析(MS)と核磁気共鳴(NMR)分光法の両方が、生体サンプル中の代謝物プロファイリングの主要な分析プラットフォームとして浮上しています。これらの方法は、代謝物16,17,18の標的および非標的の両方の同定および定量に使用されます。各プラットフォームにはそれぞれ長所と短所がありますが、NMRの非破壊的な性質は、さまざまなin vivo研究や、特にメタボロミクス研究の初期段階における未知の化合物の構造解析に適しています。MSの前に必要なサンプルの分画、誘導体化、イオン化はバイアスを引き起こし、しばしばサンプル損失を引き起こし、NMR分光法が最小限のサンプル調製またはサンプル調製なしで捕捉できる動的特徴に影響を与える可能性があります。NMRの主な制限は、MSと比較して感度が低いことであり、MSは検出限界が低いため、存在量が少ない代謝物を検出するのが困難です19。しかし、高分解能超伝導磁石、極低温冷却NMRプローブ、感度を向上させる技術などの進歩により、この制限は緩和されました20,21,22。ゲノミクスとプロテオミクスの補完的なアプローチとして、NMR分光法を用いた代謝プロファイリングは、好ましい手法として注目を集めている23,24,25。NMRは、サンプル調製、再現性、再現性が最小限に抑えられているため、感度の課題にもかかわらず、代謝物の固有の動的特徴を捕捉するための貴重なツールとなっています26。
いくつかの研究グループはメタボロミクスを成功裏に実施し、ARDS28、29、30、31、32、33、肺炎34、敗血症7、胆石35、膵炎36などのさまざまな疾患の患者の調節不全の代謝プロファイルを特定している.重症患者を対象としたNMRベースのメタボロミクス研究は、全身性炎症反応症候群(SIRS)から集中治療室(ICU)の死亡率と罹患率の主な原因である多臓器機能不全症候群(MODS)への進行を追跡するのに役立っています37。Stringerらによる研究では、血漿サンプルを使用して、敗血症誘発性急性肺損傷(ALI)患者の代謝変化を対照患者と比較して調べました38。このパイロット研究で上昇した主要な代謝物は、関与する代謝経路と臨床スコアとの関連を反映しています。この研究は、ARDS32の肺損傷メカニズムの初期段階から敗血症を区別するために、血清メタボロミクスに拡張されました。さらに、ARDSの別の研究では、急性肺損傷/ ARDSと健康なコントロールを明確に区別する強力な血清バイオマーカーが特定され、肺損傷の急性発症に対応する全身代謝変化に関する洞察が得られました39,40。
NMR分光法は、代謝フィンガープリントに関する堅牢で偏りのない情報を提供するハイスループットで自動化された分析技術であり、根底にある病態生理学を明らかにします38。NMRデータの臨床応用と生物学的解釈は、豊富な情報を含む高品質のスペクトルを取得することにかかっています。したがって、正確で均一で、適切に定式化されたデータの収集、処理、および分析を確保することが重要です。したがって、この研究の目的は、代謝物の同定と定量のためにNMRベースのメタボロミクスの重要なステップを活用することです。この試験では、適切なサンプルの選択、収集と保存、サンプルの処理と調製、データの取得と分析、目的の代謝物の特定と定量化、そして最終的には臨床状況での結果の解釈による関連性のある洞察を導き出すなど、臨床メタボロミクス研究に必要なプロトコルの主要なステップに焦点を当てています(図1)。これらの各ステップは、メタボロミクスにおけるNMR分光法を活用して、重要な生物学的および臨床的洞察を明らかにするために不可欠です。
プロトコル
倫理的承認(IECコード:2022-71-PhD-126)は、ラクナウのSanjay Gandhi Postgraduate Institute of Medical Sciences(SGPGIMS)のIECから取得されました。患者またはその親族から書面によるインフォームドコンセントが得られ、研究目的でデータを公開しました。さらに、研究は機関のガイドラインに従って実施されました。
1. 研究デザインと倫理的クリアランス
- 各グループのサンプルサイズを決定します。参加者の選考基準を慎重に検討し、設定してください。さらに、研究デザインでヒトまたは動物のサンプルを含める必要がある場合は、サンプル収集を開始する前に、適切な機関倫理委員会(IEC)から倫理的許可を得てください。
- 動脈血中の酸素分圧 (PaO2) と吸気酸素分率 (FiO2) (P/F) の比率に基づくベルリン 2012 の定義の診断基準に基づいて、軽度、中等度、重度に分類された ARDS 患者を選択します。P/F 範囲が 300-200 の ARDS 患者は軽度、200-100 は中等度、100 未満は重度38 と見なされます。ここでは、軽度と中等度のARDS患者の2つのグループに焦点を当てています( 表1を参照)。
注:この研究では、血液サンプルから得られた血清を使用してNMRベースのメタボロミクスを実行します。この研究では、参照のために含まれた参加者の平均年齢は42歳(±10.9歳)で、性別分布は男性2人、女性6人でした。
| カテゴリ | P/Fレシオ |
| 軽度 | 300-200 |
| 適度 | 200-100 |
| 重度 | 100-0 |
表1:ARDS患者の分類。
2. サンプルの選別、採取、処理
- 以下で説明する手順に従って、血清の単離を行います。
- 実験のために、プレーン(添加物なし)と滅菌バイアルに入った滅菌針を使用して、動脈から2 mLの血液サンプルを採取します。サンプルを室温で30分間凝固させます。この研究では、軽度のARDSと診断された4人の患者と、中等度のARDSと診断された4人の患者が選択されました。
- サンプルを3100 x g で15分間遠心分離し、血清25を分離します。血清を滅菌微量遠心チューブに移し、少量(すなわち、400-500μL)のアリコートを作成し、それらを適切に標識し(患者名、CR番号、日別ポイントなど)、将来の分析のために-80°Cで保存します。
注:この時点で、実験は都合の良いときに一時停止および再開できます。
- NMR実験の前に、以下の手順で血清サンプルを処理します。
- NMR実験を行う前に、サンプルを解凍してください。血清のアリコート250μLを生理食塩水リン酸緩衝液250μL(100%D2O、0.9%NaCl、50 mMリン酸ナトリウム緩衝液、pH 7.4を含む)と混合して、pHの変動を最小限に抑えます。
- 混合物を数秒間(~15秒)ボルテックスして、均一に混合されていることを確認します。最後に、調製したサンプルを、0.05 mMの濃度でプロピオン酸トリメチルシリル(TSP)を含む同軸インサートを備えたきれいなNMRチューブに移します。これは、キャリブレーションおよび定量41の外部標準として機能します。
注:参考までに、分析種と相互作用しない不活性で非反応性の化合物を使用します。その他の一般的に使用される参照化合物には、テトラメチルシラン(TMS)およびトリメチルシリルプロパンスルホン酸ナトリウム(DSS)が含まれます。
3. NMR実験
注:血清サンプル中の小分子の同定に焦点が当てられているため、高分子シグナルを抑制するCarr-Purcell-Meiboom-Gill(CPMG)パルスシーケンスを使用しました。この研究のすべての血清サンプルは、極低温冷却された三重共鳴TCI 5 mm広帯域逆プローブヘッドとシールドされたzグラジエントを備えた800 MHz NMR分光計を使用して記録されました。
- サンプルを磁石に入れます。コマンドwrpa(実験番号を記載)を記述し、Enterキーを押して、cpmgパルスプログラムを使用した陽子実験を設定します。「 タイトル」オプションをクリックして、患者の名前と必要なその他の詳細を伝えます。
- コマンド「Lock」を書き込み、Enterキーを押して、オプション「90%H2O」および「10%D2O」をさらに選択して、磁場をロックします。ATMMでプローブを手動で調整および一致させることで、無線周波数パルスの効率を最適化し、感度を最大化します。トプシムをクリックして1Dグラデーションシミングを実行します。
- 5秒の緩和遅延、12ppmのスペクトル掃引幅、300μsのエコー時間など、以下の集録パラメータを設定します。これらのパラメータはすべて、上部パネルで [Acquisition Parameters ]オプションを選択することで簡単に変更できます。
- 指数ウィンドウ関数を使用して 0.3 Hz の線幅拡張を適用し、128 回のスキャンで 64,000 ポイントにデータを収集します。これらのパラメータは、上部パネルで [Acquisition Parameters ]オプションを選択することで、同様に変更できます。 [Acquisition Parameters]をクリックすると、仕様のリストが表示され、前述の属性と他のパラメータを最適化中に変更できます。
- NMRプロセッシングおよび分析ツールを使用してNMRスペクトルを取得します。最終的なスペクトルが得られたら、コマンド apk と absn を書き込み、Enter キーを押して、それぞれ位相補正とベースライン補正を実行します。上部パネルにある [Calibrate Axis ]オプションをクリックし、TSPピークを自動または手動で0 ppmにキャリブレーションします。
- 取得したデータをシステムからワークステーションに転送し、そこでさらなる処理と分析が行われます。
注:実験フェーズは完了し、一時停止できます。データの前処理、分析、解釈を便利に行うことができます。
4. データの前処理
- NMRプロセッシングおよび分析ツール、およびNMR代謝物定量ソフトウェアを使用してデータ前処理を行います。
- 時には自動修正だけでは不十分で、その場合は手動修正も必要になります。位相補正を手動で行うには、NMR処理および解析ツールの上部メニューバーにある 「プロセス 」オプションをクリックします。次に、[ 位相の調整]をクリックし、マウスをドラッグして、スペクトルが位相補正されるまで観察します。完了したら、同じメニューバーにある[ 保存して戻る ]オプション(アイコンの形で表示)をクリックします。
- 手動ベースライン(Whittaker法)補正を行うには、NMR代謝物定量ソフトウェアのプロセッサーモジュールでスペクトルファイルを開きます。 「ベースライン補正」オプションを選択します。次に、オプション「 Whittaker Method」を選択します。次に、スペクトル全体のピークの基部にドットを配置します(通常は血清の場合、またはスペクトル内の代謝物の利用可能性に応じて0.0〜10 ppm)ベースを調整します。すべてのスペクトル ファイルのベースラインが補正されたら、それらを 1 つのフォルダーに保存します。さらに、プロセッサーまたはNMR代謝物定量ソフトウェアのプロファイラーモジュールを使用して、特定の形式で保存された同じファイルを使用して、さらなる処理を行うことができます。
- 同じソフトウェアのプロファイラーモジュールを使用してビニングを実行します。「 ツール」オプションを選択し、「 バッチ処理」を選択し、「 スペクトルビニング」を選択します。
- 統計分析に使用する最終的なビニングシートを生成するには、スペクトルビニングをクリックした後、ベースライン補正されたすべてのスペクトルファイルを含むフォルダを選択します。
- スペクトルを、0.01〜0.04 ppmの範囲で定義されたスペクトル幅を持つ同じサイズのビンのセット数に分割します(研究に従って最適化できます)。このプロセスにより、スペクトルデータが簡素化および整理され、より一貫性のある分析が容易になります。バケットのサイズと開始および終了のppm値を指定し、出力ビニングシートを保存するフォルダを指定します。
- スペクトル干渉42,43,44を防ぐために、水と溶媒TSP(4.8-5.2および0.0-0.7 ppm)に対応する化学シフト領域を除外します。
- このスプレッドシートには、1 つの行にサンプル名が含まれ、他の行には異なるビン値と ppm が含まれています。このシートを統計分析に使用する前に、サンプルを分析用のグループに整理し、カンマ区切り(CSV)形式でのみ保存して変更してください。
注:たとえば、軽度のグループと中等度のARDS患者グループを比較する場合、データを軽度と中等度のラベルで整理し、シリアル番号を追加し、ファイルをCSV形式で保存しました。
5. 統計分析
注: 前の手順で取得したビニング シートは、統計分析の入力ファイルとして機能します。本研究では、メタボロミクス統計解析ソフトウェアの統計解析(一因子)モジュールを用いた。
- 統計分析の前に、sum、log、およびParetoを使用して正規化ステップを実行しますが、異なる研究45によって異なる場合があります。
- 単変量解析と多変量解析を実行して、異なるグループを区別し、同定された代謝物の精度を判断します。
- 主成分分析 (PCA)、部分最小二乗判別分析 (PLS-DA)、および直交部分最小二乗判別分析 (OPLS-DA) を実行して、代謝プロファイルの違いの原因となるすべての代謝物について、予測の変数重要度 (VIP) スコアをもたらすグループ間の識別を観察します。
- スチューデントt検定やANOVAなどの単変量法を実行し、p値が0.05未満であるという基準に基づいて有意な代謝物を特定します。どちらの試験でも、代謝物の相対濃度の違いを示すボックスウィスカープロットが得られます。
- バイオマーカー分析を実行すると、レシーバー動作特性曲線(AUROC)グラフの下の面積が得られます。0.8 > の AUC (曲線下面積) 値は有意な46 と見なされます。
- 基準 VIP スコア>1、ボンフェローニ補正 p 値<0.0544、および AUC > 0.846 を使用して、有意な代謝物の最終リストを選択します。
- 以下に説明するように代謝物を特定します。.
- ビニングは、名前ではなくppm値の形で重要な代謝物を提供します。Biological Magnetic Resonance Bank(BMRB)47やHuman Metabolome Database(HMDB)などのデータベースを使用して、特定のppm値に対応する代謝物を特定します。
- これら以外に、以前に公開された文献42,44を同定および検証に使用する。
- NMR代謝物定量ソフトウェアのプロファイラーモジュールを使用して、同定された代謝物とそのppm値を確認します。
6. NMR代謝物定量ソフトウェアによる代謝物の定量
注:ソフトウェアのプロファイラーモジュールは、同定された代謝物を定量するために広く使用されています。
- 定量を開始する前に、プロセッサーモジュールを使用して、研究用に選択した参照化合物(この研究で使用したtsp)をキャリブレーションします。「 CSI(化学形状インジケーター)のキャリブレーション」オプションをクリックします。参照化合物の濃度(この研究では0.05 mM)に言及してください。次に、ソフトウェアピークをスペクトル基準ピークにドラッグし、その高さと幅を適切に調整します。スペクトル基準ピークがソフトウェアのピークに適合したら、[ Accept]をクリックします。
- プロファイラーモジュールで目的のスペクトルを開き、研究に適した化合物ライブラリを選択します。
- 画面下部に代謝物のリストが表示されます。このリストから目的の代謝物を選択します。選択すると、選択した代謝物の可能なppmがスペクトルの上隅に表示されます。
- 上隅の Ppm値 を選択し、スペクトルを代謝物の特定のppmスケールにズームインします。
- この時点で、2つのピークが画面に表示されます。1つは記録されたデータを表し、もう1つはソフトウェアのピークを表す点線で表示されます。両方のピークを揃えます。ソフトウェアのピークをドラッグし、サンプルに存在するピークと一致するように高さと位置を調整します。
- 適切に整列すると、代謝物の濃度は、その代謝物のmM単位の見出しの下に表示された値から取得できます(図1)。
- すべての代謝物とサンプルについて同じ手順に従い、定量化された値をスプレッドシートにエクスポートします。出力ファイルを生成するには、[ツール]メニューの[バッチ操作]オプションをクリックします。出力ファイルを保存するフォルダを指定します。
7. パスウェイ解析
注:分析後に同定された有意な代謝物は、疾患群の結果に直接影響を与える主要な経路を決定するために利用されます。この目的のために、メタボロミクス統計解析ソフトウェアのパスウェイ解析モジュールとKEGGデータベースが一般的に使用されます。
- 得られた重要な代謝物のリストをモジュールに入力します。パスの名前を含むリストが生成されます。
- インパクトファクターが0.1より大きいという基準に基づいて、最終的な経路を選択します。有意な影響値は、疾患関連の経路を示しています30。
- 得られた経路を徹底的に分析し、疾患の重症度に関連する調節不全を解明します。この方法の欠点の1つは、有意な代謝物が3未満の場合に調節不全の経路を特定するのが非効率的であることです。
- これに対処するために、同定された代謝物およびその疾患の重症度との相関関係は、以前に発表された文献を使用して決定することもできる。徹底的な文献調査を行い、同定された代謝物があなたの興味と同じ傾向をたどっているかどうかを観察してください。
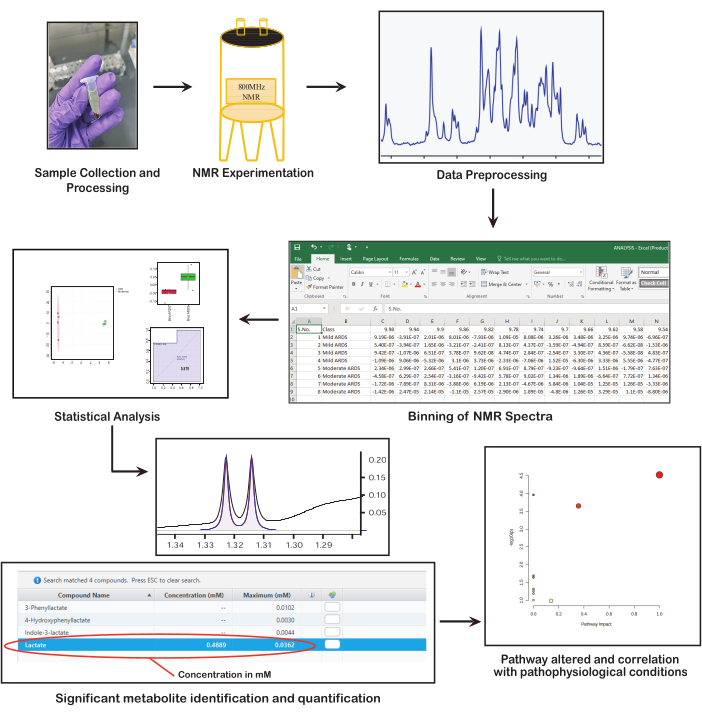
図1:NMRベースのメタボロミクスにおける基本的なステップ。 この図は、NMRベースのメタボロミクスにおける主要なステップである、サンプルの収集と調製、NMR分光法の実施、データの前処理、統計分析の実施、代謝物の同定と定量化、生物学的意義の解釈を示しています。 この図の拡大版を表示するには、ここをクリックしてください。
結果
メタボロミクス研究を実施するには、サンプルサイズと分析する特定のグループを決定することが不可欠です。適切なサンプルサイズを選択することは、疾患の重症度と正確に相関する有意な結果を得るために不可欠である48。しかし、この特定の研究では、主に参照を目的としていたNMRベースのメタボロミクスを使用した代謝物の同定と定量に関?...
ディスカッション
メタボロミクスは、疾患中に乱れる代謝サイクルを標的として、代謝物を効率的に同定・定量します。結果の品質は、メタボロミクスアプローチの各ステップの細心の注意を払って実行することに依存します。サンプルの選択と収集から経路の特定まで、すべての段階が、病気の原因となる主要な要因を正確に特定するために重要です。メタボロミクスを行う前に、...
開示事項
著者は、競合する金銭的利益を宣言しません。
謝辞
ASは、Academy of Scientific and Innovative Research(AcSIR)の登録を認めています(登録番号10BB22A71002)。ASは、フェローシップについて国防研究開発機構(DRDO)も認めています。私たちは、学内プロジェクト(CBMR/IMR/0008/2021)を通じて800 MHz NMR分光計施設と資金を提供してくれた生物医学研究センター(CBMR)に感謝します。また、SGPGIMSのクリティカルケア医学科(CCM)の継続的なサポートにも感謝しています。私たちは、多くの看護師の助けだけでなく、最も重要なことに、この研究に登録された患者に感謝します。この研究は、生物医学研究センター(CBMR)の学内プロジェクト(CBMR/IMR/0008/2021)と学外プロジェクト(No.LSRB/01/15001/LSRB-404/PEE&BS/2023) の
資料
| Name | Company | Catalog Number | Comments |
| Centirfuge | Sigma aldrich | 3-18KS | |
| Chenomx NMR suite | NMR Suite, v9, Chenomx Inc., Edmonton, Canada | NMR metabolite quantification software | |
| Co-axial insert | Sigma aldrich | Z278513 | |
| Deuterim oxide | Sigma aldrich | 151882 | |
| Eppendorf tubes | Tarsons | 500020 | |
| Metaboanalyst | Wishart Research Group | Metabolomics statistical analysis software | |
| NMR tube | Wilmad | Z412007 | 5mm diameter |
| Pipette | Eppendorf research plus | 3123000039 | 0-100 μl |
| Sample collection vials | Tarsons cryo chill vials | 523194 | |
| Sodium azide | Sigma aldrich | S2002 | |
| Sodium chloride crystal | Sigma aldrich | S9625 | |
| Sodium phosphate dibasic | Sigma aldrich | 567550 | |
| Sodium phosphate monobasic | Sigma aldrich | S0751 | |
| Topspin 3.6.4 | Bruker | NMR processing and analysis tool | |
| Tsp salt | Sigma aldrich | 269913 |
参考文献
- Rubenfeld, G. D. Confronting the frustrations of negative clinical trials in acute respiratory distress syndrome. Ann. Am. Thorac. Soc. 12 (Supplement 1), S58-S63 (2015).
- Ware, L. B., et al. Prognostic and pathogenetic value of combining clinical and biochemical indices in patients with acute lung injury. Chest. 137 (2), 288-296 (2010).
- Johnson, C. H., Julijana, I., Gary, S. Metabolomics: beyond biomarkers and towards mechanisms. Nat Rev Mol Cell Biol. 17 (7), 451-459 (2016).
- Patti, G. J., Oscar, Y., Gary, S. Metabolomics: the apogee of the omics trilogy. Nat Rev Mol Cell Biol. 13 (4), 263-269 (2012).
- Nicholson, J. K., John, C. L. Metabonomics. Nature. 455 (7216), 1054-1056 (2008).
- Schnackenberg, L. K., Beger, R. D. Monitoring the health to disease continuum with global metabolic profiling and systems biology. Pharmacogenomics. 7 (7), 1077-1086 (2006).
- Pandey, S., Siddiqui, M. A., Azim, A., Sinha, N. Metabolic fingerprint of patients showing responsiveness to treatment of septic shock in intensive care unit. Magn Reson Mater Phys Biol Med. 36 (4), 659-669 (2023).
- Mamas, M., Dunn, W. B., Neyses, L., Goodacre, R. The role of metabolites and metabolomics in clinically applicable biomarkers of disease. Arch. Toxicol. 85, 5-17 (2011).
- Dunn, W. B., Broadhurst, D. I., Atherton, H. J., Goodacre, R., Griffin, J. L. Systems level studies of mammalian metabolomes: the roles of mass spectrometry and nuclear magnetic resonance spectroscopy. Chem Soc Rev. 40 (1), 387-426 (2011).
- Dettmer, K., Bruce, D. H. Metabolomics--a new exciting field within the" omics" sciences. Environ Health Perspect. 112 (7), A396-A397 (2004).
- Goodacre, R., Vaidyanathan, S., Dunn, W. B., Harrigan, G. G., Kell, D. B. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 22 (5), 245-252 (2004).
- Lindon, J. C., Nicholson, J. K., Holmes, E., Everett, J. R. Metabonomics: metabolic processes studied by NMR spectroscopy of biofluids. Concepts Magn Reson Educ J. 12 (5), 289-320 (2000).
- Bernini, P., et al. Individual human phenotypes in metabolic space and time. J Proteome Res. 8 (9), 4264-4271 (2009).
- Nicholson, J. K., et al. Metabolic phenotyping in clinical and surgical environments. Nature. 491 (7424), 384-392 (2012).
- Vinayavekhin, N., Homan, E. A., Saghatelian, A. Exploring disease through metabolomics. ACS Chem Biol. 5 (1), 91-103 (2010).
- Dunn, W. B., Ellis, D. I. Metabolomics: current analytical platforms and methodologies. TrAC Trends Anal Chem. 24 (4), 285-294 (2005).
- Dunn, W. B., Bailey, N. J. C., Johnson, H. E. Measuring the metabolome: current analytical technologies. Analyst. 130 (5), 606-625 (2005).
- Bingol, K. Recent advances in targeted and untargeted metabolomics by NMR and MS/NMR methods. High-throughput. 7 (2), 9 (2018).
- Emwas, A. H. M. The strengths and weaknesses of NMR spectroscopy and mass spectrometry with particular focus on metabolomics research. Methods Mol Biol. 1277, 161-193 (2015).
- Markley, J. L., et al. The future of NMR-based metabolomics. Curr Opin Biotechnol. 43, 34-40 (2017).
- Marchand, J., Martineau, E., Guitton, Y., Dervilly-Pinel, G., Giraudeau, P. Multidimensional NMR approaches towards highly resolved, sensitive and high-throughput quantitative metabolomics. Curr Opin Biotechnol. 43, 49-55 (2017).
- Eghbalnia, H. R., et al. Increasing rigor in NMR-based metabolomics through validated and open source tools. Curr Opin Biotechnol. 43, 56-61 (2017).
- Vignoli, A., et al. High-throughput metabolomics by 1D NMR. Angew Chem Int Ed. 58 (4), 968-994 (2019).
- Sundekilde, U. K., Eggers, N., Bertram, H. C. NMR-based metabolomics of food. Methods Mol Biol. 2037, 335-344 (2019).
- Garcia-Perez, I., et al. Identifying unknown metabolites using NMR-based metabolic profiling techniques. Nat Protoc. , 1-30 (2020).
- Dona, A. C., et al. A guide to the identification of metabolites in NMR-based metabonomics/metabolomics experiments. Comput Struct Biotechnol J. 14, 135-153 (2016).
- Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N., Siddiqui, M. H. Metabolomics: an emerging potential approach to decipher critical illnesses. Biophys Chem. 267, 106462 (2020).
- McNicholas, B. A., et al. Impact of early acute kidney injury on management and outcome in patients with acute respiratory distress syndrome: a secondary analysis of a multicenter observational study. Crit. Care Med. 47 (9), 1216-1225 (2019).
- Ambruso, S. L., et al. Inter-organ communication in homeostasis and disease: Lung metabolomics after ischemic acute kidney injury reveals increased oxidative stress, altered energy production, and ATP depletion. Am J Physiol Lung Cell Mol Physiol. 321 (1), L50 (2021).
- Viswan, A., et al. Metabolomics based predictive biomarker model of ARDS: A systemic measure of clinical hypoxemia. PloS One. 12 (11), e0187545 (2017).
- Metwaly, S. M., Brent, W. W. Systems biology ARDS research with a focus on metabolomics. Metabolites. 10 (5), 207 (2020).
- Stringer, K. A., et al. Metabolic consequences of sepsis-induced acute lung injury revealed by plasma 1H-nuclear magnetic resonance quantitative metabolomics and computational analysis. Am J Physiol Lung Cell Mol Physiol. 300 (1), L4-L11 (2011).
- Singh, A., Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N. Unveiling Pathophysiological Insights: Serum Metabolic Dysregulation in Acute Respiratory Distress Syndrome Patients with Acute Kidney Injury. J Proteome Res. 23 (10), 4216-4228 (2024).
- Slupsky, C. M. NMR-based analysis of metabolites in urine provides rapid diagnosis and etiology of pneumonia. Biomark Med. 4 (2), 195-197 (2010).
- Siddiqui, M. A., et al. NMR spectroscopy-based analysis of gallstones of cancerous and benign gallbladders from different geographical regions of the Indian subcontinent. Plos One. 18 (6), e0286979 (2023).
- Li, J., et al. 1HNMR-based metabolomic profile of rats with experimental acute pancreatitis. BMC Gastroenterol. 14, 1-7 (2014).
- Mao, H., et al. Systemic metabolic changes of traumatic critically ill patients revealed by an NMR-based metabonomic approach. J Proteome Res. 8 (12), 5423-5430 (2009).
- Stringer, K. A., Jones, A. E., Puskatich, M. A., Karnovsky, A., Serkova, N. J. 1H-nuclear magnetic resonance (NMR)-detected lipids associated with apoptosis differentiate early acute respiratory distress syndrome (ARDS) from sepsis. C63. LUNG INJURY AND REPAIR: TWO TO TANGO. Am J Resp Crit Care Med. 189, A5000 (2014).
- Viswan, A., Singh, C., Kayastha, A. M., Azim, A., Sinha, N. An NMR based panorama of the heterogeneous biology of acute respiratory distress syndrome (ARDS) from the standpoint of metabolic biomarkers. NMR Biomed. 33 (2), e4192 (2020).
- Singh, C., et al. Metabolic profiling of human lung injury by 1 H high-resolution nuclear magnetic resonance spectroscopy of blood serum. Metabolomics. 11, 166-174 (2015).
- Pandey, S., Siddiqui, M. A., Trigun, S. K., Azim, A., Sinha, N. Gender-specific association of oxidative stress and immune response in septic shock mortality using NMR-based metabolomics. Mol Omics. 18 (2), 143-153 (2022).
- Nagana, G. A., Gowda, Y. N., Raftery, D. Expanding the limits of human blood metabolite quantitation using NMR spectroscopy. Anal Chem. 87 (1), 706-715 (2015).
- Gowda, G. A. N., Raftery, D. Quantitating metabolites in protein precipitated serum using NMR spectroscopy. Anal Chem. 86 (11), 5433-5440 (2014).
- Gowda, G. A. N., Raftery, D. NMR-based metabolomics. Adv Exp Med Biol. 1280, 19-37 (2021).
- Xia, J., Psychogios, N., Young, N., Wishart, D. S. MetaboAnalyst: a web server for metabolomic data analysis and interpretation. Nucleic Acids Res. 37 (suppl_2), W652-W660 (2009).
- orbacıoğlu, &. #. 3. 5. 0. ;. K., Aksel, G. Receiver operating characteristic curve analysis in diagnostic accuracy studies: A guide to interpreting the area under the curve value. Turk J Emerg Med. 23 (4), 195 (2023).
- Ulrich, E. L., et al. BioMagResBank. Nucleic Acids Res. 36 (suppl_1), D402-D408 (2007).
- Nyamundanda, G., Gormley, I. C., Fan, Y., Gallagher, W. M., Brennan, L. MetSizeR: selecting the optimal sample size for metabolomic studies using an analysis based approach. BMC bioinformatics. 14, 1-8 (2013).
- Beckonert, O., et al. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts. Nat Protoc. 2 (11), 2692 (2007).
- Dona, A. C., et al. Precision high-throughput proton NMR spectroscopy of human urine, serum, and plasma for large-scale metabolic phenotyping. Anal Chem. 86 (19), 9887-9894 (2014).
- Ala-Korpela, M. Potential role of body fluid 1H NMR metabonomics as a prognostic and diagnostic tool. Expert Rev Mol Diagn. 7 (6), 761-773 (2007).
- Takeda, I., et al. Understanding the human salivary metabolome. NMR Biomed. 22 (6), 577-584 (2009).
- Callejon-Leblic, B. e. m., García-Barrera, T., Pereira-Vega, A., Gómez-Ariza, J. L. Metabolomic study of serum, urine and bronchoalveolar lavage fluid based on gas chromatography mass spectrometry to delve into the pathology of lung cancer. J Pharm Biomed Anal. 163, 122-129 (2019).
- Van Oort, P. M. P., et al. Exhaled breath metabolomics for the diagnosis of pneumonia in intubated and mechanically-ventilated intensive care unit (ICU)-patients. Int J Mol Sci. 18 (2), 449 (2017).
- Serkova, N. J., Niemann, C. U. Pattern recognition and biomarker validation using quantitative 1H-NMR-based metabolomics. Expert Rev Mol Diagn. 6 (5), 717-731 (2006).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation times. Rev Sci Instrum. 29 (8), 688-691 (1958).
- Xi, Y., Rocke, D. M. Baseline correction for NMR spectroscopic metabolomics data analysis. BMC bioinformatics. 9, 1-10 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved