需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用基于 NMR 的代谢组学鉴定和定量危重患者紊乱的代谢物
摘要
核磁共振 (NMR) 波谱用于识别各种疾病患者代谢物的失调。该技术允许量化紊乱的代谢物,揭示病理生理学的见解。在这里,我们描述了基于 NMR 的方法用于患者代谢表征的分步程序。
摘要
代谢组学正在成为反映个体对病理生理状况反应的重要方法。核磁共振 (NMR) 波谱已发展成为一种工具,用于识别患有急性呼吸窘迫综合征 (ARDS)、严重急性胰腺炎 (SAP)、急性肾损伤 (AKI) 和败血症等疾病的危重患者的代谢失调。研究组和对照组血清样本的波谱数据使用 800 MHz NMR 波谱仪记录,并使用 NMR 处理和分析工具进行处理。此外,还进行了严格的统计分析,例如单变量和多变量测试,以查明重要的代谢物,然后使用 NMR 代谢物定量软件准确识别和量化这些代谢物。此外,通路分析突出了导致疾病严重程度的紊乱生化循环。通过这种全面的方法,研究人员旨在更深入地了解与这些危重疾病相关的代谢改变,从而可能为更好地了解疾病和改进诊断和治疗策略铺平道路。
引言
尽管在全球范围内不断努力提供有效的疾病诊断,但靶向治疗仍未发挥其真正的潜力。转录组学、蛋白质组学等各种方法已经鉴定出几种生物标志物,但由于缺乏敏感性和特异性,这些生物标志物没有具有足够的临床效用 1,2。靶向治疗是一些多因素疾病的一大挑战,最终会导致更高的死亡率。需要更好地了解存在广泛异质性的复杂疾病的潜在机制和病理生理学。因此,在这方面,代谢组学的发展彻底改变了治疗的发展,最终可能有助于为各种危重疾病量身定制治疗方案,例如急性呼吸窘迫综合征 (ARDS)、败血症和严重急性胰腺炎 (SAP)。
代谢组学是一种全面的方法,旨在识别和定量各种生物流体、细胞或组织提取物中的小分子量分子(代谢物,如氨基酸、脂质、肽、有机酸和维生素)。这些代谢物的重量通常小于 1500 Da,在生化过程中发挥着积极作用,反映了生物体生物状态的逐步轮廓。它们包括关键酶促过程的底物、生物途径中的中间体和细胞代谢的副产物。因此,代谢组学可以捕获饮食影响、药物相互作用和疾病状态的详细指纹。代谢物变化是代谢和生物途径的高度敏感指标,允许与表型表达和由此产生的病理生理学异常相关 3,4。代谢物的初始变化可以作为疾病严重程度的早期指标,而时间变化可能有助于监测治疗效果、疾病进展和临床结果5,6,7。因此,代谢组学通过临床、生理和生化终点重新定义疾病来增强临床测定和各种其他组学方法 8,9,10,11,12,13。代谢组学的分析能力用于通过改变代谢物浓度来监测和确定疾病易感性14,15。
在此背景下,质谱 (MS) 和核磁共振 (NMR) 波谱已成为生物样品中代谢物分析的主要分析平台。这些方法用于代谢物的靶向和非靶向鉴定和定量 16,17,18。每个平台都有其优点和局限性,但 NMR 的非破坏性使其更适合用于各种体内研究和未知化合物结构的表征,尤其是在代谢组学研究的初始阶段。MS 之前所需的样品分馏、衍生化和电离可能会引入偏倚,并经常导致样品损失,从而影响 NMR 波谱在最少或无需样品制备的情况下可以捕获的动态特征。与 MS 相比,NMR 的主要局限性是其灵敏度较低,而 MS 的检测限较低,因此难以检测丰度较低的代谢物19。然而,高分辨率超导磁体、低温冷却 NMR 探针和提高灵敏度的技术等进步缓解了这一限制 20,21,22。作为基因组学和蛋白质组学的补充方法,使用 NMR 波谱的代谢分析作为一种首选技术越来越受欢迎 23,24,25。尽管存在灵敏度挑战,但 NMR 的最小样品制备、重现性和可重复性使其成为捕获代谢物固有动态特征的宝贵工具26。
几个研究小组已经成功地进行了代谢组学研究,确定了各种疾病27 患者的代谢失调特征,例如 ARDS28、29、30、31、32、33、肺炎34、败血症7、胆结石35 和胰腺炎36.基于 NMR 的危重患者代谢组学研究有助于追踪从全身炎症反应综合征 (SIRS) 到多器官功能障碍综合征 (MODS) 的进展,这是重症监护病房 (ICU) 死亡率和发病率的主要原因37。在 Stringer 等人的一项研究中,与对照组患者相比,血浆样本用于检查败血症诱导的急性肺损伤 (ALI) 患者的代谢改变38。在这项初步研究中发现升高的关键代谢物反映了所涉及的代谢途径及其与临床评分的关联。这项研究扩展到血清代谢组学,以区分脓毒症与 ARDS32 中肺损伤机制的早期阶段。此外,ARDS 的另一项研究确定了有效的血清生物标志物,这些标志物可以明确区分急性肺损伤/ARDS 和健康对照,从而深入了解与肺损伤急性发作相对应的全身代谢变化39,40。
NMR 波谱是一种高通量和自动化分析技术,可提供有关代谢指纹图谱的可靠且无偏差的信息,揭示潜在的病理生理学38。NMR 数据的临床应用和生物学解释取决于获得包含丰富信息的高质量波谱。因此,确保准确、一致和完善的数据收集、处理和分析至关重要。因此,本研究的目的是利用基于 NMR 的代谢组学的基本步骤来鉴定和定量代谢物。本研究强调了临床代谢组学研究所需的方案的关键步骤(图 1),例如选择合适的样本、收集和储存、样本处理和制备、数据采集和分析、识别和量化感兴趣的代谢物,并最终在临床环境中解释结果以获得相关见解。这些步骤中的每一个对于在代谢组学中利用 NMR 波谱来揭示重要的生物学和临床见解都是必不可少的。
研究方案
伦理批准(IEC 代码:2022-71-PhD-126)已获得勒克瑙桑贾伊甘地医学科学研究生院 (SGPGIMS) 的 IEC。从患者或其亲属那里获得书面知情同意书,以执行研究并发布数据用于研究目的。此外,该研究是按照机构指南进行的。
1. 研究设计和伦理审查
- 确定每个组的样本数量。仔细审查并建立参与者的选择标准。此外,如果研究设计要求包含人类或动物样本,则在开始样本采集之前从适当的机构伦理委员会 (IEC) 获得伦理许可。
- 根据 2012 年柏林定义的诊断标准,根据动脉血氧分压 (PaO2) 与吸入氧分数 (FiO2) (P/F) 的比率,选择分为轻度、中度和重度的 ARDS 患者。P/F 范围为 300-200 的 ARDS 患者被认为是轻度的,200-100 是中度的,低于 100 的被认为是严重的38。在这里,我们关注两组:轻度和中度 ARDS 患者(见 表 1)。
注:该研究涉及使用从血液样本中获得的血清进行基于 NMR 的代谢组学。在本研究中,纳入参考的参与者平均年龄为 42 岁 (± 10.9 岁),性别分布为 2 名男性和 6 名女性。
| 类别 | P/F 比率 |
| 轻微 | 300-200 |
| 温和 | 200-100 |
| 严重 | 100-0 |
表 1:ARDS 患者的分类。
2. 样品选择、采集和处理
- 按照下述步骤进行血清分离。
- 对于实验,使用无菌针头在普通(无添加剂)和无菌小瓶中从动脉中收集 2 mL 血样。将样品在室温下凝固 30 分钟。在本研究中,选择了 4 例诊断为轻度 ARDS 的患者和 4 例中度 ARDS 患者。
- 将样品以 3100 x g 离心 15 分钟以分离血清25。将血清转移到无菌微量离心管中,制成较小体积的等分试样(即 400-500 μL),适当标记它们(患者姓名、CR 编号、日期等),并将它们储存在 -80 °C 以备将来分析。
注意:此时,可以在方便的时间暂停和恢复实验。
- 在NMR实验之前,使用以下步骤处理血清样品。
- 在进行 NMR 实验之前解冻样品。将 250 μL 等分试样的血清与 250 μL 磷酸盐水缓冲溶液(含有 100% D2O、0.9% NaCl、50 mM 磷酸钠缓冲液,pH 7.4)混合,以尽量减少 pH 值变化。
- 涡旋混合物几秒钟 (~15 s),以确保其均匀混合。最后,将制备好的样品转移到带有浓度为 0.05 mM 的丙酸三甲硅烷酯 (TSP) 的同轴插件的干净 NMR 管中,该插件用作校准和定量的外部标准品41。
注:作为参考,使用不与分析物相互作用的惰性和非反应性化合物。其他常用的参比化合物包括四甲基硅烷 (TMS) 和三甲基硅烷基丙磺酸钠 (DSS)。
3. 核磁共振实验
注:重点是鉴定血清样品中的小分子,因此,我们使用了抑制大分子信号的 Carr-Purcell-Meiboom-Gill (CPMG) 脉冲序列。本研究中的所有血清样品均使用 800 MHz NMR 波谱仪记录,该波谱仪配备低温冷却三共振 TCI 5 mm 宽带反向探头头和屏蔽 z 梯度。
- 将样品放入磁力架中。编写命令 wrpa (提及实验编号) 并按 Enter 键,以使用 cpmg 脉冲程序设置质子实验。通过单击选项提及患者的姓名和其他必要的详细信息 标题.
- 通过编写命令锁定来锁定磁场,按 Enter,然后进一步选择选项 90% H2O 和 10% D2O。通过 ATMM 手动调谐和匹配探头,以优化射频脉冲的效率并最大限度地提高灵敏度。通过单击 topshim 执行 1D 梯度填充。
- 设置以下采集参数,包括 5 s 的弛豫延迟、12 ppm 的频谱扫描宽度和 300 μs 的回波时间。所有这些参数都可以通过选择上部面板中的 Acquisition Parameters 选项轻松修改。
- 使用指数窗口函数应用 0.3 Hz 的线展宽,在 128 次扫描中将数据收集到 64,000 个点中。这些参数可以通过选择上部面板中的 Acquisition Parameters 选项进行类似修改。通过单击 Acquisition Parameters,将出现一个规格列表,并且可以在优化期间修改上述属性以及其他参数。
- 使用 NMR 处理和分析工具获取 NMR 波谱。获得最终光谱后,编写命令 apk 和 absn ,然后按 enter 键分别执行相位校正和基线校正。单击上面板上的 "校准轴" 选项,然后自动或手动将 TSP 峰校准为 0 ppm。
- 将采集的数据从系统传输到工作站,在那里进行进一步的处理和分析。
注意:实验阶段现已完成,可以暂停。可以方便地进行数据预处理、分析和解释。
4. 数据预处理
- 使用 NMR 处理和分析工具以及 NMR 代谢物定量软件进行数据预处理。
- 有时,只有自动校正是不够的,在这种情况下,手动校正也是必要的。要手动执行相位校正,请单击 NMR 处理和分析工具顶部菜单栏上的 "处理 "选项。然后单击 Adjust Phase(调整相位),拖动鼠标,并观察直到光谱经过相位校正。完成后,单击同一菜单栏中的 Save and Return 选项(以图标形式显示)。
- 要执行手动基线(Whittaker 方法)校正,请在 NMR 代谢物定量软件的处理器模块中打开光谱文件。选择选项 Baseline Correction。然后选择选项 Whittaker 方法。现在将点放在整个光谱中峰的底部(通常在血清的情况下为 0.0 至 10 ppm,或者根据光谱中代谢物的可用性)以调整碱基。校正基线后,所有光谱文件都已校正,请将它们保存在一个文件夹中。使用处理器或 NMR 代谢物定量软件的轮廓模块,可以使用以特定格式保存的相同文件进行进一步处理。
- 使用同一软件的 profiler 模块执行分箱。选择选项 工具 ,然后选择 Batch Process,然后选择 Spectral Binning 。
- 要生成用于统计分析的最终分箱表,请在单击光谱分箱后选择包含所有基线校正光谱文件的文件夹。
- 将光谱分成一定数量的等大小分箱,具有定义的光谱宽度,范围为 0.01 至 0.04 ppm(可根据研究进行优化)。此过程可简化和组织光谱数据,从而促进更一致的分析。指定存储段大小以及开始和结束 ppm 值,并指定将保存输出分箱表的文件夹。
- 排除与水和溶剂 TSP(4.8-5.2 和 0.0-0.7 ppm)相对应的化学位移区域,以防止光谱干扰 42,43,44。
- 此电子表格在一行中包含样品名称,在其他行中包含不同的分箱值以及 ppm。在使用此工作表进行统计分析之前,请对其进行修改,方法是将样本分组进行分析,并仅以逗号分隔 (CSV) 格式保存。
注意:例如,在比较轻度组和中度 ARDS 患者组时,我们使用轻度和中度标签排列数据,添加序列号,并将文件保存为 CSV 格式。
5. 统计分析
注意:从上一步获取的分箱表用作统计分析的输入文件。本研究采用代谢组学统计分析软件的统计分析(单因素)模块。
- 在统计分析之前,使用 sum、log 和 Pareto 执行标准化步骤,但它可能因不同的研究而异45。
- 执行单变量和多变量分析以区分不同的组并确定鉴定代谢物的准确性。
- 执行主成分分析 (PCA)、部分最小二乘判别分析 (PLS-DA) 和正交部分最小二乘判别分析 (OPLS-DA),以观察组间产生可变投影重要性 (VIP) 分数的组间差异导致代谢谱差异的每种代谢物。
- 执行单变量方法(如学生 t 检验和方差分析),以根据 p 值小于 0.05 的标准鉴定显著性代谢物。两种测试均产生箱须图,显示代谢物相对浓度的差异。
- 进行生物标志物分析,生成受试者工作特征曲线下面积 (AUROC) 图。AUC(曲线下面积)值> 0.8 被认为是显著的46。
- 使用标准 VIP 评分 >1、Bonferroni 校正的 p 值 <0.0544 和 AUC > 0.846 选择显著代谢物的最终列表。
- 如下所述鉴定代谢物。
- 分箱以 ppm 值而不是名称的形式提供重要的代谢物。使用生物磁共振组 (BMRB) 47和人类代谢组数据库 (HMDB) 等数据库识别与特定 ppm 值相对应的代谢物。
- 除此之外,使用以前发表的文献42,44 进行鉴定和验证。
- 使用 NMR 代谢物定量软件的 Profiler 模块确认鉴定的代谢物及其 ppm 值。
6. 使用 NMR 代谢物定量软件定量代谢物
注意:该软件的 Profiler 模块广泛用于量化已鉴定的代谢物。
- 在开始定量之前,使用处理器模块校准为研究选择的参比化合物(本研究中使用的 tsp)。单击选项 校准 CSI (化学形状指示剂)。提及参比化合物的浓度(本研究为 0.05 mM)。然后,将软件峰拖动到光谱参考峰上,并适当调整其高度和宽度。一旦光谱参考峰与软件的峰拟合,请单击 Accept(接受)。
- 在分析器模块中打开所需的谱图,并选择合适的化合物库进行研究。
- 在屏幕底部,将显示代谢物列表。从此列表中选择感兴趣的代谢物。选择后,所选代谢物的可能 ppm 将出现在光谱的顶角。
- 选择右上角的 Ppm 值 ,然后将光谱放大到代谢物的特定 ppm 刻度。
- 此时,屏幕上会出现两个峰值:一个代表记录的数据,另一个显示为虚线,代表软件峰值。对齐两个峰。拖动软件峰并调整其高度和位置以匹配样品中存在的峰。
- 正确对齐后,可以从该代谢物的浓度(以 mM 为单位)标题下显示的值获得代谢物的浓度(图 1)。
- 对所有代谢物和样品遵循相同的程序,并将定量值导出到电子表格中。要生成输出文件,请单击 批量作 选项下 工具 菜单。指定用于保存输出文件的所需文件夹。
7. 通路分析
注意:分析后鉴定的重要代谢物用于确定直接影响疾病组结果的主要途径。为此,通常采用代谢组学统计分析软件和 KEGG 数据库的途径分析模块。
- 将获得的重要代谢物列表输入到模块中。将生成一个包含路径名称的列表。
- 根据影响因子大于 0.1 的标准选择最终通路。显著影响值表明疾病相关途径30.
- 彻底分析产生的途径,以阐明与疾病严重程度相关的失调。这种方法的缺点之一是当重要代谢物小于 3 时,它在识别失调途径方面效率低下。
- 为了应对相同的情况,也可以使用先前发表的文献来确定已鉴定的代谢物及其与疾病严重程度的相关性。进行彻底的文献调查,并观察已鉴定的代谢物是否也遵循与您的兴趣相同的趋势。
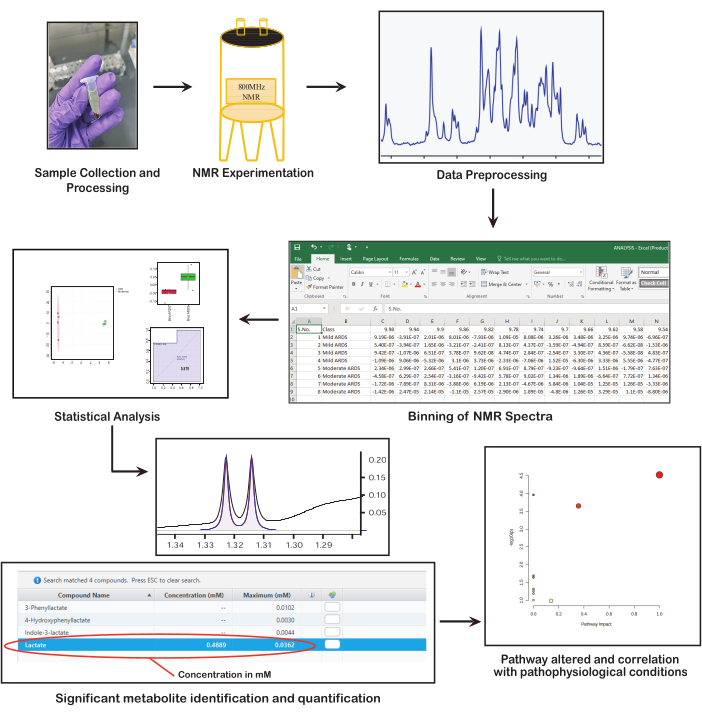
图 1:基于 NMR 的代谢组学的基本步骤。 该图介绍了基于 NMR 的代谢组学的关键步骤:收集和制备样品、进行 NMR 波谱分析、预处理数据、进行统计分析、识别和量化代谢物以及解释生物学意义。 请单击此处查看此图的较大版本。
结果
要进行代谢组学研究,必须确定样本量和将要分析的特定群体。选择适当的样本量对于获得与疾病严重程度准确相关的显着结果至关重要48。然而,在这项特定的工作中,我们使用了小样本量来演示使用基于 NMR 的代谢组学鉴定和定量代谢物所涉及的步骤,这主要是为了参考。在这项研究中,我们检查了 ARDS 轻中度患者在入住 ICU 第 1 天的代谢差异,共包...
讨论
代谢组学可有效识别和量化代谢物,靶向疾病期间变得紊乱的代谢周期。结果的质量取决于代谢组学方法中每个步骤的细致执行。从样本选择和收集到通路鉴定,每个阶段对于准确识别导致疾病的主要因素都至关重要。在进行代谢组学之前,对文献进行彻底回顾是必不可少的,并且必须在每个阶段都仔细注意。
在设计代谢组学研究时,样品的选择对?...
披露声明
作者声明没有竞争性的经济利益。
致谢
AS 感谢科学与创新研究院 (AcSIR) 的注册(注册号 10BB22A71002)。AS 还感谢国防研究与发展组织 (DRDO) 的奖学金。我们感谢生物医学研究中心 (CBMR) 通过校内项目 (CBMR/IMR/0008/2021) 提供 800 MHz NMR 波谱仪设施和资金。我们还感谢重症监护医学科 (CCM) SGPGIMS 的持续支持。我们感谢许多护士的帮助,最重要的是,感谢参加这项研究的患者。这项研究由生物医学研究中心 (CBMR) 的校内项目 (CBMR/IMR/0008/2021) 和校外项目 (No.国防研究与发展组织 (DRDO) 的 LSRB/01/15001/LSRB-404/PEE&BS/2023)。
材料
| Name | Company | Catalog Number | Comments |
| Centirfuge | Sigma aldrich | 3-18KS | |
| Chenomx NMR suite | NMR Suite, v9, Chenomx Inc., Edmonton, Canada | NMR metabolite quantification software | |
| Co-axial insert | Sigma aldrich | Z278513 | |
| Deuterim oxide | Sigma aldrich | 151882 | |
| Eppendorf tubes | Tarsons | 500020 | |
| Metaboanalyst | Wishart Research Group | Metabolomics statistical analysis software | |
| NMR tube | Wilmad | Z412007 | 5mm diameter |
| Pipette | Eppendorf research plus | 3123000039 | 0-100 μl |
| Sample collection vials | Tarsons cryo chill vials | 523194 | |
| Sodium azide | Sigma aldrich | S2002 | |
| Sodium chloride crystal | Sigma aldrich | S9625 | |
| Sodium phosphate dibasic | Sigma aldrich | 567550 | |
| Sodium phosphate monobasic | Sigma aldrich | S0751 | |
| Topspin 3.6.4 | Bruker | NMR processing and analysis tool | |
| Tsp salt | Sigma aldrich | 269913 |
参考文献
- Rubenfeld, G. D. Confronting the frustrations of negative clinical trials in acute respiratory distress syndrome. Ann. Am. Thorac. Soc. 12 (Supplement 1), S58-S63 (2015).
- Ware, L. B., et al. Prognostic and pathogenetic value of combining clinical and biochemical indices in patients with acute lung injury. Chest. 137 (2), 288-296 (2010).
- Johnson, C. H., Julijana, I., Gary, S. Metabolomics: beyond biomarkers and towards mechanisms. Nat Rev Mol Cell Biol. 17 (7), 451-459 (2016).
- Patti, G. J., Oscar, Y., Gary, S. Metabolomics: the apogee of the omics trilogy. Nat Rev Mol Cell Biol. 13 (4), 263-269 (2012).
- Nicholson, J. K., John, C. L. Metabonomics. Nature. 455 (7216), 1054-1056 (2008).
- Schnackenberg, L. K., Beger, R. D. Monitoring the health to disease continuum with global metabolic profiling and systems biology. Pharmacogenomics. 7 (7), 1077-1086 (2006).
- Pandey, S., Siddiqui, M. A., Azim, A., Sinha, N. Metabolic fingerprint of patients showing responsiveness to treatment of septic shock in intensive care unit. Magn Reson Mater Phys Biol Med. 36 (4), 659-669 (2023).
- Mamas, M., Dunn, W. B., Neyses, L., Goodacre, R. The role of metabolites and metabolomics in clinically applicable biomarkers of disease. Arch. Toxicol. 85, 5-17 (2011).
- Dunn, W. B., Broadhurst, D. I., Atherton, H. J., Goodacre, R., Griffin, J. L. Systems level studies of mammalian metabolomes: the roles of mass spectrometry and nuclear magnetic resonance spectroscopy. Chem Soc Rev. 40 (1), 387-426 (2011).
- Dettmer, K., Bruce, D. H. Metabolomics--a new exciting field within the" omics" sciences. Environ Health Perspect. 112 (7), A396-A397 (2004).
- Goodacre, R., Vaidyanathan, S., Dunn, W. B., Harrigan, G. G., Kell, D. B. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 22 (5), 245-252 (2004).
- Lindon, J. C., Nicholson, J. K., Holmes, E., Everett, J. R. Metabonomics: metabolic processes studied by NMR spectroscopy of biofluids. Concepts Magn Reson Educ J. 12 (5), 289-320 (2000).
- Bernini, P., et al. Individual human phenotypes in metabolic space and time. J Proteome Res. 8 (9), 4264-4271 (2009).
- Nicholson, J. K., et al. Metabolic phenotyping in clinical and surgical environments. Nature. 491 (7424), 384-392 (2012).
- Vinayavekhin, N., Homan, E. A., Saghatelian, A. Exploring disease through metabolomics. ACS Chem Biol. 5 (1), 91-103 (2010).
- Dunn, W. B., Ellis, D. I. Metabolomics: current analytical platforms and methodologies. TrAC Trends Anal Chem. 24 (4), 285-294 (2005).
- Dunn, W. B., Bailey, N. J. C., Johnson, H. E. Measuring the metabolome: current analytical technologies. Analyst. 130 (5), 606-625 (2005).
- Bingol, K. Recent advances in targeted and untargeted metabolomics by NMR and MS/NMR methods. High-throughput. 7 (2), 9 (2018).
- Emwas, A. H. M. The strengths and weaknesses of NMR spectroscopy and mass spectrometry with particular focus on metabolomics research. Methods Mol Biol. 1277, 161-193 (2015).
- Markley, J. L., et al. The future of NMR-based metabolomics. Curr Opin Biotechnol. 43, 34-40 (2017).
- Marchand, J., Martineau, E., Guitton, Y., Dervilly-Pinel, G., Giraudeau, P. Multidimensional NMR approaches towards highly resolved, sensitive and high-throughput quantitative metabolomics. Curr Opin Biotechnol. 43, 49-55 (2017).
- Eghbalnia, H. R., et al. Increasing rigor in NMR-based metabolomics through validated and open source tools. Curr Opin Biotechnol. 43, 56-61 (2017).
- Vignoli, A., et al. High-throughput metabolomics by 1D NMR. Angew Chem Int Ed. 58 (4), 968-994 (2019).
- Sundekilde, U. K., Eggers, N., Bertram, H. C. NMR-based metabolomics of food. Methods Mol Biol. 2037, 335-344 (2019).
- Garcia-Perez, I., et al. Identifying unknown metabolites using NMR-based metabolic profiling techniques. Nat Protoc. , 1-30 (2020).
- Dona, A. C., et al. A guide to the identification of metabolites in NMR-based metabonomics/metabolomics experiments. Comput Struct Biotechnol J. 14, 135-153 (2016).
- Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N., Siddiqui, M. H. Metabolomics: an emerging potential approach to decipher critical illnesses. Biophys Chem. 267, 106462 (2020).
- McNicholas, B. A., et al. Impact of early acute kidney injury on management and outcome in patients with acute respiratory distress syndrome: a secondary analysis of a multicenter observational study. Crit. Care Med. 47 (9), 1216-1225 (2019).
- Ambruso, S. L., et al. Inter-organ communication in homeostasis and disease: Lung metabolomics after ischemic acute kidney injury reveals increased oxidative stress, altered energy production, and ATP depletion. Am J Physiol Lung Cell Mol Physiol. 321 (1), L50 (2021).
- Viswan, A., et al. Metabolomics based predictive biomarker model of ARDS: A systemic measure of clinical hypoxemia. PloS One. 12 (11), e0187545 (2017).
- Metwaly, S. M., Brent, W. W. Systems biology ARDS research with a focus on metabolomics. Metabolites. 10 (5), 207 (2020).
- Stringer, K. A., et al. Metabolic consequences of sepsis-induced acute lung injury revealed by plasma 1H-nuclear magnetic resonance quantitative metabolomics and computational analysis. Am J Physiol Lung Cell Mol Physiol. 300 (1), L4-L11 (2011).
- Singh, A., Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N. Unveiling Pathophysiological Insights: Serum Metabolic Dysregulation in Acute Respiratory Distress Syndrome Patients with Acute Kidney Injury. J Proteome Res. 23 (10), 4216-4228 (2024).
- Slupsky, C. M. NMR-based analysis of metabolites in urine provides rapid diagnosis and etiology of pneumonia. Biomark Med. 4 (2), 195-197 (2010).
- Siddiqui, M. A., et al. NMR spectroscopy-based analysis of gallstones of cancerous and benign gallbladders from different geographical regions of the Indian subcontinent. Plos One. 18 (6), e0286979 (2023).
- Li, J., et al. 1HNMR-based metabolomic profile of rats with experimental acute pancreatitis. BMC Gastroenterol. 14, 1-7 (2014).
- Mao, H., et al. Systemic metabolic changes of traumatic critically ill patients revealed by an NMR-based metabonomic approach. J Proteome Res. 8 (12), 5423-5430 (2009).
- Stringer, K. A., Jones, A. E., Puskatich, M. A., Karnovsky, A., Serkova, N. J. 1H-nuclear magnetic resonance (NMR)-detected lipids associated with apoptosis differentiate early acute respiratory distress syndrome (ARDS) from sepsis. C63. LUNG INJURY AND REPAIR: TWO TO TANGO. Am J Resp Crit Care Med. 189, A5000 (2014).
- Viswan, A., Singh, C., Kayastha, A. M., Azim, A., Sinha, N. An NMR based panorama of the heterogeneous biology of acute respiratory distress syndrome (ARDS) from the standpoint of metabolic biomarkers. NMR Biomed. 33 (2), e4192 (2020).
- Singh, C., et al. Metabolic profiling of human lung injury by 1 H high-resolution nuclear magnetic resonance spectroscopy of blood serum. Metabolomics. 11, 166-174 (2015).
- Pandey, S., Siddiqui, M. A., Trigun, S. K., Azim, A., Sinha, N. Gender-specific association of oxidative stress and immune response in septic shock mortality using NMR-based metabolomics. Mol Omics. 18 (2), 143-153 (2022).
- Nagana, G. A., Gowda, Y. N., Raftery, D. Expanding the limits of human blood metabolite quantitation using NMR spectroscopy. Anal Chem. 87 (1), 706-715 (2015).
- Gowda, G. A. N., Raftery, D. Quantitating metabolites in protein precipitated serum using NMR spectroscopy. Anal Chem. 86 (11), 5433-5440 (2014).
- Gowda, G. A. N., Raftery, D. NMR-based metabolomics. Adv Exp Med Biol. 1280, 19-37 (2021).
- Xia, J., Psychogios, N., Young, N., Wishart, D. S. MetaboAnalyst: a web server for metabolomic data analysis and interpretation. Nucleic Acids Res. 37 (suppl_2), W652-W660 (2009).
- orbacıoğlu, &. #. 3. 5. 0. ;. K., Aksel, G. Receiver operating characteristic curve analysis in diagnostic accuracy studies: A guide to interpreting the area under the curve value. Turk J Emerg Med. 23 (4), 195 (2023).
- Ulrich, E. L., et al. BioMagResBank. Nucleic Acids Res. 36 (suppl_1), D402-D408 (2007).
- Nyamundanda, G., Gormley, I. C., Fan, Y., Gallagher, W. M., Brennan, L. MetSizeR: selecting the optimal sample size for metabolomic studies using an analysis based approach. BMC bioinformatics. 14, 1-8 (2013).
- Beckonert, O., et al. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts. Nat Protoc. 2 (11), 2692 (2007).
- Dona, A. C., et al. Precision high-throughput proton NMR spectroscopy of human urine, serum, and plasma for large-scale metabolic phenotyping. Anal Chem. 86 (19), 9887-9894 (2014).
- Ala-Korpela, M. Potential role of body fluid 1H NMR metabonomics as a prognostic and diagnostic tool. Expert Rev Mol Diagn. 7 (6), 761-773 (2007).
- Takeda, I., et al. Understanding the human salivary metabolome. NMR Biomed. 22 (6), 577-584 (2009).
- Callejon-Leblic, B. e. m., García-Barrera, T., Pereira-Vega, A., Gómez-Ariza, J. L. Metabolomic study of serum, urine and bronchoalveolar lavage fluid based on gas chromatography mass spectrometry to delve into the pathology of lung cancer. J Pharm Biomed Anal. 163, 122-129 (2019).
- Van Oort, P. M. P., et al. Exhaled breath metabolomics for the diagnosis of pneumonia in intubated and mechanically-ventilated intensive care unit (ICU)-patients. Int J Mol Sci. 18 (2), 449 (2017).
- Serkova, N. J., Niemann, C. U. Pattern recognition and biomarker validation using quantitative 1H-NMR-based metabolomics. Expert Rev Mol Diagn. 6 (5), 717-731 (2006).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation times. Rev Sci Instrum. 29 (8), 688-691 (1958).
- Xi, Y., Rocke, D. M. Baseline correction for NMR spectroscopic metabolomics data analysis. BMC bioinformatics. 9, 1-10 (2008).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。