JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
NMR 기반 대사체학을 사용한 중증 환자의 정신 나간 대사 산물의 식별 및 정량화
요약
핵자기공명(NMR) 분광법은 다양한 질병을 앓고 있는 환자의 대사 산물에서 조절 장애를 식별하는 데 사용됩니다. 이 기술을 사용하면 정신 나간 대사 산물을 정량화하여 병태 생리학적 통찰력을 얻을 수 있습니다. 여기에서는 환자의 대사 특성화를 위한 NMR 기반 접근법의 단계별 절차를 설명합니다.
초록
대사체학(Metabolomics)은 병태생리학적 상태에 대한 개인의 반응을 반영하는 중요한 접근 방식으로 부상하고 있습니다. 핵자기공명(NMR) 분광법은 급성 호흡곤란 증후군(ARDS), 중증 급성 췌장염(SAP), 급성 신장 손상(AKI) 및 패혈증과 같은 질환을 앓고 있는 중환자의 대사 조절 장애를 식별하는 도구로 발전했습니다. 연구 및 대조군의 혈청 샘플의 스펙트럼 데이터는 800MHz NMR 분광계를 사용하여 기록되고 NMR 처리 및 분석 도구를 사용하여 처리됩니다. 또한 일변량 및 다변량 테스트와 같은 엄격한 통계 분석을 수행하여 중요한 대사 산물을 정확히 찾아낸 다음 NMR 대사 산물 정량화 소프트웨어를 사용하여 정확하게 식별하고 정량화합니다. 또한 경로 분석은 질병의 심각성을 초래하는 혼란스러운 생화학적 순환을 강조합니다. 이 포괄적인 접근 방식을 통해 연구원들은 이러한 중요한 질병과 관련된 대사 변화에 대한 더 깊은 통찰력을 얻는 것을 목표로 하며, 잠재적으로 질병에 대한 더 나은 이해와 개선된 진단 및 치료 전략을 위한 길을 닦을 수 있습니다.
서문
전 세계적으로 효율적인 질병 진단을 제공하기 위한 지속적인 노력에도 불구하고 표적 치료는 여전히 진정한 잠재력을 달성하지 못했습니다. transcriptomics, proteomics(단백질체학) 등과 같은 다양한 접근법을 통해 여러 바이오마커를 확인할 수 있었지만, 민감도와 특이도가 부족하여 임상적 유용성이 충분하지 않았습니다 1,2. 표적 치료는 일부 다인자 질환에서 큰 도전 과제이며, 결국 더 높은 사망률로 이어집니다. 광범위한 이질성을 가지고 존재하는 복잡한 질병의 기저에 깔린 기전과 병태생리학에 대한 더 나은 이해가 필요합니다. 이와 관련하여 대사체학의 진화는 치료제 개발에 혁명을 일으켰으며, 이는 궁극적으로 급성 호흡곤란 증후군(ARDS), 패혈증 및 중증 급성 췌장염(SAP)과 같은 다양한 중요한 질병에 대한 치료 요법을 맞춤화하는 데 도움이 될 수 있습니다.
대사체학(Metabolomics)은 다양한 생체 유체, 세포 또는 조직 추출물에 걸쳐 저분자량 분자(아미노산, 지질, 펩타이드, 유기산 및 비타민과 같은 대사 산물)를 식별하고 정량화하는 것을 목표로 하는 포괄적인 접근 방식입니다. 일반적으로 무게가 1500Da 미만인 이러한 대사 산물은 생화학적 과정에서 적극적인 역할을 하며 유기체의 생물학적 상태의 점진적인 윤곽을 반영합니다. 여기에는 주요 효소 과정을 위한 기질, 생물학적 경로의 중간체, 세포 대사의 부산물이 포함됩니다. 결과적으로 대사체학은 식이 영향, 약물 상호 작용 및 질병 상태에 대한 상세한 지문을 포착합니다. 대사산물 변화는 대사 및 생물학적 경로에 대한 매우 민감한 지표로, 표현형 발현 및 그에 따른 병태생리학적 이상과의 상관관계를 허용합니다 3,4. 대사 산물의 초기 변화는 질병의 중증도를 나타내는 초기 지표가 될 수 있으며, 일시적인 변화는 치료 효능, 질병 진행 및 임상 결과를 모니터링하는 데 도움이 될 수 있습니다 5,6,7. 따라서 대사체학은 임상적, 생리학적, 생화학적 종말을 통해 질병을 재정의함으로써 임상 분석 및 기타 다양한 오믹스 접근법을 향상시킵니다 8,9,10,11,12,13. 대사체학의 분석 능력은 변화된 대사 산물 농도를 통해 질병 감수성을 모니터링하고 결정하는 데 사용됩니다14,15.
이러한 맥락에서 질량 분석법(MS)과 핵 자기 공명(NMR) 분광법은 모두 생물학적 샘플에서 대사 산물 프로파일링을 위한 기본 분석 플랫폼으로 부상했습니다. 이러한 방법은 대사 산물 16,17,18의 표적 및 비표적 식별 및 정량화에 모두 사용됩니다. 각 플랫폼에는 장점과 한계가 있지만 NMR의 비파괴 특성으로 인해 다양한 생체 내 연구 및 특히 대사체학 연구의 초기 단계에서 미확인 화합물의 구조 특성화에 적합합니다. MS 전에 필요한 시료 분획, 유도체화 및 이온화는 바이어스를 도입하고 종종 시료 손실을 초래하여 NMR 분광법이 최소한의 시료 전처리 또는 시료 전처리 없이 캡처할 수 있는 동적 기능에 영향을 미칠 수 있습니다. NMR의 주요 한계는 MS에 비해 감도가 낮다는 것인데, 이는 검출 한계가 낮아 덜 풍부한 대사 산물을 검출하기 어렵게 만듭니다19. 그러나 고해상도 초전도 자석, 극저온 냉각 NMR 프로브 및 감도를 향상시키는 기술과 같은 발전으로 이러한 제한이 완화되었습니다 20,21,22. 유전체학 및 단백질체학에 대한 보완적인 접근 방식으로, NMR 분광법을 사용한 대사 프로파일링이 선호되는 기술로 주목받고 있습니다 23,24,25. NMR의 최소한의 샘플 준비, 재현성 및 반복성은 감도 문제에도 불구하고 대사 산물의 고유한 동적 특징을 포착하는 데 유용한 도구입니다26.
몇몇 연구 그룹은 ARDS 28,29,30,31,32,33, 폐렴34, 패혈증 7, 담석35, 췌장염36과 같은 다양한 질병27에 대한 환자의 대사 조절 장애 프로필을 정확히 찾아내어 대사체학을 성공적으로 수행했습니다. 중환자에 대한 NMR 기반 대사체학 연구는 전신 염증 반응 증후군(SIRS)에서 중환자실(ICU) 사망률 및 이환율의 주요 원인인 다발성 장기 기능 장애 증후군(MODS)으로의 진행을 추적하는 데 중요한 역할을 했습니다37. Stringer 등의 연구에서, 혈장 샘플은 대조군 환자와 비교하여 패혈증으로 인한 급성 폐 손상(ALI) 환자의 대사 변화를 조사하는 데 사용되었습니다38. 이 파일럿 연구에서 높은 것으로 밝혀진 주요 대사 산물은 관련된 대사 경로와 임상 점수와의 연관성을 반영했습니다. 이 연구는 ARDS에서 폐 손상 메커니즘의 초기 단계와 패혈증을 구별하기 위해 혈청 대사체학으로 확장되었습니다32. 또한, ARDS에 실린 또 다른 연구에서는 급성 폐 손상/ARDS와 건강한 대조군을 뚜렷하게 구분하는 강력한 혈청 바이오마커를 확인하여, 급성 폐 손상의 발병에 해당하는 전신 대사 변화에 대한 통찰력을 제공한다39,40.
NMR 분광법은 기저 병태생리학을 밝히는 대사 지문에 대한 강력하고 편견 없는 정보를 제공하는 고처리량 및 자동화된 분석 기술입니다38. NMR 데이터의 임상 적용 및 생물학적 해석은 풍부한 정보를 포함하는 고품질 스펙트럼을 얻는 데 달려 있습니다. 따라서 정확하고 균일하며 잘 공식화된 데이터 수집, 처리 및 분석을 보장하는 것이 중요합니다. 따라서 이 연구의 목적은 대사산물의 식별 및 정량화를 위해 NMR 기반 대사체학의 필수 단계를 활용하는 것입니다. 이 연구는 적절한 시료 선택, 채취 및 보관, 시료 처리 및 준비, 데이터 수집 및 분석, 관심 대사 산물 식별 및 정량화, 궁극적으로 관련 통찰력을 도출하기 위한 임상 맥락에서 결과 해석과 같은 임상 대사체학 연구(그림 1)에 필요한 프로토콜의 주요 단계를 강조합니다. 이러한 각 단계는 대사체학에서 NMR 분광법을 활용하여 중요한 생물학적 및 임상적 통찰력을 발견하는 데 필수적입니다.
프로토콜
윤리적 승인(IEC 코드: 2022-71-PhD-126)은 러크나우에 있는 SGPGIMS(Sanjay Gandhi Postgraduate Institute of Medical Sciences)의 IEC에서 획득했습니다. 연구를 수행하고 연구 목적으로 데이터를 게시하기 위해 환자 또는 그 친척으로부터 서면 및 정보에 입각한 동의를 받았습니다. 또한, 연구는 기관의 지침에 따라 수행되었습니다.
1. 디자인 및 윤리적 정리 공부
- 각 그룹의 표본 크기를 결정합니다. 참가자에 대한 선택 기준을 주의 깊게 검토하고 설정합니다. 또한 연구 설계에 인간 또는 동물 샘플이 포함되어야 하는 경우 샘플 수집을 시작하기 전에 해당 기관 윤리 위원회(IEC)로부터 윤리적 허가를 받으십시오.
- 동맥혈 내 산소의 분압(PaO2)과 흡기산소분율(FiO2)(P/F) 비율에 기초한 Berlin 2012 정의의 진단 기준에 따라 경증, 중등도 및 중증으로 분류된 ARDS 환자를 선택합니다. P/F 범위가 300-200인 ARDS 환자는 경증, 200-100은 중등도, 100 미만은 중증으로 간주됩니다38. 여기에서는 경증 및 중등도 ARDS 환자의 두 그룹에 초점을 맞췄습니다( 표 1 참조).
참고: 이 연구는 NMR 기반 대사체학을 수행하기 위해 혈액 샘플에서 얻은 혈청을 사용하는 것을 포함합니다. 이 연구에서 참고용으로 포함된 참가자의 평균 연령은 42세(± 10.9세)였으며 성별 분포는 남성 2명, 여성 6명이었습니다.
| 범주 | P/F 비율 |
| 심하지 않음 | 300-200 |
| 온화한 | 200-100 |
| 아주 심함 | 100-0 |
표 1: ARDS 환자의 분류.
2. 시료 선별, 채취 및 처리
- 아래 설명된 단계에 따라 혈청 분리를 수행합니다.
- 실험을 위해 일반 바이알(첨가제 없음) 및 멸균 바이알에 멸균 바늘을 사용하여 동맥에서 2mL의 혈액 샘플을 수집합니다. 샘플을 실온에서 30분 동안 응고시키도록 둡니다. 본 연구에서는 경증 ARDS로 진단받은 환자 4명과 중등도 ARDS를 진단받은 환자 4명을 선정했다.
- 3100 x g 에서 15분 동안 샘플을 원심분리하여 혈청25를 분리합니다. 혈청을 멸균 마이크로 원심분리 튜브로 옮겨 더 작은 부피(즉, 400-500 μL)의 부분 표본을 만들고, 적절하게 라벨을 붙이고(환자 이름, CR 번호, 요일 지점 등), 향후 분석을 위해 -80 °C에서 보관합니다.
참고: 이 시점에서 편리한 시간에 실험을 일시 중지하고 다시 시작할 수 있습니다.
- 다음 단계를 사용하여 NMR 실험 전에 혈청 샘플을 처리합니다.
- NMR 실험을 수행하기 전에 샘플을 해동하십시오. pH 변화를 최소화하기 위해 250μL의 혈청 분취액과 250μL의 식염수 인산염수 완충액(100%D2O, 0.9% NaCl, 50mM 인산나트륨 완충액, pH 7.4 함유)을 혼합합니다.
- 혼합물이 균일하게 혼합되도록 몇 초(~15초) 동안 혼합물을 소용돌이치십시오. 마지막으로, 준비된 샘플을 0.05mM 농도의 트리메틸실릴 프로피오네이트(TSP)가 포함된 동축 인서트가 있는 깨끗한 NMR 튜브로 옮기며, 이는 보정 및 정량화를 위한 외부 표준물질역할을 합니다 41.
참고: 참고로, 분석물과 상호 작용하지 않는 불활성 및 비반응성 화합물이 사용됩니다. 일반적으로 사용되는 다른 참조 화합물로는 테트라메틸실란(TMS) 및 트리메틸실릴프로판설포네이트나트륨(DSS)이 있습니다.
3. NMR 실험
참고: 혈청 샘플에서 작은 분자를 식별하는 데 중점을 두고 있으므로 거대분자 신호를 억제하는 CPMG(Carr-Purcell-Meiboom-Gill) 펄스 시퀀스를 사용했습니다. 이 연구의 모든 혈청 샘플은 극저온 냉각 삼중 공명 TCI 5mm 광대역 역 프로브 헤드 및 차폐 z-gradient가 장착된 800MHz NMR 분광계를 사용하여 기록되었습니다.
- 샘플을 자석에 넣습니다. wrpa(실험 번호 언급) 명령을 작성하고 Enter 키를 눌러 cpmg 펄스 프로그램으로 양성자 실험을 설정합니다. 제목 옵션을 클릭하여 환자의 이름과 필요한 기타 세부 정보를 언급하십시오.
- Lock 명령을 작성 하여 자기장을 잠그고 Enter 키를 누른 다음 90% H2O 및 10% D2O 옵션을 추가로 선택합니다. ATMM을 통해 프로브를 수동으로 조정 및 일치시켜 무선 주파수 펄스의 효율성을 최적화하고 감도를 최대화합니다. topshim을 클릭하여 1D 그라디언트 shimming을 수행합니다.
- 5초의 완화 지연, 12ppm의 스펙트럼 스윕 폭, 300μs의 에코 시간을 포함하여 다음 획득 파라미터를 설정합니다. 이러한 모든 매개변수는 상단 패널에서 Acquisition Parameters 옵션을 선택하여 쉽게 수정할 수 있습니다.
- 지수 윈도우 기능을 사용하여 0.3Hz의 라인 확장을 적용하고 128회 스캔을 통해 64,000포인트로 데이터를 수집합니다. 이러한 매개변수는 상단 패널에서 Acquisition Parameters 옵션을 선택하여 유사하게 수정할 수 있습니다. Acquisition Parameters를 클릭하면 사양 목록이 나타나고 언급된 속성은 다른 매개변수와 함께 최적화 중에 수정할 수 있습니다.
- NMR 처리 및 분석 도구를 사용하여 NMR 스펙트럼을 획득합니다. 최종 스펙트럼을 얻었으면 apk 와 absn 명령을 작성하고 enter를 눌러 위상 보정과 기준선 보정을 각각 수행합니다. 상단 패널에 있는 Calibrate Axis 옵션을 클릭한 다음 TSP 피크를 자동 또는 수동으로 0ppm으로 교정합니다.
- 수집된 데이터를 시스템에서 워크스테이션으로 전송하여 추가 처리 및 분석이 수행됩니다.
참고: 이제 실험 단계가 완료되었으며 일시 중지할 수 있습니다. 데이터 전처리, 분석, 해석을 편리하게 수행할 수 있습니다.
4. 데이터 전처리
- NMR 처리 및 분석 도구와 NMR 대사 산물 정량화 소프트웨어를 사용하여 데이터 전처리를 수행합니다.
- 때로는 자동 수정만으로는 충분하지 않으며, 이 경우 수동 수정도 필요합니다. 상 보정을 수동으로 수행하려면 NMR 처리 및 분석 도구의 상단 메뉴 표시줄에서 프로세스 옵션을 클릭하십시오. 그런 다음 Adjust Phase를 클릭하고 마우스를 드래그한 다음 스펙트럼의 위상이 보정될 때까지 관찰합니다. 완료되면 동일한 메뉴 표시줄에서 사용할 수 있는 Save and Return 옵션(아이콘 형태로 표시)을 클릭합니다.
- 수동 베이스라인(Whittaker 방법) 보정을 수행하려면 NMR 대사 산물 정량화 소프트웨어의 프로세서 모듈에서 스펙트럼 파일을 엽니다. 기준선 수정 옵션을 선택합니다. 그런 다음 Whittaker 방법 옵션을 선택합니다. 이제 전체 스펙트럼(일반적으로 혈청의 경우 또는 스펙트럼에서 대사 산물의 가용성에 따라 0.0-10ppm)에서 피크의 기저부에 점을 배치하여 염기를 조정합니다. 모든 스펙트럼 파일이 기준선을 수정한 후 단일 폴더에 저장합니다. NMR 대사산물 정량화 소프트웨어의 프로세서 또는 프로파일러 모듈을 사용하여 추가 처리는 특정 형식으로 저장된 동일한 파일을 사용하여 수행될 수 있습니다.
- 동일한 소프트웨어의 프로파일러 모듈을 사용하여 비닝을 수행합니다. Tools(도구), Batch Process(배치 처리), Spectral Binning(스펙트럼 비닝) 옵션을 차례로 선택합니다.
- 통계 분석에 사용할 최종 비닝 시트를 생성하려면 스펙트럼 비닝을 클릭한 후 베이스라인이 보정된 모든 스펙트럼 파일이 포함된 폴더를 선택합니다.
- 스펙트럼을 0.01에서 0.04ppm 범위의 정의된 스펙트럼 너비를 가진 동일한 크기의 bin으로 설정합니다(연구에 따라 최적화할 수 있음). 이 프로세스는 스펙트럼 데이터를 단순화하고 구성하여 보다 일관된 분석을 용이하게 합니다. 시작 및 종료 ppm 값과 함께 버킷 크기를 지정하고 출력 binning sheet가 저장될 폴더를 지정합니다.
- 스펙트럼 간섭 42,43,44를 방지하기 위해 물과 용매 TSP(4.8-5.2 및 0.0-0.7ppm)에 해당하는 화학적 이동 영역을 제외합니다.
- 이 스프레드시트에는 한 행에는 샘플 이름이 포함되어 있고 다른 행에는 ppm과 함께 다른 bin 값이 포함되어 있습니다. 통계 분석에 이 시트를 사용하기 전에 분석을 위해 샘플을 그룹으로 구성하고 쉼표로 구분된(CSV) 형식으로만 저장하여 수정하십시오.
참고: 예를 들어, 경증 그룹과 중등도 그룹의 ARDS 환자를 비교할 때 경증 및 중등도 레이블로 데이터를 정렬하고 일련 번호를 추가한 다음 파일을 CSV 형식으로 저장했습니다.
5. 통계 분석
참고: 이전 단계에서 얻은 비닝 시트는 통계 분석을 위한 입력 파일로 사용됩니다. 본 연구에서는 대사체학 통계 분석 소프트웨어의 통계분석(one factor) 모듈을 사용하였다.
- 통계 분석 전에 sum, log 및 Pareto를 사용하여 정규화 단계를 수행하지만 이는 연구마다 다를 수 있습니다45.
- 일변량 및 다변량 분석을 수행하여 서로 다른 그룹을 구별하고 식별된 대사 산물의 정확도를 확인합니다.
- 주성분 분석(PCA), 부분 최소 제곱 판별 분석(PLS-DA) 및 직교 부분 최소 제곱 판별 분석(OPLS-DA)을 수행하여 대사 프로필의 차이를 담당하는 모든 대사 산물에 대해 VIP(Variable Importance of Projection) 점수를 산출하는 그룹 간의 구별을 관찰합니다.
- 학생용 t-검정 및 ANOVA와 같은 일변량 방법을 수행하여 p-값이 0.05 미만인 기준에 따라 유의한 대사 산물을 식별합니다. 두 테스트 모두 대사 산물의 상대 농도 차이를 보여주는 상자 수염 플롯을 생성합니다.
- AUROC(Receiver Operating Characteristic Curve) 그래프 아래 영역을 산출하는 바이오마커 분석을 수행합니다. AUC(곡선 아래 영역) 값 > 0.8은 유의미한것으로 간주됩니다 46.
- VIP score>1, Bonferroni-corrected p-value<0.0544 및 AUC > 0.846 기준을 사용하여 유의한 대사 산물의 최종 목록을 선택합니다.
- 아래 설명된 대로 대사 산물을 식별합니다.
- Binning은 이름보다는 ppm 값의 형태로 중요한 대사 산물을 제공합니다. BMRB(Biological Magnetic Resonance Bank)47및 HMDB(Human Metabolome Database)와 같은 데이터베이스를 사용하여 특정 ppm 값에 해당하는 대사 산물을 식별합니다.
- 이 외에, 식별 및 검증을 위해 이전에 출판된 문헌42,44를 사용한다.
- NMR 대사산물 정량화 소프트웨어의 프로파일러 모듈을 사용하여 식별된 대사산물을 ppm 값으로 확인합니다.
6. NMR 대사 산물 정량화 소프트웨어를 사용한 대사 산물의 정량화
참고: 소프트웨어의 프로파일러 모듈은 식별된 대사 산물을 정량화하는 데 널리 사용됩니다.
- 정량화를 시작하기 전에 프로세서 모듈을 사용하여 연구를 위해 선택된 참조 화합물(이 연구에 사용된 tsp)을 보정합니다. Calibrate CSI (chemical shape indicator) 옵션을 클릭하십시오. 기준 화합물의 농도(이 연구의 경우 0.05mM)를 언급합니다. 그런 다음, 소프트웨어 피크를 스펙트럼 기준 피크로 끌어서 놓고 높이와 너비를 적절하게 조정합니다. 스펙트럼 참조 피크가 소프트웨어의 피크에 맞으면 Accept를 클릭합니다.
- 프로파일러 모듈에서 원하는 스펙트럼을 열고 연구에 적합한 화합물 라이브러리를 선택합니다.
- 화면 아래쪽에 대사 산물 목록이 표시됩니다. 이 목록에서 관심 있는 대사 산물을 선택합니다. 선택하면 선택한 대사 산물의 가능한 ppm이 스펙트럼의 상단 모서리에 나타납니다.
- 상단 모서리에서 Ppm 값을 선택하고 스펙트럼을 대사 산물의 특정 ppm 스케일로 확대합니다.
- 이 시점에서 두 개의 피크가 화면에 나타나는데, 하나는 기록된 데이터를 나타내고 다른 하나는 소프트웨어 피크를 나타내는 점선으로 표시됩니다. 두 피크를 정렬합니다. 소프트웨어 피크를 끌어서 샘플에 있는 피크와 일치하도록 피크의 높이와 위치를 조정합니다.
- 적절하게 정렬되면 대사 산물의 농도는 해당 대사 산물에 대한 mM 단위의 표제 농도 아래에 표시된 값에서 얻을 수 있습니다(그림 1).
- 모든 대사 산물 및 샘플에 대해 동일한 절차를 따르고 정량화된 값을 스프레드시트로 내보냅니다. 출력 파일을 생성하려면 Tools 메뉴에서 Batch Operations 옵션을 클릭합니다. 출력 파일을 저장할 폴더를 지정합니다.
7. 경로 분석
참고: 분석 후 확인된 중요한 대사 산물은 질병 그룹의 결과에 직접적인 영향을 미치는 주요 경로를 결정하는 데 사용됩니다. 이러한 목적을 위해 일반적으로 대사체학 통계 분석 소프트웨어의 경로 분석 모듈과 KEGG 데이터베이스가 사용됩니다.
- 얻은 중요한 대사 산물 목록을 모듈에 입력합니다. 경로 이름이 포함된 목록이 생성됩니다.
- Impact Factor가 0.1보다 큰 기준에 따라 최종 경로를 선택합니다. 유의한 영향 값은 질병 관련 경로를 나타낸다30.
- 질병의 중증도와 관련된 조절 장애를 밝히기 위해 결과 경로를 철저히 분석합니다. 이 방법의 단점 중 하나는 유의한 대사 산물이 3 미만일 때 조절 장애 경로를 식별하는 데 비효율적이라는 것입니다.
- 이에 대처하기 위해 확인된 대사 산물 및 질병 중증도와의 상관 관계는 이전에 발표된 문헌을 사용하여 결정할 수도 있습니다. 철저한 문헌 조사를 수행하고 식별된 대사 산물도 귀하의 관심사에 따라 동일한 추세를 따르고 있는지 관찰하십시오.
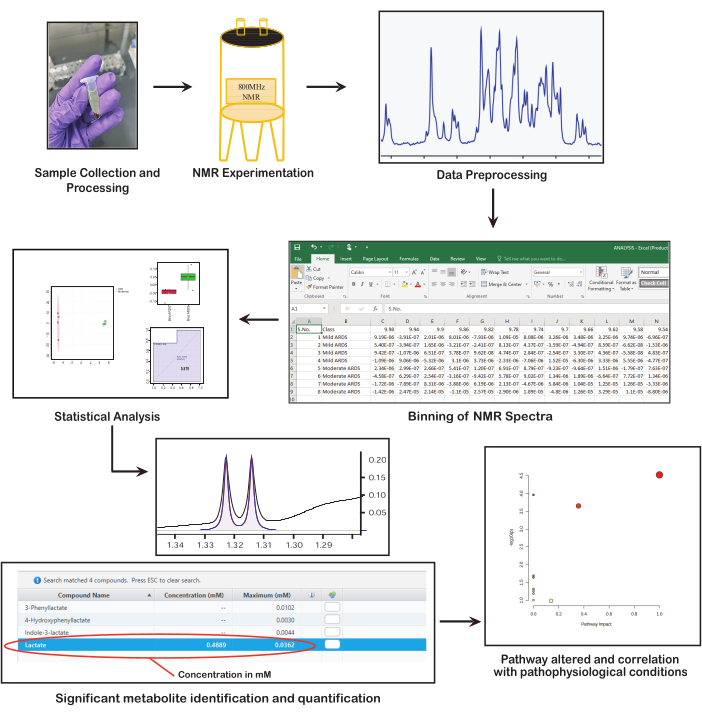
그림 1: NMR 기반 대사체학의 기본 단계. 이 그림은 NMR 기반 대사체학의 주요 단계인 샘플 수집 및 준비, NMR 분광법 수행, 데이터 전처리, 통계 분석 수행, 대사 산물 식별 및 정량화, 생물학적 중요성 해석을 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
대사체학 연구를 수행하려면 분석할 표본 크기와 특정 그룹을 결정하는 것이 중요합니다. 질병의 중증도48과 정확하게 상관관계가 있는 유의미한 결과를 얻기 위해서는 적절한 표본 크기를 선택하는 것이 필수적이다. 그러나 이 특정 작업에서는 주로 참조용으로 의도된 NMR 기반 대사체학을 사용하여 대사체의 식별 및 정량화와 관련된 단계를 보여주기 ...
토론
대사체학은 대사 산물을 효율적으로 식별하고 정량화하여 질병 중에 혼란스러워지는 대사 주기를 표적으로 합니다. 결과의 품질은 대사체학 접근법의 각 단계를 꼼꼼하게 실행하는 데 달려 있습니다. 검체 선택 및 수집에서 경로 식별에 이르기까지 모든 단계는 질병에 기여하는 주요 요인을 정확하게 식별하는 데 매우 중요합니다. 대사체학을 수행하기 전에 문헌에 대?...
공개
저자는 경쟁하는 재정적 이해관계가 없다고 선언합니다.
감사의 말
AS는 AcSIR(Academy of Scientific and Innovative Research)의 등록(등록 번호 10BB22A71002)을 인정합니다. AS는 또한 펠로우십에 대해 DRDO(Defence Research and Development Organization)를 인정합니다. 우리는 800MHz NMR 분광계 시설을 제공하고 교내 프로젝트(CBMR/IMR/0008/2021)를 통해 자금을 지원한 생물의학 연구 센터(CBMR)에 감사드립니다. 우리는 또한 SGPGIMS의 지속적인 지원에 대해 중환자의학과(CCM)에 감사드립니다. 우리는 많은 간호사들의 도움과 가장 중요하게는 이 연구에 등록된 환자들의 도움을 인정합니다. 이 연구는 생물의학 연구 센터(CBMR)의 교내 프로젝트(CBMR/IMR/0008/2021)와 교외 프로젝트(No. 국방 연구 개발 조직(DRDO)의 LSRB/01/15001/LSRB-404/PEE&BS/2023).
자료
| Name | Company | Catalog Number | Comments |
| Centirfuge | Sigma aldrich | 3-18KS | |
| Chenomx NMR suite | NMR Suite, v9, Chenomx Inc., Edmonton, Canada | NMR metabolite quantification software | |
| Co-axial insert | Sigma aldrich | Z278513 | |
| Deuterim oxide | Sigma aldrich | 151882 | |
| Eppendorf tubes | Tarsons | 500020 | |
| Metaboanalyst | Wishart Research Group | Metabolomics statistical analysis software | |
| NMR tube | Wilmad | Z412007 | 5mm diameter |
| Pipette | Eppendorf research plus | 3123000039 | 0-100 μl |
| Sample collection vials | Tarsons cryo chill vials | 523194 | |
| Sodium azide | Sigma aldrich | S2002 | |
| Sodium chloride crystal | Sigma aldrich | S9625 | |
| Sodium phosphate dibasic | Sigma aldrich | 567550 | |
| Sodium phosphate monobasic | Sigma aldrich | S0751 | |
| Topspin 3.6.4 | Bruker | NMR processing and analysis tool | |
| Tsp salt | Sigma aldrich | 269913 |
참고문헌
- Rubenfeld, G. D. Confronting the frustrations of negative clinical trials in acute respiratory distress syndrome. Ann. Am. Thorac. Soc. 12 (Supplement 1), S58-S63 (2015).
- Ware, L. B., et al. Prognostic and pathogenetic value of combining clinical and biochemical indices in patients with acute lung injury. Chest. 137 (2), 288-296 (2010).
- Johnson, C. H., Julijana, I., Gary, S. Metabolomics: beyond biomarkers and towards mechanisms. Nat Rev Mol Cell Biol. 17 (7), 451-459 (2016).
- Patti, G. J., Oscar, Y., Gary, S. Metabolomics: the apogee of the omics trilogy. Nat Rev Mol Cell Biol. 13 (4), 263-269 (2012).
- Nicholson, J. K., John, C. L. Metabonomics. Nature. 455 (7216), 1054-1056 (2008).
- Schnackenberg, L. K., Beger, R. D. Monitoring the health to disease continuum with global metabolic profiling and systems biology. Pharmacogenomics. 7 (7), 1077-1086 (2006).
- Pandey, S., Siddiqui, M. A., Azim, A., Sinha, N. Metabolic fingerprint of patients showing responsiveness to treatment of septic shock in intensive care unit. Magn Reson Mater Phys Biol Med. 36 (4), 659-669 (2023).
- Mamas, M., Dunn, W. B., Neyses, L., Goodacre, R. The role of metabolites and metabolomics in clinically applicable biomarkers of disease. Arch. Toxicol. 85, 5-17 (2011).
- Dunn, W. B., Broadhurst, D. I., Atherton, H. J., Goodacre, R., Griffin, J. L. Systems level studies of mammalian metabolomes: the roles of mass spectrometry and nuclear magnetic resonance spectroscopy. Chem Soc Rev. 40 (1), 387-426 (2011).
- Dettmer, K., Bruce, D. H. Metabolomics--a new exciting field within the" omics" sciences. Environ Health Perspect. 112 (7), A396-A397 (2004).
- Goodacre, R., Vaidyanathan, S., Dunn, W. B., Harrigan, G. G., Kell, D. B. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 22 (5), 245-252 (2004).
- Lindon, J. C., Nicholson, J. K., Holmes, E., Everett, J. R. Metabonomics: metabolic processes studied by NMR spectroscopy of biofluids. Concepts Magn Reson Educ J. 12 (5), 289-320 (2000).
- Bernini, P., et al. Individual human phenotypes in metabolic space and time. J Proteome Res. 8 (9), 4264-4271 (2009).
- Nicholson, J. K., et al. Metabolic phenotyping in clinical and surgical environments. Nature. 491 (7424), 384-392 (2012).
- Vinayavekhin, N., Homan, E. A., Saghatelian, A. Exploring disease through metabolomics. ACS Chem Biol. 5 (1), 91-103 (2010).
- Dunn, W. B., Ellis, D. I. Metabolomics: current analytical platforms and methodologies. TrAC Trends Anal Chem. 24 (4), 285-294 (2005).
- Dunn, W. B., Bailey, N. J. C., Johnson, H. E. Measuring the metabolome: current analytical technologies. Analyst. 130 (5), 606-625 (2005).
- Bingol, K. Recent advances in targeted and untargeted metabolomics by NMR and MS/NMR methods. High-throughput. 7 (2), 9 (2018).
- Emwas, A. H. M. The strengths and weaknesses of NMR spectroscopy and mass spectrometry with particular focus on metabolomics research. Methods Mol Biol. 1277, 161-193 (2015).
- Markley, J. L., et al. The future of NMR-based metabolomics. Curr Opin Biotechnol. 43, 34-40 (2017).
- Marchand, J., Martineau, E., Guitton, Y., Dervilly-Pinel, G., Giraudeau, P. Multidimensional NMR approaches towards highly resolved, sensitive and high-throughput quantitative metabolomics. Curr Opin Biotechnol. 43, 49-55 (2017).
- Eghbalnia, H. R., et al. Increasing rigor in NMR-based metabolomics through validated and open source tools. Curr Opin Biotechnol. 43, 56-61 (2017).
- Vignoli, A., et al. High-throughput metabolomics by 1D NMR. Angew Chem Int Ed. 58 (4), 968-994 (2019).
- Sundekilde, U. K., Eggers, N., Bertram, H. C. NMR-based metabolomics of food. Methods Mol Biol. 2037, 335-344 (2019).
- Garcia-Perez, I., et al. Identifying unknown metabolites using NMR-based metabolic profiling techniques. Nat Protoc. , 1-30 (2020).
- Dona, A. C., et al. A guide to the identification of metabolites in NMR-based metabonomics/metabolomics experiments. Comput Struct Biotechnol J. 14, 135-153 (2016).
- Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N., Siddiqui, M. H. Metabolomics: an emerging potential approach to decipher critical illnesses. Biophys Chem. 267, 106462 (2020).
- McNicholas, B. A., et al. Impact of early acute kidney injury on management and outcome in patients with acute respiratory distress syndrome: a secondary analysis of a multicenter observational study. Crit. Care Med. 47 (9), 1216-1225 (2019).
- Ambruso, S. L., et al. Inter-organ communication in homeostasis and disease: Lung metabolomics after ischemic acute kidney injury reveals increased oxidative stress, altered energy production, and ATP depletion. Am J Physiol Lung Cell Mol Physiol. 321 (1), L50 (2021).
- Viswan, A., et al. Metabolomics based predictive biomarker model of ARDS: A systemic measure of clinical hypoxemia. PloS One. 12 (11), e0187545 (2017).
- Metwaly, S. M., Brent, W. W. Systems biology ARDS research with a focus on metabolomics. Metabolites. 10 (5), 207 (2020).
- Stringer, K. A., et al. Metabolic consequences of sepsis-induced acute lung injury revealed by plasma 1H-nuclear magnetic resonance quantitative metabolomics and computational analysis. Am J Physiol Lung Cell Mol Physiol. 300 (1), L4-L11 (2011).
- Singh, A., Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N. Unveiling Pathophysiological Insights: Serum Metabolic Dysregulation in Acute Respiratory Distress Syndrome Patients with Acute Kidney Injury. J Proteome Res. 23 (10), 4216-4228 (2024).
- Slupsky, C. M. NMR-based analysis of metabolites in urine provides rapid diagnosis and etiology of pneumonia. Biomark Med. 4 (2), 195-197 (2010).
- Siddiqui, M. A., et al. NMR spectroscopy-based analysis of gallstones of cancerous and benign gallbladders from different geographical regions of the Indian subcontinent. Plos One. 18 (6), e0286979 (2023).
- Li, J., et al. 1HNMR-based metabolomic profile of rats with experimental acute pancreatitis. BMC Gastroenterol. 14, 1-7 (2014).
- Mao, H., et al. Systemic metabolic changes of traumatic critically ill patients revealed by an NMR-based metabonomic approach. J Proteome Res. 8 (12), 5423-5430 (2009).
- Stringer, K. A., Jones, A. E., Puskatich, M. A., Karnovsky, A., Serkova, N. J. 1H-nuclear magnetic resonance (NMR)-detected lipids associated with apoptosis differentiate early acute respiratory distress syndrome (ARDS) from sepsis. C63. LUNG INJURY AND REPAIR: TWO TO TANGO. Am J Resp Crit Care Med. 189, A5000 (2014).
- Viswan, A., Singh, C., Kayastha, A. M., Azim, A., Sinha, N. An NMR based panorama of the heterogeneous biology of acute respiratory distress syndrome (ARDS) from the standpoint of metabolic biomarkers. NMR Biomed. 33 (2), e4192 (2020).
- Singh, C., et al. Metabolic profiling of human lung injury by 1 H high-resolution nuclear magnetic resonance spectroscopy of blood serum. Metabolomics. 11, 166-174 (2015).
- Pandey, S., Siddiqui, M. A., Trigun, S. K., Azim, A., Sinha, N. Gender-specific association of oxidative stress and immune response in septic shock mortality using NMR-based metabolomics. Mol Omics. 18 (2), 143-153 (2022).
- Nagana, G. A., Gowda, Y. N., Raftery, D. Expanding the limits of human blood metabolite quantitation using NMR spectroscopy. Anal Chem. 87 (1), 706-715 (2015).
- Gowda, G. A. N., Raftery, D. Quantitating metabolites in protein precipitated serum using NMR spectroscopy. Anal Chem. 86 (11), 5433-5440 (2014).
- Gowda, G. A. N., Raftery, D. NMR-based metabolomics. Adv Exp Med Biol. 1280, 19-37 (2021).
- Xia, J., Psychogios, N., Young, N., Wishart, D. S. MetaboAnalyst: a web server for metabolomic data analysis and interpretation. Nucleic Acids Res. 37 (suppl_2), W652-W660 (2009).
- orbacıoğlu, &. #. 3. 5. 0. ;. K., Aksel, G. Receiver operating characteristic curve analysis in diagnostic accuracy studies: A guide to interpreting the area under the curve value. Turk J Emerg Med. 23 (4), 195 (2023).
- Ulrich, E. L., et al. BioMagResBank. Nucleic Acids Res. 36 (suppl_1), D402-D408 (2007).
- Nyamundanda, G., Gormley, I. C., Fan, Y., Gallagher, W. M., Brennan, L. MetSizeR: selecting the optimal sample size for metabolomic studies using an analysis based approach. BMC bioinformatics. 14, 1-8 (2013).
- Beckonert, O., et al. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts. Nat Protoc. 2 (11), 2692 (2007).
- Dona, A. C., et al. Precision high-throughput proton NMR spectroscopy of human urine, serum, and plasma for large-scale metabolic phenotyping. Anal Chem. 86 (19), 9887-9894 (2014).
- Ala-Korpela, M. Potential role of body fluid 1H NMR metabonomics as a prognostic and diagnostic tool. Expert Rev Mol Diagn. 7 (6), 761-773 (2007).
- Takeda, I., et al. Understanding the human salivary metabolome. NMR Biomed. 22 (6), 577-584 (2009).
- Callejon-Leblic, B. e. m., García-Barrera, T., Pereira-Vega, A., Gómez-Ariza, J. L. Metabolomic study of serum, urine and bronchoalveolar lavage fluid based on gas chromatography mass spectrometry to delve into the pathology of lung cancer. J Pharm Biomed Anal. 163, 122-129 (2019).
- Van Oort, P. M. P., et al. Exhaled breath metabolomics for the diagnosis of pneumonia in intubated and mechanically-ventilated intensive care unit (ICU)-patients. Int J Mol Sci. 18 (2), 449 (2017).
- Serkova, N. J., Niemann, C. U. Pattern recognition and biomarker validation using quantitative 1H-NMR-based metabolomics. Expert Rev Mol Diagn. 6 (5), 717-731 (2006).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation times. Rev Sci Instrum. 29 (8), 688-691 (1958).
- Xi, Y., Rocke, D. M. Baseline correction for NMR spectroscopic metabolomics data analysis. BMC bioinformatics. 9, 1-10 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유