Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Идентификация и количественная оценка нарушенных метаболитов у пациентов в критическом состоянии с использованием метаболомики на основе ЯМР
В этой статье
Резюме
Спектроскопия ядерного магнитного резонанса (ЯМР) используется для выявления нарушений регуляции метаболитов у пациентов с различными заболеваниями. Этот метод позволяет количественно оценить нарушенные метаболиты, раскрывая патофизиологические идеи. В данной статье мы описываем пошаговую процедуру применения подхода, основанного на ЯМР, для определения метаболических характеристик пациентов.
Аннотация
Метаболомика становится важным подходом к отражению индивидуальной реакции на патофизиологические условия. Спектроскопия ядерного магнитного резонанса (ЯМР) стала инструментом для выявления метаболических нарушений у пациентов в критическом состоянии с такими состояниями, как острый респираторный дистресс-синдром (ОРДС), тяжелый острый панкреатит (САП), острое повреждение почек (ОПП) и сепсис. Спектральные данные из образца сыворотки исследуемой и контрольной группы записываются с помощью ЯМР-спектрометра с частотой 800 МГц и обрабатываются с помощью средств обработки и анализа ЯМР. Кроме того, проводится строгий статистический анализ, такой как одномерные и многомерные тесты, чтобы точно определить значимые метаболиты, которые затем точно идентифицируются и количественно оцениваются с помощью программного обеспечения для количественного определения метаболитов ЯМР. Кроме того, анализ путей показывает нарушенные биохимические циклы, которые приводят к тяжести заболевания. С помощью этого комплексного подхода исследователи стремятся получить более глубокое представление о метаболических изменениях, связанных с этими критическими заболеваниями, потенциально прокладывая путь к лучшему пониманию болезни и улучшению диагностики и стратегий лечения.
Введение
Несмотря на постоянные усилия по обеспечению эффективной диагностики заболеваний во всем мире, таргетная терапия до сих пор не достигла своего истинного потенциала. Различные подходы, такие как транскриптомика, протеомика и т.д., привели к идентификации нескольких биомаркеров, но они не имели достаточной клинической значимости из-за недостаточной чувствительности и специфичности 1,2. Таргетная терапия является большой проблемой при некоторых мультифакториальных заболеваниях, что в конечном итоге приводит к более высокой смертности. Существует потребность в лучшем понимании основного механизма и патофизиологии сложных заболеваний, существующих с широкой гетерогенностью. Таким образом, в этом отношении развитие метаболомики произвело революцию в терапевтических разработках, что в конечном итоге может помочь в адаптации схем лечения различных критических заболеваний, таких как острый респираторный дистресс-синдром (ОРДС), сепсис и тяжелый острый панкреатит (САП).
Метаболомика — это комплексный подход, направленный на идентификацию и количественное определение молекул с малой молекулярной массой (метаболитов, таких как аминокислоты, липиды, пептиды, органические кислоты и витамины) в различных биожидкостях, клетках или тканевых экстрактах. Эти метаболиты, которые обычно весят менее 1500 Да, играют активную роль в биохимических процессах, отражая прогрессивную схему биологического состояния организма. Они включают субстраты для ключевых ферментативных процессов, промежуточные продукты в биологических путях и побочные продукты клеточного метаболизма. Следовательно, метаболомика фиксирует подробный отпечаток влияния диеты, лекарственных взаимодействий и болезненных состояний. Метаболитные изменения являются высокочувствительными индикаторами метаболизма и биологических путей, что позволяет коррелировать с фенотипическими экспрессиями и возникающими патофизиологическими аномалиями 3,4. Первоначальные вариации метаболитов могут служить ранними индикаторами тяжести заболевания, в то время как временные изменения могут помочь в мониторинге эффективности лечения, прогрессирования заболевания и клинических исходов 5,6,7. Таким образом, метаболомика улучшает клинические анализы и различные другие омиксные подходы, переопределяя заболевания через клинические, физиологические и биохимические конечные точки 8,9,10,11,12,13. Аналитические возможности метаболомики используются для мониторинга и определения восприимчивости к заболеванию путем изменения концентраций метаболитов 14,15.
В этом контексте масс-спектрометрия (МС) и спектроскопия ядерного магнитного резонанса (ЯМР) стали основными аналитическими платформами для профилирования метаболитов в биологических образцах. Эти методы используются как для целенаправленной, так и для нецелевой идентификации и количественного определения метаболитов 16,17,18. Каждая платформа имеет свои преимущества и ограничения, но неразрушающий характер ЯМР делает ее предпочтительной в различных исследованиях in vivo и для характеристики структуры неизвестных соединений, особенно на начальных этапах исследований метаболомики. Фракционирование образца, дериватизация и ионизация, необходимые перед МС, могут привести к систематическим ошибкам и часто приводить к потере образца, влияя на динамические характеристики, которые может быть зафиксирована с помощью ЯМР-спектроскопии с минимальной подготовкой образца или без нее. Основным ограничением ЯМР является его более низкая чувствительность по сравнению с МС, который обеспечивает более низкий предел обнаружения, что затрудняет обнаружение менее распространенныхметаболитов19. Тем не менее, такие достижения, как сверхпроводящие магниты с высоким разрешением, криогенное охлаждение ЯМР-зондов и методы, повышающие чувствительность, смягчили это ограничение 20,21,22. В качестве дополнительного подхода к геномике и протеомике, метаболическое профилирование с использованием ЯМР-спектроскопии набирает обороты в качестве предпочтительного метода 23,24,25. Минимальная подготовка образцов, воспроизводимость и повторяемость ЯМР делают его ценным инструментом для улавливания присущих метаболитам динамических характеристик, несмотря наего проблемы с чувствительностью.
Несколько исследовательских групп успешно провели метаболомику, точно определив нерегулируемый метаболический профиль пациентов приразличных заболеваниях, таких как ОРДС 28,29,30,31,32,33, пневмония 34, сепсис7, камни в желчном пузыре35 и панкреатит36. Метаболомные исследования пациентов в критическом состоянии на основе ЯМР сыграли важную роль в отслеживании прогрессирования от синдрома системного воспалительного ответа (SIRS) до синдрома полиорганной дисфункции (MODS), который является основной причиной смертности и заболеваемости в отделениях интенсивной терапии (ОИТ)37. В исследовании Stringer et al. образцы плазмы использовались для изучения метаболических изменений у пациентов с острым повреждением легких (ОПЛ), вызванным сепсисом, по сравнению с контрольной группой38. Ключевые метаболиты, обнаруженные в этом пилотном исследовании, отражали задействованные метаболические пути и их связь с клиническими показателями. Это исследование было расширено до метаболомики сыворотки крови для дифференциации сепсиса от ранних стадий механизмов повреждения легких при ОРДС32. Кроме того, в другом исследовании, посвященном ОРДС, были выявлены мощные сывороточные биомаркеры, которые четко дифференцируют острое повреждение легких/ОРДС и здоровые контрольные группы, что дает представление о системных метаболических изменениях, соответствующих острому началу повреждения легких39,40.
ЯМР-спектроскопия представляет собой высокопроизводительный и автоматизированный аналитический метод, который предоставляет надежную и непредвзятую информацию о метаболическом отпечатке, раскрывая лежащую в основе патофизиологию38. Клиническое применение и биологическая интерпретация данных ЯМР зависят от получения высококачественных спектров, содержащих богатую информацию. Поэтому обеспечение точного, единообразного и хорошо сформулированного сбора, обработки и анализа данных имеет решающее значение. Таким образом, цель данного исследования состоит в том, чтобы использовать основные этапы метаболомики на основе ЯМР для идентификации и количественной оценки метаболитов. В этом исследовании выделены ключевые этапы протокола, необходимого для исследования клинической метаболомики (Рисунок 1), такие как выбор подходящих образцов, сбор и хранение, обработка и подготовка образцов, сбор и анализ данных, идентификация и количественная оценка интересующего метаболита и, в конечном итоге, интерпретация результатов в клиническом контексте для получения соответствующих выводов. Каждый из этих этапов имеет важное значение для использования ЯМР-спектроскопии в метаболомике для получения важных биологических и клинических знаний.
протокол
Этическое одобрение (код IEC: 2022-71-PhD-126) было получено от IEC Института последипломного образования Санджая Ганди (SGPGIMS), Лакхнау. От пациентов или их родственников были получены письменные и информированные согласия на проведение исследования и публикацию данных в исследовательских целях. Более того, исследование проводилось в соответствии с институциональными рекомендациями.
1. Дизайн исследования и этическое обоснование
- Определите размер выборки для каждой группы. Внимательно изучите и установите критерии отбора участников. Кроме того, если план исследования требует включения образцов человека или животных, то получите этическое разрешение от соответствующего институционального этического комитета (IEC) до начала сбора образцов.
- Отдельные пациенты с ОРДС классифицируются как легкие, умеренные и тяжелые, на основе диагностических критериев Берлинского определения 2012 года, основанного на парциальном давлении кислорода в артериальной крови (PaO2) и фракции вдыхаемого кислорода (FiO2) (P/F). Пациенты с ОРДС с диапазоном P/F 300-200 оцениваются как легкие, 200-100 - как умеренные, а ниже 100 - как тяжелые38. Здесь мы сосредоточились на двух группах: пациенты с легкой и умеренной степенью тяжести ОРДС (см. таблицу 1).
ПРИМЕЧАНИЕ: Исследование включает использование сыворотки, полученной из образцов крови, для проведения метаболомики на основе ЯМР. В этом исследовании средний возраст участников, включенных для справки, составлял 42 года (± 10,9 лет), с гендерным распределением 2 мужчин и 6 женщин.
| Категория | Соотношение P/F |
| Легкая | 300-200 |
| Умеренный | 200-100 |
| Сильная | 100-0 |
Таблица 1: Категоризация пациентов с ОРДС.
2. Отбор, сбор и обработка образцов
- Выполните изоляцию сыворотки, выполнив описанные ниже действия.
- Для экспериментов соберите 2 мл образцов крови из артерий с помощью стерильной иглы в обычном (без добавок) и стерильном флаконе. Оставьте образец сворачиваться на 30 минут при комнатной температуре. Для данного исследования были отобраны 4 пациента с диагнозом ОРДС легкой степени тяжести и 4 пациента с умеренным ОРДС.
- Центрифугируйте образец при 3100 х г в течение 15 мин для отделения сыворотки25. Переложите сыворотку в стерильные микроцентрифужные пробирки, сделав аликвоты меньшего объема (т. е. 400-500 мкл), нанесите на них соответствующую маркировку (имя пациента, номер CR, точка дня рождения и т. д.) и храните при температуре -80 °C для будущего анализа.
ПРИМЕЧАНИЕ: На этом этапе эксперимент может быть приостановлен и возобновлен в удобное время.
- Обработайте образец сыворотки перед экспериментами ЯМР, выполнив следующие действия.
- Разморозьте образец перед проведением ЯМР-экспериментов. Смешайте 250 мкл аликвоты сыворотки с 250 мкл буферного раствора солевого фосфата (содержащего 100% D2O, 0,9% NaCl, 50 мМ буфера фосфата натрия, pH 7,4) для минимизации колебаний pH.
- Перемешайте смесь в течение нескольких секунд (~15 с), чтобы убедиться, что она однородно перемешана. Наконец, подготовленный образец переносят в чистую ЯМР-пробирку с коаксиальной вставкой, содержащей триметилсилилпропионат (ТСП) в концентрации 0,05 мМ, который служит внешним эталоном для калибровки и количественного определения41.
ПРИМЕЧАНИЕ: Для справки используется инертное и нереакционноспособное соединение, которое не взаимодействует с аналитами. Другие широко используемые референтные соединения включают тетраметилсилан (ТМС) и триметилсилилпропансульфонат натрия (ДСС).
3. Эксперименты с ЯМР
ПРИМЕЧАНИЕ: Основное внимание уделяется идентификации малых молекул в образцах сыворотки, поэтому мы использовали последовательность импульсов Карра-Перселла-Мейбума-Гилла (CPMG), которая подавляет сигналы макромолекул. Все образцы сыворотки в этом исследовании были зарегистрированы с помощью ЯМР-спектрометра с частотой 800 МГц, оснащенного криогенной трехрезонансной головкой TCI 5 мм с широкополосным обратным зондом и экранированным z-градиентом.
- Поместите образец в магнит. Напишите команду wrpa (укажите номер эксперимента) и нажмите enter, чтобы настроить протонный эксперимент с программой cpmg pulse. Укажите имя пациента и другие необходимые данные, нажав на опцию «Заголовок».
- Заблокируйте магнитное поле, написав команду Lock, нажмите Enter и выберите вариант 90% H2O и 10% D2O. Настройте и согласуйте пробник вручную с помощью ATMM, чтобы оптимизировать эффективность радиочастотных импульсов и максимизировать чувствительность. Проведите 1D градиентный шимминг, кликнув по топшим.
- Задайте следующие параметры сбора данных, в том числе задержку релаксации 5 с, ширину спектральной развертки 12 частей на миллион (ppm) и время эхо-сигнала 300 мкс. Все эти параметры можно легко изменить, выбрав опцию «Параметры захвата » на верхней панели.
- Примените уширение линии на 0,3 Гц с помощью функции экспоненциального окна, собирая данные в 64 000 точек за 128 сканирований. Эти параметры можно аналогичным образом изменить, выбрав опцию Параметры сбора данных на верхней панели. При нажатии на «Параметры получения» появится список спецификаций, и упомянутые атрибуты, наряду с другими параметрами, могут быть изменены в ходе оптимизации.
- Получение спектров ЯМР с помощью инструментов обработки и анализа ЯМР. Как только окончательные спектры будут получены, напишите команды apk и absn и нажмите Enter для выполнения фазовой коррекции и базовой коррекции соответственно. Нажмите на опцию «Калибровать ось» на верхней панели, а затем откалибруйте пик TSP до 0 ppm автоматически или вручную.
- Передача полученных данных из системы на рабочую станцию, где производится дальнейшая обработка и анализ.
ПРИМЕЧАНИЕ: Экспериментальная фаза завершена и может быть приостановлена. Предварительная обработка, анализ и интерпретация данных могут быть удобно выполнены.
4. Предварительная обработка данных
- Проведите предварительную обработку данных с помощью инструментов обработки и анализа ЯМР и программного обеспечения для количественного определения метаболитов ЯМР.
- Иногда одной автоматической коррекции недостаточно, и в этом случае необходима еще и ручная коррекция. Чтобы выполнить фазовую коррекцию вручную, нажмите на опцию «Процесс » в верхней строке меню инструментов обработки и анализа ЯМР. Затем нажмите на Настроить фазу, перетащите мышь и наблюдайте, пока спектры не будут скорректированы по фазе. После этого нажмите на опцию «Сохранить и вернуть » (представленную в виде значка), доступную в той же строке меню.
- Чтобы выполнить ручную коррекцию базовой линии (метод Уиттакера), откройте спектральный файл в процессорном модуле программного обеспечения для количественного определения метаболитов ЯМР. Выберите опцию Коррекция базовой линии. Затем выберите вариант Метод Уиттакера. Теперь поместите точки у основания пиков во всех спектрах (от 0,0 до 10 ppm, обычно в случае сыворотки или в зависимости от наличия метаболита в спектрах), чтобы отрегулировать основание. После того, как все спектральные файлы будут скорректированы на базовом уровне, сохраните их в одной папке. Дальнейшая обработка с использованием процессора или модуля профилирования программного обеспечения для количественного определения метаболитов ЯМР может быть выполнена с использованием того же файла, сохраненного в определенном формате.
- Выполните группирование с помощью модуля профилировщика того же программного обеспечения. Выберите опцию «Инструменты», затем «Пакетная обработка», а затем «Спектральное объединение».
- Чтобы создать окончательный лист биннинга, который будет использоваться для статистического анализа, выберите папку, содержащую все спектральные файлы с базовой коррекцией, щелкнув Спектральное биннинг.
- Разделите спектры на заданное количество ячеек одинакового размера с определенной шириной спектра, в диапазоне от 0,01 до 0,04 ppm (может быть оптимизировано в соответствии с исследованием). Этот процесс упрощает и систематизирует спектральные данные, способствуя более последовательному анализу. Укажите размер контейнера, а также начальное и конечное значения ppm и укажите папку, в которой будут сохраняться выходные листы группирования.
- Исключите области химического сдвига, соответствующие воде и растворителю TSP (4,8-5,2 и 0,0-0,7 ppm) для предотвращения спектральной интерференции 42,43,44.
- Эта таблица содержит названия образцов в одной строке и различные значения ячеек вместе с ppm в других строках. Прежде чем использовать этот лист для статистического анализа, измените его, организовав выборки в группы для анализа и сохранив его только в формате CSV.
ПРИМЕЧАНИЕ: Например, при сравнении легкой группы с умеренной группой пациентов с ОРДС мы расположили данные с метками легкой и умеренной степени тяжести, добавили серийный номер и сохранили файл в формате CSV.
5. Статистический анализ
ПРИМЕЧАНИЕ: Лист группирования, полученный на предыдущем этапе, служит входным файлом для статистического анализа. В данном исследовании был использован модуль статистического анализа (один фактор) программного обеспечения для статистического анализа метаболомики.
- Перед статистическим анализом выполните шаг нормализации с использованием сумм, логарифма и Парето, но он может варьироваться в разных исследованиях45.
- Выполнение одномерного и многомерного анализа для различения различных групп и определения точности идентифицированных метаболитов.
- Выполните анализ главных компонент (PCA), частичный дискриминантный анализ по методу наименьших квадратов (PLS-DA) и ортогональный частичный дискриминантный анализ по методу наименьших квадратов (OPLS-DA) для наблюдения за дискриминацией между группами, что дает оценку переменной важности проекции (VIP) для каждого метаболита, ответственного за разницу в метаболическом профиле.
- Используйте одномерные методы, такие как t-критерий Стьюдента и ANOVA, для выявления значимых метаболитов на основе критериев наличия p-значения менее 0,05. Оба теста позволяют получить графики с ящичковыми усами, показывающие разницу в относительной концентрации метаболитов.
- Выполните анализ биомаркеров, который дает область под графиком кривой рабочих характеристик приемника (AUROC). Значение AUC (площадь под кривой) > 0,8 считается значимым46.
- Выберите окончательный список значимых метаболитов с использованием критериев VIP score>1, скорректированного по Бонферрони p-значения<0,0544 и AUC > 0,846.
- Определите метаболиты, как описано ниже.
- Биннинг обеспечивает значительные метаболиты в виде значений ppm, а не названий. Определите метаболиты, соответствующие конкретным значениям ppm, с помощью баз данных, таких как Банк биологического магнитного резонанса (BMRB)47и База данных метаболомов человека (HMDB).
- Кроме того, используйте ранее опубликованную литературу42,44 для идентификации и валидации.
- Используйте модуль профилирования программного обеспечения для количественного определения метаболитов ЯМР для подтверждения идентифицированных метаболитов с их значениями ppm.
6. Количественная оценка метаболитов с помощью программного обеспечения для количественного определения метаболитов ЯМР
ПРИМЕЧАНИЕ: Модуль профилирования программного обеспечения широко используется для количественной оценки идентифицированных метаболитов.
- Перед началом количественного анализа откалибруйте выбранное для исследования эталонное соединение (tsp, использованное в данном исследовании) с помощью процессорного модуля. Нажмите на опцию Calibrate CSI (индикатор химической формы). Укажите концентрацию эталонного соединения (0,05 мМ для данного исследования). Затем перетащите программный пик на спектральный опорный пик и правильно отрегулируйте его высоту и ширину. Как только спектральные опорные пики совпадут с пиками программного обеспечения, нажмите «Принять».
- Откройте нужные спектры в модуле профилировщика и выберите подходящую библиотеку соединений для изучения.
- В нижней части экрана отображается список метаболитов. Выберите интересующий метаболит из этого списка. После выбора возможные ppm выбранного метаболита появятся в верхнем углу спектров.
- Выберите значение Ppm в верхнем углу и увеличьте спектр до конкретного масштаба метаболита.
- На этом этапе на экране появятся два пика: один представляет записанные данные, а другой, отображаемый в виде пунктирной линии, представляет пик программного обеспечения. Совместите оба пика. Перетащите программный пик и отрегулируйте его высоту и положение в соответствии с пиком, присутствующим в образце.
- После правильного выравнивания концентрация метаболита может быть получена из значения, отображаемого под заголовком «Концентрация в мМ» для этого метаболита (рис. 1).
- Следуйте той же процедуре для всех метаболитов и образцов и экспортируйте количественные значения в электронную таблицу. Чтобы создать выходной файл, выберите параметр «Пакетные операции » в меню «Инструменты ». Укажите нужную папку для сохранения выходного файла.
7. Анализ путей
Значимые метаболиты, выявленные после анализа, используются для определения основных путей, непосредственно влияющих на исход в группах заболеваний. Для этой цели обычно используется модуль анализа путей программного обеспечения для статистического анализа метаболомики и база данных KEGG.
- Введите полученный список значимых метаболитов в модуль. Будет сгенерирован список, содержащий названия путей.
- Выберите окончательные пути на основе критерия наличия импакт-фактора больше 0,1. Значимые значения воздействия указывают на связанные с заболеванием пути30.
- Тщательно проанализируйте полученные пути, чтобы выяснить дисрегуляцию, связанную с тяжестью заболевания. Одним из недостатков этого метода является его неэффективность в выявлении нерегулируемых путей, когда значимые метаболиты меньше 3.
- Чтобы справиться с тем же, идентифицированный метаболит и его корреляцию с тяжестью заболевания также можно определить, используя ранее опубликованную литературу. Проведите тщательный обзор литературы и посмотрите, следует ли идентифицированный метаболит той же тенденции, что и ваши интересы.
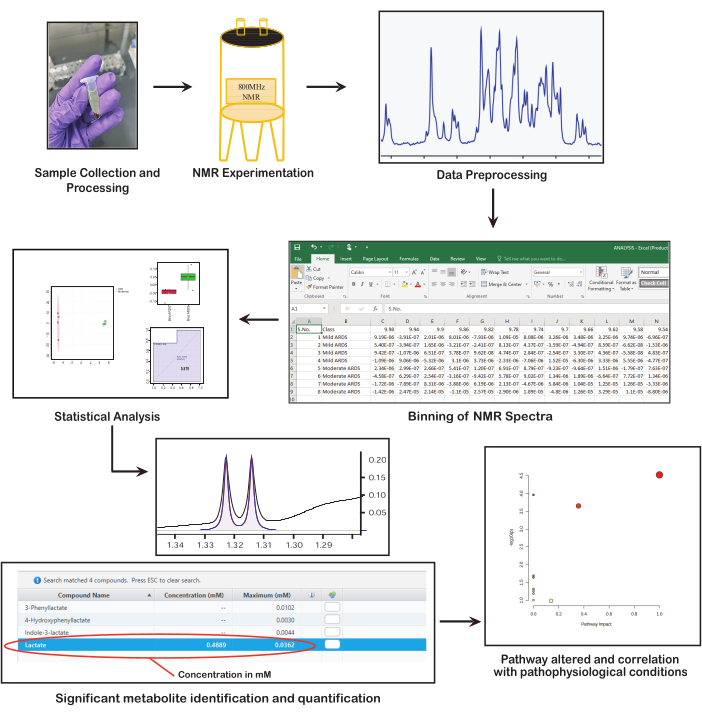
Рисунок 1: Фундаментальные шаги в метаболомике на основе ЯМР. На рисунке представлены ключевые этапы метаболомики на основе ЯМР: сбор и подготовка образцов, проведение ЯМР-спектроскопии, предварительная обработка данных, проведение статистического анализа, идентификация и количественное определение метаболитов, а также интерпретация биологической значимости. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Результаты
Для проведения метаболомного исследования важно определить размер выборки и конкретные группы, которые будут анализироваться. Выбор адекватного размера выборки имеет важное значение для получения значимых результатов, которые точно коррелируют с тяжестью заболев?...
Обсуждение
Метаболомика эффективно идентифицирует и количественно определяет метаболиты, нацеливаясь на метаболические циклы, которые нарушаются во время болезни. Качество результатов зависит от тщательного выполнения каждого этапа метаболомического подхода. Каждый этап, о?...
Раскрытие информации
Авторы заявляют об отсутствии конкурирующих финансовых интересов.
Благодарности
AS признает Академию научных и инновационных исследований (AcSIR) для регистрации (регистрационный No 10BB22A71002). AS также выражает благодарность Организации оборонных исследований и разработок (DRDO) за стипендию. Мы выражаем признательность Центру биомедицинских исследований (CBMR) за предоставление установки для ЯМР-спектрометра на частоте 800 МГц и финансирование в рамках очного проекта (CBMR/IMR/0008/2021). Мы также выражаем признательность Департаменту интенсивной терапии (CCM), SGPGIMS, за постоянную поддержку. Мы признательны за помощь многим медсестрам, а также, что самое главное, пациентам, участвующим в этом исследовании. Данное исследование финансировалось в рамках очного проекта (CBMR/IMR/0008/2021) Центра биомедицинских исследований (CBMR) и заочного проекта (No. LSRB/01/15001/LSRB-404/PEE&BS/2023) Организации оборонных исследований и разработок (DRDO).
Материалы
| Name | Company | Catalog Number | Comments |
| Centirfuge | Sigma aldrich | 3-18KS | |
| Chenomx NMR suite | NMR Suite, v9, Chenomx Inc., Edmonton, Canada | NMR metabolite quantification software | |
| Co-axial insert | Sigma aldrich | Z278513 | |
| Deuterim oxide | Sigma aldrich | 151882 | |
| Eppendorf tubes | Tarsons | 500020 | |
| Metaboanalyst | Wishart Research Group | Metabolomics statistical analysis software | |
| NMR tube | Wilmad | Z412007 | 5mm diameter |
| Pipette | Eppendorf research plus | 3123000039 | 0-100 μl |
| Sample collection vials | Tarsons cryo chill vials | 523194 | |
| Sodium azide | Sigma aldrich | S2002 | |
| Sodium chloride crystal | Sigma aldrich | S9625 | |
| Sodium phosphate dibasic | Sigma aldrich | 567550 | |
| Sodium phosphate monobasic | Sigma aldrich | S0751 | |
| Topspin 3.6.4 | Bruker | NMR processing and analysis tool | |
| Tsp salt | Sigma aldrich | 269913 |
Ссылки
- Rubenfeld, G. D. Confronting the frustrations of negative clinical trials in acute respiratory distress syndrome. Ann. Am. Thorac. Soc. 12 (Supplement 1), S58-S63 (2015).
- Ware, L. B., et al. Prognostic and pathogenetic value of combining clinical and biochemical indices in patients with acute lung injury. Chest. 137 (2), 288-296 (2010).
- Johnson, C. H., Julijana, I., Gary, S. Metabolomics: beyond biomarkers and towards mechanisms. Nat Rev Mol Cell Biol. 17 (7), 451-459 (2016).
- Patti, G. J., Oscar, Y., Gary, S. Metabolomics: the apogee of the omics trilogy. Nat Rev Mol Cell Biol. 13 (4), 263-269 (2012).
- Nicholson, J. K., John, C. L. Metabonomics. Nature. 455 (7216), 1054-1056 (2008).
- Schnackenberg, L. K., Beger, R. D. Monitoring the health to disease continuum with global metabolic profiling and systems biology. Pharmacogenomics. 7 (7), 1077-1086 (2006).
- Pandey, S., Siddiqui, M. A., Azim, A., Sinha, N. Metabolic fingerprint of patients showing responsiveness to treatment of septic shock in intensive care unit. Magn Reson Mater Phys Biol Med. 36 (4), 659-669 (2023).
- Mamas, M., Dunn, W. B., Neyses, L., Goodacre, R. The role of metabolites and metabolomics in clinically applicable biomarkers of disease. Arch. Toxicol. 85, 5-17 (2011).
- Dunn, W. B., Broadhurst, D. I., Atherton, H. J., Goodacre, R., Griffin, J. L. Systems level studies of mammalian metabolomes: the roles of mass spectrometry and nuclear magnetic resonance spectroscopy. Chem Soc Rev. 40 (1), 387-426 (2011).
- Dettmer, K., Bruce, D. H. Metabolomics--a new exciting field within the" omics" sciences. Environ Health Perspect. 112 (7), A396-A397 (2004).
- Goodacre, R., Vaidyanathan, S., Dunn, W. B., Harrigan, G. G., Kell, D. B. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 22 (5), 245-252 (2004).
- Lindon, J. C., Nicholson, J. K., Holmes, E., Everett, J. R. Metabonomics: metabolic processes studied by NMR spectroscopy of biofluids. Concepts Magn Reson Educ J. 12 (5), 289-320 (2000).
- Bernini, P., et al. Individual human phenotypes in metabolic space and time. J Proteome Res. 8 (9), 4264-4271 (2009).
- Nicholson, J. K., et al. Metabolic phenotyping in clinical and surgical environments. Nature. 491 (7424), 384-392 (2012).
- Vinayavekhin, N., Homan, E. A., Saghatelian, A. Exploring disease through metabolomics. ACS Chem Biol. 5 (1), 91-103 (2010).
- Dunn, W. B., Ellis, D. I. Metabolomics: current analytical platforms and methodologies. TrAC Trends Anal Chem. 24 (4), 285-294 (2005).
- Dunn, W. B., Bailey, N. J. C., Johnson, H. E. Measuring the metabolome: current analytical technologies. Analyst. 130 (5), 606-625 (2005).
- Bingol, K. Recent advances in targeted and untargeted metabolomics by NMR and MS/NMR methods. High-throughput. 7 (2), 9 (2018).
- Emwas, A. H. M. The strengths and weaknesses of NMR spectroscopy and mass spectrometry with particular focus on metabolomics research. Methods Mol Biol. 1277, 161-193 (2015).
- Markley, J. L., et al. The future of NMR-based metabolomics. Curr Opin Biotechnol. 43, 34-40 (2017).
- Marchand, J., Martineau, E., Guitton, Y., Dervilly-Pinel, G., Giraudeau, P. Multidimensional NMR approaches towards highly resolved, sensitive and high-throughput quantitative metabolomics. Curr Opin Biotechnol. 43, 49-55 (2017).
- Eghbalnia, H. R., et al. Increasing rigor in NMR-based metabolomics through validated and open source tools. Curr Opin Biotechnol. 43, 56-61 (2017).
- Vignoli, A., et al. High-throughput metabolomics by 1D NMR. Angew Chem Int Ed. 58 (4), 968-994 (2019).
- Sundekilde, U. K., Eggers, N., Bertram, H. C. NMR-based metabolomics of food. Methods Mol Biol. 2037, 335-344 (2019).
- Garcia-Perez, I., et al. Identifying unknown metabolites using NMR-based metabolic profiling techniques. Nat Protoc. , 1-30 (2020).
- Dona, A. C., et al. A guide to the identification of metabolites in NMR-based metabonomics/metabolomics experiments. Comput Struct Biotechnol J. 14, 135-153 (2016).
- Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N., Siddiqui, M. H. Metabolomics: an emerging potential approach to decipher critical illnesses. Biophys Chem. 267, 106462 (2020).
- McNicholas, B. A., et al. Impact of early acute kidney injury on management and outcome in patients with acute respiratory distress syndrome: a secondary analysis of a multicenter observational study. Crit. Care Med. 47 (9), 1216-1225 (2019).
- Ambruso, S. L., et al. Inter-organ communication in homeostasis and disease: Lung metabolomics after ischemic acute kidney injury reveals increased oxidative stress, altered energy production, and ATP depletion. Am J Physiol Lung Cell Mol Physiol. 321 (1), L50 (2021).
- Viswan, A., et al. Metabolomics based predictive biomarker model of ARDS: A systemic measure of clinical hypoxemia. PloS One. 12 (11), e0187545 (2017).
- Metwaly, S. M., Brent, W. W. Systems biology ARDS research with a focus on metabolomics. Metabolites. 10 (5), 207 (2020).
- Stringer, K. A., et al. Metabolic consequences of sepsis-induced acute lung injury revealed by plasma 1H-nuclear magnetic resonance quantitative metabolomics and computational analysis. Am J Physiol Lung Cell Mol Physiol. 300 (1), L4-L11 (2011).
- Singh, A., Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N. Unveiling Pathophysiological Insights: Serum Metabolic Dysregulation in Acute Respiratory Distress Syndrome Patients with Acute Kidney Injury. J Proteome Res. 23 (10), 4216-4228 (2024).
- Slupsky, C. M. NMR-based analysis of metabolites in urine provides rapid diagnosis and etiology of pneumonia. Biomark Med. 4 (2), 195-197 (2010).
- Siddiqui, M. A., et al. NMR spectroscopy-based analysis of gallstones of cancerous and benign gallbladders from different geographical regions of the Indian subcontinent. Plos One. 18 (6), e0286979 (2023).
- Li, J., et al. 1HNMR-based metabolomic profile of rats with experimental acute pancreatitis. BMC Gastroenterol. 14, 1-7 (2014).
- Mao, H., et al. Systemic metabolic changes of traumatic critically ill patients revealed by an NMR-based metabonomic approach. J Proteome Res. 8 (12), 5423-5430 (2009).
- Stringer, K. A., Jones, A. E., Puskatich, M. A., Karnovsky, A., Serkova, N. J. 1H-nuclear magnetic resonance (NMR)-detected lipids associated with apoptosis differentiate early acute respiratory distress syndrome (ARDS) from sepsis. C63. LUNG INJURY AND REPAIR: TWO TO TANGO. Am J Resp Crit Care Med. 189, A5000 (2014).
- Viswan, A., Singh, C., Kayastha, A. M., Azim, A., Sinha, N. An NMR based panorama of the heterogeneous biology of acute respiratory distress syndrome (ARDS) from the standpoint of metabolic biomarkers. NMR Biomed. 33 (2), e4192 (2020).
- Singh, C., et al. Metabolic profiling of human lung injury by 1 H high-resolution nuclear magnetic resonance spectroscopy of blood serum. Metabolomics. 11, 166-174 (2015).
- Pandey, S., Siddiqui, M. A., Trigun, S. K., Azim, A., Sinha, N. Gender-specific association of oxidative stress and immune response in septic shock mortality using NMR-based metabolomics. Mol Omics. 18 (2), 143-153 (2022).
- Nagana, G. A., Gowda, Y. N., Raftery, D. Expanding the limits of human blood metabolite quantitation using NMR spectroscopy. Anal Chem. 87 (1), 706-715 (2015).
- Gowda, G. A. N., Raftery, D. Quantitating metabolites in protein precipitated serum using NMR spectroscopy. Anal Chem. 86 (11), 5433-5440 (2014).
- Gowda, G. A. N., Raftery, D. NMR-based metabolomics. Adv Exp Med Biol. 1280, 19-37 (2021).
- Xia, J., Psychogios, N., Young, N., Wishart, D. S. MetaboAnalyst: a web server for metabolomic data analysis and interpretation. Nucleic Acids Res. 37 (suppl_2), W652-W660 (2009).
- orbacıoğlu, &. #. 3. 5. 0. ;. K., Aksel, G. Receiver operating characteristic curve analysis in diagnostic accuracy studies: A guide to interpreting the area under the curve value. Turk J Emerg Med. 23 (4), 195 (2023).
- Ulrich, E. L., et al. BioMagResBank. Nucleic Acids Res. 36 (suppl_1), D402-D408 (2007).
- Nyamundanda, G., Gormley, I. C., Fan, Y., Gallagher, W. M., Brennan, L. MetSizeR: selecting the optimal sample size for metabolomic studies using an analysis based approach. BMC bioinformatics. 14, 1-8 (2013).
- Beckonert, O., et al. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts. Nat Protoc. 2 (11), 2692 (2007).
- Dona, A. C., et al. Precision high-throughput proton NMR spectroscopy of human urine, serum, and plasma for large-scale metabolic phenotyping. Anal Chem. 86 (19), 9887-9894 (2014).
- Ala-Korpela, M. Potential role of body fluid 1H NMR metabonomics as a prognostic and diagnostic tool. Expert Rev Mol Diagn. 7 (6), 761-773 (2007).
- Takeda, I., et al. Understanding the human salivary metabolome. NMR Biomed. 22 (6), 577-584 (2009).
- Callejon-Leblic, B. e. m., García-Barrera, T., Pereira-Vega, A., Gómez-Ariza, J. L. Metabolomic study of serum, urine and bronchoalveolar lavage fluid based on gas chromatography mass spectrometry to delve into the pathology of lung cancer. J Pharm Biomed Anal. 163, 122-129 (2019).
- Van Oort, P. M. P., et al. Exhaled breath metabolomics for the diagnosis of pneumonia in intubated and mechanically-ventilated intensive care unit (ICU)-patients. Int J Mol Sci. 18 (2), 449 (2017).
- Serkova, N. J., Niemann, C. U. Pattern recognition and biomarker validation using quantitative 1H-NMR-based metabolomics. Expert Rev Mol Diagn. 6 (5), 717-731 (2006).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation times. Rev Sci Instrum. 29 (8), 688-691 (1958).
- Xi, Y., Rocke, D. M. Baseline correction for NMR spectroscopic metabolomics data analysis. BMC bioinformatics. 9, 1-10 (2008).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены