É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Identificação e quantificação de metabólitos perturbados em pacientes críticos usando metabolômica baseada em RMN
Neste Artigo
Resumo
A espectroscopia de ressonância magnética nuclear (RMN) é usada para identificar desregulação nos metabólitos em pacientes com várias doenças. Essa técnica permite a quantificação dos metabólitos perturbados, desvendando os insights fisiopatológicos. Aqui, descrevemos o procedimento passo a passo da abordagem baseada em RMN para a caracterização metabólica dos pacientes.
Resumo
A metabolômica está emergindo como uma abordagem significativa para refletir a resposta do indivíduo às condições fisiopatológicas. A espectroscopia de ressonância magnética nuclear (RMN) evoluiu como uma ferramenta para identificar desregulações metabólicas em pacientes gravemente enfermos que sofrem de condições como síndrome do desconforto respiratório agudo (SDRA), pancreatite aguda grave (SAP), lesão renal aguda (LRA) e sepse. Os dados espectrais da amostra de soro do grupo de estudo e controle são registrados usando um espectrômetro de RMN de 800 MHz e processados usando ferramentas de processamento e análise de RMN. Além disso, uma análise estatística rigorosa, como testes univariados e multivariados, é realizada para identificar metabólitos significativos, que são então identificados e quantificados com precisão usando o software de quantificação de metabólitos de RMN. Além disso, a análise de vias destaca os ciclos bioquímicos perturbados que resultam na gravidade da doença. Por meio dessa abordagem abrangente, os pesquisadores pretendem obter insights mais profundos sobre as alterações metabólicas associadas a essas doenças críticas, potencialmente abrindo caminho para uma melhor compreensão da doença e melhores estratégias de diagnóstico e tratamento.
Introdução
Apesar dos esforços contínuos para fornecer diagnóstico eficiente de doenças em todo o mundo, a terapia direcionada ainda não atingiu seu verdadeiro potencial. Várias abordagens, como transcriptômica, proteômica, etc., resultaram na identificação de vários biomarcadores, mas estes não tiveram utilidade clínica suficiente devido à falta de sensibilidade e especificidade 1,2. A terapia direcionada é um grande desafio em algumas doenças multifatoriais, levando a uma maior mortalidade. Há necessidade de uma melhor compreensão do mecanismo subjacente e da fisiopatologia de doenças complexas existentes com ampla heterogeneidade. Assim, a este respeito, a evolução da metabolômica revolucionou o desenvolvimento terapêutico, o que eventualmente pode ajudar a adaptar os regimes de tratamento para várias doenças críticas, como síndrome do desconforto respiratório agudo (SDRA), sepse e pancreatite aguda grave (SAP).
A metabolômica é uma abordagem abrangente que visa identificar e quantificar moléculas de pequeno peso molecular (metabólitos como aminoácidos, lipídios, peptídeos, ácidos orgânicos e vitaminas) em vários biofluidos, células ou extratos de tecidos. Esses metabólitos, que normalmente pesam menos de 1500 Da, desempenham papéis ativos nos processos bioquímicos, refletindo um esboço progressivo do estado biológico do organismo. Eles incluem substratos para os principais processos enzimáticos, intermediários em vias biológicas e subprodutos do metabolismo celular. Consequentemente, a metabolômica captura uma impressão digital detalhada das influências dietéticas, interações medicamentosas e estados de doença. As alterações metabólicas são indicadores altamente sensíveis do metabolismo e das vias biológicas, permitindo correlações com expressões fenotípicas e anormalidades fisiopatológicas resultantes 3,4. As variações iniciais nos metabólitos podem servir como indicadores precoces da gravidade da doença, enquanto as mudanças temporais podem ajudar no monitoramento da eficácia do tratamento, progressão da doença e resultados clínicos 5,6,7. A metabolômica, portanto, aprimora os ensaios clínicos e várias outras abordagens ômicas, redefinindo doenças por meio de desfechos clínicos, fisiológicos e bioquímicos 8,9,10,11,12,13. As capacidades analíticas da metabolômica são empregadas para monitorar e determinar a suscetibilidade à doença por meio de concentrações alteradas de metabólitos14,15.
Nesse contexto, tanto a espectrometria de massa (MS) quanto a espectroscopia de ressonância magnética nuclear (RMN) surgiram como as principais plataformas analíticas para o perfil de metabólitos em amostras biológicas. Esses métodos são usados para identificação e quantificação direcionada e não direcionada de metabólitos 16,17,18. Cada plataforma tem suas vantagens e limitações, mas a natureza não destrutiva da RMN a torna preferível em vários estudos in vivo e para a caracterização da estrutura de compostos desconhecidos, particularmente nos estágios iniciais da pesquisa metabolômica. O fracionamento, a derivatização e a ionização da amostra necessários antes da MS podem introduzir vieses e muitas vezes resultar em perda de amostra, afetando as características dinâmicas que a espectroscopia de RMN pode capturar com o mínimo ou nenhuma preparação da amostra. A principal limitação da RMN é sua menor sensibilidade em comparação com a EM, que oferece um limite inferior de detecção, dificultando a detecção de metabólitos menos abundantes19. No entanto, avanços como ímãs supercondutores de alta resolução, sondas de RMN resfriadas criogenicamente e técnicas que aumentam a sensibilidade mitigaram essa limitação 20,21,22. Como uma abordagem complementar à genômica e proteômica, o perfil metabólico usando espectroscopia de RMN está ganhando força como uma técnica preferida 23,24,25. A preparação mínima de amostras, reprodutibilidade e repetibilidade da RMN a tornam uma ferramenta valiosa para capturar as características dinâmicas inerentes aos metabólitos, apesar de seus desafios de sensibilidade26.
Vários grupos de pesquisa realizaram metabolômica com sucesso, identificando o perfil metabólico desregulado de pacientes para várias doenças27, como SDRA 28,29,30,31,32,33, pneumonia 34, sepse7, cálculos biliares35 e pancreatite36. Estudos metabolômicos baseados em RMN de pacientes críticos têm sido fundamentais para rastrear a progressão da síndrome da resposta inflamatória sistêmica (SIRS) para a síndrome de disfunção de múltiplos órgãos (MODS), que é uma das principais causas de mortalidade e morbidade em unidades de terapia intensiva (UTI)37. Em um estudo de Stringer e col., amostras de plasma foram usadas para examinar alterações metabólicas em pacientes com lesão pulmonar aguda (LPA) induzida por sepse em comparação com pacientes controles38. Os principais metabólitos encontrados elevados neste estudo piloto refletiram as vias metabólicas envolvidas e sua associação com os escores clínicos. Esta pesquisa foi estendida à metabolômica sérica para diferenciar a sepse dos estágios iniciais dos mecanismos de lesão pulmonar na SDRA32. Além disso, outro estudo em SDRA identificou biomarcadores séricos potentes que diferenciam distintamente entre lesão pulmonar aguda/SDRA e controles saudáveis, oferecendo informações sobre alterações metabólicas sistêmicas correspondentes ao início agudo da lesão pulmonar39,40.
A espectroscopia de RMN é uma técnica analítica automatizada e de alto rendimento que fornece informações robustas e imparciais sobre a impressão digital metabólica, revelando a fisiopatologia subjacente38. A aplicação clínica e a interpretação biológica dos dados de RMN dependem da obtenção de espectros de alta qualidade que contenham informações ricas. Portanto, é crucial garantir a coleta, o processamento e a análise de dados precisos, uniformes e bem formulados. Portanto, o objetivo deste estudo é alavancar as etapas essenciais da metabolômica baseada em RMN para a identificação e quantificação de metabólitos. Este estudo destaca as principais etapas do protocolo necessário para o estudo metabolômico clínico (Figura 1), como a seleção de amostras apropriadas, coleta e armazenamento, processamento e preparação de amostras, aquisição e análise de dados, identificação e quantificação do metabólito de interesse e, eventualmente, interpretação dos resultados em um contexto clínico para obter insights relevantes. Cada uma dessas etapas é essencial para alavancar a espectroscopia de RMN em metabolômica para descobrir insights biológicos e clínicos significativos.
Protocolo
A aprovação ética (código IEC: 2022-71-PhD-126) foi obtida do IEC do Instituto de Pós-Graduação em Ciências Médicas Sanjay Gandhi (SGPGIMS), Lucknow. Foram obtidos consentimentos por escrito e informados dos pacientes ou de seus familiares para a realização do estudo e publicação dos dados para fins de pesquisa. Além disso, a pesquisa foi conduzida seguindo as diretrizes institucionais.
1. Desenho do estudo e liberação ética
- Determine o tamanho da amostra para cada grupo. Revise cuidadosamente e estabeleça os critérios de seleção para os participantes. Além disso, se o desenho do estudo exigir a inclusão de amostras humanas ou animais, obtenha autorização ética do comitê de ética institucional (IEC) apropriado antes de iniciar a coleta da amostra.
- Selecione pacientes com SDRA categorizados como leves, moderados e graves, com base nos critérios diagnósticos da definição de Berlim 2012 com base na pressão parcial de oxigênio no sangue arterial (PaO2) para a fração inspirada de oxigênio (FiO2) (P/F). Os pacientes com SDRA com uma faixa de P/F de 300-200 são considerados leves, 200-100 como moderados e abaixo de 100 como graves38. Aqui, nos concentramos em dois grupos: pacientes com SDRA leve e moderada (ver Tabela 1).
NOTA: O estudo envolve o uso de soro obtido de amostras de sangue para realizar metabolômica baseada em RMN. Neste estudo, os participantes incluídos para referência tinham idade média de 42 anos (± 10,9 anos), com distribuição por sexo de 2 homens e 6 mulheres.
| Categoria | Relação P/F |
| Moderada | 300-200 |
| Moderado | 200-100 |
| Grave | 100-0 |
Tabela 1: Categorização dos pacientes com SDRA.
2. Seleção, coleta e processamento de amostras
- Realize o isolamento do soro seguindo as etapas descritas abaixo.
- Para experimentar, colete 2 mL de amostras de sangue das artérias usando uma agulha estéril em um frasco simples (sem aditivos) e estéril. Deixe a amostra coagular por 30 min em temperatura ambiente. Para este estudo, foram selecionados 4 pacientes com diagnóstico de SDRA leve e 4 pacientes com SDRA moderada.
- Centrifugue a amostra a 3100 x g por 15 min para separar o soro25. Transfira o soro para tubos de microcentrífuga estéreis, fazendo alíquotas de menor volume (ou seja, 400-500 μL), rotule-as adequadamente (nome do paciente, número CR, ponto do dia, etc.) e armazene-as a -80 ° C para análise futura.
NOTA: Neste ponto, o experimento pode ser pausado e retomado em um momento conveniente.
- Processe a amostra de soro antes dos experimentos de RMN usando as etapas a seguir.
- Descongele a amostra antes de realizar os experimentos de RMN. Misture 250 μL de alíquota do soro com 250 μL de solução tampão fosfato salino (contendo 100% D2O, 0,9% NaCl, 50 mM tampão fosfato de sódio, pH 7,4) para minimizar a variação do pH.
- Vortex a mistura por alguns segundos (~ 15 s) para garantir que esteja homogeneamente misturada. Por fim, transferir a amostra preparada para um tubo de RMN limpo com inserto coaxial contendo propionato de trimetilsilil (TSP) na concentração de 0,05 mM, que serve como padrão externo para calibração e quantificação41.
NOTA: Para referência, é utilizado um composto inerte e não reativo que não interage com os analitos. Outros compostos de referência comumente usados incluem tetrametilsilano (TMS) e trimetilsililpropanossulfonato de sódio (DSS).
3. Experimentação de RMN
NOTA: O foco está na identificação de pequenas moléculas nas amostras de soro, portanto, usamos a sequência de pulso Carr-Purcell-Meiboom-Gill (CPMG), que suprime os sinais das macromoléculas. Todas as amostras de soro neste estudo foram registradas usando um espectrômetro de RMN de 800 MHz equipado com uma cabeça de sonda inversa de banda larga TCI de ressonância tripla de 5 mm resfriada criogenicamente e gradiente z blindado.
- Coloque a amostra no ímã. Escreva o comando wrpa (mencione o número do experimento) e pressione enter para configurar um experimento de prótons com o programa de pulso cpmg. Mencione o nome do paciente e outros detalhes necessários clicando na opção Título.
- Bloqueie o campo magnético escrevendo o comando Bloquear, pressione enter e selecione a opção 90% H2O e 10% D2O mais. Ajuste e combine a sonda manualmente por ATMM para otimizar a eficiência dos pulsos de radiofrequência e maximizar a sensibilidade. Execute o calço de gradiente 1D clicando em topshim.
- Defina os seguintes parâmetros de aquisição, incluindo um atraso de relaxamento de 5 s, uma largura de varredura espectral de 12 partes por milhão (ppm) e um tempo de eco de 300 μs. Todos esses parâmetros podem ser facilmente modificados selecionando a opção Parâmetros de aquisição no painel superior.
- Aplique o alargamento de linha de 0,3 Hz usando uma função de janela exponencial, coletando dados em 64.000 pontos em 128 varreduras. Esses parâmetros podem ser modificados da mesma forma selecionando a opção Parâmetros de aquisição no painel superior. Ao clicar em Parâmetros de aquisição, uma lista de especificações aparecerá, e os atributos mencionados, juntamente com outros parâmetros, podem ser modificados durante a otimização.
- Adquira os espectros de RMN usando ferramentas de processamento e análise de RMN. Assim que os espectros finais forem obtidos, escreva o comando apk e absn e pressione enter para realizar a correção de fase e a correção da linha de base, respectivamente. Clique na opção Calibrar eixo presente no painel superior e, em seguida, calibre o pico do TSP para 0 ppm automática ou manualmente.
- Transfira os dados adquiridos do sistema para a estação de trabalho, onde são feitos processamento e análise adicionais.
NOTA: A fase experimental está concluída e pode ser pausada. O pré-processamento, análise e interpretação de dados podem ser realizados convenientemente.
4. Pré-processamento de dados
- Realize o pré-processamento de dados usando ferramentas de processamento e análise de RMN e software de quantificação de metabólitos de RMN.
- Às vezes, apenas a correção automática não é suficiente e, nesse caso, a correção manual também é necessária. Para realizar a correção de fase manualmente, clique na opção Processar na barra de menu superior das ferramentas de processamento e análise de RMN. Em seguida, clique em Ajustar fase, arraste o mouse e observe até que os espectros sejam corrigidos de fase. Feito isso, clique na opção Salvar e Devolver (presente em forma de ícone) disponível na mesma barra de menu.
- Para realizar a correção manual da linha de base (método Whittaker), abra o arquivo espectral no módulo processador do software de quantificação de metabólitos de RMN. Selecione a opção Correção de linha de base. Em seguida, selecione a opção Método Whittaker. Agora coloque os pontos na base dos picos em todo o espectro (0,0 a 10 ppm geralmente no caso do soro ou de acordo com a disponibilidade do metabólito nos espectros) para ajustar a base. Depois que todos os arquivos espectrais tiverem sido corrigidos na linha de base, salve-os em uma única pasta. O processamento posterior, usando o processador ou o módulo profiler do software de quantificação de metabólitos de RMN, pode ser realizado usando o mesmo arquivo salvo no formato específico.
- Execute o binning usando o módulo profiler do mesmo software. Selecione a opção Ferramentas, Processo em lote e, em seguida, Agrupamento espectral.
- Para gerar a planilha de compartimentalização final a ser usada para análise estatística, selecione a pasta que contém todos os arquivos espectrais corrigidos da linha de base após clicar em Compartimentalização espectral.
- Divida os espectros em um número definido de caixas de tamanhos iguais com uma largura espectral definida, variando de 0,01 a 0,04 ppm (pode ser otimizado de acordo com o estudo). Esse processo simplifica e organiza os dados espectrais, facilitando uma análise mais consistente. Especifique o tamanho do bucket junto com os valores de ppm inicial e final e designe a pasta onde as planilhas de compartimentalização de saída serão salvas.
- Exclua as regiões de deslocamento químico correspondentes à água e ao solvente TSP (4,8-5,2 e 0,0-0,7 ppm) para evitar interferência espectral 42,43,44.
- Esta planilha contém os nomes de amostra em uma linha e os diferentes valores de compartimento, juntamente com ppm nas outras linhas. Antes de usar esta planilha para análise estatística, modifique-a organizando as amostras em grupos para análise e salvando-as apenas no formato delimitado por vírgulas (CSV).
NOTA: Por exemplo, ao comparar o grupo leve com o grupo moderado de pacientes com SDRA, organizamos os dados com rótulos leves e moderados, adicionamos um número de série e salvamos o arquivo no formato CSV.
5. Análise estatística
NOTA: A planilha de compartimentalização obtida na etapa anterior serve como arquivo de entrada para análise estatística. Neste estudo, foi utilizado o módulo de análise estatística (um fator) do software de análise estatística metabolômica.
- Antes da análise estatística, execute a etapa de normalização usando soma, log e Pareto, mas pode variar em diferentes estudos45.
- Realizar análises univariadas e multivariadas para distinguir os diferentes grupos e determinar a precisão dos metabólitos identificados.
- Realize análise de componentes principais (PCA), análise discriminante de mínimos quadrados parciais (PLS-DA) e análise discriminante de mínimos quadrados parciais ortogonais (OPLS-DA) para observar a discriminação entre os grupos que produzem pontuações de importância variável de projeção (VIP) para cada metabólito responsável pela diferença no perfil metabólico.
- Realizar métodos univariados, como o teste t de Student e ANOVA, para identificar os metabólitos significativos com base nos critérios de ter um valor de p menor que 0,05. Ambos os testes produzem gráficos de bigode de caixa mostrando a diferença na concentração relativa dos metabólitos.
- Realize a análise de biomarcadores, que produz uma área sob o gráfico da curva característica de operação do receptor (AUROC). O valor da AUC (área sob a curva) > 0,8 é considerado significativo46.
- Selecione a lista final de metabólitos significativos usando os critérios pontuação VIP>1, valor de p corrigido por Bonferroni<0,0544 e AUC > 0,846.
- Identifique os metabólitos conforme descrito abaixo.
- O binning fornece metabólitos significativos na forma de valores de ppm em vez de nomes. Identifique os metabólitos correspondentes a valores específicos de ppm usando bancos de dados como o Banco de Ressonância Magnética Biológica (BMRB)47e o Banco de Dados de Metaboloma Humano (HMDB).
- Além desses, use a literatura publicada anteriormente42,44 para identificação e validação.
- Use o módulo profiler do software de quantificação de metabólitos de RMN para confirmar os metabólitos identificados com seus valores de ppm.
6. Quantificação de metabólitos usando software de quantificação de metabólitos de RMN
NOTA: O módulo profiler do software é amplamente utilizado para quantificar os metabólitos identificados.
- Antes de iniciar a quantificação, calibre o composto de referência selecionado para o estudo (tsp usado neste estudo) usando o módulo do processador. Clique na opção Calibrar CSI (indicador de forma química). Mencione a concentração do composto de referência (0,05 mM para este estudo). Em seguida, arraste o pico do software para o pico de referência espectral e ajuste sua altura e largura corretamente. Assim que os picos de referência espectral se ajustarem ao pico do software, clique em Aceitar.
- Abra os espectros desejados no módulo profiler e selecione a biblioteca de compostos apropriada para estudo.
- Na parte inferior da tela, uma lista de metabólitos é exibida. Selecione o metabólito de interesse nesta lista. Uma vez selecionado, o possível ppm do metabólito escolhido aparecerá no canto superior do espectro.
- Selecione o valor de Ppm no canto superior e amplie os espectros para a escala específica de ppm do metabólito.
- Neste ponto, dois picos aparecerão na tela: um representando os dados gravados e o outro, exibido como uma linha pontilhada, representando o pico do software. Alinhe os dois picos. Arraste o pico do software e ajuste sua altura e posição para corresponder ao pico presente na amostra.
- Uma vez devidamente alinhada, a concentração do metabolito pode ser obtida a partir do valor apresentado na rubrica concentração em mM para esse metabolito (figura 1).
- Siga o mesmo procedimento para todos os metabólitos e amostras e exporte os valores quantificados em uma planilha. Para gerar o arquivo de saída, clique na opção Operações em lote no menu Ferramentas . Especifique a pasta desejada para salvar o arquivo de saída.
7. Análise de vias
NOTA: Os metabólitos significativos identificados após a análise são utilizados para determinar as principais vias que influenciam diretamente o resultado em grupos de doenças. O módulo de análise de vias do software de análise estatística metabolômica e o banco de dados KEGG são geralmente empregados para esse fim.
- Insira a lista obtida de metabólitos significativos no módulo. Uma lista contendo os nomes dos caminhos é gerada.
- Selecione as vias finais com base em um critério de ter um fator de impacto maior que 0,1. Valores de impacto significativos indicam vias relacionadas à doença30.
- Analise minuciosamente as vias resultantes para elucidar a desregulação associada à gravidade da doença. Um dos deméritos desse método é sua ineficiência em identificar as vias desreguladas quando os metabólitos significativos são menores que 3.
- Para lidar com o mesmo, o metabólito identificado e sua correlação com a gravidade da doença também podem ser determinados usando a literatura publicada anteriormente. Faça uma pesquisa bibliográfica completa e observe se o metabólito identificado também está seguindo a mesma tendência de acordo com seu interesse.
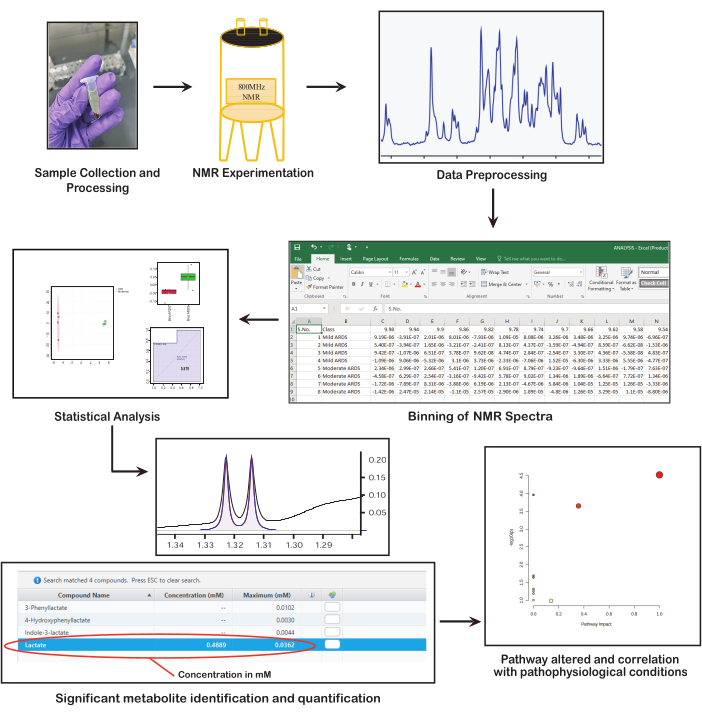
Figura 1: Etapas fundamentais na metabolômica baseada em RMN. A figura apresenta as principais etapas da metabolômica baseada em RMN: coleta e preparação de amostras, realização de espectroscopia de RMN, pré-processamento de dados, realização de análises estatísticas, identificação e quantificação de metabólitos e interpretação do significado biológico. Clique aqui para ver uma versão maior desta figura.
Resultados
Para realizar um estudo metabolômico, é essencial determinar o tamanho da amostra e os grupos específicos que serão analisados. A seleção de um tamanho amostral adequado é essencial para a obtenção de resultados significativos que se correlacionem com precisão com a gravidade da doença48. No entanto, neste trabalho em particular, usamos um pequeno tamanho de amostra para demonstrar as etapas envolvidas na identificação e quantificação de metabólito...
Discussão
A metabolômica identifica e quantifica com eficiência os metabólitos, visando os ciclos metabólicos que se tornam perturbados durante a doença. A qualidade dos resultados depende da execução meticulosa de cada etapa da abordagem metabolômica. Cada estágio, desde a seleção e coleta da amostra até a identificação da via, é fundamental para identificar com precisão os principais fatores que contribuem para a doença. Antes de realizar a metabolômica, uma revisão completa d...
Divulgações
Os autores declaram não haver interesse financeiro conflitante.
Agradecimentos
A AS agradece à Academia de Pesquisa Científica e Inovadora (AcSIR) pelo registro (Registro nº 10BB22A71002). A AS também agradece à Organização de Pesquisa e Desenvolvimento de Defesa (DRDO) pela bolsa. Agradecemos ao Centro de Pesquisa Biomédica (CBMR) por fornecer a instalação do espectrômetro de RMN de 800 MHz e financiamento por meio do projeto interno (CBMR/IMR/0008/2021). Também agradecemos ao Departamento de Medicina Intensiva (CCM), SGPGIMS, pelo apoio constante. Reconhecemos a ajuda de muitos enfermeiros, bem como, mais importante, dos pacientes inscritos neste estudo. Este estudo foi financiado pelo projeto interno (CBMR/IMR/0008/2021) do Centro de Pesquisa Biomédica (CBMR) e pelo projeto externo (No. LSRB/01/15001/LSRB-404/PEE&BS/2023) da Organização de Pesquisa e Desenvolvimento de Defesa (DRDO).
Materiais
| Name | Company | Catalog Number | Comments |
| Centirfuge | Sigma aldrich | 3-18KS | |
| Chenomx NMR suite | NMR Suite, v9, Chenomx Inc., Edmonton, Canada | NMR metabolite quantification software | |
| Co-axial insert | Sigma aldrich | Z278513 | |
| Deuterim oxide | Sigma aldrich | 151882 | |
| Eppendorf tubes | Tarsons | 500020 | |
| Metaboanalyst | Wishart Research Group | Metabolomics statistical analysis software | |
| NMR tube | Wilmad | Z412007 | 5mm diameter |
| Pipette | Eppendorf research plus | 3123000039 | 0-100 μl |
| Sample collection vials | Tarsons cryo chill vials | 523194 | |
| Sodium azide | Sigma aldrich | S2002 | |
| Sodium chloride crystal | Sigma aldrich | S9625 | |
| Sodium phosphate dibasic | Sigma aldrich | 567550 | |
| Sodium phosphate monobasic | Sigma aldrich | S0751 | |
| Topspin 3.6.4 | Bruker | NMR processing and analysis tool | |
| Tsp salt | Sigma aldrich | 269913 |
Referências
- Rubenfeld, G. D. Confronting the frustrations of negative clinical trials in acute respiratory distress syndrome. Ann. Am. Thorac. Soc. 12 (Supplement 1), S58-S63 (2015).
- Ware, L. B., et al. Prognostic and pathogenetic value of combining clinical and biochemical indices in patients with acute lung injury. Chest. 137 (2), 288-296 (2010).
- Johnson, C. H., Julijana, I., Gary, S. Metabolomics: beyond biomarkers and towards mechanisms. Nat Rev Mol Cell Biol. 17 (7), 451-459 (2016).
- Patti, G. J., Oscar, Y., Gary, S. Metabolomics: the apogee of the omics trilogy. Nat Rev Mol Cell Biol. 13 (4), 263-269 (2012).
- Nicholson, J. K., John, C. L. Metabonomics. Nature. 455 (7216), 1054-1056 (2008).
- Schnackenberg, L. K., Beger, R. D. Monitoring the health to disease continuum with global metabolic profiling and systems biology. Pharmacogenomics. 7 (7), 1077-1086 (2006).
- Pandey, S., Siddiqui, M. A., Azim, A., Sinha, N. Metabolic fingerprint of patients showing responsiveness to treatment of septic shock in intensive care unit. Magn Reson Mater Phys Biol Med. 36 (4), 659-669 (2023).
- Mamas, M., Dunn, W. B., Neyses, L., Goodacre, R. The role of metabolites and metabolomics in clinically applicable biomarkers of disease. Arch. Toxicol. 85, 5-17 (2011).
- Dunn, W. B., Broadhurst, D. I., Atherton, H. J., Goodacre, R., Griffin, J. L. Systems level studies of mammalian metabolomes: the roles of mass spectrometry and nuclear magnetic resonance spectroscopy. Chem Soc Rev. 40 (1), 387-426 (2011).
- Dettmer, K., Bruce, D. H. Metabolomics--a new exciting field within the" omics" sciences. Environ Health Perspect. 112 (7), A396-A397 (2004).
- Goodacre, R., Vaidyanathan, S., Dunn, W. B., Harrigan, G. G., Kell, D. B. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 22 (5), 245-252 (2004).
- Lindon, J. C., Nicholson, J. K., Holmes, E., Everett, J. R. Metabonomics: metabolic processes studied by NMR spectroscopy of biofluids. Concepts Magn Reson Educ J. 12 (5), 289-320 (2000).
- Bernini, P., et al. Individual human phenotypes in metabolic space and time. J Proteome Res. 8 (9), 4264-4271 (2009).
- Nicholson, J. K., et al. Metabolic phenotyping in clinical and surgical environments. Nature. 491 (7424), 384-392 (2012).
- Vinayavekhin, N., Homan, E. A., Saghatelian, A. Exploring disease through metabolomics. ACS Chem Biol. 5 (1), 91-103 (2010).
- Dunn, W. B., Ellis, D. I. Metabolomics: current analytical platforms and methodologies. TrAC Trends Anal Chem. 24 (4), 285-294 (2005).
- Dunn, W. B., Bailey, N. J. C., Johnson, H. E. Measuring the metabolome: current analytical technologies. Analyst. 130 (5), 606-625 (2005).
- Bingol, K. Recent advances in targeted and untargeted metabolomics by NMR and MS/NMR methods. High-throughput. 7 (2), 9 (2018).
- Emwas, A. H. M. The strengths and weaknesses of NMR spectroscopy and mass spectrometry with particular focus on metabolomics research. Methods Mol Biol. 1277, 161-193 (2015).
- Markley, J. L., et al. The future of NMR-based metabolomics. Curr Opin Biotechnol. 43, 34-40 (2017).
- Marchand, J., Martineau, E., Guitton, Y., Dervilly-Pinel, G., Giraudeau, P. Multidimensional NMR approaches towards highly resolved, sensitive and high-throughput quantitative metabolomics. Curr Opin Biotechnol. 43, 49-55 (2017).
- Eghbalnia, H. R., et al. Increasing rigor in NMR-based metabolomics through validated and open source tools. Curr Opin Biotechnol. 43, 56-61 (2017).
- Vignoli, A., et al. High-throughput metabolomics by 1D NMR. Angew Chem Int Ed. 58 (4), 968-994 (2019).
- Sundekilde, U. K., Eggers, N., Bertram, H. C. NMR-based metabolomics of food. Methods Mol Biol. 2037, 335-344 (2019).
- Garcia-Perez, I., et al. Identifying unknown metabolites using NMR-based metabolic profiling techniques. Nat Protoc. , 1-30 (2020).
- Dona, A. C., et al. A guide to the identification of metabolites in NMR-based metabonomics/metabolomics experiments. Comput Struct Biotechnol J. 14, 135-153 (2016).
- Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N., Siddiqui, M. H. Metabolomics: an emerging potential approach to decipher critical illnesses. Biophys Chem. 267, 106462 (2020).
- McNicholas, B. A., et al. Impact of early acute kidney injury on management and outcome in patients with acute respiratory distress syndrome: a secondary analysis of a multicenter observational study. Crit. Care Med. 47 (9), 1216-1225 (2019).
- Ambruso, S. L., et al. Inter-organ communication in homeostasis and disease: Lung metabolomics after ischemic acute kidney injury reveals increased oxidative stress, altered energy production, and ATP depletion. Am J Physiol Lung Cell Mol Physiol. 321 (1), L50 (2021).
- Viswan, A., et al. Metabolomics based predictive biomarker model of ARDS: A systemic measure of clinical hypoxemia. PloS One. 12 (11), e0187545 (2017).
- Metwaly, S. M., Brent, W. W. Systems biology ARDS research with a focus on metabolomics. Metabolites. 10 (5), 207 (2020).
- Stringer, K. A., et al. Metabolic consequences of sepsis-induced acute lung injury revealed by plasma 1H-nuclear magnetic resonance quantitative metabolomics and computational analysis. Am J Physiol Lung Cell Mol Physiol. 300 (1), L4-L11 (2011).
- Singh, A., Siddiqui, M. A., Pandey, S., Azim, A., Sinha, N. Unveiling Pathophysiological Insights: Serum Metabolic Dysregulation in Acute Respiratory Distress Syndrome Patients with Acute Kidney Injury. J Proteome Res. 23 (10), 4216-4228 (2024).
- Slupsky, C. M. NMR-based analysis of metabolites in urine provides rapid diagnosis and etiology of pneumonia. Biomark Med. 4 (2), 195-197 (2010).
- Siddiqui, M. A., et al. NMR spectroscopy-based analysis of gallstones of cancerous and benign gallbladders from different geographical regions of the Indian subcontinent. Plos One. 18 (6), e0286979 (2023).
- Li, J., et al. 1HNMR-based metabolomic profile of rats with experimental acute pancreatitis. BMC Gastroenterol. 14, 1-7 (2014).
- Mao, H., et al. Systemic metabolic changes of traumatic critically ill patients revealed by an NMR-based metabonomic approach. J Proteome Res. 8 (12), 5423-5430 (2009).
- Stringer, K. A., Jones, A. E., Puskatich, M. A., Karnovsky, A., Serkova, N. J. 1H-nuclear magnetic resonance (NMR)-detected lipids associated with apoptosis differentiate early acute respiratory distress syndrome (ARDS) from sepsis. C63. LUNG INJURY AND REPAIR: TWO TO TANGO. Am J Resp Crit Care Med. 189, A5000 (2014).
- Viswan, A., Singh, C., Kayastha, A. M., Azim, A., Sinha, N. An NMR based panorama of the heterogeneous biology of acute respiratory distress syndrome (ARDS) from the standpoint of metabolic biomarkers. NMR Biomed. 33 (2), e4192 (2020).
- Singh, C., et al. Metabolic profiling of human lung injury by 1 H high-resolution nuclear magnetic resonance spectroscopy of blood serum. Metabolomics. 11, 166-174 (2015).
- Pandey, S., Siddiqui, M. A., Trigun, S. K., Azim, A., Sinha, N. Gender-specific association of oxidative stress and immune response in septic shock mortality using NMR-based metabolomics. Mol Omics. 18 (2), 143-153 (2022).
- Nagana, G. A., Gowda, Y. N., Raftery, D. Expanding the limits of human blood metabolite quantitation using NMR spectroscopy. Anal Chem. 87 (1), 706-715 (2015).
- Gowda, G. A. N., Raftery, D. Quantitating metabolites in protein precipitated serum using NMR spectroscopy. Anal Chem. 86 (11), 5433-5440 (2014).
- Gowda, G. A. N., Raftery, D. NMR-based metabolomics. Adv Exp Med Biol. 1280, 19-37 (2021).
- Xia, J., Psychogios, N., Young, N., Wishart, D. S. MetaboAnalyst: a web server for metabolomic data analysis and interpretation. Nucleic Acids Res. 37 (suppl_2), W652-W660 (2009).
- orbacıoğlu, &. #. 3. 5. 0. ;. K., Aksel, G. Receiver operating characteristic curve analysis in diagnostic accuracy studies: A guide to interpreting the area under the curve value. Turk J Emerg Med. 23 (4), 195 (2023).
- Ulrich, E. L., et al. BioMagResBank. Nucleic Acids Res. 36 (suppl_1), D402-D408 (2007).
- Nyamundanda, G., Gormley, I. C., Fan, Y., Gallagher, W. M., Brennan, L. MetSizeR: selecting the optimal sample size for metabolomic studies using an analysis based approach. BMC bioinformatics. 14, 1-8 (2013).
- Beckonert, O., et al. Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma, serum and tissue extracts. Nat Protoc. 2 (11), 2692 (2007).
- Dona, A. C., et al. Precision high-throughput proton NMR spectroscopy of human urine, serum, and plasma for large-scale metabolic phenotyping. Anal Chem. 86 (19), 9887-9894 (2014).
- Ala-Korpela, M. Potential role of body fluid 1H NMR metabonomics as a prognostic and diagnostic tool. Expert Rev Mol Diagn. 7 (6), 761-773 (2007).
- Takeda, I., et al. Understanding the human salivary metabolome. NMR Biomed. 22 (6), 577-584 (2009).
- Callejon-Leblic, B. e. m., García-Barrera, T., Pereira-Vega, A., Gómez-Ariza, J. L. Metabolomic study of serum, urine and bronchoalveolar lavage fluid based on gas chromatography mass spectrometry to delve into the pathology of lung cancer. J Pharm Biomed Anal. 163, 122-129 (2019).
- Van Oort, P. M. P., et al. Exhaled breath metabolomics for the diagnosis of pneumonia in intubated and mechanically-ventilated intensive care unit (ICU)-patients. Int J Mol Sci. 18 (2), 449 (2017).
- Serkova, N. J., Niemann, C. U. Pattern recognition and biomarker validation using quantitative 1H-NMR-based metabolomics. Expert Rev Mol Diagn. 6 (5), 717-731 (2006).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation times. Rev Sci Instrum. 29 (8), 688-691 (1958).
- Xi, Y., Rocke, D. M. Baseline correction for NMR spectroscopic metabolomics data analysis. BMC bioinformatics. 9, 1-10 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados