Method Article
استراتيجية تداخل CRISPR المزدوجة لاستهداف التفاعلات المميتة الاصطناعية بين الحمض النووي الريبي غير المشفر في الخلايا السرطانية
In This Article
Summary
تقدم هذه الدراسة نظام CRISPRi مزدوج يستهدف الحمض النووي الريبي الطويل غير المشفر في خلايا الورم الميلانيني. إنه يتيح الضربة القاضية الجينية الاندماجية والفحص القاتل الاصطناعي ، وتحديد تفاعلات lncRNA الخاصة بالسرطان للاستراتيجيات العلاجية المحتملة.
Abstract
تمثل الحمض النووي الريبي الطويل غير المشفر (lncRNAs) فئة واسعة ومتنوعة وظيفيا من جزيئات الحمض النووي الريبي ، مع توقع أكثر من 100,000 في الجينوم البشري. على الرغم من أن lncRNAs أقل حفظا عبر الأنواع مقارنة بالجينات المشفرة للبروتين ، إلا أنها تلعب أدوارا حاسمة في تنظيم الجينات وتفاعلات الكروماتين وتطور السرطان. مشاركتهم في السرطان تجعلهم أهدافا علاجية واعدة. يوفر تداخل CRISPR (CRISPRi) ، باستخدام Cas9 غير النشط تحفيزيا المدمجة مع مثبط نسخي مثل KRAB-MeCP2 ، طريقة دقيقة لاستهداف lncRNAs النووية وتقييم وظائفها. تقدم هذه الدراسة نظام CRISPRi مزدوج باستخدام تقنيات CRISPRi المتعامدة من المكورات العنقودية الذهبية والمكورات العقدية المقيحة على أساس dCas9-KRAB-MeCP2 ، الأمثل للاستهداف الاندماجي ل lncRNAs في خلايا الورم الميلانيني البشري. يسهل البروتوكول الضربة القاضية الجينية الاندماجية أو الفحص القاتل الاصطناعي لأزواج lncRNA ، مما يوفر أداة جديدة لأبحاث السرطان. من خلال استكشاف الفتك الاصطناعي بين lncRNAs ، يمكن أن يساعد هذا النهج في تحديد تفاعلات lncRNA الحاسمة لبقاء الخلايا السرطانية ، وتقديم استراتيجيات علاجية جديدة. تم عرض وظائف النظام المزدوج ، مما يسلط الضوء على إمكاناته في تحديد تفاعلات lncRNA الحرجة الخاصة بالسرطان.
Introduction
على الرغم من أن أقل من 3٪ من الجينوم البشري يشفر البروتينات ، إلا أن ما يقرب من 80٪ من الجينوم يتمنسخها 1،2. من بين وحدات النسخ غير المشفرة ، يتم تصنيف عشرات الآلاف على أنها RNAs طويلة غير مشفرة (lncRNAs) تتجاوز 200 نيوكليوتيد ، ويقدر العدد الإجمالي لل lncRNAs البشرية بتجاوز 100,000 3,4. على عكس جينات الترميز ، فإن lncRNAs أقل حفظا عبر الأنواع. بالنظر إلى أن البشر يشتركون في 99٪ من جينومهم مع الرئيسيات مثل الشمبانزي ، يفترض أن lncRNAs لها تأثير أكبر بكثير على تطور النمطالظاهري 5،6. تشير هذه النتائج إلى وظائف خلوية مهمة ل lncRNAs. على الرغم من أن تنظيم lncRNAs وتفاعلاتها مع البروتينات المرتبطة بالحمض النووي الريبي والحمض النووي الريبي الآخر لا يزال غير مفهوم تماما ، ولم يتم بعد شرح العديد من lncRNAs بالكامل ، فمن الواضح أن lncRNAs تظهر أنماط تعبير خاصة بالخلايا والأنسجة في الصحة والمرض ، مثل السرطان7،8،9،10. وهي متورطة في وظائف متنوعة ، بما في ذلك تنظيم النسخ الجيني ، والمشاركة في تفاعلات الكروماتين11 ، ومعالجة الحمض النوويالريبي 12 ، وتثبيت الحمض النووي الريبي13 ، وتنظيم الترجمة14.
في السرطان ، تؤثر lncRNAs شديدة التنوع الخاصة بنوع الخلايا على تطور الورم والورم الخبيث من خلال تنظيم التعبير الجيني ، مما يسلط الضوء على إمكاناتها كأهداف علاجية قيمة15. بالإضافة إلى الكشف عن lncRNAs كمؤشرات حيوية في عينات الورم16 ، فإن استهداف lncRNAs الخاصة بالورم لتعطيل وظائفها النهائية يحمل إمكانات كبيرة في كل من التطبيقات السريرية والأبحاث الأساسية. تشمل الأساليب القائمة على الحمض النووي الريبي لتوضيح أدوار lncRNAs قليل النوكليوتيدات المضادة للمعنى (ASOs) ، والحمض النووي الريبي القصير ، والحمض النووي الريبي الصغيرة المتداخلة (siRNAs) 17،18. بينما يتم استخدام siRNA بشكل شائع لشاشات إسكات الجينات ، فإن الضربة القاضية القائمة على siRNA تقتصر على السيتوبلازم19. ومع ذلك ، يعمل lncRNA بشكل متكرر داخل النواة.
بدلا من ذلك ، يمكن استخدام تداخل التكرارات المتجانسة القصيرة العنقودية المتباعدة بانتظام (CRISPRi) لتثبيط lncRNAs في السرطانات البشرية20. علاوة على ذلك ، يمكن برمجة شاشات CRISPRi على مستوى الجينوم بسهولة واستهداف مجموعة واسعة من الجينات المشفرة وغير المشفرة لفحص تأثيرها الوظيفي21،22. في CRISPRi ، يتم دمج Cas9 (dCas9) الناقص التحفيزي في مجال مثبط النسخ ، مثل المجال23 للصندوق المرتبط ب Krüppel (KRAB). يتم توجيه قمع الجينات بواسطة dCas9-KRAB بواسطة دليل الحمض النووي الريبي (gRNA) إلى المنطقة ذات الاهتمام. يتحكم CRISPRi في الجينات على مستويات الحمض النووي ، مما يؤدي إلى كفاءة أعلى وأنماط ظاهرية مطلوبة لفقدان الوظيفة على عكس تداخل الحمض النووي الريبي ، النشط في مستوى ما بعد النسخ24. استجابة للفعالية المحدودة ل KRAB في إسكات الهدف ، تم إدخال اندماج KRAB و MeCP2 مع dCas9 كاستراتيجية قمع أكثر فعالية25.
على الرغم من أن إسكات الجين الفردي قد يؤثر على صلاحية السرطان ، إلا أن التفاعلات المميتة المزدوجة أو المتعددة الاصطناعية يمكن أن تنقذ الخلايا السرطانية من موتالخلايا 26. يتضمن الفتك الاصطناعي جينين أو أكثر ، يمكن لكل منهما تعويض عن وظيفة الآخر. للتغلب على المشكلات المتعلقة بشاشات الضربة القاضية أحادية الجين ، تقدم استراتيجيات كريسبر المزدوجة التي تستهدف أزواج الجينات المميتة لترميز البروتين نهجا واعدا27.
هنا ، نقدم بروتوكولا للاستخدام الاندماجي للاستهداف المتعامد القائم على CRISPRi ل lncRNA أو أزواج الحمض النووي الريبي الأخرى غير المشفرة باستخدام المكورات العنقودية الذهبية والمكورات المقيحة dCas9 المدمجة في KRAB-MeCP2 في خلايا الورم الميلانيني البشري (انظر الشكل 1). يمكن استخدام البروتوكول للضربة القاضية الكلاسيكية التوافقية CRISPR أو كفحص قائم على CRISPRi للأزواج المميتة الاصطناعية في السرطان. يسمح استخدام نوعين مختلفين من dCas9 ، SpCas9 و SaCas9 ، في نهج CRISPRi بالاستهداف المستقل للمواقع الجينومية المتميزة مع الحد الأدنى من التفاعل المتبادل ، مما يعزز الخصوصية والمرونة مع ضمان انتقائية عالية على الهدف. يتم استخدام زخارف مختلفة متجاورة للفاصل الأولي (PAMs): NGG ل SpCas9 و NNGRRT ل SaCas9. يعالج نظام dCas9 المزدوج تحديات مثل التوافق المحدود مع sgRNA من خلال تمكين التعديل الدقيق والمتزامن وتقليل المنافسة المعقدة sgRNA-RNP عند استخدام نوع واحد من dCas9. يعمل هذا الابتكار على تحسين متانة وتعدد استخدامات فحص مكتبة sgRNA المزدوجة. في الختام ، نحن نقدم نظام CRISPRi مزدوج يعمل بكامل طاقته في خلايا الورم الميلانيني كنموذج للسرطان.
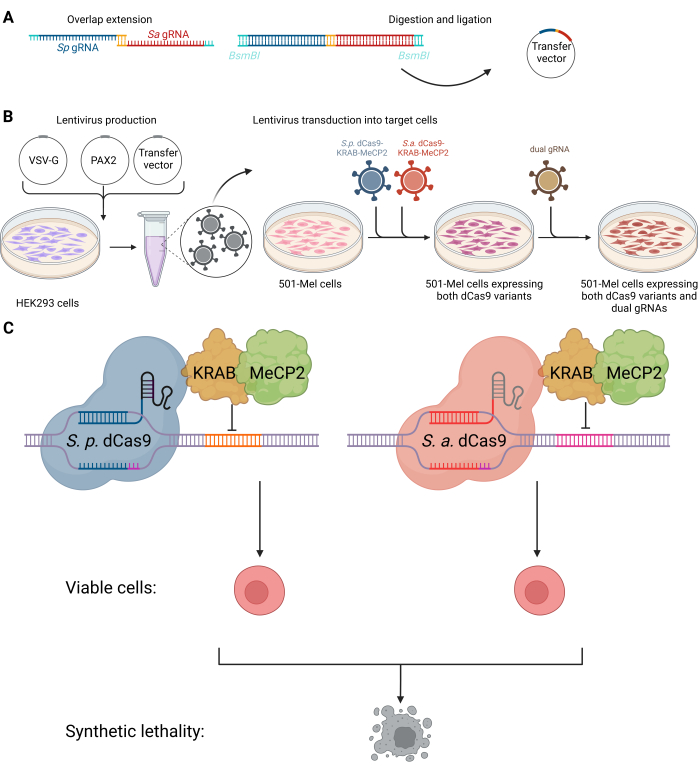
الشكل 1: مخطط نظام CRISPRi المزدوج لاستهداف التفاعلات المميتة الاصطناعية للحمض النووي الريبي غير المشفر. (أ) إجراء استنساخ ناقل الحمض النووي الريبي المزدوج. (ب) تم نقل خلايا HEK293 باستخدام بلازميد المغلف VSV-G ، وتغليف البلازميد PAX2 ، وناقل النقل لإنتاج فيروسات العدسات للنقل اللاحق إلى خلايا 501-MEL. تم دمج فيروسات العدسات التي تحتوي على المعلومات الجينية ل SpdCas9-KRAB-MeCP2 (أزرق) و SadCas9-KRAB-MeCP2 (أحمر) في خلايا 501-mel في وقت واحد. بعد اختيار المضادات الحيوية ، تم إجراء نقل ثان للفيروسات لدمج gRNAs المزدوجة المطلوبة (أخضر زيتوني). (ج) تتفاعل خلايا 501-mel التي تعبر عن متغيرات dCas9 مع gRNAs المقابلة لها لإسكات التعبير الجيني المستهدف ، مما يؤدي إلى موت الخلايا السرطانية. تم إنشاؤه باستخدام BioRender.com. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
تم توفير خط خلايا الورم الميلانيني البشري 501-ميل (RRID: CVCL_4633) من قبل مختبر Aifantis (جامعة نيويورك). تم شراء خط خلايا Lenti-X 293T HEK من Takara Bio. تمت زراعة هذه الخطوط الخلوية في وسط النسر المعدل (DMEM) عالي الجلوكوز المكمل بمصل بقري الجنين بنسبة 10٪ عند 37 درجة مئوية في جو 5٪ من ثاني أكسيد الكربون2 في ظروف معقمة. تم تمرير الخلايا حتى وصلت إلى 80٪ -90٪ التقاء. تم اختبار خطوط الخلايا بانتظام بحثا عن تلوث الميكوبلازما.
1. تصميم gRNAs باستخدام منصة تصميم gRNA عبر الإنترنت
- صمم gRNA واحدا خاصا ب SpCas9 و gRNA واحد خاص ب SaCas9 لكل lncRNA مستهدف لتقييم المتغير الذي ينتج عنه نتائج ضربة قاضية فائقة.
- استهدف lncRNAs المطلوبة ، هنا RP11-120D5.1 (RP11) و XLOC_030781 (XLOC) ، باستخدام gRNA الأكثر كفاءة لكل S. pyogenes (gRNA-sp) Cas9 و S. aureus (gRNA-sa) Cas9.
ملاحظة: يمكن استخراج المكورات الحمضة المرسوة مقيوجينات gRNAs من Petroulia et al.28. - اتبع التوصيات الخاصة بالموقع لتحديد أولويات أعلى نشاط على الهدف، معبرا عنها بدرجات عالية على الهدف، مع تقليل التطابقات خارج الهدف كما هو مقترح بواسطة أدوات تصميم gRNA.
- اختر الجينوم GRCh38 (زئبق ، الإنسان العاقل). حدد موقع PAM ل SpCas9 (NGG) و SaCas9 (NNGRRT) المحددين.
- استخدم طريقة التسجيل خارج الهدف من Hsu et al.29. ضع gRNAs داخل نافذة من -150 نقطة أساس إلى +50 نقطة أساس حول موقع بدء النسخ للحصول على الأداء الأمثل.
- اختر gRNAs ذات أعلى درجة على الهدف وخارج الهدف (انظر الجدول التكميلي 1).
- اجمع بين gRNA غير مستهدف أو مختلط للتحكم ، هنا ضد Rosa26 ، مع كل gRNA مستهدف لإنشاء ناقل gRNA مزدوج ، يستهدف lncRNA واحدا. يتيح الجمع بين gRNAs المستهدفة في بلازميد واحد التعديل الخاص بالموقع لكلا الموقعين المستهدفين.
ملاحظة: يتيح gRNA المزدوج الذي يأوي ناقلات gRNA وgRNA المستهدف ضربة قاضية ل lncRNA مستهدف واحد ، بينما تسمح نواقل gRNA المزدوجة التي تحتوي على gRNAs التي تستهدف المواقع بالضربة القاضية المتزامنة ل lncRNAs المستهدفة.
2. استنساخ الحمض النووي الريبي
- قم بإجراء تمديد متداخل PCR باستخدام جزأين من الحمض النووي يحتوي على تسلسل gRNA. استخدم التمهيدي الأمامي U6 والتمهيدي العكسي H1 (انظر الجدول التكميلي 2) للتضخيم كما هو موضح من قبل نجم وآخرون 27.
- قم بإعداد تفاعل تفاعل البوليميراز المتسلسل بسعة 50 ميكرولتر مع 2 × مزيج رئيسي عالي الدقة تفاعل البوليميراز المتسلسل مع المخزن المؤقت HF و 1 ميكرولتر من كل جزء من أجزاء الحمض النووي التي تحتوي إما على المكورات القاحية أو المكورات العنقودية الذهبية gRNA. قم بتشغيل التفاعل في ظل ظروف التدوير التالية: التمسخ الأولي عند 98 درجة مئوية لمدة 30 ثانية ، متبوعا ب 15 دورة من التمسخ عند 98 درجة مئوية لمدة 5 ثوان ، والتلدين عند 55 درجة مئوية لمدة 10 ثوان ، والاستطالة عند 72 درجة مئوية لمدة 15 ثانية.
- أضف 2.5 ميكرولتر من التمهيدي الأمامي U6 و 2.5 ميكرولتر من التمهيدي العكسي H1 بعد الانتهاء من الدورات ال 15 الأولية ، وأخضع التفاعل إلى 20 دورة إضافية في نفس الظروف ، مع تمديد نهائي لمدة 5 دقائق عند 72 درجة مئوية.
- قم بتنقية منتجات PCR باستخدام مجموعة تنقية PCR وفقا لتعليمات الشركة المصنعة.
- هضم 1 ميكروغرام من البلازميد المعدل باستخدام 1 ميكرولتر من إنزيم التقييد BsmB1 و 1 ميكرولتر من 10x عازلة لمدة 30 دقيقة عند 37 درجة مئوية في تفاعل 10 ميكرولتر.
- استنساخ منتجات تفاعل البوليميراز المتسلسل في بلازميد gRNA-Zeo-GFP المزدوج المهضوم (انظر الشكل التكميلي 1 والملف التكميلي 1) باستخدام بروتوكول تفاعل تجميع الحمض النووي HiFi كما هو موضح من قبل الآخرين30.
- قم بتحويل خلايا الإشريكية القولونية المختصة كيميائيا عن طريق خلط 50 ميكرولتر من زراعة البكتيريا مع 4 ميكرولتر من منتج التجميع وفقا لتعليمات الشركة المصنعة.
3. إنتاج فيروس العدسي
تنبيه: تعامل مع فيروسات العدسات النشطة في جميع الخطوات مع اتباع إرشادات السلامة المناسبة. قم دائما بإجراء العمل مع الفيروسات في خزانة أمان من الفئة 2 لمنع المخاطر من تكوين الهباء الجوي الذي لا مفر منه. قم بإجراء جميع الأعمال خارج خزانة الأمان ، مثل الطرد المركزي ، في حاويات ودوارات محكمة الغلق معتمدة للاستخدام مع كائنات مجموعة المخاطر 2. ارتد ملابس واقية مناسبة ، بما في ذلك معطف المختبر والقفازات التي تستخدم لمرة واحدة ونظارات السلامة ، في جميع الأوقات في منطقة العمل. يجب معالجة أسطح العمل بمحلول مطهر ، ويجب أيضا معالجة النفايات الأخرى بمحلول مطهر ، مما يسمح لها بالبقاء في الخزانة المغلقة تحت ضوء الأشعة فوق البنفسجية لمدة 240 دقيقة على الأقل. يجب شطف الماصات المصلية وأطراف الماصات المستخدمة في محلول مطهر قبل التخلص منها في زجاجات قابلة للتعقيم. بعد ذلك ، يجب التخلص من النفايات في حاوية النفايات S2. سيتم تعطيل نفايات S2 من خلال التعقيم. يمكن التعامل مع خطوط الخلايا المنقولة في ظل ظروف S1 في موعد لا يتجاوز يومين بعد التنبيغ وفقط بعد تغييرين كاملين على الأقل في وسط الاستزراع بوسط خال من الفيروسات.
- عد خلايا Lenti-X 293T HEK عن طريق خلط 10 ميكرولتر من الخلايا مع 10 ميكرولتر من محلول Trypan blue في مقياس كثافة الدم. لوحة 4 × 106 خلايا HEK من الوتر X 293T لكل لوحة 10 سم لتحقيق ~ 80٪ التقاء في اليوم التالي.
- العمل في مختبر الفئة 2 من هذه الخطوة فصاعدا. امزج 500 ميكرولتر من وسط تعداء مناسب مع ناقل نقل 11.25 ميكروغرام SpdCas9-KRAB-MeCP2 (انظر الملف التكميلي 2) ، و 5.5 ميكروغرام من بلازميد المغلف VSV-G ، و 16.5 ميكروغرام pPAX2 vector. احتضن لمدة 5 دقائق في درجة حرارة الغرفة. بالنسبة للمتغير dCas9 الثاني ، استخدم متجه النقل SadCas9-KRAB-MeCP2 (انظر الملف التكميلي 3).
- في أنبوب منفصل ، اخلطي 500 ميكرولتر من وسط المصل المخفف مع 36 ميكرولتر من كاشف PEI (المخزون: 1 مجم / مل). احتضن لمدة 5 دقائق في درجة حرارة الغرفة.
- يمزج الخلطات ويحتضن لمدة 15 دقيقة في درجة حرارة الغرفة. يضاف الخليط بعناية إلى وسط الخلية (6 مل) ويحتضن طوال الليل عند 37 درجة مئوية و 5٪ ثاني أكسيدالكربون 2. تأكد من بقاء الخلايا متصلة لتجنب انخفاض إنتاجية الفيروسات العسسية.
- تخلص من الوسيط في زجاجة نفايات قابلة للتعقيم باستخدام ماصة مصلية بعد 12-15 ساعة. من الآن فصاعدا، اشطف المواد الاستهلاكية، بما في ذلك الماصات المصلية وأطراف الماصة، بمحلول مطهر قبل التخلص منها في نفايات الأوتوكلاف. أضف 5 مل من وسط DMEM الطازج إلى الخلايا المنقولة باستخدام ماصة جديدة.
- حصاد فيروسات العدسات عن طريق نقل المادة الطافية باستخدام ماصة مصلية إلى أنبوب كنسي سعة 50 مل بعد 48 ساعة من التعداد. قم بتخزين الأنبوب الكنسي في الثلاجة. أضف 5 مل من الوسط الطازج إلى الخلايا واحتضن الخلايا لمدة 24 ساعة أخرى.
- كرر جمع فيروسات العدسات عن طريق نقل المادة الطافية إلى نفس الأنبوب الكنسي سعة 50 مل الذي يحتوي على الحصاد الأول. مرة أخرى ، أضف 5 مل من الوسط الطازج ، واحتضنه لمدة 24 ساعة ، وانقل المادة الطافية بعد ذلك إلى الأنبوب الكنسي سعة 50 مل. قم بتخزين المواد الطافية المجمعة من الحصاد الأول والثاني والثالث في الأنبوب الكنسي سعة 50 مل عند 4 درجات مئوية حتى الترشيح والطرد المركزي.
- جهاز الطرد المركزي ، المادة الطافية المركبة لمدة 5 دقائق عند 500 × جم و 4 درجات مئوية في حاويات ودوارات محكمة الغلق بالهباء الجوي ، معتمدة للاستخدام مع كائنات المجموعة 2 من أجل حبيبات الخلايا المنفصلة والحطام. قم بتصفية المادة الطافية من خلال مرشحات الحقنة المعقمة من فئة زراعة الخلايا 0.2 ميكرومتر.
- تركيز المادة الطافية المفلترة عند 1,000 × جم و 4 درجات مئوية باستخدام وحدة مرشح الطرد المركزي مع قطع مناسب للوزن الجزيئي من 100 كيلو دالتون إلى حوالي 500 ميكرولتر.
4. نقل الفيروسات العدسية واختيار الخلايا المنقولة المستقرة
تنبيه: قم دائما بإجراء العمل مع الفيروسات في خزانة أمان من الفئة 2.
- صفيحة 501-mel الخلايا في صفيحة 6 آبار في 2 × 105 خلايا / بئر مع حجم ثقافة نهائي 2 مل ، قبل يوم واحد من النقل.
- يستعاض عن وسط الاستزراع ب 2 مل من وسط DMEM المسخن مسبقا المكمل بالبوليبرين بتركيز نهائي قدره 6 ميكروغرام/مل.
- من الآن فصاعدا ، مطلوب العمل في مختبر الفئة 2. أضف 50 ميكرولتر من فيروسات العدسات التي تحتوي على SpdCas9-KRAB-MeCP2 و SadCas9-KRAB-MeCP2 في وقت واحد إلى الخلايا. اشطف المواد الاستهلاكية، بما في ذلك الماصات المصلية وأطراف الماصة، بمحلول مطهر قبل التخلص منها في نفايات الأوتوكلاف. رج الطبق برفق واحتضانه عند 37 درجة مئوية في جو 5٪ CO2 .
- تخلص من الوسط في زجاجة قابلة للتعقيم باستخدام ماصة مصلية وأضف 2 مل من الوسط الطازج الذي يحتوي على بلاستيسيدين (10 ميكروغرام / مل) وبورومايسين (2 ميكروغرام / مل) بعد 16 ساعة من النقل.
- استمر في الاختيار عن طريق استبدال الوسط بوسط جديد سعة 2 مل يحتوي على بلاستيسيدين (10 ميكروغرام / مل) وبورومايسين (2 ميكروغرام / مل) بعد يومين. التعامل مع الخلايا المنقولة في ظل ظروف S1 بعد 7 أيام من التنبيغ.
ملاحظة: يجب إجراء اختيار المضادات الحيوية حتى تموت جميع خلايا التحكم غير المنقولة. يمكن استخدام استراتيجيات الاختيار البديلة اعتمادا على شريط المقاومة في متجه النقل. إذا لزم الأمر، قم بتحسين تركيزات المضادات الحيوية باستخدام منحنى القتل. قد تكون هناك حاجة إلى اختيار استنساخ خلية واحدة من خلال قياس التدفق الخلوي أو التخفيف التسلسلي والطلاء ، متبوعا بقياس البروتين ، للحصول على مجموعات الخلايا التي تعبر عن اندماجات dCas9 بمستويات كافية.
5. القياس الكمي للبروتين وتحليل اللطخة الغربية
- عد خلايا 501-mel المستقرة عن طريق خلط 10 ميكرولتر من الخلايا مع 10 ميكرولتر من محلول Trypan Blue في مقياس كثافة الدم. أجهزة الطرد المركزي 1 × 106 من الخلايا لمدة 5 دقائق عند 500 × جم و 4 درجات مئوية. تخلص من المادة الطافية وأعد تعليق حبيبات الخلية في 1 مل من PBS المثلج.
- كرر الطرد المركزي وأعد تعليق حبيبات الخلية في 50 ميكرولتر من المخزن المؤقت RIPA الذي يحتوي على مثبطات البروتياز. احتضن على الجليد لمدة 15 دقيقة وقم بإزالة بقايا الخلية عن طريق الطرد المركزي عند 13,000 × جم لمدة 10 دقائق عند 4 درجات مئوية. قم بتخزين المادة الطافية عند -20 درجة مئوية أو استخدمها على الفور لقياس كمية البروتين كما هو موضح من قبل الآخرين31.
- امزج 10 ميكروغرام من البروتين مع 4 × LDS المؤقت ، 50 ملي مولار DTT واملأ ما يصل إلى 20 ميكرولتر بالماء المقطر. افصل البروتينات على هلام فصل بنسبة 10٪ باستخدام SDS-PAGE القائم على Tris / glycine عند 135 فولت لمدة ساعة واحدة كما هو موضح سابقا32. انقل البروتينات إلى غشاء PVDF عند 90 فولت لمدة 90 دقيقة مع نقل الخزان الرطب.
- احتضان الغشاء في المخزن المؤقت المانع (5٪ حليب خالي الدسم في TBS-T) لمدة ساعة واحدة عند 180 دورة في الدقيقة في درجة حرارة الغرفة.
- أضف 1: 10,000 جسم مضاد أحادي النسيلة للفأر مضاد ل GAPDH في المخزن المؤقت للحجب كعنصر تحكم داخلي. أضف الجسم المضاد الأساسي المضاد Cas9 (S. pyogenes) أو مضاد Cas9 (S. aureus) على أنه تخفيف 1: 1,000 في المخزن المؤقت للحجب واحتضانه طوال الليل عند 4 درجات مئوية مع اهتزاز لطيف.
- اغسل الغشاء 3 مرات باستخدام المخزن المؤقت TBST لمدة 10 دقائق لكل منهما. احتضان بجسم مضاد ثانوي مضاد للفأر HRP (1: 10،000) لمدة ساعة واحدة في درجة حرارة الغرفة.
- اغسل 3 مرات باستخدام المخزن المؤقت TBST لمدة 10 دقائق لكل منهما. الكشف عن الإشارات باستخدام ركيزة ECL ونظام التصوير.
6. اختبار الجدوى لقمع LncRNA المزدوج
تنبيه: قم دائما بإجراء العمل مع الفيروسات في خزانة أمان من الفئة 2.
- البذور 0.1 × 105 خلايا تحتوي على SadCas9-KRAB و SpdCas9-KRAB المنقولة المستقرة بعد 7 أيام من النقل في صفيحة 96 بئر في 100 ميكرولتر من وسط DMEM الدافئ مسبقا قبل يوم واحد من التنبيغ.
- في يوم التنبيغ، يستعاض عن وسط الاستزراع ب 100 ميكرولتر من وسط DMEM المسخن مسبقا المكمل بالبوليبرين بتركيز نهائي قدره 6 ميكروغرام/مل. من الآن فصاعدا ، مطلوب العمل في مختبر الفئة 2. أضف الحجم المناسب من فيروسات العدسات إلى الخلايا.
ملاحظة: يجب فحص حجم فيروسات العدسات التي تحتوي على ناقل gRNA المزدوج باستخدام GFP لإنشاء منحنى خطي عن طريق معايرة فيروسات العدس. - استبدل الوسط بوسط جديد يحتوي على 500 ميكروغرام / مل من الزيوسين ، و 5 ميكروغرام / مل بلاستيسيدين ، و 1 ميكروغرام / مل بورومايسين بعد 16 ساعة من التنبيغ.
- قم بإجراء الكشف عن التلألؤ بعد 5 أيام من التنبيغ باستخدام مقايسة صلاحية الخلية وفقا لتعليمات الشركة المصنعة.
النتائج
تم دمج أشرطة التعبير الخاصة ب SadCas9-KRAB-MeCP2 و SpdCas9-KRAB-MeCP2 في خلايا 501-mel في وقت واحد باستخدام التنبيغ بكميات متساوية من فيروسات العدس. نظرا لأن بروتوكول CRISPRi يعتمد على التعبير عن متغيرات dCas9-KRAB-MeCP2 ، فإن تحليل اللطخة الغربية مثالي لتأكيد وجودها على مستوى البروتين. بعد تخصيب الخلايا التي تم نقلها إيجابيا ، تم جمع عينات لتحليل اللطخة الغربية لتأكيد وجود أخصائيي تقويم العظام dCas9-KRAB-MeCP2. عند التحويل الناجح ، من المتوقع وجود نطاق واحد يتوافق مع بروتين الاندماج SpdCas9-KRAB-MeCP2 (202 كيلو دالتون) وبروتين الاندماج SadCas9-KRAB-MeCP2 (170 كيلو دالتون) وتم تأكيده ، كما هو موضح في الشكل 2 أ ، باستخدام الأجسام المضادة الخاصة بأخصائيي تقويم العظام Cas9 المعنيين. ومع ذلك ، إذا لم تكن اللطخة الغربية ممكنة ، فيمكن استخدام qPCR للتحقق من بروتينات الاندماج على مستوى mRNA.
بمجرد إنشاء خلايا 501-mel التي تعبر بثبات عن المثبطات الوظيفية ، تم إجراء نقل الفيروسات العدسي الثاني باستخدام ناقل gRNA مزدوج. تبين سابقا أن RP11 و XLOC يتم تنظيمهما في الثقافات قصيرة المدى للورم الميلانيني النقيلي ، وأظهر استهداف CRISPRi الفردي تأثيرهما على التكاثر وبقاء الخلية28. وهكذا ، اخترنا زوج lncRNA هذا للتقييم باعتباره تركيبة RNA اصطناعية قاتلة غير مشفرة لإثبات المفهوم ، متوقعين نمطا ظاهريا للنمو التراكمي الذي يمكن ملاحظته بسهولة في الخلايا المعدلة.
للتحقيق في تأثيرات CRISPRi المزدوج في الخلايا السرطانية باستخدام فحوصات الجدوى ، يجب قمع كل من lncRNAs المستهدفة بشكل فعال. qPCR هي الطريقة الأنسب لتأكيد هذا القمع عن طريق قياس مستويات نسخها. قمنا بتقييم وظائف بروتوكول CRISPRi المزدوج المطور من خلال تحديد مستويات الحمض النووي الريبي وفحص صلاحية الخلية عند استهداف زوج RP11 و XLOC الذي يحتمل أن يكون قاتلا. لذلك ، تم إدخال gRNAs مختلفة تستهدف RP11 و XLOC لتقليل مستويات الحمض النووي الريبي المستهدفة. تم التحقيق في قمع الحمض النووي الريبي باستخدام gRNAs الخاصة بالهدف إما ل SadCas9-KRAB-MeCP2 أو SpdCas9-KRAB-MeCP2 ، تم اختبارها بشكل فردي ومجتمعة جنبا إلى جنب مع gRNA للتحكم الذي يستهدف Rosa26 (انظر الشكل 2 ب). على الرغم من أن gRNAs التي تستهدف XLOC أو RP11 قمعت بشكل فردي أهدافها الخاصة ، إلا أن الضربة القاضية المتزامنة لكل من XLOC و RP11 لم تتحقق إلا باستخدام زوج XLOC-sa / RP11-sp.
نظرا لأن مستويات الحمض النووي الريبي XLOC و RP11 قد تم تخفيضها بنجاح في خط الخلية 501-mel مع زوج gRNA المحدد XLOC-sa / RP11-sp ، فقد تم إجراء مقايسة جدوى قمع lncRNA المزدوج (انظر الشكل 2C). بعد 5 أيام ، كانت صلاحية الخلية 81٪ و 89٪ من التحكم في Rosa26 عندما تم استخدام gRNAs المستهدفة بشكل فردي. أدى الجمع بين RP11-sp و XLOC-sa إلى انخفاض كبير إضافي مقارنة ب RP11-sp وحده ، مع بقاء الخلية بنسبة 59٪ من التحكم في Rosa26.
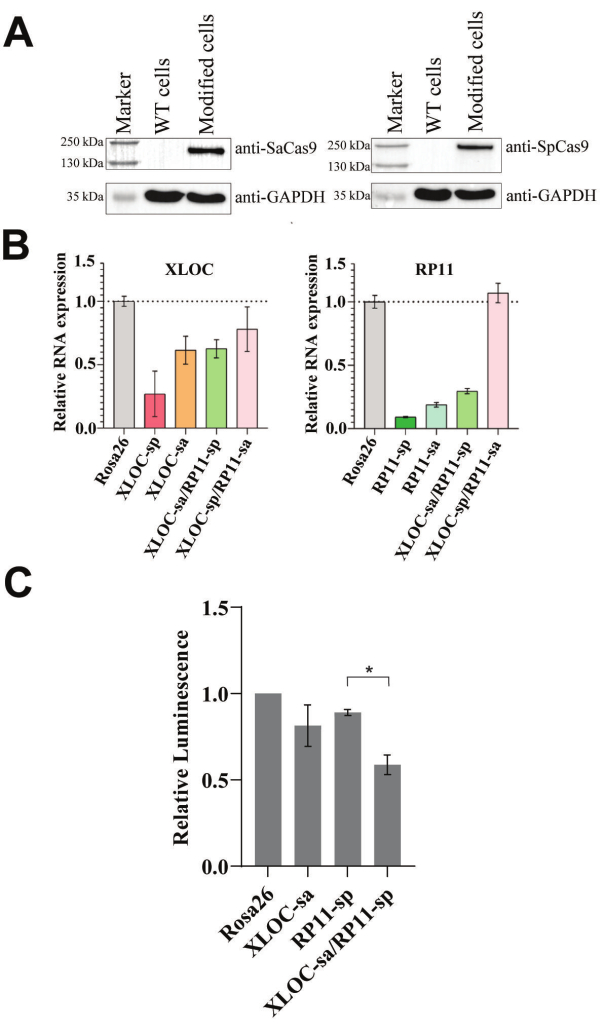
الشكل 2: وظائف نظام قمع lncRNA المزدوج. (أ) تم إجراء تحليل اللطخة الغربية للكشف عن متغيرات dCas9-KRAB-MeCP2 في خلايا 501-ميل المعدلة باستخدام الأجسام المضادة المضادة ل Cas9. تم استخدام خلايا 501-ميل من النوع البري (WT) بدون التنبيغ كعنصر تحكم (انظر الشكل التكميلي 3 ، الشكل التكميلي 4 ، الشكل التكميلي 5 ، الشكل التكميلي 6 للبقعة الخام). (ب) التحليل المستند إلى qPCR لمستويات RP11 و XLOC RNA بالنسبة لخلايا التحكم Rosa26 بعد نقل gRNAs المزدوجة إلى خلايا 501-mel المعدلة. تم استخدام GAPDH كجين التدبير المنزلي. يتم التعبير عن البيانات كمتوسط ± SD. (ج) تم إجراء مقايسة قابلية قمع lncRNA المزدوج بعد دمج gRNAs المزدوجة في خلايا 501-mel المعدلة في ثلاثة عمليات نقل مستقلة. تم تطبيع قيم التلألؤ إلى عنصر التحكم Rosa26. يتم التعبير عن نتائج الجدوى بمتوسط (ن = 3) ± SEM. تم إجراء اختبار t غير متزاوج. تشير علامة النجمة (*) إلى فرق كبير عند p < 0.05. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
الشكل التكميلي 1: خريطة البلازميد. تم إنشاء الخريطة باستخدام Benchling (https://benchling.com/). تم تعديل البلازميدات المستخدمة في البروتوكول من Addgene # 96921 و # 110824. الرجاء النقر هنا لتنزيل هذا الملف.
الشكل التكميلي 2: الشكل التكميلي 2: تحليل اللطخة الغربية ل
تعبير dCas9-KRAB. (أ) تعبير SadCas9-KRAB في خلايا 501-ميل بعد 6 أيام من التعداد. (ب) أدى النقل المتسلسل ل SadCas9-KRAB و SpdCas9-KRAB في خلايا 501-mel إلى تقليل تعبير SadCas9-KRAB بعد 3 أسابيع من التعدين. (ج) أدى النقل المتزامن ل SadCas9-KRAB و SpdCas9-KRAB في خلايا 501-mel إلى انخفاض تعبير SadCas9-KRAB بعد 3 أسابيع من التعدين. (د) يمكن ملاحظة تعبير SpdCas9-KRAB بعد النقل المتزامن ل SadCas9-KRAB و SpdCas9-KRAB. LV. حجم الفيروسات العدسية المستخدمة في ميكرولتر. الرجاء النقر هنا لتنزيل هذا الملف.
الشكل التكميلي 3: البيانات الأولية للبقعة الغربية SaCas9-KRAB. الرجاء النقر هنا لتنزيل هذا الملف.
الشكل التكميلي 4: البيانات الأولية للبقعة الغربية SpCas9-KRAB. الرجاء النقر هنا لتنزيل هذا الملف.
الشكل التكميلي 5: بيانات GAPDH_SpCas9-KRAB. الرجاء النقر هنا لتنزيل هذا الملف.
الشكل التكميلي 6: بيانات GAPDH_SaCas9-KRAB. الرجاء النقر هنا لتنزيل هذا الملف.
الجدول التكميلي 1: تسلسلات الحمض النووي الريبي الإرشادية المستخدمة. الرجاء النقر هنا لتنزيل هذا الملف.
الجدول التكميلي 2: تسلسل الحمض النووي المستخدم. الرجاء النقر هنا لتنزيل هذا الملف.
الملف التكميلي 1: ملف FASTA المزدوج grna-zeo-gfp. الرجاء النقر هنا لتنزيل هذا الملف.
الملف التكميلي 2: ملف FASTA الخاص ب dSpCas9-krab-mecp2. الرجاء النقر هنا لتنزيل هذا الملف.
الملف التكميلي 3: ملف FASTA الخاص ب dSaCas9-krab-mecp2. الرجاء النقر هنا لتنزيل هذا الملف.
Discussion
في هذه الدراسة ، قمنا بتنفيذ استراتيجية استهداف lncRNA مزدوجة في خلايا الورم الميلانيني باستخدام CRISPRi بناء على dCas9-KRAB-MeCP2 من نوعين متعامدين. باستخدام هذا النظام ، نجحنا في قمع زوج اصطناعي قاتل من lncRNAs ، RP11 و XLOC ، مما أدى إلى زيادة موت الخلايا ، على عكس ما تم قمعها بشكل فردي. ومع ذلك ، فإن أنظمة كريسبري تنطوي على خطر قمع الجينات خارج الهدف ، مما قد يؤثر على تفسير النتائج. على الرغم من عدم إجراء التحقق التجريبي ، إلا أننا قللنا من التأثيرات المحتملة خارج الهدف أثناء تصميم الحمض النووي الريبي المرسال في السيليكو.
قد يؤثر التباين في كفاءة التحويل على قابلية التكاثر. لذلك ، قمنا بتحسين التنبيغ من خلال تطبيق كميات متساوية من فيروسات العدسات ، والتي تم تحديدها من خلال معايرة منحنى قياسية باستخدام تركيزات متفاوتة من الفيروسات العدسية. ومع ذلك ، فإن زيادة حجم الحمولة للناقل المصمم حديثا قد تفرض قيودا على كفاءة تغليف الفيروسات العدسية ، مما قد يقلل من كفاءة التحويل ، خاصة في خطوط الخلايا التي يصعب تحويلها. نتيجة لذلك ، قد يتطلب تحقيق نقل كاف وتعبير قوي ل dCas9 عيارات فيروسية عالية في حالات معينة. بالإضافة إلى ذلك ، يمكن التحايل على الضربة القاضية غير الكافية عن طريق المراقبة المتكررة لمستويات بروتين dCas9-KRAB-MeCP2 وتعبير الحمض النووي الريبي لل lncRNAs المستهدفة. بدلا من ذلك ، هناك إصدارات dCas9 متوفرة مع نظام قامع واحد فقط للتغلب على هذا القيد ، ومع ذلك ، مع تكلفة انخفاض الكفاءة25.
أظهرت مجموعة XLOC-sa-RP11-sp فقط نشاطا ناجحا ، في حين أن التكوين العكسي ل XLOC-sp-RP11-sa كان غير فعال. التفسير المعقول لهذا الاختلاف في فعالية الضربة القاضية هو التفاعل بين خصوصية PAM وإمكانية الوصول إلى الكروماتين33. بالنظر إلى أن SaCas9 و SpCas9 يتعرفان على تسلسلات PAM المميزة ، فإن الموقع المستهدف ل SpCas9 في اتجاه XLOC-sp-RP11-sa قد يفتقر إلى PAM الأمثل ، وبالتالي تقليل تقارب الارتباط والنشاط اللاحق. علاوة على ذلك ، يمكن أن يؤثر الترتيب المكاني لمتغيرات بروتين الاندماج dCas9 على بنية الكروماتين وإمكانية الوصولإليه 34،35 ، مما قد يحد من توظيف آلية النسخ أو العوامل التنظيمية المرتبطة بها في تكوين XLOC-sp-RP11-sa. تشير هذه النتائج إلى أن الوضع النسبي ل SaCas9 و SpCas9 في سياق جيني معين قد يؤثر بشكل حاسم على فعاليتهما الوظيفية ، وهو جانب يستدعي مزيدا من التحقيق لاستراتيجيات التعديل المحسنة القائمة على كريسبر.
البروتوكول المطور قابل للتكيف لاستهداف أو فحص أزواج الحمض النووي الريبي الاصطناعية المميتة الأخرى غير المشفرة عن طريق تعديل متجه الحمض النووي الريبي المزدوج. بمجرد أن يعبر خط الاهتمام الخلوي بثبات عن كلا متغيري dCas9 ، تصبح مجموعة متنوعة من الأساليب ممكنة. بينما يمكن استخدام RNAi و ASOs و shRNAs لإسكات الجينات ، فإن CRISPRi تقدم منهجية فحص أكثر وضوحا ، ويمكن أيضا تصور دمجها مع المركبات الكيميائية الصغيرة أو المثبطات. من خلال استبدال ناقل gRNA المزدوج بمكتبة gRNA مزدوجة ، يسمح هذا البروتوكول بالفحص المنهجي للأزواج المميتة الاصطناعية أو التبعيات الجينية الأخرى في الخلايا السرطانية ، وبالتالي تعزيز دقة استهداف الخلايا السرطانية.
على الرغم من أننا حققنا تكامل متغيرات dCas9-KRAB-MeCP2 من خلال نقل الفيروسات العدسية ، إلا أنه يمكن أيضا استخدام طرق تعدين مستقرة بديلة لدمج الإنزيمات المرغوبة في خلايا 501-mel. هذا مهم بشكل خاص ، حيث ، في تجربتنا ، يمكن أن يحدث إسكات التركيبات الكبيرة مثل dCas9-KRAB-MeCP2 بمرور الوقت36،37. بدلا من ذلك ، تبين أن dCas9-KRAB هو بديل أصغر23. في تجربتنا ، أدى التنبيغ باستخدام بروتينات الاندماج الأصغر dCas9-KRAB المشتقة من أخصائيي تقويم العظام Cas9 إلى تقليل التعبير عن البروتين أثناء الزراعة المطولة (انظر الشكل التكميلي 2). لذلك ، يجب مراقبة التعبير المتسق للبروتين بانتظام في النقاط الزمنية المتكررة. بالإضافة إلى ذلك ، قد يؤدي تحويل بروتينين اندماجيين كبيرين جنبا إلى جنب مع علامات الاختيار وأشرطة تعبير gRNA المزدوجة إلى إجهاد خلوي أو قد يصل إلى قدرات التعبئة الفيروسية.
قد يكون تحقيق كفاءة ضربة قاضية بنسبة 100٪ في CRISPRi المزدوج أمرا صعبا بسبب تصميم gRNA دون المستوى الأمثل أو تعبير dCas9 غير الكافي. لمعالجة هذا الأمر ، يجب اختبار العديد من gRNAs لكل هدف باستخدام أدوات تصميم متقدمة لتحديد التسلسلات الأكثر فعالية. بالإضافة إلى ذلك ، فإن تحسين تعبير dCas9-KRAB-MeCP2 من خلال تصميم المتجه المحسن وتوصيف خطوط الخلايا النسيلة يمكن أن يعزز كفاءة الضربة القاضية.
تشمل الاستراتيجيات البديلة لدمج نظام كريسبري استخدام نواقل الينقولاز أو التكامل الموجه بكريسبر لتحقيق إدخال مستقر في مواقع الجينوم الملاذ الآمن ، والتي تشتهر بالحفاظ على التعبير المستقر بمرور الوقت38. ومع ذلك ، فإن العديد من أنواع الخلايا ، بما في ذلك خلايا الورم الميلانيني ، تواجه صعوبة في نقل39 ، مما يجعل نقل الفيروسات العدسية هو الطريقة المفضلة. قد يوفر التثقيب الكهربائي للإنزيمات القائمة على الترانسبوزاز أو Cas وسيلة بديلة لدمج تركيبات dCas9-KRAB-MeCP2 في خلايا الورم الميلانيني40. سيكون تنفيذ نظام خروج المغلوب المحفز مفيدا لتنشيط تعبير dCas9-KRAB-MeCP2 حسب الحاجة بدلا من التعبير التأسيسي لتقليل الإجهاد الخلوي.
أخيرا ، لا يقتصر النهج المطور على خلايا الورم الميلانيني ولكن يمكن تمديده إلى أي نوع من الخلايا حيث يتم التحقيق في الفتك الاصطناعي. من المفترض صراحة توسيع نطاق هذا النهج نحو طريقة فحص مكتبة gRNA. ومع ذلك ، نظرا لزيادة التعقيد الاندماجي ، على سبيل المثال ، مع أكثر من ثلاثة gRNAs لكل زوج جيني ، بما في ذلك الضوابط وتغطية 300 x على الأقل ، يمكن استهداف جزء فرعي فقط من lncRNAs المعبر عنها عمليا. لذلك ، يجب على المستخدمين الاختيار المسبق للمرشحين بعناية من خلال مراعاة عوامل مثل مستويات تعبير lncRNA وأهميتها المعروفة في سياقات خلوية محددة قبل متابعة فحص المكتبة. ومع ذلك ، فإن التنوع الاندماجي لهذا النهج يجعله أداة قيمة للتحقيق في التفاعلات المميتة الاصطناعية والتبعيات الجينية الأخرى ل lncRNAs عبر أنواع مختلفة من السرطان وأمراض أخرى محتملة. يؤدي هذا إلى توسيع مجموعة الأدوات المتوفرة حاليا لدراسة تفاعلات الشبكة هذه في الخلايا.
Disclosures
يتم تمويل Jochen Imig حاليا من قبل شركة Pfizer Inc. و CGCIII برعاية شركة Pfizer Inc. و Merck KGaA و AstraZeneca PLC. لم يكن للرعاة أي دور في تصميم الدراسة أو تنفيذها أو تفسيرها أو كتابتها. جميع المؤلفين ليس لديهم تضارب في المصالح للإفصاح عنهم.
Acknowledgements
نعرب عن تقديرنا لستيفان راونسر (معهد ماكس بلانك لعلم وظائف الأعضاء الجزيئية ، دورتموند) للوصول إلى مساحة مختبر السلامة الحيوية من المستوى 2. شكر خاص لإريك وانغ على مساهماته الفكرية ولجميع الأعضاء السابقين والحاليين في مختبر Imig على مناقشاتهم القيمة. يتم تمويل Jochen Imig حاليا من قبل شركة Pfizer Inc. في CGC III.
Materials
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
References
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved