Method Article
אסטרטגיית הפרעות CRISPR כפולות למיקוד אינטראקציות קטלניות סינתטיות בין RNA לא מקודדים בתאים סרטניים
In This Article
Summary
מחקר זה מציג מערכת CRISPRi כפולה המכוונת ל-RNA ארוכים שאינם מקודדים בתאי מלנומה. הוא מאפשר הפלת גנים קומבינטוריים ובדיקה קטלנית סינתטית, זיהוי אינטראקציות lncRNA ספציפיות לסרטן לאסטרטגיות טיפוליות פוטנציאליות.
Abstract
RNAs ארוכים שאינם מקודדים (lncRNAs) מייצגים קבוצה עצומה ומגוונת מבחינה תפקודית של מולקולות RNA, עם למעלה מ-100,000 צפויות בגנום האנושי. למרות ש-lncRNAs נשמרים פחות בין מינים בהשוואה לגנים מקודדי חלבון, הם ממלאים תפקידים קריטיים בוויסות גנים, אינטראקציות כרומטין והתקדמות סרטן. המעורבות שלהם בסרטן הופכת אותם למטרות טיפוליות מבטיחות. הפרעות CRISPR (CRISPRi), המשתמשות ב-Cas9 לא פעיל קטליטית המותך עם מדכא שעתוק כגון KRAB-MeCP2, מציעה שיטה מדויקת למיקוד lncRNAs גרעיניים ולהערכת תפקודיהם. מחקר זה מציג מערכת CRISPRi כפולה המשתמשת בטכנולוגיות CRISPRi אורתוגונליות מ-Staphylococcus aureus ו-Streptococcus pyogenes המבוססות על dCas9-KRAB-MeCP2, המותאמת למיקוד קומבינטורי של lncRNAs בתאי מלנומה אנושיים. הפרוטוקול מקל על הפלת גנים קומבינטוריים או סינון קטלני סינתטי של זוגות lncRNA, ומספק כלי חדשני לחקר הסרטן. על ידי חקר קטלניות סינתטית בין lncRNAs, גישה זו יכולה לסייע בזיהוי אינטראקציות lncRNA קריטיות להישרדות תאי סרטן, ולהציע אסטרטגיות טיפוליות חדשות. הפונקציונליות של המערכת הכפולה מודגמת, ומדגישה את הפוטנציאל שלה בזיהוי אינטראקציות lncRNA קריטיות ספציפיות לסרטן.
Introduction
למרות שפחות מ-3% מהגנום האנושי מקודד חלבונים, כ-80% מהגנום מועתק 1,2. מבין יחידות השעתוק הלא מקודדות, עשרות אלפים מסווגים כ-RNA ארוך שאינו מקודד (lncRNAs) העולה על 200 נוקלאוטידים, והמספר הכולל של lncRNAs אנושיים מוערך בלמעלה מ-100,000 3,4. בניגוד לגנים מקודדים, lncRNAs נשמרים פחות בין מינים. בהתחשב בכך שבני אדם חולקים 99% מהגנום שלהם עם פרימטים כמו שימפנזים, ההשערה היא של-lncRNA יש השפעה הרבה יותר גדולה על האבולוציה הפנוטיפית 5,6. ממצאים אלה מצביעים על תפקודים תאיים חשובים של lncRNAs. למרות שהרגולציה של lncRNAs והאינטראקציות שלהם עם חלבונים קושרי RNA ו-RNA אחרים נותרו לא מובנים לחלוטין, ו-lncRNAs רבים עדיין לא הוסברו במלואם, ברור ש-lncRNAs מציגים דפוסי ביטוי ספציפיים לתאים ולרקמות בבריאות ובמחלות, כגון סרטן 7,8,9,10. הם מעורבים בפונקציות מגוונות, כולל ויסות שעתוק גנים, מעורבות באינטראקציות כרומטין11, עיבוד RNA12, ייצוב RNA13 וויסות תרגום14.
בסרטן, lncRNAs מגוונים מאוד ספציפיים לסוג התא משפיעים על התפתחות הגידול והגרורות על ידי ויסות ביטוי הגנים, ומדגישים את הפוטנציאל שלהם כמטרות טיפוליות חשובות15. מעבר לזיהוי lncRNAs כסמנים ביולוגיים בדגימות גידול16, התמקדות ב-lncRNAs ספציפיים לגידול כדי לשבש את תפקודיהם במורד הזרם טומנת בחובה פוטנציאל משמעותי הן ביישומים קליניים והן במחקר בסיסי. גישות מבוססות RNA להבהרת התפקידים של lncRNAs כוללות אוליגונוקלאוטידים אנטיסנס (ASOs), RNA סיכת ראש קצרה ו-RNA מפריע קטן (siRNA)17,18. בעוד ש-siRNA משמש בדרך כלל למסכי השתקת גנים, הנוקדאון מבוסס siRNA מוגבל לציטופלזמה19. עם זאת, lncRNA פועל לעתים קרובות בתוך הגרעין.
לחלופין, ניתן להשתמש בהפרעות פלינדרומיות קצרות (CRISPRi) כדי לעכב lncRNAs בסרטן אנושי20. יתר על כן, ניתן לתכנת בקלות מסכי CRISPRi ברחבי הגנום ולמקד למגוון רחב של גנים מקודדים ולא מקודדים כדי לבחון את השפעתם התפקודית21,22. ב-CRISPRi חסר קטליטי Cas9 (dCas9) מאוחה לתחום מדכא שעתוק, כגון תחום הקופסה הקשורה לקרופל (KRAB)23. דיכוי גנים על ידי dCas9-KRAB מונחה על ידי RNA מנחה (gRNA) לאזור העניין. CRISPRi שולט בגנים ברמות ה-DNA, מה שמוביל ליעילות גבוהה יותר ולפנוטיפים רצויים של אובדן תפקוד בניגוד להפרעות RNA, הפעיל ברמה שלאחר השעתוק24. בתגובה ליעילות המוגבלת של KRAB בהשתקת מטרה, מיזוג של KRAB ו-MeCP2 עם dCas9 הוצג כאסטרטגיית דיכוי יעילה יותר25.
למרות שהשתקת גן בודד עשויה להשפיע על כדאיות הסרטן, אינטראקציות סינתטיות כפולות או מרובות קטלניות יכולות להציל תאים סרטניים ממוות תאי26. קטלניות סינתטית מערבת שני גנים או יותר, שכל אחד מהם יכול לפצות על תפקודו של השני. כדי להתגבר על בעיות עם מסכי נוקדאון של גן בודד, אסטרטגיות CRISPR כפולות המכוונות לזוגות גנים מקודדי חלבון קטלניים מציעות גישה מבטיחה27.
כאן, אנו מציגים פרוטוקול לשימוש קומבינטורי של מיקוד מבוסס CRISPRi אורתוגונלי של lncRNA או זוגות RNA אחרים שאינם מקודדים באמצעות Staphylococcus aureus ו-Streptococcus pyogenes dCas9 שהתמזגו ל-KRAB-MeCP2 בתאי מלנומה אנושיים (ראה איור 1). ניתן להשתמש בפרוטוקול להפלת CRISPR קלאסית קומבינטורית או כהקרנה מבוססת CRISPRi של זוגות קטלניים סינתטיים בסרטן. השימוש בשני מינים שונים של dCas9, SpCas9 ו-SaCas9, בגישת CRISPRi מאפשר מיקוד עצמאי של מיקומים גנומיים מובחנים עם תגובתיות צולבת מינימלית, שיפור הספציפיות והגמישות תוך הבטחת סלקטיביות גבוהה על המטרה. נעשה שימוש במוטיבים סמוכים שונים של פרוטו-ספייסר (PAMs): NGG עבור SpCas9 ו-NNGRRT עבור SaCas9. מערכת ה-dCas9 הכפולה מתמודדת עם אתגרים כגון תאימות sgRNA מוגבלת על ידי מתן אפנון מדויק בו-זמני והפחתת תחרות מורכבת sgRNA-RNP בעת שימוש בסוג יחיד של dCas9. חידוש זה משפר את החוסן והרבגוניות של סינון ספריית sgRNA כפולה. לסיכום, אנו מספקים מערכת CRISPRi כפולה מתפקדת במלואה בתאי מלנומה כמודל סרטני.
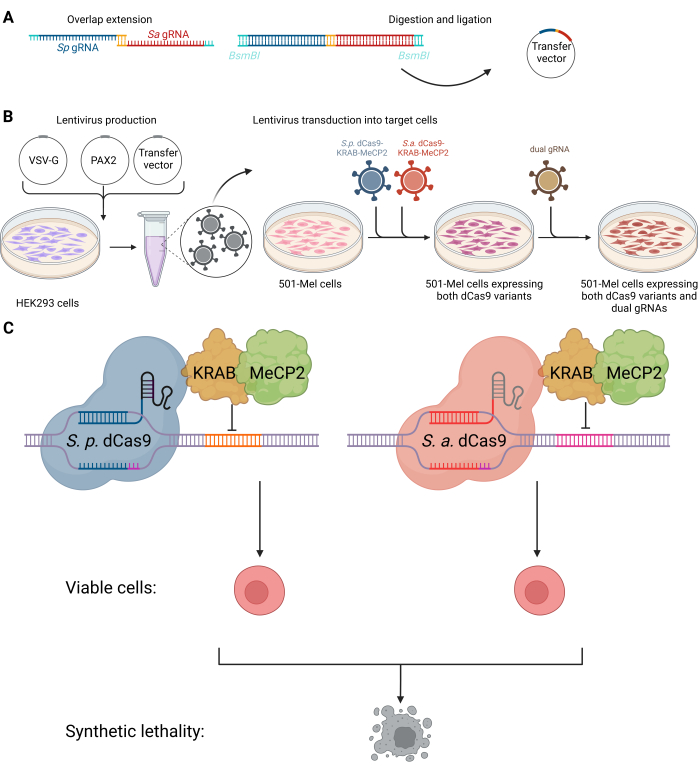
איור 1: סכימה של מערכת CRISPRi הכפולה להתמקדות באינטראקציות קטלניות סינתטיות של RNA לא-מקודדים. (A) הליך שיבוט של וקטור ה-gRNA הכפול. (ב) תאי HEK293 הועברו עם פלסמיד מעטפת VSV-G, אריזת פלסמיד PAX2 וקטור העברה לייצור לנטי-וירוסים להעברה לאחר מכן לתאי 501-mel. לנטי-וירוסים המכילים את המידע הגנטי עבור SpdCas9-KRAB-MeCP2 (כחול) ו-SadCas9-KRAB-MeCP2 (אדום) שולבו בתאי 501-mel בו זמנית. לאחר בחירת האנטיביוטיקה, בוצעה התמרה שנייה של לנטי-ויראלי כדי לשלב את ה-gRNA הכפול הרצוי (ירוק זית). (C) תאי 501-mel המבטאים את גרסאות dCas9 מקיימים אינטראקציה עם ה-gRNAs המתאימים להם כדי להשתיק את ביטוי גן המטרה, וכתוצאה מכך מוות של תאי סרטן. נוצר עם BioRender.com. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Protocol
קו תאי המלנומה האנושית 501-mel (RRID: CVCL_4633) סופק באדיבות על ידי מעבדת Aifantis (אוניברסיטת ניו יורק). קו התאים Lenti-X 293T HEK נרכש מ-Takara Bio. קווי תאים אלה גודלו במדיום הנשר המותאם (DMEM) של Dulbecco בתוספת 10% סרום בקר עוברי (FBS) ב-37 מעלות צלזיוס באטמוספירה של 5% CO2 בתנאים סטריליים. התאים עברו עד שהגיעו למפגש של 80%-90%. קווי תאים נבדקו באופן קבוע לזיהום מיקופלזמה.
1. תכנון gRNAs באמצעות פלטפורמת עיצוב gRNA מקוונת
- תכנן gRNA אחד ספציפי ל-SpCas9 ו-gRNA אחד ספציפי ל-SaCas9 עבור כל lncRNA יעד כדי להעריך איזה וריאנט מניב תוצאות נוק-דאון מעולות.
- כוון את ה-lncRNAs הרצויים, כאן RP11-120D5.1 (RP11) ו-XLOC_030781 (XLOC), תוך שימוש ב-gRNA היעיל ביותר עבור כל S. pyogenes (gRNA-sp) Cas9 ו-S. aureus (gRNA-sa) Cas9.
הערה: ניתן לחלץ את ה-S. pyogenes gRNAs מ-Petroulia et al.28. - עקוב אחר ההמלצות הספציפיות לאתר כדי לתעדף את הפעילות הגבוהה ביותר ביעד, המתבטאת בציונים גבוהים ביעד, תוך מזעור התאמות מחוץ ליעד כפי שהוצע על ידי כלי עיצוב gRNA.
- בחר את הגנום GRCh38 (hg, הומו ספיינס). בחר את אתר ה-PAM עבור SpCas9 (NGG) ו-SaCas9 (NNGRRT) הספציפיים.
- השתמש בשיטת הניקוד מחוץ למטרה מ-Hsu et al.29. מקם את ה-gRNAs בחלון של -150 bp עד +50 bp סביב אתר התחלת השעתוק לביצועים מיטביים.
- בחר gRNAs עם הציון הגבוה ביותר ביעד ומחוץ ליעד (ראה טבלה משלימה 1).
- שלב gRNA בקרה לא מכוון או מקושקש, כאן נגד Rosa26, עם כל gRNA מטרה כדי ליצור וקטור gRNA כפול, המכוון ל-lncRNA אחד. השילוב של gRNAs מטרה בפלסמיד אחד מאפשר שינוי ספציפי לאתר של שני אתרי היעד.
הערה: וקטור ה-gRNA הכפול המכיל gRNA בקרת gRNA ו-gRNA מטרה מאפשר נוק-דאון של lncRNA מטרה אחת, בעוד וקטורי gRNA כפולים המכילים gRNAs המכוונים לאתרים מאפשרים נוק-דאון בו-זמנית של lncRNAs ממוקדים.
2. שיבוט gRNA
- בצע PCR הרחבת חפיפה באמצעות שני מקטעי DNA המכילים את רצף ה-gRNA. השתמש בפריימר U6 קדימה ובפריימר ההפוך H1 (ראה טבלה משלימה 2) להגברה כמתואר על ידי Najm et al.27.
- הגדר תגובת PCR של 50 מיקרוליטר עם 25 מיקרוליטר של תערובת מאסטר PCR בנאמנות גבוהה פי 2 עם מאגר HF ו-1 מיקרוליטר מכל מקטע DNA המכיל את S. pyogenes או S. aureus gRNA. הפעל את התגובה בתנאי הרכיבה הבאים: דנטורציה ראשונית ב-98 מעלות צלזיוס למשך 30 שניות, ואחריה 15 מחזורי דנטורציה ב-98 מעלות צלזיוס למשך 5 שניות, חישול ב-55 מעלות צלזיוס למשך 10 שניות, והתארכות ב-72 מעלות צלזיוס למשך 15 שניות.
- הוסף 2.5 מיקרוליטר של פריימר U6 קדימה ו-2.5 מיקרוליטר של פריימר הפוך H1 לאחר השלמת 15 המחזורים הראשונים, והכפיף את התגובה ל-20 מחזורים נוספים באותם תנאים, עם הארכה סופית למשך 5 דקות ב-72 מעלות צלזיוס.
- יש לטהר את מוצרי ה-PCR באמצעות ערכת טיהור PCR בהתאם להוראות היצרן.
- עכל 1 מיקרוגרם של הפלסמיד שהשתנה באמצעות 1 מיקרוליטר של אנזים הגבלה BsmB1 ו-1 מיקרוליטר של מאגר 10x למשך 30 דקות ב-37 מעלות צלזיוס בתגובה של 10 מיקרוליטר.
- שכפל את תוצרי ה-PCR לפלסמיד הכפול המעוכל gRNA-Zeo-GFP (ראה איור משלים 1 וקובץ משלים 1) באמצעות פרוטוקול תגובה של הרכבת DNA HiFi כפי שתואר על ידי אחרים30.
- הפוך תאי E. coli בעלי יכולת כימית על ידי ערבוב של 50 מיקרוליטר של תרבית חיידקים עם 4 מיקרוליטר של מוצר הרכבה בהתאם להוראות היצרן.
3. ייצור Lentivirus
זהירות: טפל בנגיפי לנטי פעילים בכל השלבים תוך שמירה על הנחיות הבטיחות המתאימות. בצע תמיד עבודה עם וירוסים בארון בטיחות Class 2 כדי למנוע סכנות מהיווצרות אירוסול בלתי נמנעת. בצע את כל העבודות מחוץ לארון הבטיחות, כגון צנטריפוגה, במיכלים אטומים לאירוסול ורוטורים המאושרים לשימוש עם אורגניזמים מקבוצת סיכון 2. ללבוש ביגוד מגן מתאים, כולל מעיל מעבדה, כפפות חד פעמיות ומשקפי מגן, בכל עת באזור העבודה. יש לטפל במשטחי העבודה בתמיסת חיטוי, ויש לטפל גם בפסולת אחרת בתמיסת חיטוי, המאפשרת להם להישאר בארון האטום תחת אור UV למשך 240 דקות לפחות. יש לשטוף פיפטות סרולוגיות משומשות וקצות פיפטות בתמיסת חיטוי לפני השלכתם בבקבוקים הניתנים לחיטוי. לאחר מכן, יש להשליך את הפסולת למיכל הפסולת S2. פסולת S2 תושבת באמצעות חיטוי. ניתן לטפל בקווי תאים מותמרים בתנאי S1 לא לפני יומיים לאחר ההתמרה ורק לאחר לפחות שני שינויים מלאים של מדיום התרבית עם מדיום נטול וירוסים.
- ספור תאי Lenti-X 293T HEK על ידי ערבוב של 10 מיקרוליטר תאים עם 10 מיקרוליטר של תמיסה כחולה טריפן בהמוציטומטר. צלחת 4 x 106 תאי Lenti-X 293T HEK לכל צלחת של 10 ס"מ כדי להשיג ~80% מפגש למחרת.
- עבוד במעבדה Class 2 משלב זה ואילך. מערבבים 500 מיקרוליטר של מדיום טרנספקציה מתאים עם וקטור העברה של 11.25 מיקרוגרם SpdCas9-KRAB-MeCP2 (ראה קובץ משלים 2), 5.5 מיקרוגרם פלסמיד מעטפת VSV-G ו-16.5 מיקרוגרם וקטור pPAX2. דוגרים למשך 5 דקות בטמפרטורת החדר. עבור גרסת dCas9 השנייה, השתמש בווקטור ההעברה SadCas9-KRAB-MeCP2 (ראה קובץ משלים 3).
- בשפופרת נפרדת, מערבבים 500 מיקרוליטר של מדיום מופחת בסרום עם 36 מיקרוליטר של מגיב PEI (מלאי: 1 מ"ג/מ"ל). דוגרים למשך 5 דקות בטמפרטורת החדר.
- שלב את התערובות ודגר למשך 15 דקות בטמפרטורת החדר. מוסיפים את התערובת בזהירות טיפתית למדיום התא (6 מ"ל) ודוגרים למשך הלילה בחום של 37 מעלות צלזיוס ו-5% CO2. ודא שהתאים נשארים מחוברים כדי למנוע ירידה בתפוקת לנטי-ויראלי.
- השליכו את המדיום לבקבוק פסולת הניתן לחיטוי באמצעות פיפטה סרולוגית לאחר 12-15 שעות. מעתה ואילך, שטפו חומרים מתכלים, כולל פיפטות סרולוגיות וקצות פיפטות, בתמיסת החיטוי לפני השלכתם לפסולת החיטוי. הוסף 5 מ"ל של מדיום DMEM טרי לתאים שהועברו באמצעות פיפטה חדשה.
- קצור לנטי-וירוסים על ידי העברת הסופרנטנט באמצעות פיפטה סרולוגית לצינור קנוני של 50 מ"ל 48 שעות לאחר הטרנספקציה. אחסן את הצינור הקנוני במקרר. הוסף 5 מ"ל של מדיום טרי לתאים ודגר על התאים למשך 24 שעות נוספות.
- חזור על איסוף ה-lentiviruses על ידי העברת ה-supernatant לאותו צינור קנוני של 50 מ"ל המכיל את הקציר הראשון. שוב, הוסיפו 5 מ"ל של מדיום טרי, דגרו למשך 24 שעות, והעבירו את הסופרנטנט לאחר מכן לצינור הקנוני של 50 מ"ל. אחסן סופרנטנטים משולבים של הקציר הראשון, השני והשלישי בצינור הקנוני של 50 מ"ל בטמפרטורה של 4 מעלות צלזיוס עד לסינון וצנטריפוגה.
- צנטריפוגה הסופרנטנט המשולב למשך 5 דקות ב-500 x g ו-4 °C במיכלים ורוטורים אטומים לאירוסול המאושרים לשימוש עם אורגניזמים מקבוצת סיכון 2 כדי לגלול תאים מנותקים ופסולת. סנן את הסופרנטנט דרך מסנני מזרק סטריליים בדרגת תרבית תאים של 0.2 מיקרומטר.
- רכז את הסופרנטנט המסונן ב-1,000 x g ו-4 °C באמצעות יחידת סינון צנטריפוגלית עם חתך משקל מולקולרי מתאים של 100 kDa עד כ-500 μL. אחסן כמויות נגיף של 50-100 מיקרוליטר ב-80 מעלות צלזיוס עד לשימוש נוסף.
4. התמרת לנטי-וירוס ובחירה של תאים יציבים שעברו טרנספטציה
התראה: בצע תמיד עבודה עם וירוסים בארון בטיחות Class 2.
- צלחת 501-mel תאים בצלחת של 6 בארות ב-2 x 105 תאים / באר עם נפח תרבית סופי של 2 מ"ל, יום אחד לפני ההעברה.
- החלף את מדיום התרבית ב-2 מ"ל של מדיום DMEM שחומם מראש בתוספת פוליברן בריכוז סופי של 6 מיקרוגרם/מ"ל.
- מעתה נדרשת עבודה במעבדה מסוג 2. הוסף 50 מיקרוליטר של lentiviruses המכילים SpdCas9-KRAB-MeCP2 ו-SadCas9-KRAB-MeCP2 בו זמנית לתאים שטפו חומרים מתכלים, כולל פיפטות סרולוגיות וקצות פיפטות, בתמיסת החיטוי לפני השלכתם לפסולת החיטוי. יש לנער את הצלחת בעדינות ולדגור בטמפרטורה של 37 מעלות צלזיוס באווירה של 5% CO2 .
- השליכו את המדיום לבקבוק הניתן לחיטוי באמצעות פיפטה סרולוגית והוסיפו 2 מ"ל של מדיום טרי המכיל בלסטיצידין (10 מיקרוגרם/מ"ל) ופורומיצין (2 מיקרוגרם/מ"ל) 16 שעות לאחר ההעברה.
- המשך בבחירה על ידי החלפת המדיום במדיום טרי של 2 מ"ל המכיל בלסטיצידין (10 מיקרוגרם/מ"ל) ופורומיצין (2 מיקרוגרם/מ"ל) לאחר יומיים. טפל בתאים שעברו התמרה בתנאי S1 לאחר 7 ימים לאחר ההעברה.
הערה: יש לבצע בחירת אנטיביוטיקה עד שכל תאי הבקרה שלא עברו התמרה מתו. ניתן להשתמש באסטרטגיות בחירה חלופיות בהתאם לקלטת ההתנגדות בווקטור ההעברה. במידת הצורך, מטב את ריכוזי האנטיביוטיקה באמצעות עקומת הריגה. ייתכן שתידרש בחירת שיבוט משוכללת של תא בודד באמצעות ציטומטריית זרימה או דילול וציפוי סדרתי, ולאחר מכן כימות חלבון, כדי להשיג אוכלוסיות תאים המבטאות היתוך dCas9 ברמות מספיקות.
5. כימות חלבונים וניתוח כתמים מערביים
- ספור תאי 501-mel יציבים על ידי ערבוב של 10 מיקרוליטר של תאים עם 10 מיקרוליטר של תמיסת טריפאן כחול בהמוציטומטר. צנטריפוגה 1 x 106 מהתאים למשך 5 דקות ב 500 x גרם ו -4 מעלות צלזיוס. השליכו את הסופרנטנט והשעו מחדש את כדור התא ב-1 מ"ל של PBS קר כקרח.
- חזור על הצנטריפוגה והשהה מחדש את גלולת התא ב-50 מיקרוליטר של מאגר RIPA המכיל מעכבי פרוטאז. דגרו על קרח למשך 15 דקות והסירו פסולת תאים על ידי צנטריפוגה ב-13,000 x גרם למשך 10 דקות ב-4 מעלות צלזיוס. אחסן את הסופרנטנט בטמפרטורה של -20 מעלות צלזיוס או השתמש מיד לכימות חלבון כפי שתואר על ידי אחרים31.
- מערבבים 10 מיקרוגרם חלבון עם 4 x LDS buffer, 50 מ"מ DTT וממלאים עד 20 מיקרוליטר במים מזוקקים. הפרד את החלבונים על ג'ל הפרדה של 10% על ידי שימוש ב-SDS-PAGE מבוסס טריס/גליצין ב-135 וולט למשך שעה אחת כפי שתואר קודם לכן32. העבירו חלבונים לקרום PVDF ב-90 וולט למשך 90 דקות עם העברת מיכל רטוב.
- דגרו את הממברנה במאגר חוסם (5% חלב ללא שומן ב-TBS-T) למשך שעה אחת ב-180 סל"ד בטמפרטורת החדר.
- הוסף 1:10,000 נוגדן חד שבטי של עכבר נגד GAPDH במאגר החוסם כבקרה פנימית. הוסיפו את הנוגדן העיקרי anti-Cas9 (S. pyogenes) או anti-Cas9 (S. aureus) כדילול של 1:1,000 במאגר החוסם ודגרו למשך הלילה ב-4 מעלות צלזיוס עם ניעור עדין.
- שוטפים את הממברנה 3x עם מאגר TBST למשך 10 דקות כל אחד. דגירה עם נוגדן HRP משני נגד עכבר (1:10,000) למשך שעה בטמפרטורת החדר.
- יש לשטוף 3 פעמים עם מאגר TBST למשך 10 דקות כל אחד. זיהוי אותות באמצעות מצע ECL ומערכת הדמיה.
6. בדיקת כדאיות דיכוי LncRNA כפול
התראה: בצע תמיד עבודה עם וירוסים בארון בטיחות Class 2.
- זרע 0.1 x 105 תאים המכילים SadCas9-KRAB ו- SpdCas9-KRAB יציבים 7 ימים לאחר התמרה בצלחת של 96 בארות ב -100 מיקרוליטר של מדיום DMEM מחומם מראש יום אחד לפני ההעברה.
- ביום ההעברה, החלף את מדיום התרבות ב-100 מיקרוליטר של מדיום DMEM שחומם מראש בתוספת פוליברן בריכוז סופי של 6 מיקרוגרם/מ"ל. מעתה נדרשת עבודה במעבדה מסוג 2. הוסף את הנפח המתאים של lentiviruses לתאים.
הערה: יש לבחון את נפח ה-lentiviruses המכילים את וקטור ה-gRNA הכפול באמצעות GFP כדי ליצור עקומה ליניארית על ידי טיטרציה של lentivirus. - החלף את המדיום במדיום טרי המכיל 500 מיקרוגרם/מ"ל זאוצין, 5 מיקרוגרם/מ"ל בלסטיצידין ו-1 מיקרוגרם/מ"ל פורומיצין 16 שעות לאחר ההעברה.
- בצע זיהוי זוהר 5 ימים לאחר התמרה באמצעות בדיקת כדאיות תאים בהתאם להוראות היצרן.
תוצאות
קלטות הביטוי של SadCas9-KRAB-MeCP2 ו-SpdCas9-KRAB-MeCP2 שולבו בתאי 501-mel בו זמנית באמצעות התמרה עם כמויות שוות של לנטי-וירוסים. מכיוון שפרוטוקול CRISPRi זה מסתמך על ביטוי של גרסאות dCas9-KRAB-MeCP2, ניתוח כתמים מערביים הוא אידיאלי לאישור נוכחותם ברמת החלבון. לאחר העשרה של תאים שעברו טרנספטציה חיובית, נאספו דגימות לניתוח כתמים מערביים כדי לאשר את נוכחותם של האורתולוג dCas9-KRAB-MeCP2. לאחר התמרה מוצלחת, צפויה פס יחיד המתאים לחלבון ההיתוך SpdCas9-KRAB-MeCP2 (202 kDa) וחלבון ההיתוך SadCas9-KRAB-MeCP2 (170 kDa) ואושר, כפי שמוצג באיור 2A, תוך שימוש בנוגדנים ספציפיים לאורתולוגים של Cas9 המתאימים. עם זאת, אם הכתם המערבי אינו בר ביצוע, ניתן להשתמש ב-qPCR כדי לאמת את חלבוני ההיתוך ברמת ה-mRNA.
לאחר שנוצרו תאי 501-mel המבטאים ביציבות את המדכאים הפונקציונליים, בוצעה התמרה לנטי-ויראלית שנייה באמצעות וקטור gRNA כפול. RP11 ו-XLOC הוכחו בעבר כמווסתים בתרביות קצרות טווח של מלנומה גרורתית, ומיקוד CRISPRi בודד הוכיח את השפעתם על התפשטות והישרדות התאים28. לפיכך, בחרנו בזוג lncRNA זה להערכה כשילוב RNA סינתטי קטלני שאינו מקודד להוכחת רעיון, וצפינו פנוטיפ צמיחה מצטבר וניתן לצפייה בקלות בתאים שהשתנו.
כדי לחקור את ההשפעות של CRISPRi כפול בתאי סרטן באמצעות מבחני כדאיות, יש לדכא ביעילות את שני ה-lncRNA הממוקדים. qPCR היא השיטה המתאימה ביותר לאשר את ההדחקה הזו על ידי מדידת רמות התעתיק שלהם. הערכנו את הפונקציונליות של פרוטוקול ה-CRISPRi הכפול שפותח על ידי קביעת רמות RNA ובחינת כדאיות התא בעת התמקדות בצמד RP11 ו-XLOC שעלול להיות קטלני. לכן, gRNAs שונים המכוונים ל-RP11 ו-XLOC הוצגו כדי להפחית את רמות ה-RNA היעד. דיכוי ה-RNA נחקר באמצעות gRNAs ספציפיים למטרה עבור SadCas9-KRAB-MeCP2 או SpdCas9-KRAB-MeCP2, שנבדקו בנפרד ובשילוב לצד gRNA בקרה המכוון ל-Rosa26 (ראה איור 2B). למרות ש-gRNAs המכוונים ל-XLOC או RP11 דיכאו ביעילות את המטרות שלהם, הפלה בו-זמנית של XLOC ו-RP11 הושגה רק עם צמד XLOC-sa/RP11-sp.
מכיוון שרמות ה-RNA של XLOC ו-RP11 הופחתו בהצלחה בקו התאים של 501 מל עם זוג ה-gRNA שנבחר XLOC-sa/RP11-sp, בוצעה בדיקת כדאיות דיכוי lncRNA כפול (ראה איור 2C). לאחר 5 ימים, כדאיות התאים הייתה 81% ו-89% מקבוצת הביקורת של Rosa26 כאשר נעשה שימוש ב-gRNA המטרה בנפרד. שילוב RP11-sp ו-XLOC-sa הביא להפחתה משמעותית נוספת בהשוואה ל-RP11-sp בלבד, עם כדאיות התא ב-59% מביקורת Rosa26.
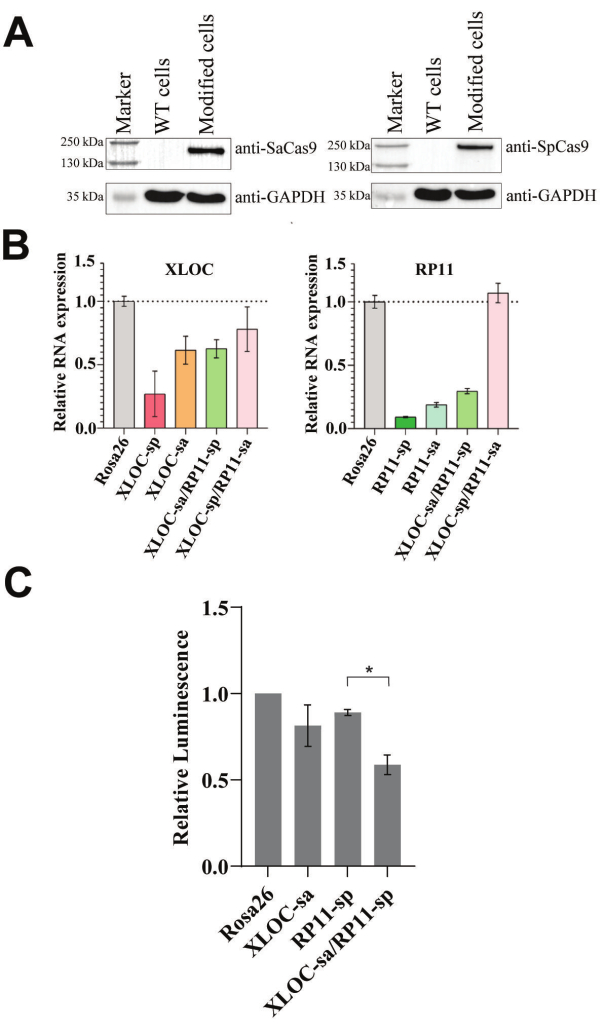
איור 2: הפונקציונליות של מערכת הדיכוי הכפולה lncRNA. (A) ניתוח כתמים מערביים בוצע כדי לזהות את גרסאות dCas9-KRAB-MeCP2 בתאי 501-mel שעברו שינוי באמצעות נוגדנים נגד Cas9. 501 תאי פרא (WT) ללא התמרה שימשו כביקורת (ראה איור משלים 3, איור משלים 4, איור משלים 5, איור משלים 6 עבור הכתם הגולמי). (B) ניתוח מבוסס qPCR של רמות RP11 ו-XLOC RNA ביחס לתאי בקרה Rosa26 לאחר התמרה של gRNA כפול לתאי 501-mel ששונו. GAPDH שימש כגן משק הבית. הנתונים מבוטאים כממוצע ± SD. (C) בדיקת כדאיות הדיכוי הכפול של lncRNA בוצעה לאחר שילוב gRNAs כפולים בתאי 501-mel ששונו בשלוש התמרות עצמאיות. ערכי הזוהר נורמלו לבקרת Rosa26. תוצאות הכדאיות מבוטאות כממוצע (n = 3) ± SEM. בוצעה בדיקת t לא מזווגת. כוכבית (*) מציינת הבדל משמעותי ב-p <-0.05. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
איור משלים 1: מפת פלסמיד. המפה נוצרה באמצעות Benchling (https://benchling.com/). הפלסמידים המשמשים בפרוטוקול שונו מ-Addgene #96921 ו-#110824. אנא לחץ כאן להורדת קובץ זה.
תרשים משלים 2: תרשים משלים 2: ניתוח כתמים מערביים של
dCas9-KRAB. (A) ביטוי SadCas9-KRAB בתאי 501-mel 6 ימים לאחר הטרנספקציה. (B) התמרה רציפה של SadCas9-KRAB ו-SpdCas9-KRAB בתאי 501-mel הובילה לביטוי מופחת של SadCas9-KRAB 3 שבועות לאחר הטרנספקציה. (C) התמרה סימולטנית של SadCas9-KRAB ו-SpdCas9-KRAB בתאי 501-mel הביאה לביטוי מופחת של SadCas9-KRAB 3 שבועות לאחר הטרנספקציה. (D) ניתן היה לצפות בביטוי SpdCas9-KRAB לאחר התמרה בו-זמנית של SadCas9-KRAB ו-SpdCas9-KRAB. LV. נפח ה-lentiviruses המשמשים ב-μL. אנא לחץ כאן להורדת קובץ זה.
איור משלים 3: נתונים גולמיים של כתם מערבי SaCas9-KRAB. אנא לחץ כאן להורדת קובץ זה.
איור משלים 4: נתונים גולמיים של כתם מערבי SpCas9-KRAKB. אנא לחץ כאן להורדת קובץ זה.
איור משלים 5: נתונים גולמיים של GAPDH_SpCas9-KRAKB. אנא לחץ כאן להורדת קובץ זה.
איור משלים 6: נתונים גולמיים של GAPDH_SaCas9-KRAKB. אנא לחץ כאן להורדת קובץ זה.
טבלה משלימה 1: רצפי RNA מנחים משומשים. אנא לחץ כאן להורדת קובץ זה.
טבלה משלימה 2: רצפי DNA משומשים. אנא לחץ כאן להורדת קובץ זה.
קובץ משלים 1: קובץ FASTA של dual-grna-zeo-gfp. אנא לחץ כאן להורדת קובץ זה.
קובץ משלים 2: קובץ FASTA של dSpCas9-krab-mecp2. אנא לחץ כאן להורדת קובץ זה.
קובץ משלים 3: קובץ FASTA של dSaCas9-krab-mecp2. אנא לחץ כאן להורדת קובץ זה.
Discussion
במחקר זה, יישמנו אסטרטגיית מיקוד lncRNA כפולה בתאי מלנומה באמצעות CRISPRi המבוססת על dCas9-KRAB-MeCP2 משני מינים אורתוגונליים. תוך שימוש במערכת זו, דיכאנו בהצלחה זוג סינתטי קטלני של lncRNAs, RP11 ו-XLOC, מה שהוביל למוות מוגבר של תאים, בניגוד למצב שבו הם דוכאו בנפרד. עם זאת, מערכות CRISPRi טומנות בחובן סיכון לדיכוי גנים מחוץ למטרה, מה שעלול להשפיע על פירוש התוצאות. למרות שלא בוצע אימות ניסוי, צמצמנו את ההשפעות הפוטנציאליות מחוץ למטרה במהלך תכנון gRNA בסיליקו.
שונות ביעילות ההתמרה עשויה להשפיע על יכולת השחזור. לכן, ייעלנו את ההתמרה על ידי יישום כמויות שוות של לנטי-וירוסים, שנקבעו באמצעות כיול עקומה סטנדרטי באמצעות ריכוזי לנטי-וירוס משתנים. עם זאת, גודל המטען המוגדל של הווקטור המעוצב החדש עשוי להטיל מגבלות על יעילות האריזה הלנטיויראלית, מה שעלול להפחית את יעילות ההעברה, במיוחד בקווי תאים קשים להתמרמר. כתוצאה מכך, השגת התמרה מספקת וביטוי חזק של dCas9 עשויים לדרוש טיטרים נגיפיים גבוהים במקרים מסוימים. בנוסף, ניתן לעקוף לא מספיק על ידי ניטור תכוף של רמות חלבון dCas9-KRAB-MeCP2 וביטוי RNA של lncRNAs ממוקדים. לחלופין, ישנן גרסאות dCas9 זמינות עם מערכת מדכא אחת בלבד כדי להתגבר על מגבלה זו, עם זאת, עם עלות של יעילות מופחתת25.
רק השילוב XLOC-sa-RP11-sp הראה פעילות מוצלחת, בעוד שתצורת XLOC-sp-RP11-sa ההפוכה לא הייתה יעילה. הסבר מתקבל על הדעת להבדל זה ביעילות הנוקדאון הוא יחסי הגומלין בין ספציפיות PAM לנגישות כרומטין33. בהתחשב בכך ש-SaCas9 ו-SpCas9 מזהים רצפי PAM מובחנים, אתר היעד של SpCas9 בכיוון XLOC-sp-RP11-sa עשוי להיות חסר PAM אופטימלי, ובכך להפחית את זיקת הקישור שלו ואת הפעילות שלאחר מכן. יתר על כן, הסידור המרחבי של שתי גרסאות חלבון ההיתוך dCas9 יכול להשפיע על ארכיטקטורת הכרומטין והנגישות34,35, מה שעלול להגביל את הגיוס של מכונות השעתוק או גורמים רגולטוריים נלווים בתצורת XLOC-sp-RP11-sa. ממצאים אלה מצביעים על כך שהמיקום היחסי של SaCas9 ו-SpCas9 בהקשר גנומי נתון עשוי להשפיע באופן קריטי על יעילותם התפקודית, היבט המצדיק חקירה נוספת לאסטרטגיות אפנון מבוססות CRISPR אופטימליות.
הפרוטוקול שפותח ניתן להתאמה למיקוד או סינון של זוגות RNA סינתטיים קטלניים אחרים שאינם מקודדים על ידי שינוי וקטור ה-gRNA הכפול. ברגע שקו תאים מעניין מבטא ביציבות את שתי גרסאות ה-dCas9, מגוון גישות הופכות לאפשריות. בעוד שניתן להשתמש ב-RNAi, ASOs ו-shRNAs להשתקת גנים, CRISPRi מציעה מתודולוגיית סינון פשוטה יותר, וניתן להעלות על הדעת גם את השילוב שלה עם תרכובות כימיות קטנות או מעכבים. על ידי החלפת וקטור ה-gRNA הכפול בספריית gRNA כפולה, פרוטוקול זה מאפשר סינון שיטתי של זוגות קטלניים סינתטיים או תלות גנטית אחרת בתאים סרטניים, ובכך משפר את הדיוק של מיקוד תאים סרטניים.
למרות שהשגנו אינטגרציה של גרסאות dCas9-KRAB-MeCP2 באמצעות התמרה לנטי-ויראלית, ניתן להשתמש בשיטות טרנספקציה יציבות חלופיות כדי לשלב את האנזימים הרצויים בתאי 501-mel. זה רלוונטי במיוחד, מכיוון שמניסיוננו, השתקה של מבנים גדולים כגון dCas9-KRAB-MeCP2 יכולה להתרחש לאורך זמן36,37. לחלופין, dCas9-KRAB הוכח כחלופה קטנה יותר23. מניסיוננו, התמרה עם חלבוני ההיתוך הקטנים יותר dCas9-KRAB שמקורם באורתולוג Cas9 הובילה לביטוי חלבון מופחת במהלך גידול ממושך (ראה איור משלים 2). לכן, יש לעקוב באופן קבוע אחר ביטוי חלבון עקבי בנקודות זמן חוזרות. בנוסף, התמרת שני חלבוני היתוך גדולים יחד עם סמני בחירה וקלטות ביטוי gRNA כפולות עלולה לגרום ללחץ תאי או להגיע ליכולות אריזה לנטי-ויראליות.
השגת יעילות של 100% ב-CRISPRi כפול יכולה להיות מאתגרת עקב עיצוב gRNA לא אופטימלי או ביטוי dCas9 לא מספיק. כדי לטפל בכך, יש לבדוק מספר gRNAs לכל יעד באמצעות כלי תכנון מתקדמים כדי לזהות את הרצפים היעילים ביותר. בנוסף, אופטימיזציה של ביטוי dCas9-KRAB-MeCP2 באמצעות עיצוב וקטורי משופר ואפיון של קווי תאים משובטים יכולה לשפר את יעילות ההפלה.
אסטרטגיות חלופיות לשילוב מערכת CRISPRi כוללות שימוש בווקטורי טרנספוזאז או אינטגרציה מכוונת CRISPR כדי להשיג החדרה יציבה לאתרים גנומיים של נמל מבטחים, הידועים בשמירה על ביטוי יציב לאורך זמן38. עם זאת, סוגי תאים רבים, כולל תאי מלנומה, מאתגרים לטרנספט39, מה שהופך את התמרת הלנטי-ויראלית לשיטה המועדפת. אלקטרופורציה של אנזימים מבוססי טרנספוזאז או Cas עשויה להציע אמצעי חלופי לשילוב מבני dCas9-KRAB-MeCP2 בתאי מלנומה40. יישום מערכת נוקאאוט הניתנת להשראה יהיה יתרון להפעלת ביטוי dCas9-KRAB-MeCP2 לפי הצורך ולא ביטוי מכונן כדי למזער את הלחץ התאי.
לבסוף, הגישה שפותחה אינה מוגבלת לתאי מלנומה אלא ניתן להרחיב אותה לכל סוג תא שבו נחקרת קטלניות סינתטית. גישה זו נועדה במפורש להיות מורחבת לקראת שיטת הסינון של ספריית gRNA. עם זאת, בשל המורכבות הקומבינטורית המוגברת, למשל, עם יותר משלושה gRNAs לכל זוג גנים, כולל בקרות וכיסוי של לפחות פי 300, ניתן למקד באופן מעשי רק חלק תת-טרנסקריפטומי של lncRNAs מבוטאים. לכן, על המשתמשים לבחור בקפידה מועמדים על ידי התחשבות בגורמים כגון רמות ביטוי lncRNA והרלוונטיות הידועה שלהם בהקשרים סלולריים ספציפיים לפני שתמשיך בסינון הספרייה. עם זאת, הרבגוניות הקומבינטורית של גישה זו הופכת אותה לכלי רב ערך לחקירת אינטראקציות קטלניות סינתטיות ותלות גנטית אחרת של lncRNAs על פני סוגי סרטן שונים ומחלות אחרות פוטנציאליות. זה מרחיב את ערכת הכלים הזמינה כיום לחקר אינטראקציות רשת אלה בתאים.
Disclosures
Jochen Imig ממומן כיום על ידי CGCIII על ידי Pfizer Inc. CGCIII ממומן על ידי Pfizer Inc., Merck KGaA ו-AstraZeneca PLC. לנותני החסות לא היה כל תפקיד בעיצוב, ביצוע, פרשנות או כתיבה של המחקר. לכל המחברים אין ניגודי אינטרסים לחשוף.
Acknowledgements
אנו מודים לסטפן ראונסר (מכון מקס פלאנק לפיזיולוגיה מולקולרית, דורטמונד) על הגישה לחלל מעבדה ברמה 2 של בטיחות ביולוגית. תודה מיוחדת לאריק וואנג על תרומתו האינטלקטואלית ולכל חברי מעבדת אימיג בעבר ובהווה על הדיונים החשובים שלהם. Jochen Imig ממומן כיום על ידי Pfizer Inc. ב-CGC III.
Materials
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
References
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved