Method Article
Estrategia dual de interferencia CRISPR para atacar interacciones letales sintéticas entre ARN no codificantes en células cancerosas
* Estos autores han contribuido por igual
En este artículo
Resumen
Este estudio presenta un sistema dual CRISPRi dirigido a ARN largos no codificantes en células de melanoma. Permite la eliminación combinatoria de genes y el cribado letal sintético, identificando las interacciones de lncRNA específicas del cáncer para posibles estrategias terapéuticas.
Resumen
Los ARN largos no codificantes (lncRNA) representan una clase vasta y funcionalmente diversa de moléculas de ARN, con más de 100.000 previstas en el genoma humano. Aunque los lncRNAs están menos conservados en todas las especies en comparación con los genes que codifican proteínas, desempeñan un papel fundamental en la regulación génica, las interacciones de la cromatina y la progresión del cáncer. Su implicación en el cáncer los convierte en prometedoras dianas terapéuticas. La interferencia CRISPR (CRISPRi), que utiliza Cas9 catalíticamente inactivo fusionado con un represor transcripcional como KRAB-MeCP2, ofrece un método preciso para dirigirse a los lncRNA nucleares y evaluar sus funciones. Este estudio presenta un sistema CRISPRi dual que utiliza tecnologías CRISPRi ortogonales de Staphylococcus aureus y Streptococcus pyogenes basadas en dCas9-KRAB-MeCP2, optimizado para la diana combinatoria de lncRNAs en células de melanoma humano. El protocolo facilita la eliminación combinatoria de genes o el cribado letal sintético de pares de lncRNA, proporcionando una herramienta novedosa para la investigación del cáncer. Al explorar la letalidad sintética entre lncRNAs, este enfoque puede ayudar a identificar las interacciones de lncRNAs críticas para la supervivencia de las células cancerosas, ofreciendo nuevas estrategias terapéuticas. Se demuestra la funcionalidad del sistema dual, lo que pone de manifiesto su potencial en la identificación de interacciones críticas de lncRNA específicas del cáncer.
Introducción
Aunque menos del 3% del genoma humano codifica proteínas, aproximadamente el 80% del genoma está transcrito 1,2. Entre las unidades transcripcionales no codificantes, decenas de miles se clasifican como ARN largos no codificantes (lncRNA) que superan los 200 nucleótidos, y se estima que el número total de lncRNAs humanos supera los 100.000 3,4. A diferencia de los genes codificantes, los lncRNAs están menos conservados en todas las especies. Dado que los humanos comparten el 99% de su genoma con primates como los chimpancés, se plantea la hipótesis de que los lncRNAs tienen una influencia mucho mayor en la evolución fenotípica 5,6. Estos hallazgos indican importantes funciones celulares de los lncRNAs. Aunque la regulación de los lncRNAs y sus interacciones con las proteínas de unión al ARN y otros ARN sigue sin comprenderse completamente, y muchos lncRNAs aún no se han anotado completamente, está claro que los lncRNAs exhiben patrones de expresión específicos de células y tejidos en la salud y la enfermedad, como el cáncer 7,8,9,10. Están implicados en diversas funciones, incluida la regulación de la transcripción génica, la participación en las interacciones de la cromatina11, el procesamiento del ARN12, la estabilización del ARN13 y la regulación de la traducción14.
En el cáncer, los lncRNAs específicos de un tipo de célula muy diversos influyen en el desarrollo tumoral y la metástasis mediante la regulación de la expresión génica, lo que pone de manifiesto su potencial como dianas terapéuticas valiosas15. Más allá de la detección de lncRNAs como biomarcadores en muestras tumorales16, dirigirse a lncRNAs específicos del tumor para interrumpir sus funciones posteriores tiene un potencial significativo tanto en aplicaciones clínicas como en investigación básica. Los enfoques basados en ARN para dilucidar las funciones de los lncRNAs incluyen oligonucleótidos antisentido (ASO), ARN de horquilla corta y ARN interferentes pequeños (siRNAs)17,18. Si bien el siRNA se utiliza comúnmente para las pantallas de silenciamiento de genes, la eliminación basada en siRNA se restringe al citoplasma19. Sin embargo, el lncRNA opera con frecuencia dentro del núcleo.
Alternativamente, la interferencia de repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPRi) se puede utilizar para inhibir los lncRNAs en cánceres humanos20. Además, las pantallas CRISPRi de todo el genoma pueden programarse fácilmente y dirigirse a una amplia gama de genes codificantes y no codificantes para examinar su impacto funcional21,22. En CRISPRi, un Cas9 catalítico deficiente (dCas9) se fusiona con un dominio represor transcripcional, como el dominio23 de la caja asociada a Krüppel (KRAB). La represión génica por dCas9-KRAB es guiada por un ARN guía (ARNg) a la región de interés. CRISPRi controla los genes a nivel del ADN, lo que conduce a una mayor eficiencia y a los fenotipos deseados de pérdida de función, en contraste con la interferencia del ARN, que está activa en el nivel postranscripcional24. En respuesta a la limitada eficacia de KRAB en el silenciamiento de dianas, se introdujo una fusión de KRAB y MeCP2 con dCas9 como una estrategia de represión más efectiva25.
Aunque el silenciamiento de un solo gen puede afectar la viabilidad del cáncer, las interacciones letales sintéticas, duales o múltiples, pueden rescatar a las células cancerosasde la muerte celular. La letalidad sintética involucra dos o más genes, cada uno de los cuales puede compensar la función del otro. Para superar los problemas con las pantallas de eliminación de un solo gen, las estrategias duales de CRISPR dirigidas a pares de genes letales que codifican proteínas ofrecen un enfoque prometedor27.
Aquí, presentamos un protocolo para el uso combinatorio de la focalización ortogonal basada en CRISPRi de lncRNA u otros pares de ARN no codificantes utilizando Staphylococcus aureus y Streptococcus pyogenes dCas9 fusionados con KRAB-MeCP2 en células de melanoma humano (ver Figura 1). El protocolo se puede utilizar para la eliminación combinada de CRISPR clásico o como detección basada en CRISPRi de pares letales sintéticos en el cáncer. El uso de dos especies diferentes de dCas9, SpCas9 y SaCas9, en el enfoque CRISPRi permite la orientación independiente de distintos loci genómicos con una reactividad cruzada mínima, lo que mejora la especificidad y la flexibilidad, al tiempo que garantiza una alta selectividad en el objetivo. Se utilizan diferentes motivos adyacentes protoespaciadores (PAMs): NGG para SpCas9 y NNGRRT para SaCas9. El sistema dual dCas9 aborda desafíos como la compatibilidad limitada de sgRNA al permitir una modulación precisa y simultánea y reducir la competencia compleja sgRNA-RNP cuando se usa un solo tipo de dCas9. Esta innovación mejora la robustez y versatilidad del cribado de bibliotecas duales de sgRNA. En conclusión, proporcionamos un sistema CRISPRi dual completamente funcional en células de melanoma como modelo de cáncer.
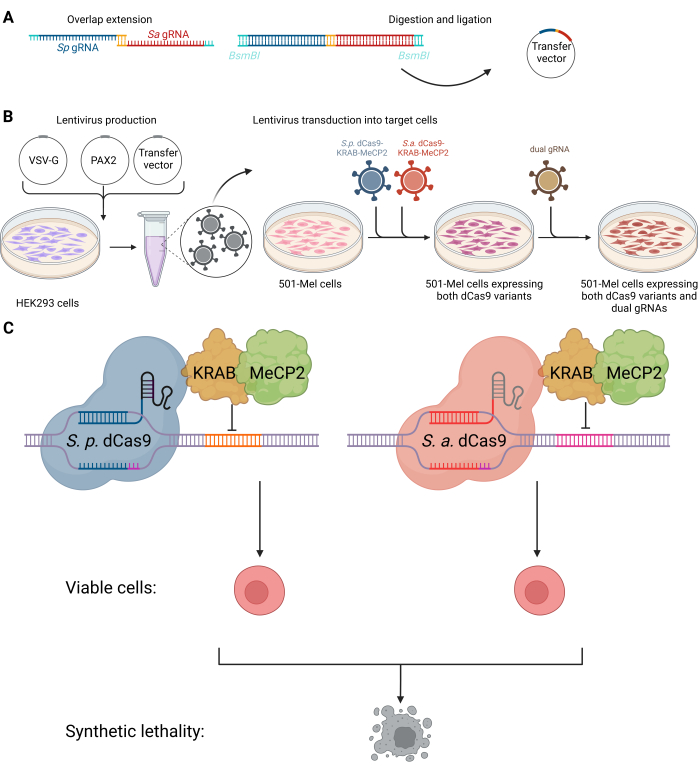
Figura 1: Esquema del sistema dual CRISPRi para dirigirse a las interacciones letales sintéticas de ARN no codificantes. (A) Procedimiento de clonación del vector dual de ARNg. (B) Las células HEK293 se transfectaron con el plásmido de envoltura VSV-G, el plásmido de empaquetamiento PAX2 y el vector de transferencia para producir lentivirus para su posterior transducción a células de 501 mel. Los lentivirus que contenían la información genética de SpdCas9-KRAB-MeCP2 (azul) y SadCas9-KRAB-MeCP2 (rojo) se integraron simultáneamente en células de 501 mel. Tras la selección del antibiótico, se realizó una segunda transducción lentiviral para integrar los gRNAs duales deseados (verde oliva). (C) Las células 501-mel que expresan las variantes dCas9 interactúan con sus ARNg correspondientes para silenciar la expresión génica diana, lo que resulta en la muerte de las células cancerosas. Creado con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
La línea celular de melanoma humano 501-mel (RRID: CVCL_4633) fue amablemente proporcionada por el Laboratorio Aifantis (Universidad de Nueva York). La línea celular Lenti-X 293T HEK fue comprada a Takara Bio. Estas líneas celulares se cultivaron en el medio de alta glucosa Eagle's Medium (DMEM) modificado de Dulbecco suplementado con un 10% de suero fetal bovino (FBS) a 37 °C en una atmósfera de 5% de CO2 en condiciones estériles. Las células fueron transitadas hasta que alcanzaron un 80%-90% de confluencia. Las líneas celulares se sometieron a pruebas periódicas para detectar la contaminación por micoplasma.
1. Diseño de ARNg utilizando una plataforma de diseño de ARNg en línea
- Diseñe un ARNg específico para SpCas9 y un ARNg específico para SaCas9 para cada lncRNA diana para evaluar qué variante produce resultados de eliminación superiores.
- Diríjase a los lncRNAs deseados, aquí RP11-120D5.1 (RP11) y XLOC_030781 (XLOC), utilizando el gRNA más eficiente para cada S. pyogenes (gRNA-sp) Cas9 y S. aureus (gRNA-sa) Cas9.
NOTA: Los ARNg de S. pyogenes pueden extraerse de Petroulia et al.28. - Siga las recomendaciones específicas del sitio para priorizar la actividad más alta en el objetivo, expresada por puntuaciones altas en el objetivo, mientras minimiza las coincidencias fuera del objetivo según lo sugerido por las herramientas de diseño de ARNg.
- Elija el genoma GRCh38 (hg, Homo sapiens). Seleccione el sitio PAM para los específicos SpCas9 (NGG) y SaCas9 (NNGRRT).
- Utilice el método de puntuación fuera del objetivo de Hsu et al.29. Coloque los ARNg dentro de una ventana de -150 pb a +50 pb alrededor del sitio de inicio transcripcional para un rendimiento óptimo.
- Elija los ARNg con la puntuación más alta en el objetivo y fuera del objetivo (consulte la Tabla complementaria 1).
- Combine un ARNg de control codificado o no dirigido, en este caso contra Rosa26, con cada ARNg objetivo para crear un vector de ARNg dual, dirigido a un ARNnc. La combinación de ARNg diana en un plásmido permite la modificación específica del sitio de ambos sitios diana.
NOTA: El vector de ARNg dual que alberga ARNg de control y ARNg diana permite la eliminación de un lncRNA objetivo, mientras que los vectores de ARNg duales que contienen ARNg dirigidos a sitios permiten la eliminación simultánea de lncRNA objetivo.
2. Clonación de ARNg
- Realice una PCR de extensión superpuesta utilizando dos fragmentos de ADN que contengan la secuencia de ARNg. Utilice el cebador directo U6 y el cebador inverso H1 (véase la Tabla complementaria 2) para la amplificación como se describe en Najm et al.27.
- Configure una reacción de PCR de 50 μL con 25 μL de 2x Mezcla maestra de PCR de alta fidelidad con tampón HF y 1 μL de cada fragmento de ADN que contenga el ARNg de S. pyogenes o S. aureus . Ejecute la reacción en las siguientes condiciones de ciclo: desnaturalización inicial a 98 °C durante 30 s, seguida de 15 ciclos de desnaturalización a 98 °C durante 5 s, recocido a 55 °C durante 10 s y elongación a 72 °C durante 15 s.
- Añadir 2,5 μL de cebador directo U6 y 2,5 μL de cebador inverso H1 después de completar los 15 ciclos iniciales, y someter la reacción a 20 ciclos adicionales en las mismas condiciones, con una extensión final de 5 min a 72 °C.
- Purifique los productos de PCR utilizando un kit de purificación de PCR de acuerdo con las instrucciones del fabricante.
- Digiera 1 μg del plásmido modificado utilizando 1 μL de enzima de restricción BsmB1 y 1 μL de tampón 10x durante 30 min a 37 °C en una reacción de 10 μL.
- Clone los productos de PCR en el plásmido dual-gRNA-Zeo-GFP digerido (ver Figura Suplementaria 1 y Archivo Suplementario 1) utilizando un protocolo de reacción de ensamblaje de ADN de alta fidelidad como se describe en otros30.
- Transforme las células de E. coli químicamente competentes mezclando 50 μL de cultivo de bacterias con 4 μL de producto de montaje de acuerdo con las instrucciones del fabricante.
3. Producción de lentivirus
PRECAUCIÓN: Manipule los lentivirus activos en todos los pasos mientras sigue las pautas de seguridad adecuadas. Siempre realice el trabajo con virus en un gabinete de seguridad de Clase 2 para evitar peligros de formación inevitable de aerosoles. Realice todos los trabajos fuera del gabinete de seguridad, como la centrifugación, en recipientes herméticos a los aerosoles y rotores aprobados para su uso con organismos del Grupo de Riesgo 2. Use ropa protectora adecuada, incluida una bata de laboratorio, guantes desechables y gafas de seguridad, en todo momento en el área de trabajo. Las superficies de trabajo deben tratarse con una solución desinfectante, y otros residuos también deben tratarse con una solución desinfectante, lo que les permite permanecer en el gabinete sellado bajo luz ultravioleta durante al menos 240 minutos. Las pipetas serológicas y las puntas de pipeta utilizadas deben enjuagarse con una solución desinfectante antes de desecharlas en frascos esterilizables en autoclave. Posteriormente, los residuos deben eliminarse en el contenedor de residuos S2. Los residuos S2 se inactivarán mediante autoclave. Las líneas celulares transducidas pueden manipularse en condiciones S1 no antes de 2 días después de la transducción y solo después de al menos dos cambios completos del medio de cultivo con un medio libre de virus.
- Cuente las células Lenti-X 293T HEK mezclando 10 μL de células con 10 μL de solución de azul de tripán en un hemocitómetro. Placa 4 x 106 celdas Lenti-X 293T HEK por placa de 10 cm para lograr ~80% de confluencia al día siguiente.
- Trabaje en un laboratorio de Clase 2 a partir de este paso. Mezcle 500 μL de un medio de transfección adecuado con el vector de transferencia SpdCas9-KRAB-MeCP2 de 11,25 μg (véase el archivo complementario 2), el plásmido de envoltura VSV-G de 5,5 μg y el vector pPAX2 de 16,5 μg. Incubar durante 5 min a temperatura ambiente. Para la segunda variante de dCas9, utilice el vector de transferencia SadCas9-KRAB-MeCP2 (véase el archivo complementario 3).
- En un tubo aparte, mezcle 500 μL de medio sérico reducido con 36 μL de reactivo de PEI (stock: 1 mg/mL). Incubar durante 5 min a temperatura ambiente.
- Combine las mezclas e incube durante 15 minutos a temperatura ambiente. Añadir la mezcla con cuidado gota a gota al medio celular (6 mL) e incubar durante la noche a 37 °C y 5% de CO2. Asegúrese de que las células permanezcan unidas para evitar la reducción del rendimiento lentiviral.
- Deseche el medio en un frasco de residuos esterilizable en autoclave con una pipeta serológica después de 12-15 h. A partir de ahora, enjuague los consumibles, incluidas las pipetas serológicas y las puntas de pipeta, con la solución desinfectante antes de desecharlos en la basura del autoclave. Añada 5 mL de medio DMEM fresco a las células transfectadas con una pipeta nueva.
- Recolectar lentivirus transfiriendo el sobrenadante mediante una pipeta serológica a un tubo canónico de 50 mL 48 h después de la transfección. Guarde el tubo canónico en la nevera. Añadir 5 mL de medio fresco a las células e incubar las células durante 24 h más.
- Repita la recolección de lentivirus transfiriendo el sobrenadante al mismo tubo canónico de 50 mL que contiene la primera cosecha. De nuevo, añadir 5 mL de medio fresco, incubar durante 24 h y transferir el sobrenadante después al tubo canónico de 50 mL. Almacene los sobrenadantes combinados de la primera, segunda y tercera cosecha en el tubo canónico de 50 mL a 4 °C hasta la filtración y centrifugación.
- Centrifugar el sobrenadante combinado durante 5 min a 500 x g y 4 °C en recipientes herméticos a los aerosoles y rotores aprobados para su uso con organismos del Grupo de Riesgo 2 para granular células desprendidas y residuos. Filtre el sobrenadante a través de filtros de jeringa de grado de cultivo de células estériles de 0,2 μm.
- Concentre el sobrenadante filtrado a 1.000 x g y 4 °C utilizando una unidad de filtro centrífuga con un límite de peso molecular adecuado de 100 kDa a aproximadamente 500 μL. Almacene las alícuotas de virus de 50-100 μL a -80 °C hasta su uso posterior.
4. Transducción de lentivirus y selección de células transfectadas estables
PRECAUCIÓN: Siempre realice trabajos con virus en un gabinete de seguridad de Clase 2.
- Placa de 501 mel celdas en una placa de 6 pocillos a 2 x 105 celdas/pocillo con un volumen de cultivo final de 2 mL, 1 día antes de la transducción.
- Sustituya el medio de cultivo por 2 mL de medio DMEM precalentado suplementado con polibreno a una concentración final de 6 μg/mL.
- A partir de ahora, se requiere trabajar en un laboratorio de Clase 2. Añadir 50 μL de lentivirus que contengan SpdCas9-KRAB-MeCP2 y SadCas9-KRAB-MeCP2 simultáneamente a las células. Enjuague los consumibles, incluidas las pipetas serológicas y las puntas de pipeta, con la solución desinfectante antes de desecharlos en la basura del autoclave. Agite la placa suavemente e incube a 37 °C en una atmósfera con 5% deCO2 .
- Deseche el medio en un frasco esterilizable en autoclave con una pipeta serológica y agregue 2 mL de medio fresco que contenga blasticidina (10 μg/mL) y puromicina (2 μg/mL) 16 h después de la transducción.
- Continúe la selección reemplazando el medio con medio fresco de 2 mL que contenga blasticidina (10 μg/mL) y puromicina (2 μg/mL) después de 2 días. Manipule las células transducidas en condiciones S1 después de 7 días después de la transducción.
NOTA: La selección de antibióticos debe realizarse hasta que todas las células de control no transducidas hayan muerto. Se pueden utilizar estrategias de selección alternativas en función del casete de resistencia en el vector de transferencia. Si es necesario, optimice las concentraciones de antibióticos utilizando una curva de eliminación. Es posible que sea necesaria una selección elaborada de clones unicelulares mediante citometría de flujo o dilución y siembra en serie, seguida de cuantificación de proteínas, para obtener poblaciones celulares que expresen fusiones de dCas9 a niveles suficientes.
5. Cuantificación de proteínas y análisis de Western blot
- Cuente las células transfectadas estables de 501 mel mezclando 10 μL de células con 10 μL de solución de azul de tripán en un hemocitómetro. Centrifugar 1 x 106 de las celdas durante 5 min a 500 x g y 4 °C. Deseche el sobrenadante y vuelva a suspender el pellet celular en 1 mL de PBS helado.
- Repita la centrifugación y vuelva a suspender el pellet celular en 50 μL de tampón RIPA que contiene inhibidores de la proteasa. Incubar en hielo durante 15 min y eliminar los restos celulares mediante centrifugación a 13.000 x g durante 10 min a 4 °C. Almacene el sobrenadante a -20 °C o utilícelo inmediatamente para la cuantificación de proteínas como se describe en otros31.
- Mezcle 10 μg de proteína con 4 x tampón LDS, 50 mM de DTT y llene hasta 20 μL con agua destilada. Separe las proteínas en un gel de separación al 10% mediante el uso de SDS-PAGE a base de Tris/glicina a 135 V durante 1 h como se describió anteriormente32. Transfiera las proteínas a una membrana de PVDF a 90 V durante 90 min con una transferencia en tanque húmedo.
- Incubar la membrana en tampón de bloqueo (5% de leche descremada en TBS-T) durante 1 h a 180 rpm a temperatura ambiente.
- Agregue el anticuerpo monoclonal de ratón anti-GAPDH 1:10.000 en el tampón de bloqueo como control interno. Añadir el anticuerpo primario anti-Cas9 (S. pyogenes) o anti-Cas9 (S. aureus) en forma de dilución 1:1.000 en el tampón de bloqueo e incubar durante la noche a 4 °C agitando suavemente.
- Lave la membrana 3 veces con tampón TBST durante 10 minutos cada una. Incubar con anticuerpo secundario anti-ratón HRP (1:10.000) durante 1 h a temperatura ambiente.
- Lavar 3 veces con tampón TBST durante 10 minutos cada una. Detecte señales utilizando ECL Substrate y un sistema de imágenes.
6. Ensayo de viabilidad de represión de LncRNA dual
PRECAUCIÓN: Siempre realice trabajos con virus en un gabinete de seguridad de Clase 2.
- Siembra 0,1 x 105 células que contienen SadCas9-KRAB y SpdCas9-KRAB transfectados estables 7 días después de la transducción en una placa de 96 pocillos en 100 μL de medio DMEM precalentado 1 día antes de la transducción.
- El día de la transducción, sustituya el medio de cultivo por 100 μL de medio DMEM precalentado suplementado con polibreno a una concentración final de 6 μg/mL. A partir de ahora, se requiere trabajar en un laboratorio de Clase 2. Agregue el volumen apropiado de lentivirus a las células.
NOTA: El volumen de lentivirus que contiene el vector de ARNg dual debe examinarse utilizando GFP para generar una curva lineal mediante la valoración de los lentivirus. - Reemplace el medio con medio fresco que contenga 500 μg/mL de zeocina, 5 μg/mL de blasticidina y 1 μg/mL de puromicina 16 h después de la transducción.
- Realice la detección de luminiscencia 5 días después de la transducción utilizando un ensayo de viabilidad celular de acuerdo con las instrucciones del fabricante.
Resultados
Los casetes de expresión de SadCas9-KRAB-MeCP2 y SpdCas9-KRAB-MeCP2 se integraron simultáneamente en células de 501 mel utilizando transducción con cantidades iguales de lentivirus. Dado que este protocolo CRISPRi se basa en la expresión de variantes de dCas9-KRAB-MeCP2, el análisis de Western blot es ideal para confirmar su presencia a nivel de proteína. Tras el enriquecimiento de las células transfectadas positivamente, se recogieron muestras para el análisis de Western blot con el fin de confirmar la presencia de los ortólogos dCas9-KRAB-MeCP2. Tras una transducción exitosa, se espera una sola banda correspondiente a la proteína de fusión SpdCas9-KRAB-MeCP2 (202 kDa) y la proteína de fusión SadCas9-KRAB-MeCP2 (170 kDa) y se confirmó, como se muestra en la Figura 2A, utilizando anticuerpos específicos para los ortólogos Cas9 respectivos. Sin embargo, si la transferencia de Western no es factible, se puede utilizar la qPCR para verificar las proteínas de fusión a nivel de ARNm.
Una vez que se generaron células de 501 mel que expresaban de manera estable los represores funcionales, se realizó una segunda transducción lentiviral utilizando un vector de ARNg dual. Previamente se demostró que RP11 y XLOC estaban regulados al alza en cultivos a corto plazo de melanoma metastásico, y la focalización individual de CRISPRi demostró su impacto en la proliferación y la supervivencia celular28. Por lo tanto, seleccionamos este par de lncRNA para su evaluación como una combinación de ARN no codificante letal sintético de prueba de concepto, anticipando un fenotipo de crecimiento acumulativo y fácilmente observable en las células modificadas.
Para investigar los efectos de CRISPRi dual en células cancerosas mediante ensayos de viabilidad, ambos lncRNAs dirigidos deben ser reprimidos de manera efectiva. La qPCR es el método más adecuado para confirmar esta represión mediante la medición de sus niveles de transcripción. Evaluamos la funcionalidad del protocolo dual CRISPRi desarrollado mediante la determinación de los niveles de ARN y el examen de la viabilidad celular cuando se dirige al par RP11 y XLOC potencialmente letal. Por lo tanto, se introdujeron diferentes ARNg dirigidos a RP11 y XLOC para reducir los niveles de ARN diana. La represión del ARN se investigó utilizando ARNg específicos de la diana para SadCas9-KRAB-MeCP2 o SpdCas9-KRAB-MeCP2, probados individualmente y en combinación junto con un ARNg de control dirigido a Rosa26 (ver Figura 2B). Aunque los ARNg dirigidos a XLOC o RP11 individualmente suprimieron eficazmente sus respectivos objetivos, la eliminación simultánea de XLOC y RP11 solo se logró con el par XLOC-sa/RP11-sp.
Dado que los niveles de ARN XLOC y RP11 se redujeron con éxito en la línea celular de 501 mel con el par de ARNg XLOC-sa/RP11-sp seleccionado, se realizó un ensayo de viabilidad de represión de ARNl dual (ver Figura 2C). A los 5 días, la viabilidad celular fue del 81% y del 89% del control Rosa26 cuando se utilizaron los ARNg diana individualmente. La combinación de RP11-sp y XLOC-sa dio lugar a una reducción significativa adicional en comparación con RP11-sp solo, con una viabilidad celular del 59% del control Rosa26.
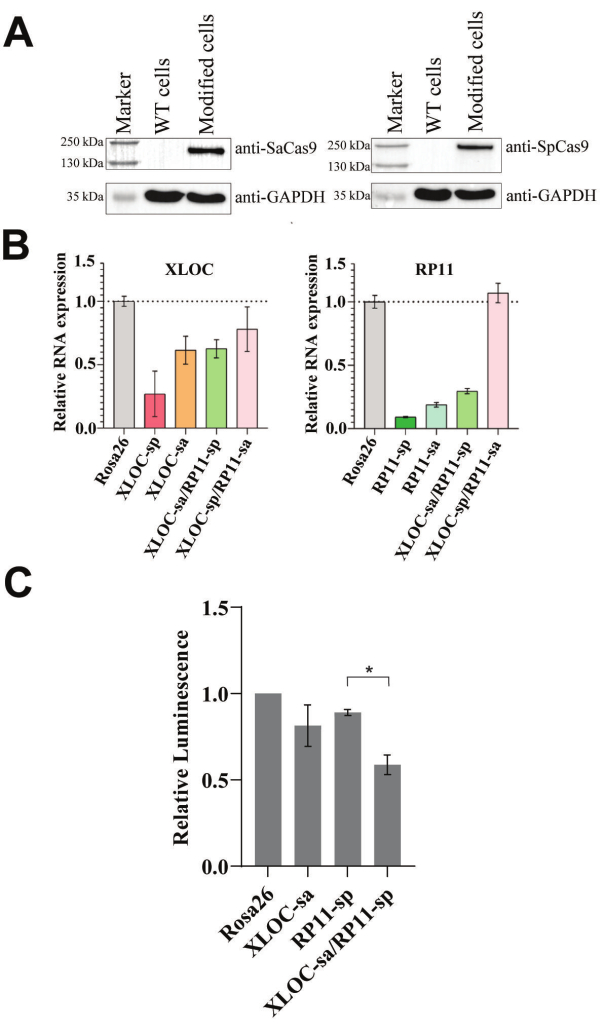
Figura 2: Funcionalidad del sistema dual de represión de lncRNA. (A) Se realizó un análisis de Western blot para detectar las variantes de dCas9-KRAB-MeCP2 en células 501-mel modificadas utilizando anticuerpos anti-Cas9. Se utilizaron células de tipo salvaje (WT) de 501 mel sin transducción como control (véase la Figura suplementaria 3, la Figura suplementaria 4, la Figura suplementaria 5, la Figura suplementaria 6 para el blot sin procesar). (B) análisis basado en qPCR de los niveles de ARN RP11 y XLOC en relación con las células de control Rosa26 después de la transducción de ARNg duales en células 501-mel modificadas. Se utilizó GAPDH como gen de limpieza. Los datos se expresan como media ± DE. (C) El ensayo de viabilidad de represión de ARNc dual se realizó después de integrar ARNg duales en células 501 mel modificadas en tres transducciones independientes. Los valores de luminiscencia se normalizaron al control Rosa26. Los resultados de viabilidad se expresan como la media (n = 3) ± SEM. Se realizó una prueba t no apareada. El asterisco (*) indica una diferencia significativa a p < 0,05. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1: Mapa de plásmidos. El mapa fue creado con Benchling (https://benchling.com/). Los plásmidos utilizados en el protocolo fueron modificados a partir de Addgene #96921 y #110824. Haga clic aquí para descargar este archivo.
Figura suplementaria 2: Figura suplementaria 2: Análisis de Western blot de
Expresión dCas9-KRAB. (A) Expresión de SadCas9-KRAB en células de 501 mel 6 días después de la transfección. (B) La transducción secuencial de SadCas9-KRAB y SpdCas9-KRAB en células de 501 mel condujo a una reducción de la expresión de SadCas9-KRAB 3 semanas después de la transfección. (C) La transducción simultánea de SadCas9-KRAB y SpdCas9-KRAB en células de 501 mel resultó en una reducción de la expresión de SadCas9-KRAB 3 semanas después de la transfección. (D) La expresión de SpdCas9-KRAB pudo observarse después de la transducción simultánea de SadCas9-KRAB y SpdCas9-KRAB. LV. El volumen de lentivirus utilizados en μL. Haga clic aquí para descargar este archivo.
Figura suplementaria 3: Datos brutos de Western blot SaCas9-KRAB. Haga clic aquí para descargar este archivo.
Figura complementaria 4: Datos brutos de Western blot SpCas9-KRAB. Haga clic aquí para descargar este archivo.
Figura complementaria 5: Datos brutos de GAPDH_SpCas9-KRAB. Haga clic aquí para descargar este archivo.
Figura complementaria 6: Datos brutos de GAPDH_SaCas9-KRAB. Haga clic aquí para descargar este archivo.
Tabla complementaria 1: Secuencias de ARN guía utilizadas. Haga clic aquí para descargar este archivo.
Tabla complementaria 2: Secuencias de ADN utilizadas. Haga clic aquí para descargar este archivo.
Fichero complementario 1: Fichero FASTA de dual-grna-zeo-gfp. Haga clic aquí para descargar este archivo.
Fichero complementario 2: Fichero FASTA de dSpCas9-krab-mecp2. Haga clic aquí para descargar este archivo.
Fichero complementario 3: Fichero FASTA de dSaCas9-krab-mecp2. Haga clic aquí para descargar este archivo.
Discusión
En este estudio, implementamos una estrategia de diana dual de lncRNA en células de melanoma utilizando CRISPRi basada en dCas9-KRAB-MeCP2 de dos especies ortogonales. Utilizando este sistema, reprimimos con éxito un par letal sintético de lncRNAs, RP11 y XLOC, lo que llevó a un aumento de la muerte celular, a diferencia de cuando se reprimieron individualmente. Sin embargo, los sistemas CRISPRi conllevan un riesgo de represión génica fuera del objetivo, lo que podría afectar a la interpretación de los resultados. Aunque no se llevó a cabo la validación experimental, minimizamos los posibles efectos fuera del objetivo durante el diseño de ARNg in silico.
La variabilidad en la eficiencia de la transducción puede afectar la reproducibilidad. Por lo tanto, optimizamos la transducción mediante la aplicación de cantidades iguales de lentivirus, determinadas a través de una calibración de curva estándar utilizando concentraciones variables de lentivirus. Sin embargo, el aumento del tamaño de la carga del vector de nuevo diseño puede imponer limitaciones a la eficiencia del empaquetamiento lentiviral, lo que podría reducir la eficiencia de la transducción, particularmente en líneas celulares difíciles de transducir. Como resultado, lograr una transducción suficiente y una expresión robusta de dCas9 puede requerir títulos virales altos en ciertos casos. Además, se puede evitar la eliminación insuficiente mediante la monitorización frecuente de los niveles de proteína dCas9-KRAB-MeCP2 y la expresión de ARN de lncRNAs específicos. Alternativamente, hay versiones de dCas9 disponibles con un solo sistema de represión para superar esta limitación, sin embargo, con el costo de una disminución de la eficiencia25.
Solo la combinación XLOC-sa-RP11-sp demostró una actividad exitosa, mientras que la configuración inversa XLOC-sp-RP11-sa fue ineficaz. Una explicación plausible para esta diferencia en la eficacia de la reducción es la interacción entre la especificidad de PAM y la accesibilidad a la cromatina33. Dado que SaCas9 y SpCas9 reconocen secuencias PAM distintas, el sitio objetivo para SpCas9 en la orientación XLOC-sp-RP11-sa puede carecer de un PAM óptimo, lo que reduce su afinidad de unión y su actividad posterior. Además, la disposición espacial de las dos variantes de la proteína de fusión dCas9 podría influir en la arquitectura y la accesibilidad de la cromatina34,35, lo que podría restringir el reclutamiento de la maquinaria transcripcional o los factores reguladores asociados en la configuración XLOC-sp-RP11-sa. Estos hallazgos sugieren que el posicionamiento relativo de SaCas9 y SpCas9 dentro de un contexto genómico determinado puede tener un impacto crítico en su eficacia funcional, un aspecto que justifica una mayor investigación para estrategias de modulación optimizadas basadas en CRISPR.
El protocolo desarrollado es adaptable para dirigirse o cribar otros pares de ARN no codificante letales sintéticos mediante la modificación del vector de ARNg dual. Una vez que una línea celular de interés expresa de manera estable ambas variantes de dCas9, se vuelve factible una variedad de enfoques. Si bien el ARNi, los ASO y los shRNA pueden emplearse para el silenciamiento génico, CRISPRi ofrece una metodología de cribado más sencilla, y también es concebible su combinación con pequeños compuestos químicos o inhibidores. Al sustituir el vector de ARNg dual por una biblioteca de ARNg dual, este protocolo permite el cribado sistemático de pares letales sintéticos u otras dependencias genéticas en las células cancerosas, mejorando así la precisión de la selección de células cancerosas.
Aunque logramos la integración de las variantes de dCas9-KRAB-MeCP2 a través de la transducción lentiviral, también se pueden usar métodos alternativos de transfección estable para incorporar las enzimas deseadas en células 501-mel. Esto es particularmente relevante, ya que, en nuestra experiencia, el silenciamiento de grandes constructos como dCas9-KRAB-MeCP2 puede ocurrir con el tiempo36,37. Alternativamente, se demostró que dCas9-KRAB era una alternativa más pequeña23. En nuestra experiencia, la transducción con las proteínas de fusión dCas9-KRAB más pequeñas derivadas de los ortólogos Cas9 condujo a una reducción de la expresión de proteínas durante el cultivo prolongado (ver Figura complementaria 2). Por lo tanto, la expresión constante de proteínas debe controlarse regularmente en puntos temporales recurrentes. Además, la transducción de dos grandes proteínas de fusión junto con marcadores de selección y casetes de expresión de ARNg dual puede inducir estrés celular o puede alcanzar capacidades de empaquetamiento lentiviral.
Lograr una eficiencia de knockdown del 100% en CRISPRi dual puede ser un desafío debido a un diseño de ARNg subóptimo o a una expresión insuficiente de dCas9. Para abordar esto, se deben probar múltiples ARNg por objetivo utilizando herramientas de diseño avanzadas para identificar las secuencias más efectivas. Además, la optimización de la expresión de dCas9-KRAB-MeCP2 a través de un mejor diseño vectorial y la caracterización de líneas celulares clonales puede mejorar la eficiencia de la eliminación.
Las estrategias alternativas para integrar el sistema CRISPRi incluyen el uso de vectores de transposasa o la integración dirigida por CRISPR para lograr una inserción estable en sitios genómicos de puerto seguro, que son conocidos por mantener una expresión estable a lo largo del tiempo38. Sin embargo, muchos tipos de células, incluidas las células de melanoma, son difíciles de transfectar39, lo que hace que la transducción lentiviral sea el método preferido. La electroporación de enzimas basadas en transposasa o Cas puede ofrecer un medio alternativo para integrar las construcciones dCas9-KRAB-MeCP2 en células de melanoma40. La implementación de un sistema de knockout inducible sería ventajosa para activar la expresión de dCas9-KRAB-MeCP2 según sea necesario en lugar de la expresión constitutiva para minimizar el estrés celular.
Por último, el enfoque desarrollado no se limita a las células de melanoma, sino que puede extenderse a cualquier tipo de célula en la que se esté investigando la letalidad sintética. Este enfoque está explícitamente destinado a ampliarse hacia la modalidad de cribado de bibliotecas de ARNg. Sin embargo, debido a la mayor complejidad combinatoria, por ejemplo, con más de tres ARNg por par de genes, incluidos los controles y una cobertura de al menos 300 veces, solo se puede apuntar en la práctica a una fracción subtranscriptómica de los ARNc expresados. Por lo tanto, los usuarios deben preseleccionar cuidadosamente a los candidatos teniendo en cuenta factores como los niveles de expresión de lncRNA y su relevancia conocida en contextos celulares específicos antes de proceder con el cribado de la biblioteca. Sin embargo, la versatilidad combinatoria de este enfoque lo convierte en una herramienta valiosa para investigar las interacciones letales sintéticas y otras dependencias genéticas de los lncRNAs en varios tipos de cáncer y potencialmente otras enfermedades. Esto amplía el conjunto de herramientas actualmente disponibles para estudiar estas interacciones de red en las células.
Divulgaciones
Jochen Imig es actualmente CGCIII financiada por Pfizer Inc. CGCIII está patrocinada por Pfizer Inc., Merck KGaA y AstraZeneca PLC. Los patrocinadores no tuvieron ningún papel en el diseño, ejecución, interpretación o redacción del estudio. Todos los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos a Stefan Raunser (Instituto Max Planck de Fisiología Molecular, Dortmund) por su acceso al espacio de laboratorio de Bioseguridad Nivel 2. Un agradecimiento especial a Eric Wang por sus contribuciones intelectuales y a todos los miembros pasados y presentes del laboratorio Imig por sus valiosas discusiones. Jochen Imig es actualmente CGCIII financiado por Pfizer Inc. en CGC III.
Materiales
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
Referencias
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados